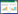文章信息
- 党柯柯, 侯金凤, 刘洪, 董元华, 李建刚
- DANG Keke, HOU Jinfeng, LIU Hong, DONG Yuanhua, LI Jiangang
- 生姜根系不同生态位细菌群落多样性特征、组成及结构差异
- Characteristics of bacterial community diversity, composition and structure in different niches of ginger root
- 生态学报. 2023, 43(4): 1691-1702
- Acta Ecologica Sinica. 2023, 43(4): 1691-1702
- http://dx.doi.org/10.5846/stxb202203150632
-
文章历史
- 收稿日期: 2022-03-15
- 网络出版日期: 2022-10-13
2. 中国科学院大学, 北京 100000
2. University of Chinese Academy of Sciences, Beijing 100000, China
植物不同生态位栖居着大量高度多样化的微生物, 在长期的协同进化过程中, 这些微生物与特定植物形成稳定特异的互利共生关系[1—3], 被称为植物的第二基因组。植物根系作为物质交换和水分运输的关键通道, 对作物的健康生长尤为重要。因此与植物根系相关的微生物组也被认为是影响作物健康、产量和品质的关键因素[4]。根系相关微生物组是指在根系不同生态位, 包括非根际、根际及根内的一类与植物根系密切相关且稳定存在的微生物组[5]。它们的主要作用是参与植物的营养吸收、物质交换、胁迫耐受及病原菌抵御等, 是植物微生态系统的重要组成部分[4]。而作为回报, 寄主植物也为微生物提供了生存空间及源源不断的能量和丰富的碳源[6—7]。根系不同生态位微环境类似于不同的“战场”, 复杂的微生物依靠对自己有利的环境特点取得优势地位, 最终形成相对稳定的微环境, 它们为维持植物健康、帮助植物适应不断变化的环境发挥着不容忽视的作用。因此, 了解根系不同生态位微生物群落的特点对植物的生长调控及病原菌防御至关重要。
根系不同生态位所分割的特定微环境会导致细菌群落的显著分化。例如, Beckers等[8]发现杨树根际细菌群落的形成似乎是一个更稳定和更受控制的过程, 而内生菌具有更强的变异性。植物根内生菌与根际环境密切相关, 研究发现[9], 将拟南芥种植在添加纤维素基质的土壤中, 发现拟南芥根内生菌40%的分类操作单元(OTUs)源于该基质细菌群落, 这可能作为植物根内选择性定殖环境微生物的重要线索。此外, 拟南芥根际细菌群落也被认为是非根际土壤细菌群落的一个子集, 随后通过宿主植物生态位分化进行过滤, 栖息在植物内部[10]。这是继植物内生菌存在源自种子和花粉的垂直传播外[11], 也存在不同生态位的水平传播的证据。栖息在宿主植物不同生态位的细菌群落存在着某种特定的平衡, 这种平衡利于宿主植物的健康生长。因此, 明确健康宿主植物特定生态位的细菌群落特点对进一步利用有益微生物保护植物健康生长有重要意义。
生姜(Zingiber officinale Roscoe)是一种比较特殊的草本植物, 其具有刺激性气味的块茎位于地下, 与根系组织相连, 块茎的产量和品质与根系微生态密切相关。与其他作物根系微生物组相比, 块茎类作物根系微生物组展现出更加丰富的种群多样化。有针对多种块茎类作物如萝卜、马铃薯、山药、红薯等的研究发现, 其内生固氮菌种类多样, 且部分具有促生和抗菌活性, 展示了块茎类作物内生菌的潜在应用价值[12]。另外, 生姜的特殊辛辣气味, 源于其自身合成的姜辣素成分, 姜辣素被证实是生姜中的一类特殊的抗菌活性成分, 对金黄色葡萄球菌、枯草芽孢杆菌、肺炎链球菌等细菌具有显著的抑制效果[13]。此外, 目前尚未发现生姜根不同生态位细菌群落特点的相关报道。因此, 本研究假设由于不同的微环境过滤作用, 非根际土壤、根际土壤及根内环境之间的细菌群落组成和群落结构存在显著差异, 另外本研究认为生姜独特的气味可能衍生出宿主特异性微生物群落。所以本文以生姜根不同生态位细菌群落为研究对象, 通过高通量测序技术, 分析其细菌多样性和组成的变化并鉴定其核心细菌, 对不同生态位细菌群落进行溯源分析, 为明确生姜的宿主-微生物相互作用提供研究基础。
1 材料与方法 1.1 供试土壤及样品采集供试土壤于2020年8月在中国科学院鹰潭红壤生态试验站采集, 该试验站位于江西省鹰潭市余江区刘家站(东经116°55′30″, 北纬28°15′20″), 为第四纪红色黏土母质发育的红壤[14]。采样田块在2018年之前处于撂荒状态, 自2018年起种植生姜。采样土壤基本理化为pH5.8、全氮(TN)0.04%、全磷(TP)0.03%、全钾(TK)1.08%、硝态氮(NN)11.27 mg/kg、速效磷(AP)2.87 mg/kg、速效钾(AK)156.45 mg/kg、有机质(OM)0.68%。
采样田块的面积为1120 m2, 为生姜重茬地块。选择蛇形采样法进行样点布置以避免土壤异质性带来的干扰。首先根据蛇形布局确定10个1 m×1 m的采样样方, 每个样方采集三个样点, 均匀混合后作为一个样品, 每个生态位得到10个样品。取样时间位于生姜块茎旺盛生长期, 采样方法参照的描述并按照生姜生长特点进行一定的改进[15]。首先选择两株生姜之间(未接触生姜根系)的垄上作为非根际土壤采样点, 小心移除表面的植物碎屑和岩石块, 收集0—20 cm的表层土壤, 置于无菌袋中并在-20℃冰箱储存。之后, 完整取出生姜植株, 轻轻抖动根系直至基本没有土壤颗粒落下, 用无菌毛刷轻轻刷下粘附在根系上的土壤并收集入无菌袋中, 即为根际土壤。根内生菌的收集经过以下步骤:根表消毒(75%酒精30 s、0.03%次氯酸钠溶液1 min、75%酒精30 s及无菌水润洗3次)、内生菌的释放(剪碎的根系:无菌磷酸缓冲液以1∶9(m/v)混合, 170 r/min震荡30 min)、无菌滤纸过滤(过滤后弃根系残留物)及离心(10000 r/min, 5 min)、收集菌体。待测样品均置于-20℃冰箱备用。用于高通量测序样品的共计30个(生态位3×样品重复10), 用于土壤理化性质的样品共计10个(非根际:样品重复10)。
1.2 样品的处理及分析样品DNA的提取选用细菌基因组DNA提取试剂盒(Sigma-Aldich, USA), 并严格按照试剂盒附带的操作步骤提取细菌基因组DNA。提取的DNA样品通过分光光度法进行定量(Nanodrop ND-2000, USA)。非根际和根际土壤样品16S rRNA基因的扩增利用通用引物515F(5′-GTGCGCCGGTAA-3′)和907R(5′-CCGTCAATTCMTTRAGTT-3′)(V4+V5区)[16]进行, 生姜根系内生菌16S rRNA基因的扩增利用通用引物799F(5′-AACMGGATTACCCKG-3′)和1193R(5′-ACGTCATCCATCCCC-3′)(V5+V7区)[17]实现。根据Illumina测序的要求, 将Illumina A接入引物5′端, 将Illumina B和标签(barcode)接入引物3′端。原始测序数据用QIIME软件进一步处理[18]。PCR反应条件为:95 ℃ 3 min;95 ℃ 1 min, 50 ℃ 1 min, 72 ℃ 1 min, 35个循环;72 ℃ 8 min。反应产物用1%琼脂糖凝胶电泳检测。PCR扩增及测序工作由上海凌恩生物科技有限公司完成。
土壤理化性质的分析参照鲁如坤[19]的《土壤农业化学分析方法》进行。其中土壤pH采用电位法(土水比1∶2.5, m/v);全氮采用凯氏定氮法(KDN-1, 雷磁, 上海)测定;全磷采用氢氧化钠熔融-钼锑抗比色法测定;全钾采用氢氧化钠熔融-火焰光度法(FP6450, 上海)测定;硝态氮采用紫外分光光度法测定[20];速效磷采用碳酸氢钠浸提-钼锑抗比色法测定;速效钾采用乙酸铵浸提-火焰光度法测定;有机质采用重铬酸钾容量法测定。
1.3 数据分析采用QIIME2(2019.4)软件对下机序列进行处理[21]。首先调用qiime cutadapt trim-paired切除序列的引物片段, 弃去未匹配引物的序列;然后通过qiime dada2 denoise-paired调用DADA2进行质控、去噪、拼接、去嵌合体。以上步骤针对每个文库分别进行分析。完成对所有文库的去噪后, 合并ASVs特征序列和ASV表格, 并去除singletons ASVs (即在全体样本中, 序列总数仅为1的ASV)。
采用R语言(R4.3)进行差异显著性分析(ANOVA, LSD)、置换多元方差分析(MRPP, P<0.05)及Mantel test检验(https://www.R-project.org/)。用Past软件进行主坐标分析(PCoA)并绘图(http://folk.uio.no/ohammer/past/)[22]。采用Cytoscape软件进行共现网络参数的构建(Spearman r>0.6, P<0.05)[23], 并用Gephi软件进行共现网络图的可视化。基于贝叶斯理论的SourceTracker分析工具对不同生态位细菌群落进行溯源分析(QIIME2)[21]。
2 结果与分析 2.1 生姜根不同生态位细菌群落测序结果及多样性分析通过序列质控、去噪、拼接、去嵌合体等处理, 非根际样品平均获得62118条有效序列, 样本ASV数为1917个;根际样品平均获得61388条有效序列, 样本ASV数为1782个;内生菌样品平均获得59692条有效序列, 样本ASV数为415个。
本文分析了细菌群落的均一性指数(Shannon index)、丰富度指数(Observed species)及系统发育多样性指数(Faith′s PD)。结果表明:生姜非根际、根际及内生菌的alpha多样性指数存在显著差异(图 1), 非根际与根际土壤细菌群落的alpha多样性指数均显著高于内生菌群落, 表明非根际及根际样品的均一性水平、物种丰富度及系统发育多样性均优于内生菌群落。
|
| 图 1 生姜非根际、根际及根内生菌alpha多样性指数 Fig. 1 Alpha diversity index of bacteria in bulk soil, rhizosphere soil, and endophyte of ginger in red soil BS, 非根际Bulk soil;RS, 根际Rhizosphere soil;EP, 根内Endosphytes;*P<0.05 (ANOVA, LSD) |
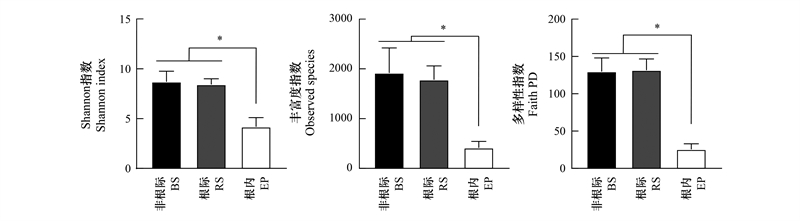
为明确根系不同生态位细菌群落在组成上的差异, 本文在ASV水平对各细菌群落的种类和丰度进行降维分析。结果表明, 第一轴解释了44.08%的细菌群落的变异度, 第二轴解释了13.43%的变异, 其中根内细菌样品在第一轴与根际及非根际样品显著分开, 而根际及非根际样品在第二轴显著分开(图 2)。MRPP分析发现, 不同生态位细菌群落结构组成差异显著(P<0.05), 换言之, 生态位是影响样品细菌群落组成的主要因素。
|
| 图 2 生姜根不同生态位细菌物种分析比较 Fig. 2 Comparison of bacterial species between bulk soil, rhizosphere soil, and endophyte |
对门水平相对丰度大于1%的细菌种类进行分析, 发现根不同生态位细菌有明显的富集规律。变形菌门(Proteobacteria)主要富集在内生菌样品中, 相对丰度为94.91%。在根际细菌中, 相对丰度显著富集的菌门为拟杆菌门(Bacteroidetes, 12.30%)和厚壁菌门(Firmicutes, 3.67%)。而酸杆菌门(Acidobacteria)、绿弯菌门(Chloroflexi)、芽单胞菌门(Gemmatimonadetes)及硝化螺旋菌门(Nitrospirae)显著富集在非根际样品中。放线菌门(Actinobacteria)、装甲菌门(Armatimonadetes)及浮霉菌门(Planctomycetes)在根际及非根际样品中均有富集。
属水平细菌组成进行分析发现, 不同生态位细菌在属水平上有明显的区分。假单胞菌属(Pseudomonas)、短波单胞菌属(Brevundimonas)、肠杆菌科未知属(Enterobacteriaceae)、寡养单胞菌属(Stenotrophomonas)、泛菌属(Pantoea)主要富集在根内, 且它们均属于变形菌门。鞘氨醇菌属(Chitinophaga)、杆菌属(Bacillus)、Parapedobacter属、Olivibacter属及金黄杆菌属(Chryseobacterium)等主要富集在根际样品中, 且它们属于拟杆菌门、厚壁菌门。其余属水平细菌主要富集在根际细菌中, 他们多数属于酸杆菌门、放线菌门及绿弯菌门。
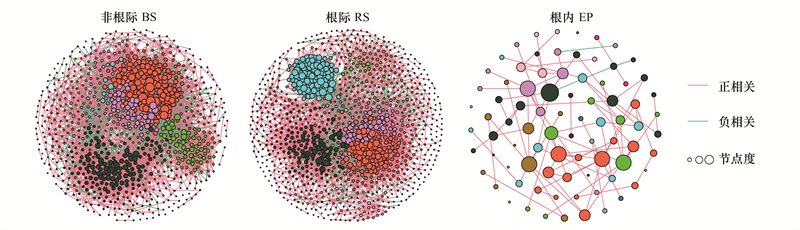
2.3 红壤生姜不同生态位细菌共现网络分析共现网络能够直观地展示微生物群落内部的相互关系。不同生态位细菌群落的共现网络差异较大, 其中内生菌群落模块化程度最高, 为0.66, 而非根际细菌仅为0.48。值得注意的是, 共现网络中细菌间的相关性以正相关为主(范围为75.25%—96.43%), 表明总体上细菌群落以合作关系为主, 其中内生菌正相关相互关系占比最高。而内生菌节点、度以及边数最少, 说明内生菌群落以更强的合作关系为主, 网络复杂度和稳定性最差。而根际细菌及非根际细菌的网络复杂度和稳定性相对较高, 其中非根际细菌网络拥有更低的负相关关系(表 1)。
| 参数 Parameter |
非根际 BS |
根际 RS |
根内 EP |
参数 Parameter |
非根际 BS |
根际 RS |
根内 EP |
|
| 模块化Modularity | 0.48 | 0.63 | 0.66 | 节点Node | 467 | 588 | 74 | |
| 正相关Positive correlation | 75.25% | 88.40% | 96.43% | 边Edge | 5858 | 4819 | 84 | |
| 节点度Node degree | 12.93 | 8.59 | 2.05 | |||||
| BS: 非根际Bulk soil;RS: 根际Rhizosphere soil;EP: 根内Endosphytes | ||||||||
|
| 图 3 生姜根不同生态位细菌共现网络图 Fig. 3 Co-occurrence network of bacteria in different niches of ginger root 一个边代表一个强大的(Spearman r>0.6)和显著(P<0.01)的相关性; 每个节点的大小与连接数(即节点度)成比例; 网络中的节点按照模块着色 |
根系不同生态位的细菌群落中具有一些独特ASVs或特征微生物, 这些微生物的种类和数量可能与环境条件和所处生态位有关。三元相图显示, 在非根际及根际样品中, 多数ASV属于变形菌门、拟杆菌门及放线菌门。而内生菌多属于变形菌门。韦恩图可比较直观地观察到非根际、根际及内生菌样品中共有相同ASV的数量关系, 三者含共有ASV为84个, 其中非根际样品和根际样品共有ASV为212个, 根际及内生菌共有ASV为132个, 非根际与内生菌共有ASV为94个。利用溯源分析对不同生态位ASVs溯源分析发现, 细菌群落的来源存在一定的相关性:根际细菌群落中检测到非根际细菌源, 约占42.8%。内生菌中检测到根际细菌源, 约占47.3%。
2.5 土壤理化条件对根系不同生态位细菌群落的影响为明确土壤理化条件对细菌群落的影响, 本研究将生姜根系不同生态位细菌群落组成与土壤理化性质进行Mantel-test检验(表 2)。检验结果表明, 非根际细菌群落受土壤pH的影响, 根际细菌群落组成受AP的影响, 而内生菌群落与土壤养分无显著相关性(P<0.05)。
| 理化因子 Soil properties |
均值 Mean |
非根际BS | 根际RS | 根内EP | |||||
| r | P | r | P | r | P | ||||
| pH | 5.80 | 0.525 | 0.007* | 0.287 | 0.074 | 0.066 | 0.339 | ||
| TN | 0.04% | 0.139 | 0.152 | -0.22 | 0.92 | 0.132 | 0.193 | ||
| TP | 0.03% | 0.03 | 0.432 | -0.178 | 0.766 | -0.357 | 0.954 | ||
| TK | 1.08% | -0.167 | 0.828 | -0.143 | 0.741 | 0.088 | 0.334 | ||
| NN | 11.27 mg/kg | -0.029 | 0.499 | 0.071 | 0.307 | 0.085 | 0.315 | ||
| AP | 2.87 mg/kg | 0.168 | 0.234 | 0.369 | 0.024* | -0.118 | 0.517 | ||
| AK | 156.45 mg/kg | 0.25 | 0.068 | 0.081 | 0.363 | 0.09 | 0.302 | ||
| OM | 0.68% | 0.258 | 0.069 | -0.251 | 0.942 | 0.223 | 0.152 | ||
| TN: 全氮Total nitrogen;TP: 全磷Total phosphorus;TK: 全钾Total potassium;NN: 硝态氮Nitrate nitrogen;AP: 速效磷Available phosphorus;AK: 速效钾Available potassium;OM: 有机质Organic matter;*P<0.05 | |||||||||
本研究发现土壤中(非根际及根际)细菌群落多样性显著高于根内生菌多样性, 这与其他寄主植物根系相关细菌群落的分布规律相似[24—27]。这是由于土壤环境具有相对丰富的养分资源, 尤其是根际土壤环境作为非根际及根表之间的活跃过渡区, 受益于根分泌物、根脱落物等营养释放, 为微生物群落的生长、发育和繁殖提供了适宜的生态位[29]。相反, 寄主内生菌环境条件相对苛刻。成功定殖到根内的细菌, 必须能够表达与细胞壁降解酶产生相关的基因并能够对一系列宿主植物先天免疫反应产生抗性, 因此能够适应宿主植物内环境的细菌种类相对较少, 内生菌的种类多样性和均匀性明显较低[28]。另外, 细菌群落内部稳定性表现为非根际微生物>根际微生物>内生菌, 这是由于三者受生姜根系的影响越来越强, 这种环境过滤作用导致细菌群落功能冗余性降低, 环境依赖性增强。
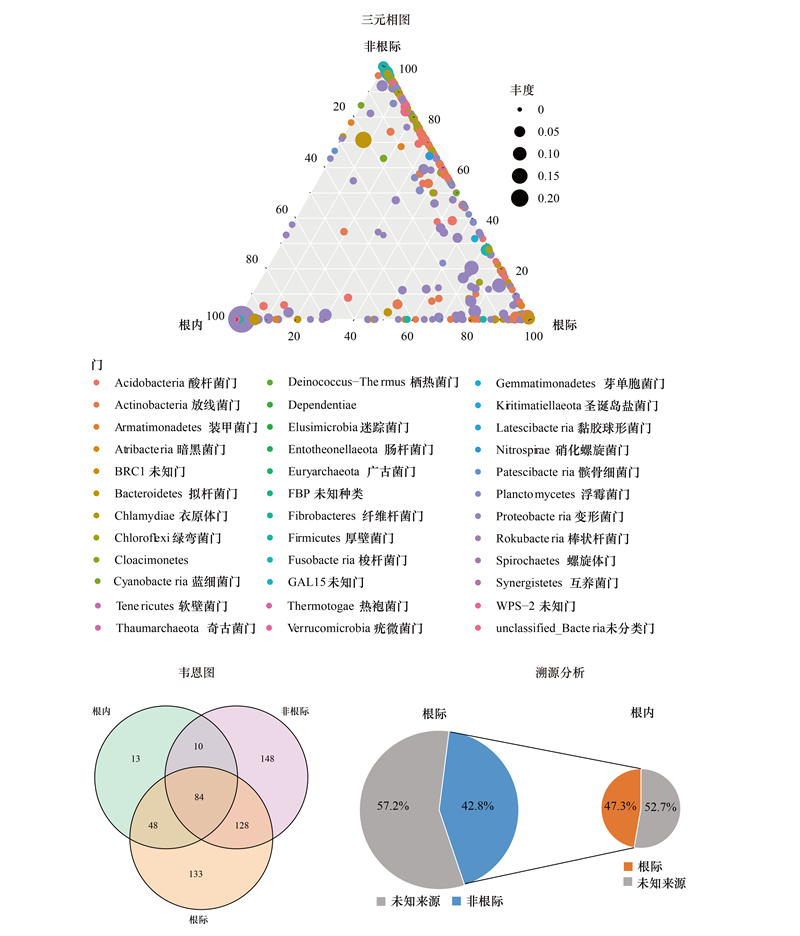
|
| 图 4 生姜根不同生态位细菌群落组成的共性及差异分析 Fig. 4 The similarities and difference of bacterial community composition in different niche of ginger root 三元相图每个点代表一个ASV, 每个点的大小、颜色和位置分别代表改ASV的相对丰度、所属细菌门和所属生态位;韦恩图数字代表了处于不同生态位共享或独有的ASV数量;溯源分析百分数代表来源占比 |
根系不同生态位优势菌种类不一。变形菌门(Proteobacteria)是生姜根内的优势门类, 这与豆科植物根内优势菌一致[29]。其中假单胞菌属(Pseudomonas)、短波单胞菌属(Brevundimonas)、寡养单胞菌属(Stenotrophomonas)及泛菌属(Pantoea)等是该门类主要贡献者, 报道显示它们在植物体内具有特定的适应性策略, 其定殖利于作物的健康生长[7—8]。如Pseudomonas、Stenotrophomonas等已经被证实作为主要的植物防御信号通路和/或能够抑制病原体的生长和毒力, 从而改善植物的胁迫等[30—32]。在根际细菌中, 拟杆菌门(Bacteroidetes)及厚壁菌门(Firmicutes)显著富集, 丰度优势主要由鞘氨醇菌属(Chitinophaga)、杆菌属(Bacillus)、金黄杆菌属(Chryseobacterium)、Parapedobacter及Olivibacter所贡献。其中Bacillus、Chitinophaga及Chryseobacterium有潜在的生防功能[33], 能够在宿主植物遭受病原菌侵袭时由根际进入根内, 协助宿主植物抵御病原真菌[34—35]。意外地是, 生姜根不同生态位优势菌的富集规律与其他植物相比, 并无显著宿主特异性, 本研究认为是根不同生态位微环境条件主导了优势菌地位, 而不是宿主基因型或特殊的挥发物, 这种观点在其他文献中亦有报道[36]。
内生菌群落的低多样性以及溯源分析结果, 均与之前报道的作物微生物组主要来自土壤环境并逐步被宿主植物富集和过滤的观点一致[9, 24—25, 37—38]。但由于宿主和土壤环境的差异, 群落多样性的变化并不一致, 如一项针对根系不同生态位的龙舌兰细菌群落的研究发现, 其根际细菌群落多样性显著高于非根际及根内细菌群落, 这是由于龙舌兰宜生长在干旱贫瘠的环境中, 根际环境由于根系分泌物及水分的渗透而获得相对丰富的代谢底物, 从而提高了根际环境的微生物多样性[39]。植物宿主对细菌网络复杂性有很强的影响, 可能源于宿主植物的定向选择降低了细菌群落功能冗余作用, 导致了群落内部的竞争作用减弱, 抗干扰能力降低[40—43]。在拟南芥根相关细菌群落分析中发现放线菌门及变形菌门为内生菌显著富集的门类, 研究者认为这两个门类下多为富营养型细菌, 在有机资源丰富的情况下有更强的竞争优势, 且该门类下植物促生菌较多[44]。当然, 不同的宿主植物、不同的土壤类型, 都对根内细菌的富集特点产生一定的影响[45—47], 但总体上得到这样的共性规律:随着宿主植物-细菌群落的“空间亲密关系”逐渐加强(从非根际到根内), 细菌群落经历了多样性的急剧丧失、群落组成相对保守的过程。而那些在近根或根内被耗尽的细菌类群, 被认为是宿主植物免疫系统遏制、或者是该生态位在营养代谢上无法满足生存需求导致的[9]。
土壤细菌群落结构在一定程度上受到土壤理化环境的影响[48—49], 但具体影响因子各不相同, 主要与特定土壤特点、采样生育期、作物种类等有关[50]。以往的研究发现, 土壤pH是影响细菌群落组成的重要因子[51—52]。如黄穗萍等[53]认为土壤pH是影响香蕉根际及非根际细菌群落的主要因素, 而本研究中, 生姜非根际细菌群落也显著受土壤pH影响, 而根际细菌及内生菌群落不受pH影响。本研究发现AP是影响根际细菌群落的关键因素, 这与曹曼曼等[50]针对铁尾矿芦苇根际细菌群落的研究一致。而内生菌群落不受土壤理化因子的影响, 本研究认为这是由于内生菌存在于生姜根系内部, 与土壤环境形成物理分隔, 因此不受土壤理化因子的直接影响, 而受植物内环境的直接干预[54]。
本研究以生姜作为宿主植物, 对细菌群落构建的选择作用、潜在细菌群落来源及富集过程提供了可靠的视角, 进一步支持了土壤微生物与宿主植物的共进化, 为后续进行促生菌及生防菌的筛选奠定了基础, 并为未来基于土壤、作物微生物组精准调控的可持续农业管理提供了重要信息。但鉴于本研究样品采集于鹰潭红壤生态试验站生姜连作地块, 未进行多品种、多区域大规模采样, 因此仍有种植品种单一、区域代表性弱等诸多不足, 而针对根系不同生态位细菌群落的研究仍需后续代表性规模采样来完善。
4 结论本研究发现, 生姜根内细菌多样性显著低于根际和非根际细菌群落。不同生态位细菌群落组成差异显著, 其中Proteobacteria是根内优势菌门, 该门类下优势菌Pseudomonas、Brevundimonas、Stenotrophomona及Pantoea显著富集。在根际细菌中, Bacteroidetes及Firmicutes显著富集, 丰度优势主要由Chitinophaga、Bacillus、Chryseobacterium等所贡献。各生态位细菌群落受不同土壤环境因子的影响, 主要表现为土壤pH与非根际细菌群落密切相关, AP与根际细菌群落组成显著相关, 而内生菌群落不受土壤理化因子影响。另外, 各生态位共现网络稳定性和复杂度表现为非根际>根际>根内细菌群落。虽然生姜根内、根际和非根际土壤环境中的细菌群落差异显著, 但不同生态位细菌有一定的关联性, 根际土壤细菌群落42.8%源自非根际样品, 而内生菌群落47.3%源自根际样品, 证实了生姜根系对环境细菌群落的过滤和筛选作用及细菌群落的生态位横向传播作用。总之, 本研究明确了不同生态位细菌群落的共性与差异, 为生姜细菌群落构建的选择作用、潜在有益菌来源及富集过程提供了理论指导。
| [1] |
Liu H W, Brettell L E, Qiu Z G, Singh B K. Microbiome-mediated stress resistance in plants. Trends in Plant Science, 2020, 25(8): 733-743. DOI:10.1016/j.tplants.2020.03.014 |
| [2] |
卜宣尹, 杨卫丽. 植物内生菌抑菌机制和抑菌次生代谢产物的研究进展. 现代药物与临床, 2021, 36(10): 2200-2206. |
| [3] |
陈海生, 刘守平, 杨万勤, 梁国钱. 雅鲁藏布江下游沿岸湿地建群种植物根际土壤细菌群落结构和多样性特征. 生态学报, 2022, 42(4): 1527-1537. |
| [4] |
崔晓辰. 根际微生物与土壤植物关系的研究进展. 现代农业研究, 2021, 27(5): 34-35, 49-49. DOI:10.3969/j.issn.1674-0653.2021.05.015 |
| [5] |
Müller D B, Vogel C, Bai Y, Vorholt J A. The plant microbiota: systems-level insights and perspectives. Annual Review of Genetics, 2016, 50: 211-234. DOI:10.1146/annurev-genet-120215-034952 |
| [6] |
Mendes R, Kruijt M, de Bruijn I, Dekkers E, Van Der Voort M, Schneider J H M, Piceno Y M, DeSantis T Z, Andersen G L, Bakker P A H M, Raaijmakers J M. Deciphering the rhizosphere microbiome for disease-suppressive bacteria. Science, 2011, 332(6033): 1097-1100. DOI:10.1126/science.1203980 |
| [7] |
Bulgarelli D, Schlaeppi K, Spaepen S, Ver Loren Van Themaat E, Schulze-Lefert P. Structure and functions of the bacterial microbiota of plants. Annual Review of Plant Biology, 2013, 64: 807-838. DOI:10.1146/annurev-arplant-050312-120106 |
| [8] |
Beckers B, De Beeck M O, Weyens N, Boerjan W, Vangronsveld J. Structural variability and niche differentiation in the rhizosphere and endosphere bacterial microbiome of field-grown poplar trees. Microbiome, 2017, 5: 25. DOI:10.1186/s40168-017-0241-2 |
| [9] |
Bulgarelli D, Rott M, Schlaeppi K, Ver Loren Van Themaat E, Ahmadinejad N, Assenza F, Rauf P, Huettel B, Reinhardt R, Schmelzer E, Peplies J, Gloeckner F O, Amann R, Eickhorst T, Schulze-Lefert P. Revealing structure and assembly cues for Arabidopsis root-inhabiting bacterial microbiota. Nature, 488(7409): 91-95.
|
| [10] |
Schulz B J E, Boyle C J C, Sieber T N. Microbial Root Endophytes. Berlin Heidelberg: Springer, 2006.
|
| [11] |
刘迎雪, 赵滢, 张宝香, 杨义明, 范书田, 李昌禹, 王月, 许培磊, 秦红艳, 路文鹏. 植物内生细菌来源及生物学功能研究进展. 特产研究, 2020, 42(4): 60-67. DOI:10.16720/j.cnki.tcyj.2020.04.011 |
| [12] |
李艳星, 郭平毅, 孙建光. 块根块茎类作物内生固氮菌分离鉴定、系统发育与促生特性. 中国农业科学, 2017, 50(1): 104-122. |
| [13] |
黎晓菊, 庹呈杰, 黄之镨, 张晓梅, 赵庆. 生姜中6个姜辣素类成分的抗菌活性研究. 天然产物研究与开发, 2022, 34(2): 220-225. DOI:10.16333/j.1001-6880.2022.2.006 |
| [14] |
唐贤. 不同土地利用方式和母质下红壤剖面酸化特征[D]. 长春: 吉林农业大学, 2018.
|
| [15] |
Edwards J, Johnson C, Santos-Medellín C, Lurie E, Podishetty N K, Bhatnagar S, Eisen J A, Sundaresan V. Structure, variation, and assembly of the root-associated microbiomes of rice. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(8): E911-E920. |
| [16] |
Bartram A K, Lynch M D J, Stearns J C, Moreno-Hagelsieb G, Neufeld J D. Generation of multimillion-sequence 16S rRNA gene libraries from complex microbial communities by assembling paired-end Illumina reads. Applied and Environmental Microbiology, 2011, 77(11): 3846-3852. DOI:10.1128/AEM.02772-10 |
| [17] |
Chelius M K, Triplett E W. The diversity of archaea and bacteria in association with the roots of Zea mays L. Microbial Ecology, 2001, 41(3): 252-263. DOI:10.1007/s002480000087 |
| [18] |
Caporaso J G, Kuczynski J, Stombaugh J, Bittinger K, Bushman F D, Costello E K, Fierer N, Peña A G, Goodrich J K, Gordon J I, Huttley G A, Kelley S T, Knights D, Koenig J E, Ley R E, Lozupone C A, McDonald D, Muegge B D, Pirrung M, Reeder J, Sevinsky J R, Turnbaugh P J, Walters W A, Widmann J, Yatsunenko T, Zaneveld J, Knight R. QIIME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [19] |
鲁如坤. 土壤农业化学分析方法. 北京: 中国农业科技出版社, 2000.
|
| [20] |
中华人民共和国国家质量监督检验检疫总局, 中国国家标准化管理委员会. GB/T 32737-2016土壤硝态氮的测定紫外分光光度法. 北京: 中国标准出版社, 2017.
|
| [21] |
Pruesse E, Quast C, Knittel K, Fuchs B M, Ludwig W, Peplies J, Glöckner F O. SILVA: a comprehensive online resource for quality checked and aligned ribosomal RNA sequence data compatible with ARB. Nucleic Acids Research, 2007, 35: 7188-7196. DOI:10.1093/nar/gkm864 |
| [22] |
Hammer Ø, Harper D A T, Ryan P D. PAST: Paleontological statistics software package for education and data analysis. Palaeontologia Electronica, 2001, 4(1): 1-9. |
| [23] |
Shannon P, Markiel A, Ozier O, Baliga N S, Wang J T, Ramage D, Amin N, Schwikowski B, Ideker T. Cytoscape: a software environment for integrated models of biomolecular interaction networks. Genome Research, 2003, 13(11): 2498-2504. DOI:10.1101/gr.1239303 |
| [24] |
王桥美, 严亮, 杨瑞娟, 彭文书, 季爱兵, 刘丽, 张文杰, 胡先奇. 普洱地区茶叶内生细菌与根际土壤细菌群落结构分析. 微生物学报, 2022, 62(2): 703-714. DOI:10.13343/j.cnki.wsxb.20210316 |
| [25] |
Xiong C, Zhu Y G, Wang J T, Singh B, Han L L, Shen J P, Li P P, Wang G B, Wu C F, Ge A H, Zhang L M, He J Z. Host selection shapes crop microbiome assembly and network complexity. New Phytologist, 2021, 229(2): 1091-1104. DOI:10.1111/nph.16890 |
| [26] |
Geszvain K, Visick K L. Roles of bacterial regulators in the symbiosis between Vibrio fischeri and Euprymna scolopes//Overmann J, ed. Molecular Basis of Symbiosis. Berlin, Heidelberg: Springer, 2005, 41: 277-290.
|
| [27] |
Buée M, De Boer W, Martin F, Van Overbeek L, Jurkevitch E. The rhizosphere zoo: an overview of plant-associated communities of microorganisms, including phages, bacteria, archaea, and fungi, and of some of their structuring factors. Plant and Soil, 2009, 321(1/2): 189-212. |
| [28] |
Qian X, Li H Z, Wang Y L, Wu B W, Wu M S, Chen L, Li X C, Zhang Y, Wang X P, Shi M M, Zheng Y, Guo L D, Zhang D X. Leaf and root endospheres harbor lower fungal diversity and less complex fungal co-occurrence patterns than rhizosphere. Frontiers in Microbiology, 2019, 10: 1015. DOI:10.3389/fmicb.2019.01015 |
| [29] |
Zhou Z Y, Yu M H, Ding G D, Gao G L, He Y Y. Diversity and structural differences of bacterial microbial communities in rhizocompartments of desert leguminous plants. PLoS One, 2020, 15(12): e0241057. DOI:10.1371/journal.pone.0241057 |
| [30] |
Saravanakumar D, Kavino M, Raguchander T, Subbian P, Samiyappan R. Plant growth promoting bacteria enhance water stress resistance in green gram plants. Acta Physiologiae Plantarum, 2011, 33(1): 203-209. DOI:10.1007/s11738-010-0539-1 |
| [31] |
Chen Y, Jing W, Nan Y, Wen Z Y, Sun X P, Chai Y R, Ma Z H. Wheat microbiome bacteria can reduce virulence of a plant pathogenic fungus by altering histone acetylation. Nature Communications, 2018, 9(1): 3429. DOI:10.1038/s41467-018-05683-7 |
| [32] |
Berendsen R L, Vismans G, Yu K, Song Y, de Jonge R, Burgman W P, Burmølle M, Herschend J, Bakker P A H M, Pieterse C M J. Disease-induced assemblage of a plant-beneficial bacterial consortium. The ISME Journal, 2018, 12(6): 1496-1507. DOI:10.1038/s41396-018-0093-1 |
| [33] |
Wen T, Zhao M L, Liu T, Huang Q W, Yuan J, Shen Q R. High abundance of Ralstonia solanacearum changed tomato rhizosphere microbiome and metabolome. BMC Plant Biology, 2020, 20: 166. DOI:10.1186/s12870-020-02365-9 |
| [34] |
Carrión V J, Perez-Jaramillo J, Cordovez V, Tracanna V, De Hollander M, Ruiz-Buck D, Mendes L W, Van Ijcken W F J, Gomez-Exposito R, Elsayed S S, Mohanraju P, Arifah A, Van Der Oost J, Paulson J N, Mendes R, Van Wezel G P, Medema M H, Raaijmakers J M. Pathogen-induced activation of disease-suppressive functions in the endophytic root microbiome. Science, 2019, 366(6465): 606-612. DOI:10.1126/science.aaw9285 |
| [35] |
Yan D B, Xia P H, Song X L, Lin T, Cao H P. Community structure and functional diversity of epiphytic bacteria and planktonic bacteria on submerged macrophytes in Caohai Lake, Southwest of China. Annals of Microbiology, 2019, 69(9): 933-944. DOI:10.1007/s13213-019-01485-4 |
| [36] |
Nemergut D R, Cleveland C C, Wieder W R, Washenberger C L, Townsend A R. Plot-scale manipulations of organic matter inputs to soils correlate with shifts in microbial community composition in a lowland tropical rain forest. Soil Biology and Biochemistry, 2010, 42(12): 2153-2160. DOI:10.1016/j.soilbio.2010.08.011 |
| [37] |
Vannier N, Mony C, Bittebiere A K, Michon-Coudouel S, Biget M, Vandenkoornhuyse P. A microorganisms' journey between plant generations. Microbiome, 2018, 6: 79. DOI:10.1186/s40168-018-0459-7 |
| [38] |
Yu P, He X M, Baer M, Beirinckx S, Tian T, Moya Y A T, Zhang X C, Deichmann M, Frey F P, Bresgen V, Li C J, Razavi B S, Schaaf G, Von Wirén N, Su Z, Bucher M, Tsuda K, Goormachtig S, Chen X P, Hochholdinger F. Plant flavones enrich rhizosphere Oxalobacteraceae to improve maize performance under nitrogen deprivation. Nature Plants, 2021, 7: 481-499. DOI:10.1038/s41477-021-00897-y |
| [39] |
Coleman-Derr D, Desgarennes D, Fonseca-Garcia C, Gross S, Clingenpeel S, Woyke T, North G, Visel A, Partida-Martinez L P, Tringe S G. Plant compartment and biogeography affect microbiome composition in cultivated and native Agave species. New Phytologist, 2016, 209(2): 798-811. DOI:10.1111/nph.13697 |
| [40] |
Álvarez-Pérez J M, González-García S, Cobos R, Olego M Á, Ibañez A, Díez-Galán A, Garzón-Jimeno E, Coque J J R. Use of endophytic and rhizosphere Actinobacteria from grapevine plants to reduce nursery fungal graft infections that lead to young grapevine decline. Applied and Environmental Microbiology, 2017, 83(24): e01564-17. |
| [41] |
Bai Y, Müller D B, Srinivas G, Garrido-Oter R, Potthoff E, Rott M, Dombrowski N, Münch P C, Spaepen S, Remus-Emsermann M, Hüttel B, Mchardy A C, Vorholt J A, Schulze-Lefert P. Functional overlap of the Arabidopsis leaf and root microbiota. Nature, 2015, 528(7582): 364-369. DOI:10.1038/nature16192 |
| [42] |
Zuo X H, Guo X G, Zhan Y Z, Wu D, Yang Z H, Dong W G, Huang L Q, Ren T G, Jing Y G, Wang Q H, Sun X M, Lin S J. Host selection and niche differentiation in sucking lice (Insecta: Anoplura) among small mammals in southwestern China. Parasitology Research, 2011, 108(5): 1243-1251. DOI:10.1007/s00436-010-2173-7 |
| [43] |
Rodriguez P A, Rothballer M, Chowdhury S P, Nussbaumer T, Falter-Braun P. Systems biology of plant-microbiome interactions. Molecular Plant, 2019, 12(6): 804-821. DOI:10.1016/j.molp.2019.05.006 |
| [44] |
Fierer N, Bradford M A, Jackson R B. Toward an ecological classification of soil bacteria. Ecology, 2017, 88(6): 1354-1364. |
| [45] |
姚领爱, 胡之璧, 王莉莉, 周吉燕, 黎万奎. 植物内生菌与宿主关系研究进展. 生态环境学报, 2010, 19(7): 1750-1754. DOI:10.3969/j.issn.1674-5906.2010.07.042 |
| [46] |
崔晋龙, 郭顺星, 肖培根. 内生菌与植物的互作关系及对药用植物的影响. 药学学报, 2017, 52(2): 214-221. DOI:10.16438/j.0513-4870.2016-0726 |
| [47] |
王娜. 乌头内生细菌群落结构及其与宿主生物碱合成关系研究[D]. 西安: 陕西师范大学, 2014.
|
| [48] |
张雪玲, 臧春华, 郭汝悦, 段毅超, 王延刚, 郭中信, 张振中, 任雪玲. 生物菌肥对连作障碍三七根际和根茎内细菌群落的影响. 河南农业科学, 2021, 50(12): 78-91. DOI:10.15933/j.cnki.1004-3268.2021.12.009 |
| [49] |
吴希慧, 王蕊, 高长青, 高胜, 杜兰兰, KHAN A, BARMON M, 郭胜利. 土地利用驱动的土壤性状变化影响微生物群落结构和功能. 生态学报, 2021, 41(20): 7989-8002. |
| [50] |
曹曼曼, 王飞, 周北海, 陈辉伦, 袁蓉芳. 铁尾矿芦苇根际微生物和根内生菌群落分布及其限制性因子解析. 环境科学, 2021, 42(10): 4998-5009. DOI:10.13227/j.hjkx.202103052 |
| [51] |
Cao P, Zhang L M, Shen J P, Zheng Y M, Di H J, He J Z. Distribution and diversity of archaeal communities in selected Chinese soils. FEMS Microbiology Ecology, 2012, 80(1): 146-158. DOI:10.1111/j.1574-6941.2011.01280.x |
| [52] |
Fierer N, Jackson R B. The diversity and biogeography of soil bacterial communities. Proceedings of the national academy of sciences of the United States of America, 2006, 103(3): 626-631. DOI:10.1073/pnas.0507535103 |
| [53] |
黄穗萍, 金德才, 李其利, 韦绍龙, 唐利华, 黄素梅, 郭堂勋, 罗述名, 莫贱友. 香蕉根际及非根际土壤细菌群落特征及共性规律研究. 南方农业学报, 2022, 53(5): 1153-1262. |
| [54] |
Atkinson C J, Fitzgerald J D, Hipps N A. Potential mechanisms for achieving agricultural benefits from biochar application to temperate soils: a review. Plant and Soil, 2010, 337(1/2): 1-18. |
 2023, Vol. 43
2023, Vol. 43