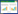文章信息
- 彭羽, 王越, 马江文, 范敏, 白岚, 周涛
- PENG Yu, WANG Yue, MA Jiangwen, FAN Min, BAI Lan, ZHOU Tao
- 基于实地调查和高光谱数据的浑善达克沙地中部植物alpha多样性遥感估测
- Assessment of plant species alpha diversity in central Hunshandak Sandland, China based on field surveys and hyperspectral data
- 生态学报. 2019, 39(13): 4883-4891
- Acta Ecologica Sinica. 2019, 39(13): 4883-4891
- http://dx.doi.org/10.5846/stxb201806221377
-
文章历史
- 收稿日期: 2018-06-22
- 网络出版日期: 2019-04-17
2. 北京师范大学地表过程与资源生态国家重点实验室, 北京 100875
2. State Key Laboratory of Earth Surface Processes and Resource Ecology, Beijing Normal University, Beijing 100875, China
遥感可以为群落物种组成、生态系统结构参数及其时空变化的研究提供丰富的信息。利用遥感技术进行植物多样性估测, 较传统样方调查法具有速度快、成本低、范围大等优势。因此, 遥感技术逐渐成为研究生物多样性的重要工具[1-3]。目前利用遥感进行植物多样性估测, 基本可分为两种方法:一是通过电磁波遥感信息直接评估单个物种、种群和物种多样性; 二是将遥感数据和环境参数应用到物种多样性预测模型中, 对物种多样性进行间接估测[4-5]。
在直接估测中, 机载遥感技术应用于植物物种的直接识别。Valérie等采用两组航空照片分别鉴定出309种树冠, 鉴定树种正确率高达87%[6]。Akasheh等利用机载多光谱数字图像(Airborne multi-spectral digital images, 空间分辨率为0.5 m)为Rio Grande River河岸带植被的物种分布进行制图[7]。在光谱信息分析方面, NDVI(Normalized Difference Vegetation Index, NDVI)被广泛应用于植物多样性的估测[8-12]。NDVI标准差与植物物种丰富度有显著相关性[3]。基于NDVI的EVI指数更适合于指示半干旱环境的植物多样性[13-14]。其他基于NDVI改进的指数还有EVI (Enhanced Vegetation Index)[15], IRI(Infra Red Index), MIRI(Middle Infra Red Index)[16-17], ARVI (Atmosphere Resistance Vegetation Index), SAVI (Soil Adjusted Vegetation Index)[15]等。由于这些光谱植被指数只应用较窄的电磁波段的遥感信息, 因此植物多样性预测精度受到一定限制[16-17]。含有更高波谱分辨率的传感器逐渐得到开发和利用。高光谱数据, 拥有上百至数百个波段信息, 涵盖可见光到近红外、中红外和远红外, 为植物多样性的高精度监测评估提供了可能。但是, 如何从大量的光谱数据中筛选关键信息, 也成为高光谱植物多样性指数估测的重要挑战。
研究发现, 采用线性回归模型筛选敏感波段数据, 能够较好地利用光谱数据预测物种多样性指数[18]。近年来, 采用主成分分析法降低多光谱数据的冗余度, 提取光谱主成分来拟合植物多样性, 也起到了很好的效果[19]。多元线性逐步回归分析, 能够从大量的变量因子中筛选出少数的重要变量因子, 已经广泛的应用于植物营养元素含量[20]、光合生理生态[21]和地上生物量[22]的遥感分析。为此, 本研究基于前人的研究结果, 应用相关性分析、主成分分析和经验波段选择法, 筛选敏感波段, 对选择的敏感波段进行多元逐步线性回归, 比较不同方法的拟合效果, 探索高光谱数据对于植物物种多样性估测的应用潜力, 为植物多样性的遥感估测提供参考。
1 研究地点研究区位于浑善达克沙地中部内蒙古自治区正蓝旗(42°55′—43°5′N; 115°10′—115°35′E)。正蓝旗共包含3个镇、8个苏木和3个国营牧场, 2016年草场面积占到了72.3%, 植被种类相对丰富。该区属于中温带半干旱大陆性季风气候, 主要由固定沙丘、半固定沙丘、流动沙丘和丘间低地、湿地等景观组成(图 1)。研究区植物物种多以沙生草本植物为主。
|
| 图 1 研究地点及样地示意图 Fig. 1 The map shows study area and sample locations |
本研究在2017年7月到8月在浑善达克沙地中部内蒙古正蓝旗进行, 在固定沙丘、半固定沙丘、流动沙丘及低地4种生境的代表性区域共选取8个样地(图 1), 样地大小为30 m×30 m。每个样地沿对角线设置15个直径为0.8 m的圆形草本样方, 共计120个样方。每个样方进行GPS定位, 统计每一样方内草本植物的种类、数量、高度和盖度等数据, 计算植物多样性指数。本文采用常用的4个物种多样性指数:丰富度(Richness, R)、Shannon-Wiener指数(H)、Simpson优势度指数(D)、Pielou均匀度指数(E)。各样地植物群落参数见表 1。
| 样地序号 Sample location code |
优势物种 Dominant species |
样方平均H指数 Average H at plot level |
样方平均盖度 Average cover at plot level |
| 1 | 沙蒿(Artemisia desertorum), 沙米(Ajriophyllum squarrosum) | 0.783 | 6.223 |
| 2 | 沙蒿(Artemisia desertorum), 冷蒿(Artemisia frigida) | 1.262 | 15.051 |
| 3 | 沙蒿(Artemisia desertorum), 冰草(Agropyron cristatum) | 1.836 | 36.113 |
| 4 | 扁穗苔草(Blysmocarex nudicarpa), 拂子茅(Calamagrostis epigeios) | 2.272 | 47.783 |
| 5 | 沙蒿(Artemisia desertorum), 褐沙蒿(Artemisia halodendron) | 0.575 | 5.076 |
| 6 | 沙蒿(Artemisia desertorum), 冷蒿(Artemisia frigida) | 0.832 | 12.334 |
| 7 | 灰绿藜(Chenopodium glaucum), 冰草(Agropyron cristatum) | 1.755 | 31.675 |
| 8 | 扁穗苔草(Blysmocarex nudicarpa), 披碱草(Elymus dahuricus) | 2.063 | 44.806 |
采用手持式地物光谱仪FieldSpec H2 (ASD Corp., U.S.A.)采集各个样方的冠层光谱。仪器光谱范围为325—1075 nm, 光谱分辨率为3 nm, 采样间隔为1 nm。光谱测量在风力小于3级的晴朗天气下进行, 采集时间为北京时间10:00—15:00。测量人员着黑色服装, 避免遮挡阳光, 减少光谱干扰。光谱传感器探头垂直向下, 保持在冠层上方1.8 m内, 确保冠层充满仪器的视场角, 每一样本重复测量10次取均值。测量前均同步测量参考白板反射的辐射光谱用于标定, 并定时进行系统优化, 以消除环境变化所带来的影响。
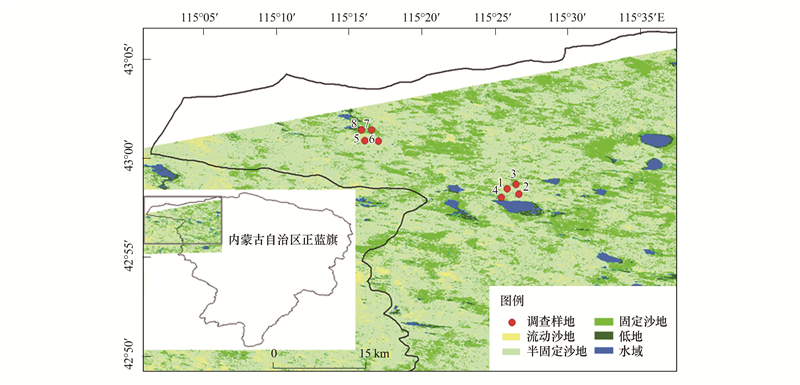
将获得的高光谱数据经滤波和去除噪音后, 根据下式计算一阶微分反射比(FD)。
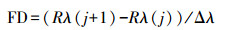
|
式中, FD是波段j和j+1之间中点处波长值为i的一阶微分反射比, Rλ(j)是波段j的反射率, Rλ(j+1)是波段j+1的反射率, Δλ是波长j到j+1的间隔。FD分析用以消除土壤等环境背景值的影响, 提取不同植物群落的光谱特征[23-24], 用于拟合植物物种多样性指数。
2.3 数据分析方法为了降低数据冗余, 提取能够反映植物多样性的重要光谱信息, 本文采用3种方法选择高光谱敏感波段。(1)Pearson相关分析法, 对采集得到的375—1025 nm间波段的FD值与该样方的植物多样性指数进行双变量相关性分析, 筛选出显著相关的波段。(2)主成分分析法(PCA), 对采集的375—1025 nm波段的FD值进行主成分分析, 根据各个波段对主成分的权重系数确定敏感波段。(3)经验波段选取法, 应用已有研究结果发现的对植物多样性敏感波段, 选择这些经验波段。
通过以上3种方法, 分别筛选出待拟合的敏感波段。以敏感波段的FD值为自变量, 植物多样性指数为响应变量, 利用逐步回归分析方法, 将90个植物样方高光谱数据和植物多样性指数进行回归。对建立的多元逐步回归方程进行基于相关性(R2)、显著性(P值)、残差(RMSE)、共线性程度(VIF)和方程复杂性(变量数)[25-27]的比较, 筛选出最优的回归方程。最后, 利用最优回归模型, 将30个未参与回归建模的样地高光谱数据代入, 进行植物多样性的拟合, 并与实际测得的多样性指数进行验证, 评估回归模型的质量。滤波和FD在ViewSpec Pro 6.0 (Analytical Spectral Devices Inc., USA)中完成, 相关分析、PCA和MSLR分析在SPSS 23.0和Excel 2010中完成。
3 结果与分析 3.1 相关性和主成分分析相关性分析结果(表 2)表明, 与H指数显著相关的波段最少, 与R显著相关的最多。与R和E指数显著相关的波段多位于可见光和近红外范围, 波段较宽; 而与H和D指数显著相关的波段相对较窄。
| 多样性指数 Diversity indices |
显著相关的波段及范围/nm The significantly related wavebands and ranges |
| 物种丰富度R Richness |
377—383, 388—392, 398—405, 409—427, 433—515, 521—525, 530—591, 602—627, 629—631, 634—644, 647—655, 659—666, 671—674, 687—689, 692—749, 752—756, 775, 776, 778, 779, 805—809, 824, 825, 827, 828, 837—840, 844—847, 885—889, 901—907, 920, 922, 942, 943, 972, 973, 1013, 1014 |
| Shannon-Wiener指数H Shannon-Wiener index |
412, 413, 415, 421, 434, 454—456, 463, 464, 474, 475, 481, 491, 498, 504, 508—510, 520, 521, 523, 524, 532, 535, 589, 590, 623—625, 631, 632, 662, 669, 670, 764, 790, 792—794, 823, 829—830, 845, 846, 887, 888, 892, 893, 906, 909, 927, 940, 965—967, 972, 973, 990, 991, 1001—1002, 1020—1021 |
| Simpson指数D Simpson index |
401, 402, 408—413, 454—456, 508, 509, 631, 632, 651, 653, 662, 663, 669—671, 681, 682, 688—690, 792—794, 809, 810, 829, 835, 848, 892, 893, 908, 923, 924, 966, 967, 972, 973, 991, 998, 1019—1021 |
| Pielou指数E Pielou index |
378—380, 387—392, 399—406, 410—414, 416—420, 425—427, 435—439, 442—446, 448—453, 457—467, 476—487, 493—499, 511—515, 518—520, 530—532, 536—539, 543—587, 603—617, 630—656, 659—666, 668—673, 687—756, 793—795, 799—801, 806—810, 907—909, 920—928, 965—967, 1019—1021 |
PCA结果发现, 第一主成分(61.56%)和第二主成分(35.90%)能够解释光谱数据变异率的97.46%, 因此第一、二主成分有足够的代表性。为了降低数据冗余, 本文根据不同波段数值在第一、二主成分线性组合中的权重系数, 进一步筛选特征波段。为了筛选到个数较少而贡献值较大的特征波段, 本文参考特征波段数/样方数 < 1的标准[28], 从651个波段中选取了90个特征波段。波长范围为500—658 nm, 权重系数均在0.7065以上(图 2)。
|
| 图 2 植物多样性敏感波段在第一、第二主成分的权重系数 Fig. 2 The weight coefficients of sensitive wavebands in the first and second principle components |
以特征波段的FD值为自变量, 植物多样性指数为响应变量, 进行逐步回归分析, 得到了136个逐步回归方程。在进行相关性(R2)、显著性(P值)、残差(RMSE)、共线性程度(VIF)和方程复杂性(变量数)比较后, 筛选出12个最优回归方程(表 3)。在筛选出的12个逐步回归方程中, 相关性(R2)最高的为基于相关性波段分析的H指数回归方程, 共线性较高的为基于经验波段的E指数回归方程, 但是12个回归方程的共线性都很低(VIF < 10)。这些回归方程的显著性和相关性较高, 残差值和共线性值较低, 可以作为植物多样性指数的拟合模型。
| 波段选择方法 Bands selected method |
提取的波段 Wavebands selected |
多样性指数 Diversity indices |
回归方程 MLSR equations |
P值 P |
决定系数R2 | 均方根误差 RMSE |
方差膨胀因子 VIF |
| 相关性法 Pearson′s correaltion |
表 1 | R | 3.715+2334.78FD825+3620.91FD838+2761.77FD567+644.78FD903+5420.26FD445-3130.89FD454+4792.25FD450-1945.65FD401-3237.21FD674 | 0.000 | 0.843 | 0.875 | 2.80 |
| H | 0.3837+1086.42FD632+728.73FD662+437.55FD829+71.67FD965+938.93FD434+39.69FD991+26.14FD1001-99.48FD906+227.96FD846 | 0.000 | 0.865 | 0.040 | 2.11 | ||
| D | 0.7216-158.87FD688+1052.61FD631+330.46FD848-324.1FD413+14.99FD1021+220.32FD409-217.22FD794 | 0.000 | 0.866 | 0.01 | 2.62 | ||
| E | 0.5736-97.32FD687+803.72FD631-362.1FD611+134.02FD392-95.17FD795+535.47FD651-129.41FD809 | 0.000 | 0.842 | 0.01 | 2.999 | ||
| 主成分法 Principal Component |
497, 501523, 590-654, 658 | R | 9.237+7294.31FD601-4345.57FD628+10834.57FD502-2834.71FD514-7289.54FD503-4689.03FD634+2125.06FD658 | 0.000 | 0.697 | 1.572 | 5.917 |
| Analysis, PCA | H | 0.8857+2840.36FD632+1036.53FD501-1355.45FD503-1480.98FD633+1269.44FD650-1659.22FD611+1171.38FD612 | 0.000 | 0.822 | 0.051 | 5.23 | |
| D | 0.4442+641.62FD632-101.86FD508+301.95FD511-336.13FD610+190.04FD607+775.51FD593-423.87FD627+606.51FD651-346.96FD504 | 0.000 | 0.742 | 0.017 | 5.346 | ||
| E | 0.271+702.53FD632-246.82FD508+369.43FD518+767.37FD651-648.93FD503+632.66FD593-235.02FD627+389.57FD501-295.2FD599 | 0.000 | 0.815 | 0.012 | 3.49 | ||
| 经验波段法[23, 30-31] Empirical bands by published literatures |
420—440, 450—470, 510—570, 630—650, 760—900 | R | 2.989+2167.91FD825+3350.27FD829+4764.05FD451-2136.78FD850+1093.48FD886+2302.81FD844+2120.79FD637-1206.78FD878+1593.45FD785 | 0.000 | 0.853 | 0.757 | 2.494 |
| H | 0.161+2362.17FD632+561.61FD838-511.46FD436+1019.63FD649-364.44FD850-418.82FD857+1157.84FD468-1071.97FD633+332.78FD526 | 0.000 | 0.831 | 0.047 | 3.02 | ||
| D | 0.337+607.77FD632+180.69FD838+307.61FD835-322.1FD435+198.39FD862+202.16FD848+241.96FD560-127.57FD878-170.52FD793 | 0.000 | 0.856 | 0.01 | 2.27 | ||
| E | 0.613+166.75FD632+208.65FD838-264.73FD435+104.88FD882+253.37FD848-127.79FD851-140.97FD520+485.84FD631-155.2FD794 | 0.000 | 0.833 | 0.011 | 8.425 | ||
| RMSE: Root-Mean-Square Error; VIF: Variance Inflation Factor | |||||||
将筛选出来的12个最优回归方程, 代入到另外30个植物样方进行验证。结果表明(图 3), 回归方程表现差异很大。对于通过主成分方法筛选出来的特征波段的回归方程, 拟合效果最好, 其中H、D、E指数拟合显著(r=0.40*, r=0.49**, r=0.65**)。而通过特征波段进行逐步回归的方程拟合效果较差, 通过相关性波段进行回归的方程, 仅有E指数拟合显著。
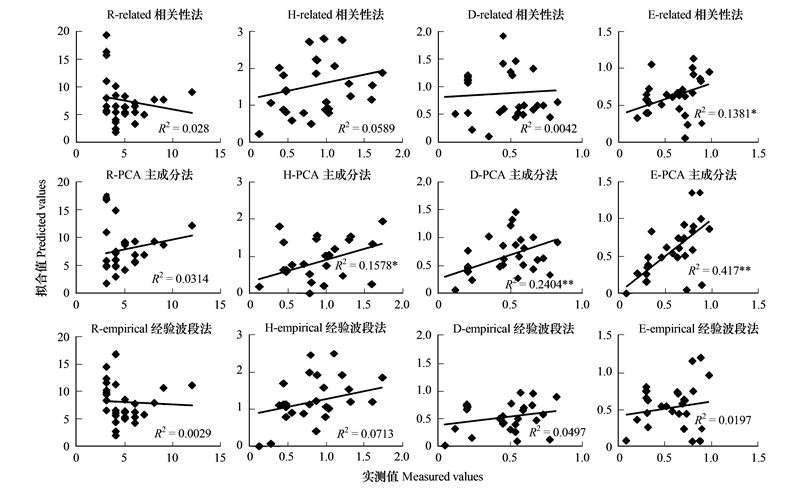
|
| 图 3 植物多样性指数实测值(横坐标)与拟合值(纵坐标)的相关性示意图 Fig. 3 The Linear regression of the field measured values (x-axis) and predicted values (y-axis) for plant species diversity indices in the central Hunshandak Sandland, Northern China 由上到下的图形分别表示经由相关性分析、PCA和经验筛选的波段进行逐步回归分析得到的拟合值与实测值之间的关系 |
高光谱拟合植物多样性指数具有较高的复杂性, 受多重因素影响。不同生境类型的光谱FD曲线表现不同(图 4)。流动沙地FD曲线在400—680 nm处明显高于其他3种沙地类型, 在近红外波段(680—760 nm), FD值表现为丘间低地>固定沙地>半固定沙地>流动沙地。对实测值-拟合值相关性(图 4)的进一步分析表明, 植被盖度适中的固定沙地R, H和D指数的拟合值准确度较高, 其中又以PCA波段选择法拟合度较高。
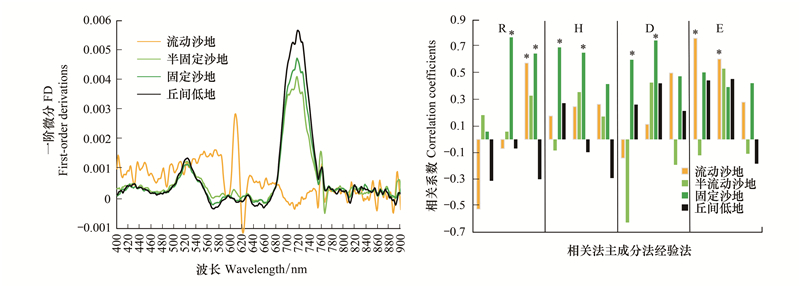
|
| 图 4 不同生境沙地类型光谱FD曲线以及实测alpha多样性指数与相关性分析、PCA和经验波段法拟合多样性指数之间的相关系数 Fig. 4 The FD curves of four kinds of sandy dunes, and the Pearson′s correlation coefficients between filed measured alpha-diversity indices and predicted values based on selected wavebands through correlation, PCA and empirical method, respectively *表示相关系数显著性(P < 0.05) |
特征波段选择一直是高光谱遥感数据分析的中心问题。本文通过相关性分析法、PCA和经验指数筛选法选择了潜在的敏感波段, 并经进一步的逐步回归分析, 选择了少量的贡献值最大的波段, 进行了拟合效果比较。相关性分析对高光谱数据每一个波段的FD值进行了分析, 缩小了波段范围。相比其他方法建立的回归方程, 该方程变量的共线性程度最低, 说明特征波段之间的冗余信息较少。但是该法在验证中表现为不稳定, 未能取得较好效果。其原因可能是由于数百个波段参数之间相关性分析中产生了交互影响效应, 单个波段的贡献难以通过相关性分析识别出来[20-21]。说明相关性分析选择的高相关波段, 不一定是含有关键光谱信息的特征波段。而根据文献研究筛选的敏感波段进行拟合的结果也不理想。由于没有直接利用高光谱数据模拟植物多样性指数的相关研究, 经验波段选择的依据多是空间分辨率高于3 m的机载高光谱遥感图像的分析结果[22, 29-30]。本文是基于微尺度(0.8 m)植物样方的高光谱数据进行的拟合分析, 尺度的转换以及传感器的差异可能使选择出来的特征波段难以适应。多项研究结果表明, 高空间分辨率的遥感数据能够提高植物多样性的模拟效果[31]。因此不同分辨率遥感影像筛选的特征波段范围会有差异。与相关性分析和经验法选择波段相比, 基于主成分分析法选择特征波段的回归方程得到了最好的多样性指数预测值。主成分分析能够识别少量的含有大量光谱特征信息的特征波段, 在其他传感器遥感数据研究中也取得较好效果[19], 表现出较高的稳定性。
本文优选的光谱模型验证精度表现一般, 其原因可能在于植物多样性遥感反演的复杂性。首先, 研究地点是沙质草地, 传感器接受的植被信号受沙地强烈的反射光影响。本文筛选的主成分波段范围为500—658 nm, 也说明可见光是收集样方光谱的主要部分。对于流动沙地和半固定沙地, 对FD值的影响更明显, 拟合精度不高。其次, 研究地点生境多样, 复杂的环境条件也会降低模型准确度。本研究地点为流动沙地、半固定沙地、固定沙地和低地, 样方植被盖度范围为5%—48%, 收集到的样方光谱数据差异性较高。本研究也通过分析四种生境条件下光谱模型的拟合度, 发现同一光谱模型在不同生境会表现出明显的差异。为了开发适宜性广的光谱模型, 没有区分不同生境分别建模。因为在实际应用中, 大面积的草地生态系统类型是多样性生境类型的混合, 研究的重点应该是开发能够拟合大面积生态系统的普适性的遥感模型, 发挥遥感进行大尺度植物多样性指数估算的优势。其三, 沙地植物群落多样性的光谱特征, 还受样方土壤类型、土壤含水量、群落复杂性、群落盖度、地表凋落物、植株阴影等因素的影响[32]。研究表明, 群落不同植物的高度差异、物种多样性组成以及群落分层特征都将影响群落的光谱特征。例如, 相同物种组成的植物群落, 由于植株间盖度、发育阶段和叶片倾角的不同, 而呈现出不同的光谱多样性指数[32]。本研究也表明, 植被盖度中等的固定沙地的植物多样性指数模型拟合效果较好(图 4)。如何将这些影响因素全部纳入高光谱模型, 尚需开展系列控制条件下的大量定量实验研究。其四, 不同植物多样性指数的拟合效果也有差异。H, D, E指数拟合效果较好的原因, 一是这些指数的计算考虑到了不同物种的多度, 同时这些指数也不受取样单位的影响, 开发的光谱模型能够反映这些特征。与之相反, 物种丰富度的拟合效果较差。其原因可能是物种丰富度仅考虑单位样方的种类数而没有考虑其多度和比例[33], 难以用光谱模型表达。一个样方的植物群落光谱特征不仅受物种种类影响, 也受物种组成比例的影响, 因此丰富度指数与光谱模型的契合度较差。
由于植物物种多样性空间分布具有尺度效应, 不考虑尺度效应分析生物多样性可能会导致错误的结论[19, 34]。遥感数据由于具有粒度效应和幅度效应[12, 35-36], 可以和植物多样性空间尺度效应结合起来进行分析, 探讨适合于植物多样性拟合的最优粒度和幅度。从微尺度到大尺度的多尺度研究有利于消除一般规则下的错误判断, 从而正确评估物种多样性。随着高分辨率高光谱卫星影像和近地面无人机高光谱遥感技术的发展, 高光谱遥感数据和影像直接监测植物物种多样性表现出了较大的应用潜力。本文基于敏感波段选择方法, 比较了不同选择方法光谱模型拟合效果的差异, 为高光谱应用于植物多样性的遥感估测提供了初步参考。未来的研究可以利用不同空间尺度(从0.3 m到60 m的系列空间分辨率)的高光谱遥感数据, 对不同空间尺度的植物多样性的遥感模型特征进行比较, 揭示植物多样性遥感模型的尺度转换机制。同时, 开展不同生境类型(流动沙地、半流动沙地、固定沙地和低地)、不同季节和不同群落条件(盖度、生活型、高度)下植物多样性光谱特征的时空差异研究, 探究普适性广和精确度高的遥感模型, 促进植物多样性参数的快速和精准估测。
| [1] |
郭中伟, 李典谟, 甘雅玲. 森林生态系统生物多样性的遥感评估. 生态学报, 2001, 21(8): 1369-1384. DOI:10.3321/j.issn:1000-0933.2001.08.023 |
| [2] |
方彬, 陈波, 张元. 生物多样性遥感监测尺度选择及制图研究. 地理与地理信息科学, 2007, 23(6): 78-81. DOI:10.3969/j.issn.1672-0504.2007.06.018 |
| [3] |
徐文婷, 吴炳方. 遥感用于森林生物多样性监测的进展. 生态学报, 2005, 25(5): 1199-1204. DOI:10.3321/j.issn:1000-0933.2005.05.037 |
| [4] |
胡海德, 李小玉, 杜宇飞, 郑海峰, 都本绪, 何兴元. 生物多样性遥感监测方法研究进展. 生态学杂志, 2012, 31(6): 1591-1596. |
| [5] |
郭庆华, 刘瑾, 李玉美, 翟秋萍, 王永财, 吴芳芳, 胡天宇, 万华伟, 刘慧明, 申文明. 生物多样性近地面遥感监测——应用现状与前景展望. 生物多样性, 2016, 24(11): 1249-1266. DOI:10.17520/biods.2016059 |
| [6] |
Valérie T, Marie-Pierre J. Tree species identification on large-scale aerial photographs in a tropical rain forest, French Guiana-application for management and conservation. Forest Ecology and Management, 2006, 225(1/3): 51-61. |
| [7] |
Akasheh O Z, Neale C M U, Jayanthi N H. Detailed mapping of riparian vegetation in the middle Rio Grande River using high resolution multi-spectral airborne remote sensing. Journal of Arid Environments, 2008, 72(9): 1734-1744. DOI:10.1016/j.jaridenv.2008.03.014 |
| [8] |
Lauver C L. Mapping species diversity patterns in the Kansas shortgrass region by integrating remote sensing and vegetation analysis. Journal of Vegetation Science, 1997, 8(3): 387-394. DOI:10.2307/3237328 |
| [9] |
Nagendra H, Gadgil M. Satellite imagery as a tool for monitoring species diversity:an assessment. Journal of Applied Ecology, 1999, 36(3): 388-397. DOI:10.1046/j.1365-2664.1999.00406.x |
| [10] |
Gould W. Remote sensing of vegetation, plant species richness, and regional biodiversity hotspots. Ecological Applications, 2000, 10(6): 1861-1870. DOI:10.1890/1051-0761(2000)010[1861:RSOVPS]2.0.CO;2 |
| [11] |
Muldavin E H, Neville P, Harper G. Indices of grassland biodiversity in the Chihuahuan desert ecoregion derived from remote sensing. Conservation Biology, 2001, 15(4): 844-855. DOI:10.1046/j.1523-1739.2001.015004844.x |
| [12] |
Rocchini D. Effects of spatial and spectral resolution in estimating ecosystem α-diversity by satellite imagery. Remote Sensing of Environment, 2007, 111(4): 423-434. DOI:10.1016/j.rse.2007.03.018 |
| [13] |
John R, Chen J Q, Lu N, Guo K, Liang C Z, Wei Y F, Noormets A, Ma K P, Han X G. Predicting plant diversity based on remote sensing products in the semi-arid region of Inner Mongolia. Remote Sensing of Environment, 2008, 112(5): 2018-2032. DOI:10.1016/j.rse.2007.09.013 |
| [14] |
Waring R H, Coops N C, Fan W, Nightingale J M. MODIS enhanced vegetation index predicts tree species richness across forested ecoregions in the contiguous U.S.A. Remote Sensing of Environment, 2006, 103(2): 218-226. DOI:10.1016/j.rse.2006.05.007 |
| [15] |
Cabacinha C D, de Castro S S. Relationships between floristic diversity and vegetation indices, forest structure and landscape metrics of fragments in Brazilian Cerrado. Forest Ecology and Management, 2009, 257(10): 2157-2165. DOI:10.1016/j.foreco.2009.02.030 |
| [16] |
Bawa K, Rose J, Ganeshaiah K N, Barve N. Assessing biodiversity from space:an example from the western Ghats, India. Conservation Ecology, 2002, 6(2): 7. |
| [17] |
Nagendra H, Rocchini D, Ghate R, Sharma B, Pareeth S. Assessing plant diversity in a dry tropical forest:comparing the utility of Landsat and Ikonos satellite images. Remote Sensing, 2010, 2(2): 478-496. DOI:10.3390/rs2020478 |
| [18] |
Mohammadi J, Shataee S. Possibility investigation of tree diversity mapping using Landsat ETM+ data in the Hyrcanian forests of Iran. Remote Sensing of Environment, 2010, 114(7): 1504-1512. DOI:10.1016/j.rse.2010.02.004 |
| [19] |
Turtureanu P D, Palpurina S, Becker T, Dolnik C, Ruprecht E, Sutcliffe L M E, Szabó A, Dengler J. Scale-and taxon-dependent biodiversity patterns of dry grassland vegetation in Transylvania. Agriculture, Ecosystems & Environment, 2014, 182: 15-24. |
| [20] |
Yao X, Huang Y, Shang G Y, Zhou C, Cheng T, Tian Y C, Cao W X, Zhu Y. Evaluation of six algorithms to monitor wheat leaf nitrogen concentration. Remote Sensing, 2015, 7(11): 14939-14966. DOI:10.3390/rs71114939 |
| [21] |
Stratoulias D, Balzter H, Zlinszky A, Tóth V R. Assessment of ecophysiology of lake shore reed vegetation based on chlorophyll fluorescence, field spectroscopy and hyperspectral airborne imagery. Remote Sensing of Environment, 2015, 157: 72-84. DOI:10.1016/j.rse.2014.05.021 |
| [22] |
Yue J B, Feng H L, Yang G J, Li Z H. A comparison of regression techniques for estimation of above-ground winter wheat biomass using near-surface spectroscopy. Remote Sensing, 2018, 10(1): 66. |
| [23] |
Chen S S, Li D, Wang Y F, Peng Z P, Chen W Q. Spectral characterization and prediction of nutrient content in winter leaves of litchi during flower bud differentiation in southern China. Precision Agriculture, 2011, 12(5): 682-698. DOI:10.1007/s11119-010-9213-2 |
| [24] |
Abdel-Rahman E M, Ahmed F B, Ismail R. Random forest regression and spectral band selection for estimating sugarcane leaf nitrogen concentration using EO-1 Hyperion hyperspectral data. International Journal of Remote Sensing, 2013, 34(2): 712-728. DOI:10.1080/01431161.2012.713142 |
| [25] |
Thenkabail P S, Smith R B, De Pauw E. Hyperspectral vegetation indices and their relationships with agricultural crop characteristics. Remote Sensing of Environment, 2000, 71(2): 158-182. DOI:10.1016/S0034-4257(99)00067-X |
| [26] |
Lucas K L, Carter G A. The use of hyperspectral remote sensing to assess vascular plant species richness on Horn Island, Mississippi. Remote Sensing of Environment, 2008, 112(10): 3908-3915. DOI:10.1016/j.rse.2008.06.009 |
| [27] |
Yu K, Leufen G, Hunsche M, Noga G, Chen X P, Bareth G. Investigation of leaf diseases and estimation of chlorophyll concentration in seven barley varieties using fluorescence and hyperspectral indices. Remote Sensing, 2013, 6(1): 64-86. DOI:10.3390/rs6010064 |
| [28] |
Peón J, Recondo C, Fernández S, Calleja J F, de Miguel E, Carretero L. Prediction of topsoil organic carbon using airborne and satellite hyperspectral imagery. Remote Sensing, 2017, 9(12): 1211. DOI:10.3390/rs9121211 |
| [29] |
Jones T G, Coops N C, Gergel S E, Sharma T. Employing measures of heterogeneity and an object-based approach to extrapolate tree species distribution data. Diversity, 2014, 6(3): 396-414. DOI:10.3390/d6030396 |
| [30] |
Möckel T, Dalmayne J, Schmid B C, Prentice H C, Hall K. Airborne hyperspectral data predict fine-scale plant species diversity in grazed dry grasslands. Remote Sensing, 2016, 8(2): 133. DOI:10.3390/rs8020133 |
| [31] |
Rocchini D, Delucchi D L, Palmer M W. Disentangling the role of remotely sensed spectral heterogeneity as a proxy for North American plant species richness. Community Ecology, 2014, 15(1): 37-43. DOI:10.1556/ComEc.15.2014.1.4 |
| [32] |
Gholizadeh H, Gamon J A, Zygielbaum A I, Wang R, Schweiger A K, Cavender-Bares J. Remote sensing of biodiversity:soil correction and data dimension reduction methods improve assessment of α-diversity (species richness) in prairie ecosystems. Remote Sensing of Environment, 2018, 206: 240-253. DOI:10.1016/j.rse.2017.12.014 |
| [33] |
Leutner B. F.. Modelling forest α-diversity and floristic composition-on the added value of Lidar plus hyperspectral remote sensing. Remote Sensing, 2012, 4(9): 2818-2845. DOI:10.3390/rs4092818 |
| [34] |
彭羽, 卿凤婷, 米凯, 薛达元. 生物多样性不同层次尺度效应及其耦合关系研究进展. 生态学报, 2015, 35(2): 577-583. |
| [35] |
Rocchini D, He K S, Oldeland J, Wesuls D, Neteler M. Spectral variation versus species β-diversity at different spatial scales:a test in African highland savannas. Journal of Environmental Monitoring, 2010, 12(4): 825-831. DOI:10.1039/b921835a |
| [36] |
Rocchini D, McGlinn D, Ricotta C, Neteler M, Wohlgemuth T. Landscape complexity and spatial scale influence the relationship between remotely sensed spectral diversity and survey-based plant species richness. Journal of Vegetation Science, 2011, 22(4): 688-698. DOI:10.1111/jvs.2011.22.issue-4 |
 2019, Vol. 39
2019, Vol. 39