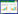文章信息
- 王太, 杜岩岩, 杨濯羽, 张艳萍, 娄忠玉, 焦文龙.
- WANG Tai, DU Yanyan, YANG Zhuoyu, ZHANG Yanping, LOU Zhongyu, JIAO Wenlong.
- 基于线粒体控制区的嘉陵裸裂尻鱼种群遗传结构分析
- Population genetic structure of Schizopygopsis kialingensis inferred from mitochondrial D-loop sequences
- 生态学报. 2017, 37(22): 7741-7749
- Acta Ecologica Sinica. 2017, 37(22): 7741-7749
- http://dx.doi.org/10.5846/stxb201609141862
-
文章历史
- 收稿日期: 2016-09-14
- 修订日期: 2016-12-07
青藏高原作为全球遗传多样性的重要区域, 通过对鱼类生物地理学的研究在探讨青藏高原的地质变迁和水系演化方面具有现实意义。裂腹鱼亚科(Schizothracinae)鱼类是青藏高原鱼类的重要类群之一, 它们对高原环境表现出了很强的适应性, 成为在高原上分布范围最广、分布海拔最高的类群, 最高可分布到海拔5600 m, 是鲤科鱼类中适应高寒环境的一个特化类群[1], 它和鳅科高原鳅属鱼类共同构成了青藏高原鱼类区系的主体。
嘉陵裸裂尻鱼(Schizopygopsis kialingensis)属裂腹鱼亚科裸裂尻鱼属(Schizopygopsis), 主要分布在嘉陵江上游支流白龙江, 以底栖无脊椎动物为食。繁殖期在每年6月河流解冻之后, 产卵场位于水深1米以内的缓流处。近年来, 随着自然地理气候的变迁、人类活动的加剧, 涉水工程的建设、水体污染和长期过度捕捞等对嘉陵裸裂尻鱼“三场”及栖息地、洄游通道等造成了严重影响, 再加上其性成熟晚, 分布区域狭窄, 生长速度缓慢等自身因素限制了种群的发展[2], 种群数量急剧减少, 栖息地呈现出一种破碎化状态, 已经被列入甘肃省重点保护野生动物名录。目前, 嘉陵裸裂尻鱼的相关研究主要集中在形态学[3]、资源分布[4]和分类地位[5]方面, 遗传多样性评估方面的研究还未见有报道。
线粒体DNA具有结构简单、母系遗传、几乎不发生重组和进化速度较快的特点, 是研究动物群体遗传学的理想分子标记[6][7]。线粒体DNA控制区序列为线粒体非编码区序列, 进化速度较快, 是分析鱼类种群遗传结构的优良标记。赵凯等[8]采用D-loop和cytb联合序列分析了的祁连山裸鲤的生物地理学过程, 认为在0.05—0.37Ma前祁连裸鲤种群由石羊河沿着青藏高原东北边缘由东向西扩张。孟玮等[9]采用D-loop序列片段分析了塔里木裂腹鱼的遗传结构, 认为塔里木河的5个群体间遗传分化显著。本文通过线粒体控制区序列变异分析了嘉陵裸裂尻鱼的种群遗传变化, 旨在为嘉陵裸裂尻鱼资源的管理与保护提供理论依据。
1 材料与方法 1.1 样品采集和DNA提取实验采集嘉陵裸裂尻鱼6个野生群体(见图 1), 共147尾个体, 样品数量、采集地见表 1。取右侧胸鳍2 g置于无水乙醇中固定后带回实验室进行分析, 采用酚/氯仿法提取基因组DNA。
| 种群 Populations | 地理坐标 Geographic location | 样本数 Samplesize (N) | 单倍型数 Number of haplotypes(Nh) | 多态位点 Number of polymorphic sites (Np) | 单倍型多样性 Haplotype diversity (Hd) | 核苷酸多样性 Nucleotide diversity(π) |
| 迭部益哇乡 | N 34°09.532′ E 103°10.427′ H 2616 m | 28 | 4 | 3 | 0.426±0.107 | 0.00084±0.00026 |
| 迭部腊子乡 | N 34°11.278′ E 103°52.456′ H 2210 m | 30 | 4 | 4 | 0.671±0.053 | 0.00144±0.00025 |
| 舟曲城关镇 | N 33°54.539′ E 104°00.935′ H 1592 m | 19 | 4 | 6 | 0.713±0.074 | 0.00319±0.00047 |
| 舟曲拱坝乡 | N 33°29.430′ E 104°32.525′ H 1406 m | 17 | 4 | 5 | 0.618±0.106 | 0.00204±0.00041 |
| 宕昌南河镇 | N 33°57.647′ E 104°25.316H 1777 m | 25 | 3 | 5 | 0.547±0.054 | 0.00305±0.00025 |
| 渭源锹峪乡 | N 35°00.890′ E 104°12.790H 2231 m | 28 | 2 | 1 | 0.071±0.065 | 0.00010±0.00009 |
| 合计Total | 147 | 14 | 17 | 0.810±0.018 | 0.00698±0.00044 |
|
| 图 1 嘉陵裸裂尻鱼采样点分布图 Fig. 1 Sketch map of sampling sites of Schizopygopsis kialingensis |
扩增mtDNA控制区序列的引物[8]: GEDL200: 5′-CACCCCTGGCTCCCAAAGCCAG-3′; GEDH860: 5′-AGGGGTTTGACAAGAATAACAGGA-3′, 由上海美吉生物医药科技有限公司合成。PCR反应体系为25 μL, 其中包括1 U TaqDNA聚合酶(TaKaRa), 2.5 μL 10× Taq buffer(TaKaRa, 含Mg2+), 两条引物(10 mmol/L)各1 μL, 0.5 μL dNTPs(2.5 mmol/L)1 μL DNA模板, 其余双蒸水补足。PCR反应程序为:94℃预变性5 min;94℃变性30 s, 55℃退火30 s, 72℃延伸60 s, 共30个循环;反应结束后在72℃再延伸10 min。PCR产物经琼脂糖凝胶电泳检测后送上海美吉生物工程公司纯化并双向测序, 测序引物为扩增引物。
1.3 数据分析用DnaSP 5.0软件[10]计算单倍型数、多态位点、单倍型多样性、核苷酸多样性和错配分布(Mismatch)分析(自举值为500)等;采用Arlequin 3.0软件[11]计算两两群体间的遗传分化指数FST值(重复次数10000), 用公式Nm=[(1/Fst)-1]/2计算基因流Nm[12], 并进行AMOVA分析和Tajima′s D、Fu′s Fs中性检验(重复次数10000), 获得SSD值和Raggedness值。采用矩阵相关分析(Mantel test)检验6个种群之间的遗传分化是否与地理距离相关, 检验矩阵为种群两两之间线性化的遗传分化系数Fst矩阵及采样点地理距离矩阵, 地理距离为两个GPS位点间的直线距离, 并进行ln转换, 计算相关系数r。用Mega 7.0软件[13]统计碱基组成, 并基于Kimura2-parameter模型以黄河裸裂尻鱼、极边扁咽齿鱼和齐口裂腹鱼为外群, 用邻接法(Neighbor-joining, NJ)构建分子系统树, 系统树中节点的自举置信水平应用自引导估计, 循环次数为1000次。Network 4.6.1.1软件[14]中的median-joining算法绘制单倍型最小网络关系图, 用以展示单倍型进化关系。
2 结果 2.1 序列变异及群体遗传多样性对嘉陵裸裂尻鱼的线粒体控制区序列进行比对排序后, 得到706 bp的同源序列。147尾个体共包含17个核苷酸变异位点, 其中简约信息位点16个, 单突变位点1个。碱基组成分析显示, A、T、C和G碱基平均含量分别为31.6%、31.6%、21.9%和14.9%, 其中A+T含量(63.2%)明显高于G+C含量(36.8%), 表现出明显的AT偏好和反G偏倚, 与其他脊椎动物线粒体控制区核苷酸的组成特点相一致[15]。嘉陵裸裂尻鱼群体的遗传多样性信息见表 1, 由表 1中可以看出147尾个体共检测出14个单倍型(KY092436—KY092449), 显示出较高的单倍型多样性和较低的核苷酸多样性(Hd=0.810±0.018;π=0.00698±0.00044), 其中采样点舟曲城关镇的嘉陵裸裂尻鱼单倍型多样性最高, 为0.713±0.074;采样点渭源锹峪乡的单倍型多样性最低, 为0.071±0.065。
2.2 群体遗传分化通过计算两两群体间Fst值得到的结果显示(表 2), 宕昌群体(TC)和舟曲群体(ZQ)之间差异不显著(P>0.01), 其余两两群体之间Fst值统计检验均显著。其中, 迭部群体与渭源群体间的Fst值最大(0.75132), 宕昌群体与舟曲群体间的Fst值最小(0.03368)。嘉陵裸裂尻鱼群体进行分子变异(AMOVA)分析结果显示(表 3), 44.29%的分子差异位于群体间, 55.71%的分子差异位于群体内, 群体间遗传分化极显著(Fst=0.44292;P =0.000)。按照采样点所处水系将6个地理种群分为2组, 渭源种群为1组, 其余5个种群为1组, 进行AMOVA分析显示, 38.48%的分子差异位于组间, 19.25%的分子差异位于组内群体间, 42.27%的分子差异位于群体内, 组间的遗传分化不显著(Fst=0.38481;P=0.155), 组内群体间和群体内的遗传分化极显著。根据遗传分化指数计算出种群间的基因流Nm为0.08275—7.17280, 其中只有舟曲群体与迭部群体、拱坝群体和宕昌群体, 宕昌与迭部群体之间的基因流大于1(表 4)。
| 群体属地Population geography | 迭部益哇乡 | 迭部腊子乡 | 舟曲城关镇 | 舟曲拱坝乡 | 宕昌南河镇 | 渭源锹峪乡 |
| 迭部益哇乡 | 0.000 | 0.009 | 0.000 | 0.000 | 0.000 | |
| 迭部腊子乡 | 0.44927 | 0.000 | 0.000 | 0.000 | 0.000 | |
| 舟曲城关镇 | 0.10357 | 0.23384 | 0.009 | 0.162 | 0.000 | |
| 舟曲拱坝乡 | 0.34506 | 0.35236 | 0.12884 | 0.000 | 0.000 | |
| 宕昌南河镇 | 0.16294 | 0.24908 | 0.03368 | 0.33274 | 0.000 | |
| 渭源锹峪乡 | 0.75132 | 0.62172 | 0.65131 | 0.70673 | 0.70106 |
| 变异来源 Source of variation | 自由度 df | 平方和 Sum of squares | 变异组分 Variance components | 变异百分比(%) Percentage of variation | F | P |
| 群体间Among populations | 6 | 24.773 | 0.19380Va | 44.29 | 0.44292 | 0.000 |
| 群体内Within populations | 141 | 34.370 | 0.24376Vb | 55.71 | ||
| 总体Total | 146 | 59.143 | 0.43756 | 100.00 | ||
| 分组间Among groups | 1 | 13.347 | 0.22192Va | 38.48 | 0.38481 | 0.155 |
| 组内群体间 Among populations within groups | 4 | 11.426 | 0.11103Vb | 19.25 | 0.31294 | 0.000 |
| 群体内Within populations | 141 | 34.370 | 0.24376Vc | 42.27 | 0.57733 | 0.000 |
| 总体Total | 146 | 59.143 | 0.57671 | 100.00 |
| 群体属地Population geography | 迭部益哇乡 | 迭部腊子乡 | 舟曲城关镇 | 舟曲拱坝乡 | 宕昌南河镇 | 渭源锹峪乡 |
| 迭部益哇乡 | 67.6 | 110.6 | 143.8 | 115.8 | 144.2 | |
| 迭部腊子乡 | 0.30646 | 53.0 | 84.4 | 47.9 | 105.3 | |
| 舟曲城关镇 | 2.16383 | 0.81911 | 34.8 | 27.5 | 136.3 | |
| 舟曲拱坝乡 | 0.47451 | 0.45950 | 1.69039 | 59.9 | 145.1 | |
| 宕昌南河镇 | 1.28431 | 0.75369 | 7.17280 | 0.50134 | 117.7 | |
| 渭源锹峪乡 | 0.08275 | 0.15211 | 0.13384 | 0.10374 | 0.10660 |
Mantel test检验表明, 嘉陵裸裂尻鱼种群之间遗传分化程度与地理距离存在显著的相关(r=0.6952, P < 0.0500), 说明距离隔离模型可以有效的解释嘉陵裸裂尻鱼种群目前遗传格局的形成。但是也有情况例外, 迭部嗌哇乡(DB)群体与迭部腊子乡(LZ)群体的地理距离较近, 但群体之间的遗传差异较大。
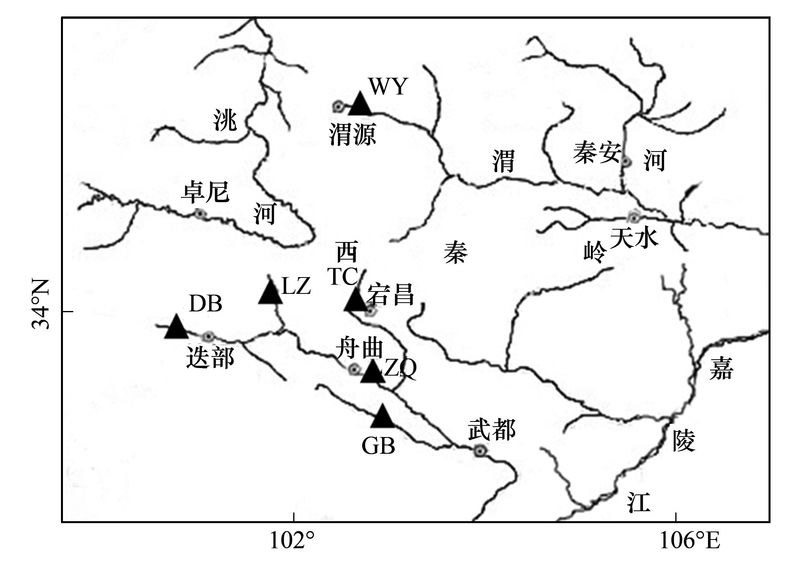
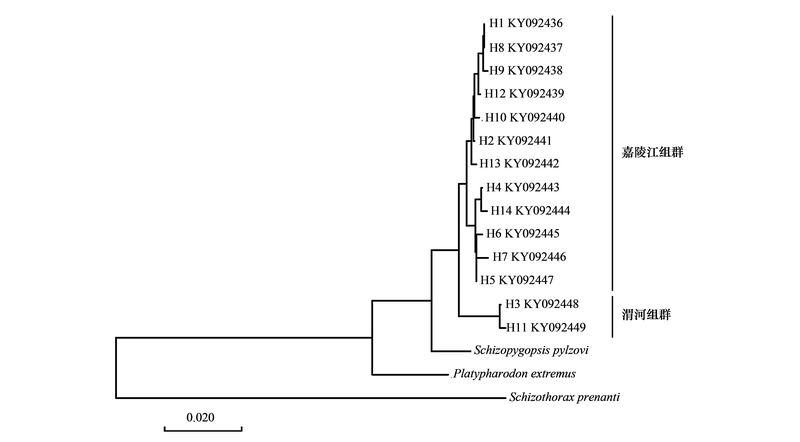
2.3 分子系统发育分析以黄河裸裂尻鱼(Schizopygopsis pylzovi)、极边扁咽齿鱼(Platypharodon extremus)和齐口裂腹鱼(Schizothorax prenanti)为外类群, 应用邻接法(NJ)构建嘉陵裸裂尻鱼14个线粒体控制区序列单倍型(KY092436-KY092449)之间的系统发育树(图 2)。结果显示, 所有的嘉陵裸裂尻鱼样本聚为一个单系, 并清楚的识别出了对应于水系的两个谱系群体, 即嘉陵江组群和渭河组群。依据Network软件以中接法(Median-joining)构建的单倍型进化网络关系图显示(图 3、图 4), 大部分单倍型之间经过一步突变, H1做为嘉陵裸裂尻鱼在嘉陵江水系的祖先单倍型, H3做为渭河水系的祖先单倍型, 并且两个谱系群体之间没有共享单倍型。单倍型进化网络图进一步支持了系统发育树的分析, 嘉陵裸裂尻鱼6个地理群体的单倍型按照嘉陵江水系和渭河水系形成两个大的类群。
|
| 图 2 基于D-loop区序列构建嘉陵裸裂尻鱼的NJ树, 节点处的数字为1000次bootstrap检验的支持率 Fig. 2 A neighbor-joining tree of part of Schizopygopsis kialingensisin resulted from D-loop sequence. Numbers at nodes are percent recovery in bootstrap analysis (1000 replicates) |
|
| 图 3 嘉陵裸裂尻鱼单倍型最小进化网络关系 Fig. 3 Haplotypes minimum spanning network for Schizopygopsis kialingensis |
|
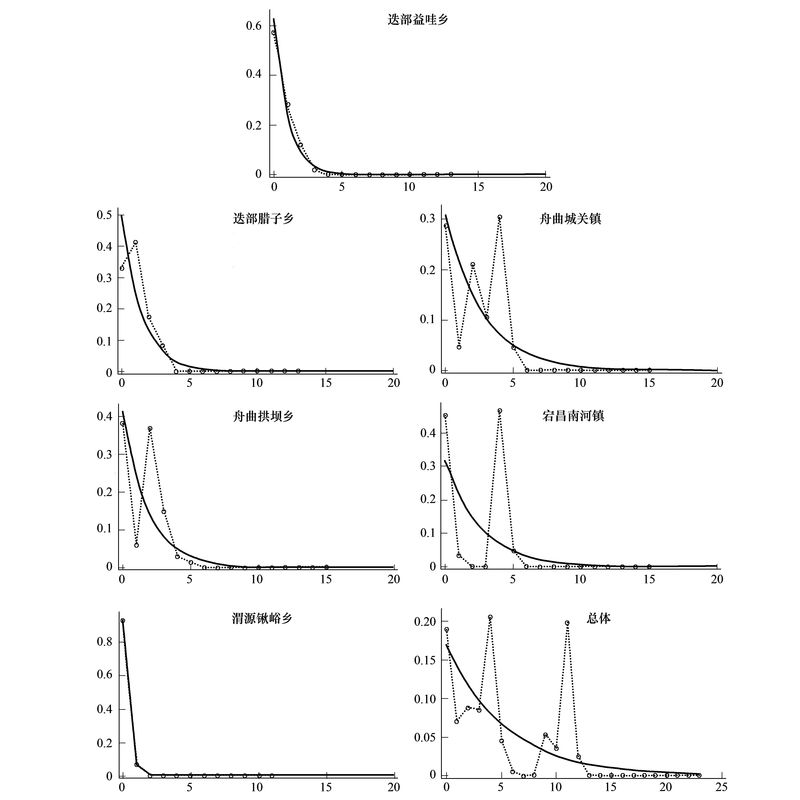
| 图 4 不同地理群体嘉陵裸裂尻鱼的核苷酸错配分布曲线 Fig. 4 The result ofmismatchdistributionpairwise haplotypes geneticdifferences forSchizopygopsis kialingensis (实线为模拟的不配对分布, 虚线为观察到的不配对分布A:DB, B:LZ, C:ZQ, D:GB, E:TC, F:WY, G:整体) |
对所扩增的147尾嘉陵裸裂尻鱼的线粒体控制区序列进行核苷酸错配分布分析、Tajima′s D和Fu′s FS中性检验来分析种群历史动态。结果显示, 迭部益哇乡群体(DB)、迭部腊子乡群体(LZ)和渭源群体(WY)的核苷酸错配分布曲线呈明显的单峰型(图 3), 但Tajima′s D和Fu′s FS检验均不显著(P>0.05), 表明这3个群体未显著偏离种群扩张模型。其余3个群体的核苷酸错配分布都呈现多峰分布, 且Tajima′s D和Fu′s FS检验均不显著, 未检测到种群扩张。将所有个体做为一个整体进行核苷酸错配分布, 呈现出多峰分布, Tajima′s D和Fu′s FS检验符合中性进化的假设, 说明嘉陵裸裂尻鱼群体在近期历史上群体大小保持稳定, 未出现显著的种群扩张。
3 讨论 3.1 遗传多样性分析种群遗传多样性是生物多样性的重要组成部分, 体现了物种对不同环境的适应能力的潜力, 遗传多样性水平、形成机制和分布格局可揭示物种起源与进化历程, 是物种进化潜能的保证, 也是物种保护工作的基础。单倍型多样性(h)和核苷酸多样性(π)是衡量一个物种群体变异程度的重要指标[16]。嘉陵裸裂尻鱼的单倍型多样性和核苷酸多样性(Hd=0.810±0.018;π=0.00698±0.00044)整体低于同亚科鱼类齐口裂腹鱼[17](Schizothorax prenanti)(Hd=0.98;π=0.019)和极边扁咽齿鱼[18](Platypharodo nextremus)(Hd=0.995;π=0.0129), 与厚唇裸重唇鱼[19](Gymnodiptychus pachycheilus)(Hd=0.844;π=0.0054)和黄河裸裂尻鱼(Schizopygopsis pylzovi)[20](Hd=0.79;π=0.0027)相当。嘉陵裸裂尻鱼6个地理群体的单倍型多样性呈现出有些群体高、有些群体低的现象, 而核苷酸多样性均较低, 甚至在迭部益哇乡(DB)和渭源(WY)群体的核苷酸多样性低于0.001。这种在不同群体的遗传多样性存在显著的地理差异的现象在淡水鱼类中被大量观察到[21, 22], 嘉陵裸裂尻鱼较低的核苷酸多样性, 可能与青藏高原第四纪以来经历了数次地质变迁和气候的改变, 使种群受到“瓶颈效应”的打击, 后部分种群又受到“创立者效应”的作用, 导致部分种群的遗传多样性贫乏。对于渭源种群(WY)来说, 长期的地理隔离也可能是造成遗传多样性水平极低的主要原因之一, 有效群体数量小引起的遗传漂变导致大量的遗传变异丢失。
| 群体属地 Population | Tajima氏D检验值 Tajima′s D | P值 P | Fu氏FS检验值 Fu′s FS | P值 P |
| 迭部益哇乡 | -0.56351 | 0.335 | -0.89198 | 0.225 |
| 迭部腊子乡 | 0.00462 | 0.568 | 0.43345 | 0.611 |
| 舟曲城关镇 | 0.97533 | 0.866 | 2.04215 | 0.863 |
| 舟曲拱坝乡 | -0.11454 | 0.475 | 0.63814 | 0.662 |
| 宕昌南河镇 | 1.75057 | 0.960 | 3.87064 | 0.953 |
| 渭源锹峪乡 | -1.15142 | 0.157 | -1.15451 | 0.056 |
| 总体Total | 0.15017 | 0.560 | 0.82298 | 0.561 |
有研究得出地理隔离或同一水域中存在不同的栖息环境限制了基因交流, 是鱼类出现种群分化的重要原因[23], 遗传分化和基因流是评价种群遗传结构的重要指标。渭源群体(WY)采自黄河流域渭河, 而其他5个地理群体为嘉陵江水系, 受地理隔离影响渭源群体与其他群体之间的Fst(0.62172—0.75132)远大于0.25, 说明嘉陵裸裂尻鱼渭源群体与其他种群之间遗传分化较大, 这与基因流Nm分析结果一致, 群体间基因流贫乏。位于嘉陵江5个水系连通地理群体之间Fst(0.03368-0.44927)较小, 相邻地理种群之间有基因交流(Nm>1), 可以抑制遗传漂变引起的遗传分化[24], 这可能与嘉陵裸裂尻鱼具有短距离洄游这一习性有关。Mantel test检验表明, 嘉陵裸裂尻鱼种群之间遗传分化程度与地理距离存在显著相关, 说明距离隔离模型可以有效解释种群目前遗传格局的形成。但是也有迭部嗌哇乡(DB)群体与迭部腊子乡(LZ)群体的地理距离较近, 但群体之间的遗传差异较大的现象, 说明地理隔离并非唯一影响嘉陵裸裂尻鱼遗传格局的因素, 推测可能是由于白龙江上游大量梯级电站的建设, 使河流失去连通性, 限制了嘉陵裸裂尻鱼的扩散, 导致两者之间的遗传分化加大。
分子变异分析(AMOVA)显示, 55.71%的变异来自于种群内, 44.29%的变异来自于种群间。种群间的遗传变异较大, 这与单倍型系统进化树(图 1)和单倍型进化网络关系图(图 2)相一致, 渭源种群单倍型独自聚为一支, 且渭源种群与其他种群之间没有共享单倍型。按照水系将嘉陵裸裂尻鱼种群划分为2组进行AMOVA分析显示, 嘉陵江水系5个地理种群间的遗传变异将为19.25%, 大部分的遗传变异来自于种群内的个体之间(42.27%), 这同样与单倍型进化树和单倍型进化网络关系图相一致, 嘉陵江水系5个地理种群间没有显著的地理种群结构。基因流估计显示各群体间的基因流水平较高, 遗传交流较频繁, 这与它们缺乏种群结构是一致的。综合这几方面研究结果都显示, 嘉陵江水系的嘉陵裸裂尻鱼没有显著的种群分化。
3.3 种群历史动态与保护嘉陵裸裂尻鱼整体、舟曲城关镇种群(ZQ)、舟曲拱坝乡种群(GB)和宕昌种群(TC)的核苷酸错配分析均呈多峰分布, Tajima′s D和Fu′s FS检验符合中性进化的假设。迭部益哇乡群体(DB)、迭部腊子乡群体(LZ)和渭源群体(WY)的核苷酸错配分布曲线呈明显的单峰型(图 3), 但Tajima′s D和Fu′s FS检验都不显著(P>0.05), 表明嘉陵裸裂尻鱼群体在近期历史上群体大小保持稳定, 未出现显著的种群扩张。在很多鱼类中都发现了单倍型多样性高而核苷酸多样性低的现象[25, 26], 被认为是小的有效群体经过一段时间的稳定后发生了扩张, 快速的种群扩张有利于提高对新突变的保持能力而导致核苷酸多样性低[27]。而嘉陵裸裂尻鱼这种单倍型多样性偏低和核苷酸多样性低的状况, 推测在第四纪冰期期间由于种群受到“瓶颈效应”打击之后, 未发生种群扩张事件, 导致遗传多样性偏低。这可能与其自身的性成熟年龄(5龄)晚, 个体小(调查最大个体全长18cm)导致的怀卵量低等生物学特性有关[1]。
鱼类种群遗传结构和谱系系统地理格局的研究结果能够为物种保护策略及渔业管理措施的制定提供科学依据[28]。嘉陵裸裂尻鱼遗传多样性总体较为丰富, 但各个地理群体内的多样性比较贫乏。受自然环境变化和涉水工程的建设等因素的影响, 可能导致嘉陵裸裂尻鱼的遗传多样性继续降低, 对复杂环境的适应能力降低, 从而进一步威胁到嘉陵裸裂尻鱼的生存。虽然目前已经建立了一些保护措施, 如建立的白龙江特有鱼类国家级水产种质资源保护区、岷江宕昌段特有鱼类国家级水产种质资源保护区和白龙江舟曲段特有鱼类省级水产种质资源保护区, 并在嘉陵江采取人工增殖放流措施。但这些措施对保护嘉陵裸裂尻鱼可能并不完善。物种的保护必须考虑到自身的遗传特性, 否则保护将可能是不成功的[29]。本研究结果表明, 嘉陵裸裂尻鱼的核苷酸多样性较低, 提示对其遗传多样性的保护是非常迫切的。根据进化上具关键意义的单元ESU(Evolutionarily Significant Unit)设置的原则[30], 我们可以将嘉陵江水系的嘉陵裸裂尻鱼种群当作一个保护管理单元进行保护, 嘉陵裸裂尻鱼渭河种群属高度分化的单倍型类群, 且遗传多样性极低, 需当作一个保护管理单元进行优先保护。
| [1] | 武云飞, 吴翠珍. 青藏高原鱼类. 成都:四川科技出版社, 1992: 463–465. |
| [2] | 武云飞. 中国裂腹鱼亚科鱼类的系统分类研究. 高原生物学集刊, 1984(3): 119–140. |
| [3] | 伍献文. 中国鲤科鱼类志. 上海:上海科学技术出版社, 1964: 137–197. |
| [4] | 王香亭. 甘肃省脊椎动物志. 兰州:甘肃省科学技术出版社, 1991: 106–107. |
| [5] | 何德奎, 陈毅锋, 陈宜瑜, 陈自明. 特化等级裂腹鱼类的分子系统发育与青藏高原隆起. 科学通报, 2003, 48(22): 2354–2362. DOI:10.3321/j.issn:0023-074X.2003.22.011 |
| [6] | Avise J C, Arnold J, Ball R M, Bermingham E, Lamb T, Neigel J E, Reeb C A, Saunders N C. Intraspecific phylogeography:the mitochondrial DNA bridge between population genetics and systematics. Annual Review of Ecology and Systematics, 1987, 18(1): 489–522. DOI:10.1146/annurev.es.18.110187.002421 |
| [7] | Sbisà E, Tanzariello F, Reyes A, Pesole G, Saccone C. Mammalian mitochondrial D-loop region structure analysis:identification of new conserved sequences and their functional and evolutionary implications. Gene, 1997, 205(1/2): 125–140. |
| [8] | Zhao K, Duan Z Y, Peng Z G, Gan X N, Zhang R Y, He S P. Phylogeography of the endemic Gymnocyprischilianensis (Cyprinidae):sequential westward colonization followed by allopatric evolution in response to cyclical Pleistocene glaciations on the Tibetan Plateau. Molecular Phylogenetics and Evolution, 2011, 59(2): 303–310. DOI:10.1016/j.ympev.2011.02.001 |
| [9] | 孟玮, 郭焱, 海萨, 杨天燕, 马燕武, 谢春刚. 塔里木裂腹鱼群体遗传结构及遗传多样性分析. 水生生物学报, 2012, 36(5): 851–857. |
| [10] | Librado P, Rozas J. DnaSP v5:a software for comprehensiveanalysis of DNA polymorphism data. Bioinformatics, 2009, 25(11): 1451–1452. DOI:10.1093/bioinformatics/btp187 |
| [11] | Excoffier L, Laval G, Schneider S. Arlequin (version. 3.0):Anintegrated software package for population genetics dataanalysis. Evolutionary Bioinformatics Online, 2007, 1: 47–50. |
| [12] | Hudson R R, Slatkin M, Maddison W P. Estimation oflevelsof gene flow from DNA sequence data. Genetics, 1992, 132(2): 583–589. |
| [13] | KumarS, Stecher G, TamuraK. MEGA7:molecularevolutionary genetics analysis version 7.0 for bigger datasets. Molecular Biology and Evolution, 2016, 33(7): 1870–1874. DOI:10.1093/molbev/msw054 |
| [14] | Bandelt H J, Forsrer P, Röhl A. Median-joining networks for inferring intraspecific phylogenies. Molecular Biology and Evolution, 1999, 16(1): 37–48. DOI:10.1093/oxfordjournals.molbev.a026036 |
| [15] | Schneider S, Excoffier L. Estimation of past demographic parameters from the distribution of pairwise differences when the mutation rates vary among sites:application to human mitochondrial DNA. Genetics, 1999, 152(3): 1079–1089. |
| [16] | NeigelJE, AviseJC. Application of a random walk model to geographic distributions of animal mitochondrial DNA variation. Genetics, 1993, 135(4): 1209–1220. |
| [17] | Song Z B, Song J, Yue B S. Population genetic diversity of Prenant's schizothoracin, Schizothora xprenanti, inferred from the mitochondrial DNA control region. Environmental Biology of Fishes, 2008, 81(3): 247–252. DOI:10.1007/s10641-007-9197-6 |
| [18] | 苏军虎, 张艳萍, 娄忠玉, 刘焕章, 焦文龙, 魏彦明. 黄河上游极边扁咽齿鱼种群遗传结构. 动物分类学报, 2012, 37(1): 29–35. |
| [19] | 苏军虎, 张艳萍, 娄忠玉, 刘焕章, 焦文龙, 杨建宝, 魏彦明. 基于线粒体控制区序列的黄河上游厚唇裸重唇鱼种群遗传结构. 生态学报, 2012, 32(13): 4191–4198. |
| [20] | 赵凯, 杨公社, 李俊兵, 何舜平. 黄河裸裂尻鱼群体遗传结构和Cyt b序列变异. 水生生物学报, 2006, 30(2): 129–133. |
| [21] | BillingtonN, HebertPDN. Mitochondrial DNA diversity in fishes and its implications for introductions. Canadian Journal of Fisheries and Aquatic Sciences, 1991, 48(S1): 80–94. DOI:10.1139/f91-306 |
| [22] | 肖武汉, 张亚平. 鱼类线粒体DNA的遗传与进化. 水生生物学报, 2000, 24(4): 384–391. |
| [23] | Wright S. Evolution in Mendelian populations. Genetics, 1931, 16(2): 97–159. |
| [24] | Wright S. Evolution and the Genetics of Populations:Variability within and among Natural Population. Chicago: University of Chicago Press, 1978: 79–103. |
| [25] | Liu G X, Zhou J, Zhou D G. Mitochondrial DNA reveals low population differentiation in elongate loach, Leptobotiaelongata (Bleeker):implications for conservation. Environmental Biology of Fishes, 2012, 93(3): 393–402. DOI:10.1007/s10641-011-9927-7 |
| [26] | Yu D, Chen M, Tang Q Y, Li X J, Liu H Z. Geological events and Pliocene climate fluctuations explain the phylogeographical pattern of the cold water fish Rhynchocyprisoxycephalus (Cypriniformes:Cyprinidae) in China. BMC Evolutionary Biology, 2014, 14: 225–225. DOI:10.1186/s12862-014-0225-9 |
| [27] | Grant W A S, Bowen B W. Shallow population histories in deep evolutionary lineages of marine fishes:insights from sardines and anchovies and lessons for conservation. Journal of Heredity, 1998, 89(5): 415–426. DOI:10.1093/jhered/89.5.415 |
| [28] | Loeschcke V, Tomiuk J, Jian S K. Conservation Genetics. Basel:Birkhäuser Verlag, 1994:37-53. |
| [29] | Frankham R. Genetics and conservation biology. Comptes Rendus Biologies, 2003, 326(S1): 22–29. |
| [30] | Moritz C. Defining 'evolutionarily significant units' for conservation. Trends in Ecology & Evolution, 1994, 9(10): 373–375. |
 2017, Vol. 37
2017, Vol. 37