文章信息
- 姬洪飞, 王颖
- JI Hongfei, WANG Ying.
- 分子生物学方法在环境微生物生态学中的应用研究进展
- Advances in molecular approach applications in microbial ecology studies
- 生态学报[J]. 2016, 36(24): 8234-8243
- Acta Ecologica Sinica[J]. 2016, 36(24): 8234-8243
- http://dx.doi.org/10.5846/stxb201506041132
-
文章历史
- 收稿日期: 2015-06-04
- 网络出版日期: 2016-04-13
2. 中国科学院水利部水土保持研究所, 杨凌 712100
2. Institute of Soil and Water Conservation, Chinese Academy of Sciences and Ministry of Water Resources, Yangling 712100, China
微生物包括细菌、古菌、真菌等,是生态系统的重要组成部分,几乎存在于所有已知的环境中,在生态系统中起着非常重要的作用,如维持生态系统功能、直接参与碳、氮、硫、磷和一些金属的生物地球化学循环[1]、加快污染环境修复进程等[2]。此外,微生物在生态系统对气候变化的响应方面也可能起着重要作用[3]。基于微生物在生态系统中所发挥的重要作用,环境和生态学研究的学者对理解生态系统中微生物的动态变化兴趣渐浓。
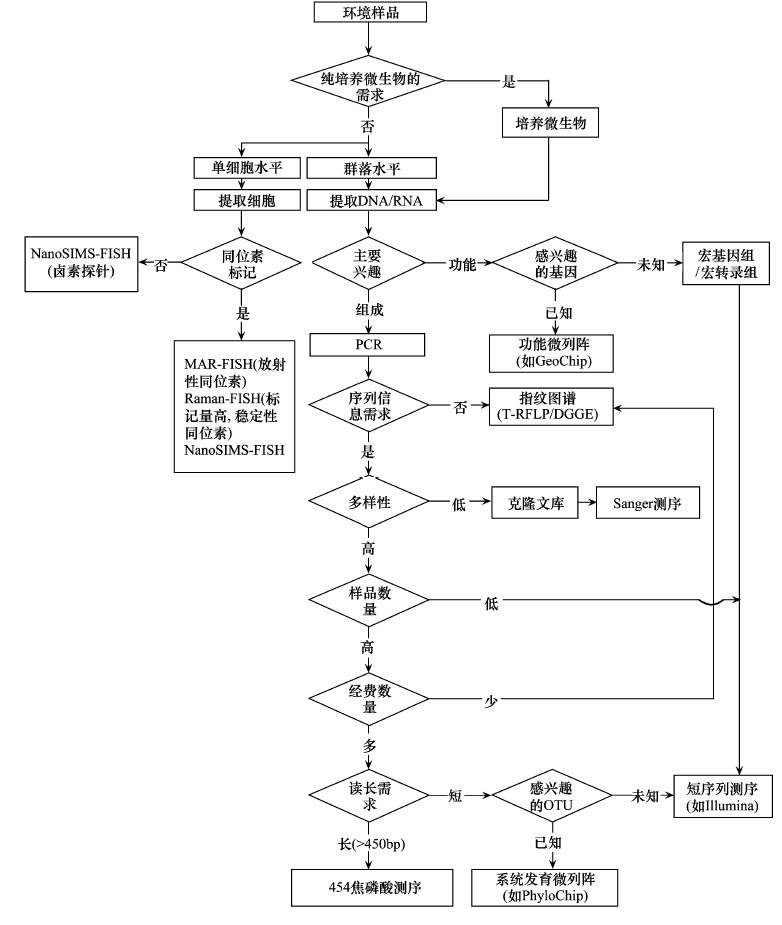
目前环境微生物生态学的研究主要集中在确定所研究的环境中有哪些微生物存在和分析微生物在所研究的环境中起什么样的作用或执行哪个生态功能[4]两个方面,此外,微生物之间以及微生物与环境因子之间的相互作用受到越来越多的关注。由于微生物个体小(平均直径约1 μm),肉眼不可见且未知,微生物的研究方法的发展加速了研究者对微生物的认知。在过去的十多年中,微生物的研究方法得到了快速发展,且商业化迅速,特别是高通量测序、宏组学和单细胞水平研究方法(如纳米二次离子质谱与荧光原位杂交相结合的方法)等(图 1)[4]技术手段使微生物研究的难度和成本都得到了显著改善。随着研究方法的发展,微生物在生态系统中的作用可以被更好的挖掘出来。然而,快速发展的微生物研究方法,以及之前已有的研究方法,给不熟悉这些技术的研究者在方法的选择上带来了困惑。因此,本文将从微生物的多样性和功能分析两个方面对相应的研究方法进行阐述,并以1—2篇文献为例具体介绍相关方法在微生物生态学研究中的应用,以期为研究者选择合适的研究方法进行环境微生物的研究提供参考。
1 环境微生物的多样性分析在很多微生物生态学的研究中,首先是分析所研究的环境中有哪些微生物,以及微生物的多样性和分布情况。微生物研究的传统方法首先是获取环境样品,然后对其进行分离培养得到微生物的单菌株。然而,很多微生物难以通过现有的培养方法得到[5],使微生物的多样性分析受到很大限制。与传统分离培养方法不同,现代分子生物学技术直接以从环境样品中提取的核酸(DNA和RNA)为分析对象,以特定的核酸片段为生物标志物,理解该环境中微生物的组成和多样性,即将所提取的DNA进行目标片段的PCR扩增,得到的PCR产物通过多种技术来鉴定其组成的多态性,如群落结构分析的指纹图谱、微列阵(Microarray)和高通量测序等。微生物多样性分析的研究中,PCR扩增的目标片段可以选择微生物的一段相对保守而又足以区分不同类群的基因区域,如细菌和古菌常用的16S rRNA[6]、真菌的18S rRNA或ITS区[7]等;同时,一些微生物的功能基因也可用于分析功能微生物的多样性,如氨氧化细菌的氨单加氧酶amoA基因(氨单加氧酶,催化硝化作用的第一步)。得到目标基因的PCR产物后,可根据需求选择下面的方法进行微生物的多样性分析(图 1)。
变性梯度凝胶电泳(DGGE)和末端限制性片段长度多态性(T-RFLP)均是通过电泳对PCR产物进行分型,从而分析微生物的多样性和相对丰富度,在分析环境中优势微生物群落(相对丰富度>1%的微生物种群)[8]的变化方面具有较多的应用(表 1)。DGGE和T-RFLP是应用于环境微生物多样性分析中较早的分子生物学方法,不仅应用方便,分辨率高,且价格低廉。DGGE主要用于分析低于500 bp的片段;T-RFLP对低于500 bp的酶切片段分型精度较高,而高于500 bp的酶切片段分型精度则不足。DGGE条带可通过切胶回收、构建克隆文库和测序来了解感兴趣的条带的系统发育信息或物种信息,而对于复杂的微生物群落组成(如土壤细菌的群落组成)的分析,T-RFLP的分辨率高于DGGE。
| 应用方法 Method | 分辨率 Resolution | 联用技术 Techniques used in combination | 样品种类 Sample types | 分析指标 Types of items analyzed | 缺点 Disadvantages |
| 变性梯度凝胶电泳/末端限制性片段长度多态性 DGGE/T-RFLP | 群落水平(相对丰富度>1%的微生物群落) | 定量PCR,克隆库 | 土壤、沉积物、水体等环境样品的DNA/RNA | 细菌、真菌、古菌以及功能菌如氨氧化菌、反硝化菌和甲烷氧化菌等的群落组成和数量 | DGGE的一个条带可能不止一个物种,对感兴趣的条带需要进行克隆测序来验证[9];T-RFLP的每个T-RF片段不一定代表一个物种, |
| 稳定同位素标记,定量PCR,克隆库 | 土壤、沉积物、水体等环境样品的DNA/RNA | 环境中活跃的细菌、真菌以及功能菌如氨氧化菌和甲烷氧化菌等的群落组成和数量 | 造成对微生物群落多样性的低估,T-RFLP在电泳过程中可能会出现一定的飘移[10],需要与克隆库相结合来确定 | ||
| 微列Microarray | 群落水平 | PhyloChip | 土壤、沉积物、水体等环境样品的DNA/RNA | 细菌、古菌以及功能菌氨氧化细菌、甲烷氧化菌和产甲烷菌的群落组成和相对丰富度 | 微列阵可以同时检测几千甚至百万条序列,但是探针的设计以及列阵中片段退火带来的偏差也可使结果不准确[11] |
| GeoChip | 土壤、沉积物、水体等环境样品的DNA/RNA | C、N循环和胁迫响应的基因的组成和丰富度 | |||
| 稳定同位素标记 | 土壤、沉积物、水体等环境样品的DNA/RNA | 环境中活跃的细菌、真菌、C、N循环和胁迫响应的基因的组成和丰富度 | |||
| 高通量测序 High-throughput sequencing | 群落水平 | 稳定同位素标记,定量PCR | 土壤、沉积物、水体等环境样品的DNA/RNA | 环境中或环境中活跃的细菌、真菌、古菌以及功能菌如氨氧化菌、反硝化菌和甲烷氧化菌等的群落组成和数量 | 454测序中,对一条序列中的同一碱基的多次重复很难做到准确定量[12];Illumina测序在测定GC含量高的序列区域时容易产生偏差[13];得到的序列多为几百个碱基或者更短,组成完 |
| 宏基因组/宏转录组 Metagenomics/ metatranscriptomics | 群落水平 | 稳定同位素标记,Illumina测序/454 焦磷酸测序 | 土壤、沉积物、水体等环境样品的DNA/RNA | 环境中整个群落或环境中活跃微生物群落的性质,包含环境微生物的种类和功能信息等 | 整的基因有一定困难;即使获得整条序列,由于很多基因的功能还不清楚,得到的序列与已知的基因或潜在功能区进行比对时,很难获得由序列到功能的准确图谱 |
| 拉曼光谱-荧光原位杂交 Raman-FISH | 单细胞水平 | 稳定同位素标记 | 纯菌、水体或水体培养物中的微生物细胞 | Raman信号检测,荧光成像,细菌,奈降解菌等 | Raman检测到光谱偏移的同位素标记需求量较高,需达到10 atom%[14];NanoSIMS不是高通量技术,且检测区域较小(1—50 μm),需要与透射电子显微镜(TEM)、扫描电子显微镜(SEM)或X射线显微镜方法等相结合来更好的选择并锁定检测目标[15]。FISH样品的自发 |
| 纳米二次离子质谱-荧光原位杂交 NanoSIMS-FISH | 单细胞水平 | CARD-FISH,HISH,稳定同位素标记 | 纯菌、沉积物、水体或水体培养物中的微生物细胞 | 元素和同位素成像(12C-,13C-,12C14N-,12C15N-,19F-),细菌、C和N循环相关的微生物等 | 荧光易影响或掩盖探针的荧光信号,可通过采用CARD-FISH来增强荧光信号[16]或采用卤素探针(与纳米二次离子质谱相结合)[17]来改善;样品的制备过程也可能会影响微生物细胞的同位素组成,如引入新的12C[18]等 |
基因微列阵用来分析样品中微生物的组成和多样性,已经用了将近20年,经历了数代的改进,常用的微列阵有系统发育芯片(PhyloChip,用于识别微生物以及微生物之间的系统发育联系,分析微生物的多样性)和功能基因芯片(GeoChip,用于研究功能基因的多样性和功能微生物的活性)。微列阵即为排满探针(用于和样品DNA进行杂交的已知短序列)的芯片,通过探针提供生物的系统发育信息或功能性质信息、或者二者均包含。当样品中的序列(纯菌、环境样品DNA或RNA的目标片段的荧光标记PCR产物或随机引物的荧光标记PCR产物)与探针进行杂交后,即可计算与探针匹配序列的相对荧光比例,从而获得微生物的多样性和相对丰富度信息。
基因微列阵的方法尤其适合识别不同时间、地点和处理之间的代表微生物或微生物群落的差别,此外,功能基因芯片还可以定量C、N、S和P循环、有机污染物降解和胁迫响应等相关的功能基因的变化。Aronson等[19]利用3代PhyloChip(16S rRNA基因微列阵,可以提供60000个左右不同OTU的信息),研究不同实验条件下松林土壤甲烷循环的相关微生物,分析了土壤中甲烷氧化菌(Methanotrophs)和产甲烷菌(Methanogens)的多样性及其与土壤中甲烷通量变化之间的联系。该方法不仅检测到了目标群落在不同时间、地点和氮水平处理之间的差异,同时发现了群落中占少数的微生物的变化,而在克隆筛选的方法或深度不够的高通量测序中,这些少数微生物的差异均可能被忽略。Cong等[20]利用GeoChip 5.0(覆盖了393个功能基因家族)分析了热带雨林土壤微生物的功能基因的组成和多样性,发现所检测到的基因类型与生物地球化学过程有关,如碳、氮和磷循环相关的功能基因,其中,碳循环相关基因包括易降解和难降解底物的相关微生物基因,在3个取样地点之间显著不同,典范对应分析和多元回归树分析均显示土壤有效氮含量与土壤微生物功能基因的组成和代谢潜力显著相关。
1.3 高通量测序Sanger测序已经用了几十年,测序质量高,序列长度达750—1000 bp。该方法无法测混合序列,需首先建立克隆文库,即将目标序列转移到寄主细胞(一般为Escherichia coli)中进行培养,形成单菌株,然后对单菌株中的目标序列进行测序,在分析微生物多样性较高的环境样品时,费时且费用高。
在过去十年中,测序方法得到了很大改进,在Sanger测序方法的基础上,用不同颜色的荧光标记四种不同的dNTP,当DNA聚合酶合成互补链时,每添加一种dNTP就会释放出相应的荧光,捕捉到的荧光信号经过特定的计算机软件处理,从而获得待测DNA的序列信息,即边合成边测序,也称为二代测序。该方法以微生物目标基因的PCR产物为样本进行测序,一个反应得到几万至几百万条序列,测序的广度和深度大大提高。目前用于环境样品中微生物多样性分析的常用方法有2种,即Roche Inc公司的454焦磷酸测序和Illumina Inc公司的MiSeq/HiSeq测序,这两种高通量测序方法通过在每个样品的引物上加标签来识别不同样品的序列,均可以同时分析多个环境样品,并得到大量序列。此外,高通量列阵(The Access Array 48.48,AA48.48,Fluidigm Corporation,South San Francisco,CA,USA)通过使用阵列芯片,可同时进行2304个PCR的扩增,将样品定量PCR和序列文库PCR在一个反应中同时完成,该PCR产物可直接用于二代测序分析,仅需5 h,与二代测序相结合,可同时得到微生物的群落组成和丰富度信息,简化二代测序样本的制备程序[21]。目前,很多研究中应用的测序方法依然是454焦磷酸测序。然而,Roche于2013年10月声明454焦磷酸测序将在2015年底停止运营,但现有的454测序仪还将继续运行。与454焦磷酸测序相比,Illumina平台测序的通量大大提高,序列长度不断改进,费用也有所降低。因此,Illumina平台测序以及其他一些测序方法(如Helicons、Pacific Biotechnology和Oxford Nanopore System的单分子测序)在今后的环境微生物研究中将会有更多的应用。
二代测序的测序广度和深度较克隆文库的方法大大提高,尤其是提高了对数量上占少数的微生物群落的覆盖,不仅能从群落水平上揭示微生物群落组成的变化,还能从更细的微生物分类水平上显示微生物群落的具体变化,在环境样品的16S rRNA、18S rRNA、真菌的ITS区和功能基因的分析中均有应用[6-7, 22-23]。寒带针叶林在全球的占地面积约为11%,占土壤有机碳储量的16%,一般认为地上部的植物残体是土壤有机碳累积的主要来源。而Clemmensen等[24]等通过分析北方森林土壤年代序列的有机碳组成发现,50%—70%的土壤有机碳储量源于根系和根系伴生微生物,且稳定同位素标记与真菌ITS区的454焦磷酸测序相结合的研究结果表明,在不同地点之间,有机碳累积差异最大的土壤剖面中,根系伴生真菌均占优势,说明根系伴生真菌在生态系统的碳动态中起重要的调节作用。Hermann-bank[21]将高通量列阵与二代测序的方法相结合,分析了肠道微生物的丰富度和组成,结果表明,不同肠道位置或腹泻情况下的肠道微生物组成和丰富度不同,腹泻与链球菌属(Streptococcus)成员的数量显著降低相关,尤其是非解乳链球菌(S. alactolyticus)。尽管目前高通量列阵与二代测序相结合的方法的应用较少,该研究依然显示了高通量列阵在定量不同样品中微生物的丰富度的同时,通过与二代测序相结合具有快速得到微生物系统发育信息的潜力。
2 环境微生物的功能性质和基因表达分析除了理解微生物的多样性和群落组成之外,很多时候还需要分析这些微生物成员的功能,如微生物的多样性与其功能性质有什么样的联系,以及微生物与其所在生态系统中的能量和养分流动是如何相互作用的等。微生物功能分析常用的方法包括稳定同位素标记与微生物DNA或RNA分析相结合的方法、功能基因微列阵(见1.2)、微生物宏基因组和宏转录组分析及单细胞水平研究方法来识别目标微生物的种类和功能的方法。
2.1 稳定同位素标记与DNA或RNA相结合将水体、土壤、沉积物以及其他生态系统中的微生物的种类和功能联系起来一直是微生物生态学研究的主要目标之一。过去二十几年的研究中,环境微生物的种类和功能研究已经得到了大量结果,而二者的直接联系,仍然是个薄弱环节[25]。稳定同位素和放射性同位素技术将不可培养的微生物种类和代谢功能相联系起来,是微生物生态学发展中的重要进步。稳定同位素探针技术(Stable isotope probing,SIP))采用稳定同位素(如13C,15N和18O等)标记的底物[26-28],使利用这些底物的微生物的标志物(DNA、RNA、蛋白质或磷脂脂肪酸(PLFA))被标记(表 2),从而从群落水平识别环境中活跃的微生物以及微生物之间的相互作用。
| 性质 Trait | 应用比较 Comparison of biomarkers′ applicability | 解析 Explanation |
| 敏感性 Sensitivity | DNA<RNA<PLFA<蛋白质 | DNA-SIP需要同位素的标记丰度较高,13C达到15%—20%,15N达到30%左右,而蛋白质标记只需1%的标记丰度;RNA的标记速度是DNA的6.5倍 |
| 培养时间 Incubation time | DNA>RNA>PLFA>蛋白质 | 培养时间与敏感性直接相关,DNA-SIP需要的培养时间最长,也更容易带来微生物交叉取食的偏差 |
| 分类学分辨率 Taxonomic resolution | DNA≈RNA>蛋白质 >PLFA | PLFA-SIP只区分较大的分类群,DNA或RNA-SIP可提供属水平或者更低水平的分类信息。与16S rRNA相比,蛋白序列的数据库现有信息受到很大程度的限制 |
| 代谢活性预测 Indication of metabolic ability | DNA<RNA<蛋白质 | 蛋白质是代谢活性最明确的指示指标,DNA只显示代谢潜力 |
| 提取容易程度 Ease of extraction | DNA≈PLFA> RNA>蛋白质 | DNA和PLFA在不同介质中均可以按照步骤提取获得,而RNA和蛋白质在环境样品中的提取有一定困难 |
| 稳定性 Stability | DNA≈PLFA> 蛋白质>RNA | DNA和PLFA相对稳定,蛋白质可能变性,mRNA非常容易降解 |
SIP实验虽然只能应用于富集培养物或环境群落的密闭培养实验,但是该方法是目前唯一可以将微生物种类和代谢功能直接联系起来的方法,还可用于分析植物和微生物之间的相互作用。其中,DNA-SIP和RNA-SIP均可将微生物的系统发育信息或功能基因信息与其代谢过程相联系起来,操作较容易,且分辨率高,在环境样品,尤其是较复杂的环境样品的分析中(如土壤)应用广泛,并常与其他DNA或RNA分析的方法相结合来分析环境样品中活跃代谢标记底物的微生物群落组成,如荧光实时定量PCR、DGGE[22]、T-RFLP[23]、微列阵[29]和高通量测序(目标基因或宏组学)[22-23, 30]等(表 1)。缺氧的水稻土壤微生物对甲烷排放的贡献占全球甲烷排放的10%—25%,而甲烷产生的主要碳源是植物残体和根系分泌物,识别活跃代谢植物残体和根系分泌物且产生甲烷的微生物种群具有重要的环境意义。Lu等[31]通过采用13CO2代替空气中的CO2对水稻进行脉冲标记,分析根际土壤活跃代谢根系分泌物的微生物,发现根际土壤中古菌Rice Cluster I 显著同化了13C,说明该类古菌在植物分泌物或残体分解产生甲烷的过程中起着重要的作用。Kim等[23]采用13C-甲苯标记海洋沉积物中微生物,通过DNA-T-RFLP分析发现,与轻DNA组分(理论上为12C-DNA)相比,重DNA组分(理论上为13C-DNA)中酶切片段为485 bp的峰占优势,系统发育上属于硫单胞杆菌属(Desulfuromonas);通过宏基因组学的方法,发现该菌除了具有严格厌氧菌的II型苯甲酰辅酶A还原酶之外,还具有兼性厌氧菌的I-型苯甲酰辅酶A还原酶,另外,还识别出了各种厌氧电子受体和编码末端氧化酶的相关基因,从而推断出该属微生物代谢甲苯的潜力。
2.2 宏基因组学和宏转录组学微生物基因组和转录组,即提取一种已知生物的DNA或RNA,不进行目标片段的PCR扩增,随机打断成几百个碱基的片段进行测序,得到全面的遗传信息,也称为鸟枪测序法。与基因组学和转录组学不同的是,宏基因组学和宏转录组学反映的是多种生物或环境中整个群落的特征,包含环境微生物的全部遗传信息,相比于16S rRNA,除了群落中各种微生物群落的分类信息外,宏组学更包含了所有微生物的基因信息,有助于对微生物群落的潜在功能进行深入分析[32]。宏基因组学的挑战有2个方面,一是从随机测序的短序列中识别和定量基因功能,二是将这些功能基因片段与其他可以作为分类信息参考的基因联系起来,一般功能基因主要通过将测得的短序列与已经发表的或已注释的基因组进行比对来识别[4],与已有的参考数据库信息直接相关。宏转录组学和宏基因组学的区别在于,宏转录组学可以实时反映微生物群落的基因表达情况,得到的信息包括样品中哪些生物在当前是活跃的,以及它们在做什么。微生物细胞和环境样品中(如土壤)RNA的滞留时间显著低于DNA,因此,环境样品中提取的RNA理论上是该样品当前生物基因表达的一个快照。深度宏组学研究常用来理解复杂微生物群落的动态,然而,这类项目(尤其是在土壤中)所需的计算资源依然受到限制,如计算机硬件和数据处理方面。目前,虽然计算所需硬件的限制仍然存在,但是数据处理方面发展迅速,如WebCarma[33],MG-RAST[34],Galaxy[35]和GenePattern[36]等工具已经得到了广泛的应用。
宏基因组学和宏转录组学呈现的是多种生物或环境中整个群落的特征,包含环境微生物的全部遗传信息;与稳定同位素标记相结合,较其他微生物多样性分析的方法,宏组学
可以更全面的反应代谢特定底物的微生物的信息。随着测序技术的发展,宏组学的方法在环境微生物的研究中已有较多的应用,包括土壤、湖泊和海洋等生态系统[23, 30, 37]。Xia等[38]采用宏基因组学和宏转录组学的方法分析了高温下纤维素厌氧降解产生甲烷的过程的微生物群落,结果表明,在纤维素高温降解过程中,热袍菌(Thermotogales)对β-糖的消耗是纤维素水解过程中的关键步骤;尽管纤维素发酵过程产生大量的乙酸,嗜乙酸甲烷八叠球菌(Methanosarcinales)的丰富度较嗜氢甲烷杆菌(Methanobacteriales)低60%,而关键基因的转录活性结果表明,乙酸降解产甲烷途径的活跃程度显著高于嗜氢产甲烷途径,说明嗜乙酸产甲烷菌在数量上占少数并不代表其代谢活性低。
2.3 单细胞水平研究方法识别目标微生物的种类和功能稳定同位素与微生物的DNA或RNA(DNA/RNA-SIP)相结合,可以从群落水平将微生物的种类、组成与其功能相联系起来,但是对微生物单细胞水平的代谢活性以及不同种类微生物细胞的代谢活性的差异等信息依然难以揭示。随着单细胞成像方法的改进,结合稳定同位素或放射性同位素标记和荧光原位杂交(Fluorescence in situ hybridization,FISH),可将微生物的数量、种类和功能以及一些微生物之间的相互作用从视觉上展现在图片中。放射性自显影技术(Microautoradiography,MAR)是最早的微生物单细胞活性的测定方法之一,灵敏度高,放射性底物的检测量最低可达10-10—10-15 mg[39],然而,该方法的应用受到标记元素的限制,标记元素需要同时具有放射性和合适的半衰期两个条件。拉曼光谱学显微技术(Raman spectroscopy,Raman)和纳米二次离子质谱(Nano-scale secondary ion mass spectrometry,nanoSIMS)技术的发展,使单细胞成像的方法得到补充。
拉曼光谱技术是根据原子质量高的重同位素(如13C和15N)产生的拉曼光谱波长较轻同位素短,来分析微生物细胞的代谢功能。例如,当微生物利用13C标记的底物以后,一些生物标志物的拉曼波长变短,如核酸、蛋白质、碳水化合物和脂类等[14]。此外,尽管每个物种均可以检测到核酸、蛋白质、碳水化合物和脂类等生物标志物,但是不同物种所产生图谱的峰高有一定差异,因此,每个物种产生的拉曼图谱不同,说明拉曼光谱技术还具有生成全细胞指纹图谱(whole-organism fingerprints)的潜力[40]。
纳米二次离子质谱可以检测任何稳定性同位素和具有合适半衰期的任何放射性同位素,常用于分析生物样品中的C、N、P、S、O、H 和卤族元素等[41],得到复杂微生物群落中微生物细胞代谢活性的成像。此外,测定同位素的比值信息(例如13C/12C),可以用于计算单细胞的元素吸收速率,分析元素的代谢途径[25],为环境中微生物的单细胞生态学和代谢潜力的研究打开了一个前所未有的窗口。该方法对同位素比值的分析具有非常高的灵敏度和准确度(理论上为1‰)[25, 42],与其他单细胞的研究方法相比,具有非常高的优势。另外,纳米二次离子质谱与荧光原位杂交的方法相结合时,探针中的荧光标记由卤素代替,即可在同一张图像中同时显示微生物的种类和功能[17],可以避免将荧光原位杂交的图像与NanoSIMS 分析的图像难以正确联系起来的问题。
拉曼光谱技术和纳米二次离子质谱的分辨率可达微米甚至纳米级别,通过放射性或稳定性同位素底物标记分析纯培养或环境微生物细胞的代谢活性,结合原位杂交来确定目标微生物的系统发育信息[43],尽管这些方法目前主要应用于相对简单的环境,如水体、纯培养微生物或富集培养物等[43-44],在土壤微生物的研究中的应用还需要更多的探索和优化,这些方法的应用增强了对环境微生物种类和功能之间联系的理解,同时为将微生物如动植物一样在单个生物水平上进行研究提供了契机。Raman-FISH作为一种单细胞水平非破坏性的方法,在微生物生态学研究中的应用还较少。到目前为止,只有13C标记的研究结果。通过13C10-萘标记结果表明,地表水中绿脓杆菌(Pseudomonas spp.)可以显著吸收同化13C10-萘,且单个绿脓杆菌细胞之间13C的含量不同[45],说明这种方法除了同时检测微生物的种类和功能外,还具有识别微生物细胞代谢异质性的潜力。NanoSIMS-FISH在微生物生态学中的应用较Raman-FISH广泛,在微生物碳、氮和硫循环的研究中均有应用[43, 46-47]。Musat等[47]研究寡营养湖水中3种厌氧光合细菌,闪囊菌(Lamprocystis purpurea)、奥氏着色菌(Chromatium okenii)和泥生绿菌(Chlorobium clathratiforme)对无机碳(H13CO\|3)和氮(15NH4+)的吸收特征,结果表明,仅占细胞总数0.3%的奥氏着色菌贡献了光合细菌碳吸收总量的70% 和氮吸收总量的40%,说明丰富度较低的奥氏着色菌在该湖水的碳氮循环过程起着主要作用。Dekas等[48]采用15N2标记培养实验与NanoSIMS-FISH分析相结合,发现厌氧甲烷氧化团聚颗粒可以进行生物固氮,且生物固氮由厌氧甲烷氧化古菌完成,此外,部分固定的氮可以转运至与其共生的非固氮的硫还原细菌中,该研究不仅揭示了全球碳、氮和硫循环之间的联系,还显示出将同位素标记技术与NanoSIMS-FISH联合使用,使微生物细胞的生理组成元素及其相关代谢和转运过程的研究可视化的潜力。
3 总结与展望微生物在全球尺度上调控着生物地球化学循环过程,影响生态系统的功能。理解生态系统功能,从群落和单细胞水平揭示自然环境中微生物的种类和功能、微生物之间以及微生物与环境因子之间的相互作用是必不可少的。分子生物学方法的发展和进步为环境微生物的研究提供了方便,但是不同方法有其内在的局限性(表 1),在方法的选择和应用上需根据研究具体情况进行选择。例如,高通量测序、宏组学和单细胞水平研究的方法均是近些年快速发展的方法,与DGGE和T-RFLP等传统方法相比,微生物的多样性或功能基因的分析的广度、深度和分辨率均有显著提高,显示出了其前所未有的优势和应用前景。然而,这些方法本身仍存在一些问题:(1)高通量测序得到的多为几百个碱基或者更短的序列,组成完整的基因有一定困难;即使获得整条序列,由于很多基因的功能还不清楚,很难获得由序列到功能的准确图谱;(2)单细胞水平研究方法中,Raman检测到光谱偏移的同位素标记量需达到10 atom%[14];FISH样品的自发荧光易影响或掩盖探针的荧光信号;样品的制备过程中,固定和脱水等步骤也可能会影响微生物细胞的同位素组成[18]等。此外,基于PCR的方法,由于引物的选择以及PCR本身的问题易对微生物的理解带来偏差,如产生假序列(chimeras)(可通过生物信息学分析移除,如Bellerophon[49]、ChimeraSlayer[50]等软件),另外,得到的信息主要以易于被PCR 扩增出的优势种群为主,而对环境中丰度较低的稀有微生物种群认识不足。未来的工作中,宏组学和单细胞水平研究的方法将有助于揭示自然环境中微生物整个群落的分类、功能信息以及丰度较低但发挥重要功能作用的微生物种群。由于目前用于阐明微生物群落组成和功能的方法均基于参考数据库,这些参考数据库主要是由克隆、Sanger测序或基因组等组成,即使新测序技术和单细胞水平技术有巨大的应用潜力,基于培养方法的研究依然要继续,如果没有这些基础数据,不仅很多序列的功能都难以辨认,用于识别环境微生物种类或功能的核酸探针的准确性也受到限制。随着微生物分子生物学方法(测序技术、单细胞水平研究技术等)的不断改进和数据分析工具的不断发展,对环境中微生物群落组成、微生物之间的相互作用的认识以及对生态系统的功能的理解也在不断增加,为检验和构思生态学理论提供新机遇[4],也为提出环境修复、自然生态系统和农业生态系统的可持续管理的新方法奠定基础。
| [1] | Barnard R, Leadley P W, Hungate B A. Global change, nitrification, and denitrification: a review. Global Biogeochemical Cycles , 2005, 19 (1) : GB1007. |
| [2] | Uhlik O, Leewis M C, Strejcek M, Musilova L, Mackova M, Leigh M B, Macek T. Stable isotope probing in the metagenomics era: a bridge towards improved bioremediation. Biotechnology Advances , 2013, 31 (2) : 154–165. DOI:10.1016/j.biotechadv.2012.09.003 |
| [3] | Mackelprang R, Waldrop M P, DeAngelis K M, David M M, Chavarria K L, Blazewicz S J, Rubin E M, Jansson J K. Metagenomic analysis of a permafrost microbial community reveals a rapid response to thaw. Nature , 2011, 480 (7377) : 368–371. DOI:10.1038/nature10576 |
| [4] | Zimmerman N, Izard J, Klatt C, Zhou J Z, Aronson E. The unseen world: environmental microbial sequencing and identification methods for ecologists. Frontiers in Ecology and the Environment , 2014, 12 (4) : 224–231. DOI:10.1890/130055 |
| [5] | Epstein S S. The phenomenon of microbial uncultivability. Current Opinion in Microbiology , 2013, 16 (5) : 636–642. DOI:10.1016/j.mib.2013.08.003 |
| [6] | Tringe S G, Hugenholtz P. A renaissance for the pioneering 16S rRNA gene. Current Opinion in Microbiology , 2008, 11 (5) : 442–446. DOI:10.1016/j.mib.2008.09.011 |
| [7] | Nilsson R H, Ryberg M, Abarenkov K, Sjökvist E, Kristiansson E. The ITS region as a target for characterization of fungal communities using emerging sequencing technologies. FEMS Microbiology Letters , 2009, 296 (1) : 97–101. DOI:10.1111/fml.2009.296.issue-1 |
| [8] | Kirk J L, Beaudette L A, Hart M, Moutoglis P, Klironomos J N, Lee H, Trevors J T. Methods of studying soil microbial diversity. Journal of Microbiological Methods , 2004, 58 (2) : 169–188. DOI:10.1016/j.mimet.2004.04.006 |
| [9] | 邢德峰, 任南琪. 应用DGGE研究微生物群落时的常见问题分析. 微生物学报 , 2006, 46 (2) : 331–335. |
| [10] | 余素林, 吴晓磊, 钱易. 环境微生物群落分析的T-RFLP技术及其优化措施. 应用与环境生物学报 , 2006, 12 (6) : 861–868. |
| [11] | Gentry T J, Wickham G S, Schadt C W, He Z, Zhou J. Microarray applications in microbial ecology research. Microbial Ecology , 2006, 52 (2) : 159–175. DOI:10.1007/s00248-006-9072-6 |
| [12] | Loman N J, Misra R V, Dallman T J, Constantinidou C, Gharbia S E, Wain J, Pallen M J. Performance comparison of benchtop high-throughput sequencing platforms. Nature Biotechnology , 2012, 30 (5) : 434–439. DOI:10.1038/nbt.2198 |
| [13] | Minoche A E, Dohm J C, Himmelbauer H. Evaluation of genomic high-throughput sequencing data generated on Illumina HiSeq and Genome Analyzer systems. Genome Biology , 2011, 12 : R112–R112. DOI:10.1186/gb-2011-12-11-r112 |
| [14] | Huang W E, Stoecker K, Griffiths R, Newbold L, Daims H, Whiteley A S, Wagner M. Raman-FISH: combining stable-isotope Raman spectroscopy and fluorescence in situ hybridization for the single cell analysis of identity and function. Environmental Microbiology , 2007, 9 (8) : 1878–1889. DOI:10.1111/emi.2007.9.issue-8 |
| [15] | Pett-Ridge J, Weber P K. NanoSIP: NanoSIMS applications for microbial biology. Methods in Molecular Biology , 2012, 881 : 375–408. DOI:10.1007/978-1-61779-827-6 |
| [16] | Eickhorst T, Tippkotter R. Improved detection of soil microorganisms using fluorescence in situ hybridization (FISH) and catalyzed reporter deposition (CARD-FISH). Soil Biology and Biochemistry , 2008, 40 (7) : 1883–1891. DOI:10.1016/j.soilbio.2008.03.024 |
| [17] | Li T L, Wu T D, Mazéas L, Toffin L, Guerquin-Kern J L, Leblon G, Bouchez T. Simultaneous analysis of microbial identity and function using NanoSIMS. Environmental Microbiology , 2008, 10 (3) : 580–588. DOI:10.1111/emi.2008.10.issue-3 |
| [18] | Peteranderl R, Lechene C. Measure of carbon and nitrogen stable isotope ratios in cultured cells. Journal of the American Society for Mass Spectrometry , 2004, 15 (4) : 478–485. DOI:10.1016/j.jasms.2003.11.019 |
| [19] | Aronson E L, Dubinsky E A, Helliker B R. Effects of nitrogen addition on soil microbial diversity and methane cycling capacity depend on drainage conditions in a pine forest soil. Soil Biology and Biochemistry , 2013, 62 : 119–128. DOI:10.1016/j.soilbio.2013.03.005 |
| [20] | Cong J, Liu X D, Lu H, Xu H, Li Y D, Deng Y, Li D Q, Zhang Y G. Available nitrogen is the key factor influencing soil microbial functional gene diversity in tropical rainforest. BMC Microbiology , 2015, 15 : 167–167. DOI:10.1186/s12866-015-0491-8 |
| [21] | Hermann-Bank M L, Skovgaard K, Stockmarr A, Larsen N, Mølbak L. The gut microbiotassay: a high-throughput qPCR approach combinable with next generation sequencing to study gut microbial diversity. BMC Genomics , 2013, 14 : 788–801. DOI:10.1186/1471-2164-14-788 |
| [22] | Lu L, Jia Z J. Urease gene-containing Archaea dominate autotrophic ammonia oxidation in two acid soils. Environmental Microbiology , 2013, 15 (6) : 1795–1809. DOI:10.1111/emi.2013.15.issue-6 |
| [23] | Kim S J, Park S J, Cha I T, Min D, Kim J S, Chung W H, Chae J C, Jeon C O, Rhee S K. Metabolic versatility of toluene-degrading, iron-reducing bacteria in tidal flat sediment, characterized by stable isotope probing-based metagenomic analysis. Environmental Microbiology , 2014, 16 (1) : 189–204. DOI:10.1111/emi.2014.16.issue-1 |
| [24] | Clemmensen K E, Bahr A, Ovaskainen O, Dahlberg A, Ekblad A, Wallander H, Stenlid J, Finlay R D, Wardle D A, Lindahl B D. Roots and associated fungi drive long-term carbon sequestration in boreal forest. Science , 2013, 339 (6127) : 1615–1618. DOI:10.1126/science.1231923 |
| [25] | Kuypers M M M, Jørgensen B B. The future of single-cell environmental microbiology. Environmental Microbiology , 2007, 9 (1) : 6–7. DOI:10.1111/emi.2007.9.issue-1 |
| [26] | Radajewski S, Ineson P, Parekh N R, Murrell J C. Stable-isotope probing as a tool in microbial ecology. Nature , 2000, 403 (6770) : 646–649. DOI:10.1038/35001054 |
| [27] | Cadisch G, Espana M, Causey R, Richter M, Shaw E, Morgan J A W, Rahn C, Bending G D. Technical considerations for the use of 15N-DNA stable-isotope probing for functional microbial activity in soils. Rapid Communications in Mass Spectrometry , 2005, 19 (11) : 1424–1428. DOI:10.1002/(ISSN)1097-0231 |
| [28] | Schwartz E. Characterization of growing microorganisms in soil by stable isotope probing with H218O. Applied and Environmental Microbiology , 2007, 73 (8) : 2541–2546. DOI:10.1128/AEM.02021-06 |
| [29] | Putkinen A, Larmola T, Tuomivirta T, Siljanen H M P, Bodrossy L, Tuittila E S, Fritze H. Peatland succession induces a shift in the community composition of Sphagnum-associated active methanotrophs. FEMS Microbiology Ecology , 2014, 88 (3) : 596–611. DOI:10.1111/fem.2014.88.issue-3 |
| [30] | Dumont M G, Pommerenke B, Casper P. Using stable isotope probing to obtain a targeted metatranscriptome of aerobic methanotrophs in lake sediment. Environmental Microbiology Reports , 2013, 5 (5) : 757–764. |
| [31] | Lu Y H, Conrad R. In situ stable isotope probing of methanogenic archaea in the rice rhizosphere. Science , 2005, 309 (5737) : 1088–1090. DOI:10.1126/science.1113435 |
| [32] | 刘莉扬, 崔鸿飞, 田埂. 高通量测序技术在宏基因组学中的应用. 中国医药生物技术 , 2013, 8 (3) : 196–200. |
| [33] | Gerlach W, Jünemann S, Tille F, Goesmann A, Stoye J. WebCARMA: a web application for the functional and taxonomic classification of unassembled metagenomic reads. BMC Bioinformatics , 2009, 10 : 430–430. DOI:10.1186/1471-2105-10-430 |
| [34] | Meyer F, Paarmann D, D'Souza M, Olson R, Glass E M, Kubal M, Paczian T, Rodriguez A, Stevens R, Wilke A, Wilkening J, Edwards R A. The metagenomics RAST server-a public resource for the automatic phylogenetic and functional analysis of metagenomes. BMC Bioinformatics , 2008, 9 : 386–386. DOI:10.1186/1471-2105-9-386 |
| [35] | Goecks J, Nekrutenko A, Taylor J, The Galaxy Team. Galaxy: a comprehensive approach for supporting accessible, reproducible, and transparent computational research in the life sciences. Genome Biology , 2010, 11 (8) : R86–R86. DOI:10.1186/gb-2010-11-8-r86 |
| [36] | Reich M, Liefeld T, Gould J, Lerner J, Tamayo P, Mesirov J P. GenePattern 2.0. Nature Genetics , 2006, 38 (5) : 500–500. DOI:10.1038/ng0506-500 |
| [37] | Fierer N, Lauber C L, Ramirez K S, Zaneveld J, Bradford M A, Knight R. Comparative metagenomic, phylogenetic and physiological analyses of soil microbial communities across nitrogen gradients. The ISME Journal , 2012, 6 : 1007–1017. DOI:10.1038/ismej.2011.159 |
| [38] | Xia Y, Wang Y B, Fang H H P, Jin T, Zhong H Z, Zhang T. Thermophilic microbial cellulose decomposition and methanogenesis pathways recharacterized by metatranscriptomic and metagenomic analysis. Science Reports , 2014, 4 : 6708–6708. DOI:10.1038/srep06708 |
| [39] | Sorokin Y I. Radioisotopic Methods in Hydrobiology. Heidelberg: Springer , 1999 . |
| [40] | Huang W E, Griffiths R I, Thompson I P, Bailey M J, Whiteley A S. Raman microscopic analysis of single microbial cells. Analytical Chemistry , 2004, 76 (15) : 4452–4458. DOI:10.1021/ac049753k |
| [41] | Boxer S G, Kraft M L, Weber P K. Advances in imaging secondary ion mass spectrometry for biological samples. Annual Review of Biophysics , 2009, 38 : 53–74. DOI:10.1146/annurev.biophys.050708.133634 |
| [42] | Orphan V J, Turk K A, Green A M, House C H. Patterns of 15N assimilation and growth of methanotrophic ANME-2 archaea and sulfate-reducing bacteria within structured syntrophic consortia revealed by FISH-SIMS. Environmental Microbiology , 2009, 11 (7) : 1777–1791. DOI:10.1111/emi.2009.11.issue-7 |
| [43] | Musat N, Foster R, Vagner T, Adam B, Kuypers M M M. Detecting metabolic activities in single cells, with emphasis on nanoSIMS. FEMS Microbiology Reviews , 2012, 36 (2) : 486–511. DOI:10.1111/j.1574-6976.2011.00303.x |
| [44] | Abraham W R. Applications and impacts of stable isotope probing for analysis of microbial interactions. Applied Microbiology and Biotechnology , 2014, 98 (11) : 4817–4828. DOI:10.1007/s00253-014-5705-8 |
| [45] | Huang W E, Ferguson A, Singer A C, Lawson K, Thompson I P, Kalin R M, Larkin M J, Bailey M J, Whiteley A S. Resolving genetic functions within microbial populations: in situ analyses using rRNA and mRNA stable isotope probing coupled with single-cell raman-fluorescence in situ hybridization. Applied and Environmental Microbiology , 2009, 75 (1) : 234–241. DOI:10.1128/AEM.01861-08 |
| [46] | 胡行伟, 张丽梅, 贺纪正. 纳米二次离子质谱技术(NanoSIMS)在微生物生态学研究中的应用. 生态学报 , 2013, 33 (2) : 348–357. |
| [47] | Musat N, Halm H, Winterholler B, Hoppe P, Peduzzi S, Hillion F, Horreard F, Amann R, Jorgensen B B, Kuypers M M M. A single-cell view on the ecophysiology of anaerobic phototrophic bacteria. Proceedings of the National Academy of Sciences of the United States of America , 2008, 105 (46) : 17861–17866. DOI:10.1073/pnas.0809329105 |
| [48] | Dekas A E, Poretsky R S, Orphan V J. Deep-sea archaea fix and share nitrogen in methane-consuming microbial consortia. Science , 2009, 326 (5951) : 422–426. DOI:10.1126/science.1178223 |
| [49] | Huber T, Faulkner G, Hugenholtz P. Bellerophon: a program to detect chimeric sequences in multiple sequence alignments. Bioinformatics , 2004, 20 (14) : 2317–2319. DOI:10.1093/bioinformatics/bth226 |
| [50] | Haas B J, Gevers D, Earl A M, Feldgarden M, Ward D V, Giannoukos G, Ciulla D, Tabbaa D, Highlander S K, Sodergren E, Methé B, DeSantis T Z, The Human Microbiome Consortium, Petrosino J F, Knight R, Birren B W. Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons. Genome Research , 2011, 21 (3) : 494–504. DOI:10.1101/gr.112730.110 |
 2016, Vol. 36
2016, Vol. 36