文章信息
- 白刃, 贺纪正, 沈菊培, 陈新, 张丽梅
- BAI Ren, HE Jizheng, SHEN Jupei, CHEN Xin, ZHANG Limei.
- 厌氧铵氧化过程中关键酶及相关分子标记在生态学研究中的应用进展
- Review of the key enzymes in the anammox process and ecological inferences derived from related molecular markers
- 生态学报[J]. 2016, 36(13): 3871-3881
- Acta Ecologica Sinica[J]. 2016, 36(13): 3871-3881
- http://dx.doi.org/10.5846/stxb201411072209
-
文章历史
- 收稿日期: 2014-11-07
- 网络出版日期: 2015-10-30
2. 中国科学院大气物理研究所, 大气边界层物理和大气化学国家重点实验室, 北京 100029;
3. 中国科学院大学, 北京 100049
2. State Key Laboratorty of Atomospheric Boundary Layer Physics and Atmospheric Chemistry, Institute of Atomspheric Physics, Chinese Academy of Sciences, Beijing 100029, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China
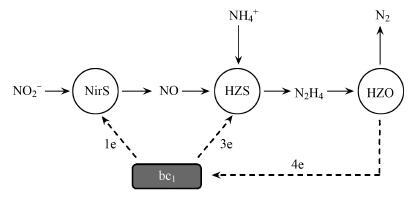
厌氧铵氧化(Anaerobic ammonium oxidation,anammox)作用是铵离子(NH4+)与亚硝酸根离子(NO2-)在厌氧条件下结合,以联氨(N2H4)为催化中间体,生成氮气和水的过程(图 1)[1]。在生态系统氮循环中,厌氧铵氧化是发现较晚的由微生物介导的含氮化合物的转化过程。早在20世纪70年代,奥地利科学家E. Broda就已经从热力学的角度推测出发生厌氧铵氧化反应过程的可能性,但直到1995年Mulder等才真正通过稳定同位素标记的手段证实了这一过程的存在。他们通过向厌氧生物反应器中加入底物14NO3-与15NH4+进行培养,证实了该过程的反应终产物主要为29N2[2]。随后的更多研究表明,厌氧铵氧化过程不仅在含氮废水的脱氮处理中起着重要作用[3],同时还是自然生态系统氮损失的重要途径之一。例如在黑海和Gulfo Dulce等海域,通过厌氧铵氧化过程产生N2导致的氮损失可占全部氮损失的20%-40%[4-5]。
目前已知的厌氧铵氧化细菌主要包括5个属Candidatus Brocadia, Candidatus Kuenenia,Candidatus Scalindua, Candidatus Jettenia和Candidatus Anammoxoglobus[4-5]均属于浮霉菌门(Planctomycetes)。由于厌氧铵氧化细菌难以培养,目前仅能获得其富集培养物。对其多样性及分布特征的研究主要是通过对环境样品中16S rRNA基因序列的分析展开的。大量基于16S rRNA基因的调查表明,厌氧化铵氧化细菌广泛分布于海洋、沉积物、淡水湖泊以及土壤生态系统中[6-8]。然而,由于目前厌氧铵氧化细菌16S rRNA基因的PCR引物相对较低的特异性,及该基因与微生物功能活性相对较弱的关联性等因素,限制了对厌氧铵氧化细菌多样性及生态学功能的进一步研究。近年来,随着对几个富集培养物的基因组学分析和酶学机制研究,及对参与厌氧铵氧化过程关键功能的酶如亚硝酸盐还原酶(Nir),联氨合成酶(HZS)和联氨氧化酶(HZO)及其编码基因的发现和揭示,使人们对于厌氧铵氧化过程的微生物代谢及遗传机制也有了更深的认识,从而使相关功能基因也逐步被作为厌氧铵氧化微生物的特异性标记并用于其多样性和生态学的研究中(表 1)。
| 酶的名称 Enzymes | 功能 Functions | 已进行宏基因组研究的菌种 Strains with whole genomes sequenced | 已验证酶活性的菌种 Strains with their enzymatic functions studied | 相关分子标记 Molecular markers | 参考文献 References | |
| 亚硝酸盐还原酶 Nitrite reductase, NirS | NO2-的还原 | Candidatus Kuenenia stuttgartiensis Candidatus Scalindua profunda Candidatus Brocadia fulgida (未检测到) | N/A | nirS | [9-11] | |
| 亚硝酸盐还原酶 Nitrite reductase, NirK | NO2-的还原 | Candidatus Jettenia asiatica KSU-1 | KSU-1 | N/A | [12-14] | |
| 联氨合成酶 Hydrazine hydrolase, HZS | N2H4的合成 | Candidatus Kuenenia stuttgartiensis Candidatus Scalindua profunda Candidatus Jettenia asiatica Candidatus Brocadia fulgida | Candidatus Kuenenia stuttgartiensis | hzsA和hzsB | [12, 14-15] | |
| 羟氨氧化酶/联氨 氧化酶Hydroxylamine oxidase,HAO/ Hydrazine synthase, HZO | NH2OH和N2H4 的氧化 | Candidatus Kuenenia stuttgartiensis Candidatus Scalindua profunda Candidatus Jettenia asiatica Candidatus Brocadia fulgida KSU-1 | NH2OH/N2H4: N2H4: | KSU-1 Candidatus Brocadia anammoxidans KSU-1 | hzoA/hzoB | [16-18] |
本文将就厌氧铵氧化过程中的几种关键酶的主要研究进展,以及各相关分子标记在生态学研究中的应用进行综合评述。以期通过对厌氧铵氧化细菌研究的进展进行追踪,为微生物生态学研究以及相关技术的研发提供有价值的信息。
1 参与厌氧铵氧化过程的关键酶厌氧铵氧化作用(NH4+ + NO2- = N2 + 2H2O)包括3个主要反应步骤,在3种酶的催化作用下完成。首先是NO2-在亚硝酸盐还原酶的作用下还原成NO,随后NO和NH4+在联氨合成酶的作用下生成联氨(N2H4),最终联氨在联氨氧化酶的催化下进一步被氧化成N2和H2O。在N2H4氧化为N2的过程中,细胞将电子转移到细胞色素bc1复合物形成质子动力势以获得能量(图1)。以下分别就这些关键酶进行介绍。
1.1 亚硝酸盐还原酶(Nir)亚硝酸盐还原酶(Nitrite reductase,Nir)介导NO2-还原为NO的过程。多数反硝化微生物中都含有Nir[19]。Nir分为含铁的细胞色素cd1亚硝酸还原酶(NirS)和含铜的亚硝酸还原酶(NirK)两种[20]。虽然负责催化相同的生化过程,但从系统发育关系上看这两种酶具有不同的演化历史。编码NirS和NirK的基因作为分子标记,一直被广泛应用于反硝化微生物的生态学研究。直到2006年,对厌氧铵氧化细菌Candidatus Kuenenia stuttgartiensis(K. stuttgartiensis)的全基因组信息分析才发现厌氧铵氧化细菌也含有nirS基因,并且推断NO是厌氧铵氧化过程的重要中间产物之一[10]。而在这之前,人们认为厌氧铵氧化细菌是直接将NO2-还原为羟氨(NH2OH)的,而没有NO这一中间产物产生[17]。2011年,Kartal等发现当向K. stuttgartiensis的培养体系中添加NO清除剂PTIO时,厌氧铵氧化过程会立即受到明显地抑制。同时用荧光探针DAF2-DA进行检测,未加PTIO的培养体系中NO与DAF2-DA反应产生荧光信号,而加PTIO的培养体系中则无荧光产生,证实了厌氧铵氧化过程中存在NO中间产物[21]。
但更进一步的研究发现,不同类群厌氧铵氧化细菌的亚硝酸盐还原途径可能存在明显差异。比如,虽然能够在K. stuttgartiensis中检测到nirS基因,但该基因的mRNA表达水平很低,并且在其蛋白质组氨基酸序列中几乎检测不到NirS[9];与此相反,在Candidatus Scalindua profunda(S. profunda)的基因组中,nirS是丰度最高的蛋白编码基因之一[11];但在与Jettenia属有着较高同源性的菌种KSU-1中,只发现了nirK基因,而没有检测到nirS基因[13]。与此类似,在Candidatus Brocadia fulgida(B. fulgida)和Candidatus Jettenia asiatica(J. asiatica)的宏基因组序列中同样没有检测到与NirS相关的基因,并且仅在后者的基因组中发现了nirK基因[12, 14]。Kartal等还猜测一些厌氧铵氧化细菌可能含有催化NO生成的其他同工酶,如对NH2OH和N2H4具有氧化功能的类羟氨氧化酶是可能的蛋白之一[9]。
1.2 联氨合成酶(HZS)厌氧铵氧化反应的第二步是NO的还原及与NH4+结合生成N2H4的过程。N2H4是目前已知的微生物中一种极罕见的代谢中间产物。尽管N2H4早已被确定为厌氧铵氧化反应的中间产物之一,基因组也显示有一种酶可能参与了这个反应过程[10],但其中的确切机制在很长一段时间内都未明确。早期的一些报导都提出这个过程可能是由联氨水解酶(Hydrazine hydrolase,HH)催化完成的[22-25],但目前除了来自对基因组序列分析预测的结论外,并没有针对HH的活性及结构的研究报导。
最新的研究结果则显示N2H4的形成实际上是通过厌氧铵氧化细菌所特有的联氨合成酶(Hydrazine synthase,HZS)的作用实现的[15]。Kartal等通过直接将NO与NH4+作为K. stuttgartiensis的底物进行厌氧培养,随之产生一定浓度的N2H4,证明了该产物是通过NO所生成的。同时,他们还从该培养体系中成功分离到一种大约为240 kDa的蛋白,该蛋白由通过基因组信息推测出的与N2H4形成关系密切的基因簇kuste2859-2861 (后分别命名为hzsC,hzsB和hzsA)所编码,命名为HZS。他们还将分离纯化出的HZS和类联氨氧化酶与NH4+和NO直接进行反应,发现HZS能催化生成N2H4,然后类联氨氧化酶再将N2H4氧化为终产物N2;而在无HZS的情况下,类联氨氧化酶则无法独立完成相应的反应过程[15]。
在基因簇kuste2859-2861编码的蛋白中,kuste2859全部为β螺旋结构,为HZS的催化位点提供了一个刚性平台。kuste2860含有两个c型细胞色素,推测其可能是催化反应的活性中心。kuste2861蛋白的N-末端和C-末端分别由β螺旋和两个c型细胞色素组成[9]。后来在B. fulgida[12]和J. asiatica[14]的基因组中也检测到与kuste2859-2861高度相似的基因簇。在S. profunda细胞内则发现了kuste2859-2861基因簇的直系同源基因,其中与kuste2861对应的部分被命名为scal01318,而与kuste2859和2860对应的部分则融合为一个整体并称为scal00025[11]。从遗传距离上看,HZS与细胞色素c过氧化物酶及色氨酸-色胺酰胺(TTQ,一种微生物的甲胺催化辅因子)生物合成酶有着较近的亲缘关系[9]。
1.3 联氨氧化酶(HZO)联氨氧化酶(Hydrazine oxidase,HZO)的发现和功能的确定也经历了一个曲折且复杂的过程。1997年,van de Graaf等利用15N同位素标记的方法证实了NH2OH和N2H4是厌氧铵氧化过程的中间产物,同时预测应该存在对NH2OH和N2H4具有氧化活性的酶[17]。由于与好氧氨氧化微生物中的羟氨氧化酶(Hydroxylamine oxidase,HAO)有着相近的功能、结构及编码基因的序列和位置,厌氧铵氧化细菌的HZO在一段时间内曾被认为与好氧氨氧化过程中的HAO是等同的[16],并在当时的文献中多被直接称为HAO或类HAO蛋白(HAO-like protein)。
1998年,Schalk等发现厌氧铵氧化生物反应器中的菌群对N2H4具有转化能力,但单独以N2H4为底物培养时厌氧铵氧化细菌无法生存[26]。他们后来又于2000年在B. anammoxidans的富集培养体系中分离纯化出一种同时具有NH2OH和N2H4氧化活性的酶。该酶为三聚体,分子量183 kDa,含有26个血红素。由于该种酶具有更强的NH2OH氧化能力,且与HAO有十分相似的活性特征,Schalk等将其归为HAO[27]。但从氨基酸序列上看,该酶与真正的HAO存在着明显差异[28]。Strous等通过对K. stuttgartiensis基因组序列的分析,同样认为厌氧铵氧化过程的HAO可能具有催化联氨氧化生成氮气的功能,并在该基因组中发现了10个与类HAO蛋白相关的基因[10]。人们随后从S. profunda[11]、 B. fulgida[12]和J. asiatica[14]的基因组中也发现与类HAO蛋白相关的直系同源基因簇,并进一步推测这个基因簇与对NH2OH和N2H4的代谢有关联。
2007年,Shimamura等从富集有菌株KSU-1的生物反应器中分离纯化出一种具有N2H4氧化活性但不具备NH2OH氧化能力的酶,将其正式命名为联氨氧化酶(HZO)[16]。该酶由2个含8个血红素的二聚体组成,分子量约62 kDa,与之前在厌氧铵氧化细菌中发现的HAO的结构不同。通过基因组信息分析,他们还发现HZO分别由hzoA和hzoB两个基因所编码的。这两个hzo基因的序列和好氧氨化细菌hao基因的相似度只有大约30%,但与K. stuttgartiensis中发现的两个hao基因有着88%和89%的相似度。在随后的研究中,Shimamura等进一步从KSU-1中分离出一种和他们先前纯化的HZO性质不同,但与之前Schalk等从B. anammoxidans中分离的HAO相似的酶,该酶对NH2OH和N2H4同时具有氧化能力且对NH2OH的氧化活性更高。同时,这种酶对N2H4的氧化活性也低于KSU-1的HZO。更有趣的是,对基因序列的进一步分析发现,编码该HAO的基因有着和KSU-1菌株hzoB基因相同的上游片段序列,显示该HAO与HZO有较近的遗传距离[23]。Klotz等进而通过蛋白序列研究了HAO与HZO在演化生物学上的关系,发现这两种酶均由反硝化细菌的硝酸盐异化还原酶NrfA演化而来,且推测它们的出现与微生物体内对NH2OH的解毒作用有关。他们认为,虽然在系统发育分析上HAO与HZO可以分别聚成单独的簇并各自具有一定的特性,但它们在功能上属于同一种酶,其本质上的区别只有在好氧氨氧化与厌氧铵氧化细菌体内对NH2OH和N2H4进行氧化时才有所表现[18, 29]。近来,通过对多个厌氧铵氧化细菌的基因组序列比较,人们已经鉴定了HAO/HZO基因簇中与N2H4的氧化相关的两个基因kustc0694和kustd1340[14],但HAO/HZO基因簇中其它各基因的功能及该基因簇在厌氧铵氧化过程中扮演的具体角色仍需要进一步研究确定。
2 厌氧铵氧化细菌的分子标记及其分子生态学研究进展通过对厌氧铵氧化细菌的代谢机理及基因组学的研究,为人们理解其多样性和生态学特征提供了重要的信息。目前,16S RNA基因已经广泛被应用于厌氧铵氧化细菌在各种生态系统中的多样性和群落结构的研究中,并取得了丰富的成果。与此同时,越来越多的研究也开始利用具有更高特异性及多样性的nirS,hzs和hzo基因。
2.1 16S rRNA基因16S RNA基因是最早用于厌氧铵氧化细菌检测的标记基因,目前报道有多对引物可用于厌氧铵氧化细菌 16S RNA基因的扩增,但各引物的覆盖率和扩增效率各有不同。如Penton较早比较了Brod541F/Brod1260R、Pla46/Amx0368等几组引物对污水处理器中厌氧铵氧化细菌的检测能力,发现Brod541F/Brod1260R对Scalindua属具有特异性扩增效果,而Pla46/Amx0368能检测到全部厌氧铵氧化细菌;随后Penton也针对多种生境进行了引物比较,发现An7F/An1388R能成功扩增3个属的类群[30];Dale通过发展了巢式PCR引物Pla46F/1037R和Amx368F/Amx820R,进一步增强了对环境样品PCR扩增的效率[31]。最近,Li比较了几组16S rRNA基因的PCR引物,发现Brod541F/Amx820R对海岸湿地、浅海和深海样品中厌氧铵氧化细菌的阳性克隆检测率最高(可达98%),而在较早研究中使用的引物An7F/An1388R的检测率则只有12%[32];Han通过比较发现引物A438f/A684r对海洋、湿地、淡水水库及生物反应器等不同环境中的厌氧铵氧化细菌有更好的扩增效果。可见,目前研究中对于厌氧铵氧化细菌16S rRNA基因PCR引物的选择和应用仍存在着较大的差异,引物之间的差异也可能导致研究结果产生一定的偏差(图 2)。

|
| 图 2 基于16S rRNA基因的厌氧铵氧化细菌系统发育树 Fig. 2 Phylogenetic tree of anammox bacteria based on 16S rRNA genes |
虽然不同的PCR扩增引物有一定的偏向性,但大量基于16S rRNA基因分析的研究结果显示,厌氧铵氧化细菌5个属Brocadia,Kuenenia,Scalindua,Jettenia和Anammoxoglobus的分布有一定的生境特异性[33]。Scalindua属主要分布于海洋生态系统中,同时它们在从陆地到海洋生态系统的过渡地带中也有较高的丰度,而其它4个属则主要在淡水和陆地生态系统中占到优势[34-35]。大量研究结果显示,在使用不同16S rRNA基因引物的情况下,无论是在海水水域或底栖物中均发现Scalindua属具有较高的多样性和优势度,且检测到的克隆子与已知的Candidatus Scalindua sorokinii,Candidatus Scalindua wagneri和Candidatus Scalindua brodae有着较高的匹配度[30, 32, 36]。而在湿地、淡水河口生态系统的研究中,5个属的16S rRNA基因均可被检测到,但以Brocadia属,Kuenenia属和Jettenia属为主,且在不同研究中检测到的优势类群也存在着较大差异[37-38]。如有研究表明,水稻田生态系统中能够检测到除Scalindua属的其余4个属的16S rRNA基因[39],也有报道进一步指出在所研究的32个农田土壤中均未检测到Scalindua属,Brocadia和Kuenenia属约则占了92%[40]。然而也有研究发现在中国东北地区的水稻土中厌氧铵氧化细菌主要是由Scalindua属为主且具有较高的多样性[41]。以上结果均暗示了陆地生态系统中厌氧铵氧化细菌16S rRNA基因的多样性和分布存在着较大的异质性,其分布格局可能受到环境理化性质、营养物质的分布和土壤类型等诸多因素的影响。
对16S rRNA基因的研究让人们对厌氧铵氧化细菌的分类地位、多样性和生态分布特征有了较为清晰的认识。但对厌氧铵氧化细菌 16S rRNA基因的定量研究仍不理想,不同研究结果间可比性差。同时,由于16S rRNA基因与微生物生态功能之间较低的关联性以及不同扩增引物之间存在的偏差,仍然需要寻找更为有效的分子标记[42]。因此,近年来越来越多的研究中开始使用3个重要的功能基因来检测厌氧铵氧化细菌的多样性、群落结构及丰度。
2.2 nirS基因厌氧铵氧化细菌的nirS基因与反硝化细菌的nirS基因有着较高的分化程度和较远的遗传距离,因此需要特异性引物进行扩增。Lam等首先基于Candidatus Schalindua的全基因组序列设计了针对该属的nirS基因特异性引物Scnir372F/Scnir845R,并用该引物对秘鲁海域最小含氧区(oxygen minimum zone,OMZ)进行了研究[43]。结果显示,通过该引物所检测到的nirS基因序列均属于Scalindua属,且该基因的丰度与之前报导该海域中16S rRNA基因的丰度以及厌氧铵氧化细菌的细胞丰度呈显著正相关关系,并且nirS基因的表达量显著高于反硝化细菌nirS基因的表达量,暗示在该生境中厌氧铵氧化细菌更占优势[43]。Jensen等对通过阿拉伯海OMZ中厌氧铵氧化细菌的细胞丰度、厌氧铵氧化细菌和反硝化细菌的nirS基因表达量的研究也得到了类似的结果[44]。
Li等针对其它4个厌氧铵氧化细菌属的nirS基因设计了特异性引物(AnnirS379F/AnnirS821R),并同时使用Schalindua属nirS特异性引物对中国东南沿海部分地区海洋生态系统进行了研究[25]。作者发现该海域中厌氧铵氧化细菌nirS基因有较高的多样性,并与16S rRNA基因和hzo基因的研究结果具有较好的一致性。该研究显示nirS在聚类树上形成Schalindua属,Kuenenia属和其他属nirS基因3个分枝,且多数序列与秘鲁海域和阿拉伯海域OMZ的Schalindua属的nirS序列有较高的相似性,但与反硝化细菌nirS基因有显著差异。其中,Schalindua属的nirS分枝可以再细分为4个簇。通过主成分分析还发现,nirS基因的组成结构随NH4/NOx环境梯度发生明显变化。Li等进一步对我国南海中的Schalindua属nirS基因的分类组成进行了细致研究,发现该基因的丰度对环境因子的响应和反硝化细菌nirS基因更为相似,与反硝化细菌的nirK基因明显不同[45]。Kirkpatrick等对黑海的研究则发现,Schalindua属nirS基因的4个簇均在5月、7月和10月间具有显著不同的表达量,同时第2、3簇在不同水域深度中的分布较为平均,而第1簇集中分布在浅表层,第4簇主要分布在深层水域[46]。这些研究结果揭示了厌氧铵氧化细菌的高度多样性及其与环境因子之间的密切关系。
目前有关厌氧铵氧化细菌nirS基因多样性的研究仍然较少。同时根据前文的引述,并非所有厌氧铵氧化细菌类群均具有nirS基因,可见该基因不能单独作为厌氧铵氧化细菌的特异性分子标记,而应该与nirK基因配合使用。考虑到Scalindua属具有nirS基因并且在海洋中有着较高的丰度和多样性,因此该基因在评估海洋生态系统厌氧铵氧化细菌的多样性中有着相对较高的参考价值。
2.3 hzs基因联氨合成酶(HZS)由hzsA,hzsB,hzsC基因簇编码,目前尚未在其它微生物的基因组中发现该基因簇,因此hzs基因可以作为厌氧铵氧化细菌的特异性分子标记用于多样性和定量检测研究。Harhangi等针对已知的5个属的厌氧铵氧化细菌hzsA基因序列设计了引物(hzsA_526F/hzsA_1857R)及PCR反应体系,并以来自废水处理器和多种自然生境的样本为模版对引物进行了评估,发现来自废水处理器样本的hzsA基因序列与已报道的B. fulgida[47], B. anammoxidans[7],J. asiatica[48]的hzsA基因有着较高的相似度。在系统发育水平上,已知的厌氧铵氧化细菌hzsA基因与16S rRNA基因也有着很高的一致性[42]。
与16S rRNA的检测结果类似,基于hzsA基因的研究结果也显示海洋生态系统中厌氧铵氧化细菌主要是由Scalindua属组成。如Borin等在地中海东部的深海中发现的hzsA基因全部属于Scalindua属[49],Russ等在深海生态系统中也仅发现了属于Scalindua属的hzsA克隆,并且发现在烃释放量较高的环境中该基因拷贝数更高[50]。Hu等研究了水稻土中hzsA的多样性,发现其中的主要类群为Brocadia属,同时也检测到少量Jettenia和Anammoxoglobus属的序列,但没有发现Kuenenia属的序列[51]。Wang等针对hzsB基因也设计了引物hzsB396F/hzsB742R,并利用该引物研究了水稻田土壤中hzsB基因的多样性和丰度。结果同样检测到的类群主要属于Brocadia属,同时还检测到若干未明确分类的簇,暗示hzsB基因可能具备更高的多样性[52]。在Wang等利用同样的引物对河口生态系统进行的研究中,5个属的序列均有发现,表明hzsB基因在河流生态系统中的多样性可能比农业土壤高[37]。
此外,Hu等利用该标记对一些来自废水处理反应器的样本进行了定量PCR检测,发现hzsA基因的拷贝数较之前研究过的16S rRNA基因拷贝数低3-6倍[53],说明相对于16S rRNA基因,hzsA基因的引物可能有更高的特异性。在一个针对我国大部分地区农田生态系统的研究中,Shen等发现hzsA基因的丰度在空间梯度上体现了较大的异质性,并主要受土壤铵态氮的影响[40]。对土壤剖面的PCR分析结果显示hzsB基因的丰度在剖面表层和深层中都相对较低,但在中等深度处(40-50 cm)最高[52]。
2.4 hzo 基因hzo基因负责编码厌氧铵氧化细菌的联氨氧化酶(HZO),目前已报道有多对针对该基因的引物被用于分子生态学研究。Quan等根据菌株KSU-1和K. stuttgartiensis的hzo基因序列首先设计了该基因的PCR引物hzoF1/hzoR1,并对几个废水处理生物反应器中的厌氧铵氧化细菌的多样性进行了研究[48]。发现通过该引物得到的氨基酸序列与KSU-1和K. stuttgartiensis的hzo基因的相似度在90%-98%之间。Schmid等通过对大量hzo基因序列进行总结分析[54],发现这些序列可以分为cluster Ⅰ,cluster Ⅱ,cluster Ⅲ 三个簇,其中cluster Ⅰ包含了KSU-1菌株,推测该簇中其它浮霉菌门的微生物也可能具有HZO的功能活性,并发现Anammoxoglobus 属和Jettenis 属与KSU-1的hzo基因有着较近的亲缘关性。Li等以hzol1F1/hzocl1R2引物研究了17个高温储油罐中hzo基因的多样性[28],发现其中丰度最高的是Scalindua属,此外,还检测到Jettenia和Anammoxoglobus属的序列。 Li等在此基础上进一步设计了针对hzo基因的引物hzoF1/hzoR1并与之前的引物进行了比较,发现新引物对多种生境中厌氧铵氧化细菌hzo基因的阳性检测率更高[32],且得到的氨基酸序列同样可聚为cluster Ⅰ,cluster Ⅱ,cluster Ⅲ三个簇。同时cluster Ⅰ还可进一步细分为subcluster Ⅰ,Ⅱ,Ⅲ和Ⅳ,其中subcluster Ⅰ和Ⅱ中的序列与菌株KSU-1, J. asiatica,以及Anammoxoglobus属的hzo的氨基酸序列很高的相似度;而subcluster Ⅲ和Ⅳ中的序列则与Scalindua属更接近。
相对于nirS和hzs基因,hzo更广泛地被应用于多样性和群落结构的研究中,且在系统发育水平上与对16S rRNA基因的研究结果有着较好的对应性。基于hzo基因的分析同样显示了厌氧铵氧化细菌的分布具有生境特异性的显著特点。Hirsch等对不同水生态系统进行的研究结果发现,hzo基因的多样性组成在地下水、深海、河口及不同河流中有显著差异。此外,hzo基因的多样性比16S rRNA基因更高,暗示hzo基因可能是厌氧铵氧化细菌多样性研究中更为有效的分子标记[6]。Hong对hzo基因的研究发现,从中国南海采集的底栖物中检测到的hzo基因全部属于Scalindua属[55],而从南海滨海地带的红树林区所采集底栖物中,除Scalindua属以外,还检测到Kuenenia属,Brocadia属和Jettenia属的hzo基因序列[56]。Kong等使用针对2个hzo基因簇的特异性引物对哥斯达黎加海域的OMZ进行了研究,发现hzo基因的表达量在表层中最高,尤其以cluster Ⅰ的hzo基因表达量更高。此外,大量hzo基因序列在系统发育树上形成一个新的簇(命名为cluster 2x),与已知的厌氧铵氧化细菌hzo基因相距较远,而有关该簇更精确的分类学地位暂不明确[57]。此外,Wang等以hzo基因为标靶,在我国东北地区的水稻土中检测到大量属于Scalindua属的hzo基因序列,这些Scalindua属的序列占到主要优势且多样性较高[41],但该结果与基于16S rRNA基因调查得出的陆地生态系统中厌氧铵氧化细菌以Brocadia和Kuenenia两个属为主的结论差异较大[38-40](表 2)。
| 目标基因 Target genes | 引物名称 Primers | 引物序列(5'-3') Sequences of the primers | 参考文献 References |
| 16S rRNA | Pla46(用于浮霉菌门) | GACTTGCATGCCTAATCC | [18] |
| 1037R(用于浮霉菌门) | CGACAAGGAATTTCGCTAC | ||
| Amx368F | TTCGCAATGCCCGAAAGG | ||
| Amx820R | AAAACCCCTCTACTTAGTGCCC | ||
| An7F | GGCATGCAAGTCGAACGAGG | [8] | |
| An1388R | GCTTGACGGGCGGTGTG | ||
| Brod541F | GAGCACGTAGGTGGGTTTGT | ||
| Brod1260R | GGATTCGCTTCACCTCTCGG | ||
| A438f | GTCRGGAGTTADGAAATG | [32] | |
| A684r | ACCAGAAGTTCCACTCTC | ||
| Scalindua-nirS | Scnir372F | TGTAGCCAGCATTGTAGCGT | [48] |
| Scnir845R | TCAAGCCAGACCCATTTGCT | ||
| An-nirS | AnnirS379F | TCTATCGTTGCATCGCATTT | [25] |
| AnnirS821R | GGATGGGTCTTGATAAACA | ||
| hzsA | hzsA_382F | GGYGGDTGYCAGATATGGG | [41] |
| hzsA_1353R | ATAHCCTTCMACHTTCACCT | ||
| hzsA_1597F | WTYGGKTATCARTATGTAG | ||
| hzsA_1857R | AAABGGYGAATCATARTGGC | ||
| hzsB | hzsB_396F | ARGGHTGGGGHAGYTGGAAG | [52] |
| hzsB_742R | GTYCCHACRTCATGVGTCTG | ||
| hzo | hzocl1F1 | TGYAAGACYTGYCAYTGG | [54] |
| hzocl1R2 | ACTCCAGATRTGCTGACC | ||
| hzocl1F2 | TGYAAGACYTGYCAYTGGG | ||
| Ana-hzo1F | TGTGCATGGTCAATTGAAAG | [29] | |
| Ana-hzo2R | ACCTCTTCWGCAGGTGCAT | ||
| hzoF1 | TGTGCATGGTCAATTGAAAG | [31] | |
| hzoR1 | CAACCTCTTCWGCAGGTGCATG | ||
| hzoAB1F | GAAGCNAAGGCNGTAGAAATTATCAC | [56] | |
| hzoAB1R | CTCTTCNGCAGGTGCATGATG | ||
| hzoAB4F | TTGARTGTGCATGGTCTAWTGAAAG | ||
| hzoAB4R | GCTGACCTGACCARTCAGG |
综上所述,在厌氮铵氧化细菌被发现不到20a的时间里,人们对其多样性、生态分布和发展相应的分子标记用于其生态学研究等方面都取得了较多的进展,且通过酶学和基因组测序分析揭示了不同厌氧铵氧化细菌的代谢方式及其遗传机理。但从以上综述中也可看出,目前对厌氧铵氧化细菌的代谢途径的理解大多停留在对于基因组数据分析的推测层面,一些关键基因的功能及其功能酶的生物化学特性仍有待进一步确认和验证。同时也有许多具体问题亟待解答:如各种酶在不同类群间是否存在结构和代谢功能上的差异?厌氧铵氧化细菌的HAO在其代谢途径中扮演了什么角色?它与HZO之间的具体关系是什么?有些厌氧铵氧化细菌体内是否含有Nir的同工酶可以催化生成NO?
在分子生态学研究方面,虽然基于16S rRNA基因的研究为认识厌氧铵氧化细菌的分类地位、多样性及分布特征方面提供了重要信息,但该基因扩增引物的代表性和特异性仍有待继续验证,且由于与厌氧铵氧化细菌的真正功能活性缺乏直接的联系,使该基因在进行定量分析的研究中价值有限。新近发现的厌氧铵氧化细菌功能基因nirS、hzo、hzsA和hzsB,虽然在一定程度上弥补了16S rRNA基因的不足,但目前对功能基因的研究刚起步,针对这些基因扩增的引物的特异性和有效性仍存在争议。此外,16S rRNA基因研究结果显示目前已知的厌氧铵氧化细菌仅分布于浮霉菌目的5个属,而对nirS、hzo、hzsA和hzsB基因的研究显示这些基因可能具有更高的多样性。功能基因与16S rRNA基因所代表的微生物之间的聚类关系如何对应,以及功能基因多样性与其真正的生态功能之间如何关联等问题尚不清晰。对这些问题的解答均需要大量的分子生态学和氮素生物地球化学调查及分析。
| [1] | Burgin A J, Hamilton S K.Have we overemphasized the role of denitrification in aquatic ecosystems? A review of nitrate removal pathways.Frontiers in Ecology and the Environment,2007,5(2): 89–96. |
| [2] | Mulder A, Van De Graaf A A, Robertson L A, Kuenen J G.Anaerobic ammonium oxidation discovered in a denitrifying fluidized bed reactor.FEMS Microbioly Ecology,1995,16(3): 177–183. |
| [3] | Li Z G, Ma Y G, Hira D, Fujii T, Furukawa K.Factors affecting the treatment of reject water by the anammox process.Bioresource Technology,2011,102(10): 5702–5708. |
| [4] | Dalsgaard T, Canfield D E, Petersen J, Thamdrup B, Acuña-González J.N2 production by the anammox reaction in the anoxic water column of Golfo Dulce, Costa Rica.Nature,2003,422(6932): 606–608. |
| [5] | Van Niftrik L A, Fuerst J A, Damsté J S S, Kuenen J G, Jetten M S M, Strous M.The anammoxosome:an intracytoplasmic compartment in anammox bacteria.FEMS Microbiology Letters,2004,233(1): 7–13. |
| [6] | Hirsch M D, Long Z T, Song B.Anammox bacterial diversity in various aquatic ecosystems based on the detection of hydrazine oxidase genes (hzoA/hzoB).Microbial Ecology,2011,61(2): 264–276. |
| [7] | LamP, LavikG, JensenM M, VanDe Vossenberg J, SchmidM, WoebkenD, GutiérrezD, AmannR, JettenM S M, KuypersM M.Revising the nitrogen cycle in the Peruvian oxygen minimum zone.Proceedings of the National Academy of Science of the United States of America,2009,106(12): 4752–4757. |
| [8] | Schmid M C, Maas B, Dapena A, Van De Pas-Schoonen K, Van De Vossenberg J, Kartal B, Van Niftrik L, Schmidt I, Cirpus I, Kuenen J G, Wagner M, Sinninghe Damsté J S, Kuypers M, Revsbech N P, Mendez R, Jetten M S M, Strous M.Biomarkers for in situ detection of anaerobic ammonium-oxidizing (anammox) bacteria.Applied and Environmental Microbiology,2005,71(4): 1677–1684. |
| [9] | Kartal B, De Almeida N M, Maalcke W J, Op Den Camp H J M, Jetten M S M, Keltjens J T.How to make a living from anaerobic ammonium oxidation.FEMS Microbiology Reviews,2013,37(3): 428–461. |
| [10] | Strous M, Pelletier E, Mangenot S, Rattei T, Lehner A, Taylor M W, Horn M, Daims H, Bartol-Mavel D, Wincker P, Barbe V, Fonknechten N, Vallenet D, Segurens B, Schenowitz-Truong C, Médigue C, Collingro A, Snel B, Dutilh B E, Op Den Camp H J M, Van Der Drift C, Cirpus I, Van De Pas-Schoonen K T, Harhangi H R, Van Niftrik L, Schmid M, Keltjens J, Van De Vossenberg J, Kartal B, Meier H, Frishman D, Huynen M A, Mewes H W, Weissenbach J, Jetten M S M, Wagner M, Le Paslier D.Deciphering the evolution and metabolism of an anammox bacterium from a community genome.Nature,2006,440(7085): 790–794. |
| [11] | Van De Vossenberg J, Woebken D, Maalcke W J, Wessels H J C T, Dutilh B E, Kartal B, Janssen-Megens E M, Roeselers G, Yan J, Speth D, Gloerich J, Geerts W, Van Der Biezen E, Pluk W, Francoijs K J, Russ L, Lam P, Malfatti S A, Tringe S G, Haaijer S C, Op Den Camp H J M, Stunnenberg H G, Amann R, Kuypers M M M, Jetten M S M.The metagenome of the marine anammox bacterium ‘Candidatus Scalindua profunda’ illustrates the versatility of this globally important nitrogen cycle bacterium.Environmental Microbiology,2013,15(5): 1275–1289. |
| [12] | Gori F, Tringe S G, Kartal B, Marchiori E, Jetten M S M.The metagenomic basis of anammox metabolism in Candidatus ‘Brocadia fulgida’.Biochemical Society Transactions,2011,39(6): 1799–1804. |
| [13] | Hira D, Toh H, Migita C T, Okubo H, Nishiyama T, Hattori M, Furukawa K, Fujii T.Anammox organism KSU-1 expresses a NirK-type copper-containing nitrite reductase instead of a NirS-type with cytochrome cd1.FEBS Letters,2012,586(11): 1658–1663. |
| [14] | Hu Z Y, Speth D R, Francoijs K J, Quan Z X, Jetten M S M.Metagenome analysis of a complex community reveals the metabolic blueprint of anammox bacterium "Candidatus jettenia asiatica".Frontiers in Microbiology,2012,3 366–366. |
| [15] | Kartal B, Maalcke W J, De Almeida N M, Cirpus I, Gloerich J, Geerts W, Op Den Camp H J M, Harhangi H R, Janssen-Megens E M, Francoijs K J, Stunnenberg H G, Keltjens J T, Jetten M S M, Strous M.Molecular mechanism of anaerobic ammonium oxidation.Nature,2011,479(7371): 127–130. |
| [16] | Shimamura M, Nishiyama T, Shigetomo H, Toyomoto T, Kawahara Y, Furukawa K, Fujii T.Isolation of a multiheme protein with features of a hydrazine-oxidizing enzyme from an anaerobic ammonium-oxidizing enrichment culture.Applied and Environmental Microbiology,2007,73(4): 1065–1072. |
| [17] | Van De Graaf A A, De Bruijn P, Robertson L A, Jetten M S M, Kuenen J G.Metabolic pathway of anaerobic ammonium oxidation on the basis of 15N studies in a fluidized bed reactor.Microbiology,1997,143(7): 2415–2421. |
| [18] | Klotz M G, Schmid M C, Strous M, Op Den Camp H J M, Jetten M S M, Hooper A B.Evolution of an octahaem cytochrome c protein family that is key to aerobic and anaerobic ammonia oxidation by bacteria.Environmental Microbiology,2008,10(11): 3150–3163. |
| [19] | Zumft W G.Cell biology and molecular basis of denitrification.Microbiology and Molecular Biology Reviews,1997,61(4): 533–616. |
| [20] | Zumft W G.Nitric oxide reductases of prokaryotes with emphasis on the respiratory, heme-copper oxidase type.Journal of Inorganic Biochemistry,2005,99(1): 194–215. |
| [21] | Egli K, Bosshard F, Werlen C, Lais P, Siegrist H, Zehnder A J B, Van der Meer J R.Microbial composition and structure of a rotating biological contactor biofilm treating ammonium-rich wastewater without organic carbon.Microbial Ecology,2003,45(4): 419–432. |
| [22] | Karlsson R, Karlsson A, Bäckman O, Johansson B R, Hulth S.Identification of key proteins involved in the anammox reaction.FEMS Microbiology Letters,2009,297 87–94. |
| [23] | Shimamura M, Nishiyama T, Shinya K, Kawahara Y, Furukawa K, Fujii T.Another multiheme protein, hydroxylamine oxidoreductase, abundantly produced in an anammox bacterium besides the hydrazine-oxidizing enzyme.Journal of Bioscience and Bioengineering,2008,105(3): 243–248. |
| [24] | Jetten M S M, Niftrik L, Strous M, Kartal B, Keltjens J T, Op Den Camp H J M.Biochemistry and molecular biology of anammox bacteria.Critical Reviews in Biochemistry and Molecular Biology,2009,44(2/3): 65–84. |
| [25] | Li M, Ford T, Li X Y, Gu J D.Cytochrome cd1-containing nitrite reductase encoding gene nirS as a new functional biomarker for detection of anaerobic ammonium oxidizing (Anammox) bacteria.Environmental Science & Technology,2011,45(8): 3547–3553. |
| [26] | Schalk J, Oustad H, Kuenen J G, Jetten M S M.The anaerobic oxidation of hydrazine:a novel reaction in microbial nitrogen metabolism.FEMS Microbiology Letters,1998,158(1): 61–67. |
| [27] | Schalk J, De Vries S, Kuenen J G, Jetten M S M.Involvement of a novel hydroxylamine oxidoreductase in anaerobic ammonium oxidation.Biochemistry,2000,39(18): 5405–5412. |
| [28] | Li H, Chen S, Mu B Z, Gu J D.Molecular detection of anaerobic ammonium-oxidizing (Anammox) bacteria in high-temperature petroleum reservoirs.Microbial Ecology,2010,60(4): 771–783. |
| [29] | Klotz M G, Stein L Y.Nitrifier genomics and evolution of the nitrogen cycle.FEMS Microbiology Letters,2008,278(2): 146–156. |
| [30] | Penton C R, Devol A H, Tiedje J M.Molecular evidence for the broad distribution of anaerobic ammonium-oxidizing bacteria in freshwater and marine sediments.Applied and Environmental Microbiology,2006,72(10): 6829–6832. |
| [31] | Dale O R, Tobias C R, Song B.Biogeographical distribution of diverse anaerobic ammonium oxidizing (Anammox) bacteria in Cape Fear River Estuary.Environmental Microbiology,2009,11(5): 1194–1207. |
| [32] | Li M, Hong Y G, Klotz M G, Gu J D.A comparison of primer sets for detecting 16S rRNA and hydrazine oxidoreductase genes of anaerobic ammonium-oxidizing bacteria in marine sediments.Applied Microbiology and Biotechnology,2010,86(2): 781–790. |
| [33] | Humbert S, Tarnawski S, Fromin N, Mallet M P, Aragno M, Zopfi J.Molecular detection of anammox bacteria in terrestrial ecosystems:distribution and diversity.The ISME Journal,2010,4(3): 450–454. |
| [34] | Schmid M C, Risgaard-Petersen N, Van De Vossenberg J, Kuypers M M M, Lavik G, Petersen J, Hulth S, Thamdrup B, Canfield D, Dalsgaard T, Rysgaard S, Sejr M K, Strous M, Op Den Camp H J M, Jetten M S M.Anaerobic ammonium-oxidizing bacteria in marine environments:widespread occurrence but low diversity.Environmental Microbiology,2007,9(6): 1476–1484. |
| [35] | Yang Q X, Jia Z J, Liu R Y, Chen J J.Molecular diversity and anammox activity of novel planctomycete-like bacteria in the wastewater treatment system of a full-scale alcohol manufacturing plant.Process Biochemistry,2007,42(2): 180–187. |
| [36] | Amano T, Yoshinaga I, Okada K, Yamagishi T, Ueda S, Obuchi A, Sako Y, Suwa Y.Detection of anammox activity and diversity of anammox bacteria-related 16S rRNA genes in coastal marine sediment in Japan.Microbes and Environments,2007,22(3): 232–242. |
| [37] | Wang S Y, Zhu G B, Peng Y Z, Jetten M S M, Yin C Q.Anammox bacterial abundance, activity, and contribution in riparian sediments of the Pearl River Estuary.Environmental Science & Technology,2012,46(16): 8834–8842. |
| [38] | Long A, Heitman J, Tobias C, Philips R Song B.Co-occurring anammox, denitrification, and codenitrification in agricultural soils.Applied and Environmental Microbiology,2013,79(1): 168–176. |
| [39] | Zhu G B, Wang S Y, Wang Y, Wang C X, Risgaard-Petersen N, Jetten M S M, Yin C Q.Anaerobic ammonia oxidation in a fertilized paddy soil.The ISME Journal,2011,5(12): 1905–1912. |
| [40] | Shen L D, Liu S, Lou L P, Liu W P, Xu X Y, Zheng P, Hu B L.Broad distribution of diverse anaerobic ammonium-oxidizing bacteria in Chinese agricultural soils.Applied and Environmental Microbiology,2013,79(19): 6167–6172. |
| [41] | Wang J, Gu J D.Dominance of Candidatus Scalindua species in anammox community revealed in soils with different duration of rice paddy cultivation in Northeast China.Applied Microbiology and Biotechnology,2013,97(4): 1785–1798. |
| [42] | Harhangi H R, Le Roy M, Van Alen T, Hu B L, Groen J, Kartal B, Tringe S G, Quan Z X, Jetten M S M, Op Den Camp H J M.Hydrazine synthase, a unique phylomarker with which to study the presence and biodiversity of anammox bacteria.Applied and Environmental Microbiology,2012,78(3): 752–758. |
| [43] | Van De Vossenberg J, Rattray J E, Geerts W, Kartal B, Van Niftrik L, Van Donselaar E G, Sinninghe Damsté J S S, Strous M, Jetten M S M.Enrichment and characterization of marine anammox bacteria associated with global nitrogen gas production.Environmental Microbiology,2008,10(11): 3120–3129. |
| [44] | Jensen M M, Lam P, Revsbech N P, Nagel B, Gaye B, Jetten M S M, Kuypers M M M.Intensive nitrogen loss over the omani Shelf due to anammox coupled with dissimilatory nitrite reduction to ammonium.The ISME Journal,2011,5(10): 1660–1670. |
| [45] | Li M, Hong Y, Cao H, Klotz M G, Gu J D.Diversity, abundance, and distribution of NO-forming nitrite reductase-encoding genes in deep-sea subsurface sediments of the South China Sea.Geobiology,2013,11(2): 170–179. |
| [46] | Kirkpatrick J B, Fuchsman C A, Yakushev E, Staley J T, Murray J W.Concurrent activity of anammox and denitrifying bacteria in the Black Sea.Frontiers in Microbiology,2012,3 256–256. |
| [47] | Kartal B, Van Niftrik L, Rattray J, Van De Vossenberg J L C M, Schmid M C, Sinninghe Damsté J, Jetten M S M, Strous M.Candidatus ‘Brocadia fulgida’:an autofluorescent anaerobic ammonium oxidizing bacterium.FEMS Microbiology Ecology,2008,63(1): 46–55. |
| [48] | Quan Z X, Rhee S K, Zuo J E, Yang Y, Bae J W, Park J R, Lee S T, Park Y H.Diversity of ammonium-oxidizing bacteria in a granular sludge anaerobic ammonium-oxidizing (anammox) reactor.Environmental Microbiology,2008,10(11): 3130–3139. |
| [49] | Borin S, Mapelli F, Rolli E, Song B, Tobias C, Schmid M C, De Lange G J, Reichart G J, Schouten S, Jetten M, Daffonchio D.Anammox bacterial populations in deep marine hypersaline gradient systems.Extremophiles,2013,17(2): 289–299. |
| [50] | Russ L, Kartal B, Op Den Camp H J M, Sollai M, Le Bruchec J, Caprais J C, Godfroy A, Sinninghe Damsté J S S, Jetten M S M.Presence and diversity of anammox bacteria in cold hydrocarbon-rich seeps and hydrothermal vent sediments of the Guaymas Basin.Frontiers in Microbiology,2013,4 219–219. |
| [51] | Hu B L, Shen L D, Liu S, Cai C, Chen T T, Kartal B, Harhangi H R, Op Den Camp H J M, Lou L P, Xu X Y, Zheng P, Jetten M S M.Enrichment of an anammox bacterial community from a flooded paddy soil.Environmental Microbiology Reports,2013,5(3): 483–489. |
| [52] | Wang Y, Zhu G B, Harhangi H R, Zhu B L, Jetten M S M, Yin C Q, Op Den Camp H J M.Co-occurrence and distribution of nitrite-dependent anaerobic ammonium and methane-oxidizing bacteria in a paddy soil.FEMS Microbiology Letters,2012,336(2): 79–88. |
| [53] | Hu B L, Zheng P, Tang C J, Chen J W, Van Der Biezen E, Zhang L, Ni B J, Jetten M S M, Yan J, Yu H Q, Kartal B.Identification and quantification of anammox bacteria in eight nitrogen removal reactors.Water Resources,2010,44(17): 5014–5120. |
| [54] | Schmid M C, Hooper A B, Klotz M G, Woebken D, Lam P, Kuypers M M M, Pommerening-Roeser A, Op Den Camp H J M, Jetten M S M.Environmental detection of octahaem cytochrome c hydroxylamine/hydrazine oxidoreductase genes of aerobic and anaerobic ammonium-oxidizing bacteria.Environmental Microbiology,2008,10(11): 3140–3149. |
| [55] | Hong Y G, Li M, Cao H L, Gu J D.Residence of habitat-specific anammox bacteria in the deep-sea subsurface sediments of the South China Sea:Analyses of marker gene abundance with physical chemical parameters.Microbial Ecology,2011,62(1): 36–47. |
| [56] | Li M, Hong Y G, Cao H L, Gu J D.Mangrove trees affect the community structure and distribution of anammox bacteria at an anthropogenic-polluted mangrove in the Pearl River Delta reflected by 16S rRNA and hydrazine oxidoreductase (HZO) encoding gene analyses.Ecotoxicology,2011,20(8): 1780–1790. |
| [57] | Kong L, Jing H, Kataoka T, Buchwald C, Liu H.Diversity and spatial distribution of hydrazine oxidoreductase (hzo) Gene in the oxygen minimum zone off costa rica.PLoS ONE,2013,8(10): e78275 |
 2016, Vol. 36
2016, Vol. 36