文章信息
- 孟学平, 申欣, 屠海淼, 朱笑琳, 赵娜娜, 程汉良, 阎斌论
- MENG Xueping, SHEN Xin, TU Haimiao, ZHU Xiaolin, ZHAO Nana, CHENG Hanliang, YAN Binlun
- 基于nad5的西施舌漳州群体遗传分化水平分析——以蛤蜊属2个物种差异水平为参照
- The genetic differentiation analysis on Coelomactra antiquata Zhangzhou populations based on nad5: taking that of two Mactra species as a reference
- 生态学报, 2015, 35(8): 2635-2642
- Acta Ecologica Sinica, 2015, 35(8): 2635-2642
- http://dx.doi.org/10.5846/stxb201306091547
-
文章历史
- 收稿日期:2013-06-09
- 修订日期:2014-05-16
2. 南京农业大学资源与环境科学学院, 江苏省海洋生物重点实验室, 南京 210095
2. Key Laboratory of Marine Biology of Jiangsu Province, College of Resources and Environmental Sciences, Nanjing Agricultural University, Nanjing 210095, China
西施舌广泛地分布于中国沿海,北至辽宁大连,南到广西北海,20年前,福建西施舌产量最多。西施舌是一种珍奇的重要渔业资源,在我国已属于濒危物种[1]。来自形态学[1, 2]、核DNA[3]和线粒体基因组部分序列[4, 5, 6]等分子生物学的资料均显示中国福建(漳州、长乐)西施舌发生了明显的分化,是西施舌的亚种或隐种;来自线粒体全基因组的比较基因组学资料也显示漳州西施舌与日照西施舌发生了明显的分化,使其与其它群体的差异达到了亚种或种间差异水平[7, 8, 9]。目前的资料为福建西施舌分类地位的重新确定提供了重要的依据,但是,就现有的研究结果,要确定福建西施舌是西施舌的亚种或腔蛤蜊属(Coelomactra)的新种,资料尚显不足。线粒体DNA编码的基因保守性不同,在群体遗传差异分析中的解析力也不同。NADH脱氢酶亚基5基因(nad5)在双壳类中相对保守,但较核糖体16S rRNA基因(16S rRNA)、细胞色素c氧化酶亚基1基因(cox1)变异稍大,更能显示群体变异水平。本研究基于nad5核苷酸序列分析中国西施舌4个代表性自然群体遗传差异,同时以蛤蜊科(Mactridae)蛤蜊属(Mactra)的中国蛤蜊(Mactra chinesis)和四角蛤蜊(Mactra veneriformis)间、双壳类部分属属内不同物种间nad5差异水平为参照,对福建西施舌分化水平的界定可提供更有说服力的证据,研究结果对西施舌种质资源的保护和可持续利用及良种选育具有重要意义。
1 材料和方法 1.1 实验材料实验用样本73个,其中西施舌55个,分别采自福建漳州(14个)、连云港(9个)、山东日照(20个)和广西北海(12个)沿海;中国蛤蜊(8个)采自山东日照;四角蛤蜊(10个)采自江苏连云港海域。样本经鉴定后,取闭壳肌75%乙醇固定备用,或用新鲜组织直接提取DNA。
1.2 DNA提取及PCR扩增取乙醇保存的或新鲜组织,用SDS、蛋白酶K裂解组织,用酚-氯仿法抽提蛋白质,乙醇沉淀获得总DNA。将日照[9]、漳州西施舌、中国蛤蜊和四角蛤蜊nad5核苷酸序列用Clustalx软件进行比对,根据相对保守区,设计兼并引物,Thm-4S-ND5F: GRG AGT CNT AYT CYG CTG G:Thm-4S-ND5R:TGA ACR CCV CCW CTR CAM GC。PCR体系25 μL,模板20—50 ng,dNTP(10 mmol/dm3 each)0.5 μL,Taq DNA聚合酶 [D0090,生工生物工程(上海)股份有限公司(上海生工)] 0.3 μL(5U/μL),95℃预变性5 min后,进行35个循环扩增:94℃变性45 s,52—55 ℃退火30 s,72℃延伸1 min。循环结束后,72℃延伸10 min。
1.3 PCR产物测序及序列比对分析PCR产物经0.8%琼脂糖凝胶电泳检测后,送上海生工进行双向测序。PCR产物经切胶回收,在ABI公司的3730xl DNA Analyzer测序仪上测序,试剂为BigDye terminator v3.1。
利用DNAStar软件包中的SeqMan软件对序列进行组装拼接,用看图软件Chromas分析测序峰图,用人工的方法对序列进行校对;查找、切除引物序列,获得用于比对分析的nad5片段核苷酸序列;用SPSS 17.0进行碱基含量差异显著性分析;用DnaSP v5.1软件进行单倍型确定;用Clustalx进行序列对位排列,MEGA4.0进行核苷酸序列差异分析:统计变异位点、简约信息位点,根据K2-P (Kimura 2-parameter)计算遗传距离,用邻接法(Neighber-Joining,NJ)构建系统进化树,系统树节点处自举置信水平用bootstrap估计,验证次数设置为1000;用Arlequin ver 3.1进行分子方差分析(AMOVA),群体间遗传分化指数(FST)计算采用pairwise difference模型。
1.4 双壳类4个属属内种间遗传距离分析从GenBank下载双壳类(Bivalve)巨蛎属(Crassostrea)8个种、贻贝属(Mytilus)4个种、竹蛏属(Solen)2个种、文蛤属(Meretrix)2个种线粒体全基因组,从中析出nad5全序列,截取与本研究扩增序列的同源部分,计算属内种间的遗传距离(K2-P),作为漳州西施舌与非漳州西施舌差异水平判断的参照。物种拉丁文、GenBank序列号见表 5及本文讨论部分。
2 结果 2.1 nad5序列核苷酸含量差异分析本研究共获得西施舌日照、连云港、北海、漳州群体,中国蛤蜊日照群体、四角蛤蜊连云港群体nad5序列73条,拼接后去除引物序列、两端取齐后得到480 bp的DNA片段,用于序列比对与分析。
西施舌4个群体共55条nad5片段,各群体nad5 碱基T、C、A、G平均含量(%)如表 1,由表 1可见漳州西施舌碱基T和A的含量极显著低于其它3个群体(P<0.01),G含量显著高于其它3个群体,C含量4个群体差异不显著。而北海、日照和连云港群体间4种碱基含量差异均不显著(P>0.05)。四角蛤蜊/中国蛤蜊共18条序列,其T、C、A、G平均含量(%)分别为46.27/42.24、13.94/15.68、20.65/20.86、19.15/21.22,AT含量为66.92/63.10,其中T、A和G含量(%)差异极显著(P<0.01),A含量差异不显著(P>0.05)。
| 群体 Population | 碱基含量 Base percentage/% | ||||||||||
| T | C | A | G | ||||||||
| 小写字母表示在0.05水平上差异显著;大写字母表示在0.01水平上差异显著 | |||||||||||
| 北海 | 42.3aA | 14.0 aA | 22.8aA | 20.9bB | |||||||
| 日照 | 42.3aA | 14.0 aA | 22.7 aA | 21.1bB | |||||||
| 连云港 | 42.2aA | 14.1 aA | 22.7 aA | 21.0bB | |||||||
| 漳州 | 40.8bB | 14.0 aA | 20.1 bB | 25.1 aA | |||||||
序列经DnaSP分析,从73个序列中共检测到44种单倍型(Hap) (表 2),其中西施舌有29种Haps,四角蛤蜊10种,中国蛤蜊5种;西施舌中,漳州群体有5种Haps,日照群体15种,连云港群体8种,北海群体3种。其中Hap2由连云港群体和北海群体共享,Hap21由连云港群体和日照群体共享,漳州群体与其它3个群体无共享单倍型。44种Haps序列比对共获得480个比对位点,无插入/缺失碱基,西施舌4个群体29个Haps变异位点(V)122个,占25.4%,简约信息位点(Pi)108个(图 1),占22.5%,北海、日照、连云港群体合并计算,V 位点39 个,占8.1%,Pi 位点14 个,占2.9%;中国蛤蜊和四角蛤蜊共有V 位点99 个,占20.6%,Pi 位点91 个(图 1),占19.0%;3 种贝类合并计算,V 位点 197 个,Pi 位点191 个,分别占41.0%和39.8%。
| 单倍型 Hap | 群体Population | 单倍型 Hap | 群体Population | 单倍型Hap | 群体Population | 单倍型Hap | 群体PopulationMve | 单倍型Hap | 群体PopulationMch | |||||||||
| ZZ | RZ | LYG | BH | ZZ | RZ | LYG | BH | ZZ | RZ | LYG | BH | |||||||
| pop.: population,Hap: haplotype;ZZ:漳州,RZ:日照,LYG:连云港;BH:北海;Mve:四角蛤蜊,Mch:中国蛤蜊 | ||||||||||||||||||
| Hap1 | 6 | Hap11 | 1 | Hap21 | 2 | 1 | Hap30 | 1 | Hap40 | 4 | ||||||||
| Hap2 | 2 | 2 | Hap12 | 2 | Hap22 | 1 | Hap31 | 1 | Hap41 | 1 | ||||||||
| Hap3 | 4 | Hap13 | 2 | Hap23 | 1 | Hap32 | 1 | Hap42 | 1 | |||||||||
| Hap4 | 2 | Hap14 | 1 | Hap24 | 1 | Hap33 | 1 | Hap43 | 1 | |||||||||
| Hap5 | 6 | Hap15 | 1 | Hap25 | 1 | Hap34 | 1 | Hap44 | 1 | |||||||||
| Hap6 | 4 | Hap16 | 1 | Hap26 | 1 | Hap35 | 1 | |||||||||||
| Hap7 | 1 | Hap17 | 2 | Hap27 | 1 | Hap36 | 1 | |||||||||||
| Hap8 | 1 | Hap18 | 1 | Hap28 | 1 | Hap37 | 1 | |||||||||||
| Hap9 | 1 | Hap19 | 1 | Hap29 | 1 | Hap38 | 1 | |||||||||||
| Hap10 | 1 | Hap20 | 2 | Hap39 | 1 | |||||||||||||
| 73 | 14 | 2 | 2 | 12 | 14 | 4 | 7 | 10 | 8 | |||||||||

|
| 图 1 蛤蜊科3种贝类NAD5基因片段核苷酸序列简约信息位点比对 Fig. 1 NAD5 gene fragments comparision of three species in family Mactridae Hap: 单倍型; Hap4—8: 漳州群体; Hap9—23: 日照群体; Hap1—3: 北海群体; Hap2,21,24—29: 连云港群体; Hap30—39:四角蛤蜊群体; Hap40—44:中国蛤蜊群体 |
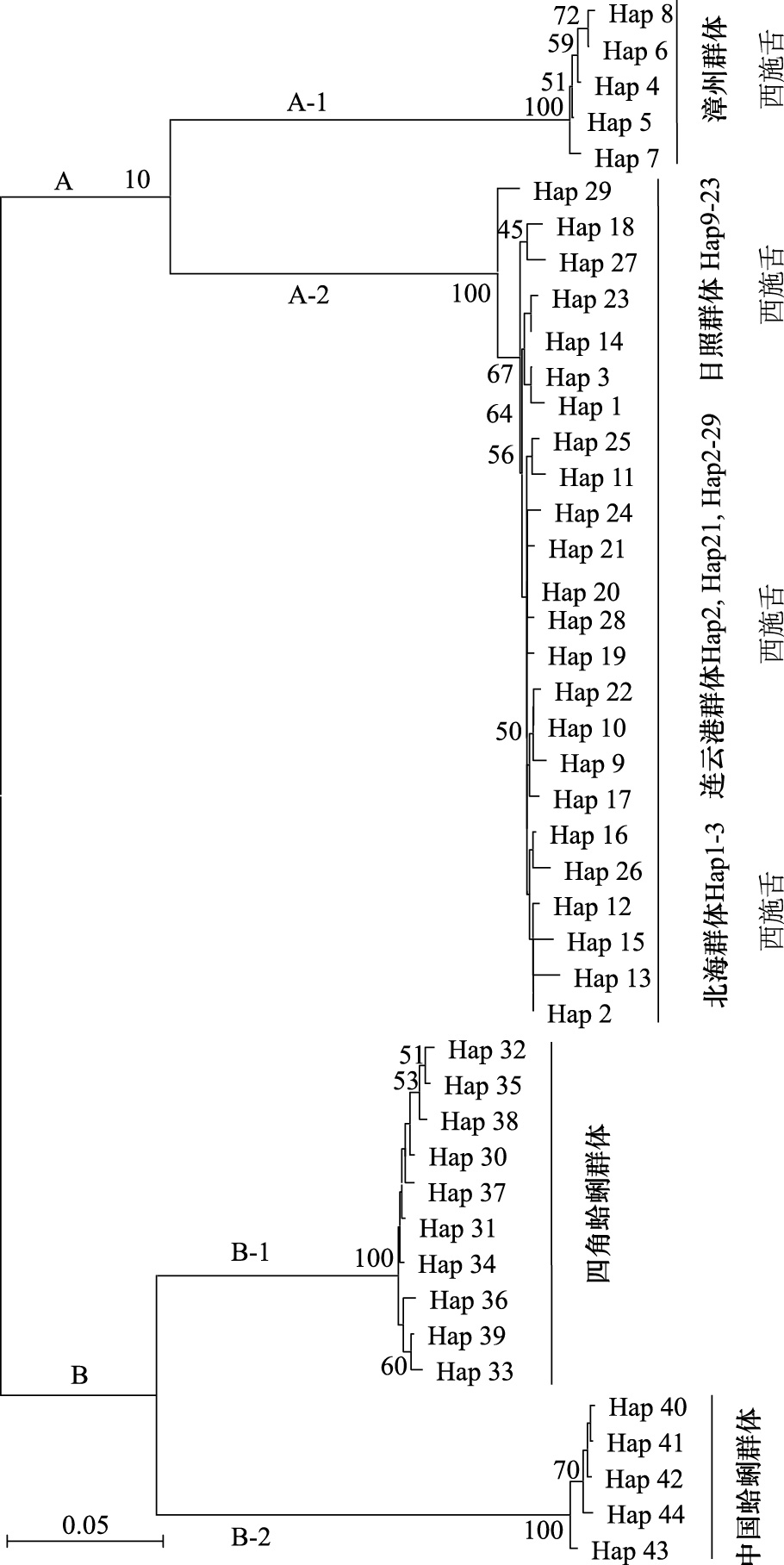
将44种Haps分为4组,日照、连云港、北海群体分为一组(Gp1),漳州群体分为另一组(Gp2),四角蛤蜊群体(Gp3)、中国蛤蜊群体(Gp4)各分为一组,计算组内和组间遗传距离(K2-P)。Gp2组内遗传距离(D)平均为0.006;Gp1组内D值平均为0.010;Gp3组内D值平均为0.009,Gp4组内D值平均为0.006。Gp1和Gp2间的D值为0.250—0.251,Gp3和Gp4间的D值为0.220,Gp1、Gp2与Gp3、Gp4之间的D值在0.304—0.365之间。计算Gp1、Gp2间的遗传距离(BG)与Gp1、Gp2组内遗传距离(WG)的比值(BG/WG),结果显示BG/WG值为25.1—41.8,Gp3、Gp4间的BG/BW比值为24.4—36.7(表 3)。日照与北海群体间的遗传距离只有0.009,远远小于漳州与其它群体的遗传距离。西施舌漳州群体和非漳州群体的BG/BW值在四角蛤蜊和中国蛤蜊的BG/BW比值范围内,说明西施舌漳州群体与日照、连云港、北海群体间的差异达到种间差异的水平。
| 组 Group | 群体 Population | Gp1 | Gp2 | Gp3 | WP | BG / WG | |||
| 连云港 | 北海 | 日照 | |||||||
| BG、WP:组间、组内遗传距离(between groups,within group genetic distances), BG / WG:组间与组内遗传距离比值(the ratio of BG/WG); Gp(group):组,A:Gp1与Gp2间,B:Gp3与Gp4间 | |||||||||
| Gp1 | 西施舌 | 连云港 | 0.010 | ||||||
| 北海 | 0.011 | 25.1—41.8A | |||||||
| 日照 | 0.012 | 0.009 | |||||||
| Gp2 | 漳州 | 0.250 | 0.250 | 0.251 | 0.006 | ||||
| Gp3 | 四角蛤蜊 | 0.304 | 0.311 | 0.305 | 0.328 | 0.009 | 24.4—36.7B | ||
| Gp4 | 中国蛤蜊 | 0.363 | 0.365 | 0.361 | 0.364 | 0.220 | 0.006 | ||
用AMOVA方法分析西施舌组间、群体间和群体内遗传变异来源,结果显示(表 4),95.55%的变异来源于组间,即漳州组与混合组(连云港、日照、北海)间,说明组间群体遗传分化很大,3.56%的变异来源于群体内,只有0.89%的变异来源于组内群体间,提示西施舌漳州群体发生了极大的遗传分化。漳州群体与日照、连云港和北海群体间的FST分别为0.967(P<0.01)、0.963(P<0.001)和0.976(P<0.01),说明漳州西施舌与日照、连云港和北海群体遗传分化很大,而北海与日照、北海与连云港间的FST分别为0.258(P<0.01)和0.191(P<0.01),日照与连云港群体间的FST为0.013(P>0.05),这两个群体间的遗传分化很小,两组间的FST为0.964(P<0.01),四角蛤蜊与中国蛤蜊间的FST为0.965(P<0.01)。
| 变异来源 Source of variation | 遗传变异元素 Variance components | 占总变异的百分数/% Percentage of variation | P |
| 组间Among groups | 47.59063 Va | 95.55 | <0.001 |
| 组内群体间Among populations Within groups | 0.44483 Vb | 0.89 | <0.001 |
| 群体内 Within populations | 1.77211 Vc | 3.56 | <0.001 |
基于nad5的 44种单倍型构建了西施舌不同群体与四角蛤蜊、中国蛤蜊的系统发生NJ树(图 2),由图 2可见,进化树拓扑结构形成2大支(A支,B支),A支又分为2小支(A-1,A-2),B也分为2小支(B-1,B-2)。日照、连云港、北海群体不同的单倍型交叉聚为支持率(100%)较高的一小支(A-2),漳州群体不同单倍型聚为另一小支(A-1)(100%)。中国蛤蜊、四角蛤蜊不同单倍型分别聚为两小支(B-1和B-2)(100%)。图 2可见,漳州西施舌与日照、连云港、北海西施舌相聚,形成清晰的两支,说明漳州西施舌与日照、连云港、北海西施舌发生了明显的分化。
|
| 图 2 基于nad5的蛤蜊科3种贝类的NJ树 Fig. 2 The NJ tree based on nad5 of three species in family Mactridae A支:西施舌4个群体,B支:四角蛤蜊、中国蛤蜊各1个群体,Hap: 单倍型 |
本人对日照、漳州西施舌线粒体全基因组组成及结构进行了比较[7],发现两地西施舌tRNAs组成、非编码区组成有明显差异,但是线粒体基因组结构只反映了2个个体间的差异,是否一个群体中其它不同的个体间的差异也达到如此高的水平,需要对更多的线粒体全基因组进行比对,但全基因组测序耗资颇大,为了能达到既经济、又能获得大量有效信息资料,因此,决定扩大取样量,将线粒体基因组个别基因或非编码区序列进行比较。本人前期工作中及国内其它学者对线粒体DNA的16S rRNA和 cox1、细胞色素b基因(cob)进行了群体间的比对。线粒体DNA编码的基因中,16S rRNA 和cox1常被用来进行高[10]、低[11]阶元种类群体差异分析。nad5核苷酸较16S和cox1变异稍大,用于遗传差异分析相对灵敏,此外,nad5分子量相对大,便于设计引物,因此,本研究选择了nad5作为标记,可放大差异,便于观察。目前,关于群体基因核苷酸差异值的种间、种内差异界限尚无标准,且不同的动物的相同基因保守性不同,因此,也不便定标准。但对于同一个基因的种间差异与种内个体间差异之比值相对稳定,可作为重要的参考指标。Hebert[12, 13]提出了10×规则:认为cox1在群体间的差异若是群体内差异的10倍以上,则认为两群体可能是2个种。前期工作中基于cox1的福建群体与非福建群体间的差异与福建群体内或非福建群体内的差异之比为17.8—23.5,远远大于10。基于nad5基因种间差异界限目前尚不明确,本研究基于nad5的漳州群体与非漳州群体间的差异与群体内个体间的差异之比为25.1—41.8,远远大于10。若参考基于cox1的10×规则,此差异也达到种间差异水平。为了有一个可参考的值,本研究计算了与西施舌同一个科(Mactridae)的蛤蜊属(Mactra)中国蛤蜊和四角蛤蜊间的差异与群体内差异比值(24.4—36.7),此比值与漳州群体、非漳州群体间与群体内之比值相似,nad5资料证明漳州西施舌已经分化为一个新种。
本研究基于nad5片段(与本研究采用的西施舌nad5片段同源)分析了双壳类巨蛎属、贻贝属、文蛤属、竹蛏属4个属属内种间遗传距离(D)(K2-P),结果显示巨蛎属8个物种间的D值为0.053—0.379,多数在0.150—0.269之间(表 5)。贻贝属4个物种间的D值为0.011—0.317,多数在0.218—0.291之间(表 5)。竹蛏属的大竹蛏(Solen grandis,NC_016665)和长竹蛏(Solen strictus,NC_017616)之间的D值为0.210。文蛤属的中华文蛤(Meretrix petechialis,NC_012767)和丽文蛤(Meretrix lusoria NC_014809)间的遗传距离为0.090。而西施舌混合群体(北海、日照和连云港)与漳州群体间的遗传距离为0.250—0.251(表 3),此值在巨蛎属8种贝类、贻贝属4种贝类种间遗传距离范围内;大于文蛤属2种贝类、竹蛏属2种贝类间的遗传距离,这从侧面证明西施舌漳州群体与其它3个群体的差异达到了种间差异水平。
| 样本序号 Sample No. | 物种名及其注册号 Species and submition No. | 样本序号 Sample No. | ||||||||||
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 9 | 10 | 11 | |||
| 1 | C. nippona NC_015248 | |||||||||||
| 2 | C. hongkongensis NC_011518 | 0.158 | ||||||||||
| 3 | C. ariakensis NC_012650 | 0.157 | 0.150 | |||||||||
| 4 | C. gigas NC_001276 | 0.245 | 0.212 | 0.219 | ||||||||
| 5 | C. angulata NC_012648 | 0.221 | 0.203 | 0.213 | 0.053 | |||||||
| 6 | C. sikamea NC_012649 | 0.216 | 0.206 | 0.206 | 0.154 | 0.156 | ||||||
| 7 | C. iredalei NC_013997 | 0.242 | 0.253 | 0.257 | 0.269 | 0.259 | 0.250 | |||||
| 8 | C. virginica NC_007175 | 0.368 | 0.371 | 0.378 | 0.378 | 0.378 | 0.342 | 0.379 | ||||
| 9 | M.galloprovincialis NC_006886 | |||||||||||
| 10 | M. edulis NC_006161 | 0.011 | ||||||||||
| 11 | M. trossulus NC_007687 | 0.218 | 0.227 | |||||||||
| 12 | M. californianus NC_015993 | 0.284 | 0.291 | 0.317 | ||||||||
西施舌连云港与北海群体、连云港与日照群体间均有共享nad5单倍型,说明3个群体间有基因交流现象,但漳州群体与上述3个群体间无共享单倍型,说明无基因交流现象,存在生殖隔离,其原因有待进一步研究;本研究分析了群体间nad5核苷酸组成差异显著性,发现西施舌漳州与非漳州群体的T、A、G 3种核苷酸含量差异极显著(P<0.01),而中国蛤蜊与四角蛤蜊的也有 3种核苷酸(T、C、G)含量差异极显著(P<0.01);AMOVA分析结果显示西施舌漳州群体与非漳州群体的FST=0.964,而中国蛤蜊与四角蛤蜊间的FST=0.965,两FST值接近。碱基组成显著差异及分化程度之高均证实漳州西施舌发生了极大的遗传分化。
我国学者孔令峰[1]对福建长乐、山东即墨、日照和江苏启东西施舌4个群体贝壳的12个变量数据分析结果显示,长乐群体与北方的3个群体发生了高度遗传分化;刘德经[2]对长乐和江苏如东野生西施舌群体贝壳的5个数量性状分析结果认为,2个群体西施舌的差异达到亚种水平。此资料为漳州西施舌分类地位的确定提供了形态学的证据;综合本研究结果、前期工作的核基因组DNA标记(ITS1[3]和RAPD[14])和线粒体基因组标记(cox1[5]和cob[6])及全基因组资料[7, 8]、结合上述形态学资料,提示中国福建西施舌与其它群体间的差异达到了种间差异水平,应该是腔蛤蜊属的新种。目前腔蛤蜊属只有两个种,即西施舌和古氏腔蛤蜊(Coelomactra cumingii),这2个种均为濒危物种[15],cox1分析结果[15]也认为福建西施舌与日照西施舌可能是姊妹种,因此,作者建议对福建西施舌进行重命名,并对其种质资源加强管理和保护,以确保此优良资源的可持续利用。
| [1] | Kong L F, Li Q, Qiu Z X. Genetic and morphological differentiation in the clam Coelomactra antiquata (Bivalvia: Veneroida) along the coast of China. Journal of Experimental Marine Biology and Ecology, 2007, 343(1): 110-117. |
| [2] | 刘德经, 朱善央. 福建与江苏西施舌群体形态差异研究. 南方水产, 2010, 6(2): 29-34. |
| [3] | 孟学平, 高如承, 申欣, 王帅, 程汉良, 董志国, 阎斌伦. 西施舌5个地理群体ITS1序列变异及系统发生分析. 生态学报, 2010, 30(20): 5555-5561. |
| [4] | Kong L F, Li Q. Genetic evidence for the existence of cryptic species in an endangered clam Coelomactra antiquata. Marine Biology, 2009, 156(7): 1507-1515. |
| [5] | 孟学平, 申欣, 张波, 程汉良, 田美, 郑雯雯. 西施舌Cyt b基因核苷酸差异分析. 水产科学, 2010, 29(9): 537-542. |
| [6] | 孟学平, 高如承, 申欣, 郑雯雯, 赵娜娜, 程汉良, 田美. 基于COI的中国西施舌DNA条形码. 水产科学, 2011, 30(10): 626-630. |
| [7] | Meng X P, Shen X, Zhao N N, Tian M, Liang M, Hao J, Cheng H L, Yan B L, Dong Z G, Zhu X L. Mitogenomics reveals two subspecies in Coelomactra antiquata (Mollusca: Bivalvia). Mitochondrial DNA, 2013, 24(2): 102-104. |
| [8] | 孟学平, 申欣, 赵娜娜, 田美, 梁猛, 郝珏, 程汉良, 阎斌伦, 董志国. 漳州西施舌线粒体基因组全序列: 腔蛤蜊属(Coelomactra) 存在新种的证据. 海洋学报, 2013, 35(3): 213-222. |
| [9] | Meng X P, Zhao N N, Shen X, Hao J, Liang M, Zhu X L, Chen H L, Yan B L, Liu Z P. Complete mitochondrial genome of Coelomactra antiquata (Mollusca: Bivalvia): The first representative from the family Mactridae with novel gene order and unusual tandem repeats. Comparative Biochemistry and Physiology Part D: Genomics and Proteomics, 2012, 7(2): 175-179. |
| [10] | Puslednik L, Serb J M. Molecular phylogenetics of the Pectinidae (Mollusca: Bivalvia) and effect of increased taxon sampling and outgroup selection on tree topology. Molecular Phylogenetics and Evolution, 2008, 48(3): 1178-1188. |
| [11] | Mao Y L, Gao T X, Yanagimoto T, Xiao Y S. Molecular phylogeography of Ruditapes philippinarum in the Northwestern Pacific Ocean based on COI gene. Journal of Experimental Marine Biology and Ecology, 2011, 407(2): 171-181. |
| [12] | Hebert P D, Penton E H, Burns J M, Janzen D H, Hallwachs W. Ten species in one: DNA barcoding reveals cryptic species in the neotropical skipper butterfly Astraptes fulgerator. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(41): 14812-14817. |
| [13] | Hebert P D, Stoeckle M Y, Zemlak T S, Francis C M. Identification of birds through DNA barcodes. PLoS Biology, 2004, 2(10): e312. |
| [14] | 孟学平, 王帅, 高如承, 申欣, 程汉良, 田美. 西施舌群体遗传结构及分化的RAPD分析. 海洋科学, 2011, 35(2): 6-9. |
| [15] | Ni L, Li Q, Kong L F, Huang S Q, Li L J. DNA barcoding and phylogeny in the family Mactridae(Bivalvia: Heterodonta): Evidence for cryptic species. Biochemical Systematics and Ecology, 2012, 44: 164-172. |
 2015, Vol. 35
2015, Vol. 35