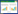文章信息
- 张洪海, 窦海龙, 陈磊, 沙未来, 赵俊杰
- ZHANG Honghai, DOU Hailong, CHEN Lei, SHA Weilai, ZHAO Junjie
- 中国狼不同地理种群遗传多样性及系统进化分析
- Genetic diversity and phylogenetic analysis of wolves from different geographical regions of China
- 生态学报, 2015, 35(6): 1869-1881
- Acta Ecologica Sinica, 2015, 35(6): 1869-1881
- http://dx.doi.org/10.5846/stxb201305221141
-
文章历史
- 收稿日期:2013-05-22
- 修订日期:2014-04-25
2. 北京师范大学生命科学学院, 北京 100875
2. College of Life Science, Beijing Normal University, Beijing 100875, China
灰狼简称狼,属于食肉目(Carnivora)、犬科(Canidae)、犬属(Canis),由于狼食谱宽、适应性强,曾在地球上广泛分布[1]。狼在我国曾一度分布于除海南省、南中国海诸岛屿以及台湾省外的广大地区,但由于人为捕杀和生境破坏,现仅集中分布于人口较为稀少的内蒙古、新疆、青海、西藏等地区[2]。我国关于狼的研究主要集中在宏观生态学方面,关于狼种群遗传多样性及系统分类地位的研究非常少,并且在狼亚种的划分上还存在着众多争议:很多学者认为中国狼或蒙古狼(Canis lupus Chanco)与欧亚狼(Canis lupus Lupus)是同一个亚种[3];中国动物志引述Pocock等和Ellerman 等的研究结论认为分布于中国境内的狼仅有蒙古狼(C. l. chanco)一个亚种[4, 5];王秀辉应用RAPAD方法将中国狼划分为5个亚种:云贵和东南集群亚种,东北河北群体亚种、甘肃群体亚种、内蒙古群体亚种和新疆群体亚种[6];罗理杨等通过测定中国各个地区狼线粒体控制区部分序列得出分布于青海、西藏、云南的狼种群属于西南种群亚种,东北种群与华北种群作为一个亚种,内蒙古及新疆种群归属不能确定[7]。国内对狼遗传多样性的研究未见报道,这对于野生狼种群的管理及保护非常不利。
线粒体DNA(Mitochondrial DNA,mtDNA)不会发生遗传重组,较核DNA有更高的突变率,因此被公认为用来作为研究动物遗传进化的标记物质[8]。线粒体细胞色素b(Cytochrome b,Cyt b)基因全长大约在1100—1200 bp之间,并且不同物种间序列长度差异较大,但是在核苷酸组成上却具有偏好性。由于Cyt b基因中含有突变和保守区,并且存在较慢和较快的进化的密码子位点,所以Cyt b基因是对动物系统进化研究的较好的分子标记。犬科动物线粒体控制区(Control region displacement loop,D-loop)全长1240—1360 bp,其中存在3个保守序列框(Conserved sequence block):CSB Ⅰ、CSB Ⅱ和CSB Ⅲ[9]。起始位点位于CSB Ⅰ区5′端的第一高变区(Hypervariable region,HVR Ⅰ)序列变异速率较快,常被用来进行种群遗传结构及系统地理分析[10]。近些年来,保护生物学研究已经深入到分子水平,要求使用分子实验技术从大量动物样本中提取DNA以获取动物的遗传信息。在采集分析样品的过程中,传统的取样方法(通过杀死动物来获取新鲜的组织或捕获动物抽取血液)对于野外动物种群特别是濒危珍稀动物种群较难实现。因此本研究采集了来自青海、新疆及内蒙、吉林4个地区55个样品,且多数样品采用非损伤取样的方法获得。通过分子生物学手段成功获得44个个体线粒体D-Loop区HVR Ⅰ序列和40个线粒体Cyt b部分序列,对其进行遗传学分析,探讨各地种群遗传多样性高低并分析其系统进化地位。
1 材料与方法 1.1 样品采集本研究于2011年12月至2012年6月分别在青新疆阿勒泰地区、内蒙古西旗、内蒙古达赉湖自然保护区、吉林图门市共采集样品55份,且样品多数来自于野外捕捉的圈养狼(样本详细资料见表 1)。所有粪便样品一律置于-20 ℃冰箱中保存,肌肉组织置于-80 ℃冰箱中保存,采集皮毛组织均为自然风干后置于常温干燥环境下保存,血液样品加入抗凝剂后使用生物低温采样箱运送至实验室-80℃冰箱中保存。
|
地理种群 Geographic group | 样品来源地 Sample origin site | 样品采集地 Sample collecting site | 样品数 Sample number | 样品类型 Sample type |
| 青海种群 | 青海刚察 | 中国科学院西高所 | 6 | 皮毛 |
| Qinghai population | 青海湖区 | 青海动物园 | 10 | 粪便 |
| 新疆种群 | 新疆阿勒泰地区 | 新疆青河县富疆狼园 | 8 | 粪便 |
| Xinjiang population | 新疆青河县 | 新疆青河县富疆狼园 | 3 | 血液 |
| 内蒙种群 | 内蒙达赉湖自然保护区 | 内蒙达赉湖自然保护区 | 3 | 肌肉组织 |
| Inner mongolia population | 内蒙西旗 | 哈尔滨野狼湖狼园 | 3 | 粪便 |
| 吉林种群 | 内蒙呼伦贝尔 | 内蒙呼伦贝尔 | 20 | 粪便 |
| Jilin population | 吉林图门市 | 哈尔滨野狼湖狼园 | 2 | 粪便 |
| 总计Total | 55 |
由于在分子生物学研究中,应用毛皮或者粪便样品为DNA 来源时,获得的DNA 含量相对于其他组织样品较低,可能会对后续实验造成影响以致无法获得准确的结果,因此针对这种情况,本研究采用重复提取的方法,对同一样品分别独立操作两次,获得的DNA分别置于-20℃冰箱保存并用于后续实验。粪便中总DNA的提取使用粪便提取试剂盒(QIAamp DNA Stool Mini Kit,USA)。血液,毛皮及肌肉组织DNA的提取使用血液及组织提取试剂盒(QIAamp DNeasy Blood & Tissue Kit,USA)。
1.2.2 PCR扩增狼线粒体控制区HVR Ⅰ引物采用犬科动物mtDNA D-loop第一高变区的序列长度为582 bp通用引物对[11]:
L15404: 5′-CTCTTGCTCCACCATCAGC-3′
H16118: 5′ -AAACTATATGTCCTGAAACC-3′
根据Cyt b内部的保守位点,选择性的扩增了位于细胞色素b后半部分长度大约767 bp的序列,其下游包含一段tRNApro序列。引物设计使用软件Primer 5.0及Oligo 7.5。所得引物序列为:
L14666:5′ -AGAGTGAATCTGAGGCGGCTT-3′
H15432:5′ -GCTTTGGGTGCTGATGGTGG-3′
狼控制区HVRⅠ和Cyt b 区PCR反应体系相同: Taq 酶0.25 μL (Takara),10 × buffer 4 μL,10 mmol/LdNTPs (Takara)3 μL,前/后引物各0.8 μL,BSA (Takara)0.25 μL,以及模板DNA 1 μL,加灭菌超纯水至50 μL。
狼控制区第一高变区(HVR Ⅰ)PCR 反应条件为:94 ℃ 预变性4 min; 接下来40个循环(94 ℃ 30 s,53 ℃ 45 s,72℃ 45 s),最后72 ℃ 延伸5 min。Cyt b区PCR反应条件为:94 ℃ 预变性4 min,接下来40个循环(94 ℃ 30 s,57 ℃ 45 s,72 ℃ 45 s),最后72 ℃ 延伸5 min。
1.2.3 PCR产物的回收及测序PCR产物采用1.0%的常规琼脂糖凝胶电泳检测,上样量为5 μL PCR产物加1 μL 6×Loading Buffer(Takara),100 V电压电泳30 min,凝胶经EB染色后置于紫外下观察将目的条带切下,用凝胶回收试剂盒(QIAquick Gel Extraction Kit,USA)回收纯化后送至北京三博远志测序公司进行测序。
1.2.4 序列分析测序所得序列结果在Genbank上经Blast比对后确定序列来源于狼。用Clustal X软件进行DNA序列排列[12],并辅以人工校对,以去掉扩增的Cyt b序列中所包含的线粒体tRNApro序列。基于Kimura双参数距离模型(Kimura-2-Parameter,K2P),通过MEGA 5.0计算各地理种群狼控制区HVR Ⅰ和Cyt b序列变异碱基比例、变异类型、转换和颠换比(R)及遗传距离等基础数据[13]。基于狼Cyt b序列,利用遗传距离和分子钟的分歧时间计算公式为t=D/2a(t为分歧时间,D为遗传距离,a为分子钟)并结合已经校正的哺乳动物细胞色素b序列的每百万年2%分子钟来计算中国各地计算各地理种群进化分歧时间[14, 15]。基于狼控制区HVR Ⅰ序列用DnaSP 5.0[16]软件划分单倍型,将单倍型序列上传至Genbank,单倍型序列登陆号见表 4,并计算核苷酸多样性(Nucleotide diversity,Pi)和单倍型多样性 (Haplotype diversity,Hd),进而推算各地种群遗传多样性高低。为了研究中国各地狼的系统发育,结合本研究中发现的中国狼线粒体HVRⅠ单倍型及从Genbank中下载已有中国狼研究中20个mtDNA控制区序列,然后重新划分单倍型,用邻接法 (Neighbour-joining,NJ)以 Genbank下载的郊狼线粒体控制区序列(Canis latrans,Genank登录号:NC_008093)为外群构建单倍型的系统发生树,其中各节点的支持率根据各序列bootstrap[17]1000次重抽样后数据计算。根据单倍型变异位点,应用软件Network 4.610 (http://www.fluxus-engineering.com/sharenet.htm)构建基于Median-Joining法简约网络(Statistical parsimony network)关系图,以期与已有研究进行比较。为了解中国狼在世界范围内的系统进化地位,下载了123个世界各地狼线粒体控制区单倍型(附表),并以8个郊狼单倍型序列(附表)为外群使用NJ法构建系统发育树。
2 结果 2.1 线粒体控制区HVRⅠ基因序列及其变异实验获得44个中国狼线粒体控制区第一高变区583 bp序列,其中内蒙西旗,内蒙达赉湖自然保护区,青海湖区,青海刚察,新疆青河县样品全部扩增测序成功(表 2)。通过比对44个线粒体控制区HVR Ⅰ序列发现共有变异位点51个,占分析序列长度的8.76%,插入缺失位点11个,碱基替换位点40个(图 1)。研究表明,通过扩增总DNA的方法有可能扩增出核基因组中线粒体基因插入拷贝[18, 19, 20],即线粒体假基因(Numts)。由于线粒体假基因和线粒体基因分歧度较大,这时应考虑青海种群是否存在这样的假基因。将青海种群线粒体控制区HVR Ⅰ基因分别与Genbank中下载先前研究的中国青海狼序列(FJ032363)以及中国内蒙狼序列(GQ374438)比对,发现青海狼与中国西藏狼序列相似度超过97.5%,而与中国内蒙狼序列比对结果与表 4结果相符,证明青海种群确实与其他种群存在较大的分歧。其中转换/颠换之比R为5.71,说明序列突变未达到饱和状态[21]。
|
| 图 1 中国部分地区狼线粒体第一高变区多态位点分布图 Fig. 1 Variable sites distribution of HVR Ⅰ in mtDNA of China Wolf |
各地区种群的线粒体控制区HVR Ⅰ基因碱基组成分析:A,T,C,G平均含量分别是26.29%、30.24%、16.62%和26.85%,其中A+T含量为56.53%,符合脊椎动物线粒体基因组成特点,低于犬科动物线粒体全序列A+T含量(60.3%—60.5%)[22],表明HVR Ⅰ相对于其他保守的线粒体基因组区段来说较易突变。
|
地理种群 Geographic group | 样品来源地 Sample origin site | 编号 Number | 样品数 Sample number | 测序成功样品数 Successful sequencing sample No. | |
| 细胞色素b Cytochrome b | 控制区高变区 Hypervariable region | ||||
| 青海种群 | 青海刚察 | G | 6 | 5 | 6 |
| Qinghai population | 青海湖区 | HQ | 10 | 7 | 10 |
| 新疆种群 | 新疆阿勒泰地区 | A | 8 | 7 | 7 |
| Xinjiang population | 新疆青河县 | Q | 3 | 3 | 3 |
| 内蒙种群 | 内蒙达赉湖自然保护区 | D | 3 | 2 | 3 |
| Inner mongolia population | 内蒙西旗 | X | 3 | 2 | 2 |
| 内蒙呼伦贝尔 | H | 22 | 13 | 12 | |
| 吉林种群 Jilin population | 吉林图门市 | T | 2 | 1 | 1 |
| 总计Total | 8 | 55 | 40 | 44 | |
实验共获得40个狼线粒体Cyt b序列(表 2)。将扩增测序所得到40条的长度为767 bp的序列经Clustal X[2]比对去掉含有的tRNApro序列后得到582 bp的Cyt b部分序列。经分析后发现,没有发现碱基插入及缺失现象,表明这些Cyt b序列来自于线粒体,而不是核基因中的线粒体假基因[23]。使用MEGA 5.0分析这40条Cyt b序列发现其碱基组成为:T 29.0%、C 29.6%、A 29.9%、G 11.6%。582个碱基位点中共发现变异位点31个,占分析序列的5.33%,且变异位点全部为简约信息位点,未发现单核苷酸变异位点。40个个体总的转换颠换比(R)为28.68,各地理种群转换颠换比均大于2.0,与动物线粒体碱基替换模式结论相符[18]。狼Cyt b部分序列G+C含量为41.2%,与脊椎动物基因序列G+C含量在40%—50%之间的结论相符[24]。密码子第一位点G+C含量(50.2%)明显高于第二位点(40.2%)和第三位点(38.2%)的含量,表明密码子第二三位点开始倾向于富含AT(表 3)。
|
碱基类型 Base type | 全序列 Complete sequence | 第一位点 The 1st position | 第二位点 The 2nd position | 第三位点 The 3rd position |
| 胸腺嘧啶(Thymine,T) | 29.0 | 22.0 | 44.0 | 21.0 |
| 胞嘧啶(Cytosine,C) | 29.6 | 29.7 | 24.2 | 34.7 |
| 腺嘌呤(Adenine,A) | 29.9 | 28.0 | 21.1 | 40.5 |
| 鸟嘌呤(Guanine,G) | 11.6 | 20.5 | 10.8 | 3.5 |
使用软件DnaSP 5.0对44个成功获得目的个体样本片段序列进行分析,共确定16个单倍型,其中青海种群内部青海刚察与青海湖区种群共享3个单倍型(QH2,QH3,QH4),新疆种群内部新疆阿勒泰地区及青河县种群共享1个单倍型XJ2,内蒙种群内部内蒙达赖湖自然保护区,西旗及呼伦贝尔种群共享2个单倍型NM2和NM4,吉林图门与内蒙呼伦贝尔种群共享1个单倍型NM3(表 4)。吉林图门与内蒙呼伦贝尔种群之间共享1个单倍群,说明两个种群之间表现为共同的母系来源或存在基因交流。
|
单倍型 Haplotypes | 多态位点 11111111 1111111133 3333334445 2334555568 9903356666 7778889912 4556685894 3077067898 3605853458 0253684564 3080545686 | 个体数 Individuals |
Genbank登录号 Genbank Accession No. | 编号 No. |
| NM1 | ACTGTGGTTG TATTCTTCTT ATCTAACGAC | H10 | KC414564 | W1 |
| NM2 | CCTACTCTTA | H1—H4,H6,H7,H11,H12,X1,D1, | KC414565 | W2 |
| NM3 | .......... .G........ ........G. .........G | D3 | KC414566 | W3 |
| NM4 | .......... .C........ ........G. .........G | H5,T | KC414567 | W4 |
| NM5 | ........C. .......... G......... T....C..CG | H8,X2,D2 | KC414568 | W5 |
| QH1 | T......... .G........ ........G. .........G | H9 | KC414569 | W6 |
| QH2 | GT....AG.. ..CCTCCTCC .C.C....GT ...G.CTGC. | G1 | KC414570 | W7 |
| QH3 | GT...AAG.. ..CCTCCTCC .C.C....GT ...G..T.C. | G2,HQ9 | KC414571 | W8 |
| QH4 | GT....AG.. ..CCTCCTCC .C.C....GT ...G.CT.C. | G3,G5,G6,HQ4,HQ6,HQ7 | KC414572 | W9 |
| QH5 | GT....AG.. .GCCTCCTCC .C.CG...GT ...G.CT.C. | G4,HQ2,HQ10 | KC414573 | W10 |
| QH6 | GT....AG.. .GCCTCCTCC .C.CGGT.GT ...G.CT.C. | HQ1 | KC414574 | W11 |
| QH7 | GT....AG.. ..CCTCCTCC .C.CG...GT .T.G.CT.C. | HQ3 | KC414575 | W12 |
| QH8 | GTGA..AA.C A.CCTCCTCC .C.CT...GT ...G.CT.C. | HQ5 | KC414576 | W13 |
| XJ1 | GT....AG.. ..CCTCCTCC .C.C....GT .T.G.CT.C. | HQ8 | KC414577 | W14 |
| XJ2 | .......... .G........ ....G..A.. .......... | A1,A2,A3,A7 | KC414578 | W15 |
| XJ3 | .......... .......... ..T.G..A.. ..C.TCT.CG | A4,A6,Q1,Q2,Q3 | KC414579 | W16 |
| ....C...C. .......... G......... T....C..CG | A5 |
由于吉林图门种群数量小于3,不符合Tajima 检验[25]要求至少3条同源原序列的要求,所以选取符合条件的青海,内蒙和新疆种群进行遗传多样性分析。使用DnaSP 5.0软件对分布在中国青海、内蒙、新疆3个种群44个个体测得的线粒体控制区HVR Ⅰ进行遗传分析后得出,中国种群单倍型多态性Hd为0.897,核苷酸多态性Pi为0.02278。新疆种群的核苷酸多样性为0.942,明显的高于另外两个种群,说明新疆种群遗传多样性较高,遗传资源丰富。通过Tajima′s D 和Fu and Li′s D 值检测结果无显著差异(P>0.10)表明这3个中国狼种群相对于中性进化的歧异度没有明显的偏离(表 5)。
|
地理种群 Geographic group | 个体数 Number | 单倍型多样性 Haplotype diversity (Hd) | 核苷酸多样性 Nucleotide diversity (Pi) | 核苷酸差异多样性 Average number of nucleotide differences(K) | Tajima 检验 Tajima′s D | Fu and Li 检验 Fu and Li′s D |
|
青海种群 Qinghai population | 16 | 0.842±0.075 | 0.00453±0.00108 | 2.64167 | -1.49407* | -1.70774* |
|
新疆种群 Xinjiang population | 10 | 0.644±0.101 | 0.00942±0.00177 | 5.55556 | 0.56153* | -0.13773* |
|
内蒙种群 Inner mongolia population | 17 | 0.647±0.120 | 0.00511±0.00150 | 3.01471 | -0.27259* | 0.19016* |
| 总体 Total | 43 | 0.897±0.028 | 0.02278±0.00122 | 13.23699 | 1.05547* | -0.29042* |
| * P >0.10无显著差异 | ||||||
从Genbank下载的中国各地区的基于线粒体控制区序列20个灰狼的线粒体控制区序列(附表)。由于下载的序列长度存在差异,所以我们将所有序列剪辑后得到长度为582 bp的线粒体控制区序列。基于582 bp的序列重新划分中国狼单倍型,发现28个单倍型,并重新对其进行编号,其中NM2(W2),NM4 (W4),QH2 (W7),QH3 (W8),QH4 (W9),XJ3 (W16)与以前研究中发现的单倍型完全匹配(附表)。
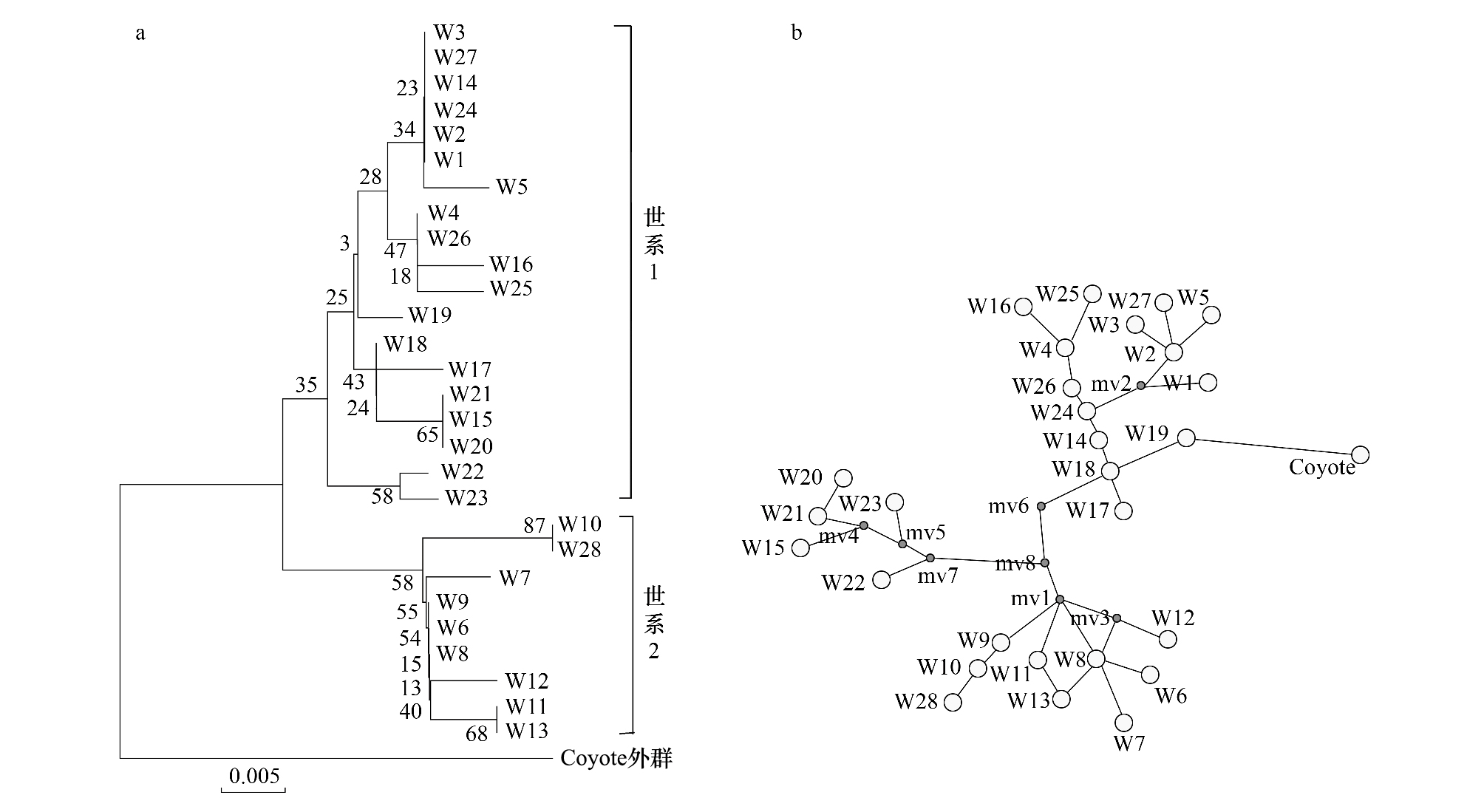
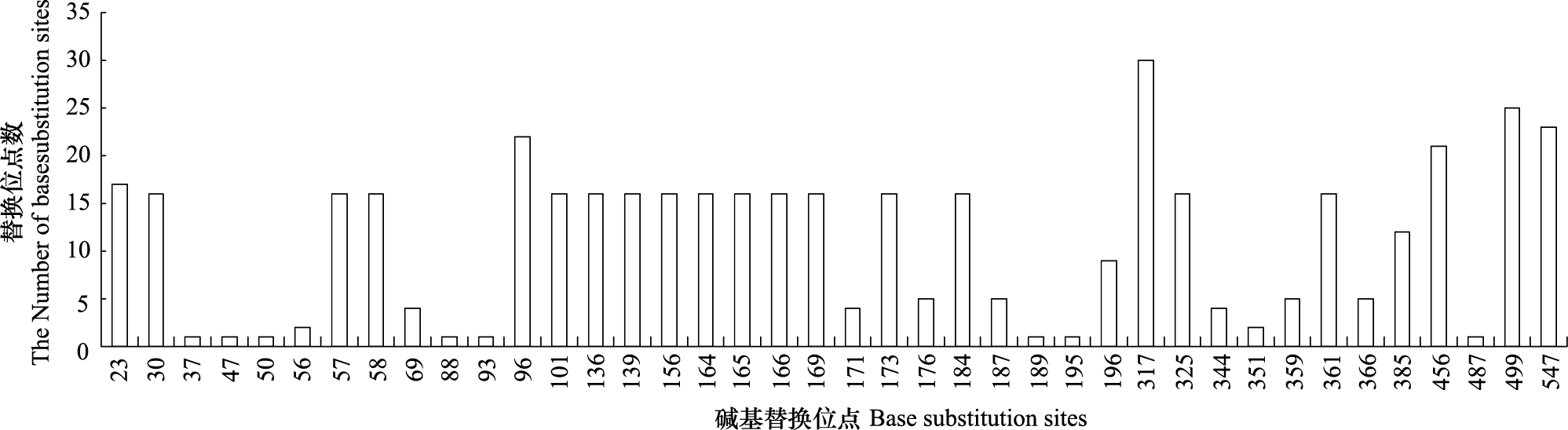
|
| 图 2 基于中国狼mtDNA控制区28单倍型序列构建的NJ树(a)和基于中国狼mtDNA控制区28单倍型序列构建的网络结构图(b) Fig. 2 Neighbor-joining tree of 28 mtDNA haplotypes based on mtDNA D-loop of China wolf (a)and Network of 28 mtDNA haplotypes based on mtDNA D-loop of China wolf (b) 圆圈代表的是单倍型,红点代表的是假定的中间单倍型,线代表一个替位突变,圈的大小与单倍型频率的大小成正比 |
通过对Genbank下载的20条中国狼线粒体控制区序列与比研究发现的单倍型对后重新定义28个单倍型。通过比对后发现本研究发现的线粒体控制区单倍型与Tsuda,K等[26]在蒙古乌兰巴托附近发现的狼单倍型共享3个单倍型,与罗理杨[7]等发现的单倍型共享4个单倍型(附表)。使用NJ法构建28个中国狼单倍型系统进化树(图 2),除青海种群外,其他地区或亚地区的的狼单倍型并没有聚集在一起,表明地区或亚地区狼在遗传学方面没有独特的遗传结构[7]。通过进化树不难看出,青海种群单倍型与除青藏高原外的单倍型基于遗传距离分属两个不同的世系,这与罗理杨等所得的结果一致[7]。把得到的28个中国狼单倍型与Genbank上下载的123个灰狼单倍型序列构建NJ树发现,位于青藏高原及其周边地区的狼与喜马拉雅山脉西侧的印度狼亲缘关系较近并为一支,而广布于中国其他地区的狼单倍型与欧亚狼亲缘关系较近,且没有表现出明显的与地理分布有关的差异(图 3)。
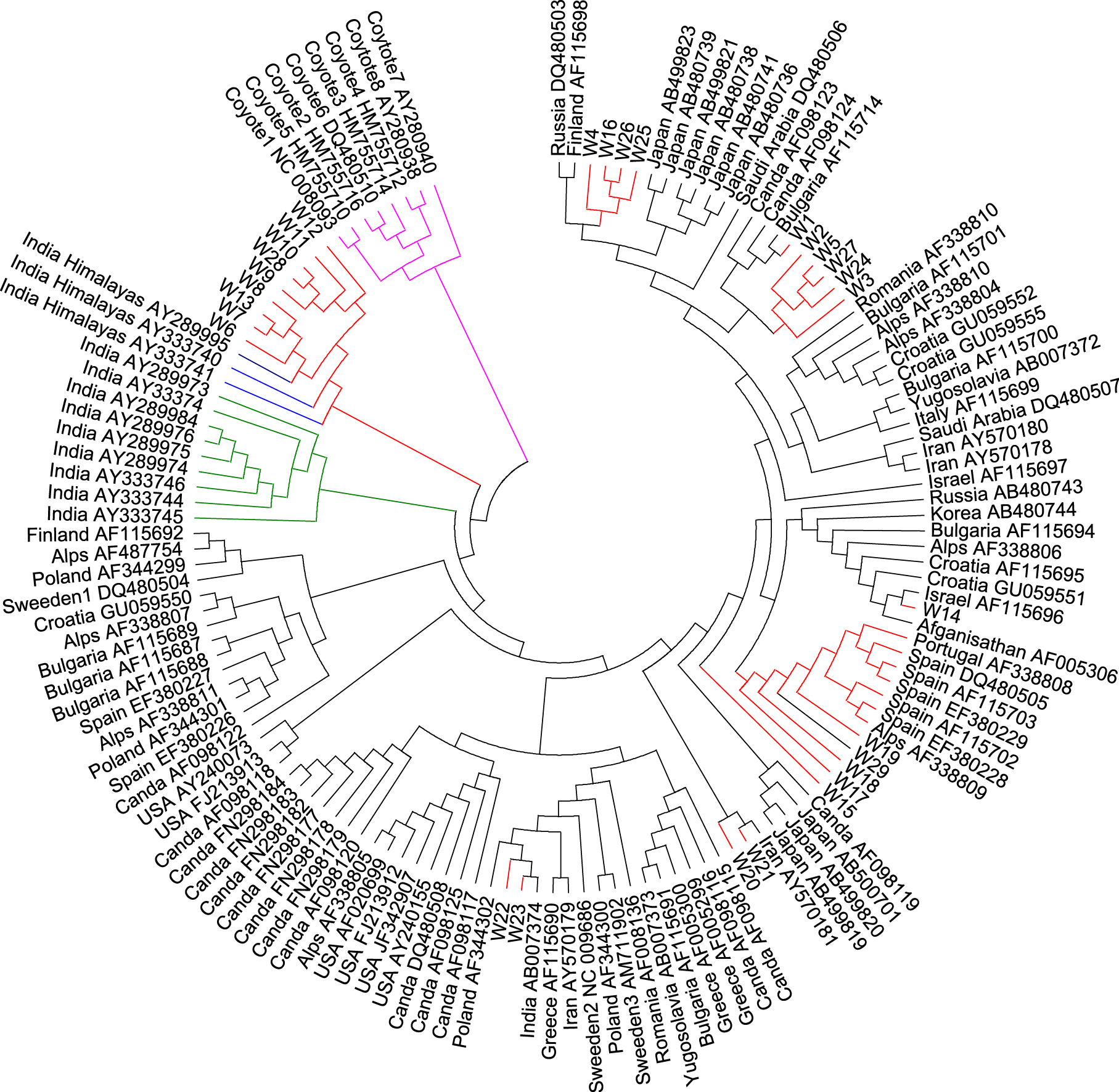
|
| 图 3 基于从Genbank下载的225bp的线粒体控制区序列,使用K2P碱基替换模型构建的环形NJ树 Fig. 3 Neighbor-joining original tree,computed using a nucleotide substitution model with Kimura 2 parameter distances,illustrating the phylogenetic relationship among wolf 225bp mtDNA control region haplotypes registered in GenBank 红线表示中国狼单倍型,粉红线代表郊狼外群,绿线代表分布于印度Canis.lupus.pallipes亚种 |
基于Cyt b序列,利用已经校正的哺乳动物细胞色素b序列的每百万年2%分子钟来计算中国各地狼进化分歧时间。基于Kimura双参数距离模型,计算中国各地狼的遗传距离,其中内蒙、新疆与吉林种群遗传距离数值范围为0.002—0.006 MYA Bp,其种群分歧时间大约为0.05—0.15 MYA Bp。青海狼与本研究其他地理种群遗传距离较远,遗传距离为0.043—0.048,由此得出青海种群与其他地理种群分歧时间大约在1.08—1.2 MYA Bp,巧合的是这与青藏高原受地质作用的急速隆起时间(0.9—1.1 MY)相近[27]。
3 讨论狼分布范围广,野外活体采样难度很大。本文尝试从粪便中成功的提取总DNA,为今后通过使用粪便样品对狼进行遗传进化研究提供了依据。本研究从55个样品中成功的提取和扩增出出44个个体的线粒体HVR Ⅰ和40个Cyt b序列,其中未成功提取出DNA的样品全部为粪便样品,而且主要为内蒙呼伦贝尔地区样品。未提取出来的原因可能是采集的粪便样品时间过长,样品中的DNA降解或者粪便样品中含有可抑制后续反应的酶[28]。
对种群进行遗传多样性评估,不仅有利于了解种群结构及动态,而且为制定种群保护和管理措施提供依据。通常考虑一个种群遗传多样的高低主要是通过对比种群内部单倍型间的遗传距离和核苷酸多态性,然而由于核苷酸多态性充分考虑到了单倍型在种群内的比例,所以一般使用核苷酸多态性来衡量一个种群遗传多样性的高低[29]。本研究利用线粒体HVR Ⅰ序列对中国4个地理狼种群进行遗传多样性检测,发现平均核苷酸多态性为2.27%,与世界其他地区狼相比遗传多样性是较高的,如Randi等[30]通过分析mtDNA控制区序列发现保加利亚狼核苷酸多样性为0.26%,Pilot[31]等在东欧十多个国家采集到643只狼样品,并成功地鉴定出21个线粒体DNA单倍型,得出其核苷酸多样性为1.7%,Tomislav等[32]分析了91只狼的线粒体控制区序列得出克罗地亚狼遗传多样性为1.8%,从而说明中国狼相对于世界其他国家和地区来说线粒体D-loop区存在丰富的多样性。
通过构建的中国狼分子系统发育树可以看出,青海种群与西藏狼明显的并为一支,而其他地区的狼则没有按照地理区划分成不同的谱系,同时各个谱系间含有共同的单倍型,说明其遗传基础一致,存在着基因交流,这与北美狼亚种分化的研究是一致的[33]。t检验也显示了吉林,内蒙,新疆种群之间的遗传距离无显著差异,而与青海种群差异明显。其中青海种群拥有较多的单倍型,且通过世界狼系统进化树不难看出,青海种群拥有的单倍型较为特殊,不与除青藏高原及周边地区外的中国其他地区的狼单倍型相似,而是与喜马拉雅山山脉西部位于印度等国家地区的狼接近,通过青海种群的分布区域考虑,青海种群位于青藏高原东北部的青海湖周边地区,北面为昆仑山和祁连山,东面为横断山脉,平均海拔在3000m以上,山地可以达到4000m以上,这些天然的地理屏障阻碍了青海种群与青藏高原外的其他地理种群进行基因交流,长期的地理隔离造成青海种群独立进化形成了隔离种群。而青海种群与西藏狼,云南狼共享单倍型,说明了青海种群与西藏云南种群在起源进化上关系比较近。
长久以来,研究认为中国狼只有一个亚种即C.l.Chanco[4, 5]。Tsuda K等[26]通过研究狼的3个亚种(C. l. lupus,C. l. pallipes and C. l. Chanco) 发现中国狼(C. l. Chanco)的线粒体控制区变异较其他两个亚种较大,而我们通过对比线粒体控制区部分序列发现,除青海地区序列与另外两个亚种序列差异较大外,其他地区种群单倍型与另外两个亚种差别不大。Ramesh K Aggarwal 等[34]利用Tsuda K 等[26]从蒙古国乌兰巴托发现的狼的线粒体单倍型与印度西部喜马拉雅山脉狼进行比较,得出印度西部喜马拉雅山脉狼是与蒙古狼是完全不同的,并推测其是世界上最古老的狼世系,这与的研究结果一致。基于线粒体DNA的研究结果,Dinesh等[35]认为可以将位于印度喜马拉雅山脉附近狼种群从中国狼或蒙古狼亚种(C. l. Chanco)中剥离出来一起划归为西藏亚种(C. l. langier)。通过本文研究,并结合以前形态学及解剖学上的研究结果[36]推测青藏高原种群的狼可能与印度喜马拉雅山附近的狼同属于西藏狼亚种(C. l. langier),当然这只是推测,具体的情况还需要从解剖生理学,基因组学乃至古生物学的证据来证实。同时通过构建的系统进化树(图 3)也能看到除青藏高原及周边地区的狼外中国其他地区的狼单倍型广布于欧亚大陆乃至北美大陆狼分支中,说明这些地区的狼起源进化关系较为复杂。
4 结论基于线粒体控制区部分序列测定及分析,研究结果表明中国狼较世界其他国家地区拥有较高的遗传多样性。系统进化分析表明中国狼可以分为两个世系,且位于青藏高原及其周边地区的狼与中国其他地区的狼遗传结构差异较大,且其遗传多样性较其他地区低。推测其在进化上与中国其他地区狼较早出现分歧,并形成独立进化种群。利用线粒体Cyt b部分序列推算各地理种群的进化分歧时间得出,位于青藏高原种群与其他地理种群分歧时间大约在1.1 MY前,与青藏高原受地质作用急速隆起时间相近。
致谢: 中国科学院西北高原生物所、青海动物园、哈尔滨野狼湖狼园、新疆富疆狼园、内蒙古达赉湖自然保护区提供研究样品,东北林业大学高中信教授、曲阜师范大学刘广帅同学为采集样品提供指导与帮助,特此致谢。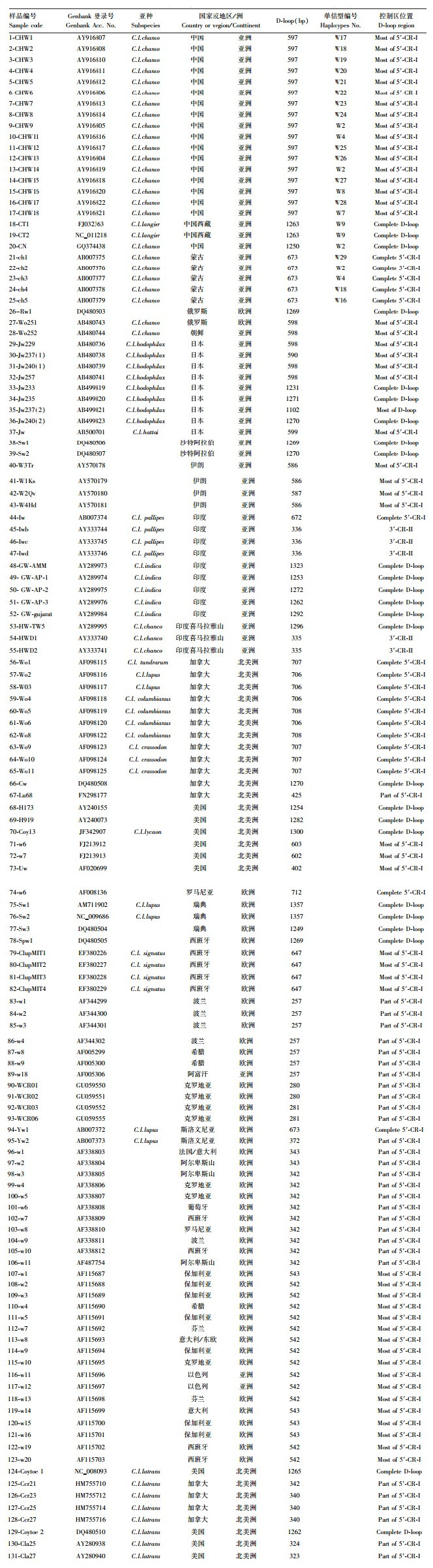 |
| [1] | Wilson D E,Reeder D M. Mammal Species of the World(MSW): A Taxonomic and Geographic Reference. 3rd ed. Baltimore: Johns Hopkins University Press, 2005. |
| [2] | 张洪海,张培玉,王振龙,车启来. 世界狼的分布、种群数量及保护现状.曲阜师范大学学报,1999,25(1):101-103. |
| [3] | Busch R H. The Wolf Almanac. New York: The Lyons Press:1993, 241-241. |
| [4] | Ellerman J R, Morrison-Scott R C.S. Checklist of Palaearctic and Indian Mammals 1758-1960. London: British Museum (Natural History) publications,1951. |
| [5] | Pocock R I. The Fauna of British India, Including Ceylon and Burma, Mammalia(2 vols, Primates and Carnivora Only). London: Taylor and Francis, 1940 |
| [6] | 王秀辉,高中信,于学伟,崔岩. 狼亚种分化的RAPD分子证据.东北林业大学学报,2000,28(5):110-113. |
| [7] | 罗理杨,金煌,杨公社.中国狼的种内系统发育关系与亚种分化.东北林业大学学报, 2003,31(5):81-83. |
| [8] | Avis J C,Arnold J,Ball R M,Birmingham E,Lamb T,Nigel J E,Reba C A,Saunders N C. Intraspecific phylogeography: The mitochondrial DNA bridge between population genetics and systematic. Annual Review of Ecology and Systematic,1987,18(1): 489-522. |
| [9] | Kim K S, Lee S E, Jeong H W, Ha J H. The complete nucleotide sequence of the domestic dog (Canis familiars) mitochondrial genome. Molecular Phylogenetics and Evolution, 1998, 10(2): 210- 220. |
| [10] | Lunt D H,Whipple L E,Hyman B C. Mitochondrial DNA variable number tandem repeats(VNTRs): utility and problems in molecular ecology. Molecular Ecology, 1998, 7(11):1441-1455. |
| [11] | ROERDINK A R,ALDSTADT J H. Sensitive method for the determination of roxarsone using solid-phase micro extraction with multi-detector gas chromatography. Journal of Chromatography A, 2004, 1057(1/2): 177-183. |
| [12] | Jeanmougin F,Thompson J D,Gouy M,Higgins D G,Gibson T J. Multiple sequence alignment with Clustal X. Trends in Biochemical Sciences, 1998, 23 (10): 403-405. |
| [13] | Tamura K,Dudley J,Nei M,Kumar S. MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version. Molecular Biology and Evolution, 2007, 24 (8):1596-1599. |
| [14] | Li W H,Gokobori T,Nei M. Pseudogenes as a paradigm of neutral evolution. Nature, 1981, 292(5820): 237-239. |
| [15] | Wayne R K, Geffen E, Girman D J, Koepfli K P, Lau LM, Marshall C R. Molecular systematics of the canidae. Systematic Biology, 1997, 46(4): 622-653. |
| [16] | Librado P,Rozas J. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data. Bioinformatics, 2009, 25 (11): 1451-1452. |
| [17] | Felsenstein J. Confidence limits on phylogenies: An approach using the bootstrap.Evolution,1985, 39(4): 783-791. |
| [18] | Irwin D M,Koeher T D, Wilson A C. Evolution of the cytochromeb gene of mammals. Journal of Molecular Evolution, 1991, 32(2): 128-144. |
| [19] | Woischnik M, Moraes C T. Pattern of organization of human mitochondrial pseudogenes in the nuclear genome . Genome Research, 2002, 12 (6): 885-893. |
| [20] | Tourmen Y, Baris O, Dessen P, Jacques C, Malthiery Y, Reynar P. Structure and chromosomal distribution of human mitochondrial pseudogenes. Genomics, 2002, 80 (1): 71-75. |
| [21] | Knight A,Mindell D P. Substitions bias,weighting of DNA sequence evolution,and the phylogenetic positions of Fea's viper. Systems Biology,1993,42(1): 18-31. |
| [22] | 陈磊,张洪海,马建章. 蒙古狼线粒体基因组序列分析及其系统发育地位.生态学报,2010,30(6):1463-1471. |
| [23] | Stadelmann B,Jacobs D S,Schoeman C, Ruedi M. Phylogeny of African Myotis bats (Chiroptera, Vespertilionidae) inferred from cytochrome b sequences. Acta Chiropterologica, 2004, 6(2): 177-192. |
| [24] | Sueoka N. On the genetic basis of variation and heterogeneity of DNA base composition. Proceedings of the National Academy of Sciences of the United States of America, 1962, 48(4): 582-592. |
| [25] | Tajima F .Statistical method for testing the neutral mutation hypothesis by DNA polymorphism. Genetics,1989,123(3): 585-595. |
| [26] | Tsuda K, Kikkawa Y, Yonekawa H, Tanabe Y. Extensive interbreeding occurred among multiple matriarchial ancestors during the domestication of dogs: evidence from interand intraspecies polymorphisms in the d-loop region of mitochondrial DNA between dogs and wolves. Genes & Genetic System, 1997, 72(4): 229-238. |
| [27] | Sun J, Liu T. Stratigraphic evidence for the uplift of the Tibetan Plateau between -1.1 and -0.9 myr ago. Quaternary Resarch,2000,54(3): 309-320. |
| [28] | 刘艳华,张明海. 基于线粒体Cty b 基因的西藏马鹿种群遗传多样性研究.生态学报,2011,31(7): 1976-1981. |
| [29] | Neigel J E,Avise J C. Application of a random walk model to geographic distributions of animal mitochondrial DNA variation.Geneties,1993,135 (4): 1209-1220. |
| [30] | Randi E, Lucchini V, Christensen M F, Mucci N, Funk S M, Dolf G, Loeschke V. Mitochondrial DNA variability in Italian and East European wolves: detecting the consequences of small population size and hybridization. Conservation Biology, 2000,14(2): 464-473. |
| [31] | Pilot M, Jedrzejewski W, Branicki W, Sidorovich W E, Jedrzejewska B, Stachura K, Funk S M. Ecological factors influence population genetic structure of European grey wolves. Molecular Ecology, 2006,15(14): 4533-4553. |
| [32] | Gomer či c ' T, Sindi či c ' M, Galov A, Arbanasi c ' H, Kusak J, Kocijan I, Gomer či c ' 1 M D, Huber D. High genetic variability of the grey wolf (Canis lupus L.) population from croatia as revealed by mitochondrial DNA control region sequences. Zoological Studies, 2010,49(6):816-823. |
| [33] | Vila C, Amorim J R, Leonard J A, Posada D, Castroviejo J, Petrucci-Fonseca F, Crandall K A, Ellegren H, Wayne R K. Mitochondrial DNA Phylogeography and Population History of the GreyWolf Canis lupus. Molecular Evolution, 1999, 8(12): 2089-2103. |
| [34] | Aggarwal R K, Ramadevi J, Singh L. Ancient origin and evolution of the Indian wolf: evidence from mitochondrial DNA typing of wolves from Trans-Himalayan region and Pennisular India. Genome Biology, 2003, 4: 6-6. |
| [35] | Sharma D K, Maldonado J E, Jhala Y V, Fleischer R C. Ancient wolf lineages in India. Biology letters, 2004, 271(S3): S1-S4. |
| [36] | Pocock R I, F R S, F Z S. The races of Canis lupus. Proceedings of the Zoological Society of London, 1935, 105(3): 647-686. |
 2015, Vol. 35
2015, Vol. 35