文章信息
- 夏允, 徐玲琳, 杨柳明, 杨智杰, 范跃新, 杨玉盛
- XIA Yun, XU Linglin, YANG Liuming, YANG Zhijie, FAN Yuexin, YANG Yusheng
- 模拟氮沉降对中亚热带米槠天然林土壤解磷微生物群落和功能潜力的影响
- Effects of simulated nitrogen deposition on soil microbial community and functional potential of phosphate-solubilizing microorganisms in a subtropical Castanopsis carlesii forest
- 生态学报. 2024, 44(4): 1727-1736
- Acta Ecologica Sinica. 2024, 44(4): 1727-1736
- http://dx.doi.org/10.20103/j.stxb.202303210543
-
文章历史
- 收稿日期: 2023-03-21
- 网络出版日期: 2023-11-27
2. 福建师范大学福建省植物生理生态重点实验室, 福州 350007;
3. 福建师范大学地理研究所, 福州 350007
2. Fujian Provincial Key Laboratory for Plant Ecophysiology, Fujian Normal University, Fuzhou 350007, China;
3. Institute of Geography, Fujian Normal University, Fuzhou 350007, China
磷是限制陆地生态系统初级生产力的关键养分。特别是热带亚热带地区, 由于土壤高度风化, 大部分磷被铁铝氧化物固定, 导致该区域的森林生态系统普遍存在磷限制[1]。此外, 亚热带地区还是全球最严重的氮沉降区域之一, 大量的氮输入改变了生态系统的氮磷平衡, 进一步加重亚热带森林土壤的磷限制[2—3]。因此, 厘清氮沉降下土壤磷周转过程及其调控机制, 对预测亚热带森林生产力如何适应全球变化具有重要意义。
土壤微生物在磷循环过程中发挥关键作用, 被认为是驱动土壤磷周转的主导因素[4]。其中, 能够促进有机磷分解或无机磷解吸附, 进而提高土壤磷有效性的微生物被称为解磷微生物[5]。目前已知的细菌、放线菌和真菌等均具有溶解磷的功能。如假单胞菌属(Pseudomonas)、伯克霍尔德菌属(Burkholderia)、芽孢杆菌属(Bacillus)、肠杆菌属(Enterobacter)、欧文氏菌属(Erwinia)、沙雷菌属(Serratia)、不动杆菌属(Acinetobacter)和根瘤菌属(Rhizobium)等均具有溶解磷的功能[6—7]。与细菌类似, 部分真菌(如曲霉菌(Aspergillus)和青霉菌(Penicillium))同样具有活化磷的能力[8]。已有研究发现氮沉降会改变微生物群落结构, 如基于全球数据的Meta分析发现长期氮添加会降低土壤微生物生物量和群落多样性, 对微生物产生负面效应[9—10]。此外, 控制实验也发现氮沉降导致功能微生物(如固氮细菌、氨氧化细菌和丛枝菌根真菌)丰度显著下降[11—13], 但也有研究发现氮添加并未改变土壤微生物群落结构[14]。说明氮沉降对土壤微生物群落结构的影响还存在不确定性。
通过合成磷酸酶促进有机磷矿化是解磷微生物驱动土壤磷转化和提高土壤磷有效性的重要途径之一[15]。由于不同磷酸酶的编码基因并不一致, 携带不同编码基因的解磷微生物合成磷酸酶的能力和种类存在差异[16]。一般认为, 酸性磷酸酶主要来源于真菌和植物根系, 是酸性土壤中参与矿化磷的主要酶;碱性磷酸酶主要来自碱性土壤微生物, 在多种有机磷化合物中裂解酯键, 从而释放磷酸盐[17]。已有研究表明磷酸酶的产生取决于土壤有机质含量、pH值、植物和微生物氮磷养分需求等[18—20]。比如, 众多研究发现施氮后土壤磷酸酶的增加与氮沉降加剧土壤磷限制有关[21—22]。但也有研究发现氮沉降对磷酸酶的影响因土壤养分状况而异, 氮输入抑制了受氮限制土壤中磷酸酶的分泌进而加重了磷限制, 但提高了受磷限制土壤中磷酸酶含量来缓解了磷限制[23]。在亚热带低磷土壤中, 氮沉降是否会提高磷酸酶的含量以及增加哪些磷酸酶的含量来缓解磷限制仍不清楚。此外, 已有研究主要通过测定酸性磷酸酶、碱性磷酸酶以及磷酸二酯酶等酶活性的变化表征土壤磷矿化潜力, 而事实上植酸酶、磷酸单脂酶等也是驱动有机磷矿化的关键水解酶, 对土壤磷转化十分重要。但由于难以直接观测, 且测定过程受环境变化以及实验方法的干扰, 导致较难揭示土壤中不同磷酸酶活性的真实变化。随着生物信息学工具的兴起, Langille等(2013)提出的PICRUSt功能预测手段提供了新的思路, 该方法基于系统发育(从高通量测序技术中获得)和功能相联系的假设, 预测宏基因组的功能组成[24], 可用于揭示土壤酶编码基因的相对变化。尽管PICRUSt功能预测无法表征土壤酶活性的绝对数量, 但可提供土壤磷转化酶编码基因的种类和相对丰度等信息, 间接表征土壤磷酸酶的种类和活性, 有助于更为清晰地认识外界环境干扰下土壤磷循环的微生物机理。
本研究选择亚热带森林顶级群落的建群种米槠天然林为研究对象, 依托长期氮沉降观测研究平台, 通过16S rRNA和ITS测序, 结合PICRUSt功能预测手段, 探究氮添加对亚热带森林土壤解磷微生物群落丰度和功能潜力的影响。旨在揭示氮沉降背景下, 驱动土壤磷转化的关键微生物类群以及促进有机磷分解的主要磷酸酶种类, 为理解氮沉降下驱动亚热带森林土壤磷循环的微生物类群和机制提供知识储备。
1 材料和方法 1.1 研究区概况试验样地位于福建三明森林生态系统与全球变化国家站(26°11′N, 117°8′E)。以米槠天然林为研究对象, 设置长期氮沉降观测样地。样地本底条件基本一致, 采用随机区组设计。根据亚热带大气氮沉降的背景值约38 kg N hm-2 a-1[25], 设置对照(CT, 0 kg N hm-2 a-1)、低氮(LN, 40 kg N hm-2 a-1)和高氮(HN, 80 kg N hm-2 a-1)3个处理, 每个处理4个重复。每个样方大小为20 m×20 m, 样方之间用PVC板进行阻隔, 间隔10 m以上。于2012年11月开始, 每月月初以喷洒NH4NO3溶液的方式施氮。为消除外加水的影响, 对照样方喷洒等量去离子水。
于2018年4月用土钻钻取0—10 cm和10—20 cm土层的土样, 每块样地随机取5个点混匀, 将采集的土壤样品用冰袋储存运回实验室, 将鲜土过2 mm筛后分成三份, 一部分放在-20℃冰箱保存, 用于提取DNA, 一部分置于4℃冰箱低温储存, 用于测定DOC和矿质氮等指标;另一部分自然风干, 用于测定土壤磷组分和其他基本理化指标。
1.2 土壤理化性质的测定土壤pH采用玻璃电极在悬浮液(土水比1∶2.5)中测定。土壤有机碳(SOC)和全氮(TN)采用高温氧化法利用碳氮元素分析仪(Elementar Vario EL III;Elementar, Langenselbod, Germany)测定。可溶性有机碳(DOC)采用去离子水浸提(土水质量比为4∶1)利用TOC分析仪(Multi N / C 3100, Analytikjena, German)测定。采用0.5 mol/L的KCl浸提土样后用连续流动分析仪(Skalar San++, Skalar, 荷兰)测定土壤铵态氮(NH4+-N)和硝态氮(NO3--N)含量。
1.3 土壤磷组分参照Hedley(1982)等制定以及Tiessen和Moir(1993)改进的顺序提取方法来测定土壤磷组分[26—27]。依次用转化为碳酸氢盐形式(2 cm × 3 cm)的阴离子交换树脂膜条, 0.5 mol/L NaHCO3、0.1 mol/L NaOH、1 mol/L HCl溶液浸提, 并将0.1 mol/L NaOH溶液经过超声振荡浸提得到NaOHs溶液。使用流动分析仪(Skalar San++, Skalar, 荷兰)测定每种提取物中无机磷(Pi)的浓度。对于0.5 mol/L NaHCO3, 0.1 mol/L NaOH和1 mol/L HCl的浸提液, 用浓硫酸和高氯酸消煮得到各浸提物的总磷(TP), 有机磷(Po)则为每种提取物的TP与Pi的差值。Klotzbücher等(2019)发现, 只有前两个提取步骤(使用HCO3形式的阴离子交换树脂和碳酸氢盐)代表了不同生物利用度的磷组分[28]。因此, 将磷组分划分为树脂磷(Resin-P)、NaHCO3提取磷(NaHCO3-P)、总可提取有机磷(TPo, NaHCO3-Po、NaOH-Po、NaOHs-Po和HCl-Po的总和)和总可提取无机磷(TPi, NaHCO3-Pi、NaOH-Pi、NaOHs-Pi和HCl-Pi的总和)。
1.4 DNA提取、PCR扩增和Illumina MiSeq测序从-20℃储存的冻土中分别称取0.25 g样品, 使用MoBio PowersoilTM DNA分离试剂盒(MO BIO Laboratories, Carlsbad, CA, 193 USA)提取微生物DNA, 然后使用1%的琼脂糖凝胶电泳和Thermo Nano Drop One分光光度计(ND-2000, Thermo Fisher Scientific, Waltham, MA, USA)对提取的DNA进行可视化和定量。以稀释10倍的DNA为模板进行PCR扩增。使用引物组341F/806R和ITS1F/ITS2R分别扩增细菌16S rRNA基因和真菌ITS区。扩增过程:94℃初始变性5 min, 然后94℃ 30 s, 52℃ 30 s, 72℃ 30 s, 在72℃下延伸10 min, 最后在4℃下保持, 直到停止。每个样本3个重复, 并将同一样本的PCR产物进行混合, 使用Illumina Miseq平台(Majorbio Bio-Pharm Technology Co.206 Ltd., Shanghai, China)对PCR产物进行处理和量化, 并使用QIIME程序对基因序列进行定量分析[29]。
1.5 数据处理将测序得到的基因序列进行质控和过滤, 去除长度<200 bp的序列, 分别使用SILVA和UNITE数据库鉴定细菌和真菌分类[30]。在97%相似度水平下将优化序列聚类为操作分类单元(Operational taxonomic unit OUT), 使用Microsoft Excel 2016软件统计每个OTU的相对丰度信息以及微生物在门和属水平的分类信息。采用PICRUSt功能预测探索和检验施氮处理后磷酸酶基因丰度的变化, 筛选后的BLAST基因片段通过比对KEGG数据库进行功能注释[31]。使用R包“psych”分别计算环境因子与解磷微生物、酶活性的相关性, 并借助“ggplot2”包制图。文中主要借助R.4.2.1软件和Origin 2018软件完成统计分析和制图。
2 结果和分析 2.1 土壤理化性质施氮后土壤pH呈下降趋势(P>0.05, 表 1), 与CT相比, LN和HN处理在0—10 cm土层均降低3.72%, 在10—20 cm土层分别降低0.97%和1.45%。施氮后SOC、TN、DOC含量在不同土层均无显著差异(P>0.05)。但施氮显著增加了两个土层的NO3--N含量, 且HN>LN>CT(P<0.05), 而NH4+-N含量未发生显著变化。
| 指标 Indicator |
0—10 cm | 10—20 cm | |||||
| CT | LN | HN | CT | LN | HN | ||
| pH (H2O) | 4.03±0.05ab | 3.88±0.18b | 3.88±0.06b | 4.14±0.08a | 4.1±0.11ab | 4.08±0.05ab | |
| 土壤有机碳Soil organic carbon/(g/kg) | 33.46±7.7a | 35.41±5.25a | 31.37±4.14a | 15.26±1.54b | 15.38±4.72b | 14.37±2.67b | |
| 可溶性有机碳 Dissolved organic carbon/(mg/kg) |
92.05±34.23a | 96.76±25.85a | 75.68±24.5ab | 42.22±7.84b | 31.34±12.39b | 33.93±8.79b | |
| 总氮Total nitrogen/(g/kg) | 2.36±0.31a | 2.41±0.51a | 2.34±0.4a | 1.09±0.07b | 1.06±0.17b | 1.07±0.13b | |
| 铵态氮Ammonium nitrogen/(mg/kg) | 24.16±3.57a | 27.79±8.82a | 23.21±7.39a | 18.5±3.41a | 16.62±4.63a | 24.84±9.91a | |
| 硝态氮Nitrate nitrogen/ (mg/kg) | 1.68±0.46cd | 5.7±1.09b | 7.91±1.69a | 1.22±0.21d | 3.32±0.49c | 6.91±0.61ab | |
| CT:对照处理Control treatment;LN:低氮处理Low nitrogen treatment;HN:高氮处理High nitrogen treatment;同行不同字母表示处理间差异显著(P<0.05), 表中数据为平均值±标准差(n=4) | |||||||
施氮后, 0—10 cm土层Resin-P、NaHCO3-P和TPo均显著降低(P<0.05, 表 2), 与CT处理相比, LN处理下分别降低23.53%、25.62%、27.63%, HN处理下分别降低48.24%、24.35%、20.05%。同时, TPi在不同处理间没有显著差异。施氮后不同磷组分在10—20 cm土层的不同处理间均没有显著差异(P>0.05)。
| 磷组分 P fraction |
0—10 cm | 10—20 cm | |||||
| CT | LN | HN | CT | LN | HN | ||
| 树脂提取磷 Resin extractable phosphorus |
1.70±0.14a | 1.30±0.14b | 0.88±0.07c | 0.30±0.03d | 0.34±0.02d | 0.36±0.13d | |
| NaHCO3提取磷 NaHCO3 extractable phosphorus |
18.15±1.87a | 13.50±1.49b | 13.73±2.00b | 7.88±0.45c | 7.65±1.78c | 7.43±0.45c | |
| 总可提取有机磷 Total extractable organic phosphorus |
69.49±6.12a | 50.29±2.82b | 55.56±3.53b | 35.90±3.35c | 33.11±5.65c | 37.27±3.29c | |
| 总可提取无机磷 Total extractable inorganic phosphorus |
42.46±2.06a | 32.29±7.23ab | 31.91±4.35ab | 26.15±1.78b | 25.53±6.48b | 24.02±7.26b | |
| 同行不同字母表示处理间差异显著(P<0.05); 表中数据为平均值±标准差(n=4) | |||||||
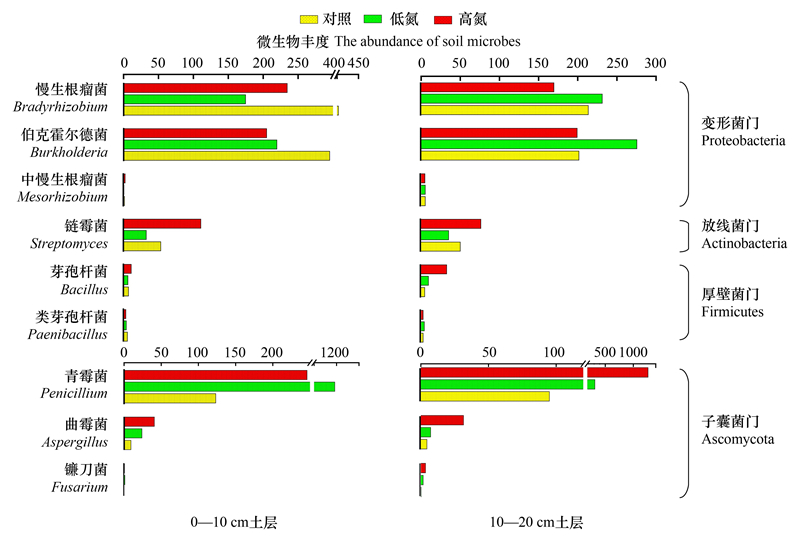
基于16S rRNA和ITS的高通量测序手段来探究氮沉降对土壤解磷微生物群落组成和丰度的影响(图 1)。结果显示米槠天然林土壤中解磷细菌主要包括变形菌门(慢生根瘤菌属(Bradyrhizobium)、伯克霍尔德菌属(Burkholderia)、中慢生根瘤菌属(Mesorhizobium))、放线菌门(链酶菌属(Streptomyces)和厚壁菌门(芽孢杆菌属(Bacillus)、类芽孢杆菌属(Paenibacillus)), 解磷真菌主要有子囊菌门(青霉菌属(Penicillium)、曲霉菌属(Aspergillus)、镰刀菌属(Fusarium))。施氮后不同种属的解磷微生物丰度变化趋势并不一致, 在0—10 cm土层, 与CT相比, HN下慢生根瘤菌和伯克霍尔德菌分别降低41.85%、69.70%, 而链霉菌、青霉菌和曲霉菌分别增加107.98%、99.59%、340.54%;在10—20 cm土层, HN下慢生根瘤菌丰度降低20.61%, 链霉菌、芽孢杆、青霉菌、曲霉菌和镰刀菌分别增加了0.53、6.22、12.29、6.00倍和13.00倍。
|
| 图 1 氮沉降对土壤解磷微生物丰度的影响 Fig. 1 Effects of nitrogen deposition on the relative abundance of soil phosphate-solubilizing microorganisms |
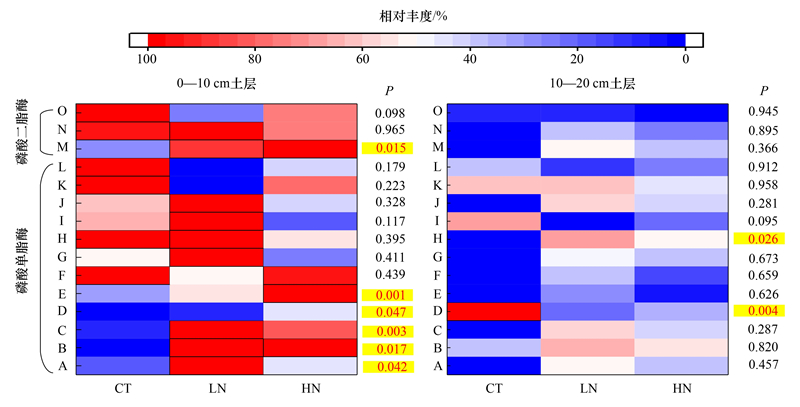
基于16S rRNA观测数据, 利用微生物OTU(最小操作分类单元)和FASTA(核苷酸序列)数据通过PICRUSt处理获取KEGG Ortholog(KO)信息(表 3), 然后通过KEGG数据库来获取编码磷酸酶类群的功能基因的变化。结果表明米槠天然林土壤中共含有15种磷酸酶的功能基因, 其中包括12种磷酸单脂酶和3种磷酸二酯酶, 施氮显著影响不同种类磷酸酶功能基因的相对丰度(图 2)。其中, 与CT相比, LN和HN处理下0—10 cm土层的酸性磷酸酶(A)、碱性磷酸酶(B)、4-植酸酶(C)、组氨醇磷酸酶(D)和3-植酸酶(E)这5种磷酸单脂酶以及二酯酶中的磷脂酶(M)功能基因相对丰度均显著增加(P<0.05)。在10—20 cm土层, HN处理下组氨醇磷酸酶和磷酸甘油酸磷酸酶(H)基因丰度显著降低(P<0.05)。
| KO编号 KO number |
基因 Gene |
酶的国际系统命名 International System of enzyme nomenclature |
磷酸酶的组成 Composition of phosphatase |
| K22913 | FIG 4 | 3.1.3.- | 5″-磷酸核糖核酸磷酸酶5″-phosphoribostamycin phosphatase |
| K01113 | phoD | 3.1.3.1 | 碱性磷酸酶D.alkaline phosphatase D |
| K01087 | otsB | 3.1.3.12 | 海藻糖磷酸酶trehalose-phosphatase |
| K05602 | hisN | 3.1.3.15 | 组氨醇磷酸酶histidinol-phosphatase |
| K22223 | pgp | 3.1.3.18 | 磷酸甘油酸磷酸酶phosphoglycolate phosphatase |
| K01078 | PHO | 3.1.3.2 | 酸性磷酸酶acid phosphatase |
| K01092 | suhB | 3.1.3.25 | 肌醇-1(或4)-单磷酸酶myo-inositol-1(or 4)-monophosphatase |
| K01093 | appA | 3.1.3.26 3.1.3.2 | 4-植酸酶/酸性磷酸酶4-phytase / acid phosphatase |
| K01096 | pgpB | 3.1.3.27 | 磷脂酰甘油磷酸酶B.phosphatidylglycerophosphatase B |
| K02203 | thrH | 3.1.3.3 2.7.1.39 | 磷酸丝氨酸/高丝氨酸磷酸转移酶phosphoserine/homoserine phosphotransferase |
| K02566 | nagD | 3.1.3.5 | 5′-核苷酸5′-nucleotidase |
| K01083 | — | 3.1.3.8 | 3植酸酶3-phytase |
| K01114 | plc | 3.1.4.3 | 磷脂酶C.phospholipase C |
| K18696 | GDE1 | 3.1.4.46 | 甘油磷酰二酯磷酸二酯酶glycerophosphoryl diester phosphodiesterase |
| K01127 | GPLD1 | 3.1.4.50 | 糖基磷脂酰肌醇磷脂酶D.glycosylphosphatidylinositol phospholipase D |
| K01523 | hisE | 3.6.1.31 | 磷酸核糖基-ATP焦磷酸水解酶phosphoribosyl-ATP pyrophosphohydrolase |
|
| 图 2 PICRUSt分析土壤磷酸酶丰度的变化 Fig. 2 PICRUSt analysis of soil phosphatase abundance changes A:酸性磷酸酶Acid phosphatase;B:碱性磷酸酶D Alkaline phosphatase D;C:4-植酸酶/酸性磷酸酶4-phytase / acid phosphatase;D:组氨醇磷酸酶Histidinol-phosphatase;E:3植酸酶3-phytase;F:肌醇-1(或4)-单磷酸酶Myo-inositol-1(or 4)-monophosphatase;G:5″-磷酸核糖核酸磷酸酶5″-phosphoribostamycin phosphatase;H:磷酸甘油酸磷酸酶Phosphoglycolate phosphatase;I:磷酸丝氨酸/高丝氨酸磷酸转移酶Phosphoserine / homoserine phosphotransferase;J:磷脂酰甘油磷酸酶B Phosphatidylglycerophosphatase B;K:海藻糖磷酸酶Trehalose-phosphatase;L:5′-核苷酸5′-nucleotidase;M:磷脂酶C Phospholipase C;N:甘油磷酰二酯磷酸二酯酶Glycerophosphoryl diester phosphodiesterase;O:糖基磷脂酰肌醇磷脂酶D Glycosylphosphatidylinositol phospholipase D。CT:对照处理Control treatment;LN:低氮处理Low nitrogen treatment;HN:高氮处理High nitrogen treatment |
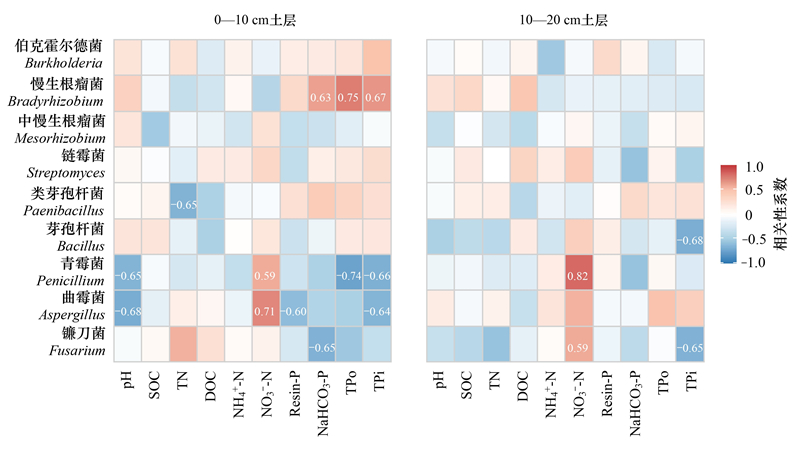
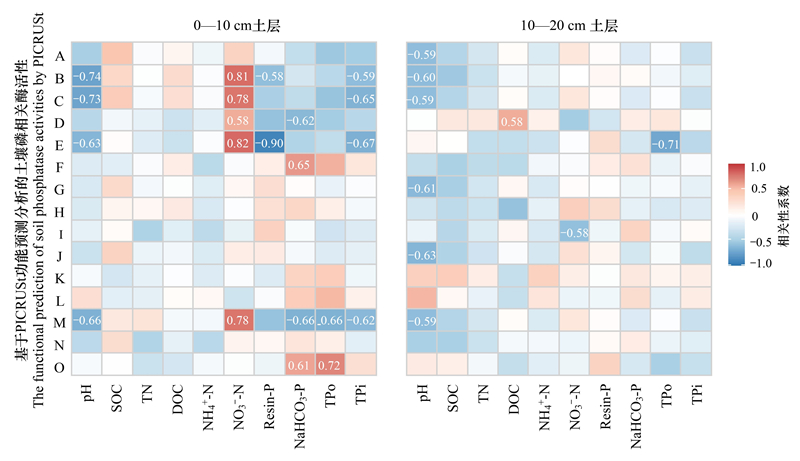
通过相关分析检验了影响土壤解磷微生物丰度和功能基因变化的关键因素, 发现施氮后显著变化的解磷真菌(青霉菌和曲霉菌)与NO3--N呈显著正相关(图 3, P<0.05), 与土壤pH和土壤磷组分(Resin-P、NaHCO3-P、TPo和TPi)呈负相关, 而解磷细菌(伯克霍尔德菌和慢生根瘤菌)与土壤磷组分呈正相关。同时, 施氮后显著变化的编码磷酸酶(碱性磷酸酶、4-植酸酶、酸性磷酸酶、组氨醇磷酸酶、3植酸酶、磷脂酶)基因丰度与NO3--N呈显著正相关, 但与pH、土壤磷(Resin-P和TPi)呈显著负相关(图 4, P<0.05)。此外, 相比10—20 cm土层, 0—10 cm土层微生物丰度和基因变化对土壤养分变化的响应更敏感。
|
| 图 3 解磷微生物丰度与环境因子的相关关系 Fig. 3 Correlation between phosphorus-solubilizing microbial abundance and environmental factors SOC:土壤有机碳Soil organic carbon;TN:总氮Total nitrogen;DOC:可溶性有机碳Dissolved organic carbon;NH4+-N:铵态氮Ammonium nitrogen;NO3--N:硝态氮Nitrate nitrogen;Resin-P:树脂磷Resin extractable phosphorus;NaHCO3-P:NaHCO3提取磷NaHCO3 extractable phosphorus;TPo:总可提取有机磷total extractable organic phosphorus;TPi:总可提取无机磷Total extractable inorganic phosphorus;图中标注数值为r值, 表明差异显著(P<0.05) |
|
| 图 4 土壤磷酸酶基因丰度与环境因子的相关关系 Fig. 4 Correlation between soil phosphatase gene abundance and environmental factors 图中标注数值为r值, 表明差异显著(P<0.05) |
施氮后表层土壤Resin-P、NaHCO3-P和TPo均显著降低, 这与亚热带毛竹林的研究结果相似[32], 施氮降低了NaHCO3-P和有机磷占总磷的比例, 并认为Po是维持亚热带地区P供应的基础, 当土壤P有效性不足时, 往往通过有机磷的矿化来满足植物和微生物的需求。Bowman等(2008)通过对温带生态系统的研究也发现施氮后土壤有效磷含量降低, 其认为可能与氮添加引起的土壤酸化有关, 由于碱性阳离子会被Al3+取代产生铝毒, 这会导致微生物活性降低进而影响土壤磷的周转, 从而导致有效磷含量降低[33]。但本研究中施氮后pH变化不明显。Fan等(2018)通过对亚热带米槠林的研究发现施氮会刺激生物的磷需求, 微生物和植物根系倾向于加速土壤有机磷的矿化来维持土壤有效磷的供应[22]。因此, 在富含微生物和植物根系的表层土壤中, 相比非生物因素, 生物因素可能在土壤磷组分变化过程中发挥更重要的作用。在10—20 cm土层中, 施氮后各处理间的磷组分差异不明显, 这可能是由于底层根系和微生物丰度较低, 对土壤磷组分的影响低于表层土壤。
3.2 氮沉降对土壤解磷微生物的影响解磷微生物相对丰度对外界环境变化较为敏感, 可指示土壤养分状况及其变化。本研究发现米槠天然林土壤解磷细菌主要包括慢生根瘤菌、伯克霍尔德菌、中慢生根瘤菌、链酶菌、芽孢杆菌和类芽孢杆菌。施氮降低了根瘤菌和伯克霍尔德菌等解磷细菌的相对丰度(图 1), 与施氮后碱性土壤解磷细菌丰度降低的研究结果一致[34]。此外, 根瘤菌和伯克霍尔德菌属除了具有解磷功能, 还会与植物根系结合形成根瘤, 促进土壤固氮并改善植物的氮吸收的能力[35]。因此, 本研究中施氮后根瘤菌和伯克霍尔德菌丰度的降低可能与土壤氮有效性的提高导致植物减少了固氮功能微生物的养分投资有关[36]。另一方面, 微生物固氮本身也是一个耗磷的过程, 而氮沉降可能会加重土壤微生物的磷限制[22], 可能是根瘤菌等固氮微生物相对丰度降低的另一个原因。如有研究发现, 施氮后参与土壤磷转化的主要微生物类群由兼具固氮功能的解磷细菌转变为非固氮功能的细菌为主[37]。
相比解磷细菌, 解磷真菌的种类较少, 但其对氮沉降的响应更敏感。本研究发现施氮后米槠天然林土壤青霉菌、曲霉菌和镰刀菌的相对丰度增加, 这可能与施氮导致植物磷需求的增加有关。植物磷需求的增加可能会改变土壤微生物群落, 增加解磷能力更强的真菌的相对丰度来提高土壤磷有效性[38]。目前, 已有研究发现曲霉素菌丝体的增加有助于促进不溶性磷酸盐的溶解和迁移。如Wang等发现接种青霉菌的作物具有更强的磷吸收能力, 其产量显著高于不接种的作物。通过对比研究发现, 青霉菌的解磷能力甚至高于芽孢杆菌[5], 如在农田生态系统中, 施氮肥后植物矿化有机磷的能力显著增加[39]。此外, 解磷微生物与环境因子的相关分析表明, 解磷真菌对土壤环境因子的响应可能更敏感, 除了氮磷养分的限制外, 由氮沉降带来的土壤酸化进程也可能会影响功能微生物类群的相对丰度。
3.3 氮沉降对微生物解磷功能的影响为了探究氮沉降背景下米槠天然林土壤中解磷微生物的功能及作用机制, 本研究借助PICURSt功能预测来分析编码磷酸酶基因及其相对丰度的变化。结果发现施氮显著增加了表层土壤中酸性磷酸酶、碱性磷酸酶、植酸酶等磷酸单脂酶和磷酸二酯酶基因的相对丰度(图 2), 表明施氮后土壤微生物编码磷酸酶的能力可能会增加。这与已有研究结果相似, 如Heuck等通过对温带森林的研究, 发现长期氮添加提高了土壤磷酸酶活性[40], 在干热河谷土壤中也发现了类似的结果[41]。但在樟子松人工林土壤中, 施氮显著降低了酸性磷酸酶、碱性磷酸酶以及磷酸酶功能基因的丰度[42], 可能与土壤类型等多种因素有关。一般认为, 酸性土壤中的大部分无机磷被铁铝氧化物固定[3], 有机磷成为潜在磷源, 在受到低磷胁迫时, 微生物可能会增加磷酸酶的合成促进有机磷矿化来提高土壤有效磷含量[5]。此外, 植物根系属性的变化也可能影响磷酸酶的分泌, 前期研究发现施氮后细根生物量和比根长均显著增加[3], 有利于提高地下碳分配, 为微生物提供更多的能量, 提高微生物合成磷酸酶的能力。此外, 施氮对土壤磷酸酶的影响还取决于施氮方式和水平。如Li等在北温带森林土壤的研究发现高氮水平下对酸性磷酸酶的分泌具有显著的促进作用, 但抑制了碱性磷酸酶的分泌[43]。
在亚热带高度风化的森林土壤中, 土壤有机磷含量较高, 占总磷的40%左右[3], 且易在磷酸酶作用下分解后释放有效磷[10]。本研究发现氮沉降后中等程度有机磷降低, 但编码磷酸酶(酸性磷酸酶、碱性磷酸酶、植酸酶)功能基因的相对丰度显著增加, 表明施氮后, 土壤微生物可能通过分泌磷酸酶促进有机磷矿化来缓解磷限制。此外, 本研究中施氮降低了根瘤菌等解磷细菌的相对丰度, 但却增加了青霉菌、曲霉菌等解磷真菌的丰度(图 1)。由于在土壤酸化等环境胁迫下, 真菌具有更强的抵抗逆境和分泌磷酸酶能力[38], 解磷真菌相对丰度的增加也可能是土壤磷酸酶基因丰度增加的重要原因之一。综上表明, 在氮沉降背景下, 植被可能会通过改变土壤微生物群落, 增加磷酸酶分泌潜力更强的微生物类群来应对和缓解低磷胁迫。
4 结论施氮显著降低了土壤Resin-P、NaHCO3-P和TPo, 加剧了土壤磷限制。并且改变了解磷微生物的组成, 降低了根瘤菌和伯克霍尔德菌等解磷细菌的相对丰度, 却增加了青霉菌和曲霉菌等解磷真菌的相对丰度。此外, 氮沉降显著提高了编码酸性磷酸酶、碱性磷酸酶和植酸酶基因的相对丰度。可见, 氮沉降加重微生物磷限制的背景下, 增加土壤解磷真菌丰度, 提高分泌磷酸酶的功能潜力, 促进有机磷分解可能是微生物缓解磷限制的重要途径。
| [1] |
Du E Z, Terrer C, Pellegrini A F A, Ahlström A, van Lissa C J, Zhao X, Xia N, Wu X H, Jackson R B. Global patterns of terrestrial nitrogen and phosphorus limitation. Nature Geoscience, 2020, 13(3): 221-226. DOI:10.1038/s41561-019-0530-4 |
| [2] |
Penuelas J, Janssens I A, Ciais P, Obersteiner M, Sardans J. Anthropogenic global shifts in biospheric N and P concentrations and ratios and their impacts on biodiversity, ecosystem productivity, food security, and human health. Global Change Biology, 2020, 26(4): 1962-1985. DOI:10.1111/gcb.14981 |
| [3] |
Fan Y X, Zhong X J, Lin F, Liu C, Yang L M, Wang M H, Chen G S, Chen Y, Yang Y S. Responses of soil phosphorus fractions after nitrogen addition in a subtropical forest ecosystem: insights from decreased Fe and Al oxides and increased plant roots. Geoderma, 2019, 337: 246-255. DOI:10.1016/j.geoderma.2018.09.028 |
| [4] |
赵盼盼, 周嘉聪, 林开淼, 林伟盛, 袁萍, 曾晓敏, 苏莹, 徐建国, 陈岳民, 杨玉盛. 不同海拔对福建戴云山黄山松林土壤微生物生物量和土壤酶活性的影响. 生态学报, 2019, 39(8): 2676-2686. DOI:10.5846/stxb201805111043 |
| [5] |
Wang Y Y, Li P S, Zhang B X, Wang Y P, Meng J, Gao Y F, He X M, Hu X M. Identification of phosphate-solubilizing microorganisms and determination of their phosphate-solubilizing activity and growth-promoting capability. BioResources, 2020, 15(2): 2560-2578. DOI:10.15376/biores.15.2.2560-2578 |
| [6] |
Dipta B, Bhardwaj S, Kaushal M, Kirti S, Sharma R. Obliteration of phosphorus deficiency in plants by microbial interceded approach. Symbiosis, 2019, 78(2): 163-176. DOI:10.1007/s13199-019-00600-y |
| [7] |
Rodriguez H, Fraga R. Phosphate solubilizing bacteria and their role in plant growth promotion. Biotechnology Advances, 1999, 17(4/5): 319-339. |
| [8] |
Elias F, Woyessa D, Muleta D. Phosphate solubilization potential of rhizosphere fungi isolated from plants in jimma zone, southwest Ethiopia. International Journal of Microbiology, 2016, 1-11. |
| [9] |
Li T, Cui L Z, Liu L L, Wang H, Dong J F, Wang F, Song X F, Che R X, Li C J, Tang L, Xu Z H, Wang Y F, Du J Q, Hao Y B, Cui X Y. Characteristics of nitrogen deposition research within grassland ecosystems globally and its insight from grassland microbial community changes in China. Frontiers in Plant Science, 2022, 13: 947279. DOI:10.3389/fpls.2022.947279 |
| [10] |
Zhang T A, Chen H Y H, Ruan H H. Global negative effects of nitrogen deposition on soil microbes. The ISME Journal, 2018, 12(7): 1817-1825. DOI:10.1038/s41396-018-0096-y |
| [11] |
Berthrong S T, Yeager C M, Gallegos-Graves L, Steven B, Eichorst S A, Jackson R B, Kuske C R. Nitrogen fertilization has a stronger effect on soil nitrogen-fixing bacterial communities than elevated atmospheric CO2. Applied and Environmental Microbiology, 2014, 80(10): 3103-3112. DOI:10.1128/AEM.04034-13 |
| [12] |
Wang C Y, Zhou J W, Liu J, Jiang K, Du D L. Responses of soil N-fixing bacteria communities to Amaranthus retroflexus invasion under different forms of N deposition. Agriculture, Ecosystems & Environment, 2017, 247: 329-336. |
| [13] |
Yan G Y, Xing Y J, Han S J, Zhang J H, Wang Q G, Mu C C. Long-time precipitation reduction and nitrogen deposition increase alter soil nitrogen dynamic by influencing soil bacterial communities and functional groups. Pedosphere, 2020, 30(3): 363-377. DOI:10.1016/S1002-0160(19)60834-9 |
| [14] |
Kim H, Kang H. The impacts of excessive nitrogen additions on enzyme activities and nutrient leaching in two contrasting forest soils. The Journal of Microbiology, 2011, 49(3): 369-375. DOI:10.1007/s12275-011-0421-x |
| [15] |
Lu J L, Jia P, Feng S W, Wang Y T, Zheng J, Ou S N, Wu Z H, Liao B, Shu W S, Liang J L, Li J T. Remarkable effects of microbial factors on soil phosphorus bioavailability: a country-scale study. Global Change Biology, 2022, 28(14): 4459-4471. DOI:10.1111/gcb.16213 |
| [16] |
Wang C Q, Xue L, Jiao R Z. Stoichiometric imbalances and the dynamics of phosphatase activity and the abundance of phoC and phoD genes with the development of Cunninghamia lanceolata (Lamb.) Hook plantations. Applied Soil Ecology, 2022, 173: 104373. DOI:10.1016/j.apsoil.2021.104373 |
| [17] |
Yadav R S, Tarafdar J C. Phytase and phosphatase producing fungi in arid and semi-arid soils and their efficiency in hydrolyzing different organic P compounds. Soil Biology and Biochemistry, 2003, 35(6): 745-751. DOI:10.1016/S0038-0717(03)00089-0 |
| [18] |
Della Mónica I F, Godoy M S, Godeas A M, Scervino J M. Fungal extracellular phosphatases: their role in P cycling under different pH and P sources availability. Journal of Applied Microbiology, 2018, 124(1): 155-165. DOI:10.1111/jam.13620 |
| [19] |
王丽君, 程瑞梅, 肖文发, 沈雅飞, 曾立雄, 杨邵, 孙鹏飞, 陈天. 三峡库区马尾松人工林土壤酶活性和微生物生物量对氮添加的季节性响应. 生态学报, 2021, 41(24): 9857-9868. DOI:10.5846/stxb202010272751 |
| [20] |
Martínez-Toledo Á, González-Mille D J, Briones-Gallardo R, Carrizalez-Yañez L, Felipe Martínez-Montoya J, de Jesús Mejía-Saavedra J, Ilizaliturri-Hernández C A. Functioning of semi-arid soils under long-term mining activity with trace elements at high concentrations. Catena, 2023, 222: 106851. DOI:10.1016/j.catena.2022.106851 |
| [21] |
Zhu F F, Yoh M, Gilliam F S, Lu X K, Mo J M. Nutrient limitation in three lowland tropical forests in Southern China receiving high nitrogen deposition: insights from fine root responses to nutrient additions. PLoS One, 2013, 8(12): e82661. DOI:10.1371/journal.pone.0082661 |
| [22] |
Fan Y X, Lin F, Yang L M, Zhong X J, Wang M H, Zhou J C, Chen Y, Yang Y S. Decreased soil organic P fraction associated with ectomycorrhizal fungal activity to meet increased P demand under N application in a subtropical forest ecosystem. Biology and Fertility of Soils, 2018, 54(1): 149-161. DOI:10.1007/s00374-017-1251-8 |
| [23] |
You C M, Wu F Z, Yang W Q, Xu Z F, Tan B, Yue K, Ni X Y. Nutrient-limited conditions determine the responses of foliar nitrogen and phosphorus stoichiometry to nitrogen addition: a global meta-analysis. Environmental Pollution, 2018, 241: 740-749. DOI:10.1016/j.envpol.2018.06.018 |
| [24] |
Langille M G I, Zaneveld J, Caporaso J G, McDonald D, Knights D, Reyes J A, Clemente J C, Burkepile D E, Vega Thurber R L, Knight R, Beiko R G, Huttenhower C. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences. Nature Biotechnology, 2013, 31(9): 814-821. DOI:10.1038/nbt.2676 |
| [25] |
Jia Y L, Yu G R, Gao Y N, He N P, Wang Q F, Jiao C C, Zuo Y. Global inorganic nitrogen dry deposition inferred from ground- and space-based measurements. Scientific Reports, 2016, 6: 19810. DOI:10.1038/srep19810 |
| [26] |
Hedley M J, Stewart J W B, Chauhan B S. Changes in inorganic and organic soil phosphorus fractions induced by cultivation practices and by laboratory incubations. Soil Science Society of America Journal, 1982, 46(5): 970-976. DOI:10.2136/sssaj1982.03615995004600050017x |
| [27] |
Tiessen H, Moir J O. Characterization of available P by sequential extraction. In: Carter MR (ed) Soil sampling and methods of analysis. Canadian Society of Soil Science. Lewis Publishers, Raton, 1993, 75-86.
|
| [28] |
Klotzbücher A, Kaiser K, Klotzbücher T, Wolff M, Mikutta R. Testing mechanisms underlying the Hedley sequential phosphorus extraction of soils. Journal of Plant Nutrition and Soil Science, 2019, 182(4): 570-577. DOI:10.1002/jpln.201800652 |
| [29] |
Baldrian P, Kolařík M, Štursová M, Kopecký J, Valášková V, Větrovský T, Žifčáková L, Šnajdr J, Rídl J, Vlčekč, Voříšková J. Active and total microbial communities in forest soil are largely different and highly stratified during decomposition. The ISME Journal, 2012, 6(2): 248-258. DOI:10.1038/ismej.2011.95 |
| [30] |
Nilsson H O, Mulchandani R, Tranberg K G, Stenram U, Wadstrom T. Helicobacter pylori detected in the liver of patients with primary cholangio- and hepatocellular carcinoma by PCR and DNA sequencing. Gut, 2000, 47: A1-A2. DOI:10.1136/gut.47.1.1 |
| [31] |
Chen S F, Zhou R C, Huang Y L, Zhang M, Yang G L, Zhong C R, Shi S H. Transcriptome sequencing of a highly salt tolerant mangrove species Sonneratia alba using Illumina platform. Marine Genomics, 2011, 4(2): 129-136. DOI:10.1016/j.margen.2011.03.005 |
| [32] |
曾泉鑫, 曾晓敏, 林开淼, 张秋芳, 程蕾, 周嘉聪, 林巧玉, 陈岳民, 徐建国. 亚热带毛竹林土壤磷组分和微生物对施氮的响应. 应用生态学报, 2020, 31(3): 753-760. |
| [33] |
Bowman W D, Cleveland C C, Halada Ĺ, Hreško J, Baron J S. Negative impact of nitrogen deposition on soil buffering capacity. Nature Geoscience, 2008, 1(11): 767-770. DOI:10.1038/ngeo339 |
| [34] |
Zheng B X, Hao X L, Ding K, Zhou G W, Chen Q L, Zhang J B, Zhu Y G. Long-term nitrogen fertilization decreased the abundance of inorganic phosphate solubilizing bacteria in an alkaline soil. Scientific Reports, 2017, 7: 42284. DOI:10.1038/srep42284 |
| [35] |
Zhong Y J, Tian J, Li X X, Liao H. Cooperative interactions between nitrogen fixation and phosphorus nutrition in legumes. The New Phytologist, 2023, 237(3): 734-745. DOI:10.1111/nph.18593 |
| [36] |
Wang Z J, Li X, Wang J H, Qi S S, Dai Z C, Du D L. Effect of nitrogen-fixing bacteria on resource investment of the root system in an invasive clonal plant under low nutritional environment. Flora, 2022, 297: 152166. DOI:10.1016/j.flora.2022.152166 |
| [37] |
徐鹏霞, 韩丽丽, 贺纪正, 罗锋, 张丽梅. 非共生生物固氮微生物分子生态学研究进展. 应用生态学报, 2017, 28(10): 3440-3450. |
| [38] |
Bowles T M, Jackson L E, Cavagnaro T R. Mycorrhizal fungi enhance plant nutrient acquisition and modulate nitrogen loss with variable water regimes. Global Change Biology, 2018, 24(1): e171-e182. |
| [39] |
Ding X D, Zhang L, Zhang S R, Feng G. Phytate utilization of maize mediated by different nitrogen forms in a plant-arbuscular mycorrhizal fungus-phosphate-solubilizing bacterium system. Journal of Plant Interactions, 2014, 9(1): 514-520. DOI:10.1080/17429145.2013.861028 |
| [40] |
Heuck C, Smolka G, Whalen E D, Frey S, Gundersen P, Moldan F, Fernandez I J, Spohn M. Effects of long-term nitrogen addition on phosphorus cycling in organic soil horizons of temperate forests. Biogeochemistry, 2018, 141(2): 167-181. DOI:10.1007/s10533-018-0511-5 |
| [41] |
樊博, 史亮涛, 潘志贤, 何光熊, 孙毅, 闫帮国. 干热河谷土壤酶活性对碳氮添加的响应. 生态学报, 2018, 38(23): 8604-8611. DOI:10.5846/stxb201709031588 |
| [42] |
Wu L H, Geng B M, Wang Y J, Zhou G W, Sun Q Y, Zhao Q. Effects of Nitrogen Addition on the Abundance of Bacterial Phosphatase Encoding Genes in the Soil of Pinus sylvestris var. mongolica Plantation. Biotechnology Bulletin, 2022, 38(11): 202-209. |
| [43] |
Li Y, Wang C M, Gao S J, Wang P, Qiu J C, Shang S S. Impacts of simulated nitrogen deposition on soil enzyme activity in a northern temperate forest ecosystem depend on the form and level of added nitrogen. European Journal of Soil Biology, 2021, 103: 103287. DOI:10.1016/j.ejsobi.2021.103287 |
 2024, Vol. 44
2024, Vol. 44