文章信息
- 傅建融, 周洁, 张艳苹, 翟欣, 李浩, 刘丽
- FU Jianrong, ZHOU Jie, ZHANG Yanping, ZHAI Xin, LI Hao, LIU Li
- 不同温度下杉叶蕨藻对风信子鹿角珊瑚附生微生物群落的影响
- Effects of Caulerpa taxifolia on microbial community structure of Acroproa hyacinthus at different temperatures
- 生态学报. 2024, 44(3): 1298-1311
- Acta Ecologica Sinica. 2024, 44(3): 1298-1311
- http://dx.doi.org/10.20103/j.stxb.202212193621
-
文章历史
- 收稿日期: 2022-12-19
- 网络出版日期: 2023-11-08
在珊瑚礁生态系统中, 珊瑚与大型底栖海藻争夺着有限的生态位[1]。由于全球气候变化, 部分珊瑚礁区逐渐向大型海藻场演替[2—3]。大型海藻通过物理、化学和微生物过程直接或间接地影响珊瑚的生长[4—5], 其物理机制主要通过慢性刷蚀使珊瑚虫螅体缩回从而增加珊瑚死亡率[6], 或过度生长阻碍幼虫附着[7];化感机制主要通过海藻自身分泌的脂溶性或水溶性的化感物质转移至珊瑚虫体内, 导致珊瑚白化[8]。
健康的珊瑚礁菌群往往处于动态平衡[9], 珊瑚虫的生理功能和珊瑚礁生态系统功能与其微生物群落结构息息相关。珊瑚依赖于共生菌科, 通过光合用固定碳的转移和溶解的无机氮和磷的同化来满足宿主能量需求[10];珊瑚虫与细菌的关联可能发挥多种功能作用, 包括硫循环、抵御病原体和应激耐受性[11—13]。然而, 环境因子的变化、物理作用或化感机制, 均可能改变珊瑚群落微生物结构[14—16]。大型海藻可以通过释放的溶解有机碳(DOC)促进珊瑚表面的细菌代谢与珊瑚竞争氧气, 从而对珊瑚的健康产生负面影响。随着珊瑚的死亡, 大型海藻将占据更多的生态位, 形成DDAM(DOC、疾病、藻类、微生物)正反馈回路[17]。此外, 大型海藻释放的萜类化合物也可能介导微生物群落结构变化而影响珊瑚生理[18—19]。因此, 大型海藻可能会减少健康珊瑚有益细菌丰度, 促进稀有微生物类群的生长, 或传播新的致病微生物以竞争生态位[20]。在珊瑚礁遭受环境压力时, 珊瑚需通过加速自身代谢以抵抗环境因子所带来的负面影响, 从而无法投入更多能量与大型海藻进行空间竞争, 二者的竞争关系也随之改变[21]。这其中也包括了珊瑚礁的微生物群落的改变, 如海洋温度升高后引发的白化效应使珊瑚优势细菌减少、病原体细菌种类增加[16], 但大型海藻在其中发挥的作用鲜有报道。
由于风信子鹿角珊瑚(Acropora hyacinthus)耐受阈值较低, 经常受到热压力等影响, 在徐闻地区是逐渐退化的枝状珊瑚种群[22—23]。在此地区大型藻类种类繁多, 其中杉叶蕨藻(Caulerpa taxifolia)是具有单萜和倍半萜类化合物的毒性海洋植物[24], 生长在各种硬质基地上, 包括大量的活珊瑚群。与杉叶蕨藻直接接触或临近的珊瑚, 部分表现出白化或损伤的痕迹, 这可能是杉叶蕨藻长期作用的影响, 也可能是海洋暖化等因素引起的复合作用。但目前对于大型海藻是否是造成珊瑚覆盖度下降的直接原因仍没有定论。因此, 为探究气候变化下大型海藻的不同竞争模式如何影响珊瑚微生物群。本研究设置不同的温度(27℃、30℃), 利用高通量测序技术对杉叶蕨藻暴露后风信子鹿角珊瑚共附生微生物、水环境微生物以及藻体表面微生物多样性及其群落结构组成与功能进行分析, 研究结果有助于理解气候变化下珊瑚—大型海藻二者的竞争机制, 以及在升温情形下其种间关系的变化, 解释大型海藻对造礁石珊瑚的潜在生态影响。
1 材料与方法 1.1 样本采集风信子鹿角珊瑚和杉叶蕨藻均采集自徐闻珊瑚礁国家自然保护区(109°55′ E, 20°16′N)。运回实验室后分别暂养在水温27℃、pH 8.1、盐度33的两个体积为200 L珊瑚养殖水箱中, 珊瑚养殖水使用珊瑚盐配置, 光源由全光谱LED珊瑚灯(Maxspect)提供, 有效辐射200 μmol m-2 s-1(光暗比12 ∶ 12 h)。暂养3个月后, 从母体截取为长4 cm的小枝, 用阿隆发水草胶固定于陶瓷底座上集中置于养殖箱中直到珊瑚正常伸展。
1.2 杉叶蕨藻暴露实验杉叶蕨藻与风信子鹿角珊瑚按两种模式设置, 一种为直接接触处理, 即海藻与珊瑚同处于一个水平通过海流对珊瑚产生摩擦;另一种为间接接触处理, 即海藻被置于外挂藻盒中。依据夏季徐闻地区(www.oceanguide.org.cn)珊瑚所经历的典型温度范围设置两种温度, 分别为环境温度27℃和升温30℃。因此本实验共设置6组, 分别为对照组—D、间接接触组—LH、直接接触组—LM、升温组—H、升温间接接触组—HH、升温直接接触组—HM, 每组放置3块珊瑚, 并设置3个平行组。根据徐闻地区近岸礁调查的, 杉叶蕨藻密度约为0.0022 g/cm3, 除对照组和升温组外, 其它4组每个缸内藻量约为25 g(0.0025 g/cm3), 由于藻类新陈代谢较快, 每日检查替换发生腐烂的藻类。每个实验缸体积为10 L(各缸每三天更换50%海水), 底滤缸35 L。4周后收集各组实验样品。
1.3 样品的收集实验结束后分别收集珊瑚表面(C)、培养水体(W)以及杉叶蕨藻表面(Z)的附生微生物。珊瑚、海藻使用PBS轻微震荡后, 使用无菌海水进行颠倒震荡10 min后收集震荡液;取5 L培养水体, 使用无菌滤膜(0.45 μm)过滤后收集滤膜。将震荡液、滤膜液氮急冻10 min后, 保存于-80℃中, 用于后续DNA提取以及测序。
1.4 珊瑚附生微生物高通量测序使用TGuide S96磁珠法基因组DNA提取试剂盒按照说明书完成核酸的提取。提取样品总DNA后, 使用微量核算酸定量仪对于提取的核酸进行浓度检测以及使用浓度1.2%的琼脂糖进行电泳质量检测。根据保守区设计得到引物(F:5′-ACTCCTACGGGAGGCAGCA-3′, R:5′-GGACTACHVGGGTWTCTAAT-3′), 在引物末端加上测序接头, 16S rRNA基因V3—V4可变区进行PCR扩增并对其产物进行纯化、定量和均一化形成测序文库, 建好的文库先进行文库质检, 质检合格的文库用Illumina Novaseq 6000进行测序。
1.5 数据处理与分析通过Trimmomatic v0.33, cutadapt 1.9.1软件对Raw Reads进行过滤以及引物序列的识别与去除, 得到Clean Reads。Usearch v10软件, 对每个样品的Clean Reads进行拼接, 根据不同区域的长度范围对拼接后数据进行长度过滤。然后使用QIIME2 2020.6软件去噪并去除嵌合体序列, 获得扩增子序列变异体(amplicon sequence variants, ASVs) [25]。以SILVA为参考数据库, 朴素贝叶斯分类器对特征序列进行分类学注释, 得到每个特征对应的物种分类信息, 将注释后的数据采用稀疏(Rarefaction)的方法进行抽平, 通过从每个样本中分别随机抽取一定数量的序列以到达统一的深度, 从而预测各样本在该测序深度下, 所能观测到的ASVs及其相对丰度。抽平后的数据用于α、β多样性分析、群落结构组成分析、BugBase与FAPROTAX功能预测分析。使用维恩图对不同分组样品进行核心ASV分析(即各组内所有样本共享的ASVs), 并采用饼图和热图可视化核心ASV组成与组间相对丰度表达。最后, 基于分析线性判别分析(linear discriminant analysis, LDA)的LefSe组间群落差异, 分析各组中差异表达的微生物。数据正态性采用Shapiro-Wilk正态性检验, 再通过方差齐性检验(残差与拟合值的视觉检验)后, 采用双因素方差分析(ANOVA), 其中“温度”和“藻类”为固定因素, 包括交互项。Tukey ′s检验与事后Fisher ′s least significant difference(LSD)检验确定不同处理间的差异。结果以平均值±标准差表示, Graphpad prism 8.0与Rstudio绘图。
2 结果 2.1 α多样性分析风信子鹿角珊瑚个体仅在升温组出现白化现象, 部分珊瑚出现死亡, 死亡率为33%。高通量测序结果表明, 去除嵌合体后共获得优化序列3369713条。Sobs指数稀释曲线趋向平坦, 结果表明测序数据量合理。大多数细菌种类被注释用于后续分析。所有样本Coverage指数均>0.99, 表明测序结果数据充足, 能反应样本有效性。α多样性分析结果如图 1所示, Shanno与Simpson指数趋势相近, 珊瑚表面附生微生物在直接与间接接触组中最高(P < 0.05), 水环境微生物中各处理组较对照组无显著差异。ACE和Chao1指数均在珊瑚表面、水环境直接与间接接触组中具有显著差异(P < 0.05)。杉叶蕨藻表面微生物各指数无显著差异。
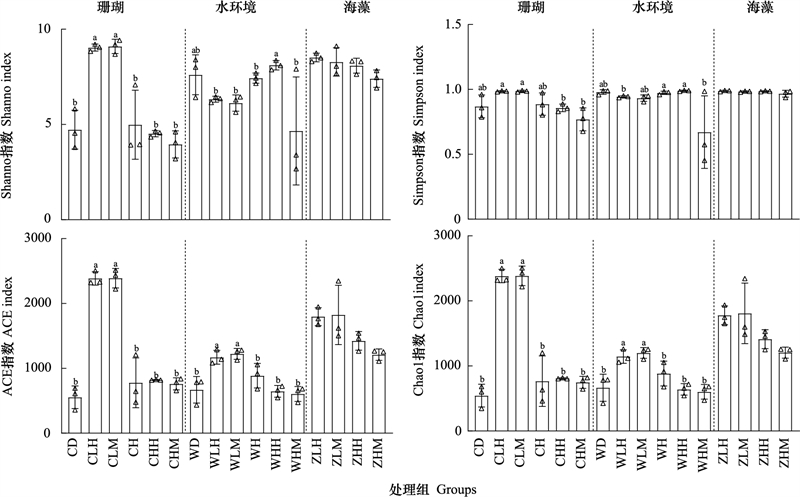
|
| 图 1 不同温度下杉叶蕨藻处理后珊瑚, 水环境, 海藻附生微生物群落α多样性 Fig. 1 α diversity of microorganism communities in corals, aquatic environment and seaweed after co-culture with Caulerpa taxifolia at different temperatures 数据以平均值±标准差的形式呈现, 不同字母代表不同处理间具有显著差异(P < 0.05);CD:珊瑚表面微生物对照组;CLH:珊瑚表面微生物间接接触组;CLM:珊瑚表面微生物直接接触组;CH:珊瑚表面微生物升温组;CHH:珊瑚表面微生物升温间接接触组;CHM:珊瑚表面微生物升温直接接触组;WD:水环境微生物对照组;WLH:水环境微生物间接接触组;WLM:水环境微生物直接接触组;WH:水环境微生物升温组;WHH:水环境微生物升温间接接触组;WHM:水环境微生物升温直接接触组;ZLH:藻体表面微生物间接接触组;ZLM:藻体表面微生物直接接触组;ZHH:藻体表面微生物升温间接接触组;ZHM:藻体表面微生物升温直接接触组;ACE:基于丰度的覆盖估计值Abundance-based coverage estimator |
基于布雷柯蒂斯距离(bray curtis distance), 采用主坐标分析(PCoA)和相关性分析(ANOSIM)评估了共生细菌群落的相似性, 如图 2所示。珊瑚表面微生物PCoA解释度占总方差的40%和15.2%, 区别于其他分组, 间接接触组与直接接触组聚为一类, 对照组自聚为一类;水环境微生物解释度为30.8%和10%, 间接接触组与直接接触组聚为一类;海藻表面微生物解释度为35.5%和17.5%, 其中不同温度样本显著区分。直接接触与间接接触两种处理模式解释度为30.1%和22.9%, 均发生重叠。以上结果说明除培养水环境外, 珊瑚、藻表面的微生物在不同温度下的β多样性区分度大。
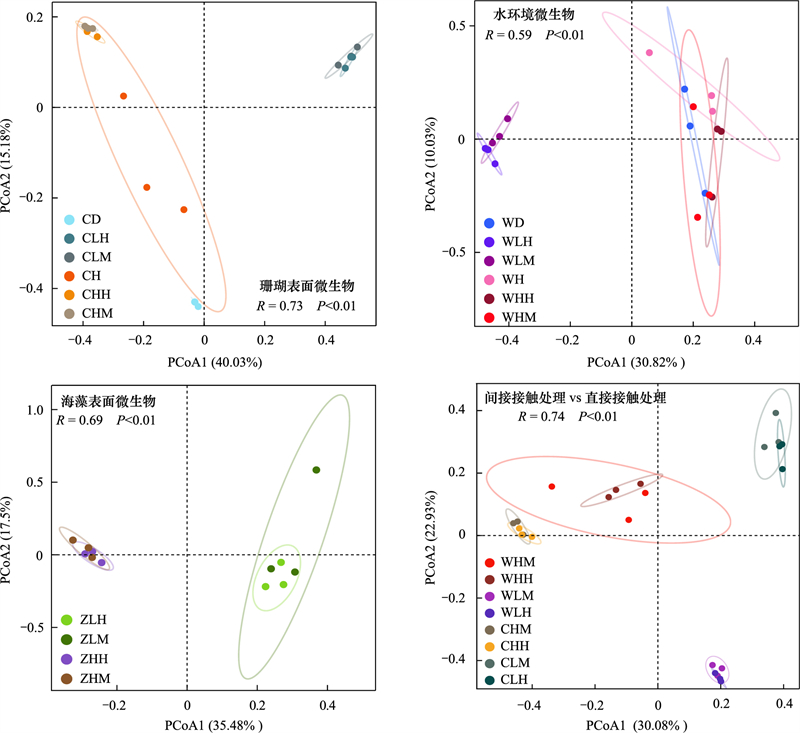
|
| 图 2 在ASV水平上微生物群落结构主坐标分析 Fig. 2 Microorganism structure at ASV level by principal coordinate analysis PCoA:主坐标分析Principal co-ordinate analysis;R:布雷柯蒂斯距离Bray curtis;ASV:扩增子序列变异体Amplicon sequence variants |
附生细菌群落组成分析结果如图 3所示。在所有样品中共检测到45个门、114个纲、337个目、712个科、1687个属以及2241个种。门水平上, 相对丰度前10的细菌门分别为变形菌门Proteobacteria(37.9%—88.4%), 拟杆菌门Bacteroidota(5.6%—32.6%), 蓝细菌门Cyanobacteria(0.5%—24.7%), 厚壁菌门Firmicutes (0.1%—12.4%), 放线菌门Actinobacteriota(0.1%—6.6%), 螺旋体门Spirochaetota(0%—9.8%), 酸杆菌门Acidobacteriota(0.02%—7.05%), 髌骨细菌门Patescibacteria(0.15%—2.36%), 疣微菌门Verrucomicrobiota(0.25%—1.64%), 以及1种未鉴定细菌(0.2%—5.8%);其余35个细菌门归为其他分类(others)。变形菌门在各组中均为优势类群。珊瑚表面, 间接接触组与直接接触组相较于对照组变形菌门与拟杆菌门相对丰度减少, 而厚壁菌门、放线菌门与螺旋体门相对丰度升高;升温组、升温间接接触组、升温直接接触组拟杆菌门相对丰度减少, 变形菌门相对丰度升高。水环境中, 直接接触组蓝细菌门相对丰度升高, 间接接触组变形菌门相对丰度升高;升温组相较于对照组微生物群落结构相似;升温间接接触组厚壁菌门以及升温直接接触组变形菌门相对丰度升高。海藻表面微生物群落结构相近, 均以变形菌门和拟杆菌门为主。
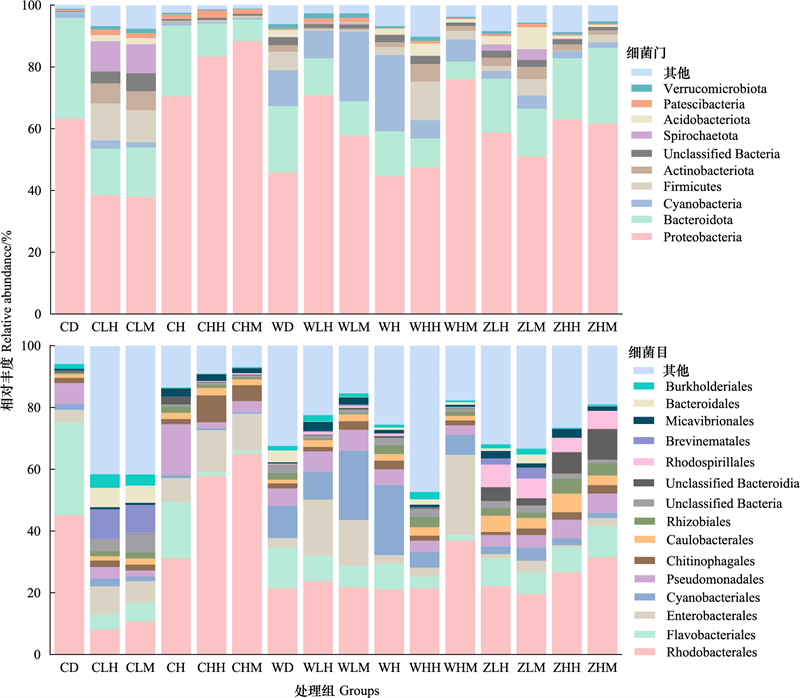
|
| 图 3 微生物群落结构柱状图 Fig. 3 Analysis of microorganism community structure Proteobacteria:变形菌门;Bacteroidota:拟杆菌门;Cyanobacteria:蓝细菌门;Firmicutes:厚壁菌门;Actinobacteriota:放线菌门;Spirochaetota:螺旋体门;Acidobacteriota:酸杆菌门;Patescibacteria:髌骨细菌门;Verrucomicrobiota:疣微菌门;Rhodobacterales:红细菌目;Flavobacteriales:黄杆菌目;Enterobacterales:肠杆菌目;Cyanobacteriales:蓝细菌目;Pseudomonadales:假单胞菌目;Chitinophagales:噬几丁质菌目;Caulobacterales:柄杆菌目;Rhizobiales:根瘤菌目;Rhodospirillales:红螺菌目;Brevinematales:螺旋体目;Micavibrionales:米卡弧菌目;Bacteroidales:拟杆菌目;Burkholderiales:伯克霍尔德氏菌目 |
目水平上, 相对丰度前15的细菌目主要包括红细菌目(Rhodobacterales), 黄杆菌目(Flavobacteriales), 肠杆菌目(Enterobacterales), 蓝细菌目(Cyanobacteriales), 假单胞菌目(Pseudomonadales), 噬几丁质菌目(Chitinophagales), 柄杆菌目(Caulobacterales), 根瘤菌目(Rhizobiales), 红螺菌目(Rhodospirillales), 螺旋体目(Brevinematales), 米卡弧菌目(Micavibrionales), 拟杆菌目(Bacteroidales), 伯克霍尔德氏菌目(Burkholderiales)以及两种未鉴定到的细菌目, 其余目归为为其他分类(others)。珊瑚表面, 间接接触组与直接接触组相较于对照组红细菌目、黄杆菌目、假单胞菌目的相对丰度分别降低34%—37%、24%—25%、3%—5%, 肠杆菌目、螺旋体目、拟杆菌目、伯克霍尔德氏菌目的相对丰度分别升高3%—5%、9%—10%、6%、2%—3%。升温组红细菌目、黄杆菌目分别降低14%、11.7%, 肠杆菌目、假单胞菌目、米卡弧菌目升高3.6%、9.6%、2%。升温直接或间接接触组黄杆菌目、假单胞菌目相对丰度降低29%、3%—5%;红细菌目、肠杆菌目、噬几丁质菌目分别升高13%—20%、8%—10%、4%—7%。水环境中, 红细菌目在升温直接接触组中上升了16%;肠杆菌目在间接接触组、直接接触组中相对丰度升高12%—15%, 升温直接接触组中升高23%;蓝细菌目在直接接触组和升温组中升高12%。海藻表面仅30℃时红细菌目相对丰度升高。
2.3.2 附生微生物功能预测如图 4所示, BugBase分析可以预测原核微生物的表型, 所有样本表型类型主要包括革兰氏阴性(20.7%—31.1%)、好氧细菌(7.7%—20.9%)、移动元件含量(11.4%—19.1%)和胁迫耐受(17.1%—26.9%)四大类。珊瑚表面, 间接接触组与直接接触组较对照组革兰氏阳性菌(4.8%—5.1%)、潜在致病菌(10.7%—11.8%)、胁迫耐受菌(21.7—22.6%)占比升高, 革兰氏阴性菌(22.5%)、好氧细菌(11%—12.4%)占比下降。水环境中, 间接接触组潜在致病菌(10.8%)占比升高。海藻表面潜在致病菌占比8.4%—9.4%。
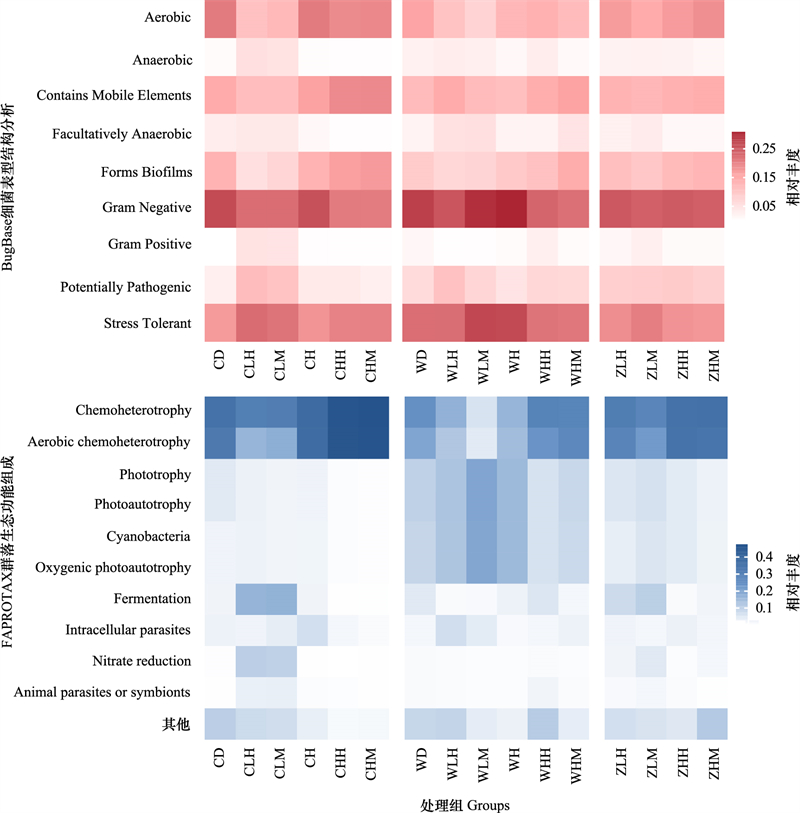
|
| 图 4 微生物群落功能分析 Fig. 4 Analysis of microorganism community function |
FAPROTAX分析可以预测原核微生物生态功能, 所有样本生态功能主要包括了化能异养(28%—48%)和需氧化能异养(22%—47%)。珊瑚表面, 间接接触组与直接接触组较对照组发酵(16.6%—17%)、硝酸盐还原(10%—10.7%)、动物寄生虫或共生体(3.3%—3.7%)占比升高;升温组胞内寄生虫(7.1%)占比升高, 而升温直接或间接接触组, 胞内寄生虫(0.9%—1.7%)占比下降。水环境中, 主要富集氧光合作用类细菌占比6.4%—19.7%。海藻表面, 动物寄生虫或共生体占比1.3%—1.7%, 胞内寄生虫占比1.9%—3%。
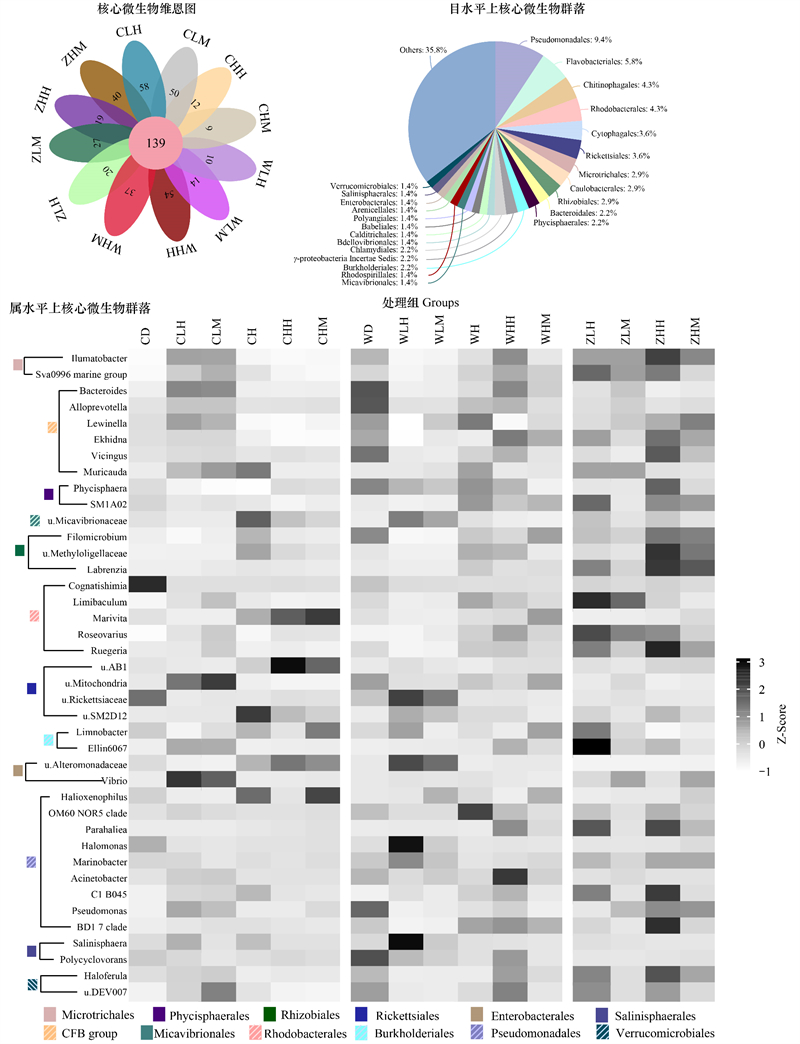
2.4 核心微生物分析对12个处理组的36个样本进行核心ASV分析, 共获得139个核心ASVs, 如图 5所示。在目水平上, 核心ASVs以假单胞菌目(9.4%)、黄杆菌目(5.8%)、噬几丁质菌目(4.3%)、红细菌目(4.3%)为主, 还包括立克次氏体菌目(Rickettsiales, 2.9%)、伯克霍尔德氏菌目(2.2%)、肠杆菌目(1.4%)、疣微菌目(Verrucomicrobiales, 1.4%)等。
|
| 图 5 核心微生物群落结构 Fig. 5 Community structure of co-expressing microorganism Others中包含小于1%的目水平微生物, 未鉴定到属类的以u.(unclassified)加科名表示; Z-score:标准分数Standard score;Microtrichales:微丝藻菌目;Phycisphaerales:浮霉菌目;Rickettsiales:立克次氏体菌目;Salinisphaerales:咸水球形菌目;CFB group:噬纤维菌-屈挠杆菌-拟杆菌群Cytophaga-Flexibacter-Bacteroides group |
构建属水平上相对丰度>1%的40个核心ASVs表达热图。珊瑚表面, 相较于对照组, 间接接触组与直接接触组Ilumatobacter、Sva0996、Muricauda、Ellin6067、Vibrio相对丰度升高, 并在海藻表面鉴定到上述微生物的丰度表达;升温组u.Micavibrionaceae、u.SM2D12、Halioxenophilus相对丰度升高。升温直接接触组Halioxenophilus相对丰度升高。水环境中, 间接接触组与直接接触组u.Alteromonadaceae、u.Micavibrionaceae丰度升高;升温组Lewinella、SM1A02、Halioxenophilus丰度升高;升温藻处理组SM1A02、Parahaliea、Haloferula丰度升高。
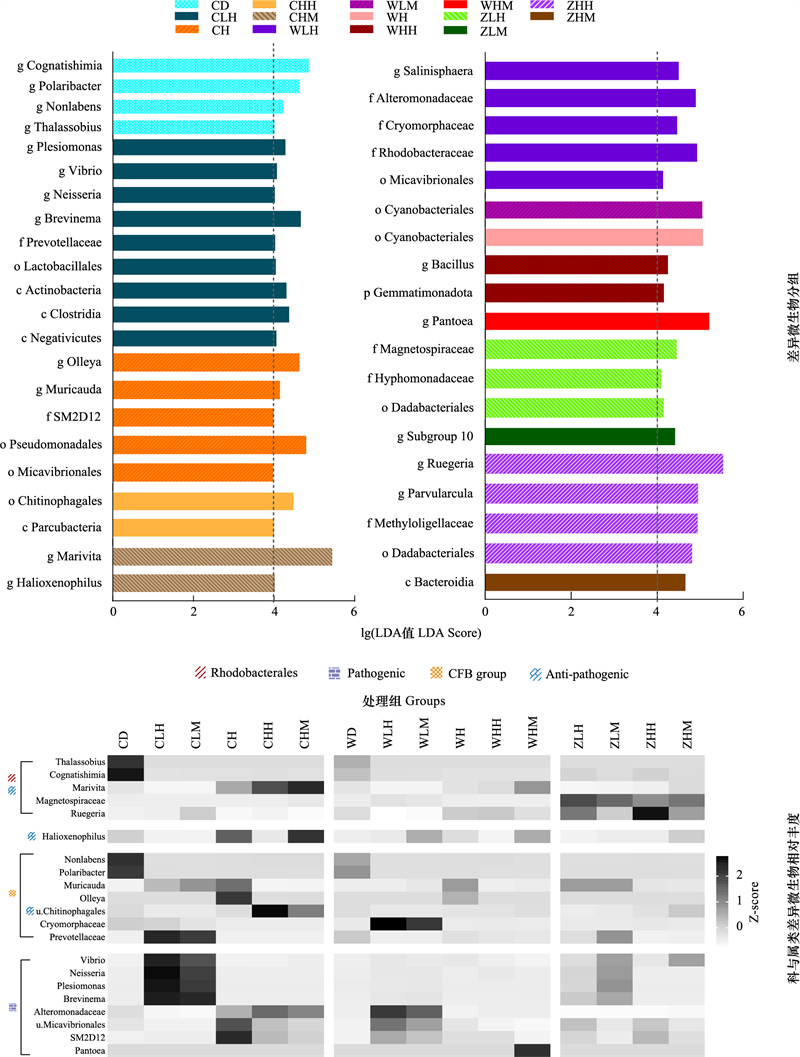
2.5 附生细菌群落的组间差异分析如图 6所示, LDA可比较各组中具有显著差异的细菌类群。珊瑚表面, 间接接触组弧菌属(Vibrio)、邻单胞菌属(Plesiomonas)、奈瑟氏菌属(Neisseria)以及螺旋体属(Brevinema);升温组立克次氏体菌目SM2D12科、米卡弧菌目、假单胞菌目;升温间接接触组噬几丁质目, 直接接触组纤维弧菌科(Cellvibrionaceae)Halioxenophilus属、玫瑰杆菌科(Roseobacteraceae)海命菌属(Marivita)相对丰度显著升高(LDA>4, P < 0.05)。水环境中, 间接接触组米卡弧菌目、红细菌目、黄杆菌目、交替单胞菌科(Alteromonadaceae);直接接触组与升温组蓝细菌目;升温间接接触组芽孢杆菌属(Bacillus), 直接接触组泛菌属(Pantoea)相对丰度显著升高(LDA>4, P < 0.05)。海藻表面, 间接接触组柄杆菌、红螺菌目, 直接接组酸杆菌门Subgroup 10属;升温间接接触组α变形菌纲类细菌, 直接接触组仅拟杆菌纲差异表达(LDA>4, P < 0.05)。
|
| 图 6 附生微生物LDA分析 Fig. 6 LDA analysis of microorganism LDA>4代表此类细菌较其他各组相对丰度具有显著差异; LDA:分析线性判别分析Linear discriminant analysis;Pathogenic:致病菌;Anti-pathogenic:益生菌 |
构建科与属水平上差异微生物相对丰度。直接或间接接触组潜在致病菌弧菌、邻单胞菌、奈瑟氏菌与螺旋体菌在珊瑚与海藻表面均有一定丰度, 而交替单胞菌、米卡弧菌、SM2D12在水环境中丰度高。升温组SM2D12、米卡弧菌等胞内寄生性病原菌丰度高, 升温直接或间接接触组中上述病原细菌丰度下降, 益生菌噬几丁质菌、海命菌、Halioxenophilus丰度上升。上述结果说明环境温度下海藻可以转移一定比例病原微生物至珊瑚表面, 热压力下海藻能抑制病原微生物在珊瑚表面的表达并提高益生菌的相对丰度。
3 讨论 3.1 环境温度下杉叶蕨藻对珊瑚菌落的影响本研究通过海藻与珊瑚共培养, 发现环境温度下珊瑚表面附生微生物α, β多样性显著区分于对照组(P < 0.05)。珊瑚微生物群落主要由变形菌门、拟杆菌门、蓝细菌门、厚壁菌门、放线菌门组成, 这与其他研究所一致[26]。在海藻作用下, 珊瑚附生菌中肠杆菌目、螺旋体菌目、伯克霍尔德氏菌目相对丰度升高。同时, 病原微生物弧菌、奈瑟氏菌、螺旋体菌和邻单胞菌相对丰度差异表达(LDA>4, P < 0.05)。
大型海藻已被证实为珊瑚致病菌的病源库, 并且在藻类作用下珊瑚附生菌落结构极易重塑[27]。Sweet[28]等证明在自然环境中患有白色综合征的珊瑚Montastraea faveolata中鉴定到丰度较高的奈瑟氏菌属、假单胞菌属、弧菌属、伯克霍尔德氏菌属, 同时, 这些微生物在丝状藻类表面同时也鉴定到, 而玫瑰杆菌属细菌则在健康的M. faveolata中有较高的丰度。弧菌属作为珊瑚疾病的病原体, 具有一系列的酶、毒素和其他毒力因子[29], 此外螺旋体科, 肠杆菌科, 奈瑟氏菌科以及伯克霍尔德氏菌科内细菌均是已被鉴定的病原微生物可能来源于藻体表面并在合适时机侵染珊瑚[28—30]。上述的病原微生物增加可能与大型海藻所释放的DOC有关, 此类营养物质在水体中增加可能会给对养分敏感或具r—选择特性的潜在致病菌“施肥”, 使它们大量繁衍而成为群落中的优势种[31—32]。但本实验过程中未发现大型海藻处理后珊瑚出现任何疾病的现象, 这可能与珊瑚所分泌的粘液具有抗菌功能有关[33]。因此, 大型海藻可以改变珊瑚的细菌群落结构但携带的致病菌短时间内未必能侵染健康珊瑚[34]。
3.2 热压力造成珊瑚白化以及微生物群落结构重塑热压力下, 珊瑚微生物群落中肠杆菌、米卡弧菌丰度上升, 立克次氏体菌、米卡弧菌差异表达(LDA>4, P < 0.05)。FAPROTAX分析发现细胞内寄生虫功能模块占比升高。热压力造成珊瑚礁白化已经被广泛报道[35], 且这种白化现象可能与弧菌的侵染[36]、虫黄藻逃逸或焦亡以及ROS毒害有关[37—38]。在暴露于海洋热浪或酸化后, 珊瑚附生微生物优势种群会从有益的细菌类群转变为细胞内致病菌以及机会性和潜在致病类群[39—41]。立克次体细菌, 在动物细胞中营细胞内寄生, 并已被证明会导致无脊椎动物疾病, 与珊瑚的健康和患病息息相关[42—43], 常见于被释放到环境中的珊瑚粘液细胞中[44], 并且此类细菌感染宿主时, 可能归因于环境因素, 如先天宿主免疫, 或由宿主微生物群抵抗系统受到热胁迫后发生紊乱[45—46]。米卡弧菌是立克次氏菌的近缘种, 在感染病宿主细胞中发挥着近似功能[47]。因此, 本研究发现, 热压力造成珊瑚白化现象可能与胞内寄生性病原菌丰度升高, 但这种影响可能不是由单一病原菌引起的, 还可能与虫黄藻逃逸和ROS毒害有关。
3.3 杉叶蕨藻与热胁迫的耦合作用热压力下, 杉叶蕨藻的“施肥效应”并不显著, 珊瑚附生菌多样性与对照组无显著差异, 但藻类缓解了珊瑚白化的现象。FAPROTAX与LDA分析发现, 相较于升温组, 杉叶蕨藻的存在使细胞内寄生虫模块的比例下降, 海命菌、u.Alteromonadaceae、Halioxenophilus、噬几丁质菌丰度上升。
大型藻类在热胁迫下可以促进珊瑚的生理功能。Brown等[48]发现, 在30℃时, 中间鹿角珊瑚(Acropora intermedia)与仙掌藻(Halimeda heteromorpha)接触导致光合作呼吸比明显增加。藻类的存在会间歇性地提供阴影, 并且大型海藻具有较粗的匍匐枝与匍匐茎, 从而保证水体自由流动与氧气交换, 减缓热压力[49—50]。热压力的缓解可能与以下两方面有关。一方面, 可能与大型海藻释放的DMSP(二甲基硫丙酸)有关。鹿角珊瑚可以通过外界获取或自身合成DMSP, DMSP所合成的DMS(二甲基硫)可以清除ROS消解氧化应激水平以及抵抗病原微生物[51—54]。因此, 藻类分泌的DMSP或许是风信子鹿角珊瑚耐受热压力的原因之一。另一方面, 红细菌目中玫瑰杆菌科细菌具有将DMSP转化为DMS的功能, 并且此类细菌由PKS-NRPS Hybrid基因所表达的蛋白可能具有抵抗病原微生物的功能[55—56]。并且, 噬几丁质菌丰度上调可能与几丁质酶的表达有关, 珊瑚暴露于病原体也会诱导产生具有多种具有防御功能的酶, 其中便包括几丁质酶[57—58]。此外, Halioxenophilus是纤维弧菌科中的一员, 研究表明纤维弧菌具有大量与抗菌化合物合成相关的基因, 在微生物群落结构变化方面发挥着重要功能[59—60]。因此, 益生菌在大型海藻作用下丰度的提高, 可能是缓解热压力所引起的病原微生物增加的原因之一。
4 结论本研究构建了27℃和30℃条件下杉叶蕨藻与风信子鹿角珊瑚直接或间接接触实验。使用高通量测序技术对珊瑚表面、培养水环境以及海藻表面微生物进行检测。结果表明, 环境温度下, 杉叶蕨藻的存在会引起珊瑚表面病原菌的增加, 并且病原菌的来源可能是海藻;升温导致珊瑚白化以及胞内寄生性病原微生物丰度显著升高;此时海藻的加入降低了寄生性病原菌的丰度, 提高了益生菌的丰度, 缓解了珊瑚白化现象。因此, 本研究认为大型海藻可以协助枝状珊瑚抵抗热压力。
| [1] |
Miller M. Coral/seaweed competition and the control of reef community structure within and between latitudes. Oceanography and Marine Biology, 1998, 36: 65-96. |
| [2] |
Hughes T P. Catastrophes, phase shifts, and large-scale degradation of a Caribbean coral reef. Science, 1994, 265(5178): 1547-1551. DOI:10.1126/science.265.5178.1547 |
| [3] |
Hughes T P, Rodrigues M J, Bellwood D R, Ceccarelli D, Hoegh-Guldberg O, McCook L, Moltschaniwskyj N, Pratchett M S, Steneck R S, Willis B. Phase shifts, herbivory, and the resilience of coral reefs to climate change. Current Biology, 2007, 17(4): 360-365. DOI:10.1016/j.cub.2006.12.049 |
| [4] |
Tanner J E. Competition between scleractinian corals and macroalgae: an experimental investigation of coral growth, survival and reproduction. Journal of Experimental Marine Biology and Ecology, 1995, 190(2): 151-168. DOI:10.1016/0022-0981(95)00027-O |
| [5] |
Bonaldo R M, Hay M E. Seaweed-coral interactions: variance in seaweed allelopathy, coral susceptibility, and potential effects on coral resilience. PLoS One, 2014, 9(1): e85786. DOI:10.1371/journal.pone.0085786 |
| [6] |
Coyer J A, Ambrose R F, Engle J M, Carroll J C. Interactions between corals and algae on a temperate zone rocky reef: mediation by sea urchins. Journal of Experimental Marine Biology and Ecology, 1993, 167(1): 21-37. DOI:10.1016/0022-0981(93)90181-M |
| [7] |
McCook L, Jompa J, Diaz-Pulido G. Competition between corals and algae on coral reefs: a review of evidence and mechanisms. Coral Reefs, 2001, 19(4): 400-417. DOI:10.1007/s003380000129 |
| [8] |
Longo G O, Hay M E. Seaweed allelopathy to corals: are active compounds on, or in, seaweeds?. Coral Reefs, 2017, 36(1): 247-253. DOI:10.1007/s00338-016-1526-9 |
| [9] |
Barott K L, Rodriguez-Mueller B, Youle M, Marhaver K L, Vermeij M J A, Smith J E, Rohwer F L. Microbial to reef scale interactions between the reef-building coral Montastraea annularis and benthic algae. Proceedings Biological Sciences, 2012, 279(1733): 1655-1664. |
| [10] |
Rädecker N, Pogoreutz C, Voolstra C R, Wiedenmann J, Wild C. Nitrogen cycling in corals: the key to understanding holobiont functioning?. Trends in Microbiology, 2015, 23(8): 490-497. DOI:10.1016/j.tim.2015.03.008 |
| [11] |
Raina J B, Tapiolas D, Willis B L, Bourne D G. Coral-associated bacteria and their role in the biogeochemical cycling of sulfur. Applied and Environmental Microbiology, 2009, 75(11): 3492-3501. DOI:10.1128/AEM.02567-08 |
| [12] |
Krediet C J, Ritchie K B, Paul V J, Teplitski M. Coral-associated micro-organisms and their roles in promoting coral health and thwarting diseases. Proceedings Biological Sciences, 2013, 280(1755): 20122328. |
| [13] |
Boilard A, Dubé C E, Gruet C, Mercière A, Hernandez-Agreda A, Derome N. Defining coral bleaching as a microbial dysbiosis within the coral holobiont. Microorganisms, 2020, 8(11): 1682. DOI:10.3390/microorganisms8111682 |
| [14] |
Diaz-Pulido G, Barrón C. CO2 enrichment stimulates dissolved organic carbon release in coral reef macroalgae. Journal of Phycology, 2020, 56(4): 1039-1052. DOI:10.1111/jpy.13002 |
| [15] |
Dubé C E, Ziegler M, Mercière A, Boissin E, Planes S, Bourmaud C A F, Voolstra C R. Naturally occurring fire coral clones demonstrate a genetic and environmental basis of microbiome composition. Nature Communications, 2021, 12: 6402. DOI:10.1038/s41467-021-26543-x |
| [16] |
Corinaldesi C, Varrella S, Tangherlini M, Dell'Anno A, Canensi S, Cerrano C, Danovaro R. Changes in coral forest microbiomes predict the impact of marine heatwaves on habitat-forming species down to mesophotic depths. Science of the Total Environment, 2022, 823: 153701. DOI:10.1016/j.scitotenv.2022.153701 |
| [17] |
Garren M. Microbial ecology: algae feed a shift on coral reefs. Nature Microbiology, 2016, 1: 16061. DOI:10.1038/nmicrobiol.2016.61 |
| [18] |
Rasher D B, Hay M E. Competition induces allelopathy but suppresses growth and anti-herbivore defence in a chemically rich seaweed. Proceedings Biological Sciences, 2014, 281(1777): 20132615. |
| [19] |
Rasher D B, Stout E P, Engel S, Kubanek J, Hay M E. Macroalgal terpenes function as allelopathic agents against reef corals. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(43): 17726-17731. |
| [20] |
Rosenberg E, Koren O, Reshef L, Efrony R, Zilber-Rosenberg I. The role of microorganisms in coral health, disease and evolution. Nature Reviews Microbiology, 2007, 5(5): 355-362. DOI:10.1038/nrmicro1635 |
| [21] |
Rölfer L, Reuter H, Ferse S C A, Kubicek A, Dove S, Hoegh-Guldberg O, Bender-Champ D. Coral-macroalgal competition under ocean warming and acidification. Journal of Experimental Marine Biology and Ecology, 2021, 534: 151477. DOI:10.1016/j.jembe.2020.151477 |
| [22] |
李锋, 沈城, 张艳苹, 周洁, 彭慧湃, 刘丽. 广东徐闻珊瑚礁国家级自然保护区珊瑚种类及覆盖率分析. 江苏农业科学, 2019, 47(24): 304-308. |
| [23] |
Fu J R, Zhou J, Zhang Y P, Liu L. Effects of Caulerpa taxifolia on physiological processes and gene expression of Acropora hyacinthus during thermal stress. Biology, 2022, 11(12): 1792. DOI:10.3390/biology11121792 |
| [24] |
Manikandan B, Ravindran J. Differential response of coral communities to Caulerpa spp. bloom in the reefs of Indian Ocean. Environmental Science and Pollution Research, 2017, 24(4): 3912-3922. DOI:10.1007/s11356-016-8136-5 |
| [25] |
Bolyen E, Rideout J R, Dillon M R, Bokulich N A, Abnet C C, Al-Ghalith G A, Alexander H, Alm E J, Arumugam M, Asnicar F, Bai Y, Bisanz J E, Bittinger K, Brejnrod A, Brislawn C J, Brown C T, Callahan B J, Caraballo-Rodríguez A M, Chase J, Cope E K, Da Silva R, Diener C, Dorrestein P C, Douglas G M, Durall D M, Duvallet C, Edwardson C F, Ernst M, Estaki M, Fouquier J, Gauglitz J M, Gibbons S M, Gibson D L, Gonzalez A, Gorlick K, Guo J R, Hillmann B, Holmes S, Holste H, Huttenhower C, Huttley G A, Janssen S, Jarmusch A K, Jiang L J, Kaehler B D, Bin Kang K, Keefe C R, Keim P, Kelley S T, Knights D, Koester I, Kosciolek T, Kreps J, Langille M G I, Lee J, Ley R, Liu Y X, Loftfield E, Lozupone C, Maher M, Marotz C, Martin B D, McDonald D, McIver L J, Melnik A V, Metcalf J L, Morgan S C, Morton J T, Naimey A T, Navas-Molina J A, Nothias L F, Orchanian S B, Pearson T, Peoples S L, Petras D, Preuss M L, Pruesse E, Rasmussen L B, Rivers A, Robeson M S, Rosenthal P, Segata N, Shaffer M, Shiffer A, Sinha R, Song S J, Spear J R, Swafford A D, Thompson L R, Torres P J, Trinh P, Tripathi A, Turnbaugh P J, Ul-Hasan S, van der Hooft J J J, Vargas F, Vázquez-Baeza Y, Vogtmann E, von Hippel M, Walters W, Wan Y H, Wang M X, Warren J, Weber K C, Williamson C H D, Willis A D, Xu Z Z, Zaneveld J R, Zhang Y L, Zhu Q Y, Knight R, Caporaso J G. Reproducible, interactive, scalable and extensible microbiome data science using QⅡME 2. Nature Biotechnology, 2019, 37(8): 852-857. DOI:10.1038/s41587-019-0209-9 |
| [26] |
van de Water J A J M, Allemand D, Ferrier-Pagès C. Host-microbe interactions in octocoral holobionts-recent advances and perspectives. Microbiome, 2018, 6(1): 64. DOI:10.1186/s40168-018-0431-6 |
| [27] |
Jenny F, Deignan Lindsey K, Bauman Andrew G, Steinberg Peter D, Diane M, Todd Peter A. Contact- and water-mediated effects of macroalgae on the physiology and microbiome of three indo-pacific coral species. Frontiers in Marine Science, 2020, 6: 831. DOI:10.3389/fmars.2019.00831 |
| [28] |
Sweet M J, Bythell J C, Nugues M M. Algae as reservoirs for coral pathogens. PLoS One, 2013, 8(7): e69717. DOI:10.1371/journal.pone.0069717 |
| [29] |
Hofer U. Coral versus Vibrio. Nature Reviews Microbiology, 2021, 19(8): 481. |
| [30] |
Defosse D L, Johnson R C, Paster B J, Dewhirst F E, Fraser G J. Brevinema andersonii gen. nov., sp. nov., an infectious spirochete isolated from the short-tailed shrew (Blarina brevicauda) and the white-footed mouse (Peromyscus leucopus). International Journal of Systematic Bacteriology, 1995, 45(1): 78-84. DOI:10.1099/00207713-45-1-78 |
| [31] |
Bruno J F, Petes L E, Drew Harvell C, Hettinger A. Nutrient enrichment can increase the severity of coral diseases. Ecology Letters, 2003, 6(12): 1056-1061. DOI:10.1046/j.1461-0248.2003.00544.x |
| [32] |
Barott K L, Rohwer F L. Unseen players shape benthic competition on coral reefs. Trends in Microbiology, 2012, 20(12): 621-628. DOI:10.1016/j.tim.2012.08.004 |
| [33] |
Ritchie K B. Regulation of microbial populations by coral surface mucus and mucus-associated bacteria. Marine Ecology Progress Series, 2006, 322: 1-14. DOI:10.3354/meps322001 |
| [34] |
Briggs A A, Brown A L, Osenberg C W. Local versus site-level effects of algae on coral microbial communities. Royal Society Open Science, 2021, 8(9): 210035. DOI:10.1098/rsos.210035 |
| [35] |
Hughes T P, Kerry J T, Álvarez-Noriega M, Álvarez-Romero J G, Anderson K D, Baird A H, Babcock R C, Beger M, Bellwood D R, Berkelmans R, Bridge T C, Butler I R, Byrne M, Cantin N E, Comeau S, Connolly S R, Cumming G S, Dalton S J, Diaz-Pulido G, Eakin C M, Figueira W F, Gilmour J P, Harrison H B, Heron S F, Hoey A S, Hobbs J P A, Hoogenboom M O, Kennedy E V, Kuo C Y, Lough J M, Lowe R J, Liu G, McCulloch M T, Malcolm H A, McWilliam M J, Pandolfi J M, Pears R J, Pratchett M S, Schoepf V, Simpson T, Skirving W J, Sommer B, Torda G, Wachenfeld D R, Willis B L, Wilson S K. Global warming and recurrent mass bleaching of corals. Nature, 2017, 543(7645): 373-377. DOI:10.1038/nature21707 |
| [36] |
Rosenberg E, Falkovitz L. The Vibrio shiloi/Oculina patagonica model system of coral bleaching. Annual Review of Microbiology, 2004, 58: 143-159. DOI:10.1146/annurev.micro.58.030603.123610 |
| [37] |
Warner M E, Fitt W K, Schmidt G W. Damage to photosystem Ⅱ in symbiotic dinoflagellates: a determinant of coral bleaching. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(14): 8007-8012. |
| [38] |
Blanckaert A C A, de Barros Marangoni L F, Rottier C, Grover R, Ferrier-Pagès C. Low levels of ultra-violet radiation mitigate the deleterious effects of nitrate and thermal stress on coral photosynthesis. Marine Pollution Bulletin, 2021, 167: 112257. DOI:10.1016/j.marpolbul.2021.112257 |
| [39] |
McDevitt-Irwin J M, Baum J K, Garren M, Vega Thurber R L. Responses of coral-associated bacterial communities to local and global stressors. Frontiers in Marine Science, 2017, 4: 262. DOI:10.3389/fmars.2017.00262 |
| [40] |
O'Brien P A, Morrow K M, Willis B L, Bourne D G. Implications of ocean acidification for marine microorganisms from the free-living to the host-associated. Frontiers in Marine Science, 2016, 3: 47. |
| [41] |
Littman R, Willis B L, Bourne D G. Metagenomic analysis of the coral holobiont during a natural bleaching event on the Great Barrier Reef. Environmental Microbiology Reports, 2011, 3(6): 651-660. DOI:10.1111/j.1758-2229.2010.00234.x |
| [42] |
Casas V, Kline D I, Wegley L, Yu Y N, Breitbart M, Rohwer F. Widespread association of a Rickettsiales-like bacterium with reef-building corals. Environmental Microbiology, 2004, 6(11): 1137-1148. DOI:10.1111/j.1462-2920.2004.00647.x |
| [43] |
Antonio D B, Andree K B, Moore J D, Friedman C S, Hedrick R P. Detection of rickettsiales-like prokaryotes by in situ hybridization in black abalone, Haliotis cracherodii, with withering syndrome. Journal of Invertebrate Pathology, 2000, 75(2): 180-182. DOI:10.1006/jipa.1999.4906 |
| [44] |
Gignoux-Wolfsohn S A, Precht W F, Peters E C, Gintert B E, Kaufman L S. Ecology, histopathology, and microbial ecology of a white-band disease outbreak in the threatened staghorn coral Acropora cervicornis. Diseases of Aquatic Organisms, 2020, 137(3): 217-237. DOI:10.3354/dao03441 |
| [45] |
Rosales S M, Miller M W, Williams D E, Traylor-Knowles N, Young B, Serrano X M. Microbiome differences in disease-resistant vs. susceptible Acropora corals subjected to disease challenge assays. Scientific Reports, 2019, 9: 18279. DOI:10.1038/s41598-019-54855-y |
| [46] |
Young B D, Serrano X M, Rosales S M, Miller M W, Williams D, Traylor-Knowles N. Innate immune gene expression in Acropora palmata is consistent despite variance in yearly disease events. PLoS One, 2020, 15(10): e0228514. DOI:10.1371/journal.pone.0228514 |
| [47] |
Davidov Y, Huchon D, Koval S F, Jurkevitch E. A new alpha-proteobacterial clade of Bdellovibrio-like predators: implications for the mitochondrial endosymbiotic theory. Environmental Microbiology, 2006, 8(12): 2179-2188. DOI:10.1111/j.1462-2920.2006.01101.x |
| [48] |
Brown K T, Bender-Champ D, Kenyon T M, Rémond C, Hoegh-Guldberg O, Dove S. Temporal effects of ocean warming and acidification on coral-algal competition. Coral Reefs, 2019, 38(2): 297-309. DOI:10.1007/s00338-019-01775-y |
| [49] |
Hauri C, Fabricius K E, Schaffelke B, Humphrey C. Chemical and physical environmental conditions underneath mat- and canopy-forming macroalgae, and their effects on understorey corals. PLoS One, 2010, 5(9): e12685. DOI:10.1371/journal.pone.0012685 |
| [50] |
Coelho V R, Fenner D, Caruso C, Bayles B R, Huang Y, Birkeland C. Shading as a mitigation tool for coral bleaching in three common Indo-Pacific species. Journal of Experimental Marine Biology and Ecology, 2017, 497: 152-163. DOI:10.1016/j.jembe.2017.09.016 |
| [51] |
Raina J B, Tapiolas D M, Forêt S, Lutz A, Abrego D, Ceh J, Seneca F O, Clode P L, Bourne D G, Willis B L, Motti C A. DMSP biosynthesis by an animal and its role in coral thermal stress response. Nature, 2013, 502(7473): 677-680. |
| [52] |
Hopkins F E, Bell T G, Yang M X, Suggett D J, Steinke M. Air exposure of coral is a significant source of dimethylsulfide (DMS) to the atmosphere. Scientific Reports, 2016, 6: 36031. |
| [53] |
Sunda W, Kieber D J, Kiene R P, Huntsman S. An antioxidant function for DMSP and DMS in marine algae. Nature, 2002, 418(6895): 317-320. DOI:10.1038/nature00851 |
| [54] |
Tout J, Jeffries T C, Petrou K, Tyson G W, Webster N S, Garren M, Stocker R, Ralph P J, Seymour J R. Chemotaxis by natural populations of coral reef bacteria. The ISME Journal, 2015, 9(8): 1764-1777. |
| [55] |
张晓华. 海洋微生物学. 青岛: 中国海洋大学出版社, 2007.
|
| [56] |
Zhang W P, Lu L, Lai Q L, Zhu B K, Li Z R, Xu Y, Shao Z Z, Herrup K, Moore B S, Ross A C, Qian P Y. Family-wide structural characterization and genomic comparisons decode the diversity-oriented biosynthesis of thalassospiramides by marine proteobacteria. Journal of Biological Chemistry, 2016, 291(53): 27228-27238. |
| [57] |
Mydlarz L D, Jones L E, Harvell C D. Innate immunity, environmental drivers, and disease ecology of marine and freshwater invertebrates. Annual Review of Ecology, Evolution, and Systematics, 2006, 37: 251-288. |
| [58] |
Mydlarz L D, Palmer C V. The presence of multiple phenoloxidases in Caribbean reef-building corals. Comparative Biochemistry and Physiology Part A, Molecular & Integrative Physiology, 2011, 159(4): 372-378. |
| [59] |
Ziegler M, Roik A, Porter A, Zubier K, Mudarris M S, Ormond R, Voolstra C R. Coral microbial community dynamics in response to anthropogenic impacts near a major city in the central Red Sea. Marine Pollution Bulletin, 2016, 105(2): 629-640. |
| [60] |
Morrow K M, Bourne D G, Humphrey C, Botté E S, Laffy P, Zaneveld J, Uthicke S, Fabricius K E, Webster N S. Natural volcanic CO2 seeps reveal future trajectories for host-microbial associations in corals and sponges. The ISME Journal, 2015, 9(4): 894-908. |
 2024, Vol. 44
2024, Vol. 44




