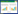文章信息
- 叶董, 王瑞, 王京, 言迫, 卓玛姐, 刘丹, 林英华
- YE Dong, WANG Rui, WANG Jing, YAN Po, ZHUO Majie, LIU Dan, LIN Yinghua
- 若尔盖高寒湿地土壤细菌群落和生态网络对围封禁牧与恢复放牧的响应
- Response of soil bacterial communities and ecological networks to grazing enclosure and re-grazing in Zoige Alpine wetland
- 生态学报. 2024, 44(24): 11229-11240
- Acta Ecologica Sinica. 2024, 44(24): 11229-11240
- http://dx.doi.org/10.20103/j.stxb.202312222786
-
文章历史
- 收稿日期: 2023-12-22
- 网络出版日期: 2024-09-09
2. 四川若尔盖湿地国家级自然保护区管理局, 阿坝藏族羌族自治州 624599
2. Administration of Zoige Wetland National Nature Reserve, Aba 624599, China
若尔盖高寒湿地位于青藏高原东部, 是我国面积最大、保存最完整的高寒泥炭沼泽湿地, 对于维持青藏高原生态系统稳定具有重要作用[1]。受全球气候变化与超载放牧等人类活动过度干扰, 若尔盖高寒湿地面积呈现不同程度萎缩与退化, 其湿地生态功能下降已引起关注[2]。过度放牧不仅改变了地上与地上生物之间的关系, 并对土壤微生物群落特征与功能产生重要影响, 导致生物多样性功能下降。因此, 评估放牧干扰对若尔盖高原湿地生态系统的影响, 制定科学放牧强度已成为生态学研究的热点之一[3]。
微生物是土壤生态系统健康的重要生物指示物, 其在土壤养分循环及其对退化地上生态系统恢复具有功能性作用[4—5]。研究表明, 土壤微生物通过参与土壤养分循环影响地上生物量与植物群落多样性。Hannula等[6]认为, 与土壤真菌相比, 土壤细菌群落受环境遗留效应的响应低, 其变化与植物群落变化密切相关。土壤细菌的相对丰度与其在生态系统中的功能作用密切相关[7], 相对丰度越高的土壤细菌适应能力越强[8], 但受到环境胁迫时, 相对丰度较低的稀有类群的适应性与耐受性均超过相对丰度较高的优势类群[9—10], 且土壤细菌种间相互作用或共存模式对环境胁迫的响应存在显著的差异性[11]。因此, 探究土壤细菌类群丰度及其相互关系对环境变化的响应, 有助于进一步认识土壤细菌群落生态过程。
围封禁牧是若尔盖高寒湿地近自然恢复恢复主要途径之一[12]。围封禁牧因禁止牦牛家畜活动等干扰, 直接或间接影响湿地地下和地上生态系统之间的相互关系, 并对湿地土壤微生物群落产生重要影响[13], 但围封禁牧对土壤细菌群落的影响因时间与小尺度空间异质性而存在异议[14—16], 且围封禁牧研究较多关注时间尺度土壤细菌群落丰度与多样性变化[17—18], 少有关注围封禁牧区恢复放牧后湿地土壤微生物群落组成、多样性与种间关系及其对干扰变化的响应[19]。网络分析是揭示种间关系或物种共存模式的重要方法[20]。虽然网络高度连接物种或网络节点物种是网络关键物种和较重要物种, 且对土壤微生物群落的复杂性与稳定性具有功能性作用, 但受气候因素、植被类型与禁牧时间等多种因素影响, 放牧与禁牧对土壤微生物群落影响存在较大的差异[21]。因此研究同一区域土壤细菌群落组成与共现网络对围封禁牧和恢复放牧的响应, 对进一步揭示围封禁牧对土壤微生物群落结构及其稳定性具有重要意义。
本研究以若尔盖湿地国家级自然保护区2016年湿地生态效益补偿(试点)项目建立的围封禁牧试验区(2017—2021年)为研究区域, 以围封禁牧区外围受持续放牧区域为对照样地, 利用16S rRNA基因的高通量扩增子测序技术, 研究围封禁牧3年、围封禁牧5年与恢复放牧1年后土壤细菌群落组成、多样性与生态网络特征, 以探究土壤细菌群落丰富类群与稀有类群与生态网络特征对禁牧与恢复放牧的响应, 为进一步认识揭示若尔盖高寒湿地土壤细菌群落对围封禁牧的适应策略提供依据。
1 材料与方法 1.1 研究区域自然概况研究区域位于若尔盖湿地国家级自然保护区, 地理位置102°29′—102°59′E, 33°25′—34°00′N。属高原寒温带湿润气候, 年均温度0.7℃, 年降水量493.6—836.7mm, 主要集中于5—8月, 约占全年降水的50%以上。年蒸发量约1260—1290mm。属高原浅丘沼泽地貌, 平均海拔3500—3600m。植被类型为沼泽植被和亚高山草甸植被, 优势植物以华扁穗草(Blysmus sinocompressus)、黑褐穗薹草(Carex atrofusca)、西藏嵩草(Kobresia tibetica)、高原嵩草(Kobresia pygmaea)、蕨麻(Argentina anserina)等为主。土壤类型为沼泽土和高原草甸土[22]。
1.2 研究方法 1.2.1 样地选择和样品采集研究以围封禁牧区中心为研究区域, 选择地势平坦、植被类型基本一致的区域为研究区, 分别设定100m×100m样地方, 在样地内沿对角设置5个10m×10m的样方。
2019年、2021年和2022年7月中旬, 在10m×10m样方内, 沿对角线设置5个20cm×20cm样方, 常规法调查样方内植物种类、高度、盖度与频度, 并计算重要值[23];采用刈割法收获样方内的植物地上生物量, 装入自封袋中, 带回实验室称其鲜重后, 置于105℃烘箱杀青后, 65℃下烘干至恒重, 称其干重量。利用直径为5cm的不锈钢土钻随机采集样方内土壤剖面0—20cm的土壤样品3—5钻, 剔除样品中可见的植物根系、凋落物、石子并混合均匀后, 一部分放入4℃保鲜箱内, 带回实验室置于-80℃保存, 用于土壤细菌群落多样性分析;另一部分带回实验, 部分样品采用烘干法测定土壤含水量, 其余样品经过自然风干后, 常规方法测量土壤有机质、全氮含量与pH(表 1)。
| 环境变量 Environment variables |
持续放牧 Grazing |
禁牧3年 3-year Non-Grazing |
禁牧5年 5-year Non-Grazing |
恢复放牧 Re-Grazing |
| 土壤pH Soil pH | 6.19±0.19b | 6.79±0.16a | 6.10±0.17b | 6.28±0.23b |
| 土壤有机质Organic matter (OM)/(g/kg) | 150.51±123.38ab | 61.44±7.45b | 90.97±19.47a | 59.49±15.87b |
| 土壤全氮Total nitrogen(TN)/(g/kg) | 9.21±6.50a | 3.99±0.20a | 4.10±0.90a | 4.47±0.88a |
| 土壤碳氮比Carbon/Nitrogen(C/N) | 8.36±1.66bc | 8.91±0.73b | 12.89±0.60a | 7.61±0.62c |
| 土壤含水量Moisture content(MC)/% | 41.05±24.17ab | 50.40±2.29a | 33.65±5.08b | 27.71±4.50b |
| 地上生物量Above-ground biomass(AB)/g (20cm×20cm) |
22.42±7.39b | 77.12±20.63a | 12.22±4.81b | 12.98±5.11b |
| 植被重要值Importance value() IV | 0.34±0.08b | 0.72±0.11a | 0.28±0.08b | 0.32±0.14b |
| 植被优势物种Dominant species (DS) | 扁穗草(Blysmus sp.)、 嵩草(Kobresia sp.)、 委陵菜(Potentilla sp.) |
披碱草(Elymus sp.)、 早熟禾(Poa sp.)、 亚菊(Ajania sp.) |
薹草(Carex sp.)、 披碱草(Elymus sp.)、 蒿(Artemisia sp.) |
披碱草(Elymus sp.)、 扁穗草(Blysmus sp.)、 附地菜(Trigonotis sp.) |
| 数据以平均值±标准误差表示, 同行不同小写字母表示差异显著(P < 0.05) | ||||
称取0.5g新鲜土壤, 使用土壤DNA试剂盒提取土壤总DNA (Fast DNA Spin kit Bio 101, Carlsbad, CA, USA)。NanoDrop 2000 UV-vis分光光度计(Thermo Scientific, Wilmington, USA)测定最终纯化后的DNA浓度;1%的琼脂糖凝胶电泳检测DNA质量。用引物338F(5′-ACTCCTACGGGAGGCAGCAG-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′)扩增细菌16S rRNA基因的V3—V4区。PCR反应体系20μL:5×Trans Start FastPfu缓冲液4μL, 正向和反向引物各0.8μL, 2.5mmol/L dNTPs 2μL, DNA聚合酶0.4μL, 模版DNA 10ng, ddH2O补足至20uL。PCR反应条件:初始95℃变性3min, 95℃变性10s、55℃退火30s、72℃延伸30s, 27个循环, 72℃延伸10min。同一样本的PCR产物混合后, 2%琼脂糖凝胶回收PCR产物;AxyPrep DNA Gel Extraction Kit(Axygen Biosciences, Union City, CA, USA)纯化PCR产物, QuantusTM Fluorometer (Promega, USA)检测、定量。NEXTflexTM Rapid DNA-Seq Kit(Bioo科技公司)建库, Miseq PE 300平台(美国Illumina公司)测序, 以上由上海美吉生物科技有限公司完成。
原始序列通过Trimmomatic软件、Flash软件质控、拼接, 并利用Uparse软件按照97%的相似度阈值对序列进行OTU聚类基础上, 通过Silva 16S rRNA数据库(v138)进行物种分类学注释, 置信度阈值为0.70。
为保证研究区域不同样本测序序列的均一性, 本研究并对全部样本OTUs按最小样本量进行样本序列抽平后, 进行后续分析。
1.3 数据分析参照Liang等[9]的方法:将全部样品中相对丰度≥0.1%和全部样品中相对丰度≥0.01%并且部分样品相对丰度≥0.1%分别定义为丰富类群(AT)和条件丰富类群(CAT);全部样品中相对丰度 < 0.01%和全部样品中相对丰度 < 0.1%并且部分样品相对丰度 < 0.01%被划分为始终稀有类群(ART)和条件稀有类群(CRT);在全部样品中0.01%≤相对丰度 < 0.1%被划分为中等丰度类群(MT);部分样品相对丰度 < 0.01%部分样品相对丰度≥0.1%被划分为条件稀有和丰富类群(CRAT)。其中, 始终丰富类群(AAT)和条件丰富类群(CAT)又被划分为丰富类群(AT), 始终稀有类群(ART)和条件稀有类群(CRT)被划分为稀有类群(RT)。
土壤细菌群落α多样性采用Chao1丰富度指数、Shannon多样性指数和Pielou均匀度指数表征。采用堆叠柱形图与Venn图分析研究区域的土壤细菌群落物种组成、共有和特有OTU, 并通过Kruskal-Wallis秩和检验判断土壤细菌丰度和多样性差异性。
基于Bray-Curtis距离矩阵的主坐标分析(principal co-ordinates analysis, PCoA)分析土壤细菌群落组成的差异;置换多因素方差分析(permutational MANOVA, PERMANOVA)与相似性分析(Analysis of similarities, ANOSIM)检验围封禁牧与恢复放牧对土壤细菌群落组成差异的显著性;Bray-Curtis相异分布指数评估围封禁牧与恢复放牧区土壤细菌群落β多样性;Mantel检验计算环境因子矩阵与土壤细菌OTU丰度矩阵的相关性, 筛选与土壤细菌群落组成显著相关的环境因子。
为了评估研究区域不同细菌群落之间的相关性, 筛选、保留同一处理中出现频率大于或等于3样本的OTUs, 并基于Spearman相关系数构建共现网络, 且当Spearman相关系数|R|>0.8, P < 0.01时, 则认为具有强相关性[24]。网络拓扑特征通过网络节点、边、平均度与平均聚集系数等表征, 并使用网络的内聚力分析评估禁牧与恢复放牧对环境压力的影响[25]。节点拓扑作用通过节点的模块内的连通性(Zi)以及模块间连通性(Pi)评估, 其中Zi≤2.5, Pi≤0.62为外围节点, Zi>2.5, Pi≤0.62为模块中心点, Zi≤2.5, Pi>0.62为连接节点, Zi < 2.5, Pi>0.62定义为网络中心点。
以上分析通过件SPSS 20.0、R(4.1.2)包“vegan”、“Hmisc”、“ggClusterNet”完成, 并通过Gephi软件对网络可视化。
2 结果与分析 2.1 土壤细菌群落多样性数据显示, 经过围封禁牧和恢复放牧土壤细菌α多样性指数均发生了显著变化(P < 0.05), 其中土壤细菌全部群落(WT)中, Chao1丰富度指数、Shannon多样性指数和Pielou均匀度指数均以NG3最大, G的Chao1指数、RG的Shannon指数与Pielou均匀度指数最小;丰富类群(AT)中, G的Chao1指数没有变化, Shannon指数、Pielou指数大小变化趋势一致, 均为NG3>NG5>G>RG;稀有类群(RT)中, Chao1丰富度指数、Shannon多样性指数和Pielou均匀度指数则均表现为NG3>RG>NG5>G(图 1)。
|
| 图 1 研究区域土壤细菌群落的α多样性指数 Fig. 1 The α diversity index of soil bacterial communities in study area 箱型图中横线为中位数, 灰点为异常值标注; WT: 全部类群Whole taxa; AT: 丰富类群Abundant taxa; RT: 稀有类群Rare taxa; G: 持续放牧Grazing; NG3:禁牧3年3-year non-grazing; NG5: 禁牧5年5-year non-grazing; RG: 恢复放牧Re-grazing; 不同小写字母表示差异显著(P < 0.05) |
Kruskal-Wallis秩和检验显示, 与对照G相比, 仅NG3土壤细菌α多样性发生显著变化(P < 0.05), 除AT的Chao1指数外, NG3的Shannon多样性指数和Pielou均匀度指数值均最高(P < 0.05), 但NG5与RG间Shannon指数差异不显著(P>0.05)(图 1)。
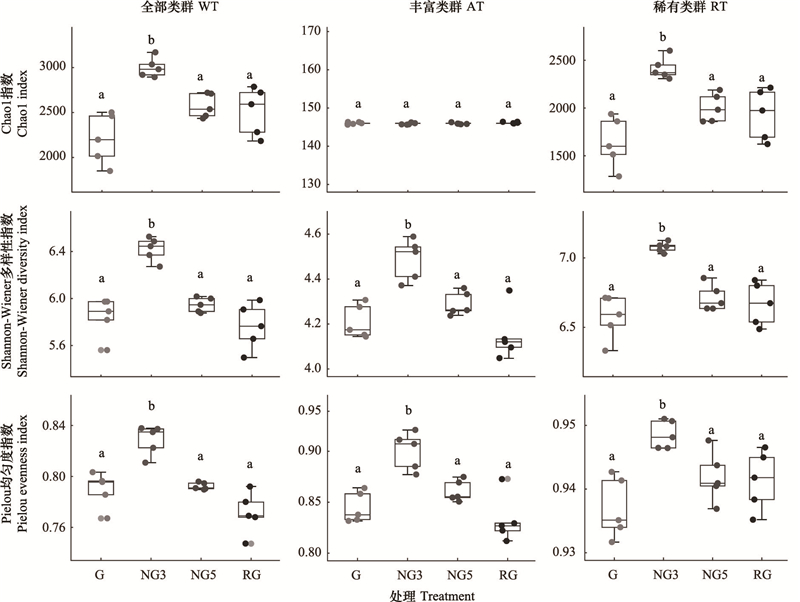
2.2 土壤细菌群落组成研究时段, 共获得了具有97%同一性的5334个OTU, 土壤样本测序共获得46细菌门, 149纲, 337目, 510科, 841属, 共1797种, 其中相对丰度均大于1%的主要优势门是放线菌门(Actinobacteriota)、变形菌门(Proteobacteria)、酸杆菌门(Acidobacteriota)、绿弯菌门(Chloroflexi)和芽单胞菌门(Gemmatimonadota), 其比例分别为27.06%、18.74%、17.62%、10.18%、2.63%;相对丰度均大于1%的主要优势属是unclassified_Vicinamibacterales、unclassified_Gaiellales、unclassified_Rokubacteriales、RB41、unclassified_Xanthobacteraceae和unclassified_Gemmatimonadaceae, 其比例分别为5.11%、5.04%、4.32%、3.91%、3.37%、2.32%(图 2)。
|
| 图 2 土壤细菌在门和属水平上群落组成 Fig. 2 Community composition of soil bacteria at phyla and genus level 编号1—5为同一处理的样品编号;Actinobacteriota: 放线菌门; Proteobacteria: 变形菌门; Acidobacteriota: 酸杆菌门; Chloroflexi: 绿弯菌门; Firmicutes: 厚壁菌门; Verrucomicrobiota疣微菌门; Gemmatimonadota: 芽单胞菌门; Bacteroidota: 拟杆菌门; Myxococcota: 粘球菌门; unclassified_Rokubacteriales: 罗库菌目未定属; unclassified_Xanthobacteraceae: 黄色杆菌科下未定属; Bacillus: 芽孢杆菌属 |
Kruskal-Wallis秩和检验显示, 除变形菌门外, 放线菌门、酸杆菌门、绿弯菌门和芽单胞菌门丰度均存在显著性差异(P < 0.05), 其中G和NG3放线菌门丰度显著低于NG5(P < 0.05), G和NG3酸杆菌门丰度显著高于NG5和RG(P < 0.05), RG绿弯菌门丰度明显小于G、NG3和NG5(P < 0.05);NG3芽单胞菌门丰度明显大于G、NG5和RG(P < 0.05)。除unclassified_Vicinamibacterales、unclassified_Rokubacteriales外, G和NG3的unclassified_Gaiellales丰度明显小于NG5、RG(P < 0.05), RG的RB41丰度明显大于G与NG3(P < 0.05), NG3的unclassified_Xanthobacteraceae丰度明显小于G与RG(P < 0.05), unclassified_Gemmatimonadaceae丰度在NG3明显大于G和NG5(P < 0.05), 表明主要土壤细菌优势菌对围封禁牧与恢复放牧的响应不同。
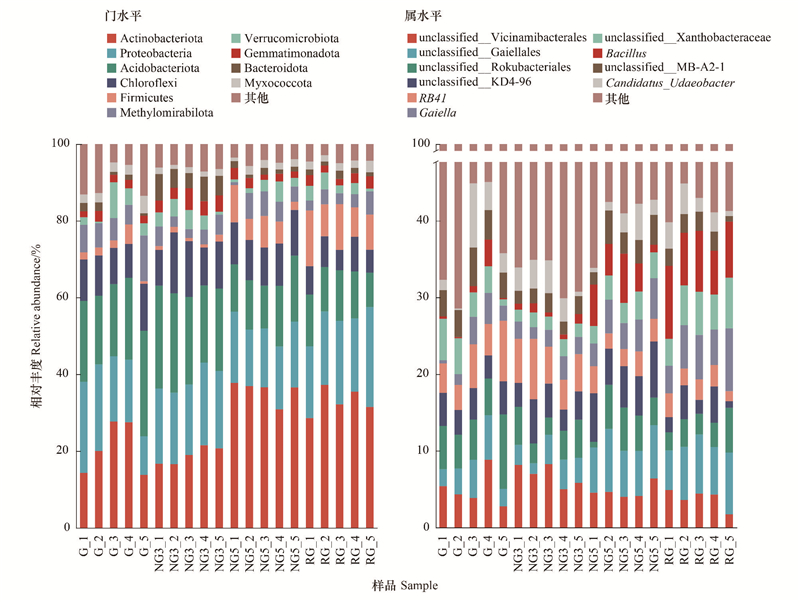
PCoA分析显示, 研究区域土壤细菌群落结构存在明显的差异(P < 0.05), 但仅NG5和RG间呈现一定聚集(图 3), 显示研究区域NG5和RG土壤细菌群落组成相似。ANOSIM和PERMANOVA分析显示, 与G相比, 围封禁牧与恢复放牧后的WT、AT与RT的群落组成发生明显改变(P < 0.05), 但NG5与RG之间WT与RT的群落组成没有明显变化(P>0.05), 并且AT在两个检验中的R值均大于RT, 表明AT的群落结构比RT更敏感, 特别是对恢复放牧的响应(表 2)。
|
| 图 3 土壤细菌群落组成差异 Fig. 3 Different composition of soil bacterial communities PCoA: 主坐标分析Principal co-ordinates analysis; 箱型图中横线为中位数, 灰点为异常值标注; 不同小写字母表示差异显著(P < 0.05) |
| 检验方法 Test method |
丰度类群 Abundance taxa |
G vs. NG3 | G vs. NG5 | G vs. RG | NG3 vs. NG5 | NG3 vs. RG | NG5 vs. RG | |
| 置换多元方差检验 | 全部类群 | R | 0.331 | 0.263 | 0.318 | 0.415 | 0.514 | 0.158 |
| PERMANOVA | P | 0.009 | 0.005 | 0.008 | 0.003 | 0.008 | 0.118 | |
| 丰富类群 | R | 0.343 | 0.299 | 0.404 | 0.513 | 0.641 | 0.241 | |
| P | 0.009 | 0.006 | 0.008 | 0.009 | 0.012 | 0.007 | ||
| 稀有类群 | R | 0.278 | 0.198 | 0.216 | 0.290 | 0.323 | 0.111 | |
| P | 0.015 | 0.027 | 0.016 | 0.012 | 0.009 | 0.426 | ||
| 相似性检验 | 全部类群 | R | 0.596 | 0.376 | 0.548 | 0.932 | 0.992 | 0.120 |
| ANOSIM | p | 0.008 | 0.012 | 0.008 | 0.010 | 0.009 | 0.192 | |
| 丰富类群 | R | 0.552 | 0.432 | 0.608 | 0.964 | 1.000 | 0.328 | |
| P | 0.005 | 0.017 | 0.006 | 0.008 | 0.005 | 0.011 | ||
| 稀有类群 | R | 0.592 | 0.392 | 0.484 | 0.880 | 0.916 | 0.024 | |
| P | 0.005 | 0.020 | 0.011 | 0.010 | 0.009 | 0.386 | ||
| PERMANOVA: 置换多元方差检验Permutational multivariate analysis of variance; ANOSIM: 相似性分析Analysis of similarities; G: 持续放牧Grazing; NG3:禁牧3年3-year Non-grazing; NG5:禁牧5年5-year Non-grazing; RG: 恢复放牧Re-grazing | ||||||||
Venn图结果显示, 土壤细菌WT、AT和RT的共有OTU数量分别为1927(36.12%)、146(17.07%)、1330(23.17%), 其中土壤细菌WT与RT的独有OTU均以NG3最多, 其次是G, RG数量最少, 且NG5、RG的独有OTU全部来自RT, 显示围封禁牧与恢复放牧独有菌群主要来自RT(图 3)。
基于Bray-Curtis相异度分布指数分析显示, 土壤细菌群落的WT、AT和RT的相异度系数变化均为以G最大, 其次是NG5, WT和AT相异系数RG最低, 而RT相异系数NG3最低。G的相异度指数显著高于NG3、NG5与RG(P < 0.05), 但RG与NG3和及NG5之间没有显著差异(P>0.05), 显示围封禁牧与恢复放牧后, 土壤细菌群落扩散能力降低, 显著降低了土壤细菌群落β多样性(图 3)。
2.3 土壤细菌群落网络特征数据显示(图 4、表 3), 共现网络总节点、总边大小均为NG3>RG>NG5>G, 反映出NG3共现网络相对最复杂, G共现网络相对最简单, 表明禁牧增加了网络的复杂度, 但随着禁牧时间延长, 其复杂度虽有所下降, 但却仍高于持续放牧对照。共现网络的边能够反映节点之间的关系, 本研究中共现网络均以正相关边为主, 在不同处理中占总边的比例从大到小为RG>G>NG5>NG3。模块化指数能够反映网络抗干扰能力的稳定性。在共现网络中, NG3的模块化指数最高, G最低, RG与NG5之间模块化指数最为接近; 而G的平均度大幅高于其他处理、平均路径长度最短, 但平均聚类系数最低;NG3的平均路径长度最大, 平均聚类系数较低; 而RG的平均聚类系数最高, 平均路径长度较短, 显示NG3网络抗干扰能力的稳定性与网络中节点之间的紧密程度均最强, 表明持续放牧状态下网络简单, 但节点之间连接更为紧密, 而网络在围封禁牧后, 网络更为复杂但节点之间连接松散。共现网络中与RT节点连接的边占网络总边的73.47%—89.63%, 且以NG3中比例最高, G中比例最低;与AT节点连接的边仅占网络总边的13.59%—21.82%, 其中G中与AT节点连接的边比例最高, 在NG3中比例最低, 表明RT是共现网络的主要组成部分, 禁牧增加了与RT连接边的比例, 减少了与AT连接边的比例。
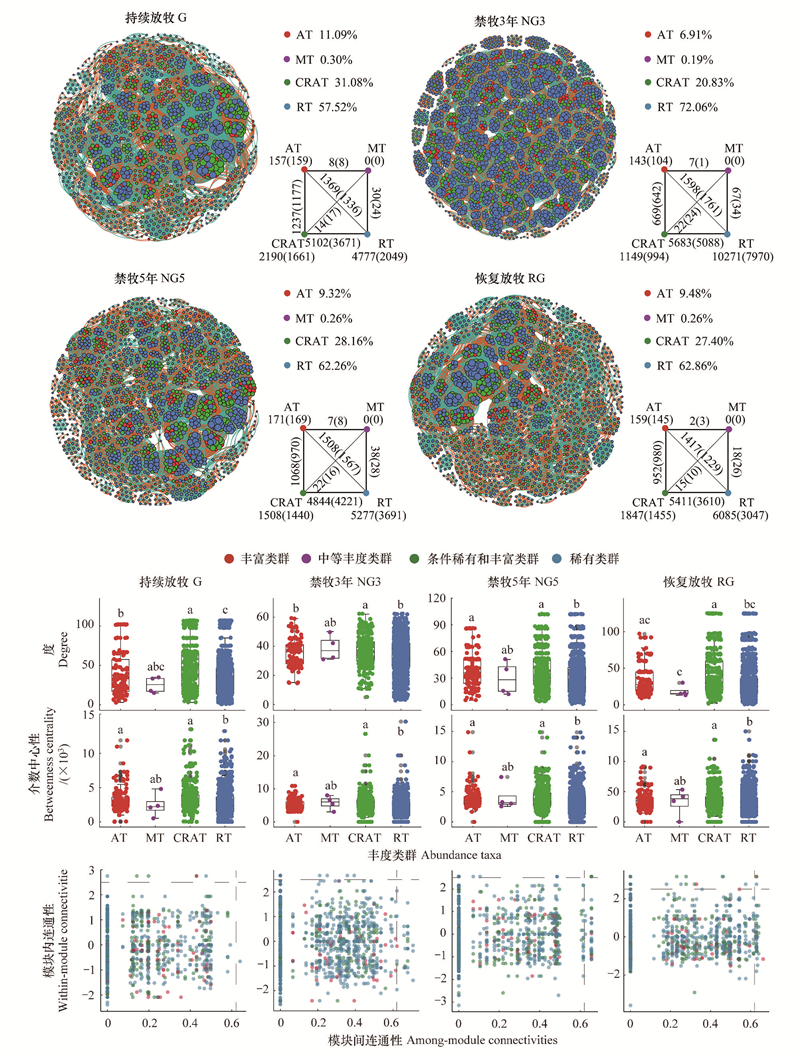
|
| 图 4 研究区域土壤细菌群落共现网络 Fig. 4 Soil bacterial community co-occurrence networks in study area AT: 丰富类群Abundant taxa; MT: 中等丰度类群Moderate taxa; CRAT: 条件稀有和丰富类群Conditionally rara and abundant taxa; RT: 稀有类群Rare taxa; 节点大小代表该节点的度, 蓝绿色线代表正相关边, 橙红色的线代表负相关边, 括号外为正相关边数量, 括号内为负相关边数量, 不同丰度类群节点拓扑属性以及拓扑角色分布, 箱型图中横线为中位数, 灰点为异常值标注, 不同小写字母表示差异显著(P < 0.05) |
| 拓朴属性 Topological characteristics |
持续放牧 Grazing |
禁牧3年 3-year Non-grazing |
禁牧5年 5-year Non-grazing |
恢复放牧 Re-grazing |
| 总节点Total nodes | 1316 | 2112 | 1566 | 1540 |
| 总边Total edges | 24986 | 36227 | 26593 | 26411 |
| 正相关边比例Positive edges percentage/% | 59.57 | 54.13 | 54.46 | 60.22 |
| 负相关边比例Negative edges percentage/% | 40.43 | 45.87 | 45.54 | 39.78 |
| 平均度Average degree | 37.97 | 34.31 | 33.96 | 34.30 |
| 平均路径长度Average path length | 6.55 | 6.86 | 6.64 | 6.64 |
| 平均聚集系数Average clustering coefficient | 0.63 | 0.63 | 0.64 | 0.65 |
| 模块化指数Modularity | 0.67 | 0.77 | 0.73 | 0.73 |
对不同丰度类群节点的拓扑属性进行比较, 发现CRAT节点的拓扑属性始终处于较高水平, 并显著高于RT, 而AT的介数中心性也始终显著高于RT(P < 0.05)(图 4), 表明共现网络中AT和CRAT在网络中的节点之间的连接中具有更重要的地位。按节点在模块内外的连通性划分关键节点, 发现绝大部分节点为外围节点, 而出现的关键节点包括连接节点、模块中心点和网络节点, 数量分别为103、56和6。在G的共现网络中共有7个关键节点, 包括6个模块中心点和1个连接节点;在NG3的共现网络中共有38个关键节点, 包括9个模块中心点和29个连接节点;在NG5的共现网络中共有49个关键节点, 包括15个模块中心点、31个连接节点和3个网络中心点;在NG5的共现网络中共有71个关键节点, 包括26个模块中心点、42个连接节点和3个网络中心点。放牧时网络的模块中心点占关键节点的比例更高, 而禁牧时连接节点比例更高, 表明放牧时在模块内的节点交流更紧密, 而禁牧时能够增加不同模块间的交流。按照丰度类群划分, 在关键节点中AT、MT、CRAT和RT占有的数量分别为14、1、49、101, 表明土壤细菌的共现网络中的相互作用关系更依赖于RT的互作模式。
2.4 影响土壤细菌群落的关键环境因子Mantel检验显示, 土壤细菌群落与pH、OM、TN、MC和AB均存在显著相关性(图 5)。其中, WT与OM、TN、MC有较强的显著相关性(P < 0.05), 其相关系数分别为0.61、0.62、0.62;AT与OM、TN、MC有较强的显著相关性(P < 0.05), 其相关系数分别为0.43、0.44、0.57;RT与OM、TN、MC有较强的显著相关性(P < 0.05), 相关系数分别为0.59、0.60、0.59。pH与WT、AT和RT的组成均有较弱的显著相关性, AB与AT的组成有着较弱的显著相关性(P < 0.05, |R| < 0.4), 表明土壤理化性质是影响土壤细菌群落各丰度类群组成的主要因素, 而地上生物量与丰富类群的群落组成有一定的联系。
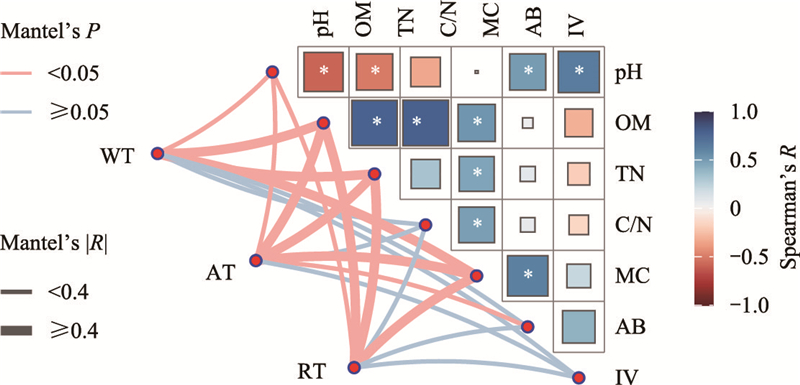
|
| 图 5 不同丰度类群组成与环境因子的相关性 Fig. 5 The correlation between different abundance taxa and environmental factors 线条颜色代表Mantel检验的显著性; 线宽代表Mantel检验R的绝对值; 颜色梯度代表Spearman相关系数, *表示相关性显著(P < 0.05); OM: 土壤有机质Organic matter; TN: 土壤全氮Total nitrogen; C/N: 土壤碳氮比Carbon/Nitrogen; MC: 土壤含水量Moisture content; AB: 地上生物量Above-ground biomass; IV: 植被重要值Importance value |
围封禁牧是调节生态系统功能恢复的有效措施[26], 本研究设置不同禁牧时间以及恢复放牧处理, 探究高寒湿地土壤细菌群落对禁牧和再次放牧干扰的响应。研究发现, 围封禁牧明显改变若尔盖高寒湿地土壤细菌群落多样性, 与持续放牧的对照G相比, 围封禁牧3年明显增加土壤细菌群落的多样性, 这与围封禁牧后, 减少了牦牛等牲畜的践踏, 地表植被组成与生物量增加有关[18]。随着禁牧时间增加到5年, 土壤细菌群落多样性已经与持续放牧G没有显著差异, 这是因为长时间禁牧与植物多样性有着强烈的负效应[27], 植物多样性的降低进而对土壤细菌群落的多样性产生了负面影响[18]。禁牧解除后恢复了放牧对植被的影响, 但与禁牧5年时相比, 土壤细菌的群落多样性以及群落全部类群的组成并没有显著变化, 这可能是因为禁牧后的土壤细菌群落已经对环境扰动具有更高的抵抗能力[28]。
土壤细菌群落对环境变化十分敏感, 并会根据外部扰动调整其生态位[29]。研究发现, 禁牧前后土壤细菌群落组成发生显著变化, 这可以用环境因子的变化解释。土壤细菌的各丰度类群的组成与土壤有机质、全氮和含水量有着较强的相关性。其中, 土壤有机质是土壤中的主要营养物质, 而植被是土壤碳氮的重要自然来源, 并且不同的植物种类在向土壤提供营养物质的数量和质量有差异[30—31]。禁牧通过植物群落及其枯落物的组成变化改变土壤有机质、全氮的含量对土壤细菌群落的组成产生了显著影响。土壤含水量是恢复沼泽化草甸的重要指标, 同时也能改变土壤好氧和厌氧条件, 是影响土壤细菌的代谢过程、群落组成的重要因素[32]。土壤pH被认为是土壤细菌群落多样性差异的驱动因素, 对土壤细菌群落的结构有着重要的影响[33], 但在本研究中, pH与土壤细菌群落各丰度类群只有较弱的显著相关性, 表明在研究区域内pH并不是影响土壤细菌群落组成的主导因子。地上生物量能够反映地上植被的生长状况, 而植被能够影响细菌群落的组成[6], 地上生物量能够与土壤细菌群落建立较强的联系[1]。本研究地上生物量仅与丰富类群的组成具有显著相关性, 这可能是丰富类群相较于全部类群和稀有类群对恢复放牧更具敏感性的原因。
土壤微生物群落的组成变化往往与其参与的生态系统功能密切相关[24]。在禁牧5年时, 土壤的有机质含量显著增加, 而放线菌门在禁牧5年时的丰度显著增加, 并且放线菌门被认为能够促进植物残留物的分解[34], 推测有机物质的积累引起了放线菌门丰度的显著变化。变形菌门在不同处理之间丰度没有出现显著差异, 可能受禁牧和放牧的影响较小。有研究发现, 变形菌门同样为能够分解难以分解的有机质, 参与能量代谢[35], 并且在不同退化程度的高寒草地中是优势物种[36]。土壤中的酸杆菌通常受土壤酸碱度的影响[37], 因此推测酸杆菌门的丰度与土壤pH变化相关, 线性回归拟合结果验证了这一点(y=2211.4x-9767, P=0.013 < 0.05)。绿弯菌通常在厌氧环境下更能发挥功能[38], 其丰度变化与土壤含水量变化相吻合。
3.2 土壤细菌网络对禁牧和恢复放牧的响应共现网络的结构可以反映微生物之间的相互作用, 相连接的节点之间可能潜在着合作、共存、捕食、竞争等关系[39]。本研究, 网络均以正相关关系为主, 放牧行为可能使得环境胁迫加剧, 土壤细菌群落可能潜藏着合作关系以应对恶劣的环境[40], 因而在进行放牧(G和RG)的网络中正相关比例较高。与Wang等[26]的研究结果不同, 本研究中禁牧降低了网络的连通性, 但增加了网络的复杂度和模块化水平以及模块中心点的比例, 表明禁牧能够缓解土壤细菌群落的环境压力, 促使更多土壤细菌进行相互作用, 但群落中种间关系整体变得松散, 而相对增加了模块内的物种之间联系的紧密程度, 增加了对环境变化的抵抗能力[25]。然而这个效果会随禁牧时间增加而下降, 这与本研究发现的围封禁牧导致植物群落简单、土壤含水量下降与土壤C/N增加有关(表 1), 其植物群落变化与王娟等[27]发现的围封禁牧导致植物群落组成趋异性演替相一致。
禁牧和放牧改变了土壤细菌群落中禁牧能够改变不同丰度类群之间的联系。本研究, 丰富类群拓扑属性更高, 占据核心地位, 但生态禁牧增加了稀有类群节点与各丰度类群边的比例, 表明禁牧能够促进土壤细菌群落中稀有类群的互作能力。禁牧带来的环境变化可能满足更多稀有类群的适宜条件[20], 使其能够与其他细菌进行互作, 有利于更多细菌种类的共存。此外, 在禁牧后土壤细菌关键节点数量增加, 并且关键节点以稀有类群为主, 表明禁牧使土壤细菌群落整体物种间相互作用的模式发生转变, 形成了以稀有类群为主的互作模式, 这些关键节点大部分隶属于嗜热油菌纲(Thermoleophilia)、放线菌纲(Actinobacteria)和Vicinamibacteria。与丰富类群相比, 稀有类群更为稳定, 在干扰环境压力变化的情况下能够维持土壤细菌群落功能[9], 这可能是禁牧后土壤细菌群落整体对干扰更具抵抗力的原因。在恢复放牧后, 网络中仍然保持着大量稀有类群的关键节点(图 4), 使恢复放牧后土壤细菌群落网络仍保持较高的复杂性。
4 结论围封禁牧显著改变了土壤细菌群落的多样性、组成和群落结构, 其中围封禁牧3年显著增加了土壤细菌群落的多样性, 稀有类群的α多样性对围封禁牧响应较为敏感。恢复放牧后, 丰度类群组成变化比全部类群更加敏感。研究显示, 禁牧能够增加土壤细菌群落生态网络的复杂性、稳定性, 并形成了以稀有类群为主的互作模式。围封禁牧和恢复放牧主要通过改变土壤性质中OM、TN和MC影响土壤细菌群落组成。综上所述, 围封禁牧有利于土壤细菌群落的稳定;丰富类群细菌对围封禁牧和放牧的敏感性, 可作为不同放牧方式响应的生物指示指标;而围封禁牧使稀有类群在细菌网络中发挥更大的作用。
| [1] |
王好才, 夏敏, 刘圣恩, 王燚, 展鹏飞, 王行. 若尔盖高原泥炭沼泽湿地土壤细菌群落空间分布及其驱动机制. 生态学报, 2021, 41(7): 2663-2675. |
| [2] |
张倚浩, 阎建忠, 程先. 气候变化与人类活动对青藏高原湿地的影响研究进展. 生态学报, 2023, 43(6): 2180-2193. |
| [3] |
Xiang S, Guo R Q, Wu N, Sun S C. Current status and future prospects of Zoige Marsh in Eastern Qinghai-Tibet Plateau. Ecological Engineering, 2009, 35(4): 553-562. DOI:10.1016/j.ecoleng.2008.02.016 |
| [4] |
Harris J. Soil microbial communities and restoration ecology: facilitators or followers?. Science, 2009, 325(5940): 573-574. DOI:10.1126/science.1172975 |
| [5] |
Wagg C, Schlaeppi K, Banerjee S, Kuramae E E, van der Heijden M G A. Fungal-bacterial diversity and microbiome complexity predict ecosystem functioning. Nature Communications, 2019, 10: 4841. DOI:10.1038/s41467-019-12798-y |
| [6] |
Hannula S E, Heinen R, Huberty M, Steinauer K, De Long J R, Jongen R, Bezemer T M. Persistence of plant-mediated microbial soil legacy effects in soil and inside roots. Nature Communications, 2021, 12(1): 5686. DOI:10.1038/s41467-021-25971-z |
| [7] |
Rivett D W, Bell T. Abundance determines the functional role of bacterial phylotypes in complex communities. Nature Microbiology, 2018, 3: 767-772. DOI:10.1038/s41564-018-0180-0 |
| [8] |
Bickel S, Or D. The chosen few—variations in common and rare soil bacteria across biomes. The ISME Journal, 2021, 15(11): 3315-3325. DOI:10.1038/s41396-021-00981-3 |
| [9] |
Liang Y T, Xiao X, Nuccio E E, Yuan M T, Zhang N, Xue K, Cohan F M, Zhou J Z, Sun B. Differentiation strategies of soil rare and abundant microbial taxa in response to changing climatic regimes. Environmental Microbiology, 2020, 22(4): 1327-1340. DOI:10.1111/1462-2920.14945 |
| [10] |
Cao H, Li S Y, He H, Sun Y Q, Wu Y C, Huang Q Y, Cai P, Gao C H. Stronger linkage of diversity-carbon decomposition for rare rather than abundant bacteria in woodland soils. Frontiers in Microbiology, 2023, 14: 1115300. DOI:10.3389/fmicb.2023.1115300 |
| [11] |
Gao C, Xu L, Montoya L, Madera M, Hollingsworth J, Chen L, Purdom E, Singan V, Vogel J, Hutmacher R B, Dahlberg J A, Coleman-Derr D, Lemaux P G, Taylor J W. Co-occurrence networks reveal more complexity than community composition in resistance and resilience of microbial communities. Nature Communications, 2022, 13: 3867. DOI:10.1038/s41467-022-31343-y |
| [12] |
Cao J J, Jiao Y M, Che R X, Holden N M, Zhang X F, Biswas A, Feng Q. The effects of grazer exclosure duration on soil microbial communities on the Qinghai-Tibetan Plateau. The Science of the Total Environment, 2022, 839: 156238. DOI:10.1016/j.scitotenv.2022.156238 |
| [13] |
Bardgett R D, Wardle D A. Herbivore‐mediated linkages between aboveground and belowground communities. Ecology, 2003, 84(9): 2258-68. DOI:10.1890/02-0274 |
| [14] |
Damgaard C. A critique of the space-for-time substitution practice in community ecology. Trends in Ecology & Evolution, 2019, 34(5): 416-421. |
| [15] |
Seaton F M, George P B L, Lebron I, Jones D L, Creer S, Robinson D A. Soil textural heterogeneity impacts bacterial but not fungal diversity. Soil Biology and Biochemistry, 2020, 144: 107766. DOI:10.1016/j.soilbio.2020.107766 |
| [16] |
李佳秀, 张青松, 吴勇, 杜子银. 围封对草地植被生长和土壤特性的影响研究进展. 中国草地学报, 2023, 45(5): 137-150. |
| [17] |
Li N, Chang R Y, Jiang H, Tariq A, Sardans J, Peñuelas J, Sun F, Zhou X M. Combined livestock grazing-exclusion and global warming decreases nitrogen mineralization by changing soil microbial community in a Tibetan alpine meadow. CATENA, 2022, 219: 106589. DOI:10.1016/j.catena.2022.106589 |
| [18] |
Wang J, Wang X T, Liu G B, Wang G L, Zhang C. Grazing-to-fencing conversion affects soil microbial composition, functional profiles by altering plant functional groups in a Tibetan alpine meadow. Applied Soil Ecology, 2021, 166: 104008. DOI:10.1016/j.apsoil.2021.104008 |
| [19] |
Wang J, Xiao Y M, Wang B, Fan B, Zhang D S, Zhou G Y. Different effects of long-term grazing exclusion and growth stages on soil fungi and bacteria in an alpine steppe on the Qinghai-Tibetan Plateau. Global Ecology and Conservation, 2023, 47: e02641. DOI:10.1016/j.gecco.2023.e02641 |
| [20] |
Jiao S, Chen W M, Wei G H. Biogeography and ecological diversity patterns of rare and abundant bacteria in oil-contaminated soils. Molecular Ecology, 2017, 26(19): 5305-5317. DOI:10.1111/mec.14218 |
| [21] |
Shu X Y, Ye Q X, Huang H, Xia L L, Tang H, Liu X Y, Wu J W, Li Y D, Zhang Y Y, Deng L J, Liu W J. Effects of grazing exclusion on soil microbial diversity and its functionality in grasslands: a meta-analysis. Frontiers in Plant Science, 2024, 15: 1366821. DOI:10.3389/fpls.2024.1366821 |
| [22] |
马广仁. 中国国际重要湿地及其生态特征. 北京: 中国林业出版社, 2016.
|
| [23] |
张金屯. 数量生态学 (3版). 北京: 科学出版社, 2018.
|
| [24] |
Wu X F, Yang J J, Ruan H, Wang S N, Yang Y R, Naeem I, Wang L, Liu L E, Wang D L. The diversity and co-occurrence network of soil bacterial and fungal communities and their implications for a new indicator of grassland degradation. Ecological Indicators, 2021, 129: 107989. DOI:10.1016/j.ecolind.2021.107989 |
| [25] |
Hernandez D J, David A S, Menges E S, Searcy C A, Afkhami M E. Environmental stress destabilizes microbial networks. The ISME Journal, 2021, 15(6): 1722-1734. DOI:10.1038/s41396-020-00882-x |
| [26] |
Wang F F, Li Z M, Fu B J, Lü Y H, Liu G P, Wang D B, Wu X. Short-term grazing exclusion alters soil bacterial co-occurrence patterns rather than community diversity or composition in temperate grasslands. Frontiers in Microbiology, 2022, 13: 824192. DOI:10.3389/fmicb.2022.824192 |
| [27] |
王娟, 张登山, 肖元明, 王博, 周国英. 物种多样性和功能性状驱动高寒草原地上生物量对长期禁牧的响应. 生态学报, 2023, 43(6): 2465-2475. |
| [28] |
Vidal A, Schucknecht A, Toechterle P, Linares D R A, Garcia-Franco N, von Heßberg A, Krämer A, Sierts A, Fischer A, Willibald G, Fuetterer S, Ewald J, Baumert V, Weiss M, Schulz S, Schloter M, Bogacki W, Wiesmeier M, Mueller C W, Dannenmann M. High resistance of soils to short-term re-grazing in a long-term abandoned alpine pasture. Agriculture, Ecosystems & Environment, 2020, 300: 107008. |
| [29] |
Yang F, Niu K C, Collins C G, Yan X B, Ji Y G, Ling N, Zhou X H, Du G Z, Guo H, Hu S J. Grazing practices affect the soil microbial community composition in a Tibetan alpine meadow. Land Degradation & Development, 2019, 30(1): 49-59. |
| [30] |
De Deyn G B, Cornelissen J H C, Bardgett R D. Plant functional traits and soil carbon sequestration in contrasting biomes. Ecology Letters, 2008, 11(5): 516-531. DOI:10.1111/j.1461-0248.2008.01164.x |
| [31] |
Moreau D, Bardgett R D, Finlay R D, Jones D L, Philippot L. A plant perspective on nitrogen cycling in the rhizosphere. Functional Ecology, 2019, 33(4): 540-552. DOI:10.1111/1365-2435.13303 |
| [32] |
Fan S, Qin J, Sun H, Jia Z, Chen Y. Alpine soil microbial community structure and diversity are largely influenced by moisture content in the Zoige wetland. International Journal of Environmental Science and Technology, 2022, 19(5): 4369-4378. DOI:10.1007/s13762-021-03287-1 |
| [33] |
Xun W B, Huang T, Zhao J, Ran W, Wang B R, Shen Q R, Zhang R F. Environmental conditions rather than microbial inoculum composition determine the bacterial composition, microbial biomass and enzymatic activity of reconstructed soil microbial communities. Soil Biology and Biochemistry, 2015, 90: 10-18. DOI:10.1016/j.soilbio.2015.07.018 |
| [34] |
Sokol N W, Slessarev E, Marschmann G L, Nicolas A, Blazewicz S J, Brodie E L, Firestone M K, Foley M M, Hestrin R, Hungate B A, Koch B J, Stone B W, Sullivan M B, Zablocki O, Soil Microbiome Consortium L L N L, Pett-Ridge J. Life and death in the soil microbiome: how ecological processes influence biogeochemistry. Nature Reviews Microbiology, 2022, 20(7): 415-430. DOI:10.1038/s41579-022-00695-z |
| [35] |
Maestre F T, Delgado-Baquerizo M, Jeffries T C, Eldridge D J, Ochoa V, Gozalo B, Quero J L, García-Gómez M, Gallardo A, Ulrich W, Bowker M A, Arredondo T, Barraza-Zepeda C, Bran D, Florentino A, Gaitán J, Gutiérrez J R, Huber-Sannwald E, Jankju M, Mau R L, Miriti M, Naseri K, Ospina A, Stavi I, Wang D L, Woods N N, Yuan X, Zaady E, Singh B K. Increasing aridity reduces soil microbial diversity and abundance in global drylands. 2015, 112(51): 15684-9.
|
| [36] |
Zhou H, Zhang D G, Jiang Z H, Sun P, Xiao H L, Wu Y X, Chen J G. Changes in the soil microbial communities of alpine steppe at Qinghai-Tibetan Plateau under different degradation levels. Science of the Total Environment, 2019, 651: 2281-2291. DOI:10.1016/j.scitotenv.2018.09.336 |
| [37] |
王光华, 刘俊杰, 于镇华, 王新珍, 金剑, 刘晓冰. 土壤酸杆菌门细菌生态学研究进展. 生物技术通报, 2016, 32(2): 14-20. |
| [38] |
Speirs L B M, Rice D T F, Petrovski S, Seviour R J. The phylogeny, biodiversity, and ecology of the chloroflexi in activated sludge. Frontiers in Microbiology, 2019, 10: 2015. DOI:10.3389/fmicb.2019.02015 |
| [39] |
Cobo-Díaz J F, Baroncelli R, Le Floch G, Picot A. Combined metabarcoding and co-occurrence network analysis to profile the bacterial, fungal and Fusarium communities and their interactions in maize stalks. Frontiers in Microbiology, 2019, 10: 261. DOI:10.3389/fmicb.2019.00261 |
| [40] |
Chen W Q, Wang J Y, Chen X, Meng Z X, Xu R, Duoji D Z, Zhang J H, He J, Wang Z G, Chen J, Liu K X, Hu T M, Zhang Y J. Soil microbial network complexity predicts ecosystem function along elevation gradients on the Tibetan Plateau. Soil Biology and Biochemistry, 2022, 172: 108766. DOI:10.1016/j.soilbio.2022.108766 |
 2024, Vol. 44
2024, Vol. 44