文章信息
- 贾元, 张琳, 吴冬秀, 宋创业, 袁伟影, 李凌浩
- JIA Yuan, ZHANG Lin, WU Dongxiu, SONG Chuangye, YUAN Weiying, LI Linghao
- 基于无人机多光谱实测数据的草地生物量反演模型比较
- Comparative analysis of grassland biomass inversion models based on unmanned aerial vehicle multispectral data
- 生态学报. 2024, 44(15): 6854-6864
- Acta Ecologica Sinica. 2024, 44(15): 6854-6864
- http://dx.doi.org/10.20103/j.stxb.202401060048
-
文章历史
- 收稿日期: 2024-01-06
- 网络出版日期: 2024-05-24
2. 国家植物园, 北京 100093;
3. 中国科学院大学, 北京 100049
2. China National Botanical Garden, Beijing 100093, China;
3. University of Chinese Academy of Sciences, Beijing 100094, China
草地是陆地生态系统的重要组成部分, 是我国畜牧业的重要生产基地, 同时在水土保持、固碳增汇、防风固沙等方面起着重要作用[1]。草地生物量是群落结构和功能的综合体现, 生物量的变化直接反映草地生态系统功能和服务状态, 同时也是陆地碳储量估算的依据[2]。因此, 对于草地生物量的快速估算和动态监测能够为草地的维护和管理提供理论依据[3]。
草地生物量获取最简单的方法是样方直接刈割法, 可以很精确的获得样方内的地上生物量数据, 但是费时费力、破坏草场, 并且受限于样方数量和空间代表性, 不适合进行大范围的生物量估算[4]。随着遥感技术的发展, 通过运用与植被相关性强的光谱与植被指数的反演, 可以实现区域甚至全球尺度上草地生物量估算[5], 如朴世龙等采用基于AVHRR构建的归一化植被指数(Normalized Difference Vegetation Index, NDVI)估算全国尺度草地植被地上生物量研究, 发现两者具有很好的相关关系, NDVI解释了生物量71%的方差, 基于模型可以快速估测我国草地植被总地上生物量, 约为146.16TgC[6];Li等利用Sentinel-2多光谱仪的光谱和纹理特征构建了升金湖湿地草地地上生物量的极端梯度提升(XGBoost)模型, 可以快速估算区域内草地生物量, 为区域生态系统的可持续管理和碳核算提供了支持[7]。
近年来, 中尺度无人机遥感技术发展迅速, 相比于传统卫星遥感, 无人机遥感具有更高的时空分辨率, 不易受到天气等因素的影响, 可以提供更为精确的模型。应用无人机近地面遥感技术, 构建更为精确的草地生物量反演模型的研究也受到越来越多的关注。模型可以分为参数模型和非参数模型两大类。第一类参数模型具有相对高的解释度, 但对变量与生物量间对应关系要求高且区域差异性较大:例如基于无人机获取的影像中提取的单个变量, 构建草地地上生物量的单变量模型, 如张正健等使用由无人机可见光波段构建了归一化绿红差异指数(Normalized Green Red Diffence Index, NGRDI)与若尔盖高寒草甸地上生物量的指数模型[8];孙世泽等人使用由无人机多光谱影像构建的比值植被指数(Ratio Vegetation Index, RVI)构建了天山山脉阴阳坡两侧的线性模型[9];Zhang等人使用无人机可见光构建了植被冠层高度模型, 使用实测地上生物量与冠层高度模型(Canopy Height Model, CHM)构建了基于CHM的对数模型[10]。还有学者组合了多种变量, 构建更为复杂的多变量模型, 如Lussem等人通过无人机可见光影像调查了德国温带草原, 使用植被指数与冠层高度组合的多项式模型来估计生物量, 为草地生物量监测提供了一个有前景的方法[11]。第二类是非参数模型, 具有较高的估测精度, 但是模型较为复杂, 几乎不具备解释性, 不同研究采用的模型也存在差异, 如基于无人机多光谱影像, Li等人采用随机森林模型反演了德克萨斯州的草原地上生物量[12];Alvarez等人调查了哥伦比亚人工草场, 构建了K近邻-草地地上生物量模型, 评估了轮牧系统和草地地上冠层特征来预测牛饲料质量和数量的预测模型方法, 以支持草地管理[13];Sharma等人使用支持向量机模型构建了南达科他州的三个人工草场的草地地上生物量预测模型[14]。
综上所述, 近地面无人机遥感技术应用到草地生物量快速准确估算的研究越来越多, 但是采用的模型种类、变量、算法差异较大, 为了对比不同模型之间的差异和对温带草原的适用性, 本研究将无人机多光谱遥感数据与在内蒙古温带草原获取的地面实测数据结合, 使用计算出的植被指数、光谱反射率作为自变量, 总结前人使用过的模型类型, 选取8类常用的参数和非参数模型, 对比分析不同模型的精度, 确定适用于我国温带草原的无人机多光谱反演生物量的最佳模型, 以期为温带草原草地资源可持续利用和草畜科学管理提供理论依据。
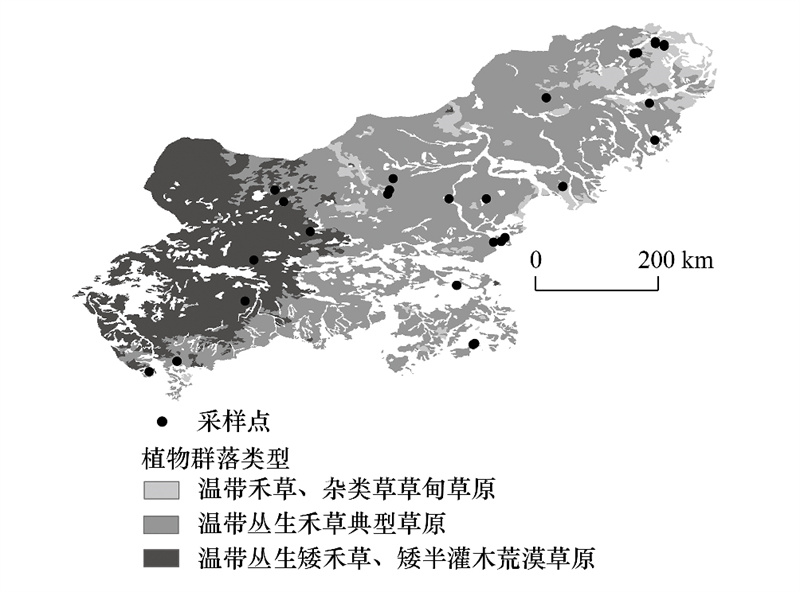
1 研究区概况本研究野外调查工作在内蒙古自治区锡林郭勒盟-乌兰察布市进行(111°29′ 15.115″—119°4′ 21.680″E, 41°37′ 49.009″—46°29′ 51.812″ N)。依据1 ∶ 100000植被图, 从东到西梯度样带上调查了荒漠草原、典型草原、草甸草原等多种群落类型[15](见图 1), 涵盖了多种植物群落类型, 包括羊草(Leymus chinensis)群落、大针茅(Stipa grandis)群落、糙隐子草(Cleistogenes squarrosa)群落、西北针茅(Stipa sareptana var. krylovii)群落、石生针茅(Stipa klemenzii)群落、碱韭(Allium polyrrhizum)群落、冷蒿(Artemisia frigida)群落、粗根鸢尾(Iris tigridia)群落等。各研究样点均在各个群系的核心区域, 具有较好的代表性。野外调查时间为2023年7—8月, 调查期间处于牧草生长旺季。
|
| 图 1 本研究采样点分布图 Fig. 1 Distribution of sampling sites of this study |
野外调查采用样带法, 从东到西, 共计56个样地, 其中分布在荒漠草原有9个样地, 在典型草原有37个样地, 在草甸草原有10个样地。地面调查采用样方法, 样方布设在选取的植被比较均匀有代表性的地段, 每块样地布设4个0.5 m×0.5 m的样方[16—18]。生物量获取采用地面刈割干重法, 即将样方内所有植物的地上部分全部收集, 然后在65 ℃烘箱里烘48 h后得到该样方的地上生物量干重, 共获得了224个地面实测调查数据以及相对应的无人机遥感影像。
无人机影像与地面样方相对应, 使用大疆无人机(型号:Matrice300RTK)搭载多光谱相机(型号:Yusense MS600pro)获取6个光谱通道影像, 光谱通道波长为450 nm、555 nm、660 nm、720 nm、750 nm、840 nm, 各波段反射率分别命名为BR、GR、RR、RE1、RE2、NIR。在地面样方布设完毕后, 地面生物量刈割之前获取多光谱影像。无人机飞行参数设置为:飞行高度为距离地面30 m, 航向重叠率和旁向重叠率均为80%, 航线速度为2.5 m/s。多光谱影像的地面分辨率为2.16 cm/pixel。原始影像处理软件为Yusense map, 对影像进行波段配准, 辐射定标, 影像拼接, 获得正射影像。
2.2 变量提取变量均通过无人机多光谱影像及其衍生出的影像中得到。使用ENVI (64-bit)中的ROI(Region of interest)工具提取出每个样方中相应变量值, 使用均值作为该样方的解释变量, 共得到了2类解释变量, 即波段变量(B)和植被指数变量(V), 共12个变量用于建模, 具体变量及计算公式如表 1。
| 变量类型Type of variable | 解释变量Explanatory variables |
| 波段变量Band | BR, GR, RR, RE1, RE2, NIR |
| 植被指数Vegetation index | NDVI, kNDVI, RVI, EVI, NDRE1, NDRE2 |
| BR: 蓝波段反射率Blue band reflectance;GR: 绿波段反射率Green band reflectance;RR: 红波段反射率Red band reflectance;RE1: 720nm处红边波段反射率Red edge band reflectance at 720nm;RE2: 750nm处红边波段反射率Red edge band reflectance at 720nm;NIR: 近红外波段反射率Near infrared band reflectance;NDVI: 归一化植被指数Normalized difference vegetation index;kNDVI: 核化归一化植被指数kernel Normalized difference vegetation index;RVI: 比值植被指数Ratio vegetation index;EVI: 增强植被指数Enhanced vegetation index;NDRE1: 归一化红边植被指数1 Normalized difference red edge vegetation index1;NDRE2: 归一化红边植被指数2 Normalized difference red edge vegetation index2 | |
选择比较了8类常用的参数和非参数模型, 分别是四类参数模型:线性模型(Linear model), 指数模型(Exponential model), 对数模型(Logarithmic model), 广义线性模型(GLM);四类非参数模型:K近邻(KNN)模型, 支持向量机(SVM)模型[19], 随机森林(RF)模型[20], 极端梯度提升模型(XGBoost)[21]。其中在构建非参数模型时, 均采用70%的数据用于训练模型, 30%用作评估模型性能的测试集;在训练完模型后, 对所有多变量模型采用十折交叉验证(10-fold Cross Validation)的方法对模型稳定性再次进行检验。
2.3.2 变量筛选针对不同模型采用多种变量筛选方法得到各类模型的最优变量:对于单变量模型和GLM模型, 采用Pearson相关性分析和逐步回归确定最优变量;对于KNN模型和SVM模型, 采用递归特征消除法[22]进行变量筛选;对于RF模型和XGBoost模型, 采用后向特征消除法[23]进行变量筛选。
2.3.3 超参优化对于非参数模型, 超参优化的方法是在设定好的搜索空间中通过评估每个组合的模型效果, 以各个组合获得的R2作为评估标准来搜索最佳超参数, 搜索后各个模型的最优超参数如表 2所示。
| 模型Models | 超参数Hyperparameters |
| KNN模型KNN model | 预测选用邻近样本=1.49, 距离度量=3, 核函数=反距离加权核函数k=1.49, distance=3, kernel=Inverse Distance Weighting |
| SVM模型SVM model | 惩罚参数=9.02, 伽马系数=0.993, 核函数=径向基函数cost=9.02, gamma=0.993, kernel=radial |
| RF模型RF model | 树的数量=300, 特征选择数量=3, 节点分裂所需的最小观测数=6, 最大深度=5 num.tree=300, mtry=3, min.node.size=6, max.depth=5 |
| XGBoost模型XGBoost model | 学习率=0.5, 叶结点最小权重和=1, 样本采样比例=1, 特征的采样比例=0.85, 列采样比例=0.75, 迭代次数=13 eta=0.5, min_child_weight=1, subsample=1, colsample_bytree=0.85, colsample_bylevel=0.75, nrounds=13 |
| KNN, K近邻K Nearest Neighbor;SVM, 支持向量机Support Vector Machine;RF, 随机森林Random Forests;XGBoost, 极限梯度提升Extreme Gradient Boosting | |
采用模型决定系数R2作为模型精度的评估标准:
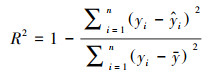
|
其中, yi为实测生物量, 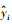
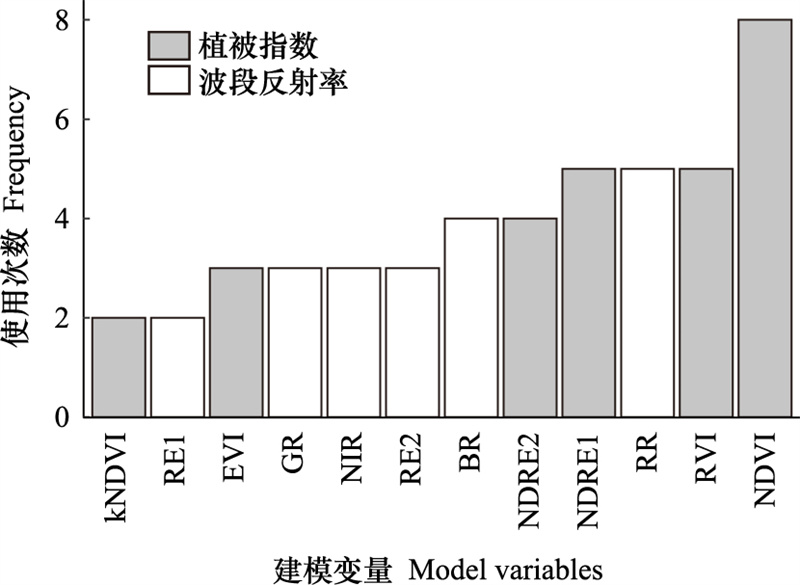
对于非参数模型, 计算了Rtrain2作为模型训练集精度以及Rtest2作为模型测试集精度, 并在构建模型后对模型进行十折交叉验证来评估模型的过拟合程度, 即将可用的数据迭代分割成一个不重叠的序列和测试集, 重新改装模型, 并对每个子集的训练和测试数据进行测试, 可以获得泛化性能的估计, 在十折交叉验证后获得Rcv2;此外还计算了Rchange2作为模型稳定性评估标准:
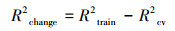
|
本研究构建的所有模型均在R studio中实现, 所有模型均满足10EPV(10 Events per variable)原则。
3 结果 3.1 生物量与各单一变量的相关性对于从影像中提取出的波段反射率与植被指数类变量, 分别与地上生物量进行单因素相关性分析, 结果发现所有变量与生物量之间相关性均达到显著水平(P < 0.05), 但波段反射率变量与生物量的相关性较低, 而植被指数中除了NDRE2和kNDVI外, 其余4类植被指数均与生物量相关性较高(见表 3)。
| BR | GR | RR | RE1 | RE2 | NIR | NDVI | NDRE1 | NDRE2 | RVI | EVI | kNDVI | |
| 生物量Biomass | -0.43 | -0.31 | -0.43 | -0.15 | 0.15 | 0.25 | 0.69 | 0.67 | 0.22 | 0.68 | 0.60 | 0.41 |
| P | *** | *** | *** | * | * | *** | *** | *** | *** | *** | *** | *** |
| BR: 蓝波段反射率Blue band reflectance;GR: 绿波段反射率Green band reflectance;RR: 红波段反射率Red band reflectance;RE1:720nm处红边波段反射率Red edge band reflectance at 720nm;RE2:750nm处红边波段反射率Red edge band reflectance at 720nm;NIR: 近红外波段反射率Near infrared band reflectance;NDVI: 归一化植被指数Normalized difference vegetation index;kNDVI: 核化归一化植被指数kernel Normalized difference vegetation index;RVI: 比值植被指数Ratio vegetation index;EVI: 增强植被指数Enhanced vegetation index;NDRE1:归一化红边植被指数1 NDRE1 Normalized difference red edge index1;NDRE2:归一化红边植被指数2 Normalized difference red edge index2;* * *, P < 0.001;* *, P < 0.01;*, P < 0.05 | ||||||||||||
由以上表 3中结果显示NDVI与生物量的相关性最高, 因此使用单变量NDVI分别与生物量构建了线性模型、对数模型和指数模型。对12个建模变量进行逐步回归的结果表明, BR, RR, RE2, NIR, NDVI, NDRE2, RVI, EVI等8个变量组合后可以达到最高的R2(见图 2), 因此用这8个变量构建GLM模型。
|
| 图 2 基于逐步回归的GLM最优变量筛选 Fig. 2 GLM optimal variable selecting based on stepwise regression BR: 蓝波段反射率Blue band reflectance;GR: 绿波段反射率Green band reflectance;RR: 红波段反射率Red band reflectance;RE1:720nm处红边波段反射率Red edge band reflectance at 720nm;RE2:750nm处红边波段反射率Red edge band reflectance at 720nm;NIR: 近红外波段反射率Near infrared band reflectance;NDVI: 归一化植被指数Normalized difference vegetation index;kNDVI: 核化归一化植被指数kernel Normalized difference vegetation index;RVI: 比值植被指数Ratio vegetation index;EVI: 增强植被指数Enhanced vegetation index;NDRE1:归一化红边植被指数1 NDRE1 Normalized difference red edge index1;NDRE2:归一化红边植被指数2 Normalized difference red edge index2 |
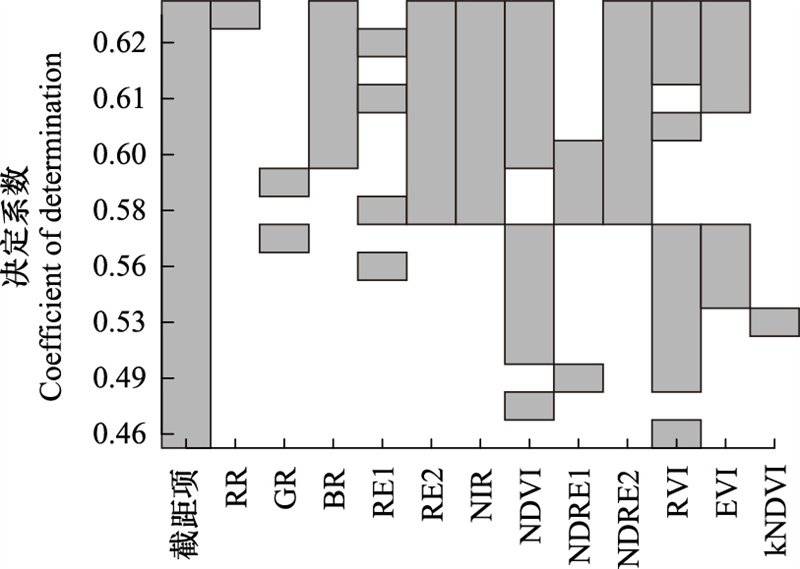
由图 3可以看出, 对于KNN模型和SVM模型, 随着变量的逐一剔除, 模型R2波动较大, R2最大值出现在12个变量均存在时, 因此KNN和SVM采用12个变量。对于RF模型和XGBoost模型, 分别在变量数为4和8时达到一个稳定状态下的最高值, 因此选取重要性前4和前8的变量用于建模, RF模型采用NDRE1, NDVI, RR和RVI 4个变量, XGBoost模型采用NDRE2, EVI, GR, BR, RVI, RR, NDVI和NDRE1 8个变量。
|
| 图 3 非参数模型精度与建模变量数折线图 Fig. 3 Non-parametric model accuracy and the number of modeling variables |
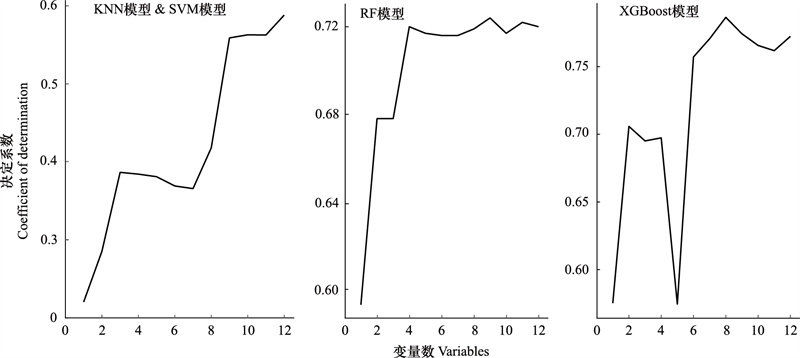
图 4结果表明, 在所有模型使用的变量中, NDVI是使用次数最多的变量, 在所有模型中都采用, 其次是RVI、NDRE1和NDRE1, 在5个模型中采用, 而kNDVI和RE1仅在KNN模型和SVM模型中使用, 使用次数较少。
|
| 图 4 各变量在研究中的使用次数 Fig. 4 Frequency of different variables usage counts |
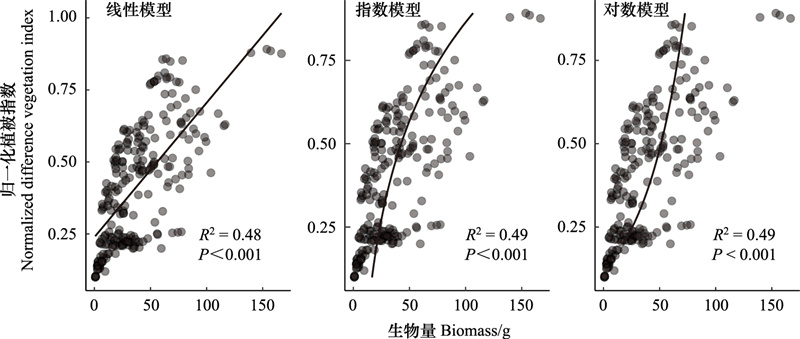
图 5比较了3种单变量参数模型的拟合效果, 结果表明使用NDVI构建的三种生物量估测模型均无法很好的收敛, 模型发生了欠拟合, R2均低于0.5。
|
| 图 5 基于NDVI的三种单变量模型 Fig. 5 Three univariate models based on NDVI |
基于逐步回归的结果构建的GLM模型获得的R2为0.63(见表 4), 相较单变量模型R2有所提升, 但十折交叉验证后Rcv2为0.50, R2下降了0.13, 说明GLM模型存在一定程度的过拟合。
| 模型
Model |
训练集R2
Rtrain2 |
测试集R2
Rtest2 |
交叉验证R2
Rcv2 |
R2变化
Rchange2 |
变量数
Number of variables |
模型变量
Variables of model |
| 线性模型
Linear model |
0.48 | 1 | NDVI | |||
| 指数模型
Exponential model |
0.49 | 1 | NDVI | |||
| 对数模型
Logarithmic model |
0.49 | 1 | NDVI | |||
| GLM模型
Generalized linear model |
0.63 | 0.50 | 0.13 | 8 | BR, RR, RE2, NIR, NDVI, NDRE2, RVI, EVI | |
| KNN模型
K nearest neighbor model |
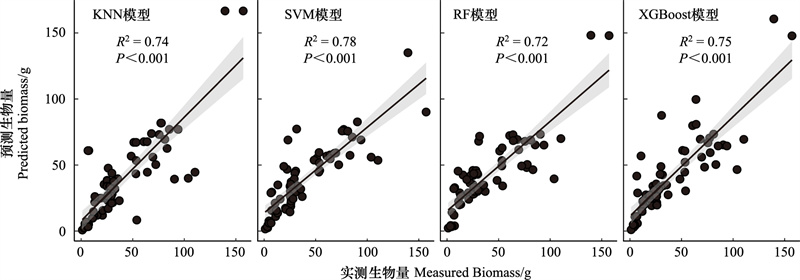
0.79 | 0.74 | 0.66 | 0.13 | 12 | BR, GR, RR, RE1, RE2, NIR, NDVI, NDRE1, NDRE2, kNDVI, RVI, EVI |
| SVM模型
Support vector machine model |
0.77 | 0.78 | 0.68 | 0.09 | 12 | BR, GR, RR, RE1, RE2, NIR, NDVI, NDRE1, NDRE2, kNDVI, RVI, EVI |
| RF模型
Random forests model |
0.72 | 0.72 | 0.69 | 0.03 | 4 | NDRE1, NDVI, RR, RVI |
| XGBoost模型
XGBoost model |
0.77 | 0.75 | 0.70 | 0.07 | 8 | NDRE2, EVI, GR, BR, RVI, RR, NDVI, NDRE1 |
基于表 2的最优超参数, 构建了4种非参数模型的最优模型, 结果如图 6和表 4所示, KNN模型获得了0.79的Rtrain2以及0.74的Rtest2, 十折交叉验证后取得了0.66的Rcv2, 所用变量为全变量;SVM模型获得了0.77的Rtrain2以及0.78的Rtest2, 十折交叉验证后取得了0.68的Rcv2, 所用变量为全变量;而RF模型使用了NDRE1, NDVI, RR, RVI等四类变量, 获得了0.72的Rtrain2以及0.72的Rtest2, 十折交叉验证后取得了0.69的Rcv2, 构建的模型相对更为鲁棒;XGBoost模型使用EVI, GR, BR, RVI, RR, NDVI, NDRE1等7个变量获得了0.77的Rtrain2以及0.75的Rtest2, 十折交叉验证后取得了0.70的Rcv2。
|
| 图 6 四种非参数模型测试集预测精度 Fig. 6 Testing accuracy of four non-parametric models on test set 阴影部分为95%置信区间 |
综合比较所有参数和非参数模型精度结果如表 4所示, 使用KNN模型获得了最高的Rtrain2, 其次是SVM模型和XGBoost模型, 这两种模型取得的Rtrain2低于KNN模型但差距不大, RF模型获得的Rtrain2则相对低于其余机器学习算法。而所有的参数模型精度则相对较低, 线性模型获得了最低的R2, 其次是指数和对数模型, 而多变量的GLM模型则取得了相对更高的R2。四种非参数模型对于30%的测试集的验证精度表现出了略微不同的趋势, 即SVM模型表现出了最高Rtest2, 其次是XGBoost模型和KNN模型, Rtest2最低的仍为RF模型。
然而仅看R2无法确定模型的稳定性, 本研究通过计算了各个模型R2的变化作为模型稳定性的评价标准, 结果表明几乎所有多变量模型均存在一定程度的过拟合, 最稳定的模型为RF模型, Rchange2仅0.03, 其次是R2下降了0.07的XGBoost模型, SVM模型的Rchange2为0.09, GLM模型和KNN模型的Rchange2为0.13。从建模变量角度而言, 三种单变量模型具有最少的变量, 其次是RF模型, 之后是XGBoost模型和GLM模型, KNN模型和SVM模型具有最多的变量。
4 讨论 4.1 变量筛选比较与优化对于草地地上生物量模型来说, 如何选取最相关变量是一个非常关键的问题。Burnham和Anderson认为, 对于一个真实的模型来说, 例如草地地上生物量模型, 存在无数个有关的变量[24]。目前在锡林郭勒盟使用遥感方法估算草地地上生物量的研究中, 出现了大量不同类型的建模变量, 可以发现使用不同的变量可以获得相似精度的生物量预测模型, 如邢晓语等人使用了NDVI, RVI, SAVI等3个变量[25];而Lyu等人则使用了降水, NDVI, EVI, 土壤性质等变量[26];而Xie等则使用了NDVI, 坡向, Landsat band1, band3, band4, band5, band7等变量[27]。而与邢晓语、Lyu、Xie等人的研究一样, 本研究中所有模型都用到了NDVI, 说明NDVI在草地生物量反演时具有重要作用。
本研究还发现除了NDVI外, RR在模型反演中也非常重要, 且是建模变量中唯一一个使用次数较多的波段反射率变量, 在所有非参数模型中均有出现。植物的反射率受到叶绿素吸收的影响, 在可见光波段存在明显的“谷-峰-谷”现象, 使用植被光谱反射率进行生物量反演的主要思路是通过植被的光谱特性来区分植被和地物。Todd等人发现红光光谱反射率对绿色, 衰老或干燥生物量很敏感[28];同样王红岩也发现HJ-1B与Landsat TM的红光波段与草地生理参量有很高的相关性[29]。虽然根据表 3的相关性发现RR与生物量相关性不高, 但RR对于植被的识别具有重要作用, 所以同样在本研究获得的模型中获得了较多使用次数, 未来可以从该思路出发拓展更多基于红波段反射率的植被指数。
4.2 参数模型比较与评价根据本研究获得的结果来看, 单变量模型与GLM模型等参数模型在精度上似乎并不占优势。使用单变量构建的地上生物量模型十分的简洁, 模型的可解释性很强, 可以很直观的体现出不同变量在模型中的作用, 但单一变量的模型对于函数形式存在较大的拘束, 容易出现欠拟合的现象, 如本研究中的三类单变量模型, 在建模时都需要假定NDVI与草地地上生物量之间符合某种确定的函数形式, 一旦选择的函数远远偏离目标参数的实际情况时, 那么所获得的模型将会缺乏实用价值的。很多使用参数方法建模的研究, 会比较多种参数模型, 如胡远宁等人将实测生物量与植被指数分别带入线性模型、指数模型、多项式模型、对数模型、乘幂模型中, 最终发现使用指数模型的R2最高[30];Hobbs也是在指数模型与线性模型之间进行对比, 最终使用了指数模型[31]。而GLM获得了较单变量模型更高的精度, 是由于使用了更多的变量, 经过不同变量的组合获得了最高的R2。Svinurai等人也比较了一元模型和多元模型, 发现多元函数获得了更高的R2[32]。
但是一般来说, 草地地上生物量很难符合某一个特定的函数形式, 因为草地地上生物量的形成是一个复杂的过程, 其中涉及到的生物因子和非生物因子多种多样, 并非单一或者几个变量可以完全解释;并且参数方法构建的模型灵活性较低, 这主要是由于参数已经固定, 模型很难运用至其它地区, 甚至Braun等人发现在热带稀树草原上, 同一个建模变量无法同时处理高生物量与低生物量区域[33], 也体现出单变量很难构建出一个普适性强的模型。
4.3 非参数模型比较与评价相较于参数模型, 非参数模型对目标函数形式不做过多的假设, 因此可以通过对训练数据进行拟合而学习出某种独一无二形式的函数, 它拥有将目标函数与更多种函数相匹配的可能, 仅需要寻找一个与目标函数尽可能较为接近的函数形式[34];并且非参数模型内部所有参数都是可以调整的, 随着模型训练集的增大, 模型内部参数也在不断发生调整, 使获得的结果更精确。目前已有许多文献发现非参数模型的精度高于参数模型[13, 35—36]。
虽然非参数模型可以获得更高的预测精度, 但却增加了模型的复杂程度, 降低了模型的可解释性, 难以提供关于模型内部决策的详细信息;并且使用非参数方法一般会涉及到大量的特征, 虽然可以使模型更拟合样本数据, 但当特征增多, 模型出现过拟合将是一个无法避免的问题[37]。对于过拟合问题, 一般可以通过使用更多的样本数据来缓解过拟合, 但在实地获取数据时, 很难预估获取多少样本量才可以缓解过拟合现象。与此同时, 当模型发生了过拟合是否应该全盘否定同样是一个值得思考的问题。Meng等人在三种机器学习模型间进行比较并选择了稳定性最弱的RF模型作为最优模型[36]。虽然该研究中RF模型出现了过拟合, 但RF模型的R2也高于另外两类模型, 并且作者在研究区各个区域获取了自2011—2016年内共2153个地面样方, 模型将一些噪声也学习得很好, 这种程度的过拟合也是提高整体估测精度的重要部分。
本研究模型对比结果也发现RF模型是最优模型, RF模型的过拟合程度最低, Rchange2仅为0.03, 是本研究获得的最鲁棒的模型。同时在以往的研究中, RF在其它机器学习模型比较的研究中往往也具有优势, 如Fan等人采用Cubist模型、GBRT模型、RF模型和XGBoost模型等四种机器学习算法构建AGB估计模型, 结果表明使用RF构建的模型性能最佳, 最有利于估计青藏高原草地的地上生物量[38];Ge等人使用随机森林、支持向量机、人工神经网络和极限学习器等四种机器学习算法分别构建我国北方草地地上生物量反演模型, 结果表明使用随机森林构建的模型是最优的草地地上生物量预测模型[39]。随机森林模型凭借其不容易过拟合, 对噪声不敏感等特点在不同地区均有良好表现, 而在本研究中, RF模型虽然并未取得最高的R2, 但也高于0.7, 并且Rchange2最小, 模型最为鲁棒, 建模所需的变量数最少, 因此综上可以认为本研究的最优模型为RF模型。
4.4 模型优化在非参数模型中, 选取合适的超参数十分重要, 本研究各个模型经过超参数调节后最终确定的超参数如表 2所示。而目前的研究中, 只有部分研究详细介绍了关于超参数的调节[17, 40]。超参数的组合对于模型R2具有重要影响, 不同的超参数组合构建的模型R2差异较大, 如RF模型, 在其它超参数不变的情况下, 随着max.depth的增大, 模型中树越深, 对数据的拟合程度越高, 可以使得模型获得更高的R2, 但是同样也会导致对新数据的泛化能力下降, 模型的过拟合程度越高, 而对于一组数据, 如何确定当前算法的最优超参数往往需要巨大的计算量, 目前研究中得到的最优模型也只是在一部分相对较好的超参数组合中计算得出的一个最佳模型, 而对于不同数据集如何确定最优超参数在某个区间也需要大量的调试与经验。
对于参数模型来说, 单变量模型除了找到一个与生物量相关性非常高的变量外, 或许很难有别的提升方法;而对于多变量的模型来说, 有学者提出了一些可以优化模型的方法。如Tran提出了一种马尔可夫链蒙特卡罗算法, 该算法可以提取出最接近于贝叶斯模型平均的模型[41], 然而, 该算法的实现目前还是一个难题, 且贝叶斯模型平均会综合考虑不同形式和参数的模型, 导致整体的复杂性会增加, 使得最终结果难以解释和理解。对于广义线性模型, Qian等人提出了一种通过预测最小拟偏差准则来衡量模型的预测能力, 当拟偏差准则最小时, 所选模型在期望条件下收敛于最优模型[42], 但是具体应用到实际模型中还存在难度。
除了模型类型外, 也可以考虑增加垂直信息以消除影像中土壤背景的影响, 使用体积法构建生物量模型, 提高模型精度。但是在草地使用的高度信息还存在不确定性, 受草原的下垫面性质影响, 无人机激光雷达容易丢失草原冠层顶部信息[43], 并且使用无人机激光雷达构建出的冠层高度模型容易低估冠层高度[44]。草地相较于森林, 更易受到风等环境因素影响, 导致点云无法准确反映草地的冠层信息, 如何在覆盖度较高的草原提取出准确的点云是未来的一个重要研究方向。
5 结论综上所述, 应用无人机近地面遥感技术是一种有效的估算区域尺度草地生物量的方法。本文比较了8种较为常用的模型, 这些模型间的差异较大, 总体上非参数模型精度较参数模型高, 非参数模型中随机森林具有最高的稳定性、相对高的精度且需要最少的变量, 是较为适用于无人机近地面遥感技术估算草地生物量的模型。除此之外, NDVI和红波段反射率在草地生物量反演中起到了重要作用。在构建生物量预测模型时, 变量筛选、模型选择与优化, 以及在建模之前选择出合适的超参数都是非常重要的, 尤其对于非参数模型应该如何避免过拟合, 以及非参数模型的优化方法研究仍是难点, 未来的研究可以从该角度进行深入。
| [1] |
彭云峰, 常锦峰, 赵霞, 石岳, 白宇轩, 李秦鲁, 姚世庭, 马文红, 方精云, 杨元合. 中国草地生态系统固碳能力及其提升途径. 中国科学基金, 2023, 37(04): 587-602. |
| [2] |
Eisfelder C, Kuenzer C, Dech S. Derivation of biomass information for semi-arid areas using remote-sensing data. International Journal of Remote Sensing, 2012, 33(9): 2937-2984. DOI:10.1080/01431161.2011.620034 |
| [3] |
郑晓翾, 赵家明, 张玉刚, 吴雅琼, 靳甜甜, 刘国华. 呼伦贝尔草原生物量变化及其与环境因子的关系. 生态学杂志, 2007, 26(4): 533-538. |
| [4] |
Lu D S. The potential and challenge of remote sensing-based biomass estimation. International Journal of Remote Sensing, 2006, 27(7): 1297-1328. DOI:10.1080/01431160500486732 |
| [5] |
Huete A, Didan K, Miura T, Rodriguez E P, Gao X, Ferreira L G. Overview of the radiometric and biophysical performance of the MODIS vegetation indices. Remote Sensing of Environment, 2002, 83(1-2): 195-213. DOI:10.1016/S0034-4257(02)00096-2 |
| [6] |
朴世龙, 方精云, 贺金生, 肖玉. 中国草地植被生物量及其空间分布格局. 植物生态学报, 2004, 04: 491-498. |
| [7] |
Li C H, Zhou L Z, Xu W B. Estimating aboveground biomass using sentinel-2 MSI data and ensemble algorithms for grassland in the Shengjin Lake wetland, China. Remote Sensing, 2021, 13(8): 1595. DOI:10.3390/rs13081595 |
| [8] |
张正健, 李爱农, 边金虎, 赵伟, 南希, 靳华安, 谭剑波, 雷光斌, 夏浩铭, 杨勇帅, 孙明江. 基于无人机影像可见光植被指数的若尔盖草地地上生物量估算研究. 遥感技术与应用, 2016, 31(1): 51-62. |
| [9] |
孙世泽, 汪传建, 尹小君, 王伟强, 刘伟, 张雅, 赵庆展. 无人机多光谱影像的天然草地生物量估算. 遥感学报, 2018, 22(05): 848-856. |
| [10] |
Zhang H F, Sun Y, Chang L, Qin Y, Chen J J, Qin Y, Du J X, Yi S H, Wang Y L. Estimation of grassland canopy height and aboveground biomass at the quadrat scale using unmanned aerial vehicle. Remote Sensing, 2018, 10(6): 851. DOI:10.3390/rs10060851 |
| [11] |
Lussem U, Bolten A, Menne J, Gnyp M L, Schellberg J, Bareth G. Estimating biomass in temperate grassland with high resolution canopy surface models from UAV-based RGB images and vegetation indices. Journal of Applied Remote Sensing, 2019, 13(3): 034525. |
| [12] |
Li Z, Angerer J P, Jaime X, Yang C H, Wu X B. Estimating rangeland fine fuel biomass in western texas using high-resolution aerial imagery and machine learning. Remote Sensing, 2022, 14(17): 4360. DOI:10.3390/rs14174360 |
| [13] |
Alvarez-Mendoza C I, Guzman D, Casas J, Bastidas M, Polanco J, Valencia-Ortiz M, Montenegro F, Arango J, Ishitani M, Selvaraj M G. Predictive modeling of above-ground biomass in Brachiaria pastures from satellite and UAV imagery using machine learning approaches. Remote Sensing, 2022, 14(22): 5870. DOI:10.3390/rs14225870 |
| [14] |
Sharma P, Leigh L, Chang J, Maimaitijiang M, Caffé M. Above-ground biomass estimation in oats using UAV remote sensing and machine learning. Sensors, 2022, 22(2): 601. DOI:10.3390/s22020601 |
| [15] |
中国科学院中国植被图编辑委员会. 中华人民共和国植被图. 北京: 地质出版社, 2007.
|
| [16] |
苗春丽, 伏帅, 刘洁, 高金龙, 高宏元, 包旭莹, 冯琦胜, 梁天刚, 贺金生, 钱大文. 基于UAV成像高光谱图像的高寒草甸地上生物量——以海北试验区为例. 草业科学, 2022, 39(10): 1992-2004. |
| [17] |
Zhang H F, Tang Z G, Wang B Y, Meng B, Qin Y, Sun Y, Lv Y, Zhang J G, Yi S. A non-destructive method for rapid acquisition of grassland aboveground biomass for satellite ground verification using UAV RGB images. Global Ecology and Conservation, 2022, 33: e01999. DOI:10.1016/j.gecco.2022.e01999 |
| [18] |
Acorsi M G, Miranda F D A, Martello M, Smaniotto D A, Sartor L R. Estimating biomass of black oat using UAV-based RGB imaging. Agronomy, 2019, 9(7): 344. DOI:10.3390/agronomy9070344 |
| [19] |
Burges C J C. A tutorial on support vector machines for pattern recognition. Data Mining and Knowledge Discovery, 1998, 2(2): 121-167. DOI:10.1023/A:1009715923555 |
| [20] |
Breiman L. Random forests. Machine Learning, 2001, 45: 5-32. DOI:10.1023/A:1010933404324 |
| [21] |
Chen T Q, Guestrin C. XGBoost: a scalable tree boosting system. Proceedings of the 22nd ACM SIGKDD International Conference on Knowledge Discovery and Data Mining. San Francisco, California, USA, 2016: 785-794.
|
| [22] |
Guyon I, Weston J, Barnhill S, Vapnik V. Gene Selection for Cancer Classification using Support Vector Machines. Machine Learning, 2002, 46(1): 389-422. |
| [23] |
Vergara J R, Estévez P A. A review of feature selection methods based on mutual information. Neural Computing and Applications, 2014, 24(1): 175-186. DOI:10.1007/s00521-013-1368-0 |
| [24] |
Burnham K, Anderson D R. Model selection and multimodel inference: a practical information-theoretic approach. New York: Springer-Verlag, 2003, 16-22. |
| [25] |
邢晓语, 杨秀春, 徐斌, 金云翔, 郭剑, 陈昂, 杨东, 王平, 朱立博. 基于随机森林算法的草原地上生物量遥感估算方法研究. 地球信息科学学报, 2021, 23(07): 1312-1324. |
| [26] |
Lyu X, Li X B, Gong J R, Li S K, Dou H S, Dang D L, Xuan X J, Wang H. Remote-sensing inversion method for aboveground biomass of typical steppe in Inner Mongolia, China. Ecological Indicators, 2021, 120: 106883. DOI:10.1016/j.ecolind.2020.106883 |
| [27] |
Xie Y C, Sha Z Y, Yu M, Bai Y F, Zhang L. A comparison of two models with Landsat data for estimating above ground grassland biomass in Inner Mongolia, China. Ecological Modelling, 2009, 220(15): 1810-1818. DOI:10.1016/j.ecolmodel.2009.04.025 |
| [28] |
Todd S W, Hoffer R M, Milchunas D G. Biomass estimation on grazed and ungrazed rangelands using spectral indices. International Journal of Remote Sensing, 1998, 19(3): 427-438. DOI:10.1080/014311698216071 |
| [29] |
王红岩, 李晓松, 张瑾, 高志海. 北京一号, 环境星, Landsat TM传感器估算草地覆盖度、叶面积指数、地上生物量比较研究. 光谱学与光谱分析, 2013, 33(10): 2803-2808. |
| [30] |
胡远宁, 冯琦胜, 陈思宇, 修丽娜, 梁天刚. 基于高分遥感数据校正的MODIS草地生物量监测模型精度评价. 草地学报, 2014, 22(04): 706-712. |
| [31] |
Hobbs T J. The use of NOAA-AVHRR NDVI data to assess herbage production in the arid rangelands of Central Australia. International Journal of Remote Sensing, 1995, 16(7): 1289-1302. DOI:10.1080/01431169508954477 |
| [32] |
Svinurai W, Hassen A, Tesfamariam E, Ramoelo A. Performance of ratio-based, soil-adjusted and atmospherically corrected multispectral vegetation indices in predicting herbaceous aboveground biomass in a Colophospermum mopane tree-shrub savanna. Grass and Forage Science, 2018, 73(3): 727-739. DOI:10.1111/gfs.12367 |
| [33] |
Braun A, Wagner J, Hochschild V. Above-ground biomass estimates based on active and passive microwave sensor imagery in low-biomass savanna ecosystems. Journal of Applied Remote Sensing, 2018, 12(4): 046027. |
| [34] |
James G, Witten D, Hastie T, Tibshirani R. An Introduction to Statistical Learning. New York: Springer, 2013: 21-24.
|
| [35] |
Meng B P, Liang T G, Yi S H, Yin J P, Cui X, Ge J, Hou M J, Lv Y Y, Sun Y. Modeling alpine grassland above ground biomass based on remote sensing data and machine learning algorithm: a case study in east of the Tibetan Plateau, China. IEEE Journal of Selected Topics in Applied Earth Observations and Remote Sensing, 2020, 13: 2986-2995. DOI:10.1109/JSTARS.2020.2999348 |
| [36] |
郭超凡, 陈泽威, 张志高. 基于最优模型选择的牧草地上生物量遥感估算研究. 草地学报, 2021, 29(05): 946-955. |
| [37] |
周志华. 机器学习. 北京: 清华大学出版社, 2016: 23-24.
|
| [38] |
Fan X Y, He G J, Zhang W Y, Long T F, Zhang X M, Wang G Z, Sun G, Zhou H K, Shang Z H, Tian D S, Li X Y, Song X N. Sentinel-2 images based modeling of grassland above-ground biomass using random forest algorithm: a case study on the Tibetan Plateau. Remote Sensing, 2022, 14(21): 5321. DOI:10.3390/rs14215321 |
| [39] |
Ge J, Hou M J, Liang T G, Feng Q S, Meng X Y, Liu J, Bao X Y, Gao H Y. Spatiotemporal dynamics of grassland aboveground biomass and its driving factors in North China over the past 20 years. Science of the Total Environment, 2022, 826: 154226. DOI:10.1016/j.scitotenv.2022.154226 |
| [40] |
卜灵心, 来全, 刘心怡. 不同机器学习算法在草原草地生物量估算上的适应性研究. 草地学报, 2022, 30(11): 3156-3164. |
| [41] |
Tran M N. A criterion for optimal predictive model selection. Communications in Statistics-Theory and Methods, 2011, 40(5): 893-906. DOI:10.1080/03610920903486798 |
| [42] |
Qian G Q, Gabor G, Gupta R P. Generalised linear model selection by the predictive least quasi-deviance criterion. Biometrika, 1996, 83(1): 41-54. DOI:10.1093/biomet/83.1.41 |
| [43] |
Zhao X X, Su Y J, Hu T Y, Cao M Q, Liu X Q, Yang Q L, Guan H C, Liu L L, Guo Q H. Analysis of UAV lidar information loss and its influence on the estimation accuracy of structural and functional traits in a meadow steppe. Ecological Indicators, 2022, 135: 108515. DOI:10.1016/j.ecolind.2021.108515 |
| [44] |
Wang D L, Xin X P, Shao Q Q, Brolly M, Zhu Z L, Chen J. Modeling aboveground biomass in Hulunber grassland ecosystem by using unmanned aerial vehicle discrete lidar. Sensors, 2017, 17(1): 180. DOI:10.3390/s17010180 |
 2024, Vol. 44
2024, Vol. 44