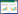文章信息
- 张春丽, 孙嘉, 李宗智, 陈俊达, 张致荣, 滕丽微, 刘振生
- ZHANG Chunli, SUN Jia, LI Zongzhi, CHEN Junda, ZHANG Zhirong, TENG Liwei, LIU Zhensheng
- 辽宁省东南部地区冬季獐肠道菌群组成及多样性差异
- Differences in gut microbial composition and diversity of water deer in winter in southeastern Liaoning Province, China
- 生态学报. 2024, 44(15): 6445-6456
- Acta Ecologica Sinica. 2024, 44(15): 6445-6456
- http://dx.doi.org/10.20103/j.stxb.202311232565
-
文章历史
- 收稿日期: 2023-11-23
- 网络出版日期: 2024-05-24
2. 辽宁省野生动物保护和疫源疫病监测中心, 大连 116013;
3. 国家林业和草原局野生动物保护学重点实验室, 哈尔滨 150040
2. Liaoning Wildlife Protection and Epidemic Disease Monitoring Center, Dalian 116013, China;
3. Key Laboratory of Conservation Biology, National Forestry and Grassland Administration, Harbin 150040, China
肠道菌群指共存于动物肠道内种类复杂、数量庞大的微生物群落[1], 与动物在营养代谢、机体健康、免疫等方面紧密相关[2]。肠道菌群与野生动物的生活史之间密不可分[3], 肠道微生物群具有适应性调节功能可以帮助动物宿主适应外界环境和食物的变化, 也可在复杂、恶劣的环境中能对宿主获取能量提供帮助[4]。
不同的栖息环境和食物组成可以直接影响动物肠道微生物的组成和结构[5—6]。郭娜等在分析不同食物组成条件下獐(Hydropotes inermis)消化道微生物组成结构后得出, 纯植食性獐消化道微生物组成结构与半植半饲的獐存在显著差异, 且具有更高的多样性[7]。黄翔等[8]对海南热带野生动植物园、大田保护区、邦溪保护区和猴猕岭坡鹿保护站的坡鹿(Cervus eldii)肠道微生物群落结构进行研究发现, 不同栖息环境下的海南坡鹿肠道微生物群落组成和结构存在显著差异, 且大田保护区内的坡鹿肠道菌群多样性显著高于另外三个地区。然而, 对于生活在东北地区栖息环境和食物组成发生改变后獐肠道微生物群落的研究却非常缺乏。
獐, 又名河麂, 喜好栖息于有水的草滩或稀疏灌丛生境, 倾向采取独居行为[9]。在《中国脊椎动物红色名录》被列为易危(VU)物种, 是我国国家II级重点保护野生动物[10]。獐曾经广泛分布于中国辽东半岛、华北平原和长江两岸地区[11]。由于栖息地丧失和人为捕猎等因素影响, 獐在东北地区的足迹逐渐消失, 近十年间主要分布在江西鄱阳湖、浙江舟山群岛等区域[12]。2017年, 李宗智等在东北地区重新发现了獐的足迹, 补充了獐在中国的重新分布范围, 为后续开展种群复壮相关研究提供基础资料[13]。目前关于獐的研究主要包括地理分布[14]、食性[15]、基因组[16]和生境偏好[17]等方面, 但仅限于我国南方分布的种群。Lin等通过对舟山群岛4个养殖场的獐种群的个体遗传信息研究发现, 每个养殖场的个体均存在于各自独立的族群, 这为后续开展獐种群有效恢复提供参考[18]。Kyung对韩国分布的獐在不同栖息地和季节条件下食物组成的变化进行了研究, 发现其相较于在灌木中, 栖息于森林地区的獐偏好取食更多的木本植物, 且冬季取食木本植物的类型多于夏季[19]。但目前仍未见基于环境变化影响的獐肠道微生物组成及多样性的相关研究, 使得目前缺乏在肠道菌群角度参考更加完善的獐种群的保护措施。
中国辽宁省大部分地区地处高纬度, 相较于其他有獐分布的如江西鄱阳湖、江苏沿海和舟山群岛等南方地区, 冬季日均温更低, 且栖息生境尤其是植被存在显著不同[20]。丁乙轩等发现吉林省地区的獐全年主要以木本植物和非禾本科植物为食[15], 与鄱阳湖獐的食性不同, 其食物组成主要以禾本科和莎草科植物为主[21]。因此生存在南北方的獐种群由于其已知的食物组成的不同, 必然会直接影响肠道微生物的差异, 导致生理、免疫、营养等不同程度的影响, 从而可能制成物种恢复的重要因素。为探究东北地区重新分布的獐的肠道菌群在寒冷气候条件下的适应机制以及对应变化, 本研究在冬季采用无损伤取样法, 收集分布在辽宁省东南部獐的新鲜粪便, 以粪便中的肠道微生物为研究对象, 通过分析寒冷季节獐肠道微生物的组成和多样性, 可以进一步了解獐在寒冷地区的种群生存适应机制, 并为后续在东北地区制定针对性的保护措施提供基础数据。
1 研究地概况本研究在辽宁省东南部的4个地区进行样本采集, 分别为隶属于本溪市桓仁满族自治县的沙尖子(SJZ)、丹东市宽甸满族自治县的下露河(XLH)和宽甸周边(KDZB)以及丹东市凤城市的凤城(FC)。研究地地理坐标为124°6′59.09″—125°37′11.27″E、40°24′43.25″—41°6′55.04″N, 位于鸭绿江北岸和西北两岸, 且4个地区的海拔高度呈梯度上升(图 1)。研究地属温带大陆性季风气候, 日照充沛, 四季分明, 全年平均气温7—11℃, 且从沿海到内陆逐渐降低, 南北气温最高相差5℃, 最高气温达30℃, 最低气温至-30℃。年平均降水量600—1100 mm, 自西北向东南递增[22]。研究区域内野生动物和植物资源较为丰富, 植被类型主要有针阔混交林、阔叶混交林、阔叶林、针叶林和灌丛等, 包括东北红豆杉(Taxus cuspidata var. cuspidata)、茶条槭(Acer ginnala)、蒙古栎(Quercus mongolica)、木贼(Equisetum hyemale)等。野生动物种类繁多, 分布有如紫貂(Martes zibellina)和原麝(Moschus moschiferus)等同营养级的被捕食者, 以及亚洲黑熊(Ursus thibetanus)和猞猁(Lynx lynx)等捕食者[15]。
|
| 图 1 研究区域及采样地点信息 Fig. 1 Study region and sampling sites in this study |
本研究基于非损伤性取样法[23]在沙尖子(SJZ)、下露河(XLH)、凤城(FC)、宽甸周边(KDZB)4个地区收集獐的新鲜粪便样本, 每个地区采集4个样本, 共16个样本。样本采集时佩戴一次性无菌手套, 为避免样品间的交叉污染, 使用消毒后的镊子将粪便样品置于50 mL离心管中, 同时记录粪便样品采集的时间和地理坐标等信息, 所有样品置于实验室-80℃冰箱中冷冻保存。
2.2 DNA提取及测序使用E.Z.N.ATM Mag-Bind Soil DNA Kit试剂盒提取粪便样本中的DNA[24], 对提取成功且通过质检的DNA使用通用引物341F:5′-CCTACGGGNGGCWGCAG-3′和805R:5′-GACTACHVGGGTATCTAATCC-3′(A)对16SrRNA基因的V3—V4区进行扩增[25]。扩增反应程序为:94℃预变性3 min;94℃变性30 s, 45℃退火20 s, 65℃延伸30 s, 25个循环;72℃延伸5 min。PCR反应体系为:2×Hieff® Robust PCR Master Mix缓冲液15 μL, Bar-PCR primer F和Primer R各1 μL, PCR products 10—20 ng, 补足H2O至30 μL。通过Illumina-HiSeq 2500平台对扩增完成的样品进行测序。
2.3 数据处理测序得到的原始序列利用Cutadapt(version 2.6)去除引物接头序列, 后通过PEAR(Version 0.9.8)拼接成一条序列[26], 使用Trimmomatic(version 0.33)对拼接后的序列进行质量过滤, 并通过Uchime(version 4.2)除去嵌合体, 获得有效序列[27]。利用Usearch(version 7.0)以97%序列一致性水平为划分标准将有效序列聚类为OTUs, 挑选出现频数最高的序列作为OTUs代表序列[28], 随后采用RDP classifier(version 2.11)贝叶斯算法对代表序列采用相对应的数据库进行分类学分析, 得到各分类水平上样本的群落组成。使用Muscle(Version 3.8.31)对多序列进行对比, 得到序列的系统发生关系。
对4组样品进行复杂度分析(Alpha Diversity), 比较獐冬季肠道微生物的多样性差异, 其中ACE、Chao1指数可以反映微生物群落丰富度, Simpson、Shannon指数反映群落多样性[29]。分别对4组样本的Alpha多样性指数进行单因素方差分析(ANOVA)和轶和检验[30], 比较其组间相关性和差异, 设置P<0.05为差异显著, P<0.01为差异极显著。同时, 对微生物群落构成进行比较分析(Beta Diversity), 使用R软件进行非度量多维度分析(NMDS)和相似性分析(ANOSIM)。NMDS可以通过点与点之间的距离来体现不同样品的差异程度, 两样本距离越接近, 表示物种组成越相似, 当stress < 0.2, 则表明样本之间存在差异[31]。ANOSIM可以对组间差异和组内差异进行检验, R值的范围在(-1, 1), R值大于0表示组间差异大于组内差异, 反之则表示组间差异小于组内差异, 设置P < 0.05表示统计具有显著性[32]。利用LEfSe软件生成线性判别分析与效应大小, 分析四组之间的差异, LDA分数的过滤值默认设置为3.5[33]。以上分析均在R(4.2.2)中进行。
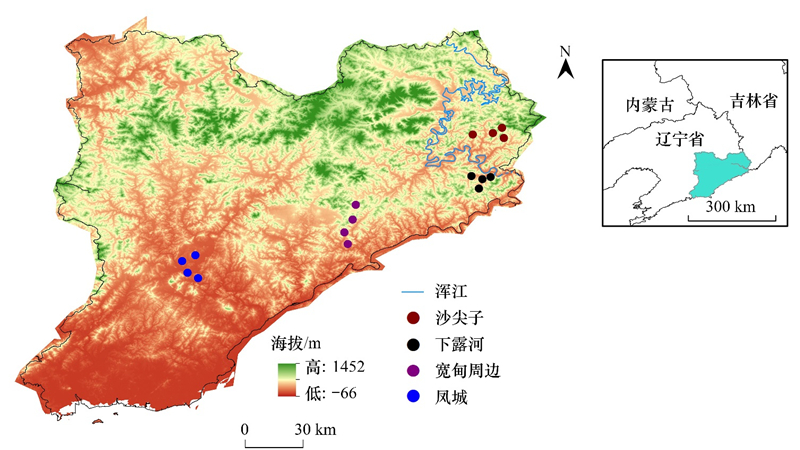
3 结果与分析 3.1 獐的肠道菌群组成测序共获得310850条原始序列, 经过质检、拼接、质控后获得有效序列309160条, 每个样品平均77290条。依据97%的序列相似性进行聚类, 共鉴定出1319个OTU, 各组样本共有OTU数量771个。凤城(FC)特有OTU数量最多, 为29个;沙尖子(SJZ)特有OTU数量最少, 为22个(图 2)。
|
| 图 2 各地区样品OTU数量 Fig. 2 The number of OTU (Operational Taxonomic Units) in each group of samples Veen diagram FC:凤城;SJZ:沙尖子;XLH:下露河;KDZB:宽甸周边 |
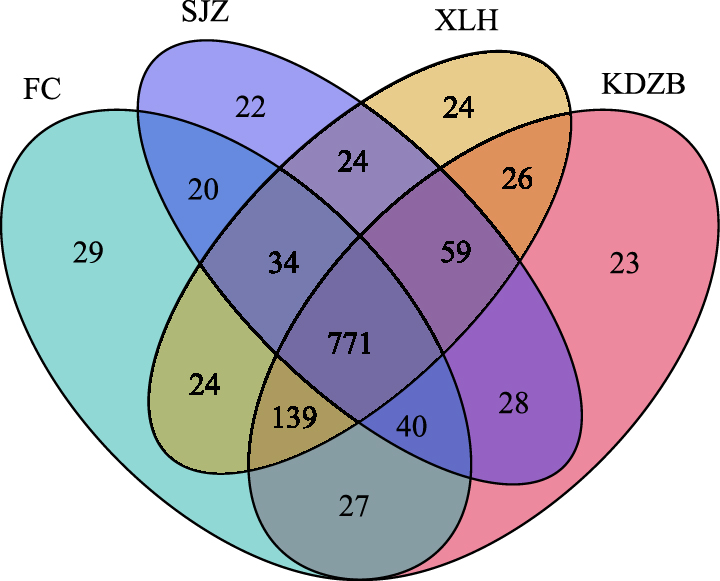
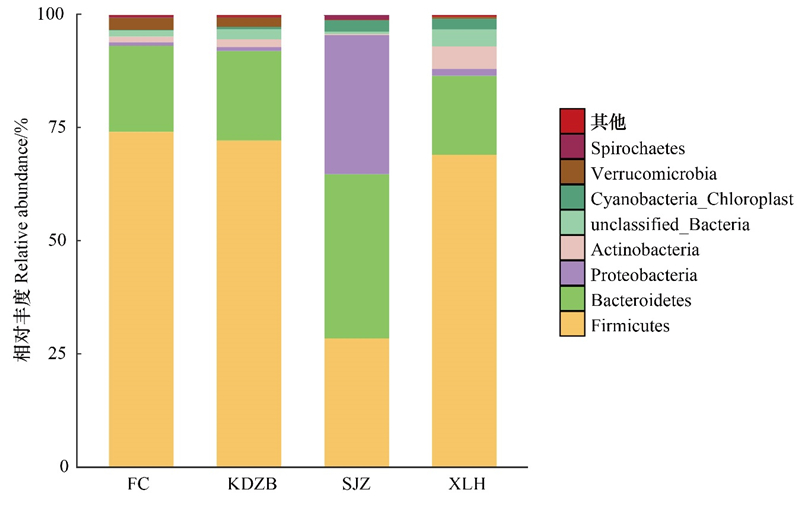
在门分类水平上, 各个地区样品共鉴定出肠道菌群18个, 其中凤城(FC)中丰度超过1%的菌门有4个, 分别为厚壁菌门(Firmicutes, 74.29%)、拟杆菌门(Bacteroidetes, 18.62%)、疣微菌门(Verrucomicrobia, 2.63%)和放线菌门(Actinobacteria, 1.28%)。宽甸周边(KDZB)中丰度超过1%的菌门有3种, 分别为厚壁菌门(79.23%)、拟杆菌门(13.82%)和疣微菌门(1.85%)。沙尖子(SJZ)中丰度超过1%的菌门有5种, 分别为拟杆菌门(35.82%)、变形菌门(Proteobacteria, 30.40%)、厚壁菌门(28.95%)、Cyanobacteria_Chloroplast(2.52%)、螺旋体门(Spirochaetes, 1.07%)。下露河(XLH)中丰度超过1%的菌门有5种, 分别为厚壁菌门(69.21%)、拟杆菌门(17.21%)、放线菌门(4.99%)、Cyanobacteria_Chloroplast(2.35%)、变形菌门(1.39%)(图 3)。
|
| 图 3 各个地区样本肠道微生物门水平下的相对丰度 Fig. 3 Relative abundance of samples at the phylum level of gut microbes in various regions FC:凤城;SJZ:沙尖子;XLH:下露河;KDZB:宽甸周边;Spirochaetes:螺旋体门;Verrucomicrobia:疣微菌门;unclassified_Bacteria:未分类细菌;Actinobacteria:放线菌门;Bacteroidetes:拟杆菌门;Firmicutes:厚壁菌门 |
厚壁菌门是凤城(FC)、下露河(XLH)、宽甸周边(KDZB)的优势菌门, 而沙尖子(SJZ)相对丰度较大的为拟杆菌门。变形菌门的丰度在沙尖子(SJZ)较高, 但在下露河(XLH)丰度较小, 在凤城(FC)和宽甸周边(KDZB)中丰度不到1%。
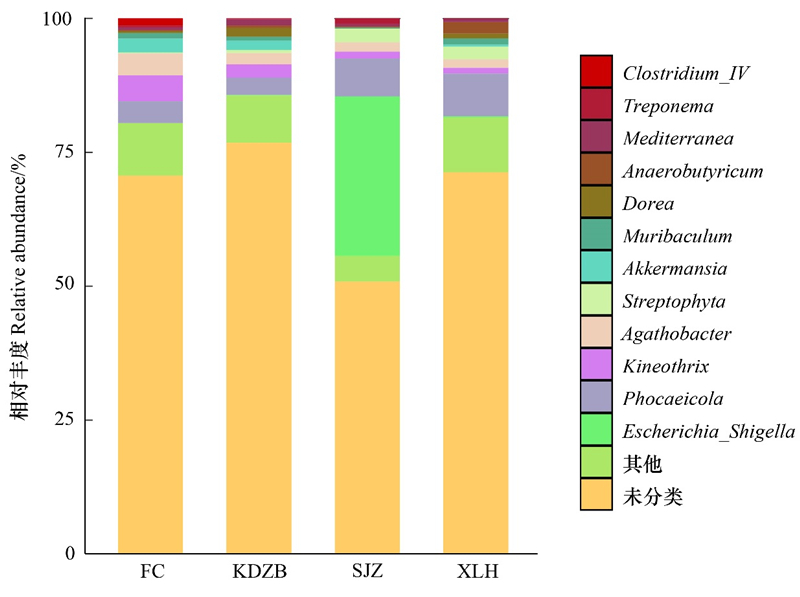
在属分类水平上, 各个地区样品共鉴定出微生物193个, 丰度超过1%的菌属共6个。其中, 凤城(FC)为丝状菌属(Kineothrix, 4.46%)、新拟杆菌属(Phocaeicola, 4.03%)、琼脂杆菌属(Agathobacter, 3.90%)、阿克曼菌属(Akkermansia, 2.53%)、梭状芽孢杆菌属(Clostridium_IV, 1.42%)和肠鼠杆菌属(Muribaculum, 1.03%)。宽甸周边(KDZB)为Phocaeicola(3.23%)、丝状菌属(2.39%)、琼脂杆菌属(1.73%)、多尔氏菌属(Dorea, 1.71%)、阿克曼菌属(1.70%)和Mediterranea(1.16%)。沙尖子(SJZ)为埃希氏志贺氏菌属(Escherichia_Shigella, 28.48%)、Phocaeicola(7.01%)、Streptophyta(2.52%)、琼脂杆菌属(1.57%)、丝状菌属(1.21%)和密螺旋体属(Treponema, 1.07%)。下露河(XLH)为Phocaeicola(7.85%)、链球菌属(2.34%)、Anaerobutyricum(2.20%)、琼脂杆菌属(1.26%)、肠鼠杆菌属(1.15%)和丝状菌属(1.06%)。埃希氏志贺氏菌属是沙尖子(SJZ)最大优势菌属, 在其它三个地区相对丰度均小于0.1%(图 4)。
|
| 图 4 各个地区样本肠道微生物属水平下的相对丰度 Fig. 4 Relative abundance of samples at the genus level of gut microbes in various regions FC:凤城;SJZ:沙尖子;XLH:下露河;KDZB:宽甸周边;Clostridium_IV:梭状芽孢杆菌属;Treponema:密螺旋体属;Dorea:多尔氏菌属;Muribaculum:肠鼠杆菌属;Akkermansia:阿克曼菌属;Agathobacter:琼脂杆菌属;Kineothrix:丝状菌属;Phocaeicola:新拟杆菌属;Escherichia_Shigella:埃希氏志贺氏菌属;Other:其它;unclassified:未分类 |
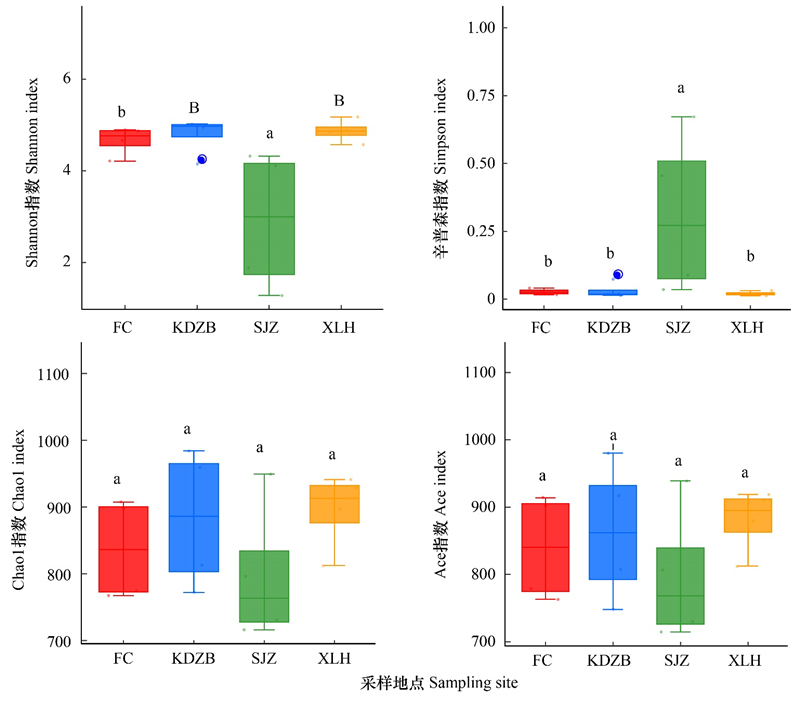
通过对4组样品进行复杂度分析(Alpha Diversity), 沙尖子(SJZ)地区獐的多样性分别与宽甸周边(KDZB)和下露河(XLH)存在极显著差异(SJZ-KDZB Shannon, P=0.007;SJZ-XLH Shannon, P=0.005), 与凤城(FC)存在显著差异(SJZ-FC Shannon, P=0.011)。Simpson指数分析与Shannon指数分析结果一致, 沙尖子(SJZ)与其它3地区的Simpson指数差异也均呈显著(SJZ-KDZB, P=0.023;SJZ-XLH, P=0.019;SJZ-FC, P=0.021)。同时, 沙尖子的Shannon指数值和丰富度指数值(ACE、Chao1)小于另外3个地区, 表明沙尖子地区的丰富度和多样性较低。(图 5)
|
| 图 5 各地区獐肠道菌群多样性差异 Fig. 5 Box plot of Alpha diversity index differences in four regions FC:凤城;SJZ:沙尖子;XLH:下露河;KDZB:宽甸周边; 相同字母表示不同地区差异不显著(P>0.05), 不同字母表示不同地区差异显著(P<0.05), 不同字母大小写不同表示差异极显著(P<0.01) |
非度量多维度分析(NMDS)结果显示, strees=0.1229 < 0.2, 具有一定代表性。除沙尖子(SJZ)地区外, 其它地区中的样品都是相对分散的混合在一起, 并未显示出明显的离散, 呈现出一定的相似性, 表明辽宁省东南部4个地区獐肠道菌群的组内结构相似, 而组间则存在着一定差异。ANOSIM分析结果与NMDS结果呈现相似, R=0.177>0, P=0.018 < 0.05, 也表明4组样品组间存在差异。其中, 沙尖子(SJZ)与其它三个地区存在显著差异(SJZ-FC, P=0.035;SJZ-KDZB, P=0.028;SJZ-XLH, P=0.04)(图 6)。

|
| 图 6 基于NMDS分析和ANOSIM分析的獐肠道菌群结构差异 Fig. 6 Differences in gut microbiota structure of water deer based on NMDS (Non-metric multidimensional scaling) analysis and ANOSIM (Analysis of similarities) analysis FC:凤城;SJZ:沙尖子;XLH:下露河;KDZB:宽甸周边 |
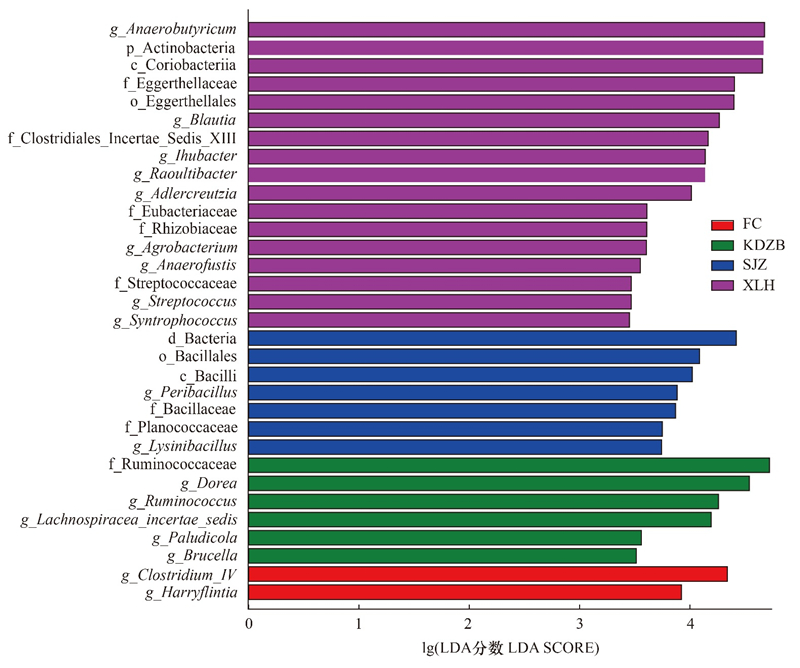
LDA直方图显示, 4个地区的獐肠道中存在着各自显著差异的细菌类群, 差异物种分属于1门2纲2目8科18属。放线菌门、Anaerobutyricum和布劳特氏菌属(Blautia)在下露河(XLH)地区分布的獐的肠道中被显著富集, 其相对丰度明显高于其它地区。凤城(FC)地区獐肠道的差异菌群仅包括梭状芽孢杆菌属(Clostridium_IV)和Harryflintia。沙尖子(SJZ)地区獐肠道中对组间差异影响最大的是近芽孢杆菌属(Peribacillus)。在宽甸周边(KDZB)中起到重要作用的是瘤胃菌科(Ruminococcaceae)、瘤胃球菌属(Ruminococcus)和多尔氏菌属(Dorea)(图 7)。
|
| 图 7 不同分类级别下各类细菌的线性判别分析LDA(Linear Discriminant Analysis)评分 Fig. 7 The LDA(Linear Discriminant Analysis)scores of various bacteria under different classification levels FC:凤城;SJZ:沙尖子;XLH:下露河;KDZB:宽甸周边;p_Actinobacteria:门_放线菌门;c_Coriobacteriia:纲_红蝽菌纲;f_Eggerthellaceae:科_伊格尔兹氏菌科;o_Eggerthellales:目_伊格尔兹氏菌目;g_Blautia:属_布劳特氏菌属;g_Adlercreutzia:属_阿德勒克罗伊茨菌属;f_Eubacteriaceae:科_优杆菌科;f_Rhizobiaceae:科_根瘤菌科;g_Agrobacterium:属_农杆菌属;g_Anaerofustis:属_粪厌氧棒形菌;f_Streptococcaceae:科_链球菌科;g_Streptococcus:属_链球菌属;g_Syntrophococcus:属_互营球菌属;o_Bacillales:目_芽孢杆菌目;c_Bacilli:纲_芽孢杆菌纲;g_Peribacillus:属_近芽孢杆菌属;f_Bacillaceae:科_芽孢杆菌科;f_Planococcaceae:科_动球菌科;g_Lysinibacillus:属_赖氨酸芽孢杆菌属;f_Ruminococcaceae:科_瘤胃菌科;g_Dorea:属_多尔氏菌属;g_Ruminococcus:属_瘤胃菌属;g_Lachnospiracea_incertae_sedis:属_毛螺菌属;g_Brucella:属_布鲁氏杆菌属;g_Clostridium_IV:属_梭状芽孢杆菌属 |
肠道菌群是动物肠道中一个复杂且动态平衡的生态网络, 对维持肠道环境的稳态和宿主的健康起到重要作用[34]。肠道菌群的失衡可能会间接影响宿主健康并且对其生活史产生不同程度的负面影响。机体内的肠道菌群与宿主在长期相互选择与适应下形成了相互制约、相互依存的关系, 两者间的相互作用是动物在生态系统中占据各自生态位和适应进化的重要因素[35]。此外, 肠道微生物群的组成结构也可以及时反映宿主饮食和环境变化等因素所产生的影响[36]。
在门分类水平中, 厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes)是冬季东北地区獐肠道中的优势菌门, 这与梅花鹿(C. nippon hortulorum)[37]、西伯利亚狍(Capreolus pygargus)[38]和白尾鹿(Odocoileus virginianus)[39]等鹿科动物肠道菌群组成研究结果基本相似, 这可能是由于厚壁菌门具有较强的降解能力, 可以帮助宿主降解食物中的纤维以促进多糖的降解, 有利于动物在食物中获得更多的能量[40];拟杆菌门具有降解碳水化合物和蛋白质等功能, 从而帮助宿主提高营养利用效率, 促进食物的消化和吸收[41]。此外, 肠道菌群的组成结构会因动物的饮食和栖息地环境等外界因素的改变而产生差异[42], Guan等在吉林省珲春市对野生和圈养梅花鹿在不同饮食条件下的肠道微生物组成进行研究发现[43], 野生梅花鹿肠道中厚壁菌门的相对丰度明显高于拟杆菌门, 而圈养梅花鹿拟杆菌门的丰度要高于厚壁菌门, 其原因就与其在不同饮食条件有关, 野生梅花鹿取食高纤维植物而圈养梅花鹿取食大多是人工投喂的玉米秸秆、杨树叶等高蛋白食物, 故圈养梅花鹿需要更多的拟杆菌门来促进碳水化合物和蛋白质等物质的降解。由于本研究在辽宁省冬季展开, 季节和地区因素导致的植物丰富度相对较低, 獐只能通过取食大量高纤维的木本植物来满足自身能量的需求, 因此厚壁菌门作为优势菌门, 在这一过程中提高了獐的食物营养吸收率, 使得其能在寒冷且食物资源缺乏的地区生存。
大部分动物肠道中的变形菌门(Proteobacteria)属于兼性厌氧菌, 会产生脂多糖和刺激性鞭毛蛋白从而促成炎症反应, 具有一定致病性[44]。相关研究表明, 在含有潜在病原微生物的水产养殖环境中, 变形菌门和Cyanobacteria_Chloroplast都是优势微生物, 这进一步表示其具有致病性[45]。在本研究结果中, 沙尖子和下露河地区分布的獐肠道中变形菌门与Cyanobacteria_Chloroplast的相对丰度均超过1%, 这可能是因为两个地区间隔浑江, 江水质量不佳, 破坏了肠道微生物群落并可能引起肠道炎症[46], 但关于浑江水质的监测仍需深入探究。此外, Sun等(2016)在冷季和暖季对西藏猕猴(Macaca mulatta)肠道微生物的差异中发现, 变形菌门的相对丰度在冷季的显著高于暖季, 因此变形菌门也可能有助于动物应对寒冷的环境, 这也与獐在寒冷地区的生存适应能力有关[45]。螺旋体门(Spirochaetes)具有降解纤维素、7碳水化合物发酵产生SCFAs等功能, 为宿主提供能量, 其在沙尖子地区獐肠道中表现出富集可能是在冬季能加快獐食物分解的速度从而产生更多能量, 但螺旋体门也属于潜在病原体, 因此有待进一步研究[48]。
在属水平下, Streptophyta在沙尖子和下露河中富集, 其是条件致病菌链球菌科(Streptococcaceae)下的一个属, 具一定的入侵能力, 会导致宿主产生许多化脓性的炎症和链球菌变态反应性疾病[49]。同时, 沙尖子地区存在的埃希氏-志贺氏菌属(Escherichia_Shigella)和密螺旋体属(Treponema)均是潜在致病菌, 也可能会导致宿主疾病的发生, 这进一步表明浑江两侧的獐可能存在相关肠道疾病的可能性。其中, 埃希氏-志贺氏菌属是水环境中常见的致病菌[50], 浑江支流贯穿沙尖子, 致病菌可能在獐饮水或取食周边植物时进入獐的体内进行累集。本研究中仅在凤城和宽甸周边的獐肠道中检测到有丰度较高的阿克曼菌属(Akkermansia), 之前有研究表明该菌属在雌性梅花鹿中富集, 具有一定的抗炎作用, 能有效降低鹿科动物孕晚期炎症感染率, 因此猜测阿克曼菌属也可能在雌性獐中富集[37]。
4.2 獐肠道菌群的多样性差异本研究通过肠道菌群Alpha多样性分析发现, 沙尖子地区的多样性低于下露河、凤城和宽甸周边。冬季东北地区积雪覆盖率高, 低海拔地区温度较高, 拥有较丰富的食物资源, 獐取食食物种类多, 其多样性也较高。沙尖子地处温度较低的高海拔地区, 獐可取食的植物资源较短缺, 因此其多样性呈现较低现象。此结果与马艳等在探究青藏高原中不同海拔高度放牧牦牛(Bos grunniens)的肠道菌群多样性结果相似, 肠道微生物群落多样性在动物体内会因海拔高度的不同而存在差异[51]。研究者普遍认为, 动物肠道菌群系统多样性和丰度越低, 机体的抵抗力和结构稳定性越差[52], 这与上述沙尖子地区肠道菌群中存在多种致病菌研究结果一致。四个地区的獐肠道微生物在丰富度上没有显著差异, 这可能是因为辽宁省东南部地区植被分布类型并未呈现显著差异, 獐取食的食物有重叠现象。该结果与Hale对哺乳动物肠道微生物研究结果相似, 即动物在高度相似的食物条件下, 肠道微生物群落会呈现出趋同现象[53]。然而沙尖子与其它三个地区之间多样性差异显著, 这与NMDS分析结果一致, 这种差异可能是由于沙尖子独特的地理位置所导致。沙尖子与其它三个地区的通路被浑江阻隔, 以致于浑江两岸的獐可能存在地理隔离现象。先前Jamtsho等在不丹东部的Sherichhu河两岸植被类型研究中表明, 地形、微生境、河道宽度等因素都可能会影响河两岸的植物物种多样性和分布格局[54]。本研究在实地调查中发现, 浑江两岸植被分布和植被类型因地形等因素存有一定差异, 这可能是辽宁省东南部4个地区样本中呈现差异的原因之一。此外, 下露河、凤城和宽甸周边采集到的粪便样本多分布于村庄附近, 其生境和取食不同于沙尖子, 这或许也是出现差异的原因之一。
4.3 不同地区獐肠道优势菌群组间差异通过LEfSe分析发现, 放线菌门和Anaerobutyricum在下露河中显著聚集, 这是因为放线菌门一般存在草食动物肠道内, 具有促进消化等功能, 而Anaerobutyricum可以促进动物益生菌生长, 与动物的体重成正相关关系[55], 因此它的富集可能意味着下露河地区的獐体重较大, 同时Anaerobutyricum属内的某些种能够调节十二指肠信号从而改善宿主的代谢机制[56], 进而可能有益于獐机体内环境的稳定。布劳特氏菌属(Blautia)、瘤胃球菌属(Ruminococcus)、多尔氏菌属(Dorea)在冬季獐体内也表现出明显富集, 布劳特氏菌属是毛螺菌科(Lachnospiraceae)下的一个属, 毛螺菌科在降解植物纤维方面具有强降解能力, 且在预防肠道炎症性疾病和代谢疾病方面起到一定作用[57], 在冬季, 毛螺菌科的富集利于獐最大程度降解粗纤维, 满足獐对能量的需求以御寒冬;瘤胃球菌属可以在反刍动物瘤胃中发酵并产生众多纤维素酶和半纤维素酶[58], 这些酶将食物中的膳食纤维转化为反刍动物所需的各种营养物质, 并在食物消化和碳水化合物代谢过程中起关键作用[59], 其在獐肠道内的富集也可以帮助促进植物纤维的消化;多尔氏菌属隶属于厚壁菌门中的梭状芽胞杆菌类(Clostridium), 其在肠道微生物中的相对丰度与动物获取能量和维持体重紧密相关[60], 较多的多尔氏菌属可能会抑制体重的减轻, 所以在辽宁省东南部地区冬季, 其可能会为獐保存更多脂肪以度过寒冬[61]。沙尖子地区獐的肠道中, 对组间差异影响最显著的是近芽孢杆菌属(Peribacillus), 有研究在其某段基因组序列中鉴定出参与脂肪酸代谢和胆汁酸生物合成的基因, 这对于调节动物肠道健康和消化至关重要[62]。梭状芽孢杆菌属(Clostridium_IV)会产生丁酸盐, 能满足结肠上皮细胞所需的营养物质和能量需求, 有助于维持肠道免疫稳态[63], 这可能会导致凤城地区獐具有较强的免疫抵抗能力, 降低其过敏和炎症等疾病的发生率。
本研究对辽宁省东南部地区冬季獐肠道菌群的组成及多样性进行了初步探究分析, 得出四个地区獐肠道中的优势菌门均为厚壁菌门和拟杆菌门。但沙尖子地区变形菌的相对丰度与厚壁菌门和拟杆菌门接近, 并且沙尖子地区与其它地区的肠道菌群多样性存在差异。该原因一方面是因为沙尖子与另三地区有浑江相隔形成地理隔离带, 另一方面可能是因为浑江两侧生境的差别而导致。东北地区冬季食物资源匮乏, 为进一步保护獐种群在该地区的恢复, 建议加强保护獐主要取食植物种类及数量密度, 满足其在寒冷环境下的生存需求。
| [1] |
Ley R E, Hamady M, Lozupone C, Turnbaugh P J, Ramey R R, Bircher J S, Schlegel M L, Tucker T A, Schrenzel M D, Knight R, Gordon J I. Evolution of Mammals and Their Gut Microbes. Science, 2008, 320(5883): 1647-1651. DOI:10.1126/science.1155725 |
| [2] |
Malmuthuge N, Griebel P J, Guan L L. The Gut Microbiome and Its Potential Role in the Development and Function of Newborn Calf Gastrointestinal Tract. Frontiers in veterinary science, 2015, 2: 36. |
| [3] |
Nicholson J K, Holmes E, Kinross J, Burcelin R, Gibson G, Jia W, Pettersson S. Host-Gut Microbiota Metabolic Interactions. Science, 2012, 336(6086): 1262-1267. DOI:10.1126/science.1223813 |
| [4] |
Hicks A L, Lee K J, Couto-Rodriguez M, Patel J, Sinha R, Guo C, Olson S H, Seimon A, Seimon T A, Ondzie A U, Karesh W B, Reed P, Cameron K N, Lipkin W I, Williams B L. Gut microbiomes of wild great apes fluctuate seasonally in response to diet. Nature communications, 2018, 9: 1786. DOI:10.1038/s41467-018-04204-w |
| [5] |
Li Z P, Wright A D G, Liu H L, Bao K, Zhang T T, Wang K Y, Cui X Z, Yang F H, Zhang Z G, Li G Y. Bacterial Community Composition and Fermentation Patterns in the Rumen of Sika Deer (Cervus nippon) Fed Three Different Diets. Microbial ecology, 2015, 69(2): 307-318. DOI:10.1007/s00248-014-0497-z |
| [6] |
Shinohara A, Nohara M, Kondo Y, Jogahara T, Nagura-Kato G A, Izawa M, Koshimoto C. Comparison of the gut microbiotas of laboratory and wild Asian house shrews (Suncus murinus) based on cloned 16S rRNA sequences. Experimental animals, 2019, 68(4): 531-539. DOI:10.1538/expanim.19-0021 |
| [7] |
郭娜. 獐的消化道微生物以及摄取盐对獐粪便微生物的影响[D]. 上海: 华东师范大学, 2019.
|
| [8] |
黄翔. 不同栖息环境下海南坡鹿肠道微生物组成差异分析[D]. 海南: 海南大学, 2022.
|
| [9] |
Peterson R O, Geist V. Deer of the World: Their Evolution, Behavior, and Ecology. The Journal of Wildlife Management, 2000, 64(2): 606. |
| [10] |
蒋志刚, 江建平, 王跃招, 张鹗, 张雁云, 李立立, 谢锋, 蔡波, 曹亮, 郑光美, 董路, 张正旺, 丁平, 罗振华, 丁长青, 马志军, 汤宋华, 曹文宣, 李春旺, 胡慧建, 马勇, 吴毅, 王应祥, 周开亚, 刘少英, 陈跃英, 李家堂, 冯祚建, 王燕, 王斌, 李成, 宋雪琳, 蔡蕾, 臧春鑫, 曾岩, 孟智斌, 方红霞, 平晓鸽. 中国脊椎动物红色名录. 生物多样性, 2016, 24(5): 501-551. |
| [11] |
Hu J, Fang S G, Wan Q H. Genetic diversity of Chinese water deer (Hydropotes inermis inermis): implications for conservation. Biochemical genetics, 2006, 44(3): 156-167. |
| [12] |
申锦. 基于3S技术研究鄱阳湖野放河麂的家域利用模式[D]. 南昌: 江西师范大学, 2021.
|
| [13] |
李宗智, 吴建平, 滕丽微, 刘振生, 王宝昆, 刘延成, 徐涛. 獐在吉林省的重新发现. 动物学杂志, 2019, 54(1): 108-112. |
| [14] |
Li Z Z, Khattak R H, Han X Z, Zhang N, Wu J P, Liu Z S, Teng L W. Distribution update of water deer (Hydropotes inermis) and prediction of their potential distribution in Northeast China. Scientific Reports, 2023, 13(1): 5610. DOI:10.1038/s41598-023-32314-z |
| [15] |
丁乙轩. 吉林白山原麝国家级自然保护区獐食性研究[D]. 哈尔滨: 东北林业大学, 2022.
|
| [16] |
Li Z Z, Zhang Z R, Mi S H, Wu J P, Xu T, Liu Z S, Teng L W. The complete mitochondrial genome of water deer in Liaoning, China. Mitochondrial DNA Part B, Resources, 2020, 5(1): 922-923. DOI:10.1080/23802359.2020.1719936 |
| [17] |
李宗智, 刘振生, 米书慧, 吴建平, 滕丽微. 白山原麝国家级自然保护区獐春夏生境选择. 生态学报, 2021, 41(4): 1625-1633. DOI:10.5846/stxb201911122385 |
| [18] |
Lin J J, Bao Y X, Liu J, Wang Y N, Zhang X. Genetic diversity and genetic structure of four cultured populations of Chinese water deer (Hydropotes inermis) in Zhoushan Archipelago. Acta Ecologica Sinica, 2013, 33(11): 3460-3469. DOI:10.5846/stxb201203120330 |
| [19] |
Lee S K, Woo C, Lee E J, Yamamoto N. Using high-throughput sequencing to investigate the dietary composition of the Korean water deer (Hydropotes inermis argyropus): a spatiotemporal comparison. Scientific Reports, 2022, 12: 22271. DOI:10.1038/s41598-022-26862-z |
| [20] |
Sun Y, Li Z Z, Chen J D, Khattak R H, Liu Z S, Teng L W. Habitat Selection: Autumn and Winter Behavioral Preferences of Water Deer (Hydropotes inermis) in Northeast China. Sustainability, 2023, 15(16): 12181. DOI:10.3390/su151612181 |
| [21] |
黄燕. 鄱阳湖区枯水期和丰水期獐(Hydropotes inermis)的食性分析[D]. 南昌: 江西师范大学, 2016.
|
| [22] |
王荣莉. 基于多源遥感数据与随机森林模型的辽宁省作物生长季干旱监测研究. 黑龙江科学, 2023, 14(18): 16-19. |
| [23] |
Fasoli S, Ferlizza E, Andreani G, Sandri C, Dondi F, Isani G. Noninvasive sampling method for urinalysis and urine protein profile in captive giraffes. Journal of Veterinary Diagnostic Investigation, 2021, 33(1): 25-34. DOI:10.1177/1040638720975370 |
| [24] |
夏凡, 杨婧, 李建, 史洋, 盖立新, 黄文华, 张经纬, 杨南, 高福利, 韩莹莹, 鲍伟东. 北京地区四个豹猫亚种群肠道菌群的组成. 生物多样性, 2022, 30(9): 100-109. |
| [25] |
Jakobsson H E, Abrahamsson T R, Jenmalm M C, Harris K, Quince C, Jernberg C, Björkstén B, Engstrand L, Andersson A F. Decreased gut microbiota diversity, delayed Bacteroidetes colonisation and reduced Th1 responses in infants delivered by caesarean section. Gut, 2014, 63(4): 559-566. DOI:10.1136/gutjnl-2012-303249 |
| [26] |
Zhang J J, Kobert K, Flouri T, Stamatakis A. PEAR: a fast and accurate Illumina Paired-End reAd mergeR. Bioinformatics, 2014, 30(5): 614-620. DOI:10.1093/bioinformatics/btt593 |
| [27] |
MagočT, Salzberg S L. FLASH: fast length adjustment of short reads to improve genome assemblies. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507 |
| [28] |
Edgar R C. UPARSE: highly accurate OTU sequences from microbial amplicon reads. Nature methods, 2013, 10: 996-998. DOI:10.1038/nmeth.2604 |
| [29] |
Eckburg P B, Bik E M, Bernstein C N, Purdom E, Dethlefsen L, Sargent M, Gill S R, Nelson K E, Relman D A. Diversity of the human intestinal microbial flora. Science, 2005, 308(5728): 1635-1638. DOI:10.1126/science.1110591 |
| [30] |
张媛媛, 赵素芬, 王运盛, 李祥翔, 蒋鹏, 刘燕, 张成林, 卢岩. 三种圈养金丝猴肠道微生物组差异分析. 野生动物学报, 2023, 44(3): 580-592. |
| [31] |
Kilroy C, Stephens T, Greenwood M, Wech J, Brown L, Matthews A, Patterson M, Patterson M. Improved predictability of peak periphyton in rivers using site-specific accrual periods and long-term water quality datasets. The Science of the Total Environment, 2020, 736: 139362. DOI:10.1016/j.scitotenv.2020.139362 |
| [32] |
Clarke K R, Somerfield P J, Gorley R N. Clustering in non-parametric multivariate analyses. Journal of Experimental Marine Biology and Ecology, 2016, 483: 147-155. DOI:10.1016/j.jembe.2016.07.010 |
| [33] |
Segata N, Izard J, Waldron L, Gevers D, Miropolsky L, Garrett W S, Huttenhower C. Metagenomic biomarker discovery and explanation. Genome biology, 2011, 12(6): R60. DOI:10.1186/gb-2011-12-6-r60 |
| [34] |
Hua Y, Cao H Q, Wang J, He F P, Jiang G S. Gut microbiota and fecal metabolites in captive and wild North China leopard (Panthera pardus japonensis) by comparsion using 16 s rRNA gene sequencing and LC/MS-based metabolomics. BMC Veterinary Research, 2020, 16(1): 363. DOI:10.1186/s12917-020-02583-1 |
| [35] |
Amato K R. Incorporating the gut microbiota into models of human and non-human primate ecology and evolution. American Journal of Physical Anthropology, 2016, 159(Suppl 61): S196-S215. |
| [36] |
Perofsky A C, Lewis R J, Meyers L A. Terrestriality and bacterial transfer: a comparative study of gut microbiomes in sympatric Malagasy mammals. The ISME journal, 2019, 13(1): 50-63. DOI:10.1038/s41396-018-0251-5 |
| [37] |
元虹懿. 东北梅花鹿夏季和冬季肠道菌群变化及其功能差异分析[D]. 哈尔滨: 东北林业大学, 2022.
|
| [38] |
Liu J Y, Liang X C, Liu Y H. Comparison of the gut microbiota composition between captive and wild Roe deer. bioRxiv, 2019, 831222. |
| [39] |
Delgado M L, Singh P, Funk J A, Moore J A, Cannell E M, Kanesfsky J, Manning S D, Scribner K T. Intestinal Microbial Community Dynamics of White-Tailed Deer (Odocoileus virginianus) in an Agroecosystem. Microbial ecology, 2017, 74(2): 496-506. DOI:10.1007/s00248-017-0961-7 |
| [40] |
Waite D W, Taylor M W. Characterizing the avian gut microbiota: membership, driving influences, and potential function. Frontiers in microbiology, 2014, 5: 223. |
| [41] |
Pitta D W, Indugu N, Kumar S, Vecchiarelli B, Sinha R, Baker L D, Bhukya B, Ferguson J D. Metagenomic assessment of the functional potential of the rumen microbiome in Holstein dairy cows. Anaerobe, 2016, 38: 50-60. DOI:10.1016/j.anaerobe.2015.12.003 |
| [42] |
Dong Z H, Zhang S, Cheng Y W, He X C, Haase I, Liang Y, Jiang Y, Wu Y J. Comparative analysis of the intestinal tract microbiota and feeding habits of five sympatric flycatchers. Avian Research, 2022, 13: 100050. DOI:10.1016/j.avrs.2022.100050 |
| [43] |
Guan Y, Yang H T, Han S Y, Feng L M, Wang T M, Ge J P. Comparison of the gut microbiota composition between wild and captive sika deer (Cervus nippon hortulorum) from feces by high-throughput sequencing. AMB Express, 2017, 7(1): 212. DOI:10.1186/s13568-017-0517-8 |
| [44] |
郭仕辉, 余永涛, 万佳宏, 毛彦妮, 张浩东, 张津慎, 田新岳, 赵清梅. 变形菌门与哺乳动物结肠肠道菌群失调相关研究进展. 中国微生态学杂志, 2022, 34(4): 479-484. |
| [45] |
Zhang H, Sun Z L, Liu B, Xuan Y M, Jiang M, Pan Y S, Zhang Y M, Gong Y, Lu X P, Yu D S, Kumar D, Hu X L, Cao G, Xue R Y, Gong C. Dynamic changes of microbial communities in Litopenaeus vannamei cultures and the effects of environmental factors. Aquaculture, 2016, 455: 97-108. DOI:10.1016/j.aquaculture.2016.01.011 |
| [46] |
Liu H, Guo X W, Gooneratne R, Lai R F, Zeng C, Zhan F B, Wang W M. The gut microbiome and degradation enzyme activity of wild freshwater fishes influenced by their trophic levels. Scientific reports, 2016, 6: 24340. DOI:10.1038/srep24340 |
| [47] |
Sun B H, Wang X, Bernstein S, Huffman M A, Xia D P, Gu Z Y, Chen R, Sheeran L K, Wagner R S, Li J H. Marked variation between winter and spring gut microbiota in free-ranging Tibetan Macaques (Macaca thibetana). Scientific Reports, 2016, 6: 26035. DOI:10.1038/srep26035 |
| [48] |
Craine J M, Dybzinski R. Mechanisms of plant competition for nutrients, water and light. Functional Ecology, 2013, 27(4): 833-840. DOI:10.1111/1365-2435.12081 |
| [49] |
Teresa S M M, Maria R C, Clara R G, Maria A T, Lucia G V. Streptococcus pyogenes as an etiological agent of acute epiglottitis. Anales de pediatria, 2023, 99(6): 438-439. DOI:10.1016/j.anpedi.2023.05.015 |
| [50] |
Motlagh A M, Yang Z J. Detection and occurrence of indicator organisms and pathogens. Water Environment Research: a Research Publication of the Water Environment Federation, 2019, 91(10): 1402-1408. DOI:10.1002/wer.1238 |
| [51] |
马艳, 向信, 樊嘉凯, 张本印. 海拔高度对青藏高原放牧牦牛肠道菌群多样性的影响. 微生物学通报, 2022, 49(2): 620-634. |
| [52] |
Singh P, Teal T K, Marsh T L, Tiedje J M, Mosci R, Jernigan K, Zell A, Newton D W, Salimnia H, Lephart P, Sundin D, Khalife W, Britton R A, Rudrik J T, Manning S D. Intestinal microbial communities associated with acute enteric infections and disease recovery. Microbiome, 2015, 3: 45. DOI:10.1186/s40168-015-0109-2 |
| [53] |
Hale V L, Tan C L, Niu K F, Yang Y Q, Knight R, Zhang Q K, Cui D Y, Amato K R. Diet Versus Phylogeny: a Comparison of Gut Microbiota in Captive Colobine Monkey Species. Microbial ecology, 2018, 75(2): 515-527. DOI:10.1007/s00248-017-1041-8 |
| [54] |
Jamtsho T, Wangchuk P, Yeshi K, Tobgay, Sridith K. Riparian plant species of Sherichhu River in Eastern Bhutan: their diversity, distribution, and ethnobotanical uses. Tropical Ecology, 2021, 62(4): 563-579. DOI:10.1007/s42965-021-00173-2 |
| [55] |
Liu J, Robinson K, Lyu W T, Yang Q, Wang J, Christensen K D, Zhang G L. Anaerobutyricum and Subdoligranulum are differentially enriched in broilers with disparate weight gains. Animals: an Open Access Journal from MDPI, 2023, 13(11): 1834. |
| [56] |
Wortelboer K, Koopen A M, Herrema H, de Vos W M, Nieuwdorp M, Kemper E M. From fecal microbiota transplantation toward next-generation beneficial microbes: The case of Anaerobutyricum soehngenii. Frontiers in Medicine, 2022, 9: 1077275. DOI:10.3389/fmed.2022.1077275 |
| [57] |
Wu X R, Chen Z Z, Dong X L, Zhao Q P, Cai J. A Novel Symbiotic Formulation Reduces Obesity and Concomitant Metabolic Syndrome in Rats by Raising the Relative Abundance of Blautia. Nutrients, 2023, 15(4): 956. DOI:10.3390/nu15040956 |
| [58] |
La Reau A J, Meier-Kolthoff J P, Suen G. Sequence-based analysis of the genus Ruminococcus resolves its phylogeny and reveals strong host association. Microbial Genomics, 2016, 2(12): e000099. |
| [59] |
La Reau A J, Suen G. The Ruminococci: key symbionts of the gut ecosystem. Journal of Microbiology, 2018, 56(3): 199-208. DOI:10.1007/s12275-018-8024-4 |
| [60] |
Korpela K, Flint H J, Johnstone A M, Lappi J, Poutanen K, Dewulf E, Delzenne N, de Vos W M, Salonen A. Gut microbiota signatures predict host and microbiota responses to dietary interventions in obese individuals. PloS one, 2014, 9(6): e90702. |
| [61] |
Sowah S A, Milanese A, Schübel R, Wirbel J, Kartal E, Johnson T S, Hirche F, Grafetstätter M, Nonnenmacher T, Kirsten R, López-Nogueroles M, Lahoz A, Schwarz K V, Okun J G, Ulrich C M, Nattenmüller J, von Eckardstein A, Müller D, Stangl G I, Kaaks R, Kühn T, Zeller G. Calorie restriction improves metabolic state independently of gut microbiome composition: a randomized dietary intervention trial. Genome Medicine, 2022, 14(1): 30. DOI:10.1186/s13073-022-01030-0 |
| [62] |
Jiang L M, Jung W Y, Park S H, Kang S W, Lee M K, Lee J S, Lee J H, Lee J Y. Draft genome sequence of Peribacillus sp. AGMB 02131 isolated from feces of a Korean cow. Korean Journal of Micorbiology, 2021, 57(1): 66-68. |
| [63] |
Sartor R B. The intestinal microbiota in inflammatory bowel diseases. Nestle Nutrition Institute workshop series, 2014, 79: 29-39. |
 2024, Vol. 44
2024, Vol. 44