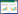文章信息
- 宋亚娜, 张珊珊, 胡太蛟, 吴明基
- SONG Yana, ZHANG Shanshan, HU Taijiao, WU Mingji
- 低甲烷排放转基因水稻对土壤微生物群落的影响
- Effect of low-methane transgenic rice on microbial communities in paddy soil
- 生态学报. 2024, 44(15): 6667-6683
- Acta Ecologica Sinica. 2024, 44(15): 6667-6683
- http://dx.doi.org/10.20103/j.stxb.202302210283
-
文章历史
- 收稿日期: 2023-02-21
- 网络出版日期: 2024-05-24
稻田是温室气体甲烷(CH4)的主要排放源之一, 其年排放量约占全球甲烷排放总量的12%—26%[1]。稻田甲烷排放是化学、物理和生物因素共同作用, 由土壤微生物驱动的甲烷产生、氧化及释放的过程[2—3]。
水稻品种遗传性状决定的光合特性、根系特性、通气组织运输能力等水稻生物学特性, 是影响稻田甲烷排放的关键因素。甲烷低排高产水稻品种的选育是减少稻田甲烷排放、实现水稻低碳生产的关键措施。近年来, 随着现代分子技术的迅速发展, 借助大麦(Hordeum vulgare L.)转录因子Hvsusiba2基因调节水稻光合产物的分配, 将Hvsusiba2基因转化水稻, 培育出了籽粒高淀粉含量且甲烷低排的转基因水稻材料[4]。与野生型水稻比较低甲烷排放转基因水稻生长季内甲烷总排放量的减排率可达到30%—50%[4—6], 实现了减少稻田甲烷排放、促进水稻增产的目标。
研究发现, 低甲烷排放转基因水稻改变了水稻光合碳的产生与分配, 提高籽粒淀粉含量的同时减少了光合碳向根系的分配, 水稻根系体积明显变小, 土壤中产甲烷菌群落发生变化、丰度显著降低, 从而降低了稻田甲烷排放量[7]。
微生物驱动了土壤物质循环及肥力演变, 微生物群落多样性直接影响土壤生态系统的稳定性与可持续性[8]。土壤养分转化、物质代谢过程均由相关功能微生物驱动进行, 如, 参与生物固氮的土壤固氮菌、氮素硝化及反硝化过程的硝化和反硝化细菌, 其中固氮菌nifH基因、氨氧化细菌amoA基因以及反硝化细菌nirS基因等功能基因的丰度与功能, 是评价土壤固氮及氮素转化的重要指标。
低甲烷排放转基因水稻促进了碳向植株地上部的运输而减少了碳向地下部的运输[4], 随根系脱落物分泌物进入土壤中的有机碳减少, 微生物生存所需碳源减少, 从而影响其生存并可能改变微生物群落的组成和功能。此外, 通过根系分泌物和作物残留物进入农田土壤的转基因作物的外源基因及其表达产物, 可能影响土壤微生物群落的多样性及功能[9]。虽然, 低甲烷排放转基因水稻能够显著实现稻田甲烷减排, 对减缓农业温室气体排放和控制全球变暖具有重要意义, 但是, 其种植是否会改变土壤微生物群落组成与功能、影响土壤微生态系统的稳定与可持续性是十分值得关注的问题, 也是转基因作物释放所必须的环境安全评价的研究内容。
目前, 高通量测序技术是土壤微生物分子生态学研究的主要手段之一[10—11]。高通量测序在稻田微生物多样性的研究中也有广泛应用, 如, 我们曾利用高通量测序技术分析了抗虫转基因水稻土壤细菌和真菌群落组成的变化[8];还有研究通过对红壤性稻田根际微生物的高通量测序, 发现施用生物黑炭的水稻根际土壤中促生菌有增加趋势[12]; 通过对细菌16S保守区的高通量测序, 发现轮作土壤微生物丰富度远高于连做[13]等。
为了揭示低甲烷排放转基因水稻对土壤微生物群落的影响, 本研究通过对土壤细菌和真菌的高通量测序, 及固氮菌nifH基因、氨氧化细菌amoA基因、反硝化细菌nirS基因和产甲烷菌mcrA基因的定量PCR分析, 初步明确了低甲烷排放转基因水稻土壤微生物群落组成与功能的变化情况, 为其应用及环境安全评价提供了理论依据。
1 材料与方法 1.1 田间试验于2019—2021年进行了3年田间定位试验, 试验地点在福建省农业科学院寿山试验站(26°11′N, 119°16′E)。本研究的田间试验获得了转基因植物中间试验安全审批(农基安办报告字[2018]第334号、农基安办报告字[2021]第2518号)。试验设置低甲烷排放转基因水稻(86R27-3)及其对应的非转基因野生型籼稻“明恢86”(MH86)的2个处理, 每个处理重复3次, 6个田间小区随机排列, 小区面积35 m2。其中, 利用大麦Hvsusiba2基因构建双T-DNA载体pCAMBIA1301-sbeIIb-hvsusiba2, 通过农杆菌方法转化水稻“明恢86”(MH86), 经过多代自交获得转Hvsusiba2基因纯合水稻株系86R27-3。试验水稻栽培的肥水和田间管理按当地常规进行。供试土壤为红壤, 基本理化性状如下: pH 5.78, 有机质含量19.50 g/kg, 全氮、磷、钾含量分别为1.19 g/kg、0.25 g/kg和18.26 g/kg, 有效氮、磷、钾含量分别为123.62 mg/kg、25.67 mg/kg和70.23 mg/kg。田间试验水稻作中稻栽培, 即5月初播种、6月初插秧及9月中下旬收获, 稻茬自然越冬, 每年种植的田间小区排列方式保持一致。
1.2 土壤样品采集在定位试验第3年于水稻分蘖期、齐穗期和成熟期采集稻田土壤, 采用5点法用土钻采集0—20 cm深度的土层土壤, 每小区采集的土壤混合为1个样品。每个样品分成2份, 分别用于土壤微生物分析和土壤理化测定。
1.3 土壤微生物DNA提取用FastDNA SPIN Kit (For Soil) (QBIOgene)的试剂盒提取土壤微生物总DNA, DNA样品-20 ℃保存待用。
1.4 土壤微生物的荧光定量PCR用Real-Time PCR System(ABI PRISM7500)扩增仪分别对固氮菌、氨氧化细菌、反硝化细菌和产甲烷菌的功能基因进行绝对荧光定量PCR分析[14—17] (表 1)。反应体系包括稀释5倍的DNA1 μL, SYBRPremix Ex TaqTM (2×)10 μL, ROX Reference DyeⅡ (50 ×)0.4 μL, 引物各1 μL (10 pmol/L生工生物工程, 上海), 超纯水补齐至总体积20μL。扩增条件为:先95 ℃预变性30 s, 然后进行40个循环, 每个循环变性95 ℃ 30 s, 退火温度见表 1、退火时间40 s, 延伸72 ℃ 40 s。通过将各个基因的PCR扩增产物克隆到pMD-18载体(TaKaRa大连)后以质粒DNA为标准品, 10倍梯度稀释质粒DNA制作标准曲线[18]。获得各功能基因的标准曲线范围分别为, nifH:9.00×108—9.00×103拷贝数/μL、amoA:1.42×108—1.42×103拷贝数/μL、nirS:4.38×108—4.38×103拷贝数/μL和mcrA:4.56×108—4.56×103拷贝数/μL, 标准曲线的R2均达到0.99, 扩增效率分别为1.02、0.99、0.98和1.01。
| 目标基因 Target gene |
引物序列 Primer sequence |
扩增长度/0.01% Amplification length |
退火温度/℃ Annealing temperature |
| 固氮菌nifH基因 Nitrogen-fixing bacterial nifH gene |
nifH-F: AAAGGYGGWATCGGYAARTCCACCAC nifH-R: TTGTTSGCSGCRTACATSGCCATCAT |
458 | 61 |
| 氨氧化细菌amoA基因 Ammonia-oxidizing bacterial amoA gene |
amoA1F: GGGGTTTCTACTGGTGGT amoA2R: CCCCTCKGSAAAGCCTTCTTC |
490 | 57 |
| 反硝化细菌nirS基因 Denitrifying bacterial nirS gene |
nirS4F: TTCRTCAAGACSCAYCCGAA nirS6R: CGTTGAACTTRCCGGT |
336 | 53 |
| 产甲烷菌mcrA基因 Methanogenic bacterial mcrA gene |
mcrA-F: GGTGGTGTMGGATTCACACARTAYGCWACAGC mcrA-R: TTCATTGCRTAGTTWGGRTAGTT |
470 | 53 |
由上海派森诺生物科技股份有限公司进行微生物基因高通量测序及云分析。细菌扩增16S rRNA V3—V4区(338F: ACTCCTACGGGAGGCAGCA/806R: GGACTACHVGGGTWTCTAAT), 真菌扩增ITS V1区(F: GGAAGTAAAAGTCGTAACAAGG/R: GCTGCGTTCTTCATCGATGC)。Illumina平台测序, 获得数据使用QIIME2 (2019.4) 软件包进行分析[19]。首先, 在97%相似度水平对高质量序列聚类, 获得门、纲、目、科和属各水平下的分类单元[8];然后进行微生物群落α-多样性分析, 包括物种丰富度指数Chao1、Observed_species, 多样性指数Shannon、Simpson, 均匀度指数Pielou_e及覆盖度指数Goods_coverage[8];依据Bray-Curtis距离进行群落组成的主坐标分析绘制二维散点图展示微生物群落β-多样性[8];同时进行组间差异的PERMANOVA, Anosim和Permdisp显著性检验[20]。选平均丰度前20位的属利用R (v3.2.0)语言和pheatmap (1.0.12)软件包绘制物种组成热图[8]。基于MetaCyc数据库使用PICRUSt2 (2.2.0)软件进行群落功能预测, 绘制依据Bray-Curtis距离的功能单元二维散点图[8];另外, 分别使用FAPROTAX (1.1)和FUNGuild (1.1a)数据库对细菌和真菌功能进行预测, 分析比较细菌及真菌主要功能群丰度差异。
1.6 植株和土壤分析收获时各小区取5株水稻分别测定单株产量、茎叶和根系干物重及有机质、氮、磷、钾养分含量。植物有机质按NY525—2012方法测定, 植物全氮、全磷和全钾测定采用硫酸双氧水消煮后, 分别进行凯氏定氮、矾钼黄比色和火焰光度检测[21]。风干土样进行有机质和有效氮磷钾含量分析, 及pH (1∶2.5 H2O浸提法)值测定。其中, 土壤有机质测定按重铬酸钾容量法, 有效氮磷钾分别采用碱解扩散法、0.5 mol/L NaHCO3浸提法和NH4OAc浸提火焰光度法[21]。
1.7 数据分析数据处理和制图使用Microsoft Excel 2007软件, 处理间的单因素方差分析(one-way ANOVA)及LSD 0.05水平显著性检验使用SPSS 17.0软件[8]。
2 结果与分析 2.1 水稻植株与土壤分析水稻植株生物量及产量测定结果显示(表 2):低甲烷排放转基因水稻(86R27-3)与野生型水稻(MH86)的植株茎叶干重及单株产量均没有明显差异, 但86R27-3的根系干重显著低于(P < 0.05) MH86;86R27-3的茎叶有机碳含量显著高于(P < 0.05)MH86, 二者根系有机碳含量差异不显著;86R27-3茎叶的全氮及全磷含量显著低于(P < 0.05)MH86, 其根系的全磷含量也显著低于MH86 (P < 0.05);植株茎叶及根系的全钾含量在86R27-3与MH86间没有明显差异。
| 水稻品种 Rice variety |
植株干重 Dry weight of plant/g |
有机碳 Organic C/% |
全氮 Total N/% |
全磷 Total P/% |
全钾 Total K/% |
单株产量 Yield of plant/g |
|||||||||
| 茎叶 Shoot |
根系 Root |
茎叶 Shoot |
根系 Root |
茎叶 Shoot |
根系 Root |
茎叶 Shoot |
根系 Root |
茎叶 Shoot |
根系 Root |
||||||
| 86R27-3 | 41.5±6.7a | 13.0±3.0b | 72.1±0.72a | 64.4±5.03a | 0.73±0.03b | 0.79±0.04a | 0.22±0.02b | 0.14±0.02b | 1.97±0.18a | 0.67±0.16a | 48.2±7.0a | ||||
| MH86 | 48.5±4.1a | 35.1±4.4a | 67.4±1.96b | 51.8±8.03a | 1.10±0.07a | 0.75±0.07a | 0.30±0.01a | 0.20±0.01a | 2.60±0.40a | 0.90±0.06a | 54.8±9.9a | ||||
| 表中数据为平均值±标准差(重复5次), 同一列数字后字母不同表示差异显著(P < 0.05) | |||||||||||||||
表 3显示了水稻土壤理化性状变化, 其中, 土壤有效氮含量在水稻分蘖期最高, 显著高于(P < 0.05)齐穗期和成熟期;各生育期内86R27-3与MH86的土壤有效氮含量没有明显差异, 但有效磷含量均为前者显著低于(P < 0.05)后者;水稻齐穗期时86R27-3的土壤速效钾含量显著低于(P < 0.05) MH86, 且水稻成熟期时土壤速效钾含量最低, 显著低于水稻分蘖期和齐穗期(P < 0.05);土壤有机碳含量在水稻不同生育期及不同品种间均无显著差异;齐穗期和成熟期的土壤pH值有所降低, 但86R27-3与MH86间没有明显差异。
| 生育期 Growth stage |
水稻品种 Rice variety |
有效氮 Availability N (Alkali-hydrolyzable N)/(mg/kg) |
有效磷 Availability P (Olsen-P)/(mg/kg) |
速效钾 Availability K (NH4OAc-K)/(mg/kg) |
有机质 Organic C/(g/kg) |
pH值 pH (H2O) |
| 分蘖期 | 86R27-3 | 121.8±25.5a | 32.4±4.5bc | 86.0±1.7b | 24.3±1.8a | 5.63±0.06a |
| Tillering stage | MH86 | 124.6±6.4a | 40.6±4.8a | 95.0±1.7ab | 23.4±3.6a | 5.60±0.00a |
| 齐穗期 | 86R27-3 | 88.2±11.1b | 28.7±2.0cd | 87.0±11.7b | 23.1±0.6a | 5.40±0.10bc |
| Heading stage | MH86 | 95.2±8.7ab | 40.3±3.3a | 107.3±15.8a | 23.8±1.9a | 5.23±0.23c |
| 成熟期 | 86R27-3 | 91.0±14.7b | 22.9±1.7d | 62.6±8.1c | 24.8±2.6a | 5.43±0.06b |
| Maturation stage | MH86 | 85.4±25.6b | 35.0±2.4ab | 49.3±4.0c | 24.3±1.0a | 5.40±0.00bc |
| 表中数据为平均值±标准差(重复3次), 同一列数字后字母不同表示差异显著(P < 0.05) | ||||||
由此可见, 与野生型水稻(MH86)比较, 低甲烷排放转基因水稻(86R27-3)根系干物重减少而植株地上部碳累积增加, 且植株磷养分含量及土壤有效磷含量均存在明显降低的趋势。
2.2 稻田土壤微生物群落结构本研究分别制定细菌16S rRNA V3—V4区和真菌ITS V1区的特定引物进行了PCR扩增及高通量测序。其中, 对16S rRNA基因的高通量测序共测得有效序列3 536 692条, 平均每个样品的测序深度为117 889条;真菌ITS基因共测得有效序列条数3 806 392条, 平均每个样品的测序深度为126 879条。
由序列的物种分类注释可见主要有10类菌占细菌总量的95%以上, 分别为变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、放线菌门(Actinobacteria)、酸杆菌门(Acidobacteria)、绿弯菌门(Chloroflexi)、拟杆菌门(Bacteroidetes)、硝化螺菌门(Nitrospirae)、浮霉菌门(Planctomycetes)、蓝藻门(Cyanobacteria)和芽单胞菌门(Gemmatimonadetes), 其中变形菌门、厚壁菌门、放线菌门、酸酐菌门和绿弯菌门依次均占细菌总量的32.7%、15.4%、14.1%、13.2%和9.8%, 是本研究稻田土壤细菌的优势种群。由表 4可见, 水稻各生育期内, 低甲烷转基因水稻(86R27-3)土壤中变形菌门和绿弯菌门的相对丰度与野生型水稻(MH86)没有明显差异;水稻分蘖期和成熟期时, 86R27-3土壤中的厚壁菌门相对丰度显著高于MH86(P < 0.05);水稻齐穗期时, 86R27-3土壤的放线菌门相对丰度显著高于(P < 0.05) MH86, 而其酸杆菌门相对丰度显著低于MH86 (P < 0.05);同时, 比较水稻不同生育期内的变化可见, 转基因与野生型水稻土壤中变形菌门和绿弯菌门相对丰度稳定, 各生育期间没有明显变化;2个品种水稻土壤厚壁菌门及放线菌门的相对丰度在水稻分蘖期、齐穗期与成熟期间依次逐渐降低, 而酸杆菌门的相对丰度在水稻分蘖期、齐穗期和成熟期间依次显著提高(P < 0.05)。由此可见, 在水稻齐穗期时86R27-3与MH86的土壤细菌相对丰度差异较大, 有2个菌门即放线菌门和酸杆菌门的相对丰度差异显著(P < 0.05), 且这2个菌门的相对丰度在水稻生育期间也存在差异。
| 生育期 Growth stage 水稻品种 Rice variety |
分蘖期 Tillering stage | 齐穗期 Heading stage | 成熟期 Maturation stage | ||||||
| 86R27-3 | MH86 | 86R27-3 | MH86 | 86R27-3 | MH86 | ||||
| 菌门相对丰度 | 变形菌门 | 31.2±1.2a | 33.3±5.9a | 32.2±0.9a | 35.2±0.7a | 33.2±0.8a | 34.9±0.4a | ||
| Relative abundance of phylum | 厚壁菌门 | 22.2±3.9a | 17.8±2.8b | 17.4±0.4b | 14.8±1.4bc | 16.6±0.6b | 12.8±0.6c | ||
| 放线菌门 | 21.3±1.2a | 19.1±6.7a | 17.6±0.6a | 11.7±0.9b | 12.5±0.6b | 9.2±0.1b | |||
| 酸酐菌门 | 6.5±1.3d | 6.9±1.4d | 9.4±0.6c | 11.5±1.2b | 13.5±0.8a | 14.8±0.7a | |||
| 绿弯菌门 | 8.9±1.0a | 11.3±3.2a | 10.0±0.7a | 9.0±1.4a | 8.9±1.0a | 11.3±1.3a | |||
| 表中数据为平均值±标准差(重复3次), 同一列数字后字母不同表示差异显著(P < 0.05) | |||||||||
真菌ITS基因测序结果表明稻田土壤中担子菌门(Basidiomycota)相对丰度最高, 均占真菌总量的52.3%, 之后依次为子囊菌门(Ascomycota)、被孢霉门(Mortierellomycota)和罗泽真菌门(Rozellomycota), 均占真菌总量的16.4%、3.1%和1.5%。不同处理比较可见(表 5), 86R27-3土壤中的担子菌门相对丰度在水稻分蘖期显著低于MH86 (P < 0.05);水稻齐穗期时86R27-3土壤子囊菌门的相对丰度显著低于MH86 (P < 0.05);在水稻分蘖期和齐穗期, 86R27-3土壤中的罗泽真菌门相对丰度均显著高于MH86 (P < 0.05);水稻各生育期内土壤被孢霉门相对丰度在86R27-3和MH86间没有明显差异。此外可见, 2个水稻材料分蘖期的土壤子囊菌门相对丰度均低于齐穗期和成熟期, 且被孢霉门相对丰度也在水稻分蘖期、齐穗期及成熟期间依次提高。上述结果表明:水稻成熟期时低甲烷排放转基因水稻(86R27-3)与野生型水稻(MH86)土壤中的主要真菌门相对丰度没有差异, 但在分蘖或齐穗期时二者土壤的担子菌门或罗泽真菌门及子囊菌门的相对丰度分别存在显著差异。
| 生育期 Growth stage 水稻品种 Rice variety |
分蘖期 Tillering stage | 齐穗期 Heading stage | 成熟期 Maturation stage | ||||||
| 86R27-3 | MH86 | 86R27-3 | MH86 | 86R27-3 | MH86 | ||||
| 菌门相对丰度/% | 担子菌门 | 45.1±14.1b | 67.1±2.8a | 54.6±6.3ab | 42.7±17.3b | 50.9±17.2ab | 53.2±5.5ab | ||
| Relative abundance of phylum | 子囊菌门 | 8.5±2.2cd | 4.9±0.1d | 12.8±3.7bc | 39.1±4.5a | 17.2±5.3b | 16.1±1.7b | ||
| 被孢霉门 | 1.4±0.3b | 1.6±0.2b | 2.2±1.5b | 1.9±1.5b | 5.2±0.3a | 6.4±0.8a | |||
| 罗泽真菌门 | 2.9±0.2a | 1.4±0.1bc | 1.6±0.1b | 1.2±0.2c | 1.2±0.3c | 1.1±0.1c | |||
| 表中数据为平均值±标准差(重复3次), 同一列数字后字母不同表示差异显著(P < 0.05) | |||||||||
使用平均丰度前20位属的丰度数据绘制聚类热图(图 1), 实现了对物种丰度分布趋势的展示。由图 1可见, 稻田土壤细菌各菌属的物种组成, 首先在水稻成熟期的86R27-3与MH86水稻品种间聚类, 其次在分蘖期的2个水稻品种间聚类, 水稻齐穗期的MH86、86R27-3依次与成熟期的2个水稻品种聚类, 最后与分蘖期聚类, 说明水稻生育期间的土壤细菌物种组成差异大于其在水稻品种间的差异。对热图中色块颜色及其代表数值的大小趋势的分析发现, 稻田土壤中2个细菌属SJA-15和Aquisphaera的热图色块颜色在86R27-3和MH86间差异较大, 表明其相对丰度在水稻分蘖期或成熟期时86R27-3均低于MH86, 而齐穗期时前者高于后者;水稻各生育期内, 土壤中甲烷氧化菌属Methylocystis的色块颜色在86R27-3和MH86间差异也较大, 且其相对丰度均为前者高于后者;此外, 在水稻齐穗期时, 86R27-3与MH86的土壤细菌属KD4-96、Mycobacterium及IMCC26256的色块颜色差异也表明其相对丰度为前者高于后者。
|
| 图 1 不同品种水稻土壤细菌和真菌群落的物种组成聚类热图 Fig. 1 Cluster heat map of bacterial or fungal communities in paddy soil with different rice variety Rt: 86R27-3分蘖期 Tillering of 86R27-3, Mt: MH86分蘖期 Tillering of MH86, Rh: 86R27-3齐穗期 Heading of 86R27-3, Mh: MH86齐穗期 Heading of MH86, Rm: 86R27-3成熟期 Maturation of 86R27-3, Mm: MH86成熟期 Maturation of MH86 |
由图 1可见, 水稻成熟期时土壤真菌属物种组成在2个水稻品种86R27-3与MH86间首先聚类, 齐穗期时2个水稻品的土壤真菌属物种组成的聚类距离最远, 说明在水稻成熟期86R27-3与MH86的土壤真菌物种组成比较相似, 而在水稻齐穗期二者差异较大。水稻齐穗期时, 土壤真菌Echria、Chaetomella、Saitozyma和Apiotrichum 4个菌属热图色差显示其相对丰度在86R27-3土壤中高于在MH86土壤中;Talaromyces、Acremonium、Paraphaeosphaeria、Hyphopichia、Kodamaea、Pseudeurotium、Fusarium、Penicillium和Lecythophora 9个菌属热图色差显示其相对丰度在MH86土壤中更高。
由此可见, 无论是土壤细菌还是土壤真菌, 其主要菌属组成在低甲烷排放转基因水稻(86R27-3)与野生型水稻(MH86)间的差异均在水稻齐穗期表现最为突出。
2.3 稻田土壤微生物群落多样性依据基因序列计算的稻田土壤细菌和真菌群落的α-多样性指数如表 6所示。由表 6可见, 2个水稻材料稻田土壤细菌群落的物种丰富度指数Chao1、Observed_species在水稻分蘖期、齐穗期和成熟期间均存在逐渐升高的趋势, 其覆盖度指数Goods_coverage则逐渐降低, 前者在水稻成熟期时显著高于(P < 0.05)分蘖期, 而后者在水稻成熟期时显著低于(P < 0.05)分蘖期;在水稻各生育期内土壤细菌群落的α-多样性指数在86R27-3与MH86间均无显著性差异。
| 生育期 Growth stage |
水稻品种 Rice variety |
丰富度指数 Chao1 |
丰富度指数 Observed_species |
Shannon多样性指数 Shannon |
Simpson多样性指数 Simpson |
均匀度指数 Pielou_e |
覆盖度指数 Goods_coverage |
| 分蘖期 | 86R27-3 | 6254±2315b | 5222±1854bc | 10.48±1.15a | 0.9969±0.0031a | 0.8517±0.0537a | 0.9704±0.0137ab |
| Tillering stage | MH86 | 6029±2166b | 5053±1527c | 10.16±1.35a | 0.9927±0.0097a | 0.8270±0.0822a | 0.9722±0.0135a |
| 齐穗期 | 86R27-3 | 8056±186ab | 6490±58abc | 11.22±0.03a | 0.9987±0.0000a | 0.8859±0.0011a | 0.9592±0.0012abc |
| Heading stage | MH86 | 8425±351ab | 6910±127ab | 11.42±0.05a | 0.9989±0.0001a | 0.8951±0.0023a | 0.9570±0.0029bc |
| 成熟期 | 86R27-3 | 8800±219a | 7082±84a | 11.21±0.03a | 0.9979±0.0001a | 0.8767±0.0015a | 0.9525±0.001c |
| Maturation stage | MH86 | 8739±189a | 6821±137ab | 11.31±0.04a | 0.9987±0.0001a | 0.8884±0.0014a | 0.9533±0.0011c |
| 表中数据为平均值±标准差(重复3次), 同一列数字后字母不同表示差异显著(P < 0.05) | |||||||
由稻田土壤真菌群落的α-多样性指数可见(表 7), 在水稻分蘖期, 86R27-3的土壤真菌群落多样性指数Shannon、Simpson及均匀度指数Pielou_e均显著高于MH86 (P < 0.05), 而水稻齐穗期和成熟期时Shannon、Simpson及Pielou_e在86R27-3与MH86间的差异不再显著;真菌群落的物种丰富度指数Chao1、Observed_species及覆盖度指数Goods_coverage在各个样品间均没有明显差异。
| 生育期 Growth stage |
水稻品种 Rice variety |
丰富度指数 Chao1 |
丰富度指数 Observed_species |
Shannon多样性指数 Shannon |
Simpson多样性指数 Simpson |
均匀度指数 Pielou_e |
覆盖度指数 Goods_coverage |
| 分蘖期 | 86R27-3 | 575±139a | 573±138a | 5.05±1.02a | 0.8192±0.1278a | 0.5564±0.1378a | 0.9999±0.0001a |
| Tillering stage | MH86 | 575±14a | 572±15a | 3.58±0.20b | 0.6064±0.0425b | 0.3907±0.0216b | 0.9999±0.0000a |
| 齐穗期 | 86R27-3 | 604±59a | 603±59a | 4.62±0.30ab | 0.7548±0.0579ab | 0.5008±0.0303ab | 0.9999±0.0000a |
| Heading stage | MH86 | 839±335a | 837±335a | 5.02±1.04a | 0.8154±0.1297a | 0.5191±0.0742ab | 0.9999±0.0001a |
| 成熟期 | 86R27-3 | 723±326a | 719±324a | 4.54±1.07ab | 0.7533±0.1362ab | 0.4798±0.0803ab | 0.9998±0.0001a |
| Maturation stage | MH86 | 617±105a | 614±104a | 4.64±0.41ab | 0.7705±0.0475ab | 0.5011±0.0309ab | 0.9999±0.0001a |
| 表中数据为平均值±标准差(重复3次), 同一列数字后字母不同表示差异显著(P < 0.05) | |||||||
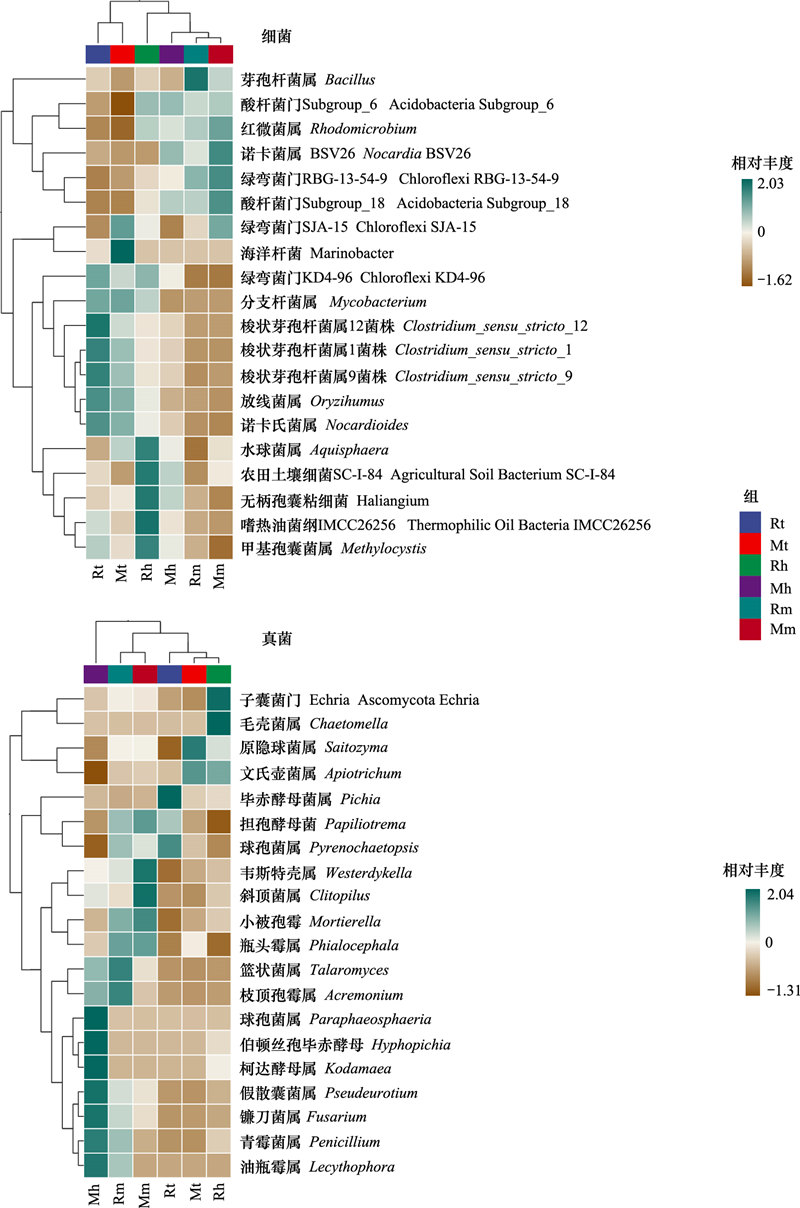
图 2为稻田土壤细菌及真菌群落β-多样性的主坐标分析。由图 2可见, 2个水稻品种的不同生育期的土壤细菌群落组成散点均在主坐标1(PCo1)上具有一定投影距离, 其中成熟期与分蘖期间的距离较大, 说明土壤细菌群落组成在水稻成熟期与分蘖期间存在一定差异;水稻分蘖期或成熟期时, 86R27-3与MH86的细菌群落组成散点在主坐标1(PCo1)或主坐标2(PCo2)上的投影没有明显距离;齐穗期时86R27-3与MH86在PCo1坐标轴上投影的距离增大, 表明此时期二者土壤细菌群落组成的差异较大一些。但各个样品组间差异的PERMANOVA, Anosim及Permdisp显著性检的P值均大于0.05 (表 8), 表明细菌群落组成在各个样品间均没有显著性差异。
|
| 图 2 不同品种水稻土壤细菌和真菌群落的主坐标分析 Fig. 2 Principal co-ordinates analysis for beta diversity of bacterial or fungal communities in paddy soil with different rice variety Rt: 86R27-3分蘖期 Tillering of 86R27-3, Mt: MH86分蘖期 Tillering of MH86, Rh: 86R27-3齐穗期 Heading of 86R27-3, Mh: MH86齐穗期 Heading of MH86, Rm: 86R27-3成熟期 Maturation of 86R27-3, Mm: MH86成熟期 Maturation of MH86 |
| 组1 Group1 |
组2 Group2 |
样本数 Sample size |
置换检验数 Permutation |
置换多元方差分析 Permanova |
相似性分析 Anosim |
置换多元离散度 Permdisp |
|||||
| F | P | F | P | F | P | ||||||
| Rt | Mt | 6 | 999 | 0.837 | 0.806 | 0.037 | 0.492 | 0.127 | 0.452 | ||
| Rt | Rh | 6 | 999 | 1.699 | 0.091 | 0.556 | 0.111 | 0.893 | 0.715 | ||
| Rt | Mh | 6 | 999 | 2.012 | 0.102 | 0.556 | 0.111 | 0.465 | 0.878 | ||
| Rt | Rm | 6 | 999 | 3.074 | 0.101 | 0.556 | 0.126 | 0.736 | 0.435 | ||
| Rt | Mm | 6 | 999 | 3.237 | 0.108 | 0.556 | 0.108 | 1.017 | 0.065 | ||
| Mt | Rh | 6 | 999 | 1.433 | 0.090 | 0.370 | 0.105 | 5.467 | 0.410 | ||
| Mt | Mh | 6 | 999 | 1.884 | 0.103 | 0.444 | 0.099 | 3.443 | 0.381 | ||
| Mt | Rm | 6 | 999 | 2.691 | 0.107 | 0.556 | 0.101 | 4.791 | 0.100 | ||
| Mt | Mm | 6 | 999 | 2.727 | 0.088 | 0.556 | 0.083 | 6.013 | 0.111 | ||
| Rh | Mh | 6 | 999 | 1.900 | 0.107 | 1.000 | 0.112 | 2.798 | 0.054 | ||
| Rh | Rm | 6 | 999 | 3.599 | 0.100 | 1.000 | 0.104 | 0.812 | 0.102 | ||
| Rh | Mm | 6 | 999 | 3.435 | 0.085 | 1.000 | 0.098 | 0.476 | 0.091 | ||
| Mh | Rm | 6 | 999 | 2.488 | 0.117 | 1.000 | 0.103 | 1.244 | 0.098 | ||
| Mh | Mm | 6 | 999 | 2.375 | 0.080 | 1.000 | 0.104 | 4.632 | 0.092 | ||
| Rm | Mm | 6 | 999 | 1.763 | 0.110 | 1.000 | 0.095 | 2.877 | 0.062 | ||
| Rt: 86R27-3分蘖期 Tillering of 86R27-3, Mt: MH86分蘖期 Tillering of MH86, Rh: 86R27-3齐穗期 Heading of 86R27-3, Mh: MH86齐穗期 Heading of MH86, Rm: 86R27-3成熟期 Maturation of 86R27-3, Mm: MH86成熟期 Maturation of MH86. Permanova:置换多元方差分析 Permutational multivariate analysis of variance;Anosim:相似性分析 Analysis of similarities;Permdisp:置换多元离散度 Permutation multivariated ispersion | |||||||||||
由图 2可见, 除个别重复外, 各个样品的土壤真菌群落组成散点在2个标轴上的投影相距都比较近, 组间差异显著性分析结果也表明真菌群落组成在各个样品间的差异也都没有达到显著性水平(表 9)。
| 组1 Group1 |
组2 Group2 |
样本数 Sample size |
置换检验数 Permutation |
置换多元方差分析 Permanova |
相似性分析 Anosim |
置换多元离散度 Permdisp |
|||||
| F | P | F | P | F | P | ||||||
| Rt | Mt | 6 | 999 | 2.002 | 0.095 | 0.556 | 0.103 | 1.315 | 0.296 | ||
| Rt | Rh | 6 | 999 | 1.724 | 0.099 | 0.519 | 0.102 | 0.788 | 0.903 | ||
| Rt | Mh | 6 | 999 | 2.151 | 0.097 | 0.259 | 0.278 | 0.138 | 1.000 | ||
| Rt | Rm | 6 | 999 | 2.423 | 0.097 | 0.259 | 0.092 | 0.324 | 0.483 | ||
| Rt | Mm | 6 | 999 | 2.644 | 0.103 | 0.556 | 0.077 | 1.114 | 0.243 | ||
| Mt | Rh | 6 | 999 | 3.761 | 0.101 | 0.667 | 0.104 | 2.335 | 0.084 | ||
| Mt | Mh | 6 | 999 | 4.063 | 0.107 | 0.574 | 0.096 | 0.724 | 0.116 | ||
| Mt | Rm | 6 | 999 | 4.279 | 0.109 | 0.556 | 0.099 | 0.642 | 0.645 | ||
| Mt | Mm | 6 | 999 | 10.149 | 0.090 | 1.000 | 0.098 | 0.324 | 0.104 | ||
| Rh | Mh | 6 | 999 | 3.011 | 0.199 | 0.333 | 0.339 | 0.276 | 0.058 | ||
| Rh | Rm | 6 | 999 | 3.554 | 0.117 | 0.556 | 0.112 | 0.141 | 1.000 | ||
| Rh | Mm | 6 | 999 | 5.668 | 0.104 | 1.000 | 0.103 | 0.889 | 0.086 | ||
| Mh | Rm | 6 | 999 | 1.571 | 0.205 | 0.111 | 0.216 | 0.037 | 0.471 | ||
| Mh | Mm | 6 | 999 | 3.021 | 0.218 | 0.333 | 0.202 | 0.541 | 0.111 | ||
| Rm | Mm | 6 | 999 | 1.821 | 0.105 | 0.407 | 0.097 | 0.423 | 0.898 | ||
| Rt: 86R27-3分蘖期 Tillering of 86R27-3; Mt: MH86分蘖期 Tillering of MH86; Rh: 86R27-3齐穗期 Heading of 86R27-3; Mh: MH86齐穗期 Heading of MH86; Rm: 86R27-3成熟期 Maturation of 86R27-3; Mm: MH86成熟期 Maturation of MH86 | |||||||||||
由上述微生物群落α及β多样性可见, 除分蘖期的土壤真菌外, 低甲烷排放转基因水稻(86R27-3)与野生型水稻(MH86)的土壤细菌或真菌的群落多样性不存在显著差异, 且群落组成也无显著差异。
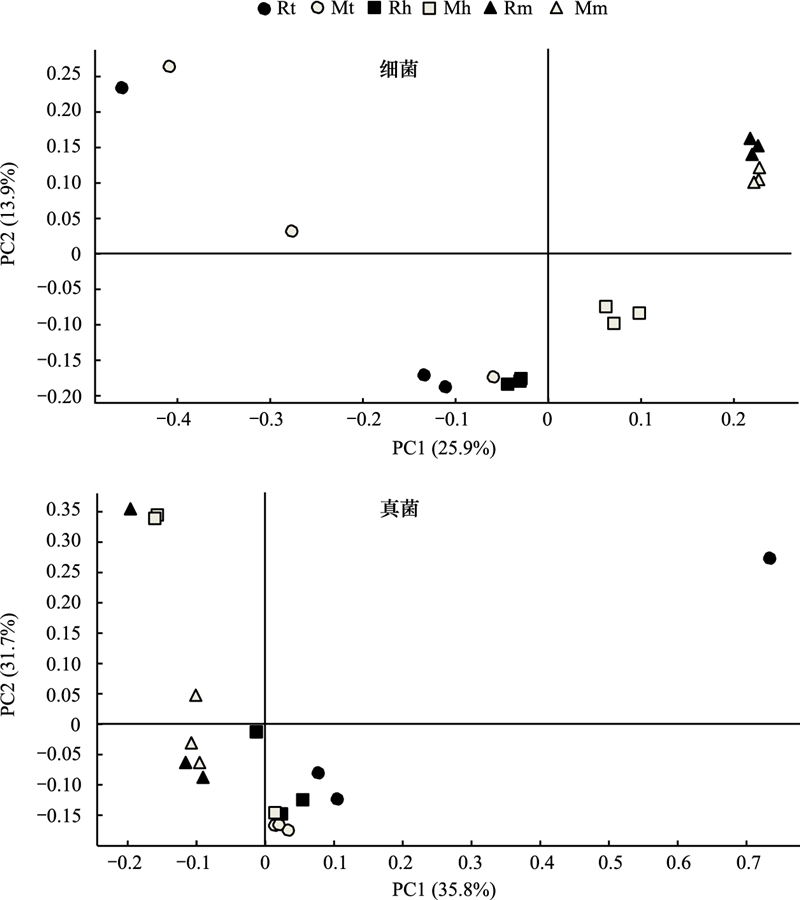
2.4 稻田土壤微生物功能群用PICRUSt2 (2.2.0)软件对土壤细菌和真菌的群落功能预测结果如图 3所示。可见, 在水稻分蘖期、齐穗期或成熟期时, 除个别重复外, 86R27-3与MH86的土壤细菌或真菌群落功能单元散点在主坐标1 (PCo1)或主坐标2 (PCo2)上的投影均比较接近, 表明86R27-3与MH86的土壤细菌或真菌群落功能组成比较相似。此外, 比较每个水稻不同生育期时可见, 86R27-3和MH86的土壤细菌或真菌群落功能单元散点的分布均有一定距离, 说明土壤细菌或真菌群落功能组成在水稻不同生育期间存在一些差异。
|
| 图 3 不同品种水稻土壤细菌和真菌群落功能单元主坐标分析 Fig. 3 Principal co-ordinates analysis for functional unit of bacterial or fungal communities in paddy soil with different rice variety Rt: 86R27-3分蘖期 Tiller of 86R27-3, Mt: MH86分蘖期 Tiller of MH86, Rh: 86R27-3齐穗期 Heading of 86R27-3, Mh: MH86齐穗期 Heading of MH86, Rm: 86R27-3成熟期 Mature of 86R27-3, Mm: MH86成熟期 Mature of MH86 |
进一步使用FAPROTAX (1.1)和FUNGuild (1.1a)数据库分别对细菌及真菌功能预测的结果如表 10、11所示。水稻分蘖期时, 预测到86R27-3稻田土壤中与硝酸盐还原、呼吸(Nitrate reduction、Nitrate respiration)、富马酸盐、硫代硫酸盐及硫呼吸(Fumarate respiration、Thiosulfate respiration、Sulfur respiration)相关的细菌功能群丰度均显著低于MH86 (P < 0.05);86R27-3齐穗期和成熟期的土壤好氧亚硝酸盐氧化(Aerobic nitrite oxidation)细菌、及其成熟期的锰氧化(Manganese oxidation)细菌功能群丰度也均显著低于MH86 (P < 0.05)。此外, 预测到稻田土壤化学异养(Chemoheterotrophy)和芳香族化合物降解(Aromatic_compound_degradation)的细菌功能群丰度在水稻分蘖期、齐穗期及成熟期间依次显著降低(P < 0.05), 水稻分蘖期土壤发酵(Fermentation)和固氮作用(Nitrogen fixation)细菌功能群丰度显著高于齐穗期和成熟期(P < 0.05), 而土壤铁呼吸(Iron respiration)及硫酸盐呼吸(Sulfate respiration)细菌功能群丰度在水稻齐穗期最高, 显著高于分蘖期和成熟期(P < 0.05)。由此可见, 与野生型水稻MH86比较, 低甲烷排放转基因水稻86R27-3土壤中一些参与氮、硫和锰代谢过程的细菌功能群丰度显著降低。
| 生育期 Growth stage 水稻品种 Rice variety |
分蘖期 Tillering stae | 齐穗期 Heading stage | 成熟期 Maturation stage | |||||
| 86R27-3 | MH86 | 86R27-3 | MH86 | 86R27-3 | MH86 | |||
| 功能组 Functional group | ||||||||
| 化学异养 Chemoheterotrophy | 17007±8354a | 23109±9038a | 9646±602b | 7268±1265b | 6053±445c | 5552±269c | ||
| 好氧化学异养 Aerobic_chemoheterotrophy | 13322±9403ab | 18381±6015a | 6995±557b | 4632±963b | 4107±328b | 3770±218b | ||
| 发酵 Fermentation | 2817±942a | 3935±863a | 1668±164b | 1664±229b | 1119±134b | 1043±100b | ||
| 食肉的或外寄生的 Predatory_or_exoparasitic | 1011±227a | 966±499a | 1138±127a | 968±226a | 845±44a | 845±106a | ||
| 甲基营养 Methylotrophy | 859±269a | 805±256a | 974±69a | 943±158a | 809±35a | 729±83a | ||
| 碳氢化合物降解 Hydrocarbon_degradation | 897±145ab | 827±239ab | 963±76a | 897±139ab | 759±47ab | 689±70b | ||
| 含硫化合物呼吸作用Respiration_of_sulfur_compounds | 665±182a | 1159±685a | 742±114a | 973±178a | 588±31a | 664±46a | ||
| 甲烷营养 Methanotrophy | 801±233a | 725±331a | 928±67a | 861±140a | 739±41a | 673±70a | ||
| 芳香族化合物降解Aromatic_compound_degradation | 1670±414a | 1258±259a | 740±52b | 486±126b | 257±27c | 196±19c | ||
| 固氮作用 Nitrogen fixation | 1668±813a | 895±233a | 553±38b | 420±135b | 361±16b | 395±27b | ||
| 铁呼吸 Iron respiration | 350±151b | 472±357b | 841±151a | 884±115a | 462±18b | 522±44b | ||
| 硫酸盐呼吸 Sulfate respiration | 384±203cd | 292±205d | 652±103ab | 895±174a | 539±41bcd | 615±40bc | ||
| 纤维素分解 Cellulolysis | 434±119ab | 391±201ab | 553±25a | 379±82ab | 301±45b | 298±35b | ||
| 光能自养 Photoautotrophy | 556±398a | 423±162a | 326±48a | 271±80a | 283±45a | 312±35a | ||
| 硝酸盐还原 Nitrate reduction | 287±48b | 1111±180a | 114±9c | 168±62c | 67±28d | 51±16d | ||
| 硝酸盐呼吸 Nitrate respiration | 223±83b | 1024±166a | 74±12c | 82±23c | 38±21d | 23±16d | ||
| 富马酸盐呼吸 Fumarate respiration | 130±21b | 972±164a | 49±8c | 39±13c | 25±6d | 23±7d | ||
| 硫代硫酸盐呼吸 Thiosulfate respiration | 192±25b | 835±138a | 60±10c | 41±13cd | 32±8d | 40±4d | ||
| 硫呼吸 Sulfur respiration | 230±36b | 833±137a | 37±7cd | 46±13c | 28±15cd | 20±13d | ||
| 好氧亚硝酸盐氧化 Aerobic nitrite oxidation | 71±38c | 71±42c | 125±24c | 290±49a | 215±22b | 302±46a | ||
| 暗氢氧化 Dark hydrogen oxidation | 200±103a | 149±75ab | 86±17bc | 67±21bc | 49±16c | 40±11c | ||
| 还原产乙酸 Reductive acetogenesis | 156±133a | 126±57ab | 84±9ab | 62±19ab | 42±14b | 40±12b | ||
| 木聚糖分解 Xylanolysis | 103±49a | 125±26a | 79±25ab | 77±41ab | 35±24b | 37±29b | ||
| 锰氧化 Manganese oxidation | 132±60a | 72±22ab | 88±4ab | 43±9bc | 39±9c | 60±8b | ||
| 表中数据为平均值±标准差(重复3次), 同一行数字后字母不同表示差异显著(P < 0.05) | ||||||||
| 生育期 Growth stage 水稻品种 Rice variety |
分蘖期Tillering stage | 齐穗期Heading stage | 成熟期Maturation stage | ||||||
| 86R27-3 | MH86 | 86R27-3 | MH86 | 86R27-3 | MH86 | ||||
| 功能群 Guild | 分类单元 Taxon | ||||||||
| 真菌寄生虫-未定义腐生物 Fungal Parasite-Undefined Saprotroph |
银耳目 Tremellales | 41184±19809b | 78171±4602a | 57654±6823ab | 45602±20801ab | 59550±22525ab | 57536±3293ab | ||
| 未定义腐生物 Undefined Saprotroph | 嗜热囊菌科 Pseudeurotium |
194±78d | 140±13d | 806±295c | 9764±2461a | 4422±1438b | 2258±1027b | ||
| 内生植物 Endophyte | 瓶霉属 Phialocephala | 1137±957b | 1690±99ab | 977±113b | 1392±589b | 2482±500a | 2422±106a | ||
| 动物内共生体腐生生物 Animal Endosymbiont-Undefined Saprotroph |
毕赤酵母属 Pichia | 5011±1766a | 701±335b | 676±249b | 245±189b | 233±172b | 407±112b | ||
| 内生菌-地衣寄生虫-未定义腐生物 Endophyte-Lichen Parasite-Undefined Saprotroph |
薄孢核菌属 Pyrenochaetopsis |
1069±956a | 459±118ab | 259±70b | 146±25b | 521±183ab | 390±83ab | ||
| 动物病原体-内生真菌-地衣寄生虫-植物病原体-土壤腐生物-木材腐生物 Animal Pathogen-Endophyte-Lichen Parasite-Plant Pathogen-Soil Saprotroph-Wood Saprotroph |
镰刀菌属 Fusarium |
60±22d | 67±17d | 113±28c | 1009±392a | 530±144ab | 300±108b | ||
| 未定义腐生物 Undefined Saprotroph | 韦斯特氏菌 Westerdykella |
217±185b | 326±10ab | 271±26b | 262±158b | 285±25b | 493±105a | ||
| 未定义腐生物 Undefined Saprotroph | 马勃科 Lasiosphaeriaceae |
376±177a | 108±18b | 285±52ab | 79±47b | 293±54ab | 280±35ab | ||
| 外生菌根未定义腐生菌 Ectomycorrhizal-Undefined Saprotroph |
革菌科 Thelephoraceae |
132±69bc | 55±22c | 104±20bc | 174±90b | 306±65a | 304±18a | ||
| 表中数据为平均值±标准差(重复3次), 同一行数字后字母不同表示差异显著(P < 0.05) | |||||||||
由表 11可见, 86R27-3稻田土壤中某些真菌功能分类单元的预测丰度显著低于MH86 (P < 0.05), 如分蘖期的未定义腐生物银耳目(Tremellales)、齐穗期的未定义腐生物嗜热囊菌科(Pseudeurotium)和镰刀菌属(Fusarium)及成熟期的未定义腐生物韦斯特氏菌(Westerdykella);而分蘖期的动物内共生体腐生生物毕赤酵母属(Pichia)和未定义腐生物马勃科(Lasiosphaeriaceae)的预测丰度则是86R27-3显著高于MH86 (P < 0.05)。此外, 2个品种水稻土壤中嗜热囊菌科(Pseudeurotium)和镰刀菌属(Fusarium)功能群预测丰度为水稻分蘖期显著低于齐穗和成熟期(P < 0.05), 而成熟期的外生菌根未定义腐生菌革菌科(Thelephoraceae)功能群丰度显著高于分蘖期和齐穗期(P < 0.05)。由此可见, 稻田土壤主要真菌功能类群丰度在水稻品种及生育期间也均存在差异。
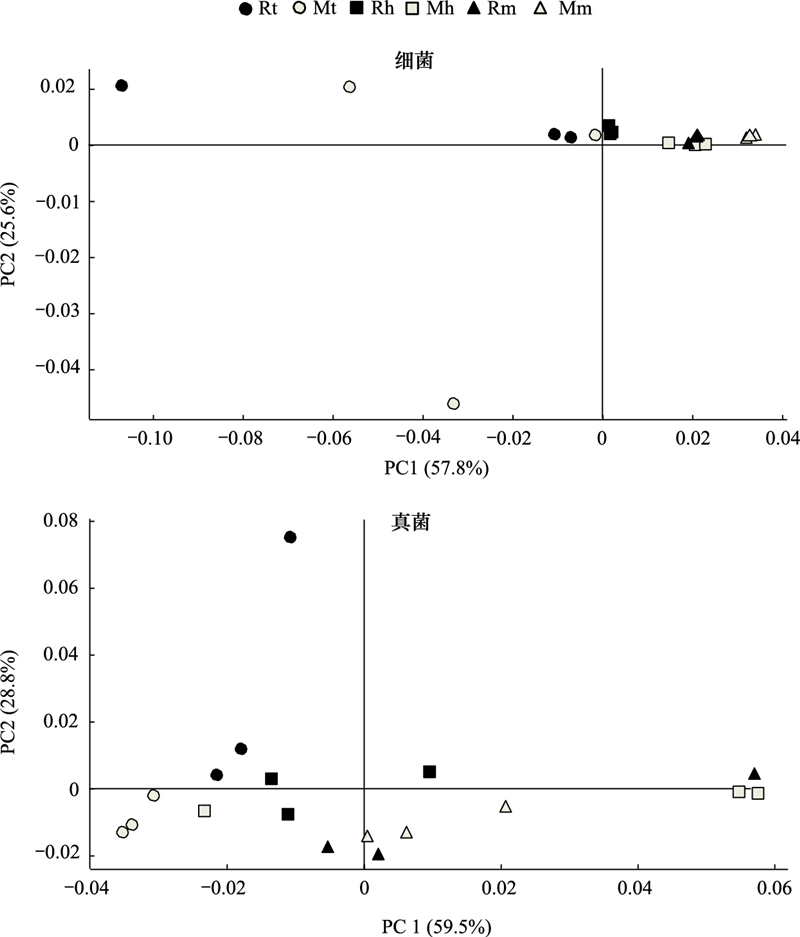
2.5 稻田土壤微生物功能基因丰度利用荧光定量PCR技术分析了稻田土壤微生物功能基因丰度, 如图 4所示。结果显示:水稻齐穗期和成熟期时86R27-3的土壤固氮菌nifH基因及反硝化细菌nirS基因的丰度均显著低于MH86 (P < 0.05), 分蘖期和成熟期时86R27-3的土壤氨氧化细菌amoA基因丰度也显著低于MH86 (P < 0.05), 水稻各个生育期内86R27-3的土壤产甲烷菌mcrA基因丰度均显著低于MH86 (P < 0.05)。可见, 在低甲烷转基因水稻86R27-3的稻田土壤中, 不仅产甲烷菌功能基因mcrA的丰度会显著降低, 与土壤固氮和氮素转化相关的微生物功能基因也存在显著降低的情况。
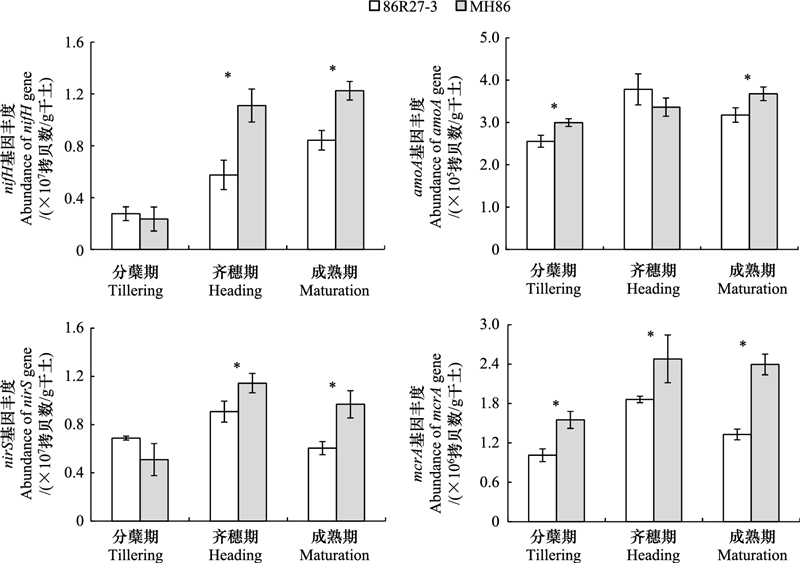
|
| 图 4 不同品种水稻土壤微生物功能基因丰度 Fig. 4 Abundance of microbial functional genes in paddy soil with different rice variety A: 固氮菌nifH基因 Nitrogen-fixing bacterial nifH gene, B: 氨氧化细菌amoA基因 Ammonia-oxidizing bacterial amoA gene, C: 反硝化细菌nirS基因 Denitrifying bacterial nirS gene, D: 产甲烷菌mcrA基因 Methanogenic bacterial mcrA gene; *代表显著差异(P < 0.05) Significantly different (P < 0.05) |
稻田是甲烷的主要农业排放源, 水稻根系分泌物及脱落物在土壤微生物的作用下可转变成为CO2、H2、乙酸盐等简单分子, 在稻田淹水的厌氧条件下, 产甲烷菌利用这些小分子底物进行无氧呼吸产生甲烷, 所产生的甲烷大部分又会被土壤中的甲烷氧化菌氧化消耗, 未被氧化的甲烷主要通过水稻植株的通气组织排向大气[22—25]。降低稻田甲烷释放己成为全世界关注的热点和难点, 以改变水稻品种特性为切入点是从源头上减少甲烷产生和释放的经济有效的方法[26]。
水稻光合碳的产生和分配影响甲烷的排放, 植物“源、库、流”调节光合产物的产生与分配[27]。“源”、“流”强弱及产量“库”容大小会影响到光合产物产生、运输及分配到根系的多少[28], 若产量即“库”容小, 就会有更多的光合产物分配到水稻根部, 进一步分泌到土壤中增加产生甲烷的原始底物, 促进甲烷的产生与释放[2]。试验摘除不同比例的水稻稻穗发现, 摘除稻穗的比例越大水稻甲烷排放量越高, 稻穗完全摘除的水稻甲烷排放量是不摘除稻穗水稻的1.5倍[29]。所以, 可通过调节提高“库”的光合碳储存能力, 促使光合碳向稻穗分配, 减少根系方向的碳分配来减少甲烷的产生。
3.2 低甲烷排放转基因水稻本研究的低甲烷排放转基因水稻利用大麦(Hordeum vulgare L.)转录因子Hvsusiba2基因调节水稻光合产物分配的特性, 将Hvsusiba2基因转化水稻后, 转基因水稻种子中与淀粉合成相关的一些基因表达显著上调[2, 4], 其成熟种子中总淀粉含量显著提高, 同时减少了碳向地下部的运输, 造成水稻根系生物量显著降低、土壤中产甲烷菌丰度也显著降低, 从而降低了稻田甲烷排放量[4—6]。
现有研究结果表明, 低甲烷排放转基因水稻主要是通过降低土壤产甲烷菌丰度实现甲烷减排。那么, 产甲烷菌丰度降低的同时, 稻田土壤中其它微生物的群落组成及功能是否发生变化、从而影响稻田土壤微生物生态系统稳定与可持续性, 是需要深入分析探讨的问题。
而且, 分别以粳稻“日本晴”和籼稻“明恢86”(MH86)为背景的转基因水稻的甲烷减排率差异较大, 前者最高可达到50%、后者30%[7]。同时研究发现粳稻“日本晴”与籼稻“明恢86”稻田土壤中的产甲烷菌群落组成存在差异, 这可能是造成转基因粳稻与籼稻的甲烷减排效果差异的原因之一[7], 由此也体现出了土壤微生物群落组成及功能的重要性。此外, 我们之前的研究也发现低甲烷排放转基因粳稻的土壤细菌群落结构与野生型水稻“日本晴”存在一定差异, 且土壤中与碳循环相关的某些微生物功能基因强度也明显降低[30]。
3.3 低甲烷排转基因水稻的土壤微生物群落微生物参与了土壤养分转化、有机质分解、腐殖质形成及污染物生物降解等土壤生化过程, 其群落组成是反映土壤环境状况、衡量土壤质量和维持土壤微生态平衡的重要指标[31—32]。不同品种植物的根系分泌物组成及数量存在差异, 由此导致了土壤微生物群落组成的差异[33]。转基因作物的外源基因有可能改变其根系分泌物、根系及植株残体的组成与数量, 从而影响土壤微生物群落组成与功能[34—35]。
本研究进一步分析了籼稻背景的低甲烷排放转基因水稻(86R27-3)与野生型籼稻“明恢86”(MH86)间稻田土壤微生物群落组成与功能的差异。通过对田间定位试验第3年的土壤细菌和真菌的高通量测序发现, 微生物群落组成的α-及β-多样性分析表明, 仅在水稻分蘖期低甲烷排放转基因水稻(86R27-3)土壤真菌群落的多样性指数Shannon、Simpson及均匀度指数Pielou_e显著高于(P < 0.05)野生型水稻(MH86), 其它生育期内的稻田土壤细菌及真菌群落组成及其多样性在86R27-3与MH86间均没有明显差异。但是, 对微生物物种组成的具体分析显示, 在某些水稻生长阶段, 一些种类的土壤细菌或真菌的相对丰度在86R27-3与MH86间存在差异。如, 细菌的厚壁菌门相对丰度在86R27-3的分蘖期或成熟期的土壤中显著提高(P < 0.05), 在齐穗期的86R27-3土壤中细菌的放线菌门相对丰度显著提高(P < 0.05), 但其酸杆菌门相对丰度显著降低(P < 0.05);真菌的担子菌门或子囊菌门的相对丰度分别在86R27-3的分蘖期或齐穗期的土壤中显著降低(P < 0.05), 但罗泽真菌门相对丰度在分蘖期和齐穗期的86R27-3土壤中均显著提高(P < 0.05)。
虽然, 某些细菌门或真菌门的相对丰度变化未能造成微生物整体群落组成及多样性的显著改变, 但对于功能更加特定的一些细菌或真菌的菌属来说, 其丰度在86R27-3与MH86间的差异仍是十分值得关注的。从物种组成热图的分析可见, 在水稻齐穗期, 稻田土壤细菌或真菌中平均丰度较高的一些菌属的相对丰度在86R27-3与MH86间存在较明显的差异, 且真菌有差异的菌属数量比细菌多;真菌的Echria等4个菌属及Talaromyces等9个菌属的相对丰度分别在86R27-3土壤中提高或降低;细菌有5个菌属SJA-15、Aquisphaera、KD4-96、IMCC26256及甲烷氧化菌属Mycobacterium的相对丰度在86R27-3土壤中提高。这些相对丰度有明显变化的细菌属或真菌属的功能的进一步分析挖掘, 对评价低甲烷排放转基因水稻土壤微生物生态功能可能更有效。如, 甲烷氧化菌属Mycobacterium的相对丰度在86R27-3土壤中的提高, 对土壤中甲烷的氧化消耗有促进作用, 从而应对稻田甲烷减排具有一定贡献。
同时, 本研究对稻田土壤细菌或真菌群落功能预测的结果显示, 低甲烷排放转基因水稻(86R27-3)土壤细菌或真菌功能群的丰度与野生型水稻(MH86)可存在显著差异。尤其是土壤细菌, 明显可见与土壤氮、硫及锰代谢相关的细菌功能群丰度在86R27-3与MH86间存在显著差异(P < 0.05), 如, 水稻分蘖期的硝酸盐还原、硝酸盐呼吸、硫代硫酸盐呼吸及硫呼吸, 水稻齐穗期和成熟期的好氧亚硝酸氧化及成熟期的锰氧化细菌功能群丰度均在86R27-3土壤中显著降低(P < 0.05)。土壤真菌中主要是一些未定义腐生物类型功能群丰度在86R27-3与MH86间存在差异, 其中, 86R27-3土壤中的银耳目、嗜热囊菌科、镰刀菌属及韦斯特氏菌功能群丰度显著降低(P < 0.05), 而其马勃科及动物内共生体腐生生物毕赤酵母属功能群丰度显著提高(P < 0.05)。说明种植低甲烷排放转基因水稻(86R27-3)对稻田土壤微生物功能群丰度存在一定影响。
另外, 本研究通过对土壤微生物功能基因的定量分析发现, 与野生型水稻(MH86)比较而言, 在低甲烷排放转基因水稻(86R27-3)土壤中, 不仅产甲烷菌功能基因mcrA丰度显著降低(P < 0.05), 其它微生物功能基因丰度也会显著降低, 如固氮菌nifH基因、氨氧化细菌amoA基因和反硝化细菌nirS基因的丰度均在不同的水稻生育期内显著降低(P < 0.05)。这些功能基因丰度的变化可能影响到稻田土壤固氮及氮素转化过程。
此外, 通过对水稻植株分析, 证明了低甲烷排放转基因水稻(86R27-3)促进碳水化合物向茎叶及籽粒运送, 减少向根系分配[4], 其根系干重显著低于野生型水稻(MH86), 但茎叶干重及单株产量没有差异, 且86R27-3的茎叶有机碳含量较高。由稻田土壤有效养分含量可见, 86R27-3的土壤有效磷含量显著低于MH86 (P < 0.05), 影响了对水稻磷营养的供给, 86R27-3的植株茎叶及根系中的磷含量也均显著降低(P < 0.05)。
综上所述, 由于低甲烷排放转基因水稻(86R27-3)根系减少, 向土壤中输送的有机物也会相应减少, 从而减少了土壤微生物生长所需的碳源, 致使产甲烷菌丰度降低的同时也降低了土壤固氮菌、氨氧化细菌及反硝化细菌功能基因丰度, 这些功能基因丰度的变化将对稻田土壤固氮及氮素转化具有潜在影响。与野生型水稻(MH86)比较, 低甲烷排放转基因水稻(86R27-3)稻田土壤微生物群落组成总体上没有显著变化, 但某些细菌或真菌的菌门或菌属的相对丰度产生变化, 且预测可见低甲烷排放转基因水稻土壤中与氮、硫及锰代谢相关的一些细菌功能群丰度的降低, 及某些未定义腐生物真菌功能群丰度的变化, 这仍是十分值得关注与深入研究的问题。此外, 本试验显示出的低甲烷排放转基因水稻(86R27-3)土壤磷养分有效性的降低, 及其植株茎叶和根系的磷养分含量的降低, 是否与土壤中影响磷素转化与吸收的微生物群落组成与丰度相关, 也有待进一步分析研究。
4 结论本试验研究结果表明:连续种植低甲烷排放转基因水稻(86R27-3)3年后, 对稻田土壤细菌或真菌的群落组成没有显著影响;但主要细菌或真菌种类的相对丰度及功能群丰度产生了变化。与野生型水稻MH86相比, 水稻齐穗期时86R27-3土壤的放线菌门(Actinobacteria)、罗泽真菌门(Rozellomycota)的相对丰度显著提高(P < 0.05), 而酸杆菌门(Acidibacteria)、子囊菌门(Ascomycota)的相对丰度显著降低(P < 0.05);参与土壤氮、硫和锰代谢的硝酸盐还原与呼吸, 硫代硫酸盐及硫呼吸, 好氧亚硝酸盐氧化及锰氧化的细菌功能群丰度在某些生长阶段的86R27-3土壤中显著降低(P < 0.05);而未定义腐生物类型的真菌功能群丰度在86R27-3土壤中有减有增, 前者如银耳目、嗜热囊菌科、镰刀菌属及韦斯特氏菌, 后者如马勃科;且微生物功能基因mcrA、nifH、amoA及nirS的丰度在86R27-3土壤中也显著降低(P < 0.05)。
| [1] |
Crippa M, Solazzo E, Guizzardi D, Monforti-Ferrario F, Tubiello F N, Leip A. Food systems are responsible for a third of global anthropogenic GHG emissions. Nature Food, 2021, 2: 198-209. DOI:10.1038/s43016-021-00225-9 |
| [2] |
单贞. 异源表达Hvsusiba2籼稻"明恢86"稻田甲烷减排研究[D]. 福州: 福建农林大学, 2017.
|
| [3] |
王明星, 李晶, 郑循华. 稻田甲烷排放及产生、转化、输送机理. 大气科学, 1998, 22(4): 600-612. DOI:10.3878/j.issn.1006-9895.1998.04.20 |
| [4] |
Su J, Hu C, Yan X, Jin Y, Chen Z, Guan Q, Wang Y, Zhong D, Jansson C, Wang F, Schnürer A, Sun C. Expression of barley SUSIBA2 transcription factor yields high-starch low-methane rice. Nature, 2015, 523: 602-606. DOI:10.1038/nature14673 |
| [5] |
单贞, 苏军, 陈在杰, 胡昌泉. 异源表达Hvsusiba2基因水稻的淀粉含量及相关酶谱分析. 福建农业学报, 2017, 32(3): 229-233. |
| [6] |
苏军, 单贞, 陈在杰. 异源表达Hvsusiba2水稻对稻田甲烷排放及土壤相关菌群的影响. 中国生态农业学报, 2018, 26(9): 1333-1342. |
| [7] |
Du L, Wang Y F, Shan Z, Shen X L, Wang F, Su J. Comprehensive analysis of SUSIBA2 rice: the low-methane trait and associated changes in soil carbon and microbial communities. Science of the Total Environment, 2021, 764: 144508. DOI:10.1016/j.scitotenv.2020.144508 |
| [8] |
宋亚娜, 陈在杰, 林艳, 胡太蛟, 吴明基, 王锋. 抗虫转基因水稻及其杂交水稻对土壤微生物群落多样性与组成的影响. 中国生态农业学报(中英文), 2024, 32(1): 15-29. |
| [9] |
杨永华. 转基因作物对土壤微生物群落的影响及主要研究策略. 农业生物技术学报, 2011, 19(1): 1-8. DOI:10.3969/j.issn.1674-7968.2011.01.001 |
| [10] |
楼骏, 柳勇, 李延. 高通量测序技术在土壤微生物多样性研究中的研究进展. 中国农学通报, 2014, 30(15): 256-260. DOI:10.11924/j.issn.1000-6850.2013-2513 |
| [11] |
郑燕, 贾仲君. 新一代高通量测序与稳定性同位素示踪DNA/RNA技术研究稻田红壤甲烷氧化的微生物过程. 微生物学报, 2013, 53(2): 173-184. |
| [12] |
陈庆荣, 王成己, 陈曦, 唐莉娜, 刘岑薇, 宋铁英, 黄毅斌. 施用烟秆生物黑炭对红壤性稻田根际土壤微生物的影响. 福建农业学报, 2016, 31(2): 184-188. |
| [13] |
张芳, 林绍艳, 徐颖洁. 水稻连作对江苏地区稻田土细菌微生物多样性的影响. 山东农业大学学报: 自然科学版, 2014, 45(2): 161-165. DOI:10.3969/j.issn.1000-2324.2014.02.01 |
| [14] |
Rösch C, Mergel A, Bothe H. Biodiversity of denitrifying and dinitrogen-fixing bacteria in an acid forest soil. Applied and Environmental Microbiology, 2002, 68(8): 3818-3829. |
| [15] |
Rotthauwe J H, Witzel K P, Liesack W. The ammonia monooxygenase structural gene amoA as a functional marker: molecular fine-scale analysis of natural ammonia-oxidizing populations. Applied and Environmental Microbiology, 1997, 63(12): 4704-4712. DOI:10.1128/aem.63.12.4704-4712.1997 |
| [16] |
Santoro A E, Boehm A B, Francis C A. Denitrifier community composition along a nitrate and salinity gradient in a coastal aquifer. Applied and Environmental Microbiology, 2006, 72(3): 2102-2109. DOI:10.1128/AEM.72.3.2102-2109.2006 |
| [17] |
Luton P E, Wayne J M, Sharp R J, Riley P W. The mcrA gene as an alternative to 16S rRNA in the phylogenetic analysis of methanogen populations in landfill. Microbiology, 2002, 148: 3521-3530. |
| [18] |
He J Z, Shen J P, Zhang L M, Zhu Y G, Zheng Y M, Xu M G, Di H J. Quantitative analyses of the abundance and composition of ammonia-oxidizing bacteria and ammonia-oxidizing archaea of a Chinese upland red soil under long-term fertilization practices. Environmental Microbiology, 2007, 9(9): 2364-2374. |
| [19] |
Caporaso J G, Bittinger K, Bushman F D, DeSantis T Z, Andersen G L, Knight R. PyNAST: a flexible tool for aligning sequences to a template alignment. Bioinformatics, 2010, 26(2): 266-267. |
| [20] |
Lozupone C, Knight R. UniFrac: a new phylogenetic method for comparing microbial communities. Applied and Environmental Microbiology, 2005, 71(12): 8228-8235. |
| [21] |
鲍士旦. 土壤农化分析 (3版). 北京: 中国农业出版社, 2000.
|
| [22] |
尉海东. 稻田甲烷排放研究进展. 中国农学通报, 2013, 29(18): 6-10. |
| [23] |
Bender M, Conrad R. Methane oxidation activity in various soils and freshwater sediments: occurrence, characteristics, vertical profiles, and distribution on grain size fractions. Journal of Geophysical Research: Atmospheres, 1994, 99(D8): 16531-16540. |
| [24] |
Wang M X, Shangguan X J, Shen R X, Reiner W, Wolfgang S. Methane production, emission and possible control measures in the rice agriculture. Advances in Atmospheric Sciences, 1993, 10(3): 307-314. |
| [25] |
Schütz H, Holzapfel-Pschorn A, Conrad R, Rennenberg H, Seiler W. A 3-year continuous record on the influence of daytime, season, and fertilizer treatment on methane emission rates from an Italian rice paddy. Journal of Geophysical Research: Atmospheres, 1989, 94(D13): 16405-16416. |
| [26] |
苏军, 王锋, 胡昌泉, 胡太蛟, 李刚, 颜静宛, 林智敏. 大麦Hvsusiba2基因在降低水稻田甲烷释放的应用: CN104593411B[P]. 2017-08-29.
|
| [27] |
兰洪国, 杜春影, 刘梦红. 水稻源库关系研究进展. 北方水稻, 2007, 37(1): 13-18. |
| [28] |
潘圣刚, 莫钊文, 田华, 聂俊, 罗一鸣, 段美洋, 唐湘如. 源库调节对水稻生长发育的影响. 广东农业科学, 2012, 39(14): 1-3. |
| [29] |
Denier Van Der Gon H A, Kropff M J, Van Breemen N, Wassmann R, Lantin R S, Aduna E, Corton T M, Van Laar H H. Optimizing grain yields reduces CH4 emissions from rice paddy fields. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(19): 12021-12024. |
| [30] |
Song Y, Su J, Wu M, Wang F. Effect of Hvsusiba2 transgenic rice on soil bacterial community and functional gene in paddy field in Fujian Province China. Journal of Agricultural and Crop Research, 2021, 9(5): 112-120. |
| [31] |
Berendsen R L, Pieterse C M J, Bakker P A H M. The rhizosphere microbiome and plant health. Trends in Plant Science, 2012, 17(8): 478-486. |
| [32] |
Bulgarelli D, Schlaeppi K, Spaepen S, Ver Loren van Themaat E, Schulze-Lefert P. Structure and functions of the bacterial microbiota of plants. Annual Review of Plant Biology, 2013, 64: 807-838. |
| [33] |
Baudoin E, Benizri E, Guckert A. Impact of artificial root exudates on the bacterial community structure in bulk soil and maize rhizosphere. Soil Biology and Biochemistry, 2003, 35(9): 1183-1192. |
| [34] |
Masoero F, Moschini M, Rossi F, Prandini A, Pietri A. Nutritive value, mycotoxin contamination and in vitro rumen fermentation of normal and genetically modified corn (cry1Ab) grown in northern Italy. Maydica, 1999, 44(3): 205-209. |
| [35] |
Song Y N, Su J, Chen R, Lin Y, Wang F. Diversity of microbial community in a paddy soil with cry1Ac/cpti transgenic rice. Pedosphere, 2014, 24(3): 349-358. |
 2024, Vol. 44
2024, Vol. 44