文章信息
- 田新民, 宋雅祺, 赵雨桐, 靳勇超
- TIAN Xinmin, SONG Yaqi, ZHAO Yutong, JIN Yongchao
- 吉林省黄泥河林区东北马鹿野外放归成效评估
- Effectiveness assessment on red deer introduction in the Huangnihe of Jilin Province
- 生态学报. 2024, 44(14): 6383-6390
- Acta Ecologica Sinica. 2024, 44(14): 6383-6390
- http://dx.doi.org/10.20103/j.stxb.202312212775
-
文章历史
- 收稿日期: 2023-12-21
- 网络出版日期: 2024-05-11
2. 吉林农业大学林学与草学学院, 长春 130118
2. College of Forestry and Grassland Science, Jilin Agricultural University, Changchun 130118, China
野外放归已成为拯救、恢复濒危物种的有效手段, 目前全球也开展了多种动物的野外放归研究与实践, 并取得了诸多进展[1]。野生动物放归后的监测与评估是检测其对野外环境适应的重要前提, 也是判断其是否建立稳定种群和放归成功以及优化放归策略的重要途径[2]。评估野生动物放归是否成功是一个复杂的过程, 多涉及其在野外环境中的行为表现[2]、生存率[3]、繁殖[4]和生境利用[5]等方面。而放归种群是否参与到野生种群的繁殖过程中往往是最直接有效的证据[6];且其对该物种种群遗传多样性的影响也是重要的检测指标, 这对野生动物种群的长期续存和进化至关重要。
东北马鹿(Cervus canadensis xanthopygus)为马鹿东北亚种, 国家二级重点保护野生动物, 是东北虎(Panthera tigris altaica)最主要的猎物之一[7]。在长期种群缩减的背景下, 近年来东北马鹿在少数重点区域呈现数量恢复、栖息地面积扩大的趋势, 但仍面临栖息地破碎化而导致的小种群孤岛化分布, 制约了其种群数量的恢复[8]。其中张广才岭南部的黄泥河林区一直是东北马鹿主要的分布区, 也是东北虎重要的栖息地[7]。历史上, 该区域的东北马鹿曾一度濒临灭绝, 虽然其种群数量近20年在缓慢恢复, 但仍面临着分布区狭小、种群数量少且被人为干扰、隔离等, 影响种群的恢复与扩散[7—10]。目前, 该区域的东北马鹿已开展了栖息地选择与评价[11—12]、冬季容纳量[12]、活动节律[13]、肠道微生物与食性[14]、种群遗传多样性与结构[8—9]等方面的研究, 为其种群数量的恢复奠定了基础。
为恢复野生东北马鹿种群, 改善隔离小种群的遗传结构, 提高遗传多样性, 维持种群的长期进化发展潜力, 黄泥河林区于2019年野外放归了5只健康圈养的东北马鹿。为有效评估东北马鹿的野外放归成效, 本研究基于非损伤性标志重捕法评估了黄泥河林区东北马鹿的种群数量;利用粪便DNA信息, 从亲权鉴定和遗传多样性方面对其野外放归成效进行了评估, 以期为东北马鹿种群的进一步科学保护与恢复提供参考, 也为其他濒危物种野外放归的监测提供借鉴。
1 材料和方法 1.1 研究地区与样本采集黄泥河林区为长白山森工集团黄泥河林业局辖区, 位于吉林省张广才岭南部, 总面积1983.62 km2, 林区内北部分布有面积415.83 km2的黄泥河国家级自然保护区。研究区中部和东南部高密度的居民点, 境内与周边的高速公路(G10、G11和G12)、铁路和国道等因素的阻隔, 已严重影响该地区马鹿种群的扩散及其与其他地区种群间的交流(图 1)。此外, 该区域的马鹿种群曾一度濒临灭绝, 近20年虽有一定的恢复, 但密度仍极低[9]。为促进马鹿种群的恢复, 2019年4月将5只圈养东北马鹿个体(4♀, 1♂)在黄泥河林区上马场林场的野化基地进行野化训练, 并于2019年9月采取自由扩散的方式进行野外放归, 其个体编号为R1—R5, 其中R1—R3均为3岁的雌性个体, R4为4岁的雌性个体, R5为4岁的雄性个体。
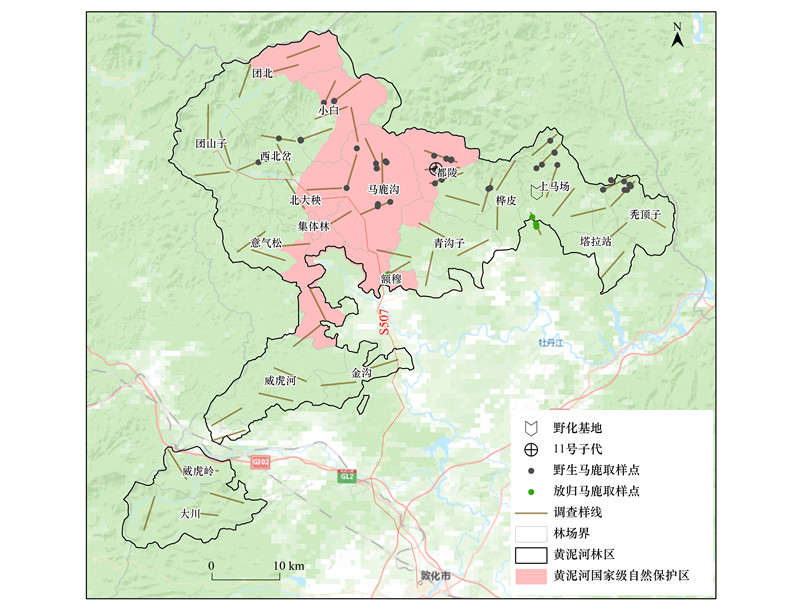
|
| 图 1 研究地区与取样位置 Fig. 1 Study area and sampling sites |
2021年12月份, 基于系统随机抽样[15—17], 在黄泥河林区的18个林场共布设60条5 km长的样线, 平均每个林场3—4条样线(图 1)。野外样本的采集, 沿调查样线主要方向寻找并跟踪马鹿足迹链, 采集粪便样本, GPS定位。根据放归个体GPS项圈定位的活动位点判断其活动区域, 并在区域附近的此降雪覆盖期间是否存在野生个体活动踪迹, 来明确区分放归与野生个体的采集样本。同一条样线区域内的同一足迹链仅取一份新鲜粪便样本, 来排除“假重捕”现象, 而在其他区域采集到的同一个体样本作为重捕样本。野外期间共收集粪便样本89份, 来自野生个体82份, 放归个体7份, 样本-80 ℃保存。
1.2 个体识别与性别鉴定采用QIAamp Fast DNA Stool Mini Kit(Qiagen)试剂盒提取粪便DNA, 利用mtDNA Cyt b引物L14724/H15149[18—19]对粪便DNA进行物种鉴定, PCR扩增、测序和NCBI数据库BLAST序列比对, 进一步核实粪便样本的来源物种。对来自马鹿的粪便DNA, 使用10对微卫星引物(T507、T530、T501、C143、T156、BM848、N、OCAM、DM45和ETH225)[8]进行个体识别, 这些位点的PCR扩增、基因分型、多态性和个体联合判别率等方面在马鹿的个体识别与遗传多样性评价中都已得到验证和应用。个体识别采用多管PCR扩增方案, 每个位点进行3—7次阳性PCR扩增, 以判定最终基因型。以上引物信息、PCR扩增、基因型检测过程参见文献[8]。将得到的马鹿样本10个微卫星基因型数据, 利用软件Excel microsatellite tool kit[20]寻找数据中相匹配的基因型, 判断不同样本来自同一个体的原则为全部等位基因相同或仅一个位点上的一个等位基因存在差异[21], 从而得到马鹿样本来源的个体数。
以X染色体ZFX与Y染色体SRY基因设计的4对引物, 基于巢式PCR进行马鹿样本的性别鉴定。第一轮扩增采用2对外引物: ZFX1-L: 5′-GGT AAG TCC TGT CGC AGC TC-3′, ZFX1-H: 5′-CGG AAA TTC CCC ATT CTA GG-3′(425 bp);MT1:5′-GCT CTA GAG AAT CCC CAA ATG-3′, DSRY1-H: 5′-ATT CGT GAG CCT GTG GTA TTG-3′(290 bp)。第二轮扩增以外引物扩增产物稀释20倍作为DNA模板, 采用2对内引物: ZFX2-L: 5′-GTT GGT TCT TTA ACG TGA ATT C-3′, ZFX2-H: 5′-GAA ATG CCT AGC TTC CAT ATC-3′(334 bp);DSRY2-L: 5′-CTG AAA AGC GAC CAT TCT TTG-3′, DSRY2-H: 5′-CAA TTT CTG TTG CCT CTT CG-3′(113 bp)[22]。每个样本进行5次以上独立平行PCR扩增, 最终以内引物扩增的电泳结果判定性别。如果至少3次出现双带(334 bp的ZFX和113 bp的SRY带)的样本记录为雄性;至少3次出现ZFX带, 所有扩增均未出现SRY带的样本记录为雌性;按此标准无法判断性别的样本将被舍弃[9, 22]。
1.3 种群数量分析根据个体识别结果, 不同地点的同一个体, 其中任何一次记为标志, 其他记录为该个体的重捕, 以此基于标志重捕法评估种群数量[15]。野外样本采集, 在同一条样线区域内的同一足迹链仅取一份样本, 避免出现“假重捕”现象。数量分析使用R软件Capwire程序包[23]完成, 软件有两种模型: ECM(even capture model)和TIRM(two innate rates model)。ECM模型的基本假设是种群中每个个体的排便率基本一致, 对每个个体的抽样强度也基本一致, 因此每个个体被捕获的概率是一致的, 即种群数量N的倒数1/N。TIRM模型的基本假设是种群中个体间被捕获的概率相差很大, 在模型拟合时按照捕捉的概率将个体分成捕捉困难(B)和容易捕捉(A)两大类[15]。最后用10000次bootstrap检验产生95%水平上N的置信区间, 并使用程序包似然比测试(likelihood ratio test)检验模型适用性, 如果P值显著偏离0, 说明个体具有相同的捕获率, ECM模型结果更为准确;如果P值趋近于0, 说明个体间捕获率相差很大, TIRM模型结果更为准确[16, 23]。
1.4 遗传多样性与亲权鉴定结合2018年本研究区内野生马鹿种群遗传多样性评估结果[8], 为评价野外放归对种群遗传多样性的影响, 本研究对两个时期的种群进行了比较分析。对于个体的mtDNA序列, 采用DnaSP 5.10[24]计算种群的单倍型数量(H)、单倍型多样性(Hd)和核苷酸多样性(Pi)。对于微卫星数据, 采用GenAlEx 6.0[25]计算位点和种群的等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)和期望杂合度(He)。等位基因数的估算可能会受到样本量的影响, 在比较不同样本量种群时常用等位基因丰富度(allelic richness, AR)来代替等位基因数。等位基因丰富度是指以种群最低样本量为基准观测到的等位基因数, 采用Allelic Diversity Analyzer(ADZE)1.0[26]以样本量小的种群个体数为基准, 计算等位基因丰富度。采用配对样本T检验对放归前、后种群遗传参数进行差异显著性检验。
采用Cervus 3.0[27]进行亲权鉴定, 以评估放归个体的繁殖情况。将性别鉴定识别出的全部雄性和雌性分别作为候选父本和母本, 取样概率均设为1.0, 其他选项均为系统默认值。为了保证鉴定结果的准确性, 所有位点全匹配的才确认亲子关系。采用Kingroup v2[28], 基于Queller和Goodnight的计算方法(QGM法)[29], 计算个体间的亲缘系数(r)作为亲权鉴定的补充[30—31]。亲缘系数范围在-1到1之间, 表示亲缘相近的程度, 0.5≤r < 1时为同父同母的兄弟姐妹关系, 0.25≤r < 0.5时为同父异母或同母异父的兄弟姐妹关系[31]。
2 结果与分析 2.1 种群数量与性比野外共采集到89份(野生个体82份, 放归个体7份)粪便样本, DNA提取与Cyt b扩增成功的样本有83份(野生个体77份, 放归个体6份), 样本利用率93.26%。物种鉴定核实, 有72份(野生个体66份, 放归个体6份)样本来自马鹿, 其余11份来自狍(Capreolus pygargus)。个体识别鉴定, 72份马鹿样本分属51只(野生个体46只(编号1—46), 放归个体5只(编号R1—R5))个体(表 1)。性别鉴定显示, 51只个体有雌性33只, 雄性18只, 雌雄性比1.83 ∶ 1;46只野生个体中雌性29只, 雄性17只, 雌雄性比1.71 ∶ 1, 野外放归增加了种群内雌性个体的比例。
| 种群 Population |
采集样本数 Collected sample numbers |
物种鉴定样本数 Species identification samples |
马鹿样本数 Red deer samples |
马鹿个体数 Identified red deer individuals |
| 野生个体Wild individuals | 82 | 77 | 66 | 46 |
| 放归个体Introduced individuals | 7 | 6 | 6 | 5 |
| 合计Total | 89 | 83 | 72 | 51 |
66份野生马鹿样本分属46只个体, 其中2只个体被采集到4次, 3只被采集到3次, 8只被采集到2次, 其余33只被采集到1次。6份放归马鹿样本分属5只个体, 其中1只个体被采集到2次, 其余4只被采集到1次。共计72份样本分属51只个体, 平均每只个体被采集到1.41次(野生个体1.44次, 放归个体1.20次)。运行Capwire程序包, 基于样本分布比较ECM和TIRM模型的适用性, 似然比测试结果显示P值显著偏离0, 说明个体被捕捉的概率相同, 因此种群数量以ECM模型的结果为依据。基于ECM模型, 统计出放归前的野生种群数量为85(95%置信区间: 63—119)只, 放归后的种群数量为96(95%置信区间: 69—152)只。野外调查的样本分布结果显示, 马鹿种群主要集中分布于黄泥河林区的北部(图 1)。
2.2 放归的遗传贡献mtDNA结果显示, 放归后种群内51只个体的Cyt b序列(425 bp)共检测到变异位点8个, 单倍型数(H)6个, 单倍型多样性(Hd)为0.678±0.059, 核苷酸多样性(Pi)为(0.435±0.043)%。微卫星结果显示, 放归后种群的平均等位基因数(Na)为(8.1±1.2)个, 有效等位基因数(Ne)为(3.9±0.5)个, 观测杂合度(Ho)为0.711±0.046, 期望杂合度(He)为0.695±0.044。2018年放归前的种群结果显示, Na为(5.2±0.7)个, Ne为(3.0±0.4)个, Ho为0.701±0.085, He为0.608±0.059(表 2)。统计发现, Na值的放归后种群极其显著高于放归前(P < 0.01), Ne值的放归后种群也显著高于放归前(P<0.05), Ho和He值也呈现放归后种群高于放归前, 但差异不显著(P>0.05)。等位基因丰富度(AR)结果显示, 放归后种群AR为6.37±0.82(不计5只放归个体的野生种群AR为5.93±0.70), 放归前为4.65±0.53, 也呈现放归后种群明显高于放归前。等位基因数(Na)显示, 本次野外放归向野生种群引入了每个位点平均2.9个“新”等位基因, 表明新的遗传物质被引入到野生种群。
| 位点Locus | Na(A) | Na(B) | Ne(A) | Ne(B) | Ho(A) | Ho(B) | He(A) | He(B) |
| T507 | 3 | 9 | 1.4 | 4.0 | 0.300 | 0.725 | 0.259 | 0.752 |
| T530 | 7 | 10 | 4.3 | 6.4 | 0.875 | 0.824 | 0.768 | 0.844 |
| T501 | 4 | 6 | 1.6 | 2.4 | 0.333 | 0.580 | 0.389 | 0.590 |
| C143 | 3 | 4 | 2.0 | 2.0 | 0.500 | 0.510 | 0.502 | 0.510 |
| T156 | 7 | 15 | 3.6 | 5.7 | 0.875 | 0.816 | 0.722 | 0.825 |
| BM848 | 6 | 8 | 3.6 | 3.9 | 0.900 | 0.763 | 0.718 | 0.742 |
| N | 2 | 2 | 1.9 | 1.8 | 0.469 | 0.500 | 0.476 | 0.444 |
| OCAM | 6 | 5 | 3.5 | 3.8 | 1.000 | 0.961 | 0.717 | 0.736 |
| DM45 | 6 | 10 | 3.8 | 3.1 | 0.828 | 0.694 | 0.738 | 0.679 |
| ETH225 | 8 | 12 | 4.7 | 5.9 | 0.931 | 0.740 | 0.788 | 0.832 |
| 平均值Mean | 5.2 | 8.1 | 3.0 | 3.9 | 7.01 | 0.711 | 0.608 | 0.695 |
| Na: 等位基因数Number of alleles;Ne: 有效等位基因数Effective number of alleles;Ho: 观测杂合度Observed heterozygosity;He: 期望杂合度Expected heterozygosity | ||||||||
5只放归个体的平均亲缘系数(r)为-0.297±0.613, 46只野生个体的r为-0.022±0.212, 放归后种群的r为-0.023±0.215, 说明放归对种群内个体的平均亲缘系数有所降低, 但二者差异不显著(P>0.05)。为了保证亲权鉴定结果的准确性, 将微卫星所有位点全部匹配才确认亲子关系。基于亲权鉴定, 在51只个体内共检测到父-母-子8对、父-子7对、母-子5对, 其共涉及到38只个体, 其余13只个体未鉴定出亲子关系。其中15对父-子的平均亲缘系数(r)为0.612(0.521—0.764), 13对母-子的r为0.628(0.522—0.853), 亲缘系数结果进一步支持了亲权鉴定检测结果的准确性。亲权鉴定繁殖证据揭示, 放归R3个体(♀)与野生2号个体(♂)在野外成功繁殖, 并产下子代11号个体(♀)。
3 讨论 3.1 种群数量与分布本研究基于非损伤性标志重捕法ECM模型评估, 得出不计放归个体数据的野生马鹿种群数量为85(63—119)只, 计入放归个体数据后的放归后种群数量为96(69—152)只。来自6份样本的5只放归个体的加入, 使平均种群数量增加了11只, 95%置信区间内的最低值数量增加了6只, 其主要与来自放归个体的采样强度较低有关。结合野生种群数量, 保守评估认为, 放归后平均马鹿种群数量为90(68—124)只, 核算其种群密度为0.045(0.034—0.063)只/km2。本研究结果, 远低于Qi等采用FMP样线法评估出的张广才岭林区马鹿平均种群密度(0.15—0.75只/km2)[7]。FMP样线法为结合调查物种的日活动距离, 记录24h内个体通过样线留下的足迹链数目来推算种群密度[7, 16]。该方法在某一个体或群反复穿过样线, 其伪重复将导致结果高估, 同时调查物种的日活动距离也会受到不同因素的影响[16, 32]。在大兴安岭驼鹿(Alces alces)调查的4种方法比较中, FMP样线法的结果高于非损伤性标志重捕法3倍以上, 并指出与传统的样带、样线法相比, 非损伤性标志重捕、大样方法所基于的原理、统计方法与结果更为科学和准确[16]。朱世兵等[12]评估出黄泥河林区马鹿种群冬季容纳量为574只(0.29只/km2), 说明该地区马鹿种群尚有较大恢复与增长的空间。对研究区18个林场的野外调查样本分布显示出, 马鹿种群主要集中分布于黄泥河林区北部的8个林场内, 朱世兵等[12]也指出该地区马鹿的适宜性栖息地也主要位于林区北部, 其与当前马鹿重点分布结果相一致。
3.2 放归的遗传贡献濒危物种的野外放归与重引入可以复壮野生种群, 提高遗传多样性, 进而维持种群的长期进化潜力[33]。mtDNA的结果显示, 放归后种群的单倍型多样性(Hd)为0.678±0.059, 核苷酸多样性(Pi)为(0.435±0.043)%, 呈现高单倍型多样性(Hd ≥0.5)与低核苷酸多样性(Pi < 0.5%), 认为是种群瓶颈效应后快速增长和突变积累的结果[34—35], 其与2018年开展的野生种群研究结论相一致[8]。微卫星的等位基因数(Na)显示, 本次野外放归向野生种群引入了每个位点平均2.9个“新”等位基因, 说明新的遗传物质被引入到野生种群。与2018年放归前结果内32只个体样本量相比, 本研究放归后的51只个体其样本量的增加可能会增大“新”等位基因的检出频率[36]。为消除样本量的影响, 以样本量小的种群个体数为基准, 计算其等位基因丰富度(AR)的结果显示, 放归后种群AR为6.37±0.82, 不计5只放归个体的野生种群AR为5.93±0.70, 均高于放归前(2018年)的AR值4.65±0.53, 也进一步说明其野外放归提高了马鹿种群的遗传多样性水平。放归后种群内平均等位基因数(Na)为(8.1±1.2)个, 有效等位基因数(Ne)为(3.9±0.5)个, Ne值极其显著低于Na(P < 0.01), 也提示种群未来依然存在等位基因丢失及遗传多样性下降的风险。
3.3 放归个体的繁殖证据经历2年多的野外放归, 5只个体均尚存活。研究认为, 建群者来源、遗传多样性、食性等因素为影响圈养动物野外放归成功的因素[33]。本研究放归个体全部来自半散放群体, 并于野化基地进行了近半年的野化训练, 其放归前已具备一定的野外适应能力, 并引入年龄均为3—4岁的青年个体;同时5只放归个体内检测到4个单倍型, 其具有较高的遗传多样性水平;马鹿为草食性物种也较其他肉食及杂食性动物更易适应野外环境。肠道微生物与食性的适应性评估也表明, 放归个体的微生物结构已趋近于野生个体, 并已形成适应性偏好食物种类[8], 说明放归个体已具备较强的野外适应性。野外放归成功的重要评价标准之一为个体参与了野外繁殖[2], 本研究亲权鉴定检测到放归R3个体(♀)与野生2号个体(♂)在野外成功繁殖, 并产下子代11号个体(♀), 其中粪球样本外部形态指标也进一步表明子代11号个体符合亚成体特征[37]。样本采集位置表明, 母本R3个体位于桦皮、上马场林场交界处, 子代11号个体位于都陵林场, 父本2号个体位于马鹿沟林场, 马鹿沟、都陵、桦皮、上马场林场为西至东相邻, 其子代与母本未在同一群体内, 可能与子代个体已近2岁性成熟分群扩散离开母仔群有关[38]。性别鉴定显示, 野生种群(46只个体)的雌雄性比1.71 ∶ 1, 放归后种群(51只个体)的雌雄性比1.83 ∶ 1, 说明野外放归提高了种群的可繁殖概率。尽管本次放归工作仅为少数个体, 但已获得许多重要和乐观的信息, 也为进一步开展野外放归及其他濒危物种的监测和研究提供了借鉴与参考。
3.4 保护与管理建议基于本次研究结果, 对黄泥河林区东北马鹿种群的保护与管理提出以下建议: (1)鉴于马鹿种群重点集中分布于林区北部, 呈现一定程度的地理隔离, 建议亟待开展黄泥河林区的栖息地修复和生态廊道构建, 促使马鹿种群向林区南部以及周边扩散与繁衍, 从而实现马鹿和东北虎种群的快速增长和扩散;(2)鉴于种群未来依然存在等位基因丢失和遗传多样性下降的风险, 建议对稀有单倍型和等位基因个体重点监测和保护, 继续开展圈养种群的野外放归, 提高个体间的基因交流;(3)增加放归群体遗传多样性水平的检测, 利于丰富和提高野外种群基因库及适应进化潜力;(4)未来将借助红外相机、GPS项圈数据开展放归个体活动节律、家域等适应性的深入研究, 为放归成效提供更为全面的信息。
| [1] |
Seddon P J, Griffiths C J, Soorae P S, Armstrong D P. Reversing defaunation: restoring species in a changing world. Science, 2014, 345(6195): 406-412. DOI:10.1126/science.1251818 |
| [2] |
Yang Z S, Gu X D, Nie Y G, Huang F, Huang Y, Dai Q, Hu Y B, Yang Y, Zhou X, Zhang H M, Yang X Y, Wei F W. Reintroduction of the giant panda into the wild: a good start suggests a bright future. Biological Conservation, 2018, 217: 181-186. DOI:10.1016/j.biocon.2017.08.012 |
| [3] |
Goossens B, Setchell J M, Tchidongo E, Dilambaka E, Vidal C, Ancrenaz M, Jamart A. Survival, interactions with conspecifics and reproduction in 37 chimpanzees released into the wild. Biological Conservation, 2005, 123(4): 461-475. DOI:10.1016/j.biocon.2005.01.008 |
| [4] |
Goossens B, Setchell J M, Vidal C, Dilambaka E, Jamart A. Successful reproduction in wild-released orphan chimpanzees (Pan troglodytes troglodytes). Primates, 2003, 44(1): 67-69. DOI:10.1007/s10329-002-0003-y |
| [5] |
Mladenovic S, Malinic M, Radic B, Vilotic D, Stamenkovic S, Gacic D. Monitoring of reintroduced red deer in the area of Tara (western Serbia): incidence of bark stripping within an acclimatisation enclosure and postrelease movements. Sumarski List, 2022, 146(9-10): 447-457. DOI:10.31298/sl.146.9-10.6 |
| [6] |
Seddon P J. Persistence without intervention: assessing success in wildlife reintroductions. Trends in Ecology & Evolution, 1999, 14(12): 503. |
| [7] |
Qi J Z, Gu J Y, Ning Y, Miquelle D, Holyoak M, Wen D S, Liang X, Liu S Y, Roberts N, Yang E Y, Lang J M, Wang F Y, Li C, Liang Z, Liu P Q, Ren Y, Zhou S C, Zhang M H, Ma J Z, Jiang G S. Integrated assessments call for establishing a sustainable meta-population of Amur tigers in northeast Asia. Biological Conservation, 2021, 261(12): 109250. |
| [8] |
田新民, 张明海. 基于粪便DNA的东北马鹿遗传多样性. 兽类学报, 2023, 43(1): 41-49. |
| [9] |
田新民. 东北马鹿种群遗传学及遗传分化驱动因素研究[D]. 哈尔滨: 东北林业大学, 2021.
|
| [10] |
田新民, 陈红, 钟林强, 黄海娇, 刘小慧, 张子栋, 杨孟平, 周绍春. 黑龙江省老爷岭南部穆棱林区马鹿种群分布数量及生境适宜性评价. 生态学报, 2022, 42(14): 5980-5989. DOI:10.5846/stxb202109172622 |
| [11] |
朱洪强, 葛志勇, 毛之夏, 刘庚, 常素慧, 李成, 张晓东, 白玉刚. 吉林黄泥河自然保护区马鹿冬季栖息地选择. 四川动物, 2012, 31(3): 478-482. |
| [12] |
朱世兵, 靳勇超, Yachmennikova Anna, Kotlov Ivan, 张士芳, 刘应竹, 张日, 张明海. 张广才岭南部东北虎猎物种群冬季营养容纳量研究. 生态学报, 2023, 43(13): 5432-5441. DOI:10.5846/stxb202109192634 |
| [13] |
马帅. 黄泥河林区三种有蹄类动物时空生态位研究[D]. 长春: 吉林农业大学, 2023.
|
| [14] |
Guo J H, Jin Y C, Tian X M, Bao H, Sun Y, Gray T, Song Y Q, Zhang M H. Diet-induced microbial adaptation process of red deer (Cervus elaphus) under different introduced periods. Frontiers in Microbiology, 2022, 13: 1033050. DOI:10.3389/fmicb.2022.1033050 |
| [15] |
田新民, 张明海. 基于粪便DNA的马鹿种群数量和性比. 生态学报, 2010, 30(22): 6249-6254. |
| [16] |
刘辉, 姜广顺, 李惠. 北方冬季有蹄类动物4种数量调查方法的比较. 生态学报, 2015, 35(9): 3076-3086. DOI:10.5846/stxb201308172102 |
| [17] |
Ebert C, Sandrini J, Welter B, Thiele B, Hohmann U. Estimating red Deer (Cervus elaphus) population size based on non-invasive genetic sampling. European Journal of Wildlife Research, 2021, 67(2): 27. DOI:10.1007/s10344-021-01456-8 |
| [18] |
Kocher T D, Thomas W K, Meyer A, Edwards S V, Pääbo S, Villablanca F X, Wilson A C. Dynamics of mitochondrial DNA evolution in animals: amplification and sequencing with conserved primers. Proceedings of the National Academy of Sciences of the United States of America, 1989, 86(16): 6196-6200. |
| [19] |
Irwin D M, Kocher T D, Wilson A C. Evolution of the cytochrome b gene of mammals. Journal of Molecular Evolution, 1991, 32(2): 128-144. DOI:10.1007/BF02515385 |
| [20] |
Park S D E. Trypanotolerance in west african cattle and the population genetic effects of selection[D]. Dublin: University of Dublin, 2001.
|
| [21] |
Bellemain E, Swenson J E, Tallmon O, Brunberg S, Taberlet P. Estimating population size of elusive animals with DNA from hunter-collected feces: four methods for brown bears. Conservation Biology, 2005, 19(1): 150-161. DOI:10.1111/j.1523-1739.2005.00549.x |
| [22] |
Yamazaki S, Motoi Y, Nagai K, Ishinazaka T, Asano M, Suzuki M. Sex determination of sika deer (Cervus nippon yesoensis) using nested PCR from feces collected in the field. Journal of Veterinary Medical Science, 2011, 73(12): 1611-1616. DOI:10.1292/jvms.11-0235 |
| [23] |
Pennell M W, Stansbury C R, Waits L P, Miller C R. Capwire: a R package for estimating population census size from non‐invasive genetic sampling. Molecular Ecology Resources, 2013, 13(1): 154-157. DOI:10.1111/1755-0998.12019 |
| [24] |
Librado P, Rozas J. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data. Bioinformatics, 2009, 25(11): 1451-1452. DOI:10.1093/bioinformatics/btp187 |
| [25] |
Peakall R, Smouse P E. GENALEX 6: genetic analysis in Excel. Population genetic software for teaching and research. Molecular Ecology Notes, 2006, 6(1): 288-295. DOI:10.1111/j.1471-8286.2005.01155.x |
| [26] |
Szpiech Z A, Jakobsson M, Rosenberg N A. ADZE: a rarefaction approach for counting alleles private to combinations of populations. Bioinformatics, 2008, 24(21): 2498-2504. DOI:10.1093/bioinformatics/btn478 |
| [27] |
Kalinowski S T, Taper M L, Marshall T C. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment. Molecular Ecology, 2007, 16(5): 1099-1106. DOI:10.1111/j.1365-294X.2007.03089.x |
| [28] |
Konovalov D A, Manning C, Henshaw M T. Kingroup: a program for pedigree relationship reconstruction and kin group assignments using genetic markers. Molecular Ecology Notes, 2004, 4(4): 779-782. DOI:10.1111/j.1471-8286.2004.00796.x |
| [29] |
Queller D C, Goodnight K F. Estimating relatedness using genetic markers. Evolution, 1989, 43(2): 258-275. DOI:10.2307/2409206 |
| [30] |
任鹏, 龚堃, 鲍毅新, 黄相相, 周晓, 韩金巧. 基于粪便DNA的小麂亲权鉴定和婚配制研究. 生态学报, 2017, 37(20): 6933-6944. DOI:10.5846/stxb201608021591 |
| [31] |
祝招玲, 刘振生, 高惠, 张致荣, 姚绪新, 刘鹏, 韩兴志, 王继飞, 滕丽微. 基于粪便DNA的贺兰山岩羊亲权鉴定和婚配制研究. 生态学报, 2019, 39(22): 8639-8647. DOI:10.5846/stxb201806261410 |
| [32] |
Stephens P A, Zaumyslova O Y, Miquelle D G, Myslenkov A I, Hayward G D. Estimating population density from indirect sign: track counts and the Formozov-Malyshev-Pereleshin formula. Animal Conservation, 2006, 9(3): 339-348. DOI:10.1111/j.1469-1795.2006.00044.x |
| [33] |
张泽钧, 张陕宁, 魏辅文, 王鸿加, 李明, 胡锦矗. 移地与圈养大熊猫野外放归的探讨. 兽类学报, 2006, 26(3): 292-299. |
| [34] |
Grant W, Bowen B. Shallow population histories in deep evolutionary lineages of marine fishes: insights from sardines and anchovies and lessons for conservation. Journal of Heredity, 1998, 89(5): 415-426. DOI:10.1093/jhered/89.5.415 |
| [35] |
Yuasa T, Nagata J, Hamasaki S, Tsuruga H, Furubayashi K. The impact of habitat fragmentation on genetic structure of the Japanese sika deer (Cervus nippon) in southern Kantoh, revealed by mitochondrial D-loop sequences. Ecological Research, 2007, 22(1): 97-106. DOI:10.1007/s11284-006-0190-x |
| [36] |
Oyler-McCance S J, Fedy B C, Landguth E L. Sample design effects in landscape genetics. Conservation Genetics, 2013, 14(2): 275-285. DOI:10.1007/s10592-012-0415-1 |
| [37] |
张明海, 田新民, 李熠. 通过粪球形态快速鉴定马鹿性别的方法与分子鉴定. 兽类学报, 2010, 30(3): 317-321. |
| [38] |
陈化鹏, 吴建平, 张明海. 黑龙江省马鹿. 哈尔滨: 东北林业大学出版社, 1997: 58-64.
|
 2024, Vol. 44
2024, Vol. 44