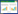文章信息
- 陈世喜, 陈梅, 李娜, 邹远超, 袁万安
- CHEN Shixi, CHEN Mei, LI Na, ZOU Yuanchao, YUAN Wanan
- 中国南海海域海洋深层水细菌多样性
- Bacterial diversity in deep sea water in the South China Sea
- 生态学报. 2024, 44(14): 6373-6382
- Acta Ecologica Sinica. 2024, 44(14): 6373-6382
- http://dx.doi.org/10.20103/j.stxb.202311172505
-
文章历史
- 收稿日期: 2023-11-17
- 网络出版日期: 2024-05-11
2. 长江上游鱼类资源保护与利用四川省重点实验室, 内江 641100;
3. 广州海洋地质调查局三亚南海地质研究所, 中国地质调查局南海地质科学院, 三亚 572024;
4. 自然资源部海底矿产资源重点实验室, 广州 511458
2. Fishes Conservation and Utilization in the Upper Reaches of the Yangtze River Key Laboratory of Sichuan Province, Neijiang Normal University, Neijiang 641100, China;
3. Sanya Institute of South China Sea Geology, Guangzhou Marine Geological Survey; Academy Of South China Sea Geological Science, China Geological Survey, Sanya 572024, China;
4. Key Laboratory of Marine Mineral Resources, Ministry of Natural Resources, Guangzhou 511458, China
地球表面面积约70%以上属于海洋, 占据整个生物圈体积的90%, 蕴藏着丰富的细菌资源[1—2]。海洋生境横向可分为远洋区和近岸海区, 纵向而言, 海洋生境中上部的100—200 m海水所对应的透光层被称为光合作用带, 光合作用带的以下区域依次为中层带、深层带、深渊带、超深渊带和深海沟[1], 并且, 海洋生境还包括海雪、海冰、所有类型的固体表面以及活的生物体等, 而这些生命环境, 无一例外的都是某些海洋细菌的生存环境[3]。海洋深层水(Deep sea water(DSW)或Deep ocean water(DOW))为海洋生境中海面约200 m以下的海水, 海洋植物无光合作用[4], 海洋深层水水层的微生物多样性报道较少。
海洋微生物总体数量庞大, 种类繁多, 截至2013年, 来自81个纲的12822种原核生物已被描述[5], 海洋细菌是海洋微生物中极其重要的组成部分, 其中变形菌门(Proteobacteria)是细菌域中最大的类群之一, 被进一步分类为α-变形菌纲(Alphaproteobacteria)、β-变形菌纲(Betaproteobacteria)、γ-变形菌纲(Gammaproteobacteria)、δ-变形菌纲(Deltaproteobacteria)和ε-变形菌纲(Epsilonproteobacteria), 对于海洋浮游细菌, α-变形菌纲和γ-变形菌纲尤为重要, 其中海水中易于培养的细菌多在γ-变形菌纲, 而依据不依赖培养的分子生物学研究发现α-变形菌纲则是海水中的最大优势细菌。尽管近年的研究发现了α-变形菌纲在种类和数量上的优势, 然而一些常见并且已发现重要功能的种属(如假单胞菌属(Pseudomonas)、弧菌属(Vibrio)、沙门氏菌属(Salmonella)及埃希氏菌属(Escherichia), 等)都在γ-变形菌纲中[6]。
南海是中国面积最大的海域, 地处热带-亚热带地区, 为西太平洋边缘海。南海海域面积为356万平方公里, 是我国唯一接近赤道的热带海区, 海洋资源最为丰富, 周围有多种生态系统分区如陆架、陆坡、岛礁、深海平原、海山、冷泉等多种生境, 蕴含着非常丰富的细菌资源, 是研究海洋细菌多样性的理想区域之一。前期研究中, 开展不同降压过程对南海深海海水中可培养细菌群落组成的影响研究表明, 深度6001 m的海洋深层水样在不同降压处理过程下细菌的丰度变化和群落组成不同[6], 其他对南海细菌的多样性研究主要集中在沉积物中[7—13], 对表层海水中细菌的多样性也有报道[14—15], 但南海海洋深层水的微生物研究资料较少, 有待进一步丰富。本文基于400 m的海洋深层水样品开展微生物生态多样性研究, 研究旨在丰富南海深海细菌的生态多样性数据, 为深层海水中微生物资源在生物学方面开发利用提供数据支持。
1 材料与方法 1.1 海水样品采集与细菌收集及采样地区域概况海洋深层水样品依据中华人民共和国国家标准GB17378.3—2007, 于2022年7月在南海深海海域内选取了1个采样点(112°35′39.6″E, 16°47′5.3″N), 采用水下高压泵法取样封存了400 m处的深层海水, 封存后立即运往实验室供实验分析用, 海上运输回实验室期间采用4℃冷藏, 陆地运输为冷链运输。采样点总水深为1050 m。
1.2 研究区域概况、海水样品采集与细菌收集深层海水样品50 mL用8 μm滤膜过滤后以0.22 μm核孔滤膜过滤, 以获得微生物细胞。测序根据所扩增的16S rRNA区域特点构建小片段文库, 基于Illumina NovaSeq测序平台对文库进行双末端测序。通过质量初筛的原始序列按照index和Barcode信息, 进行文库和样本划分, 按照Qiime2DADA2分析流程进行序列进行去引物, 质量过滤, 去噪, 拼接和去嵌合体等步骤获得最终的ASV[16—17];对样本在不同物种分类学水平的具体组成进行展示, 了解整体概况;根据ASV在不同样本中的分布, 评估每个样本的Alpha多样性水平, 并通过稀疏曲线反映测序深度是否合适;计算各样本的距离矩阵, 衡量不同样本间的Beta多样性差异及差异显著性。
1.3 细菌的高通量测序及数据统计分析对于细菌的16S rRNA基因, 选用Silva数据库(http://www.arb-silva.de)注释[18], 使用QIIME2的classify-sklearn或classify-consensus-blast算法[16, 19], 在QIIME2软件中使用默认参数, 对于每个ASVs的特征序列进行物种注释。
2 结果与分析 2.1 高通量测序的原始数据南海海洋深层水的细菌样本进行高通量测序后总计获得3057460条原始测序reads, 过滤得到3051932条过滤reads。过滤得到的reads数量占原始reads数量的比例99.82%。
2.2 门水平的细菌多样性通过与数据库进行比对, 注释了细菌域中的酸杆菌门(Acidobacteriota)的3科细菌, 放线菌门(Actinobacteriota)的5科细菌, 拟杆菌门(Bacteroidota)的29科细菌, 绿弯菌门(Chloroflexi)的2科细菌, 泉古菌门(Crenarchaeota)的9科细菌, 蓝细菌门(Cyanobacteria)的1科细菌, 厚壁菌门(Firmicutes)的118科细菌, 芽单胞菌门(Gemmatimonadota)的1科细菌, 海微菌门(Marinimicrobia (SAR406_clade))的5科细菌, 硝化螺旋菌门(Nitrospinota)的5科细菌, 浮霉菌门(Planctomycetota)的6科细菌, 变形菌门的127科细菌, 海洋细菌SAR324(Marine group B (SAR324 clade))的1科细菌, 热原体门(Thermoplasmatota)的1科细菌, 疣微菌门(Verrucomicrobiota)的1科细菌。依次的测序条数分别有21, 1306, 2233, 12, 161, 2, 26379, 794, 40, 86, 7441, 1143734, 12, 3, 21(图 1)。其中厚壁菌门中的注释的科数目最多, 变形菌门的测序数据量最大。
|
| 图 1 海洋深层水细菌在各门的种类及测序数据 Fig. 1 Species and sequencing data of deep-sea water bacteria in each phylum |
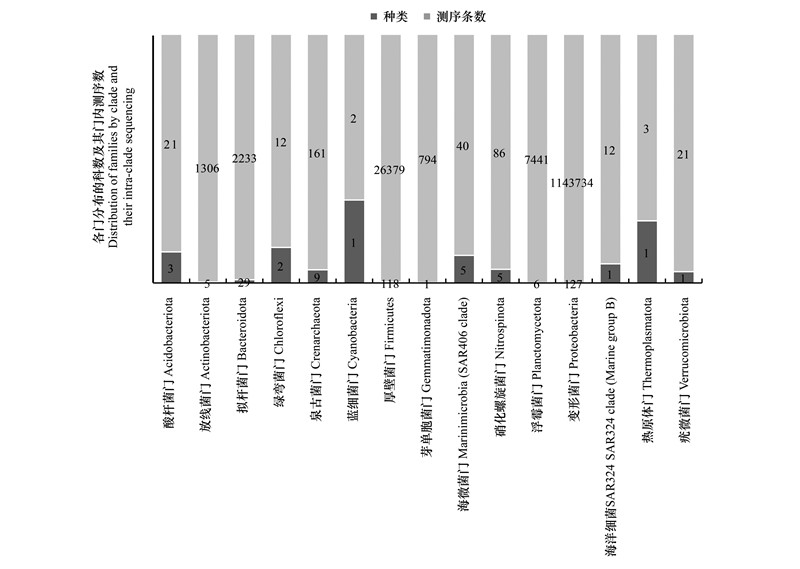
基于细菌的不同分类层级的物种丰度反映的样本的物种注释结果中门、纲、目、科、属五个分类水平各自含有的分类单元的数量, 测序数据量最大的变形菌门中的细菌主要分属于2纲、7目、11科、11属。纲水平中γ-变形菌纲, α-变形菌纲, 分别占比65%, 35%。目水平分别注释了红螺菌目(Rhodospirillales), 柄杆菌目(Caulobacterales), 根瘤菌目(Rhizobiales), 交替单胞菌目(Alteromonadales), 海洋螺杆菌目(Oceanospirillales), Nitrosococcales, 伯克氏菌目(Burkholderiales), 分别占比96%, 2%, 2%, 25%, 22%, 52%, 0.8%。科水平分别注释了毛螺菌科(Lachnospiraceae), 柄杆菌科(Caulobacteraceae), 根瘤菌科(Rhizobiaceae), 交替单胞菌科(Alteromonadaceae), 源洋菌科(Idiomarinaceae), Marinobacteraceae, 烷烃降解菌科(Alcanivoracaceae), 糖螺菌科(Saccharospirillaceae), 盐单胞菌科(Halomonadaceae), 嗜甲基菌科(Methylophagaceae), 依次分别占所在目的比例比100%, 100%, 99%, 33%, 38%, 37%, 25%, 37%, 100%, 99%。属水平分别注释了毛螺菌属(Lachnospira), 根瘤菌属(Chelativorans), 柄杆菌科未定属(unclassified Caulobacteraceae), 假交替单胞菌属(Pseudoalteromonas), 海源菌属(Idiomarina), 海杆菌属(Marinobacterium), 食烷菌属(Alcanivorax), 糖螺菌科未定属(unclassified Saccharospirillaceae), 盐单胞菌属(Halomonas), 噬甲基菌属(Methylophaga), 甲基营养菌属OM43 clade(OM43 clade), 属水平分别占变形菌门的比例为33%, 0.6%, 0.7%, 5%, 6%, 29%, 5%, 5%, 3%, 5%, 34%, 0.8%(图 2)。该海水样品在变形菌门中最为丰富的是嗜甲级菌属的细菌, 其次为毛螺菌属的细菌。
|
| 图 2 变形菌门中测序的细菌的不同分类层级的物种丰度 Fig. 2 Species abundance at different taxonomic levels of bacteria sequenced in the phylum Ascomycota 图中部分细菌分类单元由于研究较少, 暂无中文对照译名 |
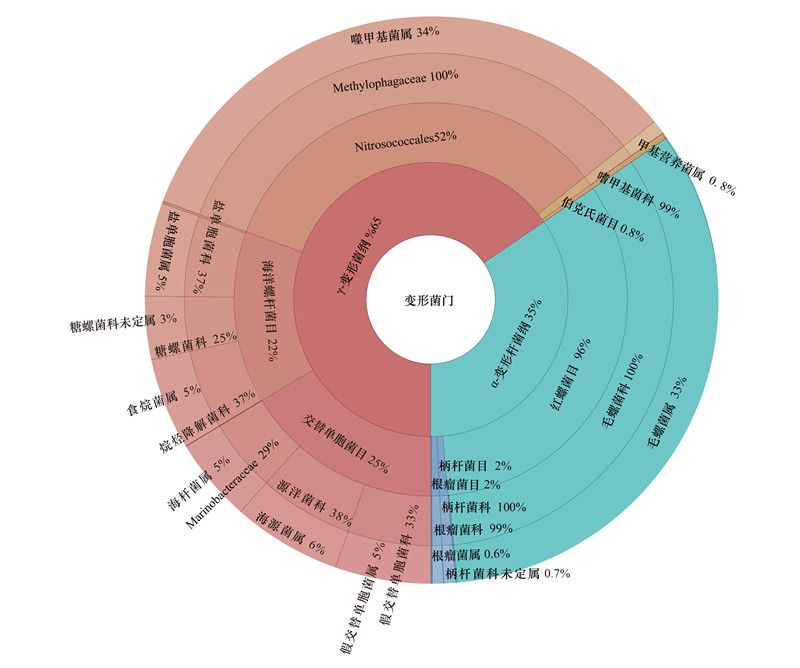
分析海洋深层水水样中注释种类最多的厚壁菌门中其细菌分属于2纲、3目、6科、25属。纲水平注释了芽孢杆菌纲(Bacilli), 梭菌纲(Clostridia), 分别占比99%, 1%。目水平注释了芽孢杆菌目(Bacillales), Paenibacillales, 毛螺菌目(Lachnospirales)。分别占比95%, 5%, 0.6%。科水平注释了芽孢杆菌科(Bacillaceae), 芽孢杆菌目未分类科(unclassified Bacillales), Marinococcaceae, 动球菌科(Planococcaceae), 类芽孢杆菌科(Paenibacillaceae), 毛螺菌科(Lachnospiraceae)。分别占比厚壁菌门的52%, 24%, 10%, 8%, 5%, 0.6%。属水平注释了芽孢杆菌目未分类属(unclassified Bacillales), 高钠芽孢杆菌属(Natribacillus), Alteribacillus, 嗜冷芽孢杆菌属(Psychrobacillus), 八叠球菌属(Sporosarcina), Paenisporosarcina, 芽孢杆菌属(Bacillus), 柯恩氏菌属(Cohnella), 两属其他的, 薄壁芽孢杆菌属(Gracilibacillus), 芽孢杆菌科未分类属(unclassified Bacillus), 海洋芽孢杆菌属(Oceanobacillus), Aeribacillus, 盐芽孢杆菌属(Halobacillus)。分别占厚壁菌门的比例为24%, 8%, 2%, 4%, 2%, 0.8%, 4%, 0.7%, 1%, 17%, 12%, 7%, 7%, 6%, 1, 0.8%(图 3)。该海水样品在厚壁菌门中最为丰富的是芽孢杆菌目未分类属及枝芽孢杆菌属的细菌。
|
| 图 3 厚壁菌门中测序的细菌的不同分类层级的物种丰度 Fig. 3 Species abundance at different taxonomic levels of bacteria sequenced in the phylum Firmicutes 图中部分细菌分类单元由于研究较少, 暂无中文对照译名 |
属以下鉴定的未知细菌的多样性很高, 说明南海深海系统中蕴含着许多潜在的新物种。在属水平以下, 水样中注释为物种的数目为类芽胞杆菌属19种、芽孢杆菌属16种、海旋菌属15种、噬甲基菌属11种、拟杆菌属12种、纤细芽胞杆菌属9种、海洋芽胞杆菌属7种、海源菌属7种、嗜油菌属6种、食碱菌属6种、unclassified_Saccharospirillaceae的2种、枝芽胞杆菌属(Virgibacillus)5种、盐单胞菌属2种、八叠球菌属1种、根瘤菌属2种、unclassified_Bacillales的3种、unclassified_Bacillaceae的5种、经黏液真杆菌属(Blautia)4种、海杆菌属4种、CL500-3的3种、unclassified_Gammaproteobacteria的3种、OM43_clade的3种、Thalassolituus的3种、拟杆菌属2种、假交替单胞菌属2种, 其他属为鉴定的单种属(图 4)。

|
| 图 4 海洋深层水样本中各属鉴定的具体种类数 Fig. 4 Specific species identified by genus in deep sea water samples |
具体可以确定的海洋深层水物种有变形菌门中α-变形菌纲、假单胞菌目、假单胞菌科的海洋解淀粉芽孢杆菌(Lentibacillus amyloliquefaciens), 以及变形菌门中γ-变形菌纲、假单胞菌目、假单胞菌科的施氏假单胞菌(Pseudomonas stutzeri), 柴油食烷菌(Alcanivorax dieselolei)和三浦半岛盐乳杆菌(Halolactibacillus miurensis);在放线菌门中鉴定了放线菌纲、棒状杆菌目、迪茨氏菌科的海迪茨氏菌(Dietzia maris)(表 1)。
| 门 Phylum |
纲 Class |
目 Order |
科 Family |
属 Genus |
种 Species |
| 变形菌门 Proteobacteria |
α-变形菌纲 Alphaproteobacteria |
假单胞菌目 Pseudomonadales |
假单胞菌科 Pseudomonadaceae |
芽孢杆菌属Bacillus | 海洋解淀粉芽孢杆菌 Lentibacillus_amyloliquefaciens |
| γ-变形菌纲 Gammaproteobacteria |
假单胞菌目 Pseudomonadales |
假单胞菌科 Pseudomonadaceae |
假单胞菌属 Pseudomonas |
施氏假单胞菌 Pseudomonas_stutzeri |
|
| 柴油食烷菌 Alcanivorax_dieselolei |
|||||
| 三浦半岛盐乳杆菌 Halolactibacillus_miurensis |
|||||
| 放线菌门 Actinobacteria |
放线菌纲 Actinobacteria |
棒状杆菌目 Corynebacteriales |
迪茨氏菌科 Dietziaceae |
迪茨氏菌属 Dietzia |
海迪茨氏菌 Dietzia_maris |
研究在南海深海海水样中变形菌门细菌种类是相对丰度最高的细菌类群, 这与国内外很多研究相一致[20—22]。研究采样点的所有测序数据比对后共注释了12门, 共计317科的细菌, 而不论基于测序数据量最大的变形菌门中的细菌主要注释仅仅11科, 或注释种类最多的厚壁菌门中其细菌仅分属于6科, 由此说明海洋深层水样品中细菌种类丰富, 且大部分仅可以被注释到门的分类水平。
属以下鉴定种相对丰度较高包括类芽胞杆菌属、芽孢杆菌属、海旋菌属、噬甲基菌属、拟杆菌属、纤细芽胞杆菌属、海洋芽胞杆菌属、产微球茎菌属、嗜油菌属、食碱菌属。这些属的细菌中, 类芽胞杆菌属前期已在南海水域被鉴定分离出一种固氮细菌[23]。芽孢杆菌属在海水中发现了地衣芽胞杆菌[24]。海旋菌属细菌可以为潜在的有机污染物降解菌[25]。噬甲基菌属可以作为固氮细菌在水质处理中发挥作用[26], 可以分泌铵态氮促进藻类生长功能[27]。拟杆菌属的深海冷泉拟杆菌可通过降解藻类多糖促进深海营养和碳循环[28]。纤细芽胞杆菌属的功能有待进一步研究。海洋芽孢杆菌次级代谢产物中具有抗生素类生物活性物质的开发利用奠定了一定的生物学和化学基础[29]。产微球茎菌属在近海有分布, 研究发现其可以产几丁质酶[30—31]。嗜油菌属细菌在墨西哥湾超过1000 m得深海处有发现, 其具有分解石油的作用[32], 并且海洋嗜油菌可以产生L-苏氨酸醛缩酶[33]。洋微生物食碱菌属具有趋化蛋白元件可以被用在环境修复中[34]。
此海洋深层水水样中目前具体可以确定的海洋深层水细菌有海洋解淀粉芽孢杆菌, 施氏假单胞菌, 柴油食烷菌和三浦半岛盐乳杆菌和迪茨氏菌。海洋解淀粉芽孢杆菌可以生产抗生素Macrolactin A[35], 并且对球等鞭金藻生长抑制效果, 或可用于赤潮藻类治理[36], 也是防治大豆疫病的潜在生防菌株[37]。施氏假单胞菌可以产卡拉胶酶[38], 亦可降解环境污染物[39—41]。柴油食烷菌可以降解卤代化合物[42], 具有很强的石油降解能力[43, 44]。三浦半岛盐乳杆菌目前应用较少。迪茨氏菌可用于降解原油[45—46], 并且具有反硝化作用[47]。对于属水平的这些优势品种或者鉴定的具体种, 它们在特定环境中具有较高竞争力和适应性, 这些细菌在生态系统中扮演着重要的角色, 具有较高的研究价值和应用价值, 可能在生态系统, 生物防治和农业应用, 工业应用, 医学和药物研究, 生物勘探和生物多样性研究中发挥作用。
4 结论研究表明海洋深层水域的细菌丰富度极高, 但对其进行分子鉴定的工作还远远不够, 有赖于细菌的纯化和培养, 这对于大量未鉴定到种的细菌的进一步鉴定及开发利用十分重要。本研究丰富了海洋深层水微生物多样性研究的基础数据, 有助于我们更深入地理解深海环境微生物多样性。加强这些细菌的研究和应用, 有助于促进环境保护、农业可持续发展和人类健康等方面的进步。
| [1] |
Whitman W B, Coleman D C, Wiebe W J. Prokaryotes: the unseen majority. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(12): 6578-6583. |
| [2] |
Lauro F M, McDougald D, Thomas T, Williams T J, Egan S, Rice S, DeMaere M Z, Ting L, Ertan H, Johnson J, Ferriera S, Lapidus A, Anderson I, Kyrpides N, Munk A C, Detter C, Han C S, Brown M V, Robb F T, Kjelleberg S, Cavicchioli R. The genomic basis of trophic strategy in marine bacteria. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(37): 15527-15533. |
| [3] |
韩妍妍, 张亚娟, 王维娜, 王安利. 海洋微生物是开发海洋药物的重要资源. 海洋科学, 2002, 26(9): 7-12. DOI:10.3969/j.issn.1000-3096.2002.09.003 |
| [4] |
Tsuchiya Y, Nakamura K, Sekikawa H, Kawamura H, Miyanishi K, Ishizu T, Yamamoto M. Subacute effects of deep-sea water from the Japan Sea on blood examination values in mice. Environmental Health and Preventive Medicine, 2002, 7(5): 189-192. DOI:10.1007/BF02898003 |
| [5] |
李越中, 陈琦. 海洋微生物资源多样性. 生物工程进展, 1998, 18(4): 34-40. |
| [6] |
Fenchel T. Marine microbiology: ecology and applications Colin Munn (with foreword by Farooq Azam). London and New York: BIOS Scientific Publishers, 2003.
|
| [7] |
李学恭, 张维佳, 周丽红, 蔡凤海, 吴龙飞. 不同降压过程对深海海水中可培养细菌群落组成的影响. 微生物学报, 2019, 59(6): 1026-1035. |
| [8] |
李涛, 王鹏, 汪品先. 南海西沙海槽沉积物细菌多样性初步研究. 地球科学进展, 2006, 21(10): 1058-1062. DOI:10.3321/j.issn:1001-8166.2006.10.009 |
| [9] |
李涛, 王鹏, 汪品先. 南海南部陆坡表层沉积物细菌和古菌多样性. 微生物学报, 2008, 48(3): 323-329. DOI:10.3321/j.issn:0001-6209.2008.03.009 |
| [10] |
李涛, 王鹏, 汪品先. 南海西沙海槽表层沉积物微生物多样性. 生态学报, 2008, 28(3): 1166-1173. DOI:10.3321/j.issn:1000-0933.2008.03.032 |
| [11] |
张浩, 吴后波, 王广华, 向文洲, 颜文. 南海北部表层沉积物中原核微生物多样性. 微生物学报, 2013, 53(9): 915-926. |
| [12] |
曾奇, 仲伟茂, 向瑶, 陈夏雨, 田新朋, 张偲, 王发左. 南海深海沉积物中52株真菌的初步分离鉴定及其代谢产物活性. 微生物学通报, 2018, 45(9): 1904-1915. |
| [13] |
Zhang Y, Su X, Chen F, Wang Y Y, Jiao L, Dong H L, Huang Y Y, Jiang H C. Microbial diversity in cold seep sediments from the northern South China Sea. Geoscience Frontiers, 2012, 3(3): 301-316. DOI:10.1016/j.gsf.2011.11.014 |
| [14] |
夏呈强, 李毅, 党延茹, 察倩倩, 贺晓艳, 秦启龙. 中印度洋与南海西部表层海水细菌多样性. 生物多样性, 2022, 30(1): 87-96. |
| [15] |
孙涛, 付婧, 周进. 南海近海浮游细菌群落结构和多样性研究. 海洋渔业, 2021, 43(6): 693-706. DOI:10.3969/j.issn.1004-2490.2021.06.006 |
| [16] |
Bokulich N A, Kaehler B D, Rideout J R, Dillon M, Bolyen E, Knight R, Huttley G A, Gregory Caporaso J. Optimizing taxonomic classification of marker-gene amplicon sequences with QⅡME 2's q2-feature-classifier plugin. Microbiome, 2018, 6(1): 90. DOI:10.1186/s40168-018-0470-z |
| [17] |
Callahan B J, McMurdie P J, Rosen M J, Han A W, Johnson A J A, Holmes S P. DADA2: high-resolution sample inference from Illumina amplicon data. Nature Methods, 2016, 13: 581-583. DOI:10.1038/nmeth.3869 |
| [18] |
Quast C, Pruesse E, Yilmaz P, Gerken J, Schweer T, Yarza P, Peplies J, Glöckner F O. The SILVA ribosomal RNA gene database project: improved data processing and web-based tools. Nucleic Acids Research, 2013, 41(D1): D590-D596. |
| [19] |
Bolyen E, Rideout J R, Dillon M R, Bokulich N A, Abnet C C, Al-Ghalith G A, Alexander H, Alm E J, Arumugam M, Asnicar F, Bai Y, Bisanz J E, Bittinger K, Brejnrod A, Brislawn C J, Brown C T, Callahan B J, Caraballo-Rodríguez A M, Chase J, Cope E K, Da Silva R, Diener C, Dorrestein P C, Douglas G M, Durall D M, Duvallet C, Edwardson C F, Ernst M, Estaki M, Fouquier J, Gauglitz J M, Gibbons S M, Gibson D L, Gonzalez A, Gorlick K, Guo J R, Hillmann B, Holmes S, Holste H, Huttenhower C, Huttley G A, Janssen S, Jarmusch A K, Jiang L J, Kaehler B D, Bin Kang K, Keefe C R, Keim P, Kelley S T, Knights D, Koester I, Kosciolek T, Kreps J, Langille M G I, Lee J, Ley R, Liu Y X, Loftfield E, Lozupone C, Maher M, Marotz C, Martin B D, McDonald D, McIver L J, Melnik A V, Metcalf J L, Morgan S C, Morton J T, Naimey A T, Navas-Molina J A, Nothias L F, Orchanian S B, Pearson T, Peoples S L, Petras D, Preuss M L, Pruesse E, Rasmussen L B, Rivers A, Robeson M S, Rosenthal P, Segata N, Shaffer M, Shiffer A, Sinha R, Song S J, Spear J R, Swafford A D, Thompson L R, Torres P J, Trinh P, Tripathi A, Turnbaugh P J, Ul-Hasan S, van der Hooft J J J, Vargas F, Vázquez-Baeza Y, Vogtmann E, von Hippel M, Walters W, Wan Y H, Wang M X, Warren J, Weber K C, Williamson C H D, Willis A D, Xu Z Z, Zaneveld J R, Zhang Y L, Zhu Q Y, Knight R, Caporaso J G. Reproducible, interactive, scalable and extensible microbiome data science using QⅡME 2. Nature Biotechnology, 2019, 37: 852-857. DOI:10.1038/s41587-019-0209-9 |
| [20] |
Sogin M L, Morrison H G, Huber J A, Mark Welch D, Huse S M, Neal P R, Arrieta J M, Herndl G J. Microbial diversity in the deep sea and the underexplored "rare biosphere". Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(32): 12115-12120. |
| [21] |
Murray A E, Grzymski J J. Diversity and genomics of Antarctic marine micro-organisms. Philosophical Transactions of the Royal Society of London Series B, Biological Sciences, 2007, 362(1488): 2259-2271. DOI:10.1098/rstb.2006.1944 |
| [22] |
Zeng Y X, Zou Y, Grebmeier J M, He J F, Zheng T L. Culture-independent and-dependent methods to investigate the diversity of planktonic bacteria in the northern Bering Sea. Polar Biology, 2012, 35(1): 117-129. DOI:10.1007/s00300-011-1044-8 |
| [23] |
张丽红, 刘艳芳, 胡青平. 中国南海一株固氮类芽孢杆菌的筛选和分离鉴定. 海洋科学, 2015, 39(3): 100-105. |
| [24] |
傅奇, 林俊杰, 甘美裕, 卢源钦, 肖玉娟. 16S与gyrB基因联合建树快速鉴定海水中的一株地衣芽胞杆菌. 海洋科学, 2020, 44(4): 90-95. |
| [25] |
Wu S S, Zhu W J, Wang C, Suo C L, Zhang W P, Li C Y, Fu H H, Zhang Y, Sun M L, Wang P. Genomic analysis of Thalassospira sp. SW-3-3 reveals its genetic potential for phthalate pollution remediation. Marine Genomics, 2022, 63: 100953. DOI:10.1016/j.margen.2022.100953 |
| [26] |
祁自忠, 杨匡, 程成, 刘子夏, 韩茵. 固定化硝化菌群联合芽孢杆菌处理对虾养殖废水. 微生物学通报, 2018, 45(9): 1922-1939. |
| [27] |
魏晓雪, 石峰, 陈子熙, 冯剑丰, 朱琳. 三角褐指藻及其藻际细菌对不同无机氮源的响应. 海洋科学, 2022, 46(1): 10-21. |
| [28] |
Zheng R K, Cai R N, Liu R, Liu G, Sun C M. Maribellus comscasis sp. nov., a novel deep-sea Bacteroidetes bacterium, possessing a prominent capability of degrading cellulose. Environmental Microbiology, 2021, 23(8): 4561-4575. |
| [29] |
高伟, 田黎, 周俊英, 史振平, 郑立, 崔志松, 李元广. 海洋芽孢杆菌(Bacillus marinus)B-9987菌株抑制病原真菌机理. 微生物学报, 2009, 49(11): 1494-1501. |
| [30] |
Wang J Q, Jin L X, Wang J X, Chan Z H, Zeng R Y, Wu J, Qu W. The first complete genome sequence of Microbulbifer celer KCTC12973T, a type strain with multiple polysaccharide degradation genes. Marine Genomics, 2022, 62: 100931. |
| [31] |
杨锦, 沈宏. 海藻酸裂解酶高产菌株Microbulbifer sp.SH-1的分离、鉴定及其产酶条件优化. 微生物学报, 2020, 60(4): 679-694. |
| [32] |
Hazen T C, Dubinsky E A, DeSantis T Z, Andersen G L, Piceno Y M, Singh N, Jansson J K, Probst A, Borglin S E, Fortney J L, Stringfellow W T, Bill M, Conrad M E, Tom L M, Chavarria K L, Alusi T R, Lamendella R, Joyner D C, Spier C, Baelum J, Auer M, Zemla M L, Chakraborty R, Sonnenthal E L, D'Haeseleer P, Holman H Y N, Osman S, Lu Z M, Van Nostrand J D, Deng Y, Zhou J Z, Mason O U. Deep-sea oil plume enriches indigenous oil-degrading bacteria. Science, 2010, 330(6001): 204-208. |
| [33] |
He Y Z, Li S Y, Wang J, Yang X R, Zhu J W, Zhang Q, Cui L, Tan Z G, Yan W P, Zhang Y, Tang L Y, Da L T, Feng Y. Discovery and engineering of the l-threonine aldolase from Neptunomonas marine for the efficient synthesis of β-hydroxy-α-amino acids via C-C formation. ACS Catalysis, 2023, 13(11): 7210-7220. |
| [34] |
唐鸿志, 王伟伟, 张莉鸽, 黄玲, 陆歆毓, 许平. 合成生物学在环境修复中的应用. 生物工程学报, 2017, 33(3): 506-515. |
| [35] |
侯东园, 吴祖芳, 张鑫. 海洋解淀粉芽孢杆菌(Bacillus amyloliquefacien)产抗生素Macrolactin A的碳源优化. 海洋与湖沼, 2015, 46(5): 1071-1077. |
| [36] |
李晓英, 陈义华, 赵裕超, 冯森磊, 李雯倩, 朱强, 董志国. 解淀粉芽孢杆菌化感作用对球等鞭金藻生长抑制效果. 海洋科学, 2021, 45(1): 62-69. |
| [37] |
林巧玲, 卢乃会, 何红, 袁越. 海洋细菌解淀粉芽胞杆菌SH-27在大豆体内的定殖动态及促生防病作用. 中国生物防治学报, 2019, 35(2): 240-246. |
| [38] |
彭小珍, 吉宏武, 刘唤明, 章超桦. 施氏假单胞菌Pseudomonas stutzeri产卡拉胶酶液体发酵条件优化. 食品研究与开发, 2007, 28(10): 7-11. |
| [39] |
兰亚红, 谢明, 陈福良, 万方浩, 周淑云. 施氏假单胞菌JHY01菌株毒死蜱降解酶的定位及其提取条件的优化. 中国生物防治, 2008, 24(4): 349-353. |
| [40] |
史延华, 任磊, 贾阳, 赵百锁, 闫艳春. 施氏假单胞菌YC-YH1的萘降解特性及产物分析. 微生物学通报, 2015, 42(10): 1866-1876. |
| [41] |
周晓黎, 孙迎雪, 沈丹丹, 杨哲, 田媛. 一株施氏假单胞菌Pseudomonas stutzeri DN-LWX19的脱氮性能. 环境工程学报, 2015, 9(1): 247-252. |
| [42] |
李安章, 邵宗泽. 柴油食烷菌B-5及其卤代烷烃脱卤酶DadA对卤代化合物的降解. 微生物学报, 2014, 54(9): 1063-1072. |
| [43] |
吴业辉, 邵宗泽. 海洋烷烃降解菌Alcanivorax sp. A-11-3的分离鉴定及其降解酶基因研究. 台湾海峡, 2008, 27(4): 427-434. |
| [44] |
吴秉奇, 刘淑杰, 陈福明, 周楚莹. 海洋石油降解菌的筛选及复合菌系的构建. 生物技术通报, 2016, 32(8): 184-193. |
| [45] |
周翔, 谢佳蓉, 梁剑平, 辛志君, 陆锡宏, 郭志廷, 郝宝成, 杨贤鹏, 赵美荣. 基于3D响应曲面法与重离子辐射优化海迪茨氏菌降解原油的研究. 环境科学学报, 2013, 33(9): 2473-2481. |
| [46] |
周翔, 梁剑平, 辛志君, 陆锡宏, 杨贤鹏, 郭志廷, 郝宝成. 12C6+重离子诱变海迪茨氏菌及其修复柴油污染土壤的研究. 核农学报, 2013, 27(6): 823-830. |
| [47] |
郭艳丽, 张培玉, 于德爽, 曲洋, 郭沙沙, 成广勇. 一株轻度嗜盐反硝化菌的分离鉴定及特性. 应用与环境生物学报, 2010, 16(3): 394-398. |
 2024, Vol. 44
2024, Vol. 44