文章信息
- 段春健, 宛颂, 樊剑波, 叶桂萍, 林永新, 贺纪正
- DUAN Chunjian, WAN Song, FAN Jianbo, YE Guiping, LIN Yongxin, HE Jizheng
- 长期施用猪粪对红壤完全氨氧化菌基因丰度的影响
- Effects of long-term pig manure applications on the abundance of comammox bacteria in an Ultisol
- 生态学报. 2023, 43(2): 849-857
- Acta Ecologica Sinica. 2023, 43(2): 849-857
- http://dx.doi.org/10.5846/stxb202107031772
-
文章历史
- 收稿日期: 2021-07-03
- 网络出版日期: 2022-09-26
2. 福建师范大学地理科学学院, 福州 350007;
3. 中国科学院南京土壤研究所, 南京 210008;
4. 闽江学院海洋研究院, 福州 350108
2. School of Geographical Sciences, Fujian Normal University, Fuzhou 350007, China;
3. Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, China;
4. Institute of Oceanography, Minjiang University, Fuzhou 350108, China
硝化作用是氮循环中的一个关键过程, 占陆地生态系统总氮通量的15%以上[1]。传统的硝化作用被认为是一个两步过程: 氨氧化细菌(Ammonia oxidizing bacteria, AOB)或氨氧化古菌(Ammonia oxidizing archaea, AOA), 先将NH3氧化成NO2-, 然后由亚硝酸盐氧化细菌(Nitrite oxidizing bacteria, NOB)将NO2-氧化成NO3-[2]。然而, 单一微生物将氨氧化成硝酸盐在理论上是可行的, 能量效率也是更高的。但直到2015年, 能够独立将氨氧化为硝酸盐的完全氨氧化菌(comammox Nitrospira)的发现, 才从根本上打破了硝化作用必须由两类微生物分工完成这一百年范式[3], 为硝化微生物的研究带来新的机遇与挑战。通过对amoA的系统发育分析, 完全氨氧化菌可以分为进化枝A(clade A)和进化枝B(clade B), 而进化枝A可以进一步分为进化枝A.1和进化枝A.2[4]。尽管已有大量研究发现完全氨氧化菌在不同生境中广泛分布, 但完全氨氧化菌进化枝A和进化枝B在土壤中的分布规律和功能活性尚不清楚, 仍需研究进一步阐述[5—6]。
迄今为止, 农田土壤氨氧化古菌和氨氧化细菌的分布规律和功能活性研究已取得长足进展。氨氧化古菌通常在酸性农田土壤中占据主导地位, 其丰度被发现与硝化活性呈显著正相关[7]; 而氨氧化细菌对氨的亲和力较低, 通常在氮含量较高的碱性土壤中更占优势[8]。然而, Huang等[9]研究发现, 氨氧化细菌在酸性土壤中也可以发挥重要作用, 表明酸性土壤中氨氧化细菌在硝化过程中的功能活性仍存在一定争议。相比于氨氧化古菌和氨氧化细菌, 人们对农田土壤完全氨氧化菌的认识则远远不足。目前, 完全氨氧化菌在农田土壤中的分布规律和功能活性存在较大争议, 例如:Li等[10]通过对澳大利亚的农田土壤研究发现完全氨氧化菌的丰度比氨氧化古菌和氨氧化细菌高, 而Xu等[11]对中国不同农田土壤的研究发现完全氨氧化菌的丰度低于氨氧化古菌和氨氧化细菌。此外, Kits等[12]发现, 完全氨氧化菌对氨的亲和力高于氨氧化古菌和氨氧化细菌, 可能在寡营养条件下更具竞争力, 而Li等[10]和Zhou等[13]则发现完全氨氧化菌在高氮的农田土壤中发挥重要功能, 表明存在喜好高氮环境的完全氨氧化菌。农田红壤广泛分布于我国南方地区, 具有酸性较强, 肥力条件差的典型特点[14]。为了提高作物产量, 大量化肥施入红壤中, 使得土壤进一步酸化, 威胁农业的可持续发展。施用有机肥是维持作物产量, 保护土壤肥力的重要手段。Lin等[15]发现, 相比于秸秆和绿肥, 施用猪粪可以更有效地促进土壤团聚体形成, 提高土壤有机质和养分含量。那么, 施用猪粪如何影响氨氧化微生物, 尤其是完全氨氧化菌丰度?主控因素为何?
基于以上科学问题, 本研究依托中国科学院鹰潭红壤生态试验站长期猪粪施用定位试验田, 通过实时荧光定量PCR的方法, 研究了长期施用猪粪对氨氧化微生物功能基因丰度的影响。旨在阐明猪粪添加对旱地红壤氨氧化微生物功能基因, 尤其是完全氨氧化菌功能基因丰度的影响。以期综合评估农田红壤氨氧化微生物对长期施用猪粪的响应, 为该地区养分管理提供数据支持。
1 材料与方法 1.1 研究区概况研究地点位于江西省鹰潭市中国科学院鹰潭红壤生态实验站(28°15′20″N, 116°55′30"E)。该地区具有典型的亚热带季风气候, 年平均降雨量为1795 mm, 年平均温度为17.6℃。土壤为第四纪发育而来的红色黏土, 属于典型的老成土(Ultisol)。其中粘粒占41.2%、粉砂占33.2%、砂粒占25.6%。
1.2 试验设计与样品采集试验样地建立于2002年, 采用夏季花生, 冬季休眠的耕种方式。试验设计采用随机区组的方法, 在田间共设4个处理, 每个处理各设计3个重复:对照不施肥(CK); 低量粪肥(M9), 施用9 Mg/hm2的猪粪; 中量粪肥(M18), 施用18 Mg/hm2的猪粪; 和高量粪肥(M27), 施用27 Mg/hm2的猪粪。猪粪在当地收集, 使用前进行3个月的存储发酵。猪粪含有机碳(SOC)157 g/kg, 总氮(TN)20.1 g/kg, 总磷(TP)12.1 g/kg, 全钾(TK)7.90 g/kg。
土壤样品采集于2019年10月, 采用蛇形采样的方法, 在各个小区内选取10个样点, 采样深度0—20 cm, 使用土钻采取10个完整的土芯, 彻底混合, 形成一个均匀的混合样品, 放置于自封袋中。后将样本放在冰上, 立即送到实验室。仔细挑去土壤中肉眼可见的有机碎片和杂质后, 将样品分为三份, 一部分样品风干, 用于测定土壤理化性质; 一部分鲜土立刻用于测定土壤净硝化速率; 另一部分新鲜土样, 放置于-80℃, 进行后续的分子生物学分析。
1.3 土壤理化性质的测定土壤基本理化性质的分析按照常规方法进行[16], 简要操作步骤如下:取风干土样, 烘干至恒重, 测定土壤含水量; 土壤pH值测定:取5 g风干土样, 加入除去CO2的去离子水, 震荡后使用pH计(FE20-FiveEasyTMpH, MettlerToledo, German)进行测定。可溶性有机碳(DOC)测定:取当量10 g新鲜土样, 按照土水比1∶5提取, 震荡、过滤后, 利用岛津碳氮分析仪(TOC Vcph, Shimadzu, Kyoto Japan)测定。土壤硝态氮(NO3--N)和铵态氮(NH4+-N)含量测定:取相当于5 g干土的新鲜土样, 加入25 mL 2 mol/L的KCl溶液, 震荡、过滤后用流动分析仪(San++ System, Skalar, Holland)测定。总氮(TN)、有机碳(SOC)测定:将自然风干后的土样, 研磨通过100目筛, 利用碳氮元素分析仪(VarioMAX, Elementar, Germany)测定。土壤速效磷(AP)含量测定:称取5 g风干土样, 利用50 mL 0.05 mol/L HCl和0.025 mol/L H2SO4混合液提取, 钼蓝比色法测定。土壤速效钾(AK)含量测定:称取5 g风干土样, 利用50 mL 1 mol/L醋酸铵提取, 火焰光度计(FP640, INASA, China)测定。
1.4 土壤净硝化速率测定土壤净硝化速率的简要测定方法[17]如下:称取当量5 g土样至120 mL培养瓶中, 加入硫酸铵, 控制氮素添加的最终浓度为50 mg/kg, 调节含水量至田间最大持水量的60%。将培养瓶分为两组, 一组立即加入25 mL 2 mol/L的KCl溶液, 震荡、离心、过滤后用流动分析仪测定NO3--N浓度, 记作C1;另一组置于25℃培养箱中, 黑暗避光条件下培养7d, 用同样的方法测定NO3--N浓度, 记作C2。最后根据下列公式计算净硝化速率:

|
其中, Rn表示净硝化速率(mg kg-1 d-1), V表示KCl的体积, C1表示培养前NO3--N浓度(mg/L), C2表示培养7d后NO3--N浓度(mg/L), W表示样品重量(g), T表示培养时间(d)。
1.5 土壤总DNA提取土壤总DNA提取利用MoBio PowerSoilTM DNA Isolation Kits(Mo Bio Laboratories, Carlsbad, CA, USA)试剂盒提取, 称取0.25 g解冻的新鲜土壤, 按照说明手册的操作步骤提取土壤总DNA。将提取的DNA放置于-80℃冰箱, 用于后续实验分析。
1.6 实时荧光定量PCR采用实时荧光定量PCR(Real-time quantitative PCR)的方法, 利用CFX96 Optical Real-Time Detection System(Bio-Rad Laboratories Inc, HerculesCA, USA)定量PCR仪, 测定氨氧化微生物的功能基因丰度。反应体系为20 μL, 包括:10 μL SYBR Premix、0.2 μL前引物、0.2 μL后引物、8.6 μL灭菌水及1 μL DNA模板, 定量PCR所使用引物序列和扩增条件如表 1所示。在定量过程中, 每个样品和标准曲线模板设置三个重复, 并设置三个阴性对照。
| 微生物 Target group |
引物序列 Primer sequences |
热循环条件 Thermal conditions |
参考文献 Reference |
| 氨氧化古菌 AOA |
Arch-amoAF: STAATGGTCTGGCTTAGACG Arch-amoAR: GCGGCCATCCATCTGTATGT |
95℃预变性5 min; 95℃变性30 s, 55℃退火30 s, 72℃延伸1 min, 40个循环; 熔解曲线:温度从65.0℃上升到95.0℃, 每5 s增加0.5℃ | [18] |
| 氨氧化细菌 AOB |
amoA-1F: GGGGTTTCTACTGGTGGT amoA-2R: CCCCTCKGSAAAGCCTTCTTC |
95℃预变性5 min; 95℃变性30 s, 55℃退火30 s, 72℃延伸1 min, 40个循环; 熔解曲线:温度从65.0℃上升到95.0℃, 每5 s增加0.5℃ | [19] |
| 完全氨氧化菌分支A Clade A |
CA377f: GTGGTGGTGGTCBAAYTA C576r: GAAGCCCATRTARTCNGCC |
95℃预变性3 min; 95℃变性10 s, 55℃退火20 s, 72℃延伸15 s, 39个循环; 熔解曲线:温度从65.0℃上升到95.0℃, 每5 s增加0.5℃ | [20] |
| 完全氨氧化菌分支B Clade B |
CB377f: GTACTGGTGGGCBAAYTT C576r: GAAGCCCATRTARTCNGCC |
95℃预变性3 min; 95℃变性10 s, 55℃退火20 s, 72℃延伸15 s, 39个循环; 熔解曲线:温度从65.0℃上升到95.0℃, 每5 s增加0.5℃ | [20] |
标准曲线的制备方法如下:首先对目的基因进行扩增, 克隆获取含有目的基因的质粒, 后将其导入大肠杆菌, 在恒温条件下培养, 然后提取质粒DNA, 用NanoDrop ND-2000检验核酸浓度, 根据质粒浓度和片段等信息计算质粒的基因拷贝数。在实时荧光定量PCR过程中, 对质粒进行10倍梯度稀释, 根据不同质粒浓度和反应的循环数(Ct值)得到标准曲线, 以此计算样本基因拷贝数。扩增过程中溶解曲线始终出现单峰, 扩增效率为90%—100%。
1.7 数据分析利用SPSS 18.0软件进行数据分析统计。处理间基本理化性质、净硝化速率和氨氧化微生物功能基因丰度的差异利用单因素方差分析(ANOVA)进行比较, 处理间的显著性利用Least significant difference(LSD)进行检验。利用皮尔森(Pearson)相关分析, 研究土壤基本理化性质、净硝化速率和功能基因丰度之间的相关关系。采用Origin 2018软件进行绘图。
2 结果 2.1 施用猪粪对土壤理化性质和净硝化速率的影响长期施用猪粪显著改变红壤基本理化性质(表 2)。CK的pH值为4.95, M9和M18分别显著降低土壤pH至4.80和4.79, 而M27则没有。CK的DOC和SOC含量分别为5.74 mg/kg和7.52 mg/kg, 长期施用猪粪提高SOC和DOC含量, 且提升量随着猪粪用量的增加而增加。CK处理的TN、NH4+和NO3-含量同样最低, 长期施用猪粪显著提高TN、NH4+和NO3-含量, 且M27处理的提升效果最好。CK处理AP含量为6.39 mg/kg, 长期施用猪粪均显著提高AP含量, 提升效果随着猪粪用量的增加而增加。CK处理的净硝化速率为0.33 mg kg-1 d-1(图 1), M9处理对硝化速率无显著影响, 而M18和M27处理显著提高土壤净硝化速率, 增幅分别达到317%和416%。
| 处理 Treatment |
pH (1∶5) |
可溶性有机碳 DOC/ (mg/kg) |
土壤有机碳 SOC/ (mg/kg) |
全氮 TN/ (g/kg) |
铵态氮 NH4+/ (mg/kg) |
硝态氮 NO3-/ (mg/kg) |
速效磷 AP/ (mg/kg) |
速效钾 AK/ (mg/kg) |
| CK | 4.95±0.06b | 5.74±0.71a | 5.68±0.33a | 0.66±0.02a | 7.52±0.09a | 5.95±0.17a | 6.39±0.97a | 76.70±12.4a |
| M9 | 4.80±0.02a | 8.22±0.74ab | 6.61±0.42ab | 0.81±0.07ab | 8.31±0.42ab | 8.86±0.84b | 28.50±5.49b | 87.50±3.36a |
| M18 | 4.79±0.01a | 10.40±1.27b | 6.73±0.25bc | 0.81±0.05b | 9.40±0.73b | 11.20±0.67c | 49.80±4.36c | 129.00±3.08b |
| M27 | 4.90±0.03ab | 14.00±0.41c | 7.67±0.12c | 0.90±0.04b | 9.62±0.30b | 11.80±0.83c | 105.00±14.5d | 150.00±11.7b |
| 平均值±标准误(n=3), 同列不同字母表示差异显著(P < 0.05); CK:对照; M9:施用9 Mg/hm2的猪粪; M18:施用18 Mg/hm2的猪粪; M27:施用27 Mg/hm2的猪粪 | ||||||||

|
| 图 1 长期施用猪粪对红壤净硝化速率的影响 Fig. 1 Effects of long-term application of pig manure on net nitrification rate in Ultisols 不同字母表示差异显著(P < 0.05); CK:对照; M9:施用9 Mg/hm2的猪粪; M18:施用18 Mg/hm2的猪粪; M27:施用27 Mg/hm2的猪粪 |
CK处理的氨氧化古菌amoA丰度为4.53×108 copies/g(图 2), M18处理显著提升氨氧化古菌amoA丰度至8.07×108 copies/g, 而M9和M27对氨氧化古菌amoA丰度无显著影响。氨氧化细菌amoA基因丰度在CK处理中含量最低, 随着猪粪用量的增加, 氨氧化细菌 amoA丰度逐步提高, 但均无显著性差异。完全氨氧化菌进化枝A的amoA丰度在CK处理中丰度为3.84×106 copies/g, M9和M18处理虽然提高了完全氨氧化菌进化枝A的amoA基因丰度, 但并未达到显著性差异, 而M27处理则显著提升完全氨氧化菌进化枝A的amoA基因丰度至1.19×107 copies/g。氨氧化古菌的amoA丰度在各个处理中均高于氨氧化细菌和完全氨氧化菌进化枝A, 高出约1—2个数量级。在实时荧光定量PCR过程中, 尽管尝试多种扩增条件, 各个处理的完全氨氧化菌进化枝B都存在很强的非特异性扩增, 凝胶电泳呈现的条带杂乱, 目标条带不存在或很弱, 因此将其排除在后续分析中。

|
| 图 2 长期施用猪粪对红壤硝化功能基因丰度的影响 Fig. 2 Effects of long-term application of pig manure on amoA gene abundances in Ultisols 不同字母表示差异显著(P<0.05); CK:对照; M9:施用9 Mg/hm2的猪粪; M18:施用18 Mg/hm2的猪粪; M27:施用27 Mg/hm2的猪粪; AOA:氨氧化古菌; AOB:氨氧化细菌; Clade A:完全氨氧化菌进化支A |
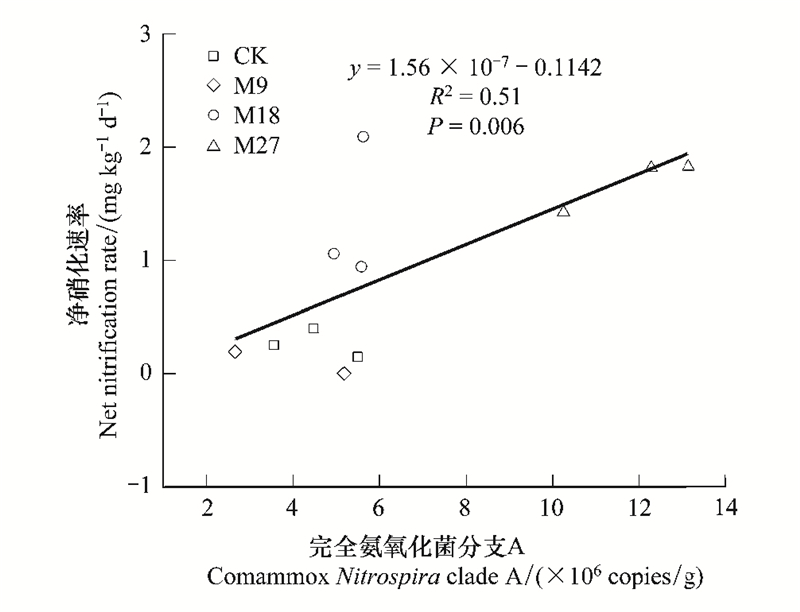
相关性分析表明(表 3), 氨氧化古菌的基因丰度与pH值呈显著负相关(P < 0.05), 与NH4+、NO3-、TN呈显著正相关(P < 0.05), 而与SOC、AP、AK无显著相关性。氨氧化细菌amoA基因丰度与DOC、AP呈显著正相关(P < 0.05), 而与其余理化性质无显著相关性。完全氨氧化菌进化枝A的amoA基因丰度与pH值无相关性, 与其他土壤理化性质呈显著正相关(P < 0.05)。逐步回归分析表明, pH值是影响氨氧化古菌amoA基因丰度的主要环境因子, 可解释其41.1%的变异; DOC是影响氨氧化细菌amoA基因丰度的主要因子, 可解释其43.0%的变异; AP是影响完全氨氧化菌进化枝A amoA基因丰度的重要因子, 可解释86.5%的变异。此外, 相关性分析发现(图 3)氨氧化古菌和氨氧化细菌的amoA基因丰度与净硝化速率无显著相关关系, 而完全氨氧化菌进化枝A的amoA基因丰度与净硝化速率呈显著正相关(P < 0.01)。
| 微生物 Target group |
pH (1∶5) |
土壤 有机碳 SOC |
可溶性 有机碳 DOC |
全氮 TN |
铵态氮 NH4+ |
硝态氮 NO3- |
速效磷 AP |
速效钾 AK |
| 氨氧化古菌AOA | -0.681* | 0.364 | 0.464 | 0.611* | 0.596* | 0.657* | 0.251 | 0.374 |
| 氨氧化细菌AOB | 0.107 | 0.694* | 0.569 | 0.521 | 0.484 | 0.527 | 0.629* | 0.403 |
| 完全氨氧化菌分支A Clade A | 0.168 | 0.877** | 0.840** | 0.739** | 0.654* | 0.698* | 0.937** | 0.782** |
| *和**分别表示P < 0.05和P < 0.01; AOA:氨氧化古菌; AOB:氨氧化细菌; Clade A:完全氨氧化菌进化枝A | ||||||||
|
| 图 3 完全氨氧化菌进化枝A amoA基因丰度与净硝化速率的相关关系 Fig. 3 Correlation between comammox Nitrospira clade A amoA gene abundance and net nitrification rate |
施用有机肥是调控土壤养分含量及其稳定性的重要农业管理措施, 猪粪作为一种重要的有机肥, 对改善土壤质地和养分含量有着较为明显的作用。本研究发现, 猪粪的施用使土壤pH值呈下降趋势, 这可能是因为猪粪中的有机质被慢慢降解, 有机官能团释放出大量H+, 导致土壤pH值下降[21]; 同时, 猪粪施用促进土壤硝化活性, 从而释放出H+, 使土壤进一步酸化, pH值降低。另一方面, 随着猪粪施用量的增加, 有机物质和腐殖质含量逐步提高, 有机质和官能团可以络合金属离子和H+, 腐殖质的缓冲作用一定程度缓解土壤酸化[22]。因此, 高量粪肥施用没有显著降低土壤pH值可能是由于未分解或半分解的有机物质的缓冲作用导致的。大量研究表明, 猪粪等有机肥的施用, 可以改善土壤结构, 增加土壤有机质和碳氮磷等元素含量[23—24]。然而, 随着猪粪的施用量的增加, 土壤中碳和养分的固持逐渐趋近于饱和[25]; 土壤中部分养分的外源输入与植物吸收、微生物利用和淋溶损失等输出达到一个动态平衡的状态[26]。因此, 相比于M18, M27并没有提高部分养分的含量。综上所述, 单独施用猪粪并不能改善红壤的酸化情况, 但可以显著提升红壤有机质和养分含量, 有利于农田红壤作物的生长发育。同时也要注意猪粪用量, 避免不必要的肥料和养分损失, 以及过量施用带来的二次污染。
3.2 施用猪粪对氨氧化微生物基因丰度的影响供试土壤中, 氨氧化古菌amoA的基因丰度比氨氧化细菌和完全氨氧化菌高出1—2个数量级, 表明在酸性红壤中, 氨氧化古菌是丰度最高的氨氧化微生物。氨氧化古菌含有酸碱平衡的目的基因和高亲和氨的获取系统[27], 该基因编码的膜系统可通过对钾离子的吸收, 产生反膜电位, 在低pH值条件下维持氨氧化古菌稳态[28], 而对NH3的高亲和力, 保证了氨氧化古菌可以在NH3浓度较低的酸性环境中生存。因此, 在酸性红壤当中, 氨氧化古菌amoA基因丰度最高。红壤氨氧化古菌amoA基因丰度主要受土壤pH值和养分条件限制, 猪粪施用后, 虽然向土壤中带入大量有机质, 分解产生NH3, 但在酸性条件下NH3易与土壤中H+结合形成NH4+, NH3的浓度与有效性并不高。氨氧化古菌对底物具有较高亲和力, 低NH3环境更有利于氨氧化古菌生长[29]。氨氧化细菌amoA基因丰度并没有随着猪粪的施加而显著提高, 这一方面是由于氨氧化细菌对NH3的亲和力较低, 更喜欢在高NH3的碱性环境中生存[7], 而酸性红壤产生的NH3大多转化成NH4+, 限制了氨氧化细菌的生长; 另一方面, 猪粪施用导致红壤pH值下降, pH值降低对氨氧化细菌的限制作用抵消了猪粪施加后养分含量提高的促进作用。完全氨氧化菌进化枝A丰度低于氨氧化古菌和氨氧化细菌, 表明在酸性农田红壤中完全氨氧化菌进化枝A的丰度并不占据优势。这与Xu等[11]在中国不同地区的五种农田土壤的发现一致。这可能是由于完全氨氧化菌的生长速率比传统氨氧化微生物低, 在土壤中的丰度一般低于氨氧化古菌和氨氧化细菌[30]。和CK相比, M9和M18处理未能显著提高完全氨氧化菌进化枝A的amoA丰度, M27则显著提高其丰度。相关性分析表明, 完全氨氧化菌进化枝A丰度与除pH值外的其他理化性质呈显著正相关(P < 0.05), 表明进化枝A丰度主要受土壤养分含量控制, 存在喜欢富营养环境的完全氨氧化菌。逐步回归分析发现, AP含量是限制完全氨氧化菌进化枝A的amoA基因丰度的主要环境因子[31], 可以解释其86.5%的变异。原因可能是磷素是土壤微生物生长的必须营养元素, 而红壤普遍缺磷[32], 完全氨氧化菌进化枝A可能对缺磷更加敏感, 因此猪粪施加后土壤AP增加可能更有利于提高完全氨氧化菌的丰度。
3.3 完全氨氧化菌与净硝化速率的关系与CK相比, M9并未显著改变土壤净硝化速率, M18和M27则显著提高土壤净硝化速率, 增幅分别达到317%和416%, 表明长期施用中量和高量猪粪可以显著提高红壤硝化速率。相关分析表明, 红壤净硝化速率与氨氧化古菌和氨氧化细菌基因丰度无显著相关性, 但与完全氨氧化菌进化枝A呈显著正相关, 表明完全氨氧化菌进化枝A可能在红壤硝化过程中扮演重要角色。迄今为止, 只有一株从水生生态系统中分离出来的完全氨氧化菌, 动力学分析表明, 其表观半饱和常数Km (NH3)约为63 nmol/L, 亲和力高于氨氧化古菌和氨氧化细菌, 在寡营养条件下更具竞争力[12, 33]。然而, 猪粪施加后, 有机质分解释放出大量的NH3, 可以提高土壤NH3的有效性。王欣丽等[34]研究发现, 添加2 Mg/hm2猪粪可提高酸性红壤的NH3浓度至300 nmol/L以上。本研究中土壤NH3的浓度远高于63 nmol/L, 且完全氨氧化菌进化枝A丰度随着猪粪用量增加而增加, 表明完全氨氧化菌进化枝A喜好的生存环境可能不局限于寡营养生境。因此, 完全氨氧化菌进化枝A可以在长期施用猪粪的酸性红壤硝化过程中发挥重要作用, 且其丰度和活性随着猪粪的施用量增加而提高。值得一提的是, 该结果是基于相关性分析得到的, 由于尚未发现完全氨氧化菌的特异性抑制剂, 该方法是研究完全氨氧化菌活性的替代方法[35], 但在未来的研究中可以通过多种手段加以验证。例如:寻找能够特异性抑制完全氨氧化菌活性的抑制剂、利用DNA-SIP(DNA-stable isotope probing)和RNA-SIP(RNA-stable isotope probing)技术测定微生物同化13CO2的活性、从土壤中分离培养完全氨氧化菌等, 验证氨氧化微生物在酸性红壤硝化过程中的功能活性。
4 结论本研究利用荧光定量PCR, 对长期施用猪粪的农田红壤氨氧化微生物丰度进行研究, 结果表明:(1)长期施用猪粪显著提高土壤有机质和各种养分含量, 且随着猪粪的施用量增加而增加。(2)农田红壤完全氨氧化菌以进化枝A为主, 而进化枝B丰度极低, 存在很强的非特性扩增, 但进化枝A的丰度显著低于氨氧化古菌和氨氧化细菌。(3)长期施用高量猪粪显著提高进化枝A丰度和净硝化速率。进化枝A丰度与土壤各种养分含量呈显著正相关, 速效磷是最主要的影响因子, 表明存在喜好富营养环境的完全氨氧化菌。(4)进化枝A丰度和净硝化速率呈显著正相关, 可能在长期施用猪粪的农田红壤硝化过程中发挥重要功能。
| [1] |
Kuypers M M M, Marchant H K, Kartal B. The microbial nitrogen-cycling network. Nature Reviews Microbiology, 2018, 16(5): 263-276. DOI:10.1038/nrmicro.2018.9 |
| [2] |
Costa E, Pérez J, Kreft J U. Why is metabolic labour divided in nitrification?. Trends in Microbiology, 2006, 14(5): 213-219. DOI:10.1016/j.tim.2006.03.006 |
| [3] |
Daims H, Lebedeva E V, Pjevac P, Han P, Herbold C, Albertsen M, Jehmlich N, Palatinszky M, Vierheilig J, Bulaev A, Kirkegaard R H, von Bergen M, Rattei T, Bendinger B, Nielsen P H, Wagner M. Complete nitrification by Nitrospira bacteria. Nature, 2015, 528(7583): 504-509. DOI:10.1038/nature16461 |
| [4] |
Xia F, Wang J G, Zhu T, Zou B, Rhee S K, Quan Z X. Ubiquity and diversity of complete ammonia oxidizers (comammox). Applied and Environmental Microbiology, 2018, 84(24): e01390-18. |
| [5] |
曹彦强, 王智慧, 莫永亮, 王梅, 蒋先军. 施肥和淹水管理对水稻土氨氧化微生物数量的影响. 土壤学报, 2019, 56(4): 1004-1011. |
| [6] |
高复云, 李雅颖, 姚槐应. 完全氨氧化菌的分子生态学研究进展. 应用生态学报, 2020, 31(8): 2831-2840. DOI:10.13287/j.1001-9332.202008.032 |
| [7] |
Wakelin S A, Colloff M J, Harvey P R, Marschner P, Gregg A L, Rogers S L. The effects of stubble retention and nitrogen application on soil microbial community structure and functional gene abundance under irrigated maize. FEMS Microbiology Ecology, 2007, 59(3): 661-670. DOI:10.1111/j.1574-6941.2006.00235.x |
| [8] |
陈竹, 郭岩彬, 孟凡乔, 邵小明, 刘宝驹, 吴文良. 施肥方式对干旱半干旱地区土壤氨氧化微生物数量和群落的影响. 生态学报, 2021, 41(11): 4577-4585. |
| [9] |
Huang X R, Zhao J, Su J, Jia Z J, Shi X L, Wright A L, Xia Z B, Jiang X J. Neutrophilic bacteria are responsible for autotrophic ammonia oxidation in an acidic forest soil. Soil Biology and Biochemistry, 2018, 119: 83-89. DOI:10.1016/j.soilbio.2018.01.016 |
| [10] |
Li C Y, Hu H W, Chen Q L, Chen D L, He J Z. Comammox Nitrospira play an active role in nitrification of agricultural soils amended with nitrogen fertilizers. Soil Biology and Biochemistry, 2019, 138: 107609. DOI:10.1016/j.soilbio.2019.107609 |
| [11] |
Xu S Y, Wang B Z, Li Y, Jiang D Q, Zhou Y T, Ding A Q, Zong Y X, Ling X T, Zhang S Y, Lu H J. Ubiquity, diversity, and activity of comammox Nitrospira in agricultural soils. Science of the Total Environment, 2020, 706: 135684. DOI:10.1016/j.scitotenv.2019.135684 |
| [12] |
Kits K D, Sedlacek C J, Lebedeva E V, Han P, Bulaev A, Pjevac P, Daebeler A, Romano S, Albertsen M, Stein L Y, Daims H, Wagner M. Kinetic analysis of a complete nitrifier reveals an oligotrophic lifestyle. Nature, 2017, 549(7671): 269-272. DOI:10.1038/nature23679 |
| [13] |
Zhou X, Wang S W, Ma S T, Zheng X K, Wang Z Y, Lu C H. Effects of commonly used nitrification inhibitors-dicyandiamide (DCD), 3, 4-dimethylpyrazole phosphate (DMPP), and nitrapyrin-on soil nitrogen dynamics and nitrifiers in three typical paddy soils. Geoderma, 2020, 380: 114637. DOI:10.1016/j.geoderma.2020.114637 |
| [14] |
赵其国, 黄国勤, 马艳芹. 中国南方红壤生态系统面临的问题及对策. 生态学报, 2013, 33(24): 7615-7622. |
| [15] |
Lin Y X, Ye G P, Kuzyakov Y, Liu D Y, Fan J B, Ding W X. Long-term manure application increases soil organic matter and aggregation, and alters microbial community structure and keystone taxa. Soil Biology and Biochemistry, 2019, 134: 187-196. DOI:10.1016/j.soilbio.2019.03.030 |
| [16] |
鲁如坤. 土壤农业化学分析方法. 北京: 中国农业科技出版社, 2000.
|
| [17] |
Chu H, Fujii T, Morimoto S, Lin X G, Yagi K, Hu J L, Zhang J B. Community structure of ammonia-oxidizing bacteria under long-term application of mineral fertilizer and organic manure in a sandy loam soil. Applied & Environmental Microbiology, 2007, 73(2): 485-491. |
| [18] |
Francis C A, Roberts K J, Beman J M, Santoro A E, Oakley B B. Ubiquity and diversity of ammonia-oxidizing archaea in water columns and sediments of the ocean. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(41): 14683-14688. DOI:10.1073/pnas.0506625102 |
| [19] |
Rotthauwe J H, Witzel K P, Liesack W. The ammonia monooxygenase structural gene amoA as a functional marker: molecular fine-scale analysis of natural ammonia-oxidizing populations. Applied and Environmental Microbiology, 1997, 63(12): 4704-4712. DOI:10.1128/aem.63.12.4704-4712.1997 |
| [20] |
Jiang R, Wang J G, Zhu T, Zou B, Wang D Q, Rhee S K, An D, Ji Z Y, Quan Z X. Use of newly designed primers for quantification of complete ammonia-oxidizing (comammox) bacterial clades and strict nitrite oxidizers in the genus Nitrospira. Applied and Environmental Microbiology, 2020, 86(20). |
| [21] |
Zhu Y M, Berry D F, Martens D C. Copper availability in two soils amended with eleven annual applications of copper-enriched hog manure. Communications in Soil Science and Plant Analysis, 1991, 22(7/8): 769-783. |
| [22] |
Park S, Kim K S, Kang D, Yoon H, Sung K. Effects of humic acid on heavy metal uptake by herbaceous plants in soils simultaneously contaminated by petroleum hydrocarbons. Environmental Earth Sciences, 2013, 68(8): 2375-2384. DOI:10.1007/s12665-012-1920-8 |
| [23] |
Liu G M, Zhang X C, Wang X P, Shao H B, Yang J S, Wang X P. Soil enzymes as indicators of saline soil fertility under various soil amendments. Agriculture, Ecosystems & Environment, 2017, 237: 274-279. |
| [24] |
Liu Z J, Rong Q L, Zhou W, Liang G Q. Effects of inorganic and organic amendment on soil chemical properties, enzyme activities, microbial community and soil quality in yellow clayey soil. PLoS One, 2017, 12(3): e0172767. DOI:10.1371/journal.pone.0172767 |
| [25] |
黄晶, 张杨珠, 高菊生, 张文菊, 刘淑军. 长期施肥下红壤性水稻土有机碳储量变化特征. 应用生态学报, 2015, 26(11): 3373-3380. |
| [26] |
杨小东, 曾希柏, 文炯, 王亚男, 白玲玉, 许望龙, 苏世鸣, 吴翠霞. 猪粪施用量对红壤旱地理化性质及酶活性的影响. 土壤学报, 2020, 57(3): 739-749. |
| [27] |
Lehtovirta-Morley L E, Sayavedra-Soto L A, Gallois N, Schouten S, Stein L Y, Prosser J I, Nicol G W. Identifying potential mechanisms enabling acidophily in the ammonia-oxidizing archaeon "Candidatus Nitrosotalea devanaterra". Applied and Environmental Microbiology, 2016, 82(9): 2608-2619. DOI:10.1128/AEM.04031-15 |
| [28] |
Herbold C W, Lehtovirta-Morley L E, Jung M Y, Jehmlich N, Hausmann B, Han P, Loy A, Pester M, Sayavedra-Soto L A, Rhee S K, Prosser J I, Nicol G W, Wagner M, Gubry-Rangin C. Ammonia-oxidising archaea living at low pH: insights from comparative genomics. Environmental Microbiology, 2017, 19(12): 4939-4952. |
| [29] |
Lu L, Han W Y, Zhang J B, Wu Y C, Wang B Z, Lin X G, Zhu J G, Cai Z C, Jia Z J. Nitrification of archaeal ammonia oxidizers in acid soils is supported by hydrolysis of urea. The ISME Journal, 2012, 6(10): 1978-1984. |
| [30] |
Lawson C E, Lücker S. Complete ammonia oxidation: an important control on nitrification in engineered ecosystems?. Current Opinion in Biotechnology, 2018, 50: 158-165. |
| [31] |
Lin Y X, Fan J B, Hu H W, Duan C J, Ye G P, Wan S, He Z Y, Zheng Y, He J Z. Differentiation of individual clusters of comammox Nitrospira in an acidic Ultisol following long-term fertilization. Applied Soil Ecology, 2022, 170: 104267. |
| [32] |
黄国勤, 赵其国. 红壤生态学. 生态学报, 2014, 34(18): 5173-5181. |
| [33] |
Shi X Z, Hu H W, Wang J Q, He J Z, Zheng C Y, Wan X H, Huang Z Q. Niche separation of comammox Nitrospira and canonical ammonia oxidizers in an acidic subtropical forest soil under long-term nitrogen deposition. Soil Biology and Biochemistry, 2018, 126: 114-122. |
| [34] |
王欣丽, 朱飞, 姚静, 姜永见, 王芸, 任丽英. 长期施肥对酸性土壤氨氧化微生物群落的影响. 植物营养与肥料学报, 2018, 24(2): 375-382. |
| [35] |
Lu X D, Taylor A E, Myrold D D, Neufeld J D. Expanding perspectives of soil nitrification to include ammonia-oxidizing archaea and comammox bacteria. Soil Science Society of America Journal, 2020, 84(2): 287-302. |
 2023, Vol. 43
2023, Vol. 43