文章信息
- 马跃维, 丁文冕, 王跃澎, 原野, 黄艳燕, 南蓬
- MA Yuewei, DING Wenmian, WANG Yuepeng, YUAN Ye, HUANG Yanyan, NAN Peng
- 马里亚纳海沟共培养细菌多样性动态变化及潜在微生物互作关系
- Dynamic changes of bacterial diversity and microbial interactions in co-culture samples from the Mariana Trench
- 生态学报. 2023, 43(19): 8122-8138
- Acta Ecologica Sinica. 2023, 43(19): 8122-8138
- http://dx.doi.org/10.20103/j.stxb.202207142006
-
文章历史
- 收稿日期: 2022-07-14
- 网络出版日期: 2023-05-16
2. 复旦大学中华古籍保护研究院, 上海 200433;
3. 上海浦东复旦大学张江科技研究院, 上海 201203
2. Institute of Chinese Ancient Books Conservation, Fudan University, Shanghai 200433, China;
3. Fudan Zhang Jiang Institute, Shanghai 201203, China
深海, 一般指1000—6000m的海洋, 占地球海洋总体积的75%[1], 6000m以下的海洋则被称为深渊[2]。除去热液口、冷泉等较为极端特殊的环境, 深海环境具有以下普遍特征:黑暗、低氧[3]、低温、高压[4]、高盐[5]、寡营养[6—7]。寡营养的极端生态环境造就了深海微生物独特的物种多样性和遗传多样性, 其间生活的微生物为了适应这种极端环境, 可以比表层同类更为有效地利用难以降解的有机物质[8]。马里亚纳海沟是世界上已知最深的海沟, 也是与南极、北极、珠穆朗玛峰齐名的“世界第四极”。其最南端的最大水深超过10900m(有报道称其具体深度为10915—10920m[9]), 此处是地球上最深的地方, 被称作“挑战者深渊”(the Challenger Deep)[10]。该区沉积物来源比较复杂, 是陆源物质、生源物质、火山物质和自生矿物共同作用的结果[11]。但由于原位保压采样等技术并未发展完全, 对该区的生态学研究与其他区域相比仍有很大空缺。
随着16S rRNA测序、SSU rRNA测序、宏基因组测序技术的发展, 研究者对马里亚纳海沟微生物的群落结构已有一定了解, 但马里亚纳海沟微生物的分离培养还存在较大空白, 已报道的可分离培养的微生物门级类群数量更是远小于分子生物学能够检测到的数量[12—16], 提示存在大比例的未培养微生物(Unculturable microorganisms)。未培养微生物是指能用分子生物学手段检测到, 但未能获得纯培养的微生物[17]。现有研究表明, 未培养微生物生长的限制因素包括:①培养基组分和生长条件不全面;②忽视了环境中微生物之间的相互作用;③实验室营养条件不合适;④检测灵敏性受限等[18—19]。已有的对马里亚纳海沟进行的微生物分离培养研究多利用常见环境及海洋微生物培养基(如R2A、2216E等)直接进行稀释涂布, 这种方法未针对深海环境对培养基进行改良, 可能忽视了深海微生物生长的营养需求;且对一些长期生活在寡营养条件下的海洋细菌而言, 未稀释的培养基中相对高浓度的底物本身可能是有毒的[18];其次, 实验室的分离培养可能破坏自然环境中微生物之间的相互作用, 物种之间的信息交流被阻断, 导致微生物的不可培养[19]。自然界中, 绝大多数微生物以组成微生物群落的形式共同生存, 它们通过相互作用来适应各种环境[20], 这类相互作用被称为微生物互作(Microbial interaction)。微生物互作分为共栖、竞争和寄生三大类[21], 可以很好地解释未培养微生物的存在, 进而对未培养微生物的分离提供新思路。目前对微生物互作的研究大多集中于微生物发酵、根际微生物、肠道微生物[22—25]等, 而对深海微生物在寡营养条件下的互作尚未见报道。故本文在对马里亚纳海沟样本使用多种改良培养基进行大规模微生物分离培养的基础上(数据未发表), 选取寡营养培养基对其进行液体共培养, 探究马里亚纳海沟样本共培养细菌多样性动态变化及其中潜在的微生物互作关系, 为深渊等深海极端环境下微生物生态系统组成及维持提供数据支撑, 也为进一步研究极端微生物生存策略提供科学指导。
1 材料与方法 1.1 实验材料本研究采用来自马里亚纳海沟的4个样本, 为“深海关键技术与装备”国家重点专项科考人员乘“张謇”号科考船于2018年12月12日及2019年12月5日至2019年12月7日期间采集所得, 航次号为TS15。具体采样信息见表 1。
| 样本编号 Sample ID |
取样地点 Sampling position |
取样日期 Sampling date |
经度 Longitude/(°E) |
纬度 Latitude/(°N) |
深度 Depth /m |
样品类型 Sample type |
| S1 | MBR04 | 2019.12.05 | 142.23 | 10.82 | 5842 | 表层沉积物 |
| S2 | MBR02 | 2018.12.12 | 142.20 | 11.33 | 10900 | 表层沉积物 |
| W1 | MBR04 | 2019.12.07 | 142.23 | 10.82 | 1000 | 海水 |
| W2 | MBR02 | 2018.12.12 | 142.20 | 11.33 | 10900 | 海水 |
| S1:表面沉积物样本1 Surface sediment sample 1;S2:表面沉积物样本2 Surface sediment sample 2;W1:海水样本1 Water sample 1;W2:海水样本2 Water sample 2 | ||||||
1/10 2216E+培养基(后文简称E):市售2216E液体培养基(青岛海博生物技术有限公司)3.76g, 氯化钠30.00g, 2g/L硝酸钾溶液(硝酸钾0.1g, 去离子水100mL)2mL, 加入去离子水定容至1L, 121℃高压灭菌20min。
1/3 R2A+培养基(后文简称R):市售R2A液体培养基(青岛海博生物技术有限公司)1.08g, 氯化钠30.00g, 2g/L硝酸钾溶液(硝酸钾0.1g, 去离子水100mL)2mL, 加入去离子水定容至1L, 121℃高压灭菌20min。
1.2.2 液体共培养将样品分别用两种改良培养基稀释至10-4(以0.2g表层沉积物样本+900μL对应培养基作为10-1)。取1mL稀释液接种于预先装有9mL对应改良培养基的15mL离心管中, 各做3组, 每组包括3管生物学重复。置于摇床, 28℃, 振荡培养。培养2d、3d、5d时, 取出对应离心管, 将3管生物学重复的管中培养物离心至同一离心管, 弃上清, 使用液氮将装有沉淀菌体的离心管急速冷冻, 置于-80℃保存, 以进行后续混样测序。为表述便捷清晰, 对上述样品以“样品来源”_“培养基简称”_“培养时间”模式命名, 如用E培养基培养了2天的沉积物S1样品命名为S1_E_2;同一样品来源、同一培养基但培养时间不同的几个样品则称为一个“样品组”。
1.2.3 RNA提取及PCR扩增菌体送至上海美吉生物公司进行细菌多样性分析。为避免原样品中残留的微生物DNA片段干扰, 本研究对各管菌体提取RNA并反转录为cDNA, 使用NanoDrop2000对cDNA进行质检, 确认其浓度、纯度、完整性。选取细菌16s rRNA V3—V4高变区作为目的片段, 使用引物338F(5′-ACTCCTACGGGAGGCAGCAG-3′)与806R(5′-GGACTACHVGGGTWTCTAAT-3′)进行PCR扩增[26]。PCR体系为:5×FastPfu缓冲液4μL, 2.5 mM dNTPs 2μL, 前引物0.8μL, 后引物0.8μL, FastPfu聚合酶0.4μL, BSA 0.2μL, 模板DNA 10 ng, 补ddH2O至20 μL。PCR反应条件为:预变性95℃ 3min, 变性95℃ 30s, 退火55℃ 30s, 延伸72℃ 45s, 29个循环;72℃延伸10min。
1.2.4 文库定量、测序及原始处理基于二代测序平台Illumina MiSeq, 对上述PCR产物进行高通量测序;获得Miseq测序得到的PE reads后, 使用fastp软件(Version 0.19.6, https://github.com/OpenGene/fastp)过滤低质量序列, 并使用FLASH软件(Version 1.2.11, https://ccb.jhu.edu/software/FLASH/index.shtml)拼接双端序列, 过滤低质量序列后得到优化序列。具体除杂方法与参数如下:①过滤reads尾部质量值20以下的碱基, 设置50bp的窗口, 如果窗口内的平均质量值低于20, 从窗口开始截去后端碱基, 过滤质控后50bp以下的reads, 去除含N碱基的reads;②根据PE reads之间的overlap关系, 将成对reads拼接成一条序列, 最小overlap长度为10bp;③拼接序列的overlap区允许的最大错配比率为0.2, 筛选不符合序列;④根据序列首尾两端的barcode和引物区分样品, 并调整序列方向, barcode允许的错配数为0, 最大引物错配数为2。
以16S rDNA相似度>97% 作为OTU聚类标准, 使用UPARSE软件(Version 7.0.1090, http://www.drive5.com/uparse/)对优化序列进行聚类, 并在聚类过程中去除嵌合体, 具体聚类步骤如下:①对优化序列提取非重复序列, 便于降低分析中间过程冗余计算量(http://drive5.com/usearch/manual/dereplication.html);②去除没有重复的单序列(http://drive5.com/usearch/manual/singletons.html);③按照97%相似性对非重复序列(不含单序列)进行OTU聚类, 在聚类过程中去除嵌合体, 得到OTU的代表序列;④将所有优化序列map至OTU代表序列, 选出与代表序列相似性在97%以上的序列, 生成OTU表格。
1.2.5 共培养菌液细菌多样性分析采用RDP classifier(置信度阈值0.7, Version 2.11, https://sourceforge.net/projects/rdp-classifier/)贝叶斯算法对97%相似水平的OTU代表序列进行分类学分析, 并分别在各个分类学水平(域、界、门、纲、目、科、属、种)与Silva数据库(Release 138, https://www.arb-silva.de/)比对, 统计各样品的群落物种组成。使用Mothur软件(Version v.1.30.2, https://www.mothur.org/wiki/Download_mothur)计算各样品的Alpha多样性指数, 包括反映群落丰富度(Community richness)的指数Sobs、Chao、Ace;反映群落多样性(Community diversity)的指数Shannon、Simpson;反映群落覆盖度(Community coverage)的指数Coverage等。用于指数评估的OTU相似水平设定为97%。使用LEfSe软件(Version 1.0)根据分类学组成对样本按照不同的分组条件进行线性判别分析(LDA), 找出对样本划分产生显著性差异影响的群落或物种。使用PICRUSt软件包(Version 1.1.0, http://picrust.github.io/picrust/)对OTU丰度表进行标准化, 根据OTU丰度计算各功能类别的丰度, 并结合KEGG数据库中原核生物的分类谱系对样品16S rDNA数据进行分级功能预测。使用jvenn网站(http://jvenn.toulouse.inra.fr/app/example.html)进行花瓣图绘制。制图及润色使用R软件(Version 3.3.1)与Adobe Illustrator CC 2018完成。
2 结果 2.1 共培养样品细菌Alpha多样性分析完成对马里亚纳海沟4份样本(W1、W2、S1、S2)、2种培养基(E、R)、3个培养时间(2d、3d、5d)共24份共培养样品的多样性数据分析, 其中W1_R_2样品多样性低于检测限, 故摒弃该样品数据。共获得1272544条有效优化序列, 平均序列长度426bp。原始数据均已上传至NCBI数据库, 项目检索号为PRJNA844887;登录号为SRR19631964—SRR19631986。在97%相似度水平对OTU进行聚类注释, 结果统计为19个门、34个纲、76个目、131个科、227个属、419个OTUs。各样品多样性稀释曲线与香农曲线均呈现先增加后逐渐平缓的趋势(图 1), 且各样品测序覆盖度均高于99.9%(表 2), 表明样品序列检测度高、测序数据量合理, 测序结果足以反映样本中绝大多数的微生物多样性信息。

|
| 图 1 马里亚纳海沟不同样本液体共培养样品细菌群落Alpha多样性动态变化 Fig. 1 Variation of α-diversity of bacterial communities in liquid co-culture samples from the Mariana Trench S1:表面沉积物样本1;S2:表面沉积物样本2;W1:海水样本1;W2:海水样本2;E: 1/10 2216E+培养基; R: 1/3 R2A+培养基 |
| 样品编号 Sample ID |
有效序列 Sequence numbers |
OTU数 OTUs |
Ace指数 Ace index |
Chao指数 Chao index |
Shannon指数 Shannon index |
Simpson指数 Simpson index |
覆盖度 Coverage |
| S1_E_2 | 57591 | 27 | 67.80683 | 36 | 0.396492 | 0.834715 | 0.999844 |
| S1_E_3 | 54462 | 14 | 16.03333 | 15.5 | 0.725494 | 0.538531 | 0.999945 |
| S1_E_5 | 54224 | 7 | 0 | 13 | 0.123327 | 0.950887 | 0.999926 |
| S2_E_2 | 34520 | 9 | 9.784 | 9 | 0.437029 | 0.805261 | 0.999971 |
| S2_E_3 | 35238 | 11 | 31.40496 | 14 | 0.459163 | 0.765197 | 0.999886 |
| S2_E_5 | 36526 | 17 | 20.94957 | 19 | 0.525836 | 0.73488 | 0.99989 |
| W1_E_2 | 46273 | 4 | 4 | 4 | 0.059262 | 0.980964 | 1 |
| W1_E_3 | 59936 | 9 | 10.2 | 9.25 | 0.420352 | 0.759927 | 0.999967 |
| W1_E_5 | 58681 | 19 | 23.31177 | 20 | 0.717391 | 0.587603 | 0.999949 |
| W2_E_2 | 60769 | 11 | 17.24 | 14 | 0.019603 | 0.995497 | 0.999934 |
| W2_E_3 | 61174 | 13 | 27.0127 | 23 | 0.026598 | 0.993379 | 0.999918 |
| W2_E_5 | 53136 | 46 | 51.62842 | 50 | 0.07566 | 0.980373 | 0.999831 |
| S1_R_2 | 64097 | 54 | 124.9464 | 129.6 | 0.051664 | 0.987876 | 0.999563 |
| S1_R_3 | 57113 | 162 | 196.5402 | 205.0435 | 0.94295 | 0.603657 | 0.999212 |
| S1_R_5 | 61318 | 4 | 13.26852 | 5 | 0.00169 | 0.999674 | 0.999967 |
| S2_R_2 | 42399 | 25 | 31.06827 | 28.75 | 0.265141 | 0.906536 | 0.999858 |
| S2_R_3 | 42726 | 3 | 4.125 | 3 | 0.001235 | 0.999766 | 0.999977 |
| S2_R_5 | 39521 | 3 | 4.801749 | 3 | 0.002 | 0.999595 | 0.999975 |
| W1_R_3 | 54311 | 327 | 330.5931 | 330.3913 | 2.43063 | 0.240922 | 0.999761 |
| W1_R_5 | 58743 | 163 | 194.3434 | 183.5833 | 1.66014 | 0.28316 | 0.999336 |
| W2_R_2 | 63671 | 4 | 4 | 4 | 0.001698 | 0.999686 | 1 |
| W2_R_3 | 65369 | 6 | 13.6 | 9 | 0.002822 | 0.999449 | 0.999954 |
| W2_R_5 | 62070 | 5 | 11.78 | 6 | 0.003987 | 0.999163 | 0.999968 |
| S1_E_2:E培养基培养2天的S1样本S1 sample cultured in E medium for 2 days;W1_R_2:R培养基培养2天的W1样本W1 sample cultured in R medium for 2 days;以此类推 | |||||||
如表 2所示, 各样品分别含有3—327个OTUs, 在使用E培养基的样品组中, 除了S1_E样品组, 其余样品组的OTU数均随培养时间增加而增加;使用R培养基的样品组中, 除了S2_R样品组, 其余样品组中的OTU数均随时间先升后降。
以Ace指数和Chao指数评估各样品细菌的丰富度, 发现W1_E、W2_E、S2_E的丰富度随培养时间增加而增加, S1_E的丰富度则随培养时间增加而降低;W1_R、W2_R、S1_R的丰富度均先升高后降低, S2_R的丰富度则先下降后略有上升。整体上, S1、W1样品组中, 使用R培养基的细菌丰富度最大值大于使用E培养基的, W2样品组中则相反, S2样品组中二者差异不大。
以Shannon指数和Simpson指数评估各样品细菌的多样性:对比各样品组多样性结果, W1_E、W2_E、S2_E中的细菌多样性随培养时间增加而增加, S1_E中的细菌多样性则随培养时间增加而降低;W1_R、S1_R中的细菌多样性先升后降, W2_R中的细菌多样性随培养时间增加而增加, S2_R中的细菌多样性则随培养时间增加而降低。整体上, 深海样品组W1、S1比深渊样品组W2、S2检测到了更高的细菌多样性;培养3d的样品往往显示出较高的细菌多样性。
2.2 样品细菌物种组成分析及样品比较分析对共培养样品中细菌群落在各个水平上进行分类学注释, 结果显示, 在门水平上(图 2), 共培养样品中占比较大的是变形菌门(Proteobacteria)和拟杆菌门(Bacteroidota)的细菌。S1_E样品组中, 拟杆菌门的相对丰度随培养时间增加而增加, 变形菌门的相对丰度则逐渐降低;W2_R样品组中, W2_R_2的优势菌门为变形菌门, W2_R_3、W2_R_5的优势菌门则为拟杆菌门;其余样品组中, 变形菌门均为优势类群, 相对丰度56.99%—99.99%不等;在W1_E_3、W1_R_3、W1_R_5样品中, 厚壁菌门(Firmicutes)均占10%以上的相对丰度;在W1_R_3样品中, 检测到了11.09%的蓝细菌门(Cyanobacteria)微生物, W1_R_5中则检测到了0.04%的蓝细菌;S1_R_3样品中检测到了0.29%的蓝细菌。整体上, W1_R_3、S1_R_3检测所获门级微生物多样性较高, 与使用Shannon指数及Simpson指数的预测相符。
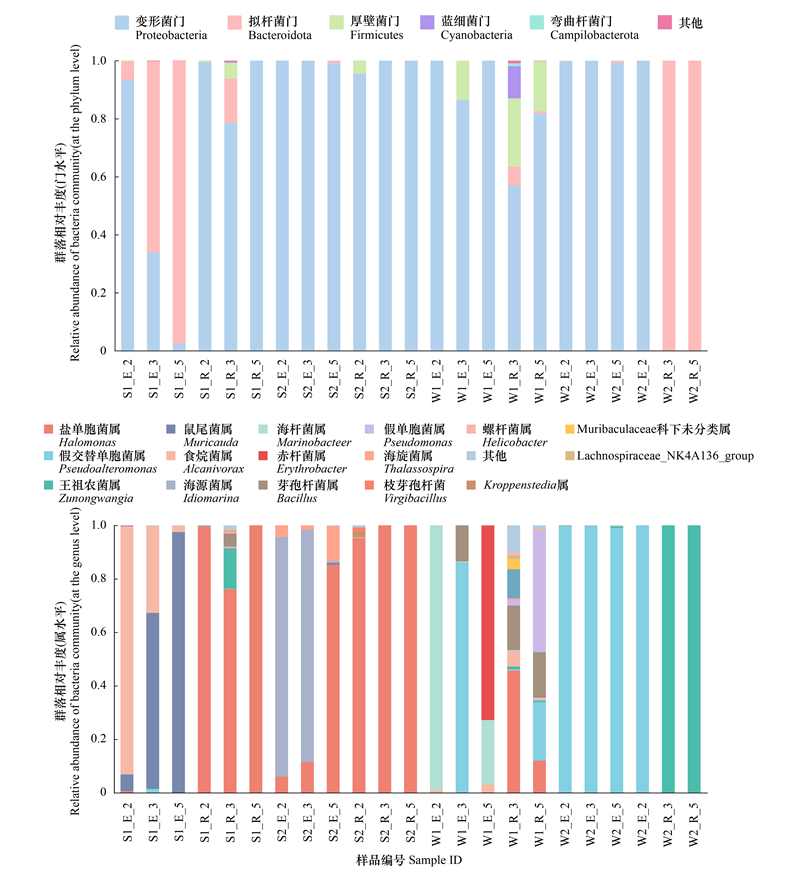
|
| 图 2 马里亚纳海沟不同样本液体共培养样品细菌相对丰度 Fig. 2 The relative abundance of bacteria community in liquid co-culture samples from the Mariana Trench S1_E_2:E培养基培养2天的S1样本;W1_R_2:R培养基培养2天的W1样本;以此类推 |
在属水平上(图 2), 整体相对丰度较高的细菌属是盐单胞菌属(Halomonas)、假交替单胞菌属(Pseudoalteromonas)、王祖农菌属(Zunongwangia)、鼠尾菌属(Muricauda)、食烷菌属(Alcanivorax), 但各样品组的动态变化趋势及各样品的优势菌属并不相同。S1_R、S2_R样品组中, 盐单胞菌在三个培养时间均为优势类群;S2_E样品组中, 盐单胞菌属在2d、3d时占比不到15%, 但在5d时成为绝对优势类群, 占比85.20%;S1_E样品组中, 鼠尾菌属相对丰度随培养时间增加而增加, 食烷菌属相对丰度则随培养时间增加而下降;W1_E_2中海杆菌属(Marinobacter)为绝对优势类群(99.04%), W1_E_3中假交替单胞菌属则为优势类群(86.12%), W1_E_5中赤杆菌属(Erythrobacter)则为优势类群(72.74%);W2_E样品组中, 假交替单胞菌属在三个培养时间均为绝对优势菌属;W2_R_2中, 假交替单胞菌属为绝对优势类群, W2_R_3、W2_R_5中, 鼠尾菌属则为绝对优势类群。整体上, 样品S1_R_3、W1_R_3、W1_R_5多样性较高。就共培养样品细菌多样性随培养时间的动态变化而言, 盐单胞菌属、假交替单胞菌属显示出了较强的竞争能力, 这种动态变化也反映了不同种属细菌在不同培养基中培养初期的生长速度差异, 以及它们适应环境并与其他微生物竞争的能力差异。
各样品共有及特有的属级类群数如图 3所示。S1样品组中, 盐单胞菌为共有属, S1_R_2、S1_R_3检测到特有属;S2样品组中, 盐单胞菌为共有属, S2_R_3、S2_R_5、S2_E_5检测到特有属;W1样品组中, 海杆菌属、芽孢杆菌属(Bacillus)、食烷菌属为共有属, W1_R_3、W1_R_5检测到较多特有属;W2样品组中, 芽孢杆菌属为共有属, W2_E_2、W2_E_3、W2_E_5、W2_R_3均检测到特有属。整体上, 使用E培养基、培养时间5d的样品, 以及使用R培养基、培养时间为3d的样品检测所获的属级类群数量较多, 也更易出现特有属。
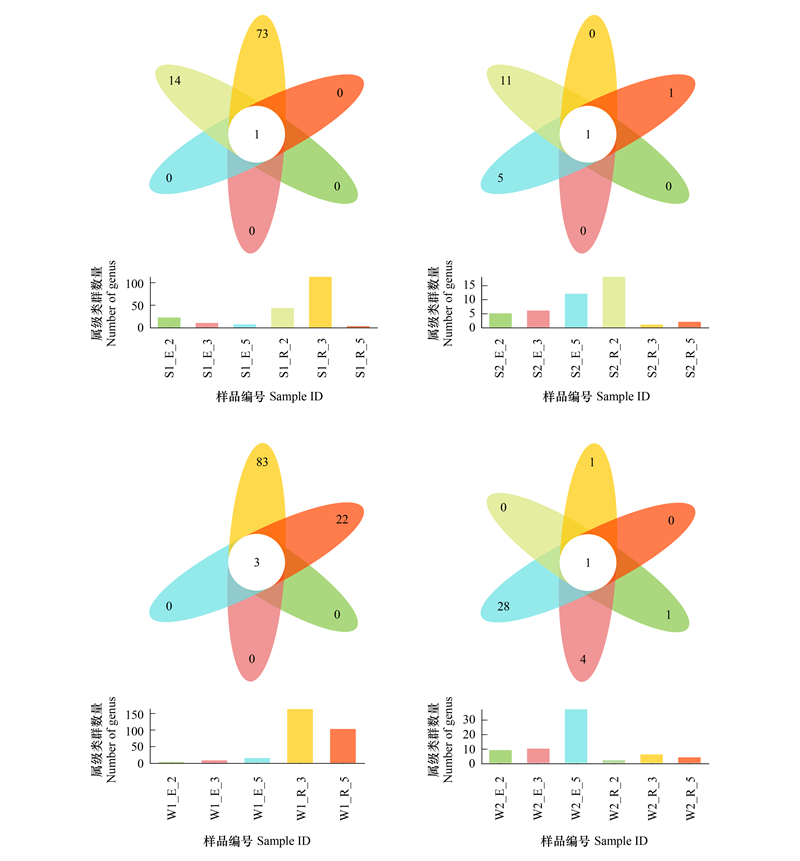
|
| 图 3 马里亚纳海沟不同样本液体共培养样品物种组成相似性及重叠情况(属水平) Fig. 3 Community composition similarity and overlap of liquid co-culture samples from the Mariana Trench (at the genus level) 花瓣图中, 花瓣里的数字表示对应分组特有的属级类群数目, 中心数字则对应所有分组共有的属级类群数目;柱形图为各样品属级类群总数 |
以培养基为分组的LEfSe多级物种差异判别分析结果显示(图 4), 交替单胞菌目(Alteromonadales)在使用E培养基的样品组中显著富集, 对组间差异有较大贡献;盐单胞菌科(Halomonadaceae)、盐单胞菌属在使用R培养基的样品组中显著富集, 对组间差异有较大贡献;蓝细菌目(Cyanobacteriales)、蓝细菌科(Cyanobacteriaceae)也在使用R培养基的样品组中显著富集, 对组间差异有一定贡献。以样本来源为分组的LEfSe多级物种差异判别分析结果显示(图 4), 厚壁菌门、芽孢杆菌纲(Bacilli)、芽孢杆菌目(Bacillales)、芽孢杆菌科(Bacillaceae)、芽孢杆菌属、海杆菌科(Marinobacteraceae)、海杆菌属、赤杆菌属在使用W1样本的样品组中显著富集, 对组间差异有较大贡献;假交替单胞菌属在使用W2样本的样品组中显著富集, 对组间差异有较大贡献;海洋螺菌目(Oceanospirillales)、食烷菌科(Alcanivoracaceae)、食烷菌属在使用S1样本的样品组中显著富集, 对组间差异有较大贡献, 鼠尾菌属、类诺卡氏菌属(Nocardioides)也显著富集于S1, 对组间差异有一定贡献;盐单胞菌科、盐单胞菌属则显著富集于使用S2样本的样品组, 对组间差异贡献较大。这些微生物类群在不同样品组中的富集及对组间差异的贡献与随培养时间的多样性动态变化相符。以深海样本(1000—6000m)与深渊样本(6000m以下)为分组的LEfSe多级物种差异判别分析结果则显示, 假单胞菌属(Pseudomonas)、海杆菌属、食烷菌属等很多微生物类群在深渊样本中显著富集(图 4)。
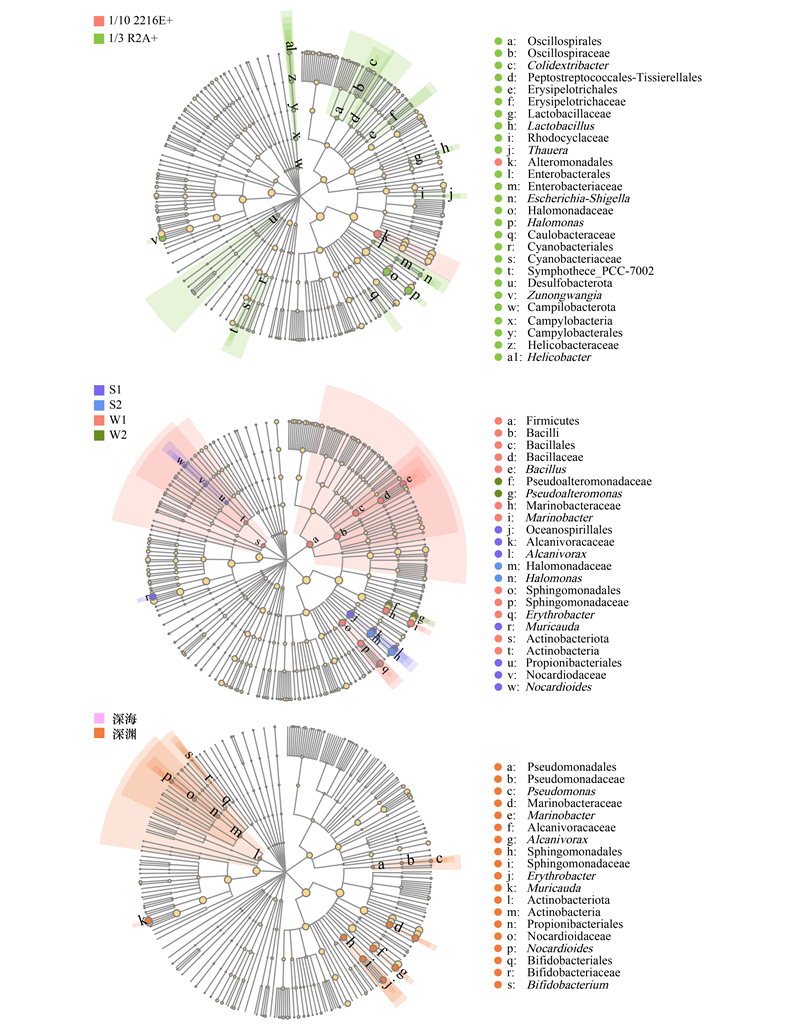
|
| 图 4 马里亚纳海沟不同样本共培养样品LEfSe多级物种层级树 Fig. 4 Hierarchical tree diagram of LEfSe multi level species in liquid co-culture samples from the Mariana Trench 紫、蓝、红、绿、橙、粉色节点表示在对应组别中显著富集, 且对组间差异存在显著影响的细菌类群;淡黄色节点表示对组间差异无显著影响的细菌类群;节点大小表征对组间差异的贡献;Oscillospirales:颤螺旋菌目;Oscillospiraceae:颤螺旋菌科;Colidextribacter:Colidextribacter属;Peptostreptococcales-Tissierellales:肽链球菌-组织菌目;Erysipelotrichales:丹毒丝菌目;Erysipelotrichaceae:丹毒丝菌科;Lactobacillaceae:乳酸杆菌科;Lactobacillus:乳杆菌属;Rhodocyclaceae:红环菌科;Thauera:索氏菌属;Alteromonadales:交替单胞菌目;Enterobacterales:肠杆菌目;Enterobacteriaceae:肠杆菌科;Escherichia-Shigella:大肠杆菌志贺菌属;Halomonadaceae:盐单胞菌科;Halomonas:盐单胞菌属;Caulobacteraceae:柄杆菌科;Cyanobacteriales:蓝细菌目;Cyanobacteriaceae:蓝细菌科;Symphothece_PCC-7002:聚球藻PCC-7002;Desulfobacterota:脱硫菌门;Zunongwangia:王祖农菌属;Campilobacterota:弯曲杆菌门;Campylobacteria:弯曲菌门;Campylobacterales:弯曲菌目;Helicobacteraceae:螺杆菌科;Helicobacter:螺杆菌属;Firmicutes:厚壁菌门;Bacilli:芽孢杆菌纲;Bacillales:芽孢杆菌目;Bacillaceae:芽孢杆菌科;Bacillus:芽孢杆菌属;Pseudoalteromonadaceae:假交替单胞菌科;Pseudoalteromonas:假交替单胞菌属;Marinobacteraceae:海杆菌科;Marinobacter:海杆菌属;Oceanospirillales:海洋螺菌目;Alcanivoracaceae:食烷菌科;Alcanivorax:食烷菌属;Sphingomonadales:鞘脂单胞菌目;Sphingomonadaceae:鞘脂单胞菌科;Erythrobacter:赤杆菌属;Muricauda:鼠尾菌属;Actinobacteriota:放线菌门;Actinobacteria:放线菌纲;Propionibacteriales:丙酸杆菌目;Nocardiodaceae:类诺卡氏菌科;Nocardioides:类诺卡氏菌属;Pseudomonadales:假单胞菌目;Pseudomonadaceae:假单胞菌科;Pseudomonas:假单胞菌属;Bifidobacteriales:双歧杆菌目;Bifidobacteriaceae:双歧杆菌科;Bifidobacterium:双歧杆菌属 |
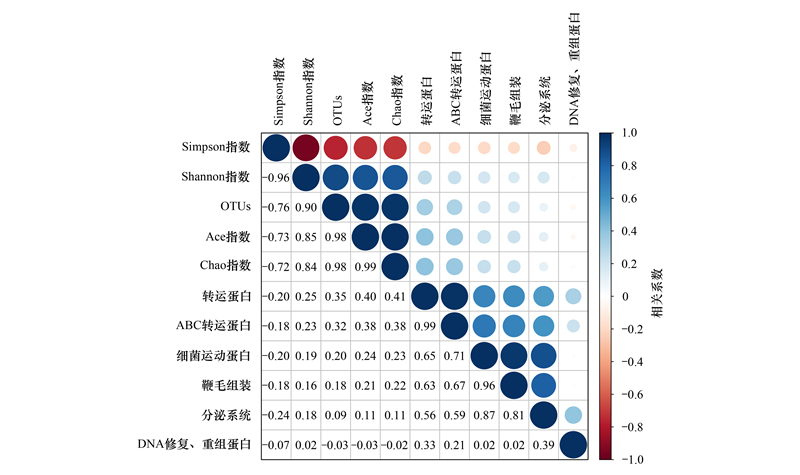
使用PICRUSt软件对共培养样品16S rDNA数据进行COG和KEGG功能注释, 共获得41个二级生物代谢预测功能, 243个三级生物代谢预测功能, 各样品中预测代谢通路的组成差异不大(图 5)。其中, 丰度较高的代谢通路大多为微生物生存的必需通路, 如DNA修复和重组蛋白、ABC转运蛋白、核糖体等。如图 6, 对于丰度前60的三级代谢通路, 不同样品组内, 各代谢通路丰度随时间的变化基本一致:W1_E、S1_R样品组中, 各代谢通路丰度先降后升;S2_R、W1_R、W2_R、W2_R样品组中, 各代谢通路丰度先升后降;S2_E样品组中, 各代谢通路丰度呈现升高趋势;S1_E样品组中, 转运蛋白、ABC转运蛋白、DNA修复与重组蛋白等维持微生物生存的基本通路丰度随培养时间增加而降低, 分泌系统、细菌运动蛋白、鞭毛组装等负责微生物运动、分泌等与外界有信息交流的通路的丰度则显示出升高趋势。共培养样品细菌多样性、细菌丰度与部分预测功能通路具有较弱的正相关性(图 7)。
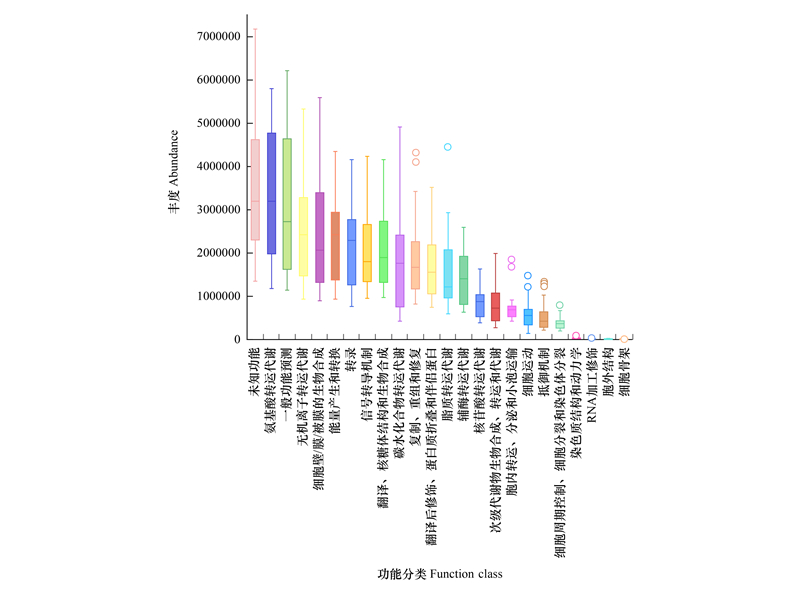
|
| 图 5 马里亚纳海沟不同样本共培养样品COG功能注释分类 Fig. 5 COG function annotation classification of liquid co-culture samples from the Mariana Trench |

|
| 图 6 马里亚纳海沟不同样本液体共培养样品KEGG注释功能热图 Fig. 6 KEGG annotated function heatmap of liquid co-culture samples from the Mariana Trench |
|
| 图 7 马里亚纳海沟样本共培养样品细菌丰度、多样性与预测功能通路丰度相关性分析 Fig. 7 Correlation between the bacterial abundance, diversity and abundance of predictive function of liquid co-culture samples from the Mariana Trench |
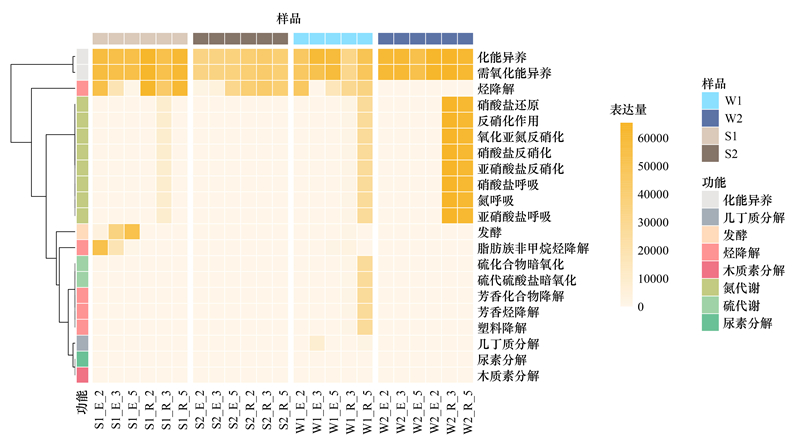
采用FAPROTAX方法对各共培养样品中的微生物群落功能表型进行预测, 并对其中碳、氢、氮、磷、硫等循环功能进行分析, 共预测到了21个微生物代谢相关的功能, 其中8个氮循环相关的功能表型、2个硫循环相关的功能表型。如图 8所示, 化能异养、需氧化能异养功能在各样品中都有较高丰度, 并在各样品组中均呈现不同的小幅度变化趋势, 暗示其可能是各微生物类群的生长所需的必需通路, 并且会随微生物群落组成变化而变化;除W2_E、W2_R样品组外的其他样品中, 烃降解功能丰度较高, 且在各样品组中随培养时间的变化趋势不同, 提示其中微生物群落组成不同及互作关系的潜在差异;W2_R_3d、W2_R_5d样品中, 硝酸盐还原、氮呼吸、亚硝酸盐呼吸、硝酸盐呼吸、硝酸盐反硝化、亚硝酸盐反硝化、氧化亚氮反硝化、反硝化作用等氮循环相关的功能表型丰度均较高;W1_R_5d样品中, 氮循环相关的功能表型显示出一定丰度, 并存在硫化合物暗氧化、硫代硫酸盐暗氧化等硫代谢通路以及芳香化合物降解通路;S1_E样品组中, 发酵这一表型的丰度随培养时间增加而增加, 脂肪族非甲烷烃降解的丰度则随培养时间增加而下降。
|
| 图 8 马里亚纳海沟不同样本共培养样品FAPROTAX功能预测热图 Fig. 8 FAPROTAX function prediction heatmap of liquid co-culture samples from the Mariana Trench |
对马里亚纳海沟各样本的共培养16S rDNA多样性分析结果与从其中分离获得的单菌落多样性有差异。与本项目前期进行的采用多种培养条件进行极限稀释液体培养研究[27](可避免细菌间的竞争)及采用6种培养基进行平板稀释法培养的研究(数据未发表)相比, 共培养样品16S rDNA多样性分析获得的15个门、30个纲、62个目、117个科、211个属是前期实验未得到的;另有1个目、9个科、19个属为共培养样品中未检测到、但分离获得单菌落的类群。
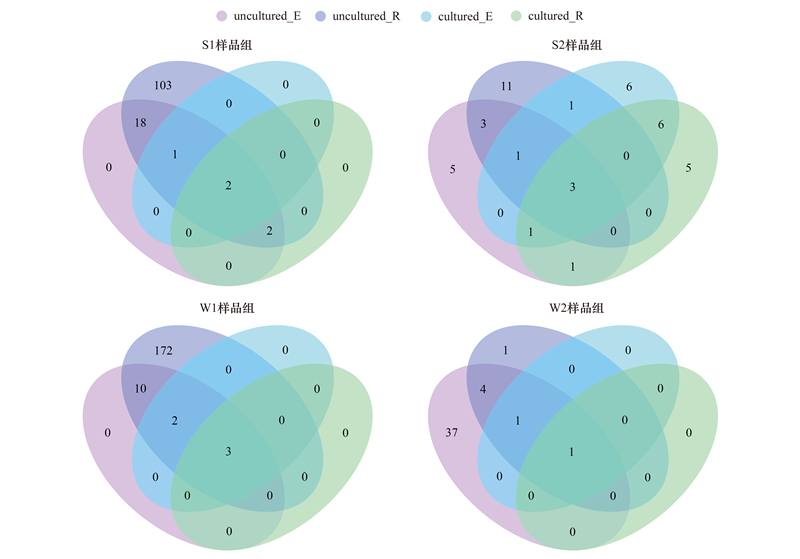
对比同样使用E、R培养基的分离培养微生物多样性结果(数据未发表)与液体共培养16S rDNA多样性结果(图 9), 各样品组中, 均仅有少量菌属为分离培养方法与共培养16S rDNA均检测到的共有属。使用R培养基的16S rDNA多样性结果在S1、S2、W1样品组中均有大量特有属, 使用E培养基的16S rDNA多样性结果则在W2样品组中有较多特有属。S1样品组中, 海杆菌属、食烷菌属为共有属;S2样品组中, 海旋菌属、盐单胞菌属、海源菌属为共有属, 17个属为共培养16S rDNA样品中为检测到的微生物类群;W1样品组中, 食烷菌属、盐单胞菌属、赤杆菌属为共有属;W2样品组中, 假交替单胞菌属为共有属;这些样品组内的共有属级类群均为深海常见类群。与分离培养微生物多样性结果相反, S2样品组共培养16S rDNA检测到的属级类群数目远小于W1、S1样品组。
|
| 图 9 可培养微生物多样性与液体共培养菌液16S rDNA多样性属级类群Venn图 Fig. 9 Venn diagram of culturable microbial diversity and 16S rDNA diversity of liquid co-culture samples uncultured_E:使用E培养基样品的16S rDNA检测结果; uncultured_R:使用R培养基样品的16S rDNA检测结果; cultured_E:使用E培养基分离获得微生物的鉴定结果; cultured_R:使用R培养基分离获得微生物的鉴定结果 |
本项目前期采用多种培养条件进行的极限稀释液体培养[27]及采用6种培养基的平板稀释法培养(数据未发表)共从马里亚纳海沟海水中分离获得4个门的细菌, 即变形菌门、拟杆菌门、放线菌门、厚壁菌门, 且变形菌门为优势类群, 这一结果与王震等人[28]的研究结果相类似;李毅等人未获得放线菌门的微生物[29];黄莹等人未分离到拟杆菌门, 但分离获得了栖热菌门[30]。然而, 对马里亚纳海沟的高通量测序结果总能鉴定出几十到几百的门级类群[13, 27, 31—32]。本项目前期研究针对马里亚纳海沟环境特征对培养基成分、浓度进行了调整, 减弱了培养基组分与实验室营养条件对微生物生长的影响, 但门水平上数量的巨大差异仍提示样品中存在大量未培养微生物, 这很可能是由于接种于平板上的微生物之间无法产生胞间联系, 从而阻断了微生物间的相互作用。
使用多种培养基的分离培养结果与共培养样品16S rDNA检测结果均检测到特有目、科、属级类群。各样品组中, 均仅有少量菌属为分离培养方法与共培养样品均检测到的共有属, 共培养样品检测到的特有属数量远多于分离培养方法分离所获。检测到16S rDNA但并未分离获得纯培养的未培养微生物类群(如蓝细菌门、酸杆菌门、绿弯菌门等)可能在生长过程中需要依赖与其他微生物的共生关系;分离获得纯培养但并未检测到16S rDNA的微生物类群(如橙色单胞菌属、伯克霍尔德菌属、环杆菌属等)则可能竞争能力不强, 无法在混合培养中生存, 需要在低竞争的条件下才能够占有自己的生态位。
3.1 共培养样品中潜在的微生物互生以蓝细菌为例, 本研究中共培养样品组W1、S1均检测到蓝细菌门的存在, W1_R_3样品中更检测到了11.09%的蓝细菌门微生物。研究表明, 蓝细菌常被发现与异养细菌共存, 它们可能为异养细菌提供内含多糖和肽的完美栖息地;异养菌则可能通过提供有益的代谢物对蓝藻产生积极的影响[33]。原绿球藻菌是一类广泛分布于海洋的蓝细菌, 并已成为一个研究自然和生物多样性规则的有用模型[34]。大部分稀释的原绿球藻菌株不能在平板上生长, 而与异养细菌共存的情况下生长效果明显变好, 能在液体培养基中以稀释培养物的形式生长, 也能在半固体琼脂糖平板上形成菌落[35]。本项目前期研究从马里亚纳海沟样品中分离获得了盐单胞菌属、假交替单胞菌属、食烷菌属、海杆菌属、鼠尾菌属、鞘脂单胞菌属等微生物, 这些类群均有与蓝细菌互作的报道[36—38], 但本研究并未分离到蓝细菌的菌株。研究表明, 在长期营养缺乏或能量胁迫情况下, 海洋蓝细菌能够通过形成代谢活性非常低的休眠细胞而存活, 但过长时间的碳、氮饥饿或重金属胁迫会使蓝细菌发生黄化, 转移到新的培养基也很难存活[35]。Roth等人发现, 与异养细菌共培养能够延长蓝细菌在恶劣环境下的存活时间, 更有利于其进入休眠状态后的复苏[39]。另外, 对S2样本的宏基因组测序检测到了少量蓝细菌的存在[27], 但由于W2、S2样本所在环境水文压力过大, 其中蓝细菌进入休眠状态便难以复苏。故本研究在较浅的W1、S1样本共培养样品中发现了蓝细菌, 而10900m深的W2、S2样本中并未检测到。综上所述, 实验室常用的稀释平板涂布法会打断蓝细菌与异养菌之间的物质交换等胞间联系, 从而使蓝细菌无法简单地在平板上形成单菌落, 马里亚纳海沟样本中其他未培养微生物的生长限制可能也基于此。
3.2 共培养样品中潜在的微生物竞争本研究使用16S rDNA测序, 检测了马里亚纳海沟样本培养2d、3d、5d的微生物相对丰度, 其结果显示出一定的动态变化。其中, 表层沉积物样本对应的共培养样品中, 盐单胞菌属常能在培养后期占据优势地位, 海源菌属、鼠尾菌属、王祖农菌属在这种动态变化中也显示出较强的竞争力。来自不同生态系统的数据表明, 细菌间的竞争是普遍的。基因组分析表明, 几乎所有的放线菌将其5%—10%的基因组用于次级代谢产物, 包括抗生素和其他潜在的破坏性分子。Freilich等人预测, 大量采样的细菌种群之间存在大量竞争, 而单向积极相互作用的实例很少[40]。竞争排斥原则(高斯定律)指出, 具有相同生态位的两个物种由于竞争而不能共存, 竞争要么导致物种灭绝, 要么导致生态位的分化[41]。Ghoul和Mitri提出, 如果一个菌株表现出导致竞争菌株适应度下降的表型, 那么它就是有竞争力的。这些表型(如分泌消化酶、生产抗生素或可抑制群体感应)的进化往往是由于生物竞争而非环境压力。竞争可能是被动的, 也可能是主动的。在被动竞争中, 一个菌株通过消耗资源伤害另一个菌株, 如限制竞争对手对营养物质的获取;而在主动竞争中, 两个竞争菌株的单个细胞通过直接和主动干扰相互拮抗, 最终目标是消灭竞争对手;从菌株特异性细菌素到更广谱抗生素和多肽的抗菌素的生产, 都是主动竞争的例子[42]。微生物的种间竞争可以通过创造新的生态位来促进多样性的增加, 也可能由于空缺生态位的减少或种群规模的减小而抑制多样性[43]。在本研究中, 盐单胞菌属、海源菌属等类群显示出较强的竞争力, 而在寡营养液体共培养过程中, 这些类群的强竞争力似乎都导向竞争物种灭绝的结果, 与前人提出的规律相符。
但也有研究表明, 在群落生态学中, 竞争和相关的竞争排斥是群落聚集的主要驱动力[41]。莫冉等人建立了微生物互养关系的动力学模型, 系统地研究了互养关系中代谢资源的利用能否促进共存以及如何促进共存, 结果表明, 物种通过利用其他竞争成员的代谢产物, 打破外界资源量对其生长的限制, 使得原本被排斥的物种得以继续生长, 之后资源的竞争使得各物种及资源达到动态平衡, 从而实现竞争物种稳定共存[44]。在本研究中, 马里亚纳海沟这一极端环境中的寡营养条件, 使得微生物可能需要通过这种代谢资源的利用来组成独特的生态系统并维持其稳定性。
3.3 功能预测结果反映微生物互作存在原因本研究对共培养样品的功能预测结果显示, 化能异养、烃类降解在各样品中均显示出较高丰度;S1_E样品组中, 微生物生存必需通路的丰度随培养时间而降低, 负责微生物运动、分泌等与外界有信息交流的通路的丰度则显示出升高趋势。研究表明, 微生物群落构建中一个关键的过程是大量“公共产品”的细胞外分泌, 包括酶、蛋白质、副产品、废物、辅因子、氨基酸和维生素。它们有益于群落中所有能够吸收它们的生物。许多微生物缺少一些代谢物, 也缺乏必要的相关途径或基因, 因此需要依赖细胞外来源, 即通过其他生物体的分泌物获得所述代谢物[41]。故我们推测, 微生物在接种初期, 可能通过基础代谢途径快速竞争营养资源以满足生长, 当资源不足时, 则可能开始分泌代谢产物或进行细菌运动以进行胞间交流。根据“黑皇后假说”, 物种之间共享的代谢产物的这种“渗漏”允许物种间进行代谢相互依赖的进化, 包括通过基因缺失重新形成物种[45]。根据本研究的结果与前人研究, 我们推测, 深海环境中的微生物可能由于寡营养压力丢弃部分基因, 从而尤其需要与其他微生物进行胞间交流来保证生长。
4 结论与展望马里亚纳海沟等极端的深海环境孕育着独特的细菌群落结构和多样性特征。本研究选取寡营养培养基对马里亚纳海沟海水及表层沉积物样本进行液体共培养, 分别在不同培养阶段取样进行高通量测序, 共检测到19个门、34个纲、76个目、131个科、227个属的细菌, 其中变形菌门和拟杆菌门为优势菌群。共培养样品细菌多样性动态变化的结果表明, 马里亚纳海沟细菌群落中存在竞争、互利共生的相互作用, 共培养法有利于揭示细菌间的互作关系。
本研究结果也表明, 对于未培养微生物的生长限制, 实验室培养打断胞间联系确实是一大原因;若想进一步分离或利用深海微生物代谢产物, 可培养方法与共培养方法结合比较合适。研究表明, 利用与未培养微生物互作较为明显的可培养菌株作为助手或可弥补某些类群的代谢缺陷, 使其被分离培养, 从而丰富可培养微生物的多样性[22]。随着原位培养技术的发展, chamber、iChip、微囊包埋等技术可以实现不打断胞间联系进行培养, 但仍有操作困难、无法选择性分离等不足[46], 将本研究的液体共培养方法与原位培养结合, 或许能有更好的应用前景。
此外, 本研究对共培养样品的功能进行预测, 结果检测到了发酵、几丁质降解、木质素降解、尿素分解等的相关通路。以木质素为例, 木质素是植物细胞壁的主要成分, 具有重要的生物资源潜力。然而, 由于其顽固性, 对它的利用仍有难度;微生物酶能够帮助木质素分解, 微生物发酵在此方向上有较大应用价值[47]。本研究不仅可以指导分离更多的深海微生物, 液体共培养本身也具有作为代谢研究及应用开发的资源潜力。
致谢: 感谢中国科学院上海生命科学研究院李轩教授, 复旦大学生命科学学院生物多样性与生态工程教育部重点实验室的杨继教授、宋志平教授、王玉国教授、李霖锋教授对本研究提出的建议与帮助。| [1] |
Bienhold C, Zinger L, Boetius A, Ramette A. Diversity and biogeography of bathyal and abyssal seafloor bacteria. PLoS One, 2016, 11(1): e0148016. DOI:10.1371/journal.pone.0148016 |
| [2] |
Gamo T, Shitashima K. Chemical characteristics of hadal waters in the Izu-Ogasawara Trench of the western Pacific Ocean. Proceedings of the Japan Academy Series B, Physical and Biological Sciences, 2018, 94(1): 45-55. DOI:10.2183/pjab.94.004 |
| [3] |
Kamjam M, Sivalingam P, Deng Z, Hong K. Deep Sea actinomycetes and their secondary metabolites. Frontiers in Microbiology, 2017, 8: 760. DOI:10.3389/fmicb.2017.00760 |
| [4] |
陈柔雯, 王可欣, 何媛秋, 田新朋, 龙丽娟. 一份南海深海沉积物样品中可培养细菌的多样性. 生物资源, 2018, 40(4): 321-333. |
| [5] |
Jamieson A J, Fujii T, Mayor D J, Solan M, Priede I G. Hadal trenches: the ecology of the deepest places on Earth. Trends in Ecology & Evolution, 2010, 25(3): 190-197. |
| [6] |
Danovaro R, Dell'Anno A, Fabiano M, Pusceddu A, Tselepides A. Deep-sea ecosystem response to climate changes: the eastern Mediterranean case study. Trends in Ecology & Evolution, 2001, 16(9): 505-510. |
| [7] |
Jin M, Gai Y B, Guo X, Hou Y P, Zeng R Y. Properties and applications of extremozymes from deep-sea extremophilic microorganisms: a mini review. Marine Drugs, 2019, 17(12): 656. DOI:10.3390/md17120656 |
| [8] |
赵昌会, 叶德赞, 魏文铃. 深海微生物的研究进展. 微生物学通报, 2006, 33(3): 142-146. |
| [9] |
Pathom-Aree W, Stach J E M, Ward A C, Horikoshi K, Bull A T, Goodfellow M. Diversity of actinomycetes isolated from Challenger Deep sediment (10, 898 m) from the Mariana Trench. Extremophiles: Life Under Extreme Conditions, 2006, 10(3): 181-189. DOI:10.1007/s00792-005-0482-z |
| [10] |
Gooday A J, Uematsu K, Kitazato H, Toyofuku T, Young J R. Traces of dissolved particles, including coccoliths, in the tests of agglutinated foraminifera from the Challenger Deep (10, 897 m water depth, western equatorial Pacific). Deep Sea Research Part Ⅰ: Oceanographic Research Papers, 2010, 57(2): 239-247. DOI:10.1016/j.dsr.2009.11.003 |
| [11] |
朱坤杰, 何树平, 陈芳, 廖志良, 王金莲, 邓希光. 马里亚纳海沟南部海域沉积物的工程地质特性及其成因. 地质学刊, 2015, 39(2): 251-257. DOI:10.3969/j.issn.1674-3636.2015.02.251 |
| [12] |
Scheckenbach F, Hausmann K, Wylezich C, Weitere M, Arndt H. Large-scale patterns in biodiversity of microbial eukaryotes from the abyssal sea floor. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(1): 115-120. |
| [13] |
Nunoura T, Nishizawa M, Hirai M, Shimamura S, Harnvoravongchai P, Koide O, Morono Y, Fukui T, Inagaki F, Miyazaki J, Takaki Y, Ken T K. Microbial diversity in sediments from the bottom of the challenger deep, the Mariana trench. Microbes and Environments, 2018, 33(2): 186-194. DOI:10.1264/jsme2.ME17194 |
| [14] |
Nunoura T, Takaki Y, Hirai M, Shimamura S, Makabe A, Koide O, Kikuchi T, Miyazaki J, Koba K, Yoshida N, Sunamura M, Ken T K. Hadal biosphere: insight into the microbial ecosystem in the deepest ocean on Earth. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(11): E1230-E1236. |
| [15] |
Li Y J, Cao W R, Wang Y, Ma Q J. Microbial diversity in the sediments of the southern Mariana Trench. Journal of Oceanology and Limnology, 2019, 37(3): 1024-1029. DOI:10.1007/s00343-019-8131-z |
| [16] |
Xue C X, Liu J W, Lea-Smith D J, Rowley G, Lin H Y, Zheng Y F, Zhu X Y, Liang J C, Ahmad W, Todd J D, Zhang X H. Insights into the vertical stratification of microbial ecological roles across the deepest seawater column on earth. Microorganisms, 2020, 8(9): 1309. DOI:10.3390/microorganisms8091309 |
| [17] |
Huys G R, Raes J. Go with the flow or solitary confinement: a look inside the single-cell toolbox for isolation of rare and uncultured microbes. Current Opinion in Microbiology, 2018, 44: 1-8. DOI:10.1016/j.mib.2018.05.002 |
| [18] |
Joint I, Mühling M, Querellou J. Culturing marine bacteria-an essential prerequisite for biodiscovery. Microbial Biotechnology, 2010, 3(5): 564-575. DOI:10.1111/j.1751-7915.2010.00188.x |
| [19] |
邢磊, 赵圣国, 郑楠, 李松励, 王加启. 未培养微生物分离培养技术研究进展. 微生物学通报, 2017, 44(12): 3053-3066. |
| [20] |
Kouzuma A, Kato S, Watanabe K. Microbial interspecies interactions: recent findings in syntrophic consortia. Frontiers in Microbiology, 2015, 6: 477. |
| [21] |
Hoek T A, Axelrod K, Biancalani T, Yurtsev E A, Liu J H, Gore J. Resource availability modulates the cooperative and competitive nature of a microbial cross-feeding mutualism. PLoS Biology, 2016, 14(8): e1002540. DOI:10.1371/journal.pbio.1002540 |
| [22] |
王冲, 万洪善, 常亚奇, 孙绪军, 马泽亮. 基于微生物互作的共培养分离技术研究进展. 生物资源, 2021, 43(6): 606-616. |
| [23] |
周袁璐, 师苑, 陆跃乐, 朱林江, 陈小龙. 传统发酵过程微生物互作研究进展. 中国食品学报, 2021, 21(11): 349-358. |
| [24] |
张新生, 卢杰. 植物根系分布与根际微生态互作研究现状. 吉林林业科技, 2021, 50(6): 36-42. |
| [25] |
许萌萌, 孙雨婷, 陈虹, 杨德琴. 互作转录组测序技术在微生物研究中的应用进展. 四川大学学报: 医学版, 2022, 53(2): 201-207. |
| [26] |
张庆芳, 杨超, 于爽, 刘春莹, 迟乃玉. 黄海海域海洋沉积物细菌多样性分析. 微生物学通报, 2020, 47(2): 370-378. |
| [27] |
Chen P, Zhou H, Huang Y Y, Xie Z, Zhang M J, Wei Y L, Li J, Ma Y W, Luo M, Ding W M, Cao J W, Jiang T, Nan P, Fang J S, Li X. Revealing the full biosphere structure and versatile metabolic functions in the deepest ocean sediment of the Challenger Deep. Genome Biology, 2021, 22(1): 207. |
| [28] |
王震, 朱祥杰, 苑志欣, 郝建华, 郑兰红. 基于多种培养基和不同培养温度的马里亚纳海沟异养细菌多样性分析. 海洋湖沼通报, 2021(1): 91-100. |
| [29] |
李毅, 党研茹, 任雪冰, 察倩倩, 秦启龙, 夏呈强, 冯焱, 李宏, 张锋. 马里亚纳海沟可培养水生细菌的多样性. 微生物学报, 2021, 61(6): 1383-1398. |
| [30] |
黄莹, 秦纹静, 戴红, 张汝毅, 全哲学. 马里亚纳海沟不同深度水样微生物分离与多样性分析. 微生物学通报, 2020, 47(9): 2720-2731. |
| [31] |
Peoples L M, Donaldson S, Osuntokun O, Xia Q, Nelson A, Blanton J, Allen E E, Church M J, Bartlett D H. Vertically distinct microbial communities in the Mariana and Kermadec trenches. PLoS One, 2018, 13(4): e0195102. |
| [32] |
Cui G J, Li J, Gao Z M, Wang Y. Spatial variations of microbial communities in abyssal and hadal sediments across the Challenger Deep. PeerJ, 2019, 7: e6961. |
| [33] |
Elisabeth Hube A, Heyduck-Söller B, Fischer U. Phylogenetic classification of heterotrophic bacteria associated with filamentous marine cyanobacteria in culture. Systematic and Applied Microbiology, 2009, 32(4): 256-265. |
| [34] |
Coleman M L, Chisholm S W. Code and context: Prochlorococcus as a model for cross-scale biology. Trends in Microbiology, 2007, 15(9): 398-407. |
| [35] |
周玉婷, 李佳霖, 赵振军, 秦松. 海洋微微型蓝藻与异养细菌相互作用的研究进展. 海洋科学, 2022, 46(4): 123-132. |
| [36] |
Sher D, Thompson J W, Kashtan N, Croal L, Chisholm S W. Response of Prochlorococcus ecotypes to co-culture with diverse marine bacteria. The ISME Journal, 2011, 5(7): 1125-1132. |
| [37] |
Morris J J, Kirkegaard R, Szul M J, Johnson Z I, Zinser E R. Facilitation of robust growth of Prochlorococcus colonies and dilute liquid cultures by helper heterotrophic bacteria. Applied and Environmental Microbiology, 2008, 74(14): 4530-4534. |
| [38] |
Coe A, Ghizzoni J, LeGault K, Biller S, Roggensack S E, Chisholm S W. Survival of Prochlorococcus in extended darkness. Limnology and Oceanography, 2016, 61(4): 1375-1388. |
| [39] |
Roth-Rosenberg D, Aharonovich D, Luzzatto-Knaan T, Vogts A, Zoccarato L, Eigemann F, Nago N, Grossart H P, Voss M, Sher D. Prochlorococcus cells rely on microbial interactions rather than on chlorotic resting stages to survive long-term nutrient starvation. mBio, 2020, 11(4): e01846-20. |
| [40] |
Ghoul M, Mitri S. The ecology and evolution of microbial competition. Trends in Microbiology, 2016, 24(10): 833-845. |
| [41] |
Mataigne V, Vannier N, Vandenkoornhuyse P, Hacquard S. Microbial systems ecology to understand cross-feeding in microbiomes. Frontiers in Microbiology, 2021, 12: 780469. |
| [42] |
Bauer M A, Kainz K, Carmona-Gutierrez D, Madeo F. Microbial wars: competition in ecological niches and within the microbiome. Microbial Cell: Graz, Austria, 2018, 5(5): 215-219. |
| [43] |
Chu X L, Zhang Q G, Buckling A, Castledine M. Interspecific niche competition increases morphological diversity in multi-species microbial communities. Frontiers in Microbiology, 2021, 12: 699190. |
| [44] |
莫冉, 宋卫信, 李群, 张锋. 代谢互养关系在维持微生物物种多样性中的作用. 生态学报, 2021, 41(16): 6506-6512. |
| [45] |
Lopez J G, Wingreen N S. Noisy metabolism can promote microbial cross-feeding. eLife, 2022, 11: 70694. |
| [46] |
张作艳, 丁立建, 何山. 未培养微生物原位培养技术研究进展. 天然产物研究与开发, 2018, 30(5): 907-913. |
| [47] |
Kamimura N, Sakamoto S, Mitsuda N, Masai E J, Kajita S. Advances in microbial lignin degradation and its applications. Current Opinion in Biotechnology, 2019, 56: 179-186. |
 2023, Vol. 43
2023, Vol. 43




