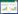文章信息
- 吴林坤, 李奇松, 李倩, 方媛, 蔡思思, 林文雄
- WU Linkun, LI Qisong, LI Qian, Fang Yuan, CAI Sisi, LIN Wenxiong
- 玉米和花生间作根际细菌群落结构与PICRUSt的功能预测
- Analysis of rhizosphere bacterial community structure and PICRUSt-predicted functional categories in maize and peanut intercropping systems
- 生态学报. 2023, 43(18): 7485-7496
- Acta Ecologica Sinica. 2023, 43(18): 7485-7496
- http://dx.doi.org/10.20103/j.stxb.202201210203
-
文章历史
- 收稿日期: 2022-01-21
- 网络出版日期: 2023-05-08
2. 福建省农业生态过程与安全监控重点实验室, 福州 350002;
3. 武夷学院茶与食品学院, 武夷山 354300
2. Fujian Provincial Key Laboratory of Agroecological Processing and Safety Monitoring, Fujian Agriculture and Forestry University, Fuzhou 350002, China;
3. College of Tea and Food Science, Wuyi University, Wuyishan 354300, China
间作是一种基于生物多样性的农业种植模式, 是实现生态农业和可持续农业的重要举措之一。其中, 禾本科/豆科作物间作是十分经典的间作模式, 节肥增产效果明显[1]。阐明禾本科/豆科作物间作的生态效益以及产量优势形成的内在机制, 对于推动现代生态农业发展、构建环境友好型可持续农业等均具有重要的理论和实践指导意义。
有关间作产量优势形成的机制研究主要聚焦于以下几个方面:(1)地上部、地下部生态位分化及种间资源互补[2];(2)土壤理化性质改善[3];(3)根系分泌物或植物挥发性物质的生态学效应[4—6];(4)土壤微生物群落结构与功能变化[7]。有研究指出, 玉米/蚕豆间作模式下, 蚕豆根系通过释放有机酸、质子来酸化根际, 从而有利于土壤难溶性磷的活化和玉米对磷的吸收利用, 而间作玉米通过竞争吸收土壤有效氮而减缓蚕豆的“氮阻遏”效应, 提高了蚕豆的根系结瘤和固氮水平[3—4, 8]。Wang等[9]研究发现, 玉米与苜蓿间作时, 苜蓿根系分泌物如羧酸盐、酸性磷酸酶能有效活化土壤难溶性磷、有机磷, 提升土壤磷有效性, 从而促进间作玉米的生长。关于根系分泌物方面, Li等[4]研究发现, 玉米的根系分泌物能够显著提升蚕豆根系黄酮类物质合成、结瘤相关基因的表达, 从而促进间作蚕豆的根系结瘤和固氮。此外, Fu等[10]研究发现, 分蘖洋葱伴生间作能够提高番茄根系分泌物对致病菌即大丽轮枝菌的抑制作用, 从而降低番茄黄萎病的发生。近年来, 有关间作产量优势的形成机制研究也开始关注土壤微生物群落与功能变化[7, 11]。有研究指出, 与单作玉米相比, 间作绿肥显著提高了土壤pH值、养分含量和酶活性, 调控了土壤微生物群落结构尤其是显著降低了潜在植物病原菌的相对丰度, 确保了长期连作条件下玉米的稳定高产[12]。Wang等[11]研究指出, 间作产量优势形成受到了土壤微生物组的介导调控, 间作能够通过抑制土传病原菌生长来降低植物-土壤负反馈效应。类似地, Sun等[13]研究发现, 与玉米间作, 可以显著提高蚕豆根际产铁载体细菌的数量, 进而抑制尖孢镰刀菌生长, 提高作物产量。此外, 也有研究指出间作模式下土壤理化性质的改善如土壤团聚体结构改善也与土壤微生物群落变化密切相关[14]。
课题组前期研究发现, 玉米/花生间作明显增加了土地当量比, 尤其是促进了玉米产量和花生根瘤的形成, 二者根际互作显著提高了两种作物根际中脲酶活性、磷酸单酯酶活性、速效磷含量等[15—16]。此外, 对玉米/花生间作模式下土壤微生物区系变化的初步分析也发现, 间作提高了玉米根际革兰氏阳性菌/革兰氏阴性菌比例, 而花生根际呈现相反趋势, 但玉米和花生根际真菌/细菌比例在间作模式下均降低[15—16], 但是对特异菌群变化规律以及群落功能变化尚缺乏系统研究。据此, 本研究设置了不同间作分隔方式(无隔、网隔、全隔), 采用高通量测序技术对不同间作模式下玉米、花生根际细菌群落结构和功能变化进行分析, 以期从微生物角度阐明玉米/花生间作产量优势形成的根际生态学机制。
1 材料与方法 1.1 试验设计及取样以“正大12号”玉米品种和“闽花8号”花生品种为试验材料进行间作, 设置了以下3种间作模式:无隔间作(即无分隔处理, 存在地下部互作过程, 二者根系可直接接触)、网隔间作(即50 μm尼龙网分隔, 存在地下部互作过程, 二者根系无法直接接触以排除空间竞争)、全隔间作(即塑料膜分隔, 无地下部互作过程)。不同间作模式下, 玉米与花生的地上部均未作分隔, 以保证不同处理之间作物群体地上部互作条件一致[15]。试验采用盆(直径35 cm、高40 cm)栽方式进行, 每盆装有13 kg的土壤(pH 5.51、有机质10.61 g/kg、总氮1.23 g/kg、总磷0.49 g/kg、总钾7.05 g/kg、速效氮40.65 mg/kg、速效磷12.62 mg/kg、速效钾31.60 mg/kg), 每盆种植玉米1株、花生3株, 玉米与花生间的株距为19 cm, 花生间的株距为13 cm。每个处理设置3个重复。整个试验期间, 不同处理之间的水肥管理措施等保持一致。
于玉米雄花期即种植60 d时进行玉米和花生根际土采集。取样前, 严格控制水份以避免过湿, 且保持各处理的土壤水份相近。取样时, 先完整挖出植株并小心分开玉米与花生根系, 抖动去除根系周围疏松土壤, 仅收集紧贴根系表面的少量土壤(每株土壤收集量尽量保持一致)作为根际土。收集的根际土过2mm筛子以去除动植物残体等。每个处理、每种植物各收集3份土壤。无隔、网隔、全隔间作模式下采集的玉米根际土分别命名为无隔玉米、网隔玉米、全隔玉米根际土, 花生类似命名。
1.2 根际土壤总DNA提取将上述采集的根际土壤利用DNA提取试剂盒BioFast soil Genomic DNA Extraction kit(BioFlux, 杭州, 中国)进行土壤DNA提取, 经1%琼脂糖凝胶电泳检测后, 利用Nanodrop 2000C Spectrophotometer (Thermo Scientific, 马萨诸塞州, USA) 进行DNA浓度测定。
1.3 高通量测序及分析质量合格的土壤DNA用于根际细菌群落高通量测序分析。利用带Barcode的特异引物341F (5′-CCTAYGGGRBGCASCAG-3′)和806R (5′-GGACTACNNGGGTATCTAAT-3′)扩增细菌16S V3—V4区[17], 并使用HiSeq2500 PE250进行测序。测序序列经拼装、质量控制过滤、去除嵌合体后获得最终的有效数据(Effective Tags)。接着, 利用Uparse软件对Effective Tags进行OTUs聚类(97%的一致性), 进一步利用Mothur方法与SILVA的SSUrRNA数据库对OTUs代表序列进行物种注释。基于KEGG数据库利用PICRUSt软件进行菌群功能预测分析。
1.4 统计分析各样品的群落组成数据经均一化处理后进行α-多样性(Observed species指数、ACE指数、Simposon多样性指数、香农-维纳多样性指数)和β-多样性分析如NMDS(非度量多维尺度, Non-metric multidimensional scaling)分析和UPGMA(非加权组平均法, Unweighted pair-group method with arithmetic means)聚类分析。采用DPS 7.05软件进行单因素方差分析(Tukey′s test, P<0.05)。韦恩图和热图绘制采用R软件(version 3.4.1)。基于细菌群落属水平相对丰度表和预测功能相对丰度表, 采用STAMP软件绘制扩展柱状图(Extended error bar)。利用R软件(version 3.4.1)计算细菌各菌属Spearman相关性系数(r>0.7、P<0.05), 再利用Gephi软件进行微生物共存网络图绘制及其拓扑特征分析。
2 结果 2.1 OTU分析与物种注释经质控处理, 从18个根际土壤样品中共获得1161405条有效序列(Effective Tags), 平均每个样品有64523条有效序列。稀释曲线分析表明当测序条数达40000条时, 曲线趋向平坦, 说明所有土壤样品的测序深度已几近饱和, 符合分析要求。以97%的一致性将有效序列聚类成为OTUs, 从无隔玉米(NM)、网隔玉米(SM)、全隔玉米(CM)、无隔花生(NP)、网隔花生(SP)、全隔花生(CP)中分别获得3041、3060、3022、3186、3066、3129个OTUs。进一步经物种注释分析发现, 90.6%、90.4%、88.9%、84.0%、68.2%、44.3%、6.1%的有效序列能成功注释到界、门、纲、目、科、属、种水平。
2.2 根际细菌群落α-多样性和β-多样性分析如表 1所示, 在玉米根际, 不同间作模式之间的丰富度指数(observed species指数、ACE指数)和多样性指数(Simposon多样性指数、香农-维纳多样性指数)均没有显著差异。在花生根际, 全隔花生的丰富度指数和多样性指数均显著高于网隔花生, 而无隔花生和网隔花生之间没有显著差异。
| α-多样性指数 α- diversity indices |
玉米Maize | 花生Peanut | |||||
| NM | SM | CM | NP | SP | CP | ||
| Observed species | 2516a | 2508 a | 2499 a | 2668 ab | 2558b | 2807a | |
| ACE | 3444.7a | 3421.0a | 3255.6a | 3589.9ab | 3414.4b | 3913.7a | |
| Simposon多样性指数 Simposon diversity index |
0.993a | 0.993a | 0.9933a | 0.992ab | 0.9913b | 0.9933a | |
| Shannon-Weiner多样性指数 Shannon-Weiner index |
8.895a | 8.8873a | 8.9747a | 9.0833ab | 8.9023b | 9.2163a | |
| 同一行不同字母表示差异显著Different letters in rows show significant differences (Tukey′s test, P<0.05, n=3); NM: 无隔玉米; SM: 网隔玉米; CM: 全隔玉米; NP: 无隔花生; SP: 网隔花生; CP: 全隔花生. | |||||||
基于OTUs相对丰度表, 对不同间作模式下玉米和花生根际细菌群落结构进行非度量多维尺度NMDS和UPGMA聚类分析, 结果显示:玉米根际的细菌群落结构明显区别于花生根际的细菌群落。而且, 对于玉米和花生两种作物, 不同间作模式之间根际细菌群落结构均差异明显。此外, 无隔和网隔玉米的根际细菌群落结构更为相似, 并区别于全隔玉米(图 1)。
|
| 图 1 不同间作模式下玉米与花生根际细菌群落非度量多维尺度分析和UPGMA聚类分析 Fig. 1 Non-metric multidimensional scaling (NMDS) ordination and UPGMA clustering analysis of bacterial communities in maize and peanut rhizosphere under different intercropping treatments 样品后的数字表示生物学重复 |
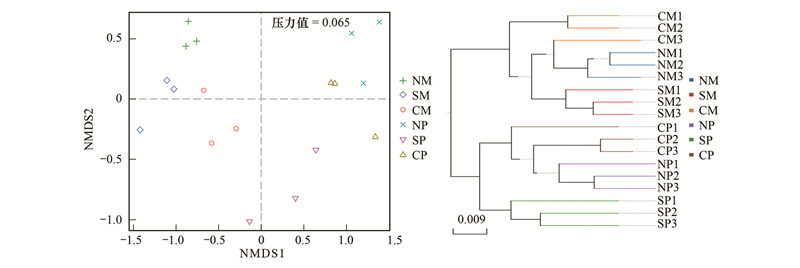
不同间作模式下, 玉米和花生根际优势细菌类群都主要包括变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、芽单胞菌门(Gemmatimonadetes)、酸杆菌门(Acidobacteria)、绿弯菌门(Chloroflexi)、拟杆菌门(Bacteroidetes)、硝化螺旋菌门(Nitrospirae)、奇古菌门(Thaumarchaeota)、疣微菌门(Verrucomicrobia)、厚壁菌门(Firmicutes)。对于玉米而言, 无隔玉米和网隔玉米根际放线菌门的相对丰度显著(P<0.05)高于全隔玉米, 而芽单胞菌门、绿弯菌门的相对丰度显著(P<0.05)低于全隔玉米。对于花生而言, 网隔花生根际放线菌门、厚壁菌门的相对丰度显著(P<0.05)高于全隔花生和无隔花生, 而芽单胞菌门、硝化螺旋菌门的相对丰度显著(P<0.05)低于全隔花生和无隔花生(图 2)。
|
| 图 2 不同间作模式下玉米与花生根际细菌群落门水平各类群相对含量变化分析 Fig. 2 Relative abundances of several bacterial phyla in maize and peanut rhizosphere under different intercropping treatments |
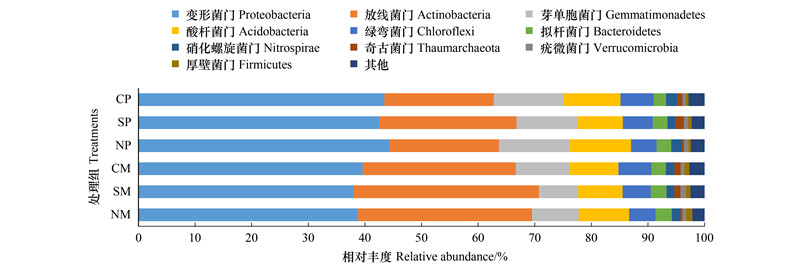
韦恩图分析还发现有952个OTUs其相对丰度在网隔、无隔玉米中高于全隔玉米, 有776个OTUs其相对丰度在网隔、无隔花生中高于全隔花生。进一步发现, 有259个OTUs不管在玉米还是花生根际均表现为网隔、无隔处理高于全隔处理, 并且主要注释为放线菌门、变形菌门、拟杆菌门、厚壁菌门、疣微菌门(图 3)。
|
| 图 3 无隔、网隔处理高于全隔处理的OTU韦恩图分析 Fig. 3 Venn diagram analysis based on the OTUs that were more abundant in no separation treatment and nylon-net separation treatment than in plastic-film separation treatment NM>CM表示无隔玉米高于全隔玉米的OTU; SM>CM表示网隔玉米高于全隔玉米的OTU; NP>CP表示无隔花生高于全隔花生的OTU; SP>CP表示网隔花生高于全隔花生的OTU |
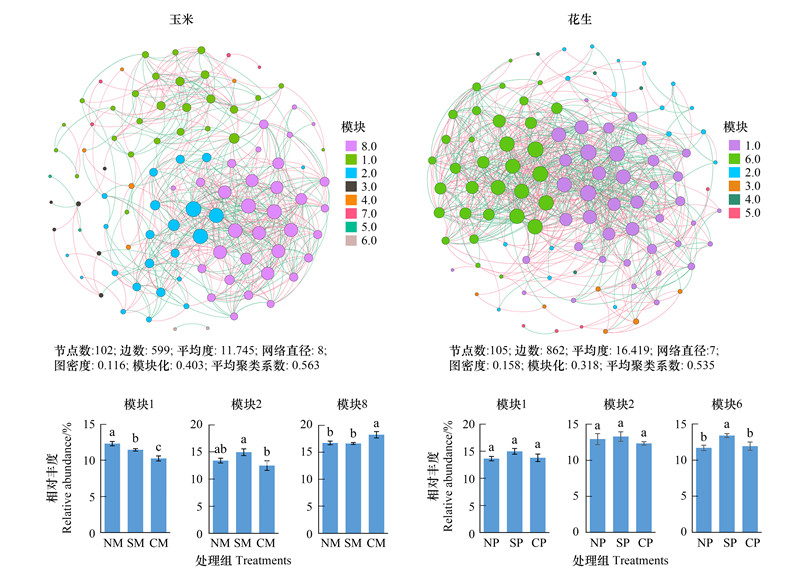
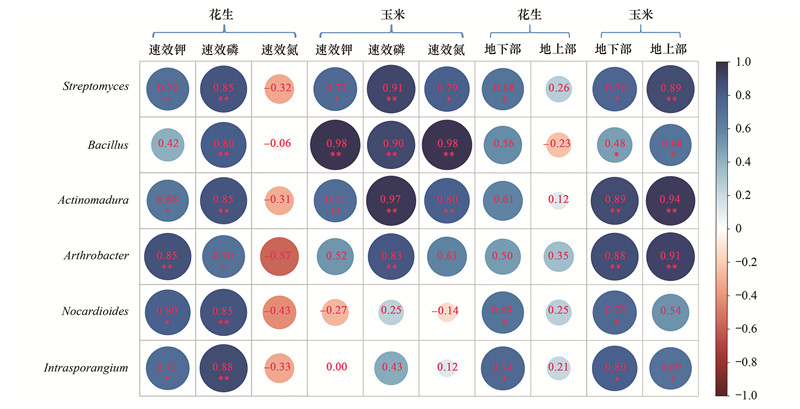
共存网络分析显示:花生根际细菌共存网络的复杂性更高, 表现在花生根际共存网络的节点数、边数、节点平均度高于玉米根际。玉米根际细菌共存网络中, 模块1、2、8的节点数较高, 为主要模块。与全隔玉米根际相比, 模块1、2中各菌属的总相对丰度在无隔和网隔玉米根际中更高, 而模块8表现出相反趋势。花生根际细菌共存网络中, 模块1、2、6的节点数较高, 为主要模块, 而且网隔花生根际模块6的总相对丰度显著高于无隔花生和全隔花生(图 4)。对玉米根际细菌共存网络的模块1、2、8中各菌属相对丰度作差异显著性(P<0.05、效应值η2>0.8)检验, 发现显著差异菌属主要属于放线菌门、变形菌门、厚壁菌门、硝化螺旋菌门等, 类似地花生根际细菌共存网络的模块1、2、6中差异菌属也属于放线菌门、变形菌门、厚壁菌门、硝化螺旋菌门(图 5)。进一步, 热图分析显示, 无隔玉米和网隔玉米根际的Streptomyces、Bacillus、Actinomadura、Arthrobacter等菌属的相对丰度都显著(P<0.05)高于全隔玉米, 而且网隔玉米根际的Ochrobactrum、Nocardioides、Intrasporangium等菌属也显著(P<0.05)高于全隔玉米, 然而Gemmatimonas、Mizugakiibacter、Rhodanobacter等呈现相反趋势。网隔花生根际Streptomyces、Actinomadura、Arthrobacter、Nocardioides、Intrasporangium等菌属的相对丰度显著(P<0.05)高于无隔或全隔花生, 这些菌属在玉米根际同样是网隔处理显著(P<0.05)高于全隔处理(图 5)。进一步, Pearson相关性分析显示, 玉米的土壤速效氮、速效磷、速效钾与Streptomyces、Bacillus、Actinomadura均呈显著正相关;花生的土壤速效磷、速效钾与Streptomyces、Actinomadura、Arthrobacter、Nocardioides、Intrasporangium等菌属丰度均呈显著正相关。此外, 玉米地上部、地下部生物量与Streptomyces、Bacillus、Actinomadura、Arthrobacter、Intrasporangium等菌属丰度呈显著正相关;花生地下部生物量与Streptomyces、Nocardioides、Intrasporangium菌属丰度呈显著正相关(图 6)。
|
| 图 4 玉米与花生根际细菌共存网络分析 Fig. 4 Co-occurrence network analysis of maize and peanut rhizosphere bacterial communities |

|
| 图 5 玉米与花生根际细菌共存网络优势模块中差异菌属热图分析 Fig. 5 Heatmap analysis of differential genera in dominant modules from maize and peanut rhizosphere bacterial networks 热图对应的值为每一行物种的相对丰度经标准化处理后得到的Z值 |
|
| 图 6 特定差异菌属与土壤速效养分、玉米花生生物量Pearson相关性分析 Fig. 6 Pearson correlation analysis between specific differential bacterial taxa and soil available nutrients and maize and peanut biomass * P<0.05; * * P<0.01 |
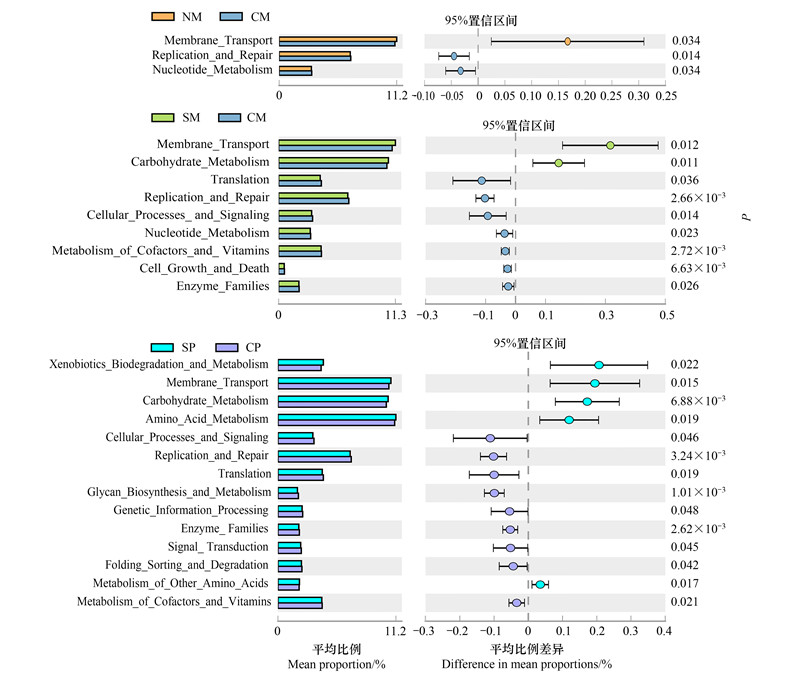
PICRUSt功能预测分析发现, 无隔玉米和网隔玉米根际的功能类如Membrane Transport(膜转运)显著高于全隔玉米, 而Repliction and Repair(复制与修复)、Nucleotide Metabolism(核酸代谢)显著低于全隔玉米。此外, 网隔玉米的Carbohydrate Metabolism(碳水化合物代谢)显著高于全隔玉米, 而Translation(蛋白质翻译)、Cellular Processes and Signaling(细胞过程与信号)、Metabolism of Cofactors and Vitamins(辅助因子和维生素代谢)、Cell Growth and Death(细胞生长与死亡)显著低于全隔玉米(图 7)。相似地, 网隔花生根际的功能类如膜转运、碳水化合物代谢显著高于全隔花生, 而复制与修复、蛋白质翻译、细胞过程与信号、辅助因子和维生素代谢等显著低于全隔花生(图 7)。
|
| 图 7 不同间作模式下玉米与花生根际细菌群落功能预测分析 Fig. 7 Function prediction analysis of bacterial community in maize and peanut rhizosphere under different intercropping treatments 过滤参数设置如下: P值>0.05, 相对丰度差异<0.02%. A: NM与CM比较; B: SM与CM比较; C: SP与CP比较. 该过滤条件下NP与CP之间无显著差异的功能类 |
植物微生物组作为植物的第二基因组, 对于宿主植物的生长发育与健康至关重要[18]。随着研究的不断深入, 越来越多研究指出豆科与禾本科间作产量优势的形成除了受到生态位分异、资源互补的驱动外, 还受到了根系分泌物和土壤微生物的介导调控, 尤其是土壤生物互作和植物-土壤反馈产生的间接促进作用扮演了重要角色[11, 19—20]。
本研究设置了三种不同的间隔方式, 即无隔间作(玉米、花生二者根系可直接接触)、网隔间作(二者根系无法直接接触但分泌物等可互相交流)、全隔间作(无地下部互作过程), 并采用高通量测序技术对不同间作模式下的玉米和花生根际细菌群落结构进行分析, 发现无隔、网隔玉米的细菌群落结构明显区别于全隔玉米, 网隔花生的细菌群落结构明显区别于全隔花生(图 1)。类似地, 课题组前期研究也发现, 无隔、网隔玉米的地上部生物量、地下部生物量、土壤速效磷含量等均显著高于全隔玉米;网隔花生的地下部生物量、根瘤数、单个根瘤重、土壤速效磷含量等均显著高于全隔花生, 而无隔花生的长势与结瘤能力介于二者之间[16]。可见, 不同分隔方式下根际细菌群落结构差异与宿主植物长势的变化趋势一致, 推测根际微生物组在玉米/花生间作产量优势的形成过程中发挥了重要作用。此外, 由于网隔处理下玉米与花生根系未发生直接接触, 但彼此的根系分泌物可互相交流, 故推测网隔与全隔处理下根际微生物组结构差异和来自对方根系分泌物的介导调控有关, 但具体调控过程与机制有待进一步研究。
3.2 间作下根际微生物组多样性及特异菌属丰度变化规律及潜在作用机理多样性指数分析发现, 玉米根际的细菌群落多样性指数在不同间作处理之间无显著性差异, 网隔花生的群落多样性指数甚至低于全隔花生(表 1)。有研究也指出, 与不同豆科绿肥作物间作, 玉米根际细菌、真菌和古菌群落的Shannon指数均无显著变化[12]。对小麦/西瓜、马铃薯/玉米间作、苜蓿/玉米间作的研究也有类似发现即间作并未提高细菌群落多样性[21—23]。有研究指出土壤生态系统功能主要取决于功能多样性而非分类多样性[24—25], 故推测玉米/花生间作产量优势的形成与根际细菌群落分类多样性变化没有直接联系, 可能更多受到了根际特异功能菌群的介导调控。本研究发现长势较好的无隔、网隔玉米其根际放线菌门及其所属类群(Streptomyces、Actinomadura、Arthrobacter)、Bacillus(属于厚壁菌门)相对丰度显著高于全隔玉米, 长势较好的网隔花生其根际放线菌门及其所属类群(Streptomyces、Actinomadura、Arthrobacter、Nocardioides、Intrasporangium)、厚壁菌门相对丰度显著高于全隔花生, 而两种作物根际芽单胞菌门呈现相反变化趋势(图 2、图 5)。而且, 在玉米和花生两种间作作物根际, 无隔、网隔处理高于全隔处理的OTU也均主要注释为放线菌门等(图 3)。这与前人研究相似, 如Gao等[26]研究发现, 烟草-花生间作提高了花生根际放线菌门、厚壁菌门丰度而降低了芽单胞菌门的相对丰度。类似地, 苜蓿/玉米间作提高了玉米根际放线菌门丰度而降低了两种作物根际芽单胞菌门丰度[23]。有大量研究表明, 土壤放线菌门、厚壁菌门细菌具有抑制病原菌生长、激活植物免疫、促进植物生长、降解复杂有机质以及增强土壤养分循环等功能[27—32]。Streptomyces、Bacillus、Arthrobacter、Actinomadura都是常见报道的植物促生菌或生防菌, 对维持土壤健康有重要作用[33—36]。还有研究表明, Streptomyces、Bacillus、Arthrobacter、Nocardioides等还具有生物固氮、溶磷解钾功能[37—42]。Pearson相关性分析也确实表明, 上述菌属丰度增加与玉米/花生间作下土壤速效养分增加有显著正相关性(图 6)。也有研究指出, 间作可以通过抑制土壤病原菌生长而缓解植物-土壤负反馈效应[5, 13, 43]。此外, 功能预测分析还发现, 无隔或网隔处理下玉米和花生根际细菌群落的膜转运、碳水化合物代谢等功能类的相对丰度显著高于全隔处理, 这可能与无隔或网隔处理下, 输入根际的根系分泌物等有机质种类和含量增多有关, 提升了根际微生物的代谢活性与功能。因此, 玉米/花生间作模式下, 这些有益菌属丰度的增加可能有利于减轻植物-土壤负反馈效应、加速养分循环与供应, 维持土壤健康, 进而促进间作植物的生长。
根系分泌物是根际微生物的重要营养与能量来源, 并且能够选择性塑造根际微生物组结构[44]。Hu等[19]利用稳定性同位素核酸探针技术研究发现, 玉米/蚕豆间作系统中, 玉米根系分泌物能够激发蚕豆根际的Arthrobacter、Bacillus、Paenibacillus等菌属生长, 进而提高蚕豆的生物固氮。Li等[45]研究发现, 与苍术间作, 引起花生根系分泌物中的酚酸类、有机酸类物质含量升高, 从而定向调控了花生根际微生物组结构, 增加了放线菌门某些类群如Pseudonocardiaceae的丰度, 而降低了镰刀菌属等病原菌丰度。也有研究指出, 与木薯间作, 能够刺激花生根系释放乙烯, 进而调控了花生根际微生物群落结构, 增加了放线菌如Catenulispora sp.的丰度, 提升了土壤质量[32]。此外, Dong等[46]研究指出, 玉米/花生间作可通过促进根际黄酮类物质分泌和Bradyrhizobium丰度来提高玉米植株氮积累量和产量。可见, 间作模式下, 根系分泌物在调控根际微生物组结构与功能中发挥了重要作用。故推测本研究中, 放线菌门、厚壁菌门、Streptomyces、Arthrobacter、Actinomadura等类群间作下丰度增加也受到了根系分泌物的介导调控, 但是具体是哪些根系分泌物起主要作用仍需进一步探究。
4 结论综上可见, 玉米/花生间作显著影响了二者根际细菌群落结构及其功能, 间作提高了作物根际放线菌门、厚壁菌门的相对丰度, 尤其是增加了Streptomyces、Bacillus、Arthrobacter、Actinomadura等常见报道的有益菌丰度。而且, 玉米/花生间作提高了膜转运、碳水化合物代谢等功能类的相对丰度而降低了复制与修复等功能类的相对丰度。本研究从土壤微生物角度阐明了玉米/花生间作下产量优势形成的根际生态学机制。但是, 后续仍需进一步关注禾本科/豆科间作下真菌群落结构变化及其生态学效应、根系分泌物的介导调控作用以及采用宏基因组学系统解析根际微生物组功能等。
| [1] |
Mudare S, Kanomanyanga J, Jiao X Q, Mabasa S, Lamichhane J R, Jing J Y, Cong W F. Yield and fertilizer benefits of maize/grain legume intercropping in China and Africa: a meta-analysis. Agronomy for Sustainable Development, 2022, 42(5): 81. DOI:10.1007/s13593-022-00816-1 |
| [2] |
张少斌, 梁开明, 郭靖, 罗颢. 基于生态位角度的农作物间套作增产机制研究进展. 福建农业学报, 2016, 31(9): 1005-1010. DOI:10.19303/j.issn.1008-0384.2016.09.020 |
| [3] |
Li L, Li S M, Sun J H, Zhou L L, Bao X G, Zhang H G, Zhang F S. Diversity enhances agricultural productivity via rhizosphere phosphorus facilitation on phosphorus-deficient soils. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(27): 11192-11196. |
| [4] |
Li B, Li Y Y, Wu H M, Zhang F F, Li C J, Li X X, Lambers H, Li L. Root exudates drive interspecific facilitation by enhancing nodulation and N2 fixation. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(23): 6496-6501. |
| [5] |
Zhang H, Mallik A, Zeng R S. Control of Panama disease of banana by rotating and intercropping with Chinese chive (Allium tuberosum rottler): role of plant volatiles. Journal of Chemical Ecology, 2013, 39(2): 243-252. DOI:10.1007/s10886-013-0243-x |
| [6] |
Zhang H, Yang Y X, Mei X Y, Li Y, Wu J Q, Li Y W, Wang H L, Huang H C, Yang M, He X H, Zhu S S, Liu Y X. Phenolic acids released in maize rhizosphere during maize-soybean intercropping inhibit Phytophthora blight of soybean. Frontiers in Plant Science, 2020, 11: 886. DOI:10.3389/fpls.2020.00886 |
| [7] |
Duchene O, Vian J F, Celette F. Intercropping with legume for agroecological cropping systems: Complementarity and facilitation processes and the importance of soil microorganisms. A review. Agriculture. Ecosystems & Environment, 2017, 240: 148-161. |
| [8] |
Li Y Y, Yu C B, Cheng X, Li C J, Sun J H, Zhang F S, Lambers H, Li L. Intercropping alleviates the inhibitory effect of N fertilization on nodulation and symbiotic N2 fixation of faba bean. Plant and Soil, 2009, 323(1): 295-308. |
| [9] |
Wang L Y, Hou B C, Zhang D S, Lyu Y, Zhang K, Li H G, Rengel Z, Shen J B. The niche complementarity driven by rhizosphere interactions enhances phosphorus-use efficiency in maize/alfalfa mixture. Food and Energy Security, 2020, 9(4): e252. DOI:10.1002/fes3.252 |
| [10] |
Fu X P, Wu X, Zhou X G, Liu S W, Shen Y H, Wu F Z. Companion cropping with potato onion enhances the disease resistance of tomato against Verticillium dahliae. Frontiers in Plant Science, 2015, 6: 726. |
| [11] |
Wang G Z, Bei S K, Li J P, Bao X G, Zhang J D, Schultz P A, Li H G, Li L, Zhang F S, Bever J D, Zhang J L. Soil microbial legacy drives crop diversity advantage: linking ecological plant-soil feedback with agricultural intercropping. Journal of Applied Ecology, 2021, 58(3): 496-506. DOI:10.1111/1365-2664.13802 |
| [12] |
Ablimit R, Li W K, Zhang J D, Gao H N, Zhao Y M, Cheng M M, Meng X Q, An L Z, Chen Y. Altering microbial community for improving soil properties and agricultural sustainability during a 10-year maize-green manure intercropping in Northwest China. Journal of Environmental Management, 2022, 321: 115859. DOI:10.1016/j.jenvman.2022.115859 |
| [13] |
Sun X Z, Zhang C C, Bei S K, Wang G Z, Geisen S, Bedoussac L, Christie P, Zhang J L. High bacterial diversity and siderophore-producing bacteria collectively suppress Fusarium oxysporum in maize/faba bean intercropping. Frontiers in Microbiology, 2022, 13: 972587. DOI:10.3389/fmicb.2022.972587 |
| [14] |
Tian X L, Wang C B, Bao X G, Wang P, Li X F, Yang S C, Ding G C, Christie P, Li L. Crop diversity facilitates soil aggregation in relation to soil microbial community composition driven by intercropping. Plant and Soil, 2019, 436(1): 173-192. |
| [15] |
Li Q S, Wu L K, Chen J, Khan M A, Luo X M, Lin W X. Biochemical and microbial properties of rhizospheres under maize/peanut intercropping. Journal of Integrative Agriculture, 2016, 15(1): 101-110. DOI:10.1016/S2095-3119(15)61089-9 |
| [16] |
Li Q S, Chen J, Wu L K, Luo X M, Li N, Arafat Y, Lin S, Lin W X. Belowground interactions impact the soil bacterial community, soil fertility, and crop yield in maize/peanut intercropping systems. International Journal of Molecular Sciences, 2018, 19(2): 622. DOI:10.3390/ijms19020622 |
| [17] |
Tan Y, Cui Y S, Li H Y, Kuang A X, Li X R, Wei Y L, Ji X L. Diversity and composition of rhizospheric soil and root endogenous bacteria in Panax notoginseng during continuous cropping practices. Journal of Basic Microbiology, 2017, 57(4): 337-344. DOI:10.1002/jobm.201600464 |
| [18] |
Berendsen R L, Pieterse C M J, Bakker P A H M. The rhizosphere microbiome and plant health. Trends in Plant Science, 2012, 17(8): 478-486. DOI:10.1016/j.tplants.2012.04.001 |
| [19] |
Hu H Y, Li H, Hao M M, Ren Y N, Zhang M K, Liu R Y, Zhang Y, Li G, Chen J S, Ning T Y, Kuzyakov Y. Nitrogen fixation and crop productivity enhancements co-driven by intercrop root exudates and key rhizosphere bacteria. Journal of Applied Ecology, 2021, 58(10): 2243-2255. DOI:10.1111/1365-2664.13964 |
| [20] |
Yu R P, Lambers H, Callaway R M, Wright A J, Li L. Belowground facilitation and trait matching: two or three to tango?. Trends in Plant Science, 2021, 26(12): 1227-1235. DOI:10.1016/j.tplants.2021.07.014 |
| [21] |
Yu H J, Chen S C, Zhang X X, Zhou X G, Wu F Z. Rhizosphere bacterial community in watermelon-wheat intercropping was more stable than in watermelon monoculture system under Fusarium oxysporum f.sp. niveum invasion. Plant and Soil, 2019, 445(1): 369-381. |
| [22] |
伏云珍, 马琨, 李倩, 李光文, 崔慧珍. 马铃薯||玉米间作对土壤细菌多样性的影响. 中国生态农业学报: 中英文, 2020, 28(11): 1715-1725. |
| [23] |
赵雅姣, 刘晓静, 吴勇, 童长春. 豆禾牧草间作根际土壤养分、酶活性及微生物群落特征. 中国沙漠, 2020, 40(3): 219-228. |
| [24] |
Chaparro J M, Sheflin A M, Manter D K, Vivanco J M. Manipulating the soil microbiome to increase soil health and plant fertility. Biology and Fertility of Soils, 2012, 48(5): 489-499. DOI:10.1007/s00374-012-0691-4 |
| [25] |
Lakshmanan V, Selvaraj G, Bais H P. Functional soil microbiome: belowground solutions to an aboveground problem. Plant Physiology, 2014, 166(2): 689-700. DOI:10.1104/pp.114.245811 |
| [26] |
Gao L, Liu X M, Du Y M, Zong H, Shen G M. Effects of tobacco-peanut relay intercropping on soil bacteria community structure. Annals of Microbiology, 2019, 69(13): 1531-1536. DOI:10.1007/s13213-019-01537-9 |
| [27] |
Fan F L, Yin C, Tang Y J, Li Z J, Song A L, Wakelin S A, Zou J, Liang Y C. Probing potential microbial coupling of carbon and nitrogen cycling during decomposition of maize residue by 13C-DNA-SIP. Soil Biology and Biochemistry, 2014, 70: 12-21. DOI:10.1016/j.soilbio.2013.12.002 |
| [28] |
Lee S M, Kong H G, Song G C, Ryu C M. Disruption of Firmicutes and Actinobacteria abundance in tomato rhizosphere causes the incidence of bacterial wilt disease. The ISME Journal, 2021, 15(1): 330-347. DOI:10.1038/s41396-020-00785-x |
| [29] |
Taschen E, Amenc L, Tournier E, Deleporte P, Malagoli P, Fustec J, Bru D, Philippot L, Bernard L. Cereal-legume intercropping modifies the dynamics of the active rhizospheric bacterial community. Rhizosphere, 2017, 3: 191-195. DOI:10.1016/j.rhisph.2017.04.011 |
| [30] |
Vanissa Tchuisseu Tchakounté G, Berger B, Patz S, Fankem H, Ruppel S. Community structure and plant growth-promoting potential of cultivable bacteria isolated from Cameroon soil. Microbiological Research, 2018, 214: 47-59. DOI:10.1016/j.micres.2018.05.008 |
| [31] |
Trivedi P, Delgado-Baquerizo M, Trivedi C, Hamonts K, Anderson I C, Singh B K. Keystone microbial taxa regulate the invasion of a fungal pathogen in agro-ecosystems. Soil Biology and Biochemistry, 2017, 111: 10-14. DOI:10.1016/j.soilbio.2017.03.013 |
| [32] |
Chen Y, Bonkowski M, Shen Y, Griffiths B S, Jiang Y J, Wang X Y, Sun B. Root ethylene mediates rhizosphere microbial community reconstruction when chemically detecting cyanide produced by neighbouring plants. Microbiome, 2020, 8(1): 4. DOI:10.1186/s40168-019-0775-6 |
| [33] |
Verma V C, Singh S K, Prakash S. Bio-control and plant growth promotion potential of siderophore producing endophytic Streptomyces from Azadirachta indica A. Juss.. Journal of Basic Microbiology, 2011, 51(5): 550-556. DOI:10.1002/jobm.201000155 |
| [34] |
Kloepper J W, Ryu C M, Zhang S A. Induced systemic resistance and promotion of plant growth by Bacillus spp. Phytopathology, 2004, 94(11): 1259-1266. DOI:10.1094/PHYTO.2004.94.11.1259 |
| [35] |
张幸博, 郭小红, 刘玉珍, 李建华, 王京, 孙晓伟, 法鹏飞, 危月辉. 烟草黑胫病生防菌LG-3的分离鉴定及防效研究. 湖北农业科学, 2021, 60(2): 82-84, 89. DOI:10.14088/j.cnki.issn0439-8114.2021.02.015 |
| [36] |
Li M S, Guo R, Yu F, Chen X, Zhao H Y, Li H X, Wu J. Indole-3-acetic acid biosynthesis pathways in the plant-beneficial bacterium Arthrobacter pascens ZZ21. International Journal of Molecular Sciences, 2018, 19(2): 443. DOI:10.3390/ijms19020443 |
| [37] |
Dahal B, NandaKafle G, Perkins L, Brözel V S. Diversity of free-Living nitrogen fixing Streptomyces in soils of the badlands of South Dakota. Microbiological Research, 2017, 195: 31-39. DOI:10.1016/j.micres.2016.11.004 |
| [38] |
Wan W J, Qin Y, Wu H Q, Zuo W L, He H M, Tan J D, Wang Y, He D L. Isolation and characterization of phosphorus solubilizing bacteria with multiple phosphorus sources utilizing capability and their potential for lead immobilization in soil. Frontiers in Microbiology, 2020, 11: 752. DOI:10.3389/fmicb.2020.00752 |
| [39] |
Wang Y J, Huang Q Q, Gao H, Zhang R Q, Yang L, Guo Y R, Li H K, Awasthi M K, Li G C. Long-term cover crops improved soil phosphorus availability in a rain-fed apple orchard. Chemosphere, 2021, 275: 130093. DOI:10.1016/j.chemosphere.2021.130093 |
| [40] |
保朱寿能. 紫茎泽兰(Ageratina adenophora)根内生固氮菌的分离及促生效应研究[D]. 昆明: 云南大学, 2017.
|
| [41] |
Ali A M, Awad M Y M, Hegab S A, El Gawad A M A, Eissa M A. Effect of potassium solubilizing bacteria (Bacillus cereus) on growth and yield of potato. Journal of Plant Nutrition, 2021, 44(3): 411-420. DOI:10.1080/01904167.2020.1822399 |
| [42] |
Boubekri K, Soumare A, Mardad I, Lyamlouli K, Hafidi M, Ouhdouch Y, Kouisni L. The screening of potassium- and phosphate-solubilizing Actinobacteria and the assessment of their ability to promote wheat growth parameters. Microorganisms, 2021, 9(3): 470. DOI:10.3390/microorganisms9030470 |
| [43] |
Wang G Z, Li H G, Christie P, Zhang F S, Zhang J L, Bever J D. Plant-soil feedback contributes to intercropping overyielding by reducing the negative effect of take-all on wheat and compensating the growth of faba bean. Plant and Soil, 2017, 415(1): 1-12. |
| [44] |
Trivedi P, Leach J E, Tringe S G, Sa T M, Singh B K. Plant-microbiome interactions: from community assembly to plant health. Nature Reviews Microbiology, 2020, 18(11): 607-621. DOI:10.1038/s41579-020-0412-1 |
| [45] |
Li X G, Yang Z, Zhang Y N, Yu L, Ding C F, Liao Y, Dai C C, Wang X X. Atractylodes lancea volatiles induce physiological responses in neighboring peanut plant during intercropping. Plant and Soil, 2020, 453(1): 409-422. |
| [46] |
Dong Q Q, Zhao X H, Zhou D Y, Liu Z H, Shi X L, Yuan Y, Jia P Y, Liu Y Y, Song P H, Wang X G, Jiang C J, Liu X B, Zhang H, Zhong C, Guo F, Wan S B, Yu H Q, Zhang Z. Maize and peanut intercropping improves the nitrogen accumulation and yield per plant of maize by promoting the secretion of flavonoids and abundance of Bradyrhizobium in rhizosphere. Frontiers in Plant Science, 2022, 13: 957336. DOI:10.3389/fpls.2022.957336 |
 2023, Vol. 43
2023, Vol. 43