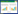文章信息
- 李苗, 陈小勇
- LI Miao, CHEN Xiaoyong
- 环境DNA技术在鱼类生态学中的应用研究进展
- Advances in the application of environmental DNA technology in fish ecology
- 生态学报. 2023, 43(17): 6951-6967
- Acta Ecologica Sinica. 2023, 43(17): 6951-6967
- http://dx.doi.org/10.20103/j.stxb.202208172362
-
文章历史
- 收稿日期: 2022-08-17
- 网络出版日期: 2023-04-28
2. 中国科学院昆明动物研究所遗传资源与进化国家重点实验室 & 云南省高黎贡山生物多样性与生态安全重点实验室, 昆明 650201;
3. 中国科学院东南亚生物多样性研究中心, 缅甸内比都 05282;
4. 云南省东南亚生物多样性保护国际联合实验室, 勐腊 666303
2. State Key Laboratory of Genetic Resources and Evolution & Yunnan Key Laboratory of Biodiversity and Ecological Conservation of Gaoligong Mountain, Kunming Institute of Zoology, Chinese Academy of Sciences, Kunming 650201, China;
3. Southeast Asia biodiversity Research Institute, Chinese Academy of Sciences, Nay Pyi Taw 05282, Myanmar;
4. Yunnan International Joint Laboratory of Southeast Asia Biodiversity Conservation, Mengla 666303, China
在全球变化的大背景之下, 生物多样性锐减是21世纪人类面临的重要挑战之一[1]。水生生态系统作为一种重要的生态系统, 其不仅能够为人类提供大量食物、工农业以及生活用水, 而且其还具有商业、航运交通以及休闲娱乐等其他诸多功能, 是人类社会快速发展必不可少的条件[2—3]。然而, 由于过度捕捞、生物入侵、水环境污染以及生态位的不断丧失和改变, 全球的水生生物多样性一直在持续下降[4]。鱼类作为水生生态系统中的重要组成部分之一, 其对于物质循环、能量流动以及生态系统的自我调节与稳定至关重要[5]。然而, 在长期高强度的人为干扰下, 全球鱼类的很大一部分正在迅速地走向灭绝[6]。据《地球生命力报告2020》的结果显示全球的地球生命力指数、物种生境指数以及生物多样性完整指数均在持续下降[7], 其中处于生物可持续水平的鱼类种群占比已由1974年的90%下降至2017年的65.8%[8]。为了能够有效的保护鱼类多样性, 对水生生态系统进行长期持续的监测至关重要。然而, 由于传统的鱼类资源调查方法存在一定的局限性而无法满足上述要求, 因而寻求一种行之有效的鱼类监测方法迫在眉睫。
近年来, 科研人员逐渐意识到环境DNA(eDNA)技术可作为传统生物监测方法的辅助手段之一, 在未来甚至能够完全取代传统方法成为一种常规化的水生生物监测方法。eDNA是指从水体、沉积物、土壤以及空气等环境样本中获取到的没有预先被分离的所有生物的总DNA[9—13]。对于鱼类等大型生物而言, 从环境中捕获到的DNA主要来源于生物体与环境之间的相互作用, 并通过皮肤细胞、毛发或鳞片的脱落以及代谢废物排泄的方式将遗传物质释放到环境中[12, 14]。科研人员可对eDNA进行采集, 并将其作为物种监测的证据[15]。相较于传统的鱼类监测方法而言, eDNA技术具有灵敏度高、经济高效、省时省力、采样受限小以及对生态系统无干扰等诸多优点[16], 因而倍受广大生态学研究领域科研人员的青睐。目前, eDNA技术已经被广泛的应用于国内外鱼类生态学的研究领域之中, 且表现出极大的潜力[17—20]。然而, 随着eDNA技术在鱼类生态学研究应用中的不断深入, 也暴露出其所存在的一些问题与缺陷, 例如:(1)eDNA技术在实际应用中操作流程的不规范导致不同研究之间的结果无法进行对比[21];(2)基因参考数据库的缺乏导致大量生境中实际存在的物种无法被进行有效的注释[22];(3)eDNA被释放后的生态学过程尚未明确, 致使研究结果中产生假阳性与假阴性错误[23]。上述问题的存在不仅揭露了eDNA技术目前在鱼类生态学研究领域内的不足之处, 同时也在一定程度指明了eDNA技术未来发展的方向以及科研工作者应该关注的焦点。
本文将系统的对eDNA技术的发展历程、分析流程、在鱼类生态学研究领域中的研究进展以及eDNA技术目前所面临的问题与挑战进行阐述, 并对eDNA技术未来的发展方向与应用前景做出了展望。通过本文, 以期能够对国内eDNA技术在鱼类生态学研究中的规范使用具有指导意义, 同时能够推动eDNA技术在国内的快速发展。
1 eDNA技术简介eDNA这一词汇最早于1987年被提出, 其最初被应用于沉积物中环境微生物学的研究[24], 而其真正得到研究人员的认可并被正式应用于生态学研究领域却是在2000年之后[25]。自Ficetola等首次应用eDNA技术对入侵物种美国牛蛙Rana catesbeiana进行监测之后[26], eDNA技术开始在水生生态系统这一研究领域得到了广泛应用, 其研究对象几乎囊括了所有水生生物类群, 包括鱼类[27—29]、两栖类[30—31]、哺乳类[32—33]、甲壳类[34—35]、水生昆虫[36—37]、微生物[38—39]以及浮游动植物[40—42]等。同时, 随着测序技术与聚合本酶链式反应(PCR)技术的发展, eDNA技术的应用由最初的单物种监测[26]发展到对整个生物群落的监测[43], 也由定性监测[26](目标种有无的监测)发展到了定量监测(生物量评估与丰度监测)[44]。总而言之, eDNA技术在水生生态系统中的应用为水生生物资源的生态调查开辟了一条新的道路。
2 eDNA技术的分析流程eDNA技术的操作流程主要包括eDNA样品采集、eDNA提取、PCR扩增、测序以及结果分析等主要步骤, 具体如图 1所示:
|
| 图 1 eDNA技术的操作流程 Fig. 1 Operation procedure of eDNA technology eDNA:环境DNA Environmental DNA;PCR:聚合酶链式反应Polymerase Chain Reaction;NGS:二代测序Next Generation Sequencing |
在鱼类生态学研究领域中eDNA样品的采集方式分为主动采集与被动采集两种, 其中主动采集方式包括水样采集与沉积物样品采集, 而被动采集则是在自然水域中寻找那些能够富集eDNA的生物作为天然的eDNA采样器。
2.1.1 水样采集与eDNA富集据现有的研究表明, eDNA水样的采集量一般集中在15 ml—10 L, 以1 L与2 L最为常见[45], 而在具体研究中水样的采集量通常会根据实验的需求有所差异。与组织DNA相比, 水环境中的DNA属于微量, 甚至是痕量, 所以在水样采集之后首先要对水体中的DNA进行富集以提高物种的检出率。关于eDNA的富集方式, 当前主要的方法有酒精沉淀法和过滤法[46], 前者主要用于小体积水样的DNA富集, 对于人造生态系统的水样采集较为适合, 而后者则主要用于大体积水样的DNA富集, 适合用来对河流与海洋等水域开阔的自然生态系统进行DNA富集。酒精沉淀法主要是用3 mol/L的醋酸钠溶液以及无水乙醇与水样混合来进行DNA富集[47], 而过滤法则是利用过滤装置对水样进行过滤将DNA截留在滤膜上以达到富集的效果[46], 在过滤过程中滤膜的合理选用至关重要。然而, 由于不同研究人员具有不同的研究目的与研究对象, 在利用过滤法进行DNA富集时所选取的滤膜也不尽相同。就目前现有的研究可知, 在进行eDNA富集时研究人员最为常用的滤膜为玻璃纤维膜、硝酸纤维膜、聚碳酸酯膜、混合纤维膜以及尼龙膜, 而最为常见的滤膜孔径则为0.45 μm与1.2 μm两种[48]。为了能够使研究结果更加精确, 作者建议在具体的研究中研究人员应该针对自己的研究对象对水样的采集量以及eDNA富集方法进行筛选, 以达到最佳的效果。
2.1.2 沉积物样品采集eDNA沉积物样品的采集方案应该根据水深、底质类型以及目标生物类群而定[49]。对于以鱼类为目标类群的研究而言, 表层沉积物样品的采样量一般集中在0.25—10 g[50], 采样设备的选用则根据生态系统的类型有所不同, 其中对于大型河流和深度为几百米以内浅海的沉积物样品一般选用抓斗进行采样[51—52], 而对于湖泊和深海等底质较为松软的沉积物样品则利用岩芯进行采样[53—54]。此外, 由于沉积物中DNA的混合不均匀, 通常还需要在采样点所在的区域内进行子样本的采集[55]。
2.1.3 eDNA的被动采集由于eDNA的主动采集方式往往需要科研人员在野外花费大量的时间采集样品, 为了能够节省野外调查的时间, 相关研究人员开始尝试从自然界中寻找天然的eDNA富集器或者设计富集能力很强的特殊材料来完成eDNA的被动采样, 诸如在海洋生态系统中以海绵作为纯天然的海水过滤器, 通过采集海绵来进行鱼类生态调查[56—58], 更有甚者是利用3D打印的羟基磷灰石作为DNA富集器将其置于采样点处来完成eDNA的被动采样[59]。然而, 上述被动采样的方式虽然在一定程度上节省了时间与经济成本, 但关于其在具体科学问题的解决方面是否合理有效还有待进一步的证实。
2.2 eDNA的提取鉴于eDNA样品成分复杂且含有较多的杂质, 为了能够提高后续实验中目标物种的检出率, 如何从样品中提取出高质量的DNA至关重要。在eDNA技术应用到水生生态系统研究领域的初期, 传统的酚-氯仿-异戊醇法以及各种商业化的试剂盒均被应用于eDNA的提取, 但提取效果参差不齐, 而后有研究人员专门对常用的几种商业化试剂盒与酚-氯仿-异戊醇法提取水环境中eDNA的效果做了对比, 发现使用商业化的试剂盒能够提取更高质量的eDNA[46]。目前最新的研究中基本上采用商业化的试剂盒来进行eDNA的提取, 其中提取水样eDNA使用较多的试剂盒为DNeasy Blood & Tissue Kit(QIAGEN)与Mo Bio Power Water DNA Extraction Kit两种[46, 60], 而提取沉积物样品eDNA使用较多的试剂盒主要为DNeasy PowerSoil DNA Isolation Kit (QIAGEN)与DNeasy PowerMax Soil Kit (QIAGEN)两种[49, 50]。
2.3 引物设计与PCR扩增PCR引物的设计和DNA条形码区段的选择在eDNA技术的应用中也是至关重要的一环, 这对后续实验结果的精确性具有决定性的作用。根据实验目的的不同, 通常可以分为目标物种的特异性监测和某特定生物类群的多样性监测。特异性监测主要是针对某一特定的物种设计特异性极强的引物从而能够在PCR过程中只扩增该物种的基因序列而不会扩增其他亲缘物种的基因序列, 多样性监测则是针对某特定的生物类群设计通用性引物, 要求能够尽可能多的覆盖该生物类群的基因序列。目前常用的引物设计软件主要有Primer Premier、Oligo、Beacon Designer、PrimerHunter以及Primer Express等[61—62], 引物设计在线网站主要有NCBI Primer-Blast、Primer3 Plus、Bath Primer、The PCR Suit、Primerbank以及PrimerX等。
目前研究人员选取的条形码区段主要集中在线粒体上, 主要原因在于与细胞核DNA(nDNA)相比, 线粒体DNA(mtDNA)具有以下三个优点[63]:(1)美国国家生物技术信息中心(NCBI)数据库中含有大量的mtDNA基因序列数据, 有利于进行eDNA的序列比对;(2)细胞中mtDNA的拷贝数要远高于nDNA的拷贝数;(3)mtDNA相对保守, 含有更多的能够进行物种鉴定的高变区以及易于设计通用引物的保守区。关于mtDNA目的基因的选择最常见的主要有(表 1)细胞色素c氧化酶亚基I (COI) 基因[80—81]、细胞色素b (Cytb) 基因[82—83]、12s核糖体RNA(12s rRNA)基因[22, 84]以及16s核糖体RNA(16s rRNA)基因[85—86]等, 而具体在研究中该选择哪个基因作为目标基因则取决于研究所选取生物类群的特征。同时, 引物的选择对eDNA监测的结果也具有一定的影响, 例如Zhang等通过Silico PCR的方法比较了22对硬骨鱼类的通用引物并将其用于北京市的河流水样中进行PCR扩增, 发现12s rRNA区段的引物能够监测到的鱼类多样性要优于其他基因区段的引物[87]。此外, Shu等类似的研究也证实了上述观点[88]。因此, 在选择PCR扩增所用的引物时要遵循以下3个原则:(1)由于eDNA降解严重, 因而引物扩增的片段不宜过长, 要保证能够尽可能多的回收样品中的DNA[89—90];(2)所选择的引物要保证具有良好的扩增效果, 既要能准确地扩增目标类群, 同时又不会发生非特异性扩增[91—92];(3)所选择引物扩增的目标区域最好能够具有完善的基因参考数据库, 能够使尽可能多的测序数据被注释到种的水平[93]。
| 目的基因 Gene of interest |
引物 Primers |
序列(5′-3′) Sequences(5′-3′) |
长度 Length |
参考文献 References |
| 12s rRNA | 12S_F1 12S_R1 |
ACTGGGATTAGATACCCC TAGAACAGGCTCCTCTAG |
106 bp | [64] |
| 12s rRNA | MiFish-U-F MiFish-U-R |
GTCGGTAAAACTCGTGCCAGC CATAGTGGGGTATCTAATCCCAGTTTG |
170 bp | [65] |
| 12s rRNA | MiFish-E-F MiFish-E2-R |
GTTGGTAAATCTCGTGCCAGC CATAGTAGGGTATCTAATCCTAGTTTG |
170 bp | [65] |
| 12s rRNA | Ac12S-F Ac12S-R |
ACTGGGATTAGATACCCCACTATG GAGAGTGACGGGCGGTGT |
385 bp | [66] |
| 12s rRNA | Am12S-F Am12S-R |
AGCCACCGCGGTTATACG CAAGTCCTTTGGGTTTTAAGC |
241 bp | [66] |
| 12s rRNA | Teleo Rdeg Teleo F |
CTTCCGGTACACTTACCRTG ACACCGCCCGTCACTCT |
100 bp | [67] |
| 12s rRNA | teleo_F teleo_R |
ACACCGCCCGTCACTCT CTTCCGGTACACTTACCATG |
100 bp | [67] |
| 12s rRNA | Taberlet-tele02-F Taberlet-tele02-R |
AAACTCGTGCCAGCCACC GGGTATCTAATCCCAGTTTG |
167 bp | [68] |
| 12s rRNA | AcMDB07-F AcMDB07-R |
GCCTATATACCGCCGTCG GTACACTTACCATGTTACGACTT |
300 bp | [69] |
| 16s rRNA | L2513 H2714 |
GCCTGTTTACCAAAAACATCA CTCCATAGGGTCTTCTCGTCTT |
202 bp | [70] |
| 16s rRNA | Ac16S-F Ac16S-R |
CCTTTTGCATCATGATTTAGC CAGGTGGCTGCTTTTAGGC |
330 bp | [66] |
| 16s rRNA | Ve16s-F Ve16s-R |
CGAGAAGACCCTATGGAGCTTA AATCGTTGAACAAACGAACC |
310 bp | [66] |
| 16s rRNA | 16S fish-specific F 16S fish-specific R |
GGTCGCCCCAACCRAAG CGAGAAGACCCTWTGGAGCTTIAG |
100 bp | [71] |
| 16s rRNA | Vert-16S-eDNAF1 Vert-16S-eDNAR1 |
AGACGAGAAGACCCYdTGGAGCTT GATCCAACATCGAGGTCGTAA |
250 bp | [72] |
| 16s rRNA | 16SF/D 16S2R-degenerate |
GACCCTATGGAGCTTTAGAC CGCTGTTATCCCTADRGTAACT |
219 bp | [73] |
| COI | PS1-F PS1-R |
ACCTGCCTGCCGTATTTGGYGCYTGRGCCG GRATAGT ACGCCACCGAGCC |
247 bp | [74] |
| Cytb | Cytb_L14841 Cytb_15149R |
AAAAACCACCGTTGTTATTCAACTA GCDCCTCARAATGAYATTTGTCCTCA |
413 bp | [75] |
| Cytb | L14735 H15149c |
AAAAACCACCGTTGTTATTCAACTA GCCCCTCAGAATGATATTTGTCCTCA |
413 bp | [76] |
| Cytb | L14912 H15149 |
TTCCTAGCCATACAYTAYAC GGTGGCKCCTCAGAAGGACATTTGKCCYCA |
285 bp | [77] |
| Cytb | Fish2bCBR Fish2CBL |
GATGGCGTAGGCAAACAAGA ACAACTTCACCCCTGCAAAC |
80 bp | [47] |
| Cytb | Fish2degCBL Fish2CBR |
ACAACTTCACCCCTGCRAAY GATGGCGTAGGCAAATAGGA |
80 bp | [47, 78] |
| Cytb | FishCBL FishCBR |
TCCTTTTGAGGCGCTACAGT GGAATGCGAAGAATCGTGTT |
130 bp | [78] |
| Mitogenomes | Actinopterygii16SLRpcr_F Actinopterygii16SLRpcr_R |
CAGGACATCCTAATGGTGCAG ATCCAACATCGAGGTCGTAAAC |
16 kb | [79] |
在应用eDNA技术进行单物种监测时, PCR产物用传统的一代测序即可, 而在用宏条形码进行物种多样性监测时, 往往用高通量测序(NGS)更为合适, 主要原因在于高通量测序速度快、数据量大且准确性高。目前, eDNA研究领域中应用的高通量测序主要为扩增子测序, 应用较多的测序平台主要为Illumina Miseq、Illumina Hiseq、Illumina NextSeq以及Roche GS FLX 454等[47, 94—96]。
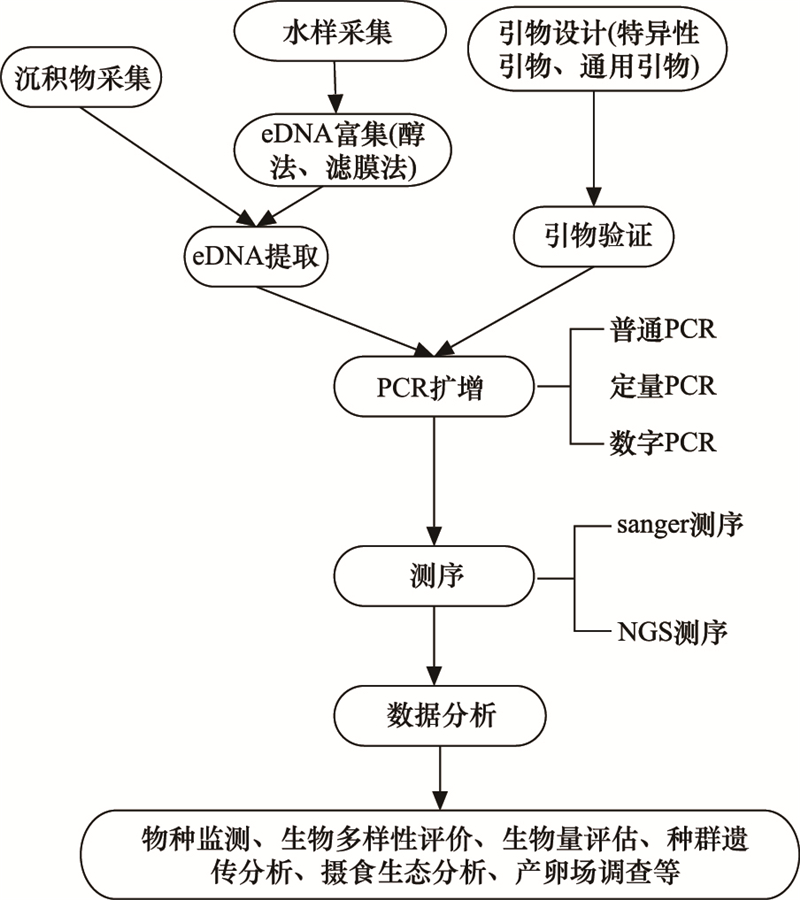
测序之后就是对数据进行分析, 一代测序的数据在NCBI上进行比对即可确定目标种的有无, 高通量测序的原始下机数据处理则较为复杂, 其主要流程包括质量控制、过滤、分子可操作分类单元(MOTU)聚类以及对代表性MOTU序列进行BLAST比对和物种注释(图 2)。目前测序数据处理的主流软件主要有Mothur[97]、OBITools[87]、Usearch[98]、Vsearch[99]、PhyloPythia[100]、Cutadapt[95]、QIIME[101]以及QIIME 2[102]等。通常来说, 对高通量测序数据的分析要求科研人员具有一定的编程基础, 但为了方便那些无编程背景的科研人员, 生物公司通常开发了高通量测序数据处理的生物信息云计算平台, 例如MiFish Pipeline (http://mitofish.aori.u-tokyo.ac.jp/)鱼类多样性在线数据分析平台等[103]。
|
| 图 2 测序数据的分析流程 Fig. 2 Analysis process of sequencing data MOTU:分子可操作分类单元Molecular Operational Taxonomic Units |
自eDNA技术被引入水生生态系统的研究领域以来, 在鱼类生态学研究中得到了广泛的应用。目前, eDNA技术主要被用来进行鱼类的目标物种监测、生物量评估、多样性监测与评价、繁殖活动监测、种群遗传分析以及摄食生态等方面的研究[23, 104]。在本部分, 将以eDNA技术在鱼类生态学研究中应用最为广泛的4个方向展开论述。
3.1 目标物种监测精确地掌握目标物种的空间分布与丰度情况对研究物种的生态习性与制定相关的保护政策至关重要[105]。对于鱼类等水生生物而言, 其所生存的环境在一定程度上增加了物种监测的困难性[48], 尤其是针对那些种群密度极小物种的调查, 诸如隐存种、濒危种、稀有种以及处于入侵早期的入侵种。同时, 由于传统方法往往很难捕获到密度极小的种群, 从而无法精确地掌握其种群动态。鉴于上述原因, 科研人员尝试利用eDNA技术进行极小密度种群的监测。目前, eDNA技术在极小密度种群监测应用中的成功案例主要有入侵鱼类与珍惜濒危鱼类的监测两个方面。
生物入侵是生物多样性丧失和全球同质化的主要原因之一[106—107]。一旦入侵成功, 入侵种会对当地的农业、林业、渔业、电力生产以及贸易产生严重的影响, 进而造成当地经济的大量损失[108]。为了能够有效地遏制生物入侵, 最佳的解决办法便是在入侵种尚未形成较大种群时就将其消除[107]。Nakao等利用eDNA技术对5个大坝库区的大口黑鲈Micropterus salmoides、小口黑鲈Micropterus dolomieu以及蓝鳃太阳鱼Lepomis macrochirus这3种入侵鱼类进行了监测, 结果表明eDNA技术可以被作为一种生物入侵监测的工具, 但同时也发现了一些问题, 例如该技术在一些eDNA浓度较低的采样点灵敏度较低[19]。因此, 未来在用eDNA技术进行入侵种的监测时, 尚有许多难题需要攻克。
珍稀濒危鱼类通常因其极小的种群密度而难以用传统的调查方法对其种群动态进行监测[107], 而掌握其种群的分布与丰度状况对于保护策略的制定至关重要[109]。许多珍稀濒危物种自种群数量急剧下降之后便很难用传统的方法对其进行监测, 这为科研人员的工作带来了极大的困难, eDNA技术的出现则使上述情况出现了转机。Cardás等利用eDNA技术对西班牙北部纳隆河和萨拉河的欧洲鳗鲡Anguilla anguilla进行了监测[110], 证实了eDNA在濒危鱼类监测方面的潜力。此外, Budd等利用eDNA技术对美国关岛西太平洋海域的路氏双髻鲨Sphyrna lewini 进行了监测, 并建立了相关的监测体系, 其中特别值得关注的是基于传统方法的调查自1970年开始便再未捕获到路氏双髻鲨[111]。由此可见, eDNA技术是一种强有力的珍稀濒危鱼类监测工具。
3.2 生物量评估生物量是生态学研究领域中重要的基础生物学参数之一[112], 准确地评估生物量对于种群大小的管理[113—114]、自然资源的保护[115]、生态系统的功能与服务[116]以及生态交互作用[117]具有十分重要的意义。自上个世纪以来, 有关动物种群生物量评估方法的研究一直是科研人员与政策制定者所关注的焦点[118]。然而, 对于鱼类等水生生物类群而言, 准确的评估其生物量是十分困难的[112]。同时, 现有的评估方法通常成本高昂、耗时耗力且对于种群数量极小的物种无法开展有效的工作[119—120]。为了解决上述问题带来的困扰, Takahara等首次提出利用eDNA技术来评估鱼类的生物量, 并将该技术应用到了鲤Cyprinus carpio的生物量评估中, 其原理主要是寻找鱼类释放到水环境中的eDNA的浓度与鱼类生物量之间的关系, 进而建立相应的生物量评估模型[112]。自此之后, 利用eDNA技术探究鱼类生物量的研究日益增多。为了摸清鱼类的生长对eDNA技术进行生物量评估的影响, Mizumoto等以日本北部的濒危物种远东哲罗鲑Parahucho perryi为研究对象, 探究了由不同生长阶段的远东哲罗鱼组成的鱼群释放eDNA的浓度与其生物量之间的关系, 结果表明在鱼类生物量恒定的情况下, 不同规格的鱼所组成的鱼群释放的DNA的浓度是相等的[121]。然而, 以往在自然水域中进行的研究, 结果通常显示eDNA的浓度与生物量之间的相关性并不是很强[119]。为了能够进一步提高利用eDNA技术进行生物量评估结果的精确性, Fukaya等提出在利用eDNA评估鱼类生物量时应充分考虑eDNA时空分布形成的机制, 并以日本竹筴鱼Trachurus japonicus为研究对象证实了只有在某些特定的假设条件下才能够利用广义线性模型来准确地评估鱼类的种群数量[122]。但时至今日, 关于eDNA在水环境中动态变化的情况尚无定论, 而这始终是阻碍利用eDNA技术评估鱼类生物量的一大障碍。因此, 在未来一段时间内, 如何利用eDNA技术精确地评估鱼类的生物量仍是该领域内的热点与难点问题之一。
3.3 生物多样性监测与评价生物多样性是衡量物种丰富程度的重要指标之一, 而生物多样性调查则是开展生物多样性评价的重要基础性工作, 如何获取精确的调查数据将会对后续的研究工作产生重要的影响。传统的鱼类多样性调查方法主要依赖于鱼类标本的采集, 诸如底拖网、围网、流刺网、地笼以及电捕鱼法等, 但上述方法往往具有一定的选择性, 不能够全面系统地对研究区域内的鱼类多样性做出精确的评价。随着分子生物学技术的不断发展, Thomsen等开创性地应用eDNA技术对丹麦海域的鱼类多样性进行了调查, 结果发现了15种鱼类, 其中包括一罕见种——欧洲沙丁鱼Sardina pilchardus。同时, 为了验证eDNA技术在鱼类多样性监测方面的效率, Thomsen等还将eDNA调查的结果与9种传统调查方法的结果进行了对比, 表明利用eDNA监测到的鱼类物种数高于或者等于传统方法, 证实了eDNA在鱼类多样性调查方面具有极大的潜力[47]。此后, 利用eDNA进行鱼类多样性调查的相关研究日益增多[65—66]。
在利用eDNA技术进行鱼类多样性研究的初期, 研究人员最为关注的是eDNA技术在鱼类监测方面的能力, 相关研究的主题主要是利用eDNA技术与传统方法同时对某特定研究区域内的鱼类多样性进行调查, 进而验证eDNA技术是否可被用来进行鱼类多样性的调查[47]。在经过大量研究的证实之后, eDNA技术逐渐开始被用来解决具体的科学问题, 例如鱼类群落时空分布的动态变化[123—124]、鱼类群落中各物种相对丰度的情况[66, 125]、鱼类群落的系统发育多样性[126]以及功能多样性[127—128]等。然而, 生物多样性监测的目的是为了进行生物多样性评价。为了得到准确的评价结果, 如何选择合适的评价指标与标准尤为重要[129—130]。尽管基于eDNA技术的监测方法能够提供比传统方法更多的可用于生物多样性评价的信息[131], 但目前尚未有一套基于eDNA技术的生物多样性评价体系。未来要想将基于eDNA技术的方法整合到现有的生物多样性监测与评价体系中, 还需要从国家与国际的层面来采取行动以制定相关的标准[131]。
3.4 鱼类繁殖活动监测精确地掌握鱼类产卵的时间与场所有助于人类更加清楚地认识鱼类的繁殖习性、准确地评估鱼类种群的繁殖力, 进而能够针对当前的资源状况制定相关的开发利用与保护策略。然而, 当前在鱼类早期资源调查中所用到的调查方法通常会导致受精卵与仔稚鱼死亡率的升高, 进而会对受威胁种与稀有种的种群繁殖造成极大的伤害。鉴于上述原因的考虑, 相关科研人员尝试利用eDNA技术来探究鱼类产卵的时间与场所。Bylemans等利用eDNA技术对澳洲麦氏鲈Macquaria australasica的产卵时间与场所进行了调查, 研究发现在繁殖期水体中nDNA的量要显著高于mtDNA的量, 而在其他时期nDNA的量与mtDNA的量大致相等。因此, 可以根据栖息环境中鱼类nDNA与mtDNA相对丰度的变化来识别产卵时间与场所[69]。Bracken等以海七鳃鳗Petromyzon marinus为研究对象, 利用qPCR对爱尔兰境内2条河流中固定点位eDNA的时空分布进行了连续3年的监测, 研究结果发现在海七鳃鳗的繁殖期, 河流中eDNA的浓度将会显著升高, 表明eDNA技术可以被用来进行鱼类繁殖活动的监测[132]。此外, 还有大量类似的研究同样支持利用eDNA技术进行鱼类繁殖活动的监测[133—135]。
尽管目前已有大量研究利用eDNA技术对鱼类繁殖活动进行了监测, 并证明eDNA技术在该方面具有一定的潜力, 但其在实际应用中仍存在些许问题。就当前利用eDNA技术进行鱼类繁殖活动监测的研究而言, 所选取的研究对象多为人类已熟知其生活习性并掌握了其繁殖时间的物种, 从而能够使科研人员根据已掌握的情况结合eDNA量的变化来推断鱼类的产卵活动, 而对那些生活习性没有被人类所掌握的鱼类来说, 则无法使用该方法来进行判断。因此, 未来应着重探究在尚未熟知鱼类生态习性的背景下如何利用eDNA技术精确地识别鱼类的繁殖时间与繁殖场所。
4 eDNA技术在鱼类生态学研究领域中面临的挑战应用eDNA技术进行鱼类调查的主要目的是为了能够精确的掌握某特定研究区域内鱼类资源的状况[79], 从而解决一些具体的科学问题。因此, 为了能够保证eDNA研究结果的精确性, 阐明影响eDNA研究结果精确性的因素至关重要[136]。目前, eDNA技术在鱼类生态学研究领域的具体应用中产生的错误主要有假阳性与假阴性两种类型, 而造成上述错误的主要原因来自于eDNA释放到环境中之后的一系列生态学过程以及eDNA分析流程中的各个方面。鉴于此, 为了能够使科研人员在应用eDNA技术的过程中尽量避免假阳性与假阴性错误的产生, 本部分将针对造成eDNA研究结果中各类错误的原因展开详细的论述并提出可能的解决方案。
4.1 eDNA技术操作流程的标准化随着eDNA技术在鱼类生态学研究领域内的日益流行, 寻求一套能够使研究结果最佳的eDNA技术操作流程与实验方案已成为科研人员关注的热点问题之一[137—138]。当前已有研究表明, 水样采集量、eDNA的富集与提取方法、PCR扩增目的基因的选择等均会对研究结果的精确性产生影响[27, 46, 139]。同时, 也有大量研究对eDNA技术的操作流程进行了优化, 但现有的研究更多的是针对某些特定的生物类群以及研究区域所做出的优化, 在一定程度上不具有普适性, 从而也就使不同区域的研究结果无法进行相应的对比[21, 48]。为了能够在更大尺度上使利用eDNA技术调查获得的数据具有可比较性, 同时能够使政策制定者在相应政策的制定上不做出误判, eDNA技术的应用必须规范化、标准化[21]。目前, 欧洲标准化委员会(CEN)已经在讨论制定欧盟成员国在使用eDNA技术进行生物监测时的标准化问题[140], 日本也成立了专门的eDNA学会并编写了一本eDNA技术标准化的研究手册[21]—“Environmental DNA Sampling and Experiment Manual” (http://ednasociety.org/en/manual)。然而, 目前国内关于eDNA领域的研究日益增多, 但不同单位的科研人员在采样以及实验方案的设计等方面存在诸多差异, 这将使不同研究之间结果的精确性无法被进行准确地评估。因此, 为了能够使eDNA技术在国内广泛的正确使用, 政府部门应该尽快出台相应的研究手册, 以确保在全国范围内使用eDNA技术获取的生物监测数据能够具有良好的对比性。
针对上述eDNA技术分析流程标准化的问题, 相关部门在编写eDNA研究手册时应着重考虑样品采集、DNA富集与提取、PCR扩增以及后续的生物信息学分析等方面标准的制定。目前, 已有不少研究专门针对eDNA技术的分析流程进行了优化, 相关研究结果可作为eDNA技术标准化的参考依据[46, 55, 141—142]。通过对现有文献的整理即可发现在鱼类生态研究领域中eDNA样品的采集应以水样采集为主, 水样采集地点要尽可能覆盖各种生境类型, 水样的采集量则以1 L或2 L为最佳且每个采样点至少采集3个平行样本[94, 143]。关于eDNA的富集方法一般推荐使用过滤的方法将DNA截留在滤膜上, 而滤膜材质与孔径的选择则主要倾向于0.45 μm的玻璃纤维滤膜[141]。富集之后则需要对eDNA进行提取, 就eDNA的提取而言当前主要推荐使用商业化的试剂盒, 其中提取效果较好的试剂盒主要为Qiagen′s DNeasy Blood & Tissue Kit与Mo Bio Power Water DNA Extraction Kit两种[46, 60]。eDNA提取之后PCR扩增测序过程中需要标准化的主要是引物的选取, 当前在关于引物的选用方面较为推荐使用12s rRNA基因的通用引物[87, 88]。测序之后需要标准化的则为生物信息学分析流程, 尽管目前已有大量用于生信分析的软件, 但不同软件在算法上存在诸多差异, 未来最好能够开发出一套专门针对鱼类eDNA高通量测序数据处理的软件与分析流程[144]。相信通过对eDNA技术的分析流程进行标准化之后, 将能够使eDNA研究结果的精确性得到极大的提升。
4.2 参考数据库的缺乏在eDNA技术成为主流的鱼类资源调查方法之前, 尚有许多方法论上的困难需要克服[65], 其中之一便是DNA序列参考数据库的不完全以及现有的数据库中存在诸多错误信息[22]。目前, eDNA研究中常用的公共参考数据库主要有NCBI的基因数据库(http://www.ncbi.nlm.nih.gov)、欧洲分子生物学实验室的数据库(EMBL)(http://www.ebi.ac.uk/embl)、生命条形码数据库(BOLD)(http://v4.boldsystems.org)、渔业生物条形码计划在线数据库(FISH-BOL)(http://www.fishbol.org)以及日本的鱼类线粒体基因组数据库(MitoFish)(http://mitofish.aori.u-tokyo.ac.jp)等。然而, 鉴于上述数据库中大量错误信息的存在与鱼类12s rRNA基因序列的极度匮乏, 科研人员逐渐意识到建立本土鱼类基因数据库的重要性, 且现已有相关研究证明相较于公共数据库而言, 本土鱼类基因数据库能提供更加精确的物种分类注释信息, 对eDNA研究结果精确性的提高具有十分重要的意义[31, 145]。
为了解决上述数据库不完全的问题, 科研人员在利用传统方法进行鱼类资源调查时应尽可能广泛地收集构建鱼类基因数据库所需的鱼类组织材料, 以确保构建的数据库能够在最大程度上涵盖某海域或流域的鱼类物种信息。同时, 为了保障数据库的精确性, 数据库中应包含每个鱼类物种的样品采集信息、分类鉴定信息以及DNA条形码信息, 尤其是对于那些形态学上较难辨认的物种要格外注意。此外, 关于数据库构建过程中条形码扩增片段的选择方面, 作者建议最好能够建立鱼类线粒体全基因组数据库以满足不同研究的需求, 即便是出于成本的考虑只能构建单基因片段的数据库, 那么也应该建立能够覆盖该目的基因全部序列的长片段DNA条形码数据库[23]。尽管拥有一个尽可能完善且精确的本土鱼类DNA条形码数据库将能够极大地提高eDNA研究结果的精确性, 但本土数据库同时也存在一个严重的缺陷——物种的覆盖度较低, 这说明在研究中如果仅使用本土数据库进行物种的分类注释, 将无法检测到数据库中尚未涵盖的某些处于入侵早期的鱼类物种信息[144], 从而也会对研究结果的精确性产生一定的影响。因此, 为了能够取得最佳的研究效果, 在进行高通量测序数据的物种分类注释时最好能够同时利用公共数据库与本土数据库。
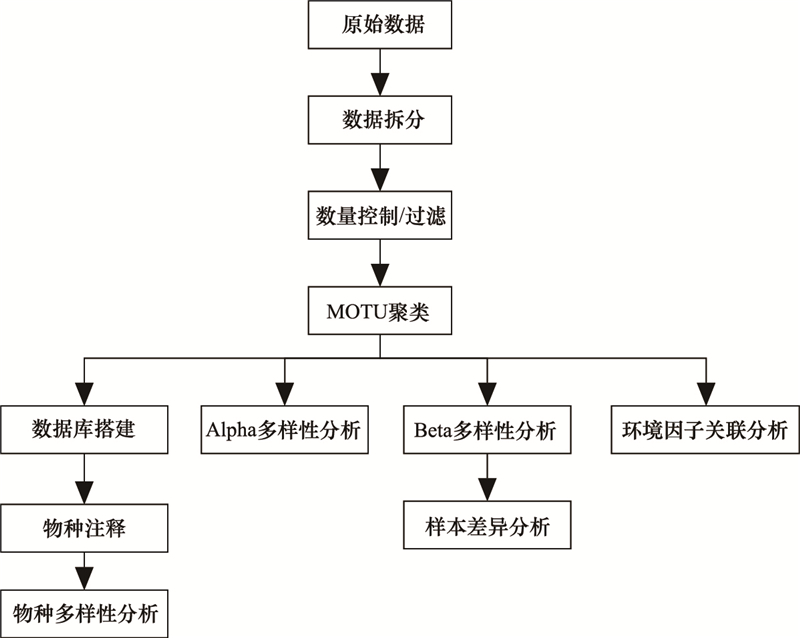
4.3 eDNA被释放后生态学过程的探究eDNA的生态学过程(图 3)主要包括eDNA在环境中的来源、状态、迁移以及最终的结局[146]。eDNA的生态是一系列物理、化学以及生物相互作用的过程, 在此过程中影响eDNA监测结果精确性的因素主要包括各类生物因素与非生物因素。
|
| 图 3 eDNA的生态 Fig. 3 The ecology of eDNA |
鱼类群落中种群数量的大小、所处的生活史阶段、新陈代谢状况、捕食者的存在以及各类洄游等均会影响环境中可检测的eDNA信号强度[23]。Maruyama等设计室内对照组实验研究发现由于代谢活动方面的差异, 幼鱼较成鱼有更高的eDNA释放率[147]。然而, 在自然水域中鱼类种群结构复杂且利用eDNA技术无法判别种群结构信息, 这将导致利用eDNA技术无法准确地推算鱼类种群的生物量。此外, Jo等发现鱼类生物量的大小也会影响eDNA的释放效率[148], 而鱼类的集群以及各种类型的洄游往往会导致鱼类种群生物量的骤增, 这也会对eDNA监测结果的精确性产生一定的干扰。因此, 为了能够使eDNA技术的监测结果更具说服力, 在后期的研究中加强鱼类eDNA释放速率方面的研究十分必要。
除上述生物因素外, 环境中的非生物因素也会严重影响eDNA的监测结果。据当前的研究表明, 影响鱼类eDNA监测效率的非生物因素主要包括水温、pH、紫外光照、盐度、浑浊度以及水文条件等, 例如Strickler等探究了紫外光照、温度以及pH对eDNA在水体中存留时间的影响, 并发现eDNA在黑暗、低温且偏碱性的水环境中能够存留的时间更久[15], 而Diaz-Ferguson和Moyer则发现水文条件的变化会影响eDNA在水环境中的分布情况[149]。尽管目前已有大量关于非生物因素对eDNA监测结果影响的方面的研究, 但基本上是基于室内控制实验且在实验设计中仅考虑了部分环境因子对eDNA生态的影响, 这与自然水体中eDNA生态的真实情况还存在一定的差距。因此, 探究所有对eDNA的生态有影响的非生物因子如何综合影响eDNA的研究结果将是科研人员未来应该重点关注的方向。
针对上述eDNA在环境中的生态学过程会对研究结果精确性产生影响这一问题, 作者建议在后期的研究中应充分发挥多学科交叉融合研究的作用, 综合运用各种生态学以及水文学模型进一步阐明eDNA的生态学过程。此外, 研究人员在具体的研究中应充分考虑eDNA的生态学过程可能会对研究结果所带来的影响, 并在前期的实验方案设计与后期的实验结果解释时将其纳入[136, 146], 从而将eDNA的生态学过程对研究结果精确性的影响降至最低。
5 总结及前景展望自eDNA技术被引入水生生态系统的研究领域以来, 经过十多年的发展其已经成为了一种强有力的鱼类资源调查手段。目前, eDNA技术已经在物种监测、鱼类多样性监测、生物量评估以及鱼类繁殖活动监测等方面得到了广泛的应用且表现出极大的潜力。同时, 相较于传统的鱼类监测方法而言, eDNA技术在监测效率、时间与经济成本以及对生态系统的干扰等方面更具优势。然而, 由于eDNA技术在操作流程上的不规范、物种注释参考数据库的不完善以及eDNA被释放进入环境之后生态学过程的不明确等原因, 科研人员在应用eDNA技术进行鱼类监测时所得研究结果的精确性将会受到一定的影响。因此, 未来要想使eDNA技术能够成为一种替代传统方法的常规化鱼类监测手段还需要克服很多困难。
在本文中, 综述了eDNA技术在鱼类生态学研究领域中的应用现状, 同时对eDNA在具体应用中所面临的问题与挑战做了相关总结, 并提出了相应的解决方案。最终, 通过对eDNA技术在鱼类生态学研究领域中相关文献的梳理可发现, 在未来的研究中要想使eDNA调查得出的结果具有更高的可信度, 科研人员应聚焦于以下几个方面:(1)不断完善鱼类mtDNA 12s rRNA基因数据库的构建。当前利用eDNA技术进行鱼类生态学的研究中, 业内普遍认为以12s rRNA基因作为目的基因能够获得的物种鉴别效果最佳, 而在全球范围内该基因的条形码数据库极度匮乏, 严重影响了高通量测序数据物种注释结果的精确性。因此, 未来亟需构建全球各地的本土鱼类12s rRNA基因参考数据库。(2)阐明eDNA的生态学过程。eDNA的生态学过程一直以来都是影响eDNA监测结果精确性的重要原因之一, 尽管当前已有大量科研人员在该方面做了许多工作, 但尚无明确的定论, 未来应考虑多学科交叉并结合各种水文学以及生态学模型全面综合的考虑各种影响eDNA生态过程的生物与非生物因素, 进一步阐明eDNA在不同生态系统中的生态学过程。(3)开发鱼类eDNA研究的生物信息学分析流程。当前用来分析鱼类eDNA宏条形码测序数据的主流软件最初大多是专门针对微生物研究而开发的, 且不同的软件在算法上存在一定的差异, 从而会在一定程度上影响最终的分析结果。因此, 未来科研人员应开发一套专门针对鱼类eDNA研究的生物信息学分析流程, 尽可能的降低不同研究中测序数据在分析过程中因算法导致的差异。(4)不断开发新技术, 早日实现eDNA监测自动化。近年来关于eDNA采样的各种新型设备层出不穷, 甚至某些在线监测设备已经被投入使用, 但上述有关设备在许多方面仍不完善, 得到的监测结果并不具备很强的说服力。科研人员未来可加强在监测设备开发方面的研究, 争取早日开发出能够放置在各种水生生态系统中自动监测并上传鱼类生态学数据的仪器设备, 以实现鱼类eDNA监测的自动化。
| [1] |
Evans N T, Lamberti G A. Freshwater fisheries assessment using environmental DNA: a primer on the method, its potential, and shortcomings as a conservation tool. Fisheries Research, 2018, 197: 60-66. DOI:10.1016/j.fishres.2017.09.013 |
| [2] |
Dodds W K, Perkin J S, Gerken J E. Human impact on freshwater ecosystem services: a global perspective. Environmental Science & Technology, 2013, 47(16): 9061-9068. |
| [3] |
程馨雨, 陶捐, 武瑞东, 陈礼强, 丁城志. 淡水鱼类功能生态学研究进展. 生态学报, 2019, 39(3): 810-822. |
| [4] |
Irfan S, Alatawi A M M. Aquatic ecosystem and biodiversity: a review. Open Journal of Ecology, 2019, 9(1): 1-13. DOI:10.4236/oje.2019.91001 |
| [5] |
Holmlund C M, Hammer M. Ecosystem services generated by fish populations. Ecological Economics, 1999, 29(2): 253-268. DOI:10.1016/S0921-8009(99)00015-4 |
| [6] |
Reid A J, Carlson A K, Creed I F, Eliason E J, Gell P A, Johnson P T J, Kidd K A, MacCormack T J, Olden J D, Ormerod S J, Smol J P, Taylor W W, Tockner K, Vermaire J C, Dudgeon D, Cooke S J. Emerging threats and persistent conservation challenges for freshwater biodiversity. Biological Reviews, 2019, 94(3): 849-873. DOI:10.1111/brv.12480 |
| [7] |
Almond R E, Grooten M, Peterson T. Living Planet Report 2020-Bending the curve of biodiversity loss. World Wildlife Fund, 2020. |
| [8] |
FAO. The state of world fisheries and aquaculture 2020. Sustainability in action. The Food and Agriculture Organization of the United Nations, 2020.
|
| [9] |
Schenekar T. The Current state of eDNA research in freshwater ecosystems: are we shifting from the developmental phase to standard application in biomonitoring?. Hydrobiologia, 2022, 1-20. |
| [10] |
Nagler M, Podmirseg S M, Ascher-Jenull J, Sint D, Traugott M. Why eDNA fractions need consideration in biomonitoring. Molecular Ecology Resources, 2022, 22(7): 2458-2470. DOI:10.1111/1755-0998.13658 |
| [11] |
Pawlowski J, Apothéloz-Perret-Gentil L, Altermatt F. Environmental DNA: what's behind the term? Clarifying the terminology and recommendations for its future use in biomonitoring. Molecular Ecology, 2020, 29(22): 4258-4264. DOI:10.1111/mec.15643 |
| [12] |
Taberlet P, Coissac E, Hajibabaei M, Rieseberg L H. Environmental DNA. Molecular Ecology, 2012, 21(8): 1789-1793. DOI:10.1111/j.1365-294X.2012.05542.x |
| [13] |
王萌, 金小伟, 林晓龙, 杜丽娜, 崔永德, 吴小平, 孙红英, 谢志才, 王新华, 王备新. 基于环境DNA-宏条形码技术的底栖动物监测及水质评价研究进展. 生态学报, 2021, 41(18): 7440-7453. |
| [14] |
Thomsen P F, Willerslev E. Environmental DNA-An emerging tool in conservation for monitoring past and present biodiversity. Biological Conservation, 2015, 183: 4-18. DOI:10.1016/j.biocon.2014.11.019 |
| [15] |
Strickler K M, Fremier A K, Goldberg C S. Quantifying effects of UV-B, temperature, and pH on eDNA degradation in aquatic microcosms. Biological Conservation, 2015, 183: 85-92. DOI:10.1016/j.biocon.2014.11.038 |
| [16] |
Bohmann K, Evans A, Gilbert M T P, Carvalho G R, Creer S, Knapp M, Yu D W, de Bruyn M. Environmental DNA for wildlife biology and biodiversity monitoring. Trends in Ecology & Evolution, 2014, 29(6): 358-367. |
| [17] |
李晓玲, 刘洋, 王丛丛, 俞晔伟, 李纲. 基于环境DNA技术的夏季东海鱼类物种多样性研究. 海洋学报, 2022, 44(4): 74-84. |
| [18] |
闫卉果, 董智玲, 马婷婷, 张连博, 王晓艳, 叶华, 姚维志, 何文平. 基于环境DNA的岩原鲤检测及生物量评估. 水产学报, 2022, 46(6): 1018-1026. |
| [19] |
Nakao R, Miyata R, Nakamura N, Muramatsu M, Okamura H, Imamura F, Akamatsu Y. Development of environmental DNA chip for monitoring the invasive alien fishes in dam reservoirs. Landscape and Ecological Engineering, 2022, 1-9. DOI:10.1007/s11355-022-00522-w?utm_content=meta |
| [20] |
Pascher K, Švara V, Jungmeier M. Environmental DNA-based methods in biodiversity monitoring of protected areas: application range, limitations, and needs. Diversity, 2022, 14(6): 463. DOI:10.3390/d14060463 |
| [21] |
Minamoto T, Miya M, Sado T, Seino S, Doi H, Kondoh M, Nakamura K, Takahara T, Yamamoto S, Yamanaka H, Araki H, Iwasaki W, Kasai A, Masuda R, Uchii K. An illustrated manual for environmental DNA research: water sampling guidelines and experimental protocols. Environmental DNA, 2021, 3(1): 8-13. DOI:10.1002/edn3.121 |
| [22] |
Miya M. Environmental DNA metabarcoding: a novel method for biodiversity monitoring of marine fish communities. Annual Review of Marine Science, 2022, 14: 161-185. DOI:10.1146/annurev-marine-041421-082251 |
| [23] |
Wang S P, Yan Z G, Hänfling B, Zheng X, Wang P X, Fan J T, Li J L. Methodology of fish eDNA and its applications in ecology and environment. Science of the Total Environment, 2021, 755: 142622. DOI:10.1016/j.scitotenv.2020.142622 |
| [24] |
Ogram A, Sayler G S, Barkay T. The extraction and purification of microbial DNA from sediments. Journal of Microbiological Methods, 1987, 7(2/3): 57-66. |
| [25] |
Rondon M R, August P R, Bettermann A D, Brady S F, Grossman T H, Liles M R, Loiacono K A, Lynch B A, MacNeil I A, Minor C, Tiong C L, Gilman M, Osburne M S, Clardy J, Handelsman J, Goodman R M. Cloning the soil metagenome: a strategy for accessing the genetic and functional diversity of uncultured microorganisms. Applied and Environmental Microbiology, 2000, 66(6): 2541-2547. DOI:10.1128/AEM.66.6.2541-2547.2000 |
| [26] |
Ficetola G F, Miaud C, Pompanon F, Taberlet P. Species detection using environmental DNA from water samples. Biology Letters, 2008, 4(4): 423-425. DOI:10.1098/rsbl.2008.0118 |
| [27] |
Kawato M, Yoshida T, Miya M, Tsuchida S, Nagano Y, Nomura M, Yabuki A, Fujiwara Y, Fujikura K. Optimization of environmental DNA extraction and amplification methods for metabarcoding of deep-sea fish. MethodsX, 2021, 8: 101238. DOI:10.1016/j.mex.2021.101238 |
| [28] |
Rivera S F, Rimet F, Vasselon V, Vautier M, Domaizon I, Bouchez A. Fish eDNA metabarcoding from aquatic biofilm samples: methodological aspects. Molecular Ecology Resources, 2022, 22(4): 1440-1453. DOI:10.1111/1755-0998.13568 |
| [29] |
王晨, 陶孟, 李爱民, 施鹏, 杨江华, 王志浩, 张效伟. 基于环境DNA宏条形码技术的秦淮河生物多样性研究. 生态学报, 2022, 42(2): 611-624. |
| [30] |
Everts T, Halfmaerten D, Neyrinck S, De Regge N, Jacquemyn H, Brys R. Accurate detection and quantification of seasonal abundance of American bullfrog (Lithobates catesbeianus) using ddPCR eDNA assays. Scientific Reports, 2021, 11(1): 11282. DOI:10.1038/s41598-021-90771-w |
| [31] |
Valdivia-Carrillo T, Rocha-Olivares A, Reyes-Bonilla H, Domínguez-Contreras J F, Munguia-Vega A. Integrating eDNA metabarcoding and simultaneous underwater visual surveys to describe complex fish communities in a marine biodiversity hotspot. Molecular Ecology Resources, 2021, 21(5): 1558-1574. DOI:10.1111/1755-0998.13375 |
| [32] |
Andruszkiewicz E A, Yamahara K M, Closek C J, Boehm A B. Quantitative PCR assays to detect whales, rockfish, and common murre environmental DNA in marine water samples of the Northeastern Pacific. PLoS One, 2020, 15(12): e0242689. DOI:10.1371/journal.pone.0242689 |
| [33] |
Dugal L, Thomas L, Jensen M R, Sigsgaard E E, Simpson T, Jarman S, Thomsen P F, Meekan M. Individual haplotyping of whale sharks from seawater environmental DNA. Molecular Ecology Resources, 2022, 22(1): 56-65. DOI:10.1111/1755-0998.13451 |
| [34] |
Gold Z, Wall A R, Curd E E, Kelly R P, Pentcheff N D, Ripma L, Barber P H, Wetzer R. eDNA metabarcoding bioassessment of endangered fairy shrimp (Branchinecta spp.). Conservation Genetics Resources, 2020, 12(4): 685-690. DOI:10.1007/s12686-020-01161-9 |
| [35] |
Li M, Shan X J, Wang W J, Ding X S, Dai F Q, Lv D, Wu H H. Qualitative and quantitative detection using eDNA technology: a case study of Fenneropenaeus chinensis in the Bohai Sea. Aquaculture and Fisheries, 2020, 5(3): 148-155. DOI:10.1016/j.aaf.2020.03.012 |
| [36] |
Maggioni D, Assandri G, Ramazzotti F, Magnani D, Pellegrino I, Valsecchi E, Galimberti A. Differential genetic variability at two mtDNA COI regions does not imply mismatches in Odonata molecular identification performances. The European Zoological Journal, 2021, 88(1): 425-435. DOI:10.1080/24750263.2021.1896795 |
| [37] |
Schmidt K J, Soluk D A, Maestas S E M, Britten H B. Persistence and accumulation of environmental DNA from an endangered dragonfly. Scientific Reports, 2021, 11: 18987. DOI:10.1038/s41598-021-98099-1 |
| [38] |
Aldeguer-Riquelme B, Ramos-Barbero M D, Santos F, Antón J. Environmental dissolved DNA harbours meaningful biological information on microbial community structure. Environmental Microbiology, 2021, 23(5): 2669-2682. DOI:10.1111/1462-2920.15510 |
| [39] |
Balasubramanian V K, Joseph Maran M I, Ramteke D, Vijaykumar D S, Kottarathail Rajendran A, Ramachandran P, Ramachandran R. Environmental DNA reveals aquatic biodiversity of an urban backwater area, southeast coast of India. Marine Pollution Bulletin, 2021, 171: 112786. DOI:10.1016/j.marpolbul.2021.112786 |
| [40] |
Hanžek N, Gligora Udovič M, Kajan K, Borics G, Várbíró G, Stoeck T, Žutinić P, Orlić S, Stanković I. Assessing ecological status in karstic lakes through the integration of phytoplankton functional groups, morphological approach and environmental DNA metabarcoding. Ecological Indicators, 2021, 131: 108166. DOI:10.1016/j.ecolind.2021.108166 |
| [41] |
Suter L, Polanowski A M, Clarke L J, Kitchener J A, Deagle B E. Capturing open ocean biodiversity: comparing environmental DNA metabarcoding to the continuous plankton recorder. Molecular Ecology, 2021, 30(13): 3140-3157. DOI:10.1111/mec.15587 |
| [42] |
Xie R L, Zhao G F, Yang J H, Wang Z H, Xu Y P, Zhang X W, Wang Z J. eDNA metabarcoding revealed differential structures of aquatic communities in a dynamic freshwater ecosystem shaped by habitat heterogeneity. Environmental Research, 2021, 201: 111602. DOI:10.1016/j.envres.2021.111602 |
| [43] |
Carvalho C S, De Oliveira M E, Rodriguez-Castro K G, Saranholi B H, Galetti P M Jr. Efficiency of eDNA and iDNA in assessing vertebrate diversity and its abundance. Molecular Ecology Resources, 2022, 22(4): 1262-1273. DOI:10.1111/1755-0998.13543 |
| [44] |
Gargan L M, Brooks P R, Vye S R, Ironside J E, Jenkins S R, Crowe T P, Carlsson J. The use of environmental DNA metabarcoding and quantitative PCR for molecular detection of marine invasive non-native species associated with artificial structures. Biological Invasions, 2022, 24(3): 635-648. DOI:10.1007/s10530-021-02672-8 |
| [45] |
Rees H C, Maddison B C, Middleditch D J, Patmore J R M, Gough K C. REVIEW: The detection of aquatic animal species using environmental DNA: a review of eDNA as a survey tool in ecology. Journal of Applied Ecology, 2014, 51(5): 1450-1459. DOI:10.1111/1365-2664.12306 |
| [46] |
Deiner K, Walser J C, Mächler E, Altermatt F. Choice of capture and extraction methods affect detection of freshwater biodiversity from environmental DNA. Biological Conservation, 2015, 183: 53-63. DOI:10.1016/j.biocon.2014.11.018 |
| [47] |
Thomsen P F, Kielgast J, Iversen L L, Møller P R, Rasmussen M, Willerslev E. Detection of a diverse marine fish fauna using environmental DNA from seawater samples. PLoS One, 2012, 7(8): e41732. DOI:10.1371/journal.pone.0041732 |
| [48] |
李苗, 单秀娟, 王伟继, 吕丁, 戴芳群, 丁小松, 吴欢欢. 中国对虾生物量评估的环境DNA检测技术的建立及优化. 渔业科学进展, 2019, 40(1): 12-19. DOI:10.19663/j.issn2095-9869.20180620001 |
| [49] |
Pawlowski J, Bruce K, Panksep K, Aguirre F I, Amalfitano S, Apothéloz-Perret-Gentil L, Baussant T, Bouchez A, Carugati L, Cermakova K, Cordier T, Corinaldesi C, Costa F O, Danovaro R, Dell'anno A, Duarte S, Eisendle U, Ferrari B J D, Frontalini F, Frühe L, Haegerbaeumer A, Kisand V, Krolicka A, Lanzén A, Leese F, Lejzerowicz F, Lyautey E, Maček I, Sagova-Marečková M, Pearman J K, Pochon X, Stoeck T, Vivien R, Weigand A, Fazi S. Environmental DNA metabarcoding for benthic monitoring: a review of sediment sampling and DNA extraction methods. Science of the Total Environment, 2022, 818: 151783. DOI:10.1016/j.scitotenv.2021.151783 |
| [50] |
Thomson-Laing G, Howarth J D, Vandergoes M J, Wood S A. Optimised protocol for the extraction of fish DNA from freshwater sediments. Freshwater Biology, 2022, 67(9): 1584-1603. DOI:10.1111/fwb.13962 |
| [51] |
Xie Y W, Wang J Z, Yang J H, Giesy J P, Yu H X, Zhang X W. Environmental DNA metabarcoding reveals primary chemical contaminants in freshwater sediments from different land-use types. Chemosphere, 2017, 172: 201-209. DOI:10.1016/j.chemosphere.2016.12.117 |
| [52] |
Stoeck T, Frühe L, Forster D, Cordier T, Martins C I M, Pawlowski J. Environmental DNA metabarcoding of benthic bacterial communities indicates the benthic footprint of salmon aquaculture. Marine Pollution Bulletin, 2018, 127: 139-149. DOI:10.1016/j.marpolbul.2017.11.065 |
| [53] |
Brannock P M, Learman D R, Mahon A R, Santos S R, Halanych K M. Meiobenthic community composition and biodiversity along a 5500 km transect of Western Antarctica: a metabarcoding analysis. Marine Ecology Progress Series, 2018, 603: 47-60. DOI:10.3354/meps12717 |
| [54] |
Lejzerowicz F, Gooday A J, Angeles I B, Cordier T, Morard R, Apothéloz-Perret-Gentil L, Lins L, Menot L, Brandt A, Levin L, Arbizu P M, Smith C, Pawłowski J. Eukaryotic biodiversity and spatial patterns in the clarion-clipperton zone and other abyssal regions: insights from sediment DNA and RNA metabarcoding. Frontiers in Marine Science, 2021, 8: 671033. DOI:10.3389/fmars.2021.671033 |
| [55] |
Bruce K, Blackman R C, Bourlat S J, Hellström M, Bakker J, Bista I, Bohmann K, Bouchez A, Brys R, Clark K, Elbrecht V, Fazi S, Fonseca V G, Hänfling B, Leese F, Mächler E, Mahon A R, Meissner K, Panksep K, Pawlowski J, Yáñez P L S, Seymour M, Thalinger B, Valentini A, Woodcock P, Traugott M, Vasselon V, Deiner K. A practical guide to DNA-based methods for biodiversity assessment, 2021.
|
| [56] |
Mariani S, Baillie C, Colosimo G, Riesgo A. Sponges as natural environmental DNA samplers. Current Biology, 2019, 29(11): R401-R402. DOI:10.1016/j.cub.2019.04.031 |
| [57] |
Turon M, Angulo-Preckler C, Antich A, Praebel K, Wangensteen O S. More than expected from old sponge samples: a natural sampler DNA metabarcoding assessment of marine fish diversity in nha trang bay (Vietnam). Frontiers in Marine Science, 2020, 7: 605148. DOI:10.3389/fmars.2020.605148 |
| [58] |
Cai W, Harper L R, Neave E F, Shum P, Craggs J, Arias M B, Riesgo A, Mariani S. Environmental DNA persistence and fish detection in captive sponges. Molecular Ecology Resources, 2022, 22(8): 2956-2966. DOI:10.1111/1755-0998.13677 |
| [59] |
Verdier H, Konecny-Dupre L, Marquette C, Reveron H, Tadier S, Grémillard L, Barthès A, Datry T, Bouchez A, Lefébure T. Passive sampling of environmental DNA in aquatic environments using 3D-printed hydroxyapatite samplers. Molecular Ecology Resources, 2022, 22(6): 2158-2170. DOI:10.1111/1755-0998.13604 |
| [60] |
Xing Y C, Gao W R, Shen Z X, Zhang Y Y, Bai J, Cai X W, Ouyang J L, Zhao Y H. A review of environmental DNA field and laboratory protocols applied in fish ecology and environmental health. Frontiers in Environmental Science, 2022, 10: 725360. DOI:10.3389/fenvs.2022.725360 |
| [61] |
Dougherty M M, Larson E R, Renshaw M A, Gantz C A, Egan S P, Erickson D M, Lodge D M. Environmental DNA (eDNA) detects the invasive rusty crayfish Orconectes rusticus at low abundances. The Journal of Applied Ecology, 2016, 53(3): 722-732. DOI:10.1111/1365-2664.12621 |
| [62] |
Agersnap S, Larsen W B, Knudsen S W, Strand D, Thomsen P F, Hesselsøe M, Mortensen P B, Vrålstad T, Møller P R. Monitoring of noble, signal and narrow-clawed crayfish using environmental DNA from freshwater samples. PLoS One, 2017, 12(6): e0179261. DOI:10.1371/journal.pone.0179261 |
| [63] |
Andújar C, Arribas P, Yu D W, Vogler A P, Emerson B C. Why the COI barcode should be the community DNA metabarcode for the Metazoa. Molecular Ecology, 2018, 27(20): 3968-3975. DOI:10.1111/mec.14844 |
| [64] |
Kelly R P, Port J A, Yamahara K M, Crowder L B. Using environmental DNA to census marine fishes in a large mesocosm. PLoS One, 2014, 9(1): e86175. DOI:10.1371/journal.pone.0086175 |
| [65] |
Miya M, Sato Y, Fukunaga T, Sado T, Poulsen J Y, Sato K, Minamoto T, Yamamoto S, Yamanaka H, Araki H, Kondoh M, Iwasaki W. MiFish, a set of universal PCR primers for metabarcoding environmental DNA from fishes: detection of more than 230 subtropical marine species. Royal Society Open Science, 2015, 2(7): 150088. DOI:10.1098/rsos.150088 |
| [66] |
Evans N T, Olds B P, Renshaw M A, Turner C R, Li Y Y, Jerde C L, Mahon A R, Pfrender M E, Lamberti G A, Lodge D M. Quantification of mesocosm fish and amphibian species diversity via environmental DNA metabarcoding. Molecular Ecology Resources, 2016, 16(1): 29-41. DOI:10.1111/1755-0998.12433 |
| [67] |
Valentini A, Taberlet P, Miaud C, Civade R, Herder J, Thomsen P F, Bellemain E, Besnard A, Coissac E, Boyer F, Gaboriaud C, Jean P, Poulet N, Roset N, Copp G H, Geniez P, Pont D, Argillier C, Baudoin J M, Peroux T, Crivelli A J, Olivier A, Acqueberge M, Le Brun M, Møller P R, Willerslev E, Dejean T. Next-generation monitoring of aquatic biodiversity using environmental DNA metabarcoding. Molecular Ecology, 2016, 25(4): 929-942. DOI:10.1111/mec.13428 |
| [68] |
Taberlet P, Bonin A, Zinger L, Coissac E. Environmental DNA: for biodiversity research and monitoring. First edition.
|
| [69] |
Bylemans J. Monitoring Freshwater Fish Communities with Environmental DNA (eDNA) Metabarcoding. University of Canberra, 2018.
|
| [70] |
Kitano T, Umetsu K, Tian W, Osawa M. Two universal primer sets for species identification among vertebrates. International Journal of Legal Medicine, 2007, 121(5): 423-427. DOI:10.1007/s00414-006-0113-y |
| [71] |
Shaw J L A, Clarke L J, Wedderburn S D, Barnes T C, Weyrich L S, Cooper A. Comparison of environmental DNA metabarcoding and conventional fish survey methods in a river system. Biological Conservation, 2016, 197: 131-138. DOI:10.1016/j.biocon.2016.03.010 |
| [72] |
Vences M, Lyra M L, Perl R G B, Bletz M C, Stanković D, Lopes C M, Jarek M, Bhuju S, Geffers R, Haddad C F B, Steinfartz S. Freshwater vertebrate metabarcoding on Illumina platforms using double-indexed primers of the mitochondrial 16S rRNA gene. Conservation Genetics Resources, 2016, 8(3): 323-327. DOI:10.1007/s12686-016-0550-y |
| [73] |
Stat M, John J, DiBattista J D, Newman S J, Bunce M, Harvey E S. Combined use of eDNA metabarcoding and video surveillance for the assessment of fish biodiversity. Conservation Biology, 2019, 33(1): 196-205. DOI:10.1111/cobi.13183 |
| [74] |
Balasingham K D, Walter R P, Mandrak N E, Heath D D. Environmental DNA detection of rare and invasive fish species in two Great Lakes tributaries. Molecular Ecology, 2018, 27(1): 112-127. DOI:10.1111/mec.14395 |
| [75] |
Kocher T D, Thomas W K, Meyer A, Edwards S V, Pääbo S, Villablanca F X, Wilson A C. Dynamics of mitochondrial DNA evolution in animals: amplification and sequencing with conserved primers. Proceedings of the National Academy of Sciences of the United States of America, 1989, 86(16): 6196-6200. |
| [76] |
Burgener M, Hübner P. Mitochondrial DNA enrichment for species identification and evolutionary analysis. Zeitschrift für Lebensmitteluntersuchung Und-Forschung A, 1998, 207(4): 261-263. DOI:10.1007/s002170050329 |
| [77] |
Gillet B, Cottet M, Destanque T, Kue K, Descloux S, Chanudet V, Hughes S. Direct fishing and eDNA metabarcoding for biomonitoring during a 3-year survey significantly improves number of fish detected around a South East Asian Reservoir. PLoS One, 2018, 13(12): e0208592. DOI:10.1371/journal.pone.0208592 |
| [78] |
Thomsen P F, Kielgast J, Iversen L L, Wiuf C, Rasmussen M, Gilbert M T P, Orlando L, Willerslev E. Monitoring endangered freshwater biodiversity using environmental DNA. Molecular Ecology, 2012, 21(11): 2565-2573. DOI:10.1111/j.1365-294X.2011.05418.x |
| [79] |
Deiner K, Renshaw M A, Li Y Y, Olds B P, Lodge D M, Pfrender M E. Long-range PCR allows sequencing of mitochondrial genomes from environmental DNA. Methods in Ecology and Evolution, 2017, 8(12): 1888-1898. DOI:10.1111/2041-210X.12836 |
| [80] |
Clarke L J, Suter L, Deagle B E, Polanowski A M, Terauds A, Johnstone G J, Stark J S. Environmental DNA metabarcoding for monitoring metazoan biodiversity in Antarctic nearshore ecosystems. PeerJ, 2021, 9: e12458. DOI:10.7717/peerj.12458 |
| [81] |
Christianson L M, Johnson S B, Schultz D T, Haddock S H D. Hidden diversity of Ctenophora revealed by new mitochondrial COI primers and sequences. Molecular Ecology Resources, 2022, 22(1): 283-294. DOI:10.1111/1755-0998.13459 |
| [82] |
Osathanunkul M, Minamoto T. A molecular survey based on eDNA to assess the presence of a clown featherback (Chitala ornata) in a confined environment. PeerJ, 2020, 8: e10338. DOI:10.7717/peerj.10338 |
| [83] |
Soh E Y C, Smith F, Gimenez M R, Yang L, Vejborg R M, Fletcher M, Halliday N, Bleves S, Heeb S, Cámara M, Givskov M, Hardie K R, Tolker-Nielsen T, Ize B, Williams P. Disruption of the Pseudomonas aeruginosa Tat system perturbs PQS-dependent quorum sensing and biofilm maturation through lack of the Rieske cytochrome bc1 sub-unit. PLoS Pathogens, 2021, 17(8): e1009425. DOI:10.1371/journal.ppat.1009425 |
| [84] |
Polanco F A, Mutis Martinezguerra M, Marques V, Villa-Navarro F, Borrero Perez G H, Cheutin M C, Dejean T, Hocdé R, Juhel J B, Maire E. Detecting aquatic and terrestrial biodiversity in a tropical estuary using environmental DNA. Biotropica, 2021, 53(6): 1606-1619. DOI:10.1111/btp.13009 |
| [85] |
Charvoz L, Apothéloz-Perret-Gentil L, Reo E, Thiébaud J, Pawlowski J. Monitoring newt communities in urban area using eDNA metabarcoding. PeerJ, 2021, 9: e12357. DOI:10.7717/peerj.12357 |
| [86] |
Mullin K E, Barata I M, Dawson J, Orozco-terWengel P. First extraction of eDNA from tree hole water to detect tree frogs: a simple field method piloted in Madagascar. Conservation Genetics Resources, 2022, 14(1): 99-107. DOI:10.1007/s12686-021-01245-0 |
| [87] |
Zhang S, Zhao J D, Yao M. A comprehensive and comparative evaluation of primers for metabarcoding eDNA from fish. Methods in Ecology and Evolution, 2020, 11(12): 1609-1625. DOI:10.1111/2041-210X.13485 |
| [88] |
Shu L, Ludwig A, Peng Z G. Environmental DNA metabarcoding primers for freshwater fish detection and quantification: in silico and in tanks. Ecology and Evolution, 2021, 11(12): 8281-8294. DOI:10.1002/ece3.7658 |
| [89] |
Deiner K, Bik H M, Mächler E, Seymour M, Lacoursière-Roussel A, Altermatt F, Creer S, Bista I, Lodge D M, De Vere N, Pfrender M E, Bernatchez L. Environmental DNA metabarcoding: Transforming how we survey animal and plant communities. Molecular Ecology, 2017, 26(21): 5872-5895. DOI:10.1111/mec.14350 |
| [90] |
Jo T, Murakami H, Masuda R, Sakata M K, Yamamoto S, Minamoto T. Rapid degradation of longer DNA fragments enables the improved estimation of distribution and biomass using environmental DNA. Molecular Ecology Resources, 2017, 17(6): e25-e33. |
| [91] |
Riaz T, Shehzad W, Viari A, Pompanon F, Taberlet P, Coissac E. ecoPrimers: inference of new DNA barcode markers from whole genome sequence analysis. Nucleic Acids Research, 2011, 39(21): e145-e145. DOI:10.1093/nar/gkr732 |
| [92] |
Bylemans J, Gleeson D M, Hardy C M, Furlan E. Toward an ecoregion scale evaluation of eDNA metabarcoding primers: a case study for the freshwater fish biodiversity of the Murray-Darling Basin (Australia). Ecology and Evolution, 2018, 8(17): 8697-8712. DOI:10.1002/ece3.4387 |
| [93] |
Collins R A, Bakker J, Wangensteen O S, Soto A Z, Corrigan L, Sims D W, Genner M J, Mariani S. Non-specific amplification compromises environmental DNA metabarcoding with COI. Methods in Ecology and Evolution, 2019, 10(11): 1985-2001. DOI:10.1111/2041-210X.13276 |
| [94] |
Hänfling B, Lawson Handley L, Read D S, Hahn C, Li J L, Nichols P, Blackman R C, Oliver A, Winfield I J. Environmental DNA metabarcoding of lake fish communities reflects long-term data from established survey methods. Molecular Ecology, 2016, 25(13): 3101-3119. DOI:10.1111/mec.13660 |
| [95] |
Banerji A, Bagley M, Elk M, Pilgrim E, Martinson J, Domingo J S. Spatial and temporal dynamics of a freshwater eukaryotic plankton community revealed via 18S rRNA gene metabarcoding. Hydrobiologia, 2018, 818(1): 71-86. DOI:10.1007/s10750-018-3593-0 |
| [96] |
Valdez-Moreno M, Ivanova N V, Elías-Gutiérrez M, Pedersen S L, Bessonov K, Hebert P D N. Using eDNA to biomonitor the fish community in a tropical oligotrophic lake. PLoS One, 2019, 14(4): e0215505. DOI:10.1371/journal.pone.0215505 |
| [97] |
Schloss P D, Westcott S L, Ryabin T, Hall J R, Hartmann M, Hollister E B, Lesniewski R A, Oakley B B, Parks D H, Robinson C J, Sahl J W, Stres B, Thallinger G G, Van Horn D J, Weber C F. Introducing mothur: open-source, platform-independent, community-supported software for describing and comparing microbial communities. Applied and Environmental Microbiology, 2009, 75(23): 7537-7541. DOI:10.1128/AEM.01541-09 |
| [98] |
Edgar R C. Search and clustering orders of magnitude faster than BLAST. Bioinformatics, 2010, 26(19): 2460-2461. DOI:10.1093/bioinformatics/btq461 |
| [99] |
Rognes T, Flouri T, Nichols B, Quince C, Mahé F. VSEARCH: a versatile open source tool for metagenomics. PeerJ, 2016, 4: e2584. DOI:10.7717/peerj.2584 |
| [100] |
Kalyuzhnaya M G, Lapidus A, Ivanova N, Copeland A C, McHardy A C, Szeto E, Salamov A, Grigoriev I V, Suciu D, Levine S R, Markowitz V M, Rigoutsos I, Tringe S G, Bruce D C, Richardson P M, Lidstrom M E, Chistoserdova L. High-resolution metagenomics targets specific functional types in complex microbial communities. Nature Biotechnology, 2008, 26(9): 1029-1034. DOI:10.1038/nbt.1488 |
| [101] |
Kuczynski J, Stombaugh J, Walters W A, González A, Caporaso J G, Knight R. Using QIIME to analyze 16S rRNA gene sequences from microbial communities. Current Protocols in Microbiology, 2012, 27(1): 1-30. |
| [102] |
Fung C, Rusling M, Lampeter T, Love C, Karim A, Bongiorno C, Yuan L L. Automation of QIIME2 metagenomic analysis platform. Current Protocols, 2021, 1(9): e254. DOI:10.1002/cpz1.254 |
| [103] |
Sato Y, Miya M, Fukunaga T, Sado T, Iwasaki W. MitoFish and MiFish pipeline: a mitochondrial genome database of fish with an analysis pipeline for environmental DNA metabarcoding. Molecular Biology and Evolution, 2018, 35(6): 1553-1555. DOI:10.1093/molbev/msy074 |
| [104] |
Yao M, Zhang S, Lu Q, Chen X Y, Zhang S-Y, Kong Y Q, Zhao J D. Fishing for fish environmental DNA: ecological applications, methodological considerations, surveying designs, and ways forward. Molecular Ecology, 2022, 31(20): 5132-5164. DOI:10.1111/mec.16659 |
| [105] |
Plough L V, Ogburn M B, Fitzgerald C L, Geranio R, Marafino G A, Richie K D. Environmental DNA analysis of river herring in Chesapeake Bay: a powerful tool for monitoring threatened keystone species. PLoS One, 2018, 13(11): e0205578. DOI:10.1371/journal.pone.0205578 |
| [106] |
Dejean T, Valentini A, Duparc A, Pellier-Cuit S, Pompanon F, Taberlet P, Miaud C. Persistence of environmental DNA in freshwater ecosystems. PLoS One, 2011, 6(8): e23398. DOI:10.1371/journal.pone.0023398 |
| [107] |
单秀娟, 李苗, 王伟继. 环境DNA(eDNA)技术在水生生态系统中的应用研究进展. 渔业科学进展, 2018, 39(3): 23-29. DOI:10.19663/j.issn2095-9869.20171025001 |
| [108] |
Sepulveda A J, Nelson N M, Jerde C L, Luikart G. Are environmental DNA methods ready for aquatic invasive species management?. Trends in Ecology & Evolution, 2020, 35(8): 668-678. |
| [109] |
Bonfil R, Palacios-Barreto P, Vargas O U M, Ricaño-Soriano M, Díaz-Jaimes P. Correction to: detection of critically endangered marine species with dwindling populations in the wild using eDNA gives hope for sawfishes. Marine Biology, 2021, 168(5): 60. DOI:10.1007/s00227-021-03862-7 |
| [110] |
Burgoa Cardás J, Deconinck D, Márquez I, Peón Torre P, Garcia-Vazquez E, Machado-Schiaffino G. New eDNA based tool applied to the specific detection and monitoring of the endangered European eel. Biological Conservation, 2020, 250: 108750. DOI:10.1016/j.biocon.2020.108750 |
| [111] |
Budd A M, Cooper M K, Le Port A L, Schils T, Mills M S, Deinhart M E, Huerlimann R, Strugnell J M. First detection of critically endangered scalloped hammerhead sharks (Sphyrna lewini) in Guam, Micronesia, in five decades using environmental DNA. Ecological Indicators, 2021, 127: 107649. DOI:10.1016/j.ecolind.2021.107649 |
| [112] |
Takahara T, Minamoto T, Yamanaka H, Doi H, Kawabata Z I. Estimation of fish biomass using environmental DNA. PLoS One, 2012, 7(4): e35868. DOI:10.1371/journal.pone.0035868 |
| [113] |
Jamieson I G, Allendorf F W. How does the 50/500 rule apply to MVPs?. Trends in Ecology & Evolution, 2012, 27(10): 578-584. |
| [114] |
Begon M, Townsend C R. Ecology: from individuals to ecosystems. John Wiley & Sons, 2020.
|
| [115] |
Luikart G, Ryman N, Tallmon D A, Schwartz M K, Allendorf F W. Estimation of census and effective population sizes: the increasing usefulness of DNA-based approaches. Conservation Genetics, 2010, 11(2): 355-373. DOI:10.1007/s10592-010-0050-7 |
| [116] |
Immell D, Anthony R G. Estimation of black bear abundance using a discrete DNA sampling device. The Journal of Wildlife Management, 2008, 72(1): 324-330. DOI:10.2193/2006-297 |
| [117] |
Krebs C. Ecology: the experimental analysis of distribution and abundance, Harper & Row, 1973.
|
| [118] |
Schwarz C J, Seber G A F. Estimating animal abundance: review Ⅲ. Statistical Science, 1999, 14(4): 427-456. |
| [119] |
Yates M C, Glaser D M, Post J R, Cristescu M E, Fraser D J, Derry A M. The relationship between eDNA particle concentration and organism abundance in nature is strengthened by allometric scaling. Molecular Ecology, 2021, 30(13): 3068-3082. DOI:10.1111/mec.15543 |
| [120] |
Yates M C, Bernos T A, Fraser D J. A critical assessment of estimating census population size from genetic population size (or vice versa) in three fishes. Evolutionary Applications, 2017, 10(9): 935-945. DOI:10.1111/eva.12496 |
| [121] |
Mizumoto H, Urabe H, Kanbe T, Fukushima M, Araki H. Establishing an environmental DNA method to detect and estimate the biomass of Sakhalin Taimen, a critically endangered Asian salmonid. Limnology, 2018, 19(2): 219-227. DOI:10.1007/s10201-017-0535-x |
| [122] |
Fukaya K, Murakami H, Yoon S, Minami K, Osada Y, Yamamoto S, Masuda R, Kasai A, Miyashita K, Minamoto T, Kondoh M. Estimating fish population abundance by integrating quantitative data on environmental DNA and hydrodynamic modelling. Molecular Ecology, 2021, 30(13): 3057-3067. DOI:10.1111/mec.15530 |
| [123] |
Stoeckle M Y, Soboleva L, Charlop-Powers Z. Aquatic environmental DNA detects seasonal fish abundance and habitat preference in an urban estuary. PLoS One, 2017, 12(4): e0175186. DOI:10.1371/journal.pone.0175186 |
| [124] |
Pont D, Rocle M, Valentini A, Civade R, Jean P, Maire A, Roset N, Schabuss M, Zornig H, Dejean T. Environmental DNA reveals quantitative patterns of fish biodiversity in large rivers despite its downstream transportation. Scientific Reports, 2018, 8: 10361. DOI:10.1038/s41598-018-28424-8 |
| [125] |
Thomsen P F, Møller P R, Sigsgaard E E, Knudsen S W, Jørgensen O A, Willerslev E. Environmental DNA from seawater samples correlate with trawl catches of subarctic, deepwater fishes. PLoS One, 2016, 11(11): e0165252. DOI:10.1371/journal.pone.0165252 |
| [126] |
Marques V, Castagné P, Fernández A P, Borrero-Pérez G H, Hocdé R, Guérin P É, Juhel J B, Velez L, Loiseau N, Letessier T B, Bessudo S, Valentini A, Dejean T, Mouillot D, Pellissier L, Villéger S. Use of environmental DNA in assessment of fish functional and phylogenetic diversity. Conservation Biology: the Journal of the Society for Conservation Biology, 2021, 35(6): 1944-1956. DOI:10.1111/cobi.13802 |
| [127] |
Aglieri G, Baillie C, Mariani S, Cattano C, Calò A, Turco G, Spatafora D, Di Franco A, Di Lorenzo M, Guidetti P, Milazzo M. Environmental DNA effectively captures functional diversity of coastal fish communities. Molecular Ecology, 2021, 30(13): 3127-3139. DOI:10.1111/mec.15661 |
| [128] |
Zhong W J, Zhang J Y, Wang Z H, Lin J Q, Huang X Y, Liu W H, Li H J, Pellissier L, Zhang X W. Holistic impact evaluation of human activities on the coastal fish biodiversity in the Chinese coastal environment. Environmental Science & Technology, 2022, 56(10): 6574-6583. |
| [129] |
万本太, 徐海根, 丁晖, 刘志磊, 王捷. 生物多样性综合评价方法研究. 生物多样性, 2007, 15(1): 97-106. DOI:10.3321/j.issn:1005-0094.2007.01.011 |
| [130] |
曾志新, 罗军, 颜立红, 邹建文, 肖绿田. 生物多样性的评价指标和评价标准. 湖南林业科技, 1999, 26(2): 26-29. |
| [131] |
Blancher P, Lefrançois E, Rimet F, Vasselon V, Argillier C, Arle J, Beja P, Boets P, Boughaba J, Chauvin C, Deacon M, Duncan W, Ejdung G, Erba S, Ferrari B, Fischer H, Hänfling B, Haldin M, Hering D, Hette-Tronquart N, Hiley A, Järvinen M, Jeannot B, Kahlert M, Kelly M, Kleinteich J, Koyuncuoǧlu S, Krenek S, Langhein-Winther S, Leese F, Mann D, Marcel R, Marcheggiani S, Meissner K, Mergen P, Monnier O, Narendja F, Neu D, Onofre Pinto V, Pawlowska A, Pawlowski J, Petersen M, Poikane S, Pont D, Renevier M S, Sandoy S, Svensson J, Trobajo R, Tünde Zagyva A, Tziortzis I, van der Hoorn B, Vasquez M I, Walsh K, Weigand A, Bouchez A. A strategy for successful integration of DNA-based methods in aquatic monitoring. Metabarcoding and Metagenomics, 2022, 6: 215-226. |
| [132] |
Bracken F S A, Rooney S M, Kelly-Quinn M, King J J, Carlsson J. Identifying spawning sites and other critical habitat in lotic systems using eDNA "snapshots": a case study using the sea lamprey Petromyzon marinus L. Ecology and Evolution, 2018, 9(1): 553-567. |
| [133] |
Tillotson M D, Kelly R P, Duda J J, Hoy M, Kralj J, Quinn T P. Concentrations of environmental DNA (eDNA) reflect spawning salmon abundance at fine spatial and temporal scales. Biological Conservation, 2018, 220: 1-11. DOI:10.1016/j.biocon.2018.01.030 |
| [134] |
Takeuchi A, Iijima T, Kakuzen W, Watanabe S, Yamada Y, Okamura A, Horie N, Mikawa N, Miller M J, Kojima T, Tsukamoto K. Release of eDNA by different life history stages and during spawning activities of laboratory-reared Japanese eels for interpretation of oceanic survey data. Scientific Reports, 2019, 9(1): 6074. DOI:10.1038/s41598-019-42641-9 |
| [135] |
Hayer C A, Bayless M F, George A, Thompson N, Richter C A, Chapman D C. Use of environmental DNA to detect grass carp spawning events. Fishes, 2020, 5(3): 27. DOI:10.3390/fishes5030027 |
| [136] |
Cristescu M E, Hebert P D N. Uses and misuses of environmental DNA in biodiversity science and conservation. Annual Review of Ecology, Evolution, and Systematics, 2018, 49(1): 209-230. DOI:10.1146/annurev-ecolsys-110617-062306 |
| [137] |
赵彦伟, 陈家琪, 董丽, 麻晓梅, 白洁, 田凯. 环境DNA技术在水生态领域应用研究进展. 农业环境科学学报, 2021, 40(10): 2057-2065. DOI:10.11654/jaes.2021-0666 |
| [138] |
邢迎春, 高婉茹, 白洁, 赵亚辉. 环境DNA在湖泊生物多样性研究中的应用. 水生生物学报, 2022, 46(1): 137-148. |
| [139] |
Geerts A N, Boets P, van den Heede S, Goethals P, van der heyden C. A search for standardized protocols to detect alien invasive crayfish based on environmental DNA (eDNA): a lab and field evaluation. Ecological Indicators, 2018, 84: 564-572. DOI:10.1016/j.ecolind.2017.08.068 |
| [140] |
Leese F, Bouchez A, Abarenkov K, Altermatt F, Borja Á, Bruce K, Ekrem T, Čiampor Jr F, Čiamporová-Zatovičová Z, Costa F O, Duarte S, Elbrecht V, Fontaneto D, Franc A, Geiger M F, Hering D, Kahlert M, Kalamujić Stroil B, Kelly M, Keskin E, Liska I, Mergen P, Meissner K, Pawlowski J, Penev L, Reyjol Y, Rotter A, Steinke D, van der Wal B, Vitecek S, Zimmermann J, Weigand A M. Why we need sustainable networks bridging countries, disciplines, cultures and generations for aquatic biomonitoring 2.0: a perspective derived from the DNAqua-net COST action. Next Generation Biomonitoring: Part 1. Amsterdam: Elsevier, 2018: 63-99.
|
| [141] |
Miya M, Sado T. Environmental DNA sampling and experimental manual version 2.1. Ed. by eDNA Methods Standardization Committee, The eDNA Society, 2019.
|
| [142] |
De Brauwer M, Chariton A, Clarke L, Cooper M, Dibattista J, Furlan E, Giblot-Ducray D, Gleeson D, Harford A, Herbert S, MacDonald A J, Miller A, Montgomery K, Mooney T, Noble L M, Rourke M, Sherman C D H, Stat M, Suter L, West K M, White N, Villacorta-Rath C, Zaiko A, Trujillo-Gonzalez A. Environmental DNA protocol development guide for biomonitoring. National eDNA Reference Centre, Canberra, 2022.
|
| [143] |
Muha T P, Robinson C V, Garcia de Leaniz C, Consuegra S. An optimised eDNA protocol for detecting fish in lentic and lotic freshwaters using a small water volume. PLoS One, 2019, 14(7): e0219218. DOI:10.1371/journal.pone.0219218 |
| [144] |
Xiong F, Shu L, Zeng H H, Gan X N, He S P, Peng Z G. Methodology for fish biodiversity monitoring with environmental DNA metabarcoding: the primers, databases and bioinformatic pipelines. Water Biology and Security, 2022, 1(1): 100007. DOI:10.1016/j.watbs.2022.100007 |
| [145] |
Gold Z, Curd E E, Goodwin K D, Choi E S, Frable B W, Thompson A R, Walker H J Jr, Burton R S, Kacev D, Martz L D, Barber P H. Improving metabarcoding taxonomic assignment: a case study of fishes in a large marine ecosystem. Molecular Ecology Resources, 2021, 21(7): 2546-2564. DOI:10.1111/1755-0998.13450 |
| [146] |
Barnes M A, Turner C R. The ecology of environmental DNA and implications for conservation genetics. Conservation Genetics, 2016, 17(1): 1-17. DOI:10.1007/s10592-015-0775-4 |
| [147] |
Maruyama A, Nakamura K, Yamanaka H, Kondoh M, Minamoto T. The release rate of environmental DNA from juvenile and adult fish. PLoS One, 2014, 9(12): e114639. DOI:10.1371/journal.pone.0114639 |
| [148] |
Jo T, Murakami H, Yamamoto S, Masuda R, Minamoto T. Effect of water temperature and fish biomass on environmental DNA shedding, degradation, and size distribution. Ecology and Evolution, 2019, 9(3): 1135-1146. DOI:10.1002/ece3.4802 |
| [149] |
Díaz-Ferguson E E, Moyer G R. History, applications, methodological issues and perspectives for the use of environmental DNA (eDNA) in marine and freshwater environments. Revista De Biologia Tropical, 2014, 62(4): 1273-1284. DOI:10.15517/rbt.v62i4.13231 |
 2023, Vol. 43
2023, Vol. 43