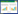文章信息
- 王倩, 刘红文, 贾淑霞, 申建国, 陈学文, 张士秀, 张延, 高燕, 梁爱珍
- WANG Qian, LIU Hongwen, JIA Shuxia, SHEN Jianguo, CHEN Xuewen, ZHANG Shixiu, ZHANG Yan, GAO Yan, LIANG Aizhen
- 保护性耕作对东北黑土微生物碳循环功能基因的影响
- Effect of conservation tillage on microbial functional genes related to carbon cycle of black soil
- 生态学报. 2023, 43(11): 4760-4771
- Acta Ecologica Sinica. 2023, 43(11): 4760-4771
- http://dx.doi.org/10.5846/stxb202110303049
-
文章历史
- 收稿日期: 2021-10-30
- 网络出版日期: 2023-02-07
2. 中国科学院大学, 北京 100049;
3. 北华大学林学院, 吉林 132013;
4. 农安县巴吉垒镇综合服务中心, 长春 130212
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. College of Forestry, Beihua University, Jilin 132013, China;
4. Bajilei Town, Nong'an County Comprehensive Service Center, Changchun 130212, China
全球陆地土壤每年向大气释放50—80.4 Pg(1015 g)碳, 是化石燃料燃烧年输入大气CO2量的11倍[1], 因此土壤碳库的微小变化都能引起大气中CO2浓度的剧烈响应[2]。农田是受人为干扰最为严重的陆地生态系统, 也最有潜力通过良好的耕作管理措施实现碳固定[3]。保护性耕作(减少土壤扰动、秸秆还田和轮作)是备受国际关注的既能满足人类需求又对环境影响最小的农业管理措施[4]。然而保护性耕作对土壤CO2排放量或速率的影响因研究区域、土壤和环境的不同而异。比如全球范围内耕作土壤的CO2排放量比免耕土壤多21%[5], 但是也有研究表明秸秆还田增加土壤CO2排放量(7%—45%)[6]。土壤类型、土壤有机碳含量、土壤输入新碳(根际分泌物、秸秆还田)的质量及可利用性是影响土壤CO2排放及碳循环的重要因素[5, 7], 因此保护性耕作通过增加碳源输入(秸秆还田)、改变土壤理化性质和微环境等影响农田生态系统碳循环过程[8], 秸秆还田被认为是增加农田生态系统土壤碳固定最有前途的方法[9—10]。
土壤微生物介导了如碳固定、甲烷代谢和碳降解等多个重要的碳循环基本过程[11], 是影响土壤碳循环和有机质(碳)稳定性的关键因素[12]。保护性耕作通过改变土壤微生物特征进而影响土壤碳存储、周转过程[13—17]。长期保护性耕作试验研究发现, 保护性耕作通过提高表层土壤微生物量, 增强土壤微生物代谢活性, 促使土壤微生物群落结构向着有利于有机碳积累的方向发展[14];通过提高土壤生物(微生物和线虫)的碳源利用效率(即增强以细菌通道为主的碳流传递效率), 促进土壤碳固存[16];还通过改变黑土团聚体中土壤生物(微生物和线虫)分布模式及相互作用强度, 增强团聚体中有机碳的累积和稳定性[17]。以往研究多侧重于保护性耕作下土壤微生物活性、生物量以及群落组成和多样性等对于土壤碳周转的影响, 然而关于土壤微生物基因组结构与碳循环之间关系的研究较少。
生物地球化学过程中的微生物功能取决于与其功能基因相关的活动或行为。Trivedi等[18]报道了功能基因与碳降解相关的酶活性密切相关。了解这些微生物功能基因能够更直接地估计微生物驱动的碳转化机制。已有学者报道, 土壤基质(如碳源)可通过驱动编码碳水化合物活性酶(CAZy)的基因转录, 影响碳循环过程[19]。随着高通量测序技术的发展和测序价格的大幅下降, 基于宏基因组的多样性分析现已成为可能。宏基因组分子技术试图分析从环境样本或纯培养中提取总DNA中存在的所有遗传信息, 相较于以往依赖于扩增引物和量化特定的基因目标来表征功能基因的技术, 利用该技术可以获得微生物遗传能力的整个视图[20—21]。Souza等[22]利用宏基因组技术研究发现, 保护性耕作改变碳代谢途径, 其中保护性耕作具有更多与芳香族化合物代谢相关的序列, 而传统耕作中具有更多与碳水化合物代谢相关的序列。Yang等[23]报道, 参与碳降解的功能基因模式与土壤温室气体排放密切相关。Jing[24]等报道, 外界变化(如氮沉降)通过改变某些碳循环功能基因的丰度改变土壤有机碳(SOC)的分解。但是, 保护性耕作下黑土碳循环有关的功能基因如何影响其碳循环过程尚不明确。
本研究以黑土区保护性耕作试验地(7年)为研究平台, 分析了耕作方式对土壤碳水化合物活性酶(CAZy)以及碳循环功能基因(碳固定、碳水化合物代谢以及甲烷代谢)的影响, 揭示保护性耕作下黑土碳循环功能基因信息;并结合作物生长季节内不同耕作处理土壤CO2释放速率的差异, 深入了解农田耕作措施对土壤碳循环过程及其功能基因的影响, 将有助于揭示土壤固碳的微生物作用机制。
1 材料与方法 1.1 研究区概况该试验地位于中国科学院东北地理与农业生态研究所长春保护性耕作观测站(44°00′N, 125°24′E), 初建于2012年。温带大陆性季风气候, 年平均降水量为515 mm, 年平均气温为0.9 ℃(1982—2009年)。试验区的土壤类型是中层典型黑土, pH值为7.3, 土壤总有机碳(土壤中不含无机碳)和总氮含量分别19.1 g/kg和1.6 g/kg。
1.2 试验设计试验采用单因素裂区随机区组设计, 处理包括免耕秸秆还田(NT)、秋翻秸秆还田(MP)和常规耕作(秸秆不还田)(CT), 每个处理四次重复, 每个小区面积为7.8 m×25 m, 种植作物和模式为玉米连作。免耕秸秆还田处理, 除了春季使用免耕播种机播种外, 不对土壤进行任何干扰, 收获后秸秆切成约30 cm小段返还土壤表面;秋翻秸秆还田处理, 包括春季整地(7.5—10 cm)、播种、中耕和秋收后翻耕(20 cm)等田间作业, 玉米秸秆在秋翻时移除试验小区, 在次年春季耕种时埋入土壤;常规耕作处理的田间管理与秋翻处理相同, 但玉米秸秆不还田。所有处理的播种时间、肥料种类和用量、田间管理措施均一致, 播种时先施89 kg N/hm2、51 kg P/hm2和78 kg K/hm2作为底肥, 并在V-6生长期再施用45 kg N/hm2作追肥。播种前后均使用除草剂和杀虫剂来控制杂草和害虫。
1.3 土壤CO2释放速率测定春天播种后, 在每个试验小区距地头10 m、垄的1/2处埋设3个高和直径均为20 cm的聚氯乙烯(PVC)环, PVC环在测量前一天插入土壤(约15 cm), 并将PVC环外围土壤轻轻压实, 以防测量中漏气。土壤CO2释放速率采用全自动土壤碳通量测量系统LI-Cor 8100(Li-Cor, 林肯,内布拉斯加州,美国)测定, 自播种之后生长季节内每两周测量一次, 测定时间为上午9:00—11:00。自试验地建立以来, 每年生长季节都测定土壤CO2释放速率。本研究以6年平均值反映不同耕作处理间土壤CO2释放速率的差异。
1.4 土壤样品采集于2019年4月份在试验区内用土钻采集0—5 cm土层的土壤, 每个小区随机采集7个土壤样点混合, 每个处理3次重复。采集的土壤样品放入带冰袋的保温箱, 并立即带回实验室。去除可见的根和石头, 过2 mm筛。一部分土壤样品在-80℃下储存, 用于DNA提取。另一部分新鲜土壤样品用于土壤含水量、氨态氮(NH4+-N)、硝态氮(NO3--N)、土壤水溶性有机碳(DOC)、溶解性总氮(DTN)的测定。另外在取样时在每个小区用3个100 cm3的环刀采集土壤样品用于测定土壤容重。
1.5 土壤样品测定土壤含水量和容重采用烘干法测定。土壤NH4+-N、NO3--N用2 mol/L氯化钾(KCL)溶液提取, 连续流动分析仪测定(SKALAR SAN++, 荷兰)。土壤DOC、DTN用0.5 mol/L K2SO4溶液浸提, TOC总有机碳分析仪测定(TOC-L CPH, SHIMADZU, 日本)。
1.6 鸟枪法宏基因组测序按照制造商的说明, 使用土壤DNA提取试剂盒(Mo Bio Laboratories, 卡尔斯巴德,加利福尼亚州,美国)提取DNA。利用1%琼脂糖凝胶电泳检查DNA质量后, 使用Covaris M220将DNA剪切成约400 bp的片段。使用NEXTFLEX Rapid DNA-Seq(Bioo Scientific, 奥斯汀,得克萨斯州,美国)构建文库, 然后在Illumina Hiseq 4000测序平台(Illumina Inc., 圣迭戈,加利福尼亚州,美国)上进行宏基因组测序(上海美吉生物医药科技有限公司)。原始数据已提交至美国国家生物技术信息中心(NCBI)的测序数据存储库(SRA)(序列号:PRJNA692995)。
使用FASTP v0.20.0(https://github.com/OpenGene/fastp)修剪原始序列读数, 以除去衔接子和低质量序列。经过质量过滤后, 由MEGAHIT v1.1.2[25]对优化序列进行拼接组装, 在拼接结果中筛选300 bp以上的重叠群(contigs)作为最终的组装结果。使用MetaGene[26](http://metagene.cb.k.u-tokyo.ac.jp/)对拼接结果中的contigs进行ORF预测。使用CD-HIT将所有预测的基因序列进行聚类(95%置信区间、90%覆盖率)[27], 并选择最长的序列作为代表性序列, 构建非冗余基因集数。使用SOAPaligner v2.21软件[28]将每个样品的优化序列与非冗余基因集进行比对(95%置信区间), 统计基因在对应样品中的丰度信息。
使用DIAMOND v 0.8.35[29]将非冗余基因集的氨基酸序列分别与NCBI非冗余蛋白(NR)数据库、碳水化合物活性酶(CAZy)数据库和京都基因与基因组百科全书(KEGG)数据库进行分类和功能注释(E值≤1×10-5)。
1.7 统计分析利用SPSS 23.0(SPSS Inc., 芝加哥,伊利诺伊州,美国), 通过方差分析(ANOVA)确定耕作方式对土壤CO2释放速率的影响;通过Levene检验确定符合方差齐性后, 利用方差分析(ANOVA)来检查不同耕作方式之间碳循环功能基因的差异性;通过相关性分析确定土壤碳循环功能基因组成(PCoA第一主成分)和土壤CO2释放速率的关系;基于R包(Pheatmap)绘制组间显著差异功能基因的聚类热图及相关性热图;基于斯皮尔曼相关(P < 0.05), 采用Gephi软件进行网络分析。主坐标分析(PCoA), 冗余分析(db-RDA), 置换多元方差分析(PERMANOVA)以及物种与功能贡献分析借助美吉云平台实现。
2 结果 2.1 耕作方式对土壤CO2释放速率的影响基于6年的平均值, 免耕土壤的平均CO2释放速率低于秋翻和常规耕作, 主要体现在生长季节前期, 免耕土壤CO2释放速率分别比秋翻低28%(5月份)、11%(6月份)和23%(7月份);比常规耕作低31%(5月份)、19%(6月份)和7%(7月份)。免耕、秋翻与常规耕作处理间7月份的土壤CO2释放速率存在显著差异(图 1)。
|
| 图 1 不同耕作方式下土壤CO2释放速率 Fig. 1 Soil CO2 efflux under different tillage treatments 不同小写字母代表耕作处理间差异显著, P < 0.05 |
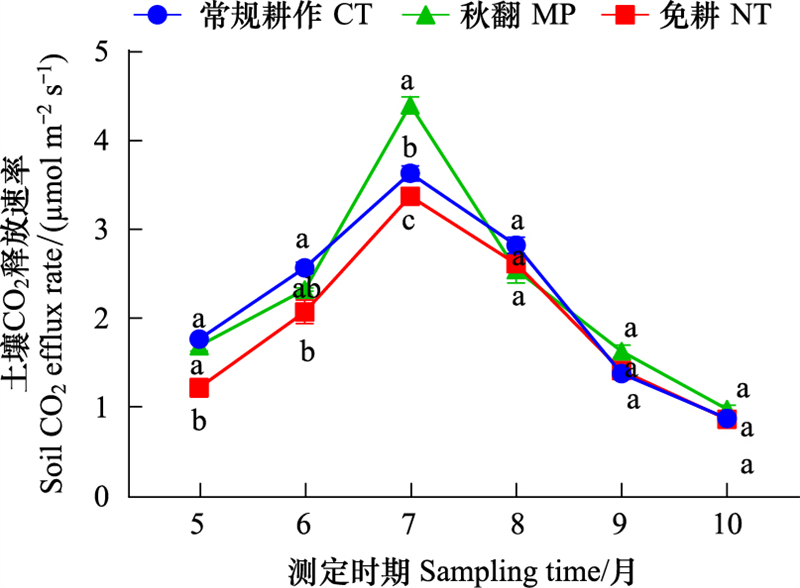
CAZy数据库中六种碳水化合物活性酶家族(糖苷水解酶(GHs)、糖基转移酶(GTs)、多糖裂解酶(PLs)、碳水化合物酯酶(CEs)、辅助活性氧化还原酶(AAs)和碳水化合物结合模块(CBMs))在三种耕作处理中均全部存在, 且以糖苷水解酶(GHs)(约58%)和糖基转移酶(GTs)(约28%)占主导(表 1)。在不同耕作处理下, 我们发现一些存在显著差异的CAZy家族(图 2), 主要集中在GHs和PLs上, 其中免耕和秋翻的糖苷水解酶类(GH102)显著高于常规耕作;而免耕的糖基转移酶(GT39)和多糖裂解酶(PL17)类低于常规耕作, 秋翻的GT39和PL17则高于常规耕作(图 2)。总体上, 免耕和秋翻处理的差异CAZy基因的相对丰度高于常规耕作(图 2)。贡献分析显示, GH102、GT39、PL17、GH5_38和GH13_17酶类的主要贡献物种包括变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、芽单胞菌门(Gemmatimonadetes)和拟杆菌门(Bacteroidetes)(图 2)。
| 糖苷水解酶 GHs |
糖基转移酶 GTs |
碳水化合物酯酶 CEs |
辅助活性氧化还原酶 AAs |
碳水化合物结合模块 CBMs |
多糖裂解酶 PLs |
|
| 免耕NT | 34156.0±1619 | 16472.6±884 | 3401.3±178 | 2026.0±208 | 600.7±36 | 672.0±7 |
| 秋翻MP | 32606.7±2621 | 15358.0±1193 | 3166.0±274 | 2224.7±234 | 524.0±24 | 706.7±94 |
| 常规CT | 32280.7±1573 | 14928.0±885 | 2990.7±193 | 2374.7±140 | 484.0±70 | 608.0±46 |
| CAZy: 碳水化合物活性酶; GHs:糖苷水解酶Glycoside Hydrolases; GTs:糖基转移酶Glycosyl Transferases; CEs:碳水化合物酯酶Carbohydrate Esterases; AAs:辅助活性氧化还原酶Auxiliary Activities; CBMs:碳水化合物结合模块Carbohydrate-Binding Modules; PLs:多糖裂解酶Polysaccharide Lyases; NT:免耕no-tillage; MP:秋翻moldboard plow;CT:常规耕作conventional tillage | ||||||
|
| 图 2 不同耕作方式下差异CAZy基因(P < 0.05), 丰度前10的优势物种(门) 对差异CAZy基因的贡献 Fig. 2 Significantly different CAZy genes among different tillage regimes, contribution analysis of the top 10 abundant phyla to the significantly different CAZy genes among three tillage treatments 糖苷水解酶类:GH102,GH5—38,GH13—17;糖基转移酶类:GT39;多糖裂解酶类: PL17,PL5—1;CAZy:碳水化合物活性酶;NT:免耕No-tillage;MP:秋翻Moldboard plow;CT:常规耕作conventional tillage |
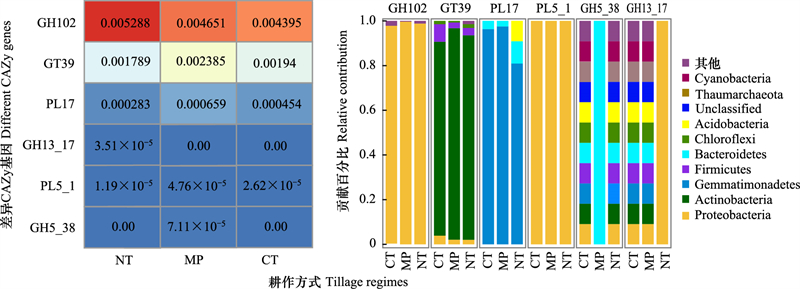
本文关注了三大重要的碳循环过程(碳固定、甲烷代谢以及碳水化合物代谢)功能基因。通过KEGG数据库注释分别得到107个、123个和697个与碳固定、甲烷代谢和碳水化合物代谢相关的功能基因(KO)类别。主坐标分析(PCoA)表明不同耕作处理土壤碳循环功能基因组成差异显著(Adonis, R2=0.45; P=0.006)(图 3), 前两轴分别解释了总功能基因组成变异的61.22%和18.89%。基于距离的冗余分析(db-RDA)分析表明土壤水溶性有机碳含量和容重是影响碳循环功能基因组成的主导土壤因子(图 3)。
|
| 图 3 不同耕作方式土壤碳循环功能基因的主坐标分析(PCoA) 及基于距离的冗余分析(db-RDA) Fig. 3 PCoA (principal co-ordinates analysis) and db-RDA (distance-based redundancy analysis) plots of the functional genes related to carbon cycling under different tillage regimes SBD:土壤容重Soil bulk density;SWC:土壤含水量Soil water content; NH4+-N:土壤氨态氮Soil ammonium (NH4+) contents;NO3--N:土壤硝态氮Soil nitrate (NO3-) contents;DOC:土壤水溶性有机碳Soil dissolved organic carbon;DTN:土壤溶解性总氮Soil dissolved total nitrogen;*环境因子影响的显著性; *表示P < 0.05;**表示P < 0.01 |
通过比较得到了不同耕作处理之间具有显著差异的功能基因(相对丰度>0.1%), 其中秋翻和常规耕作土壤碳固定、甲烷代谢以及碳水化合物代谢功能基因组成类似, 聚类在一起, 免耕单独聚为一类(图 4)。耕作处理间共有8个碳固定功能基因存在显著差异(P < 0.05)。免耕土壤K01007和K00170功能基因丰度比常规耕作低19%和21%, 比秋翻低14%和17%;而K00625、K01676、K09709、K00925和K14470碳固定功能基因的丰度则比常规耕作高约17%(12%—21%), 比秋翻高11%(3%—18%)(图 4)。免耕土壤上调的碳固定功能基因主要与土壤DOC含量、土壤容重正相关(图 4、5, P < 0.05), 而常规耕作土壤上调的碳固定功能基因(如K01007、K00170)与土壤DOC含量负相关(图 4、5, P < 0.05)。
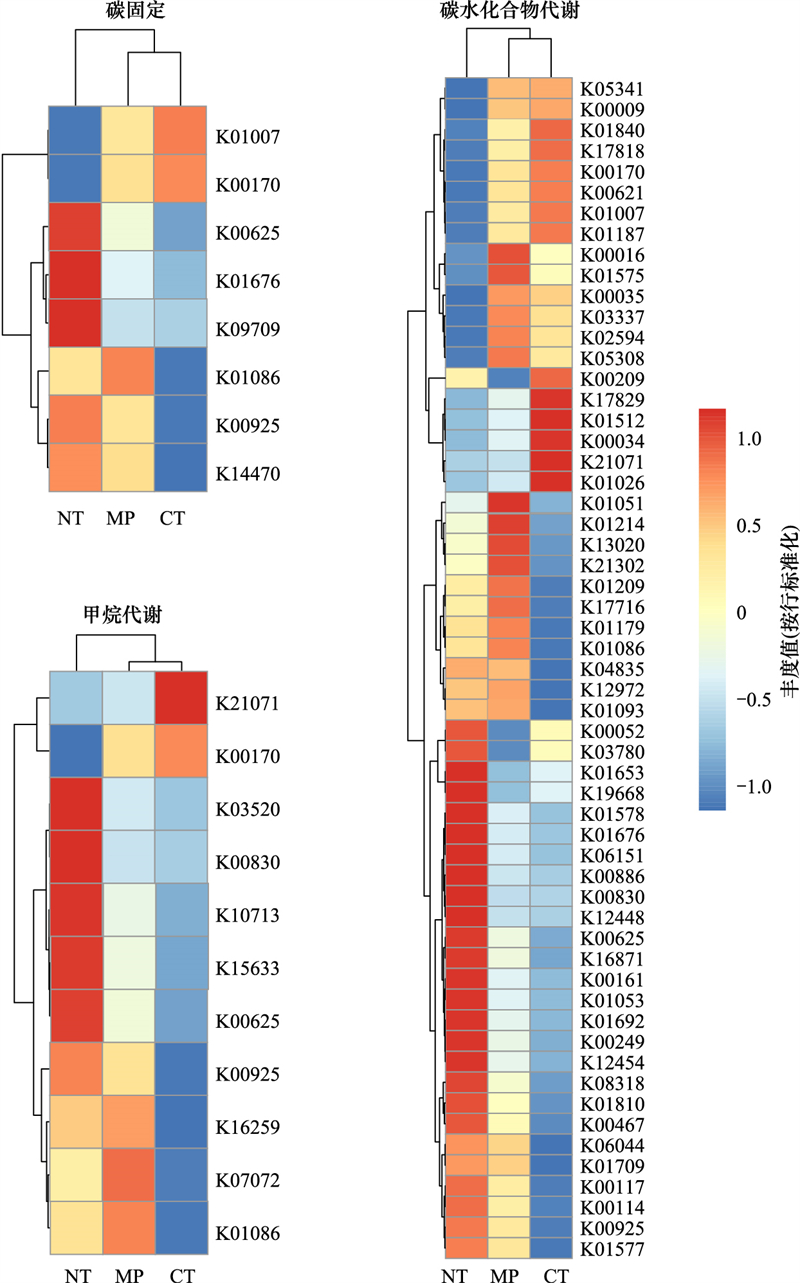
|
| 图 4 不同耕作方式下土壤碳固定, 甲烷代谢和碳水化合物代谢功能基因相对丰度聚类 Fig. 4 The clustering heatmap of the relative abundance of functional genes related to carbon fixation, methane metabolism and carbohydrate metabolism under different tillage regimes |
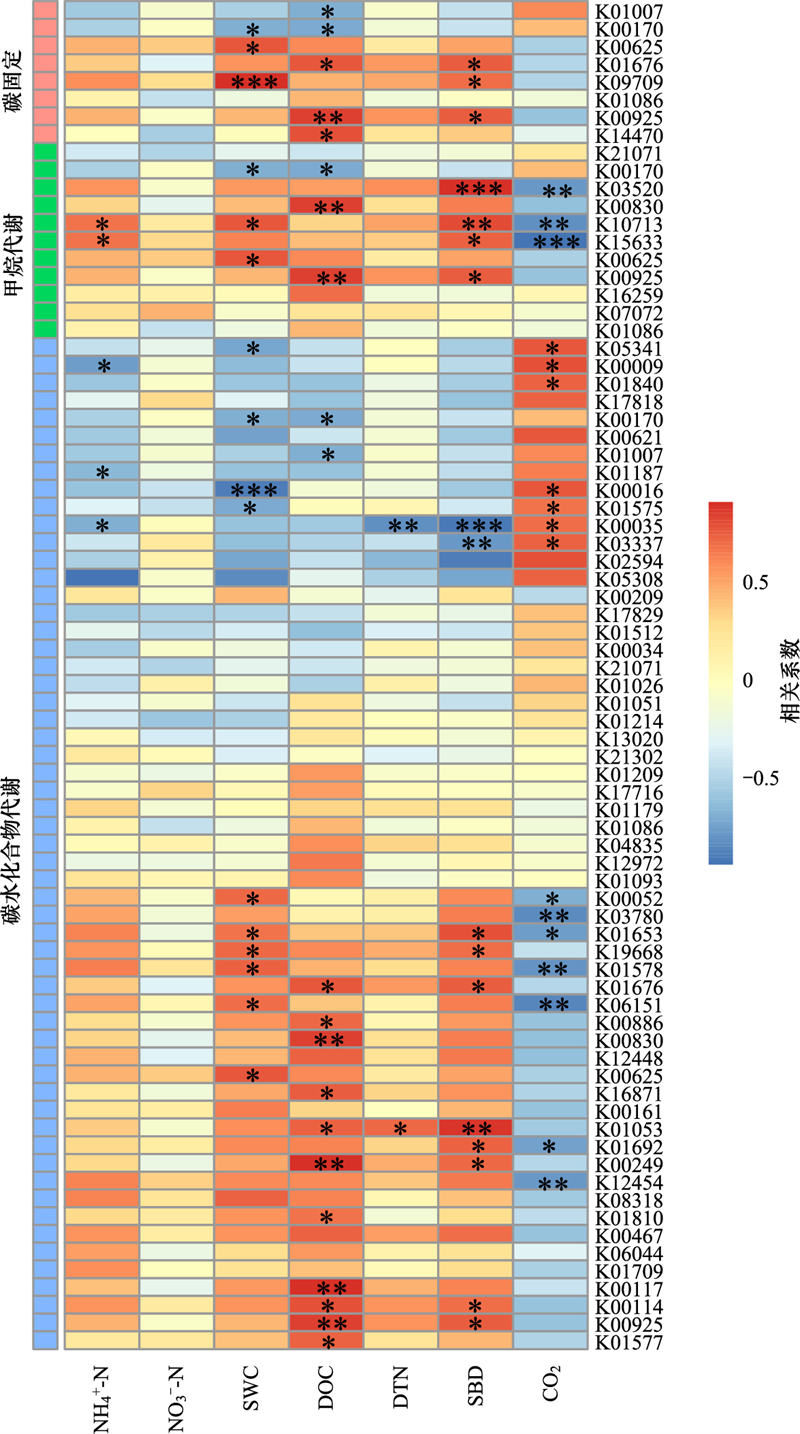
|
| 图 5 耕作处理间具有显著差异的碳循环功能基因与土壤因子的相关性 Fig. 5 Correlation heatmap of significantly different soil functional genes related to carbon cycle and soil properties under different tillage regimes *显著性检验, *表示P < 0.05; **表示P < 0.01;***表示P < 0.001 |
耕作处理间共有11个甲烷代谢功能基因存在显著差异(P < 0.05)。免耕土壤6个甲烷代谢基因的丰度(K03520、K00830、K10713、K15633、K00625和K00925)比常规耕作高15%(2%—17%), 比秋翻高10%(9%—24%);另外2个甲烷代谢基因(K00170和K21071)的丰度则比常规耕作低13%(6%—21%), 比秋翻低11%(6%—16%)(图 4)。免耕土壤上调的甲烷代谢基因主要与土壤DOC、土壤容重或土壤含水量显著正相关(图 4、图 5, P < 0.05)。
耕作处理间共有57个碳水化合物代谢功能基因存在显著差异(P < 0.05)。免耕土壤一些碳水化合物代谢基因丰度上调, 一些基因下调。例如, 免耕土壤的一些碳水化合物代谢基因的丰度(如K05341、K00009、K01840、K17818、K00170、K00621和K00035等)比常规耕作低25%(7%—74%), 比秋翻低18%(0.4%—80%);而K00052、K03780、K01653、K00117、K00114和K00925等碳水化合物功能基因的丰度则比秋翻高14%(2%—44%), 比常规耕作高23%(2%—84%)(图 4)。绝大多数免耕土壤的上调基因(包括K00886、K00830、K01676、K00117和K00114等)与土壤DOC及土壤容重显著正相关(P < 0.05), 一些上调基因(如K00052、K01653、K19668和K1578等)也与土壤含水量显著正相关(图 4、5, P < 0.05)。
通过建立物种(丰度前30的门)和土壤碳循环功能基因的相关性网络图(图 6), 根据网络图中节点的度数, 确定奇古菌(Thaumarchaeota)对土壤碳循环功能的贡献最大, 此外, 酸杆菌门(Acidobacteria), 泉古菌门(Crenarchaeota)及绿弯菌门(Chloroflexi)同样具有重要贡献。
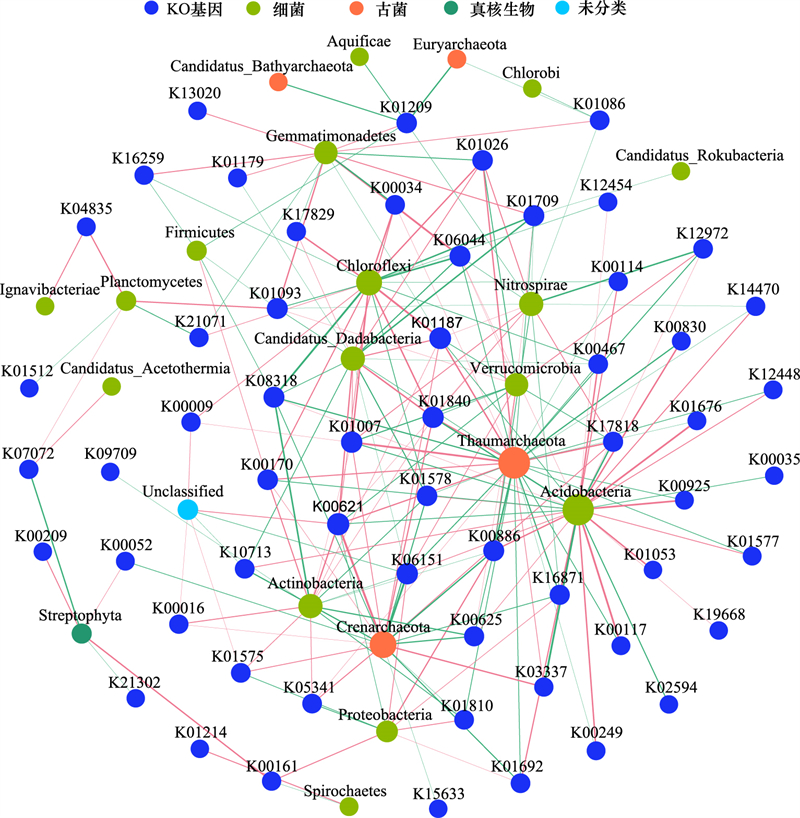
|
| 图 6 不同耕作处理下土壤碳循环功能基因与微生物门(丰度前30) 的相关性 Fig. 6 Correlation network diagram of soil C-cycling functional genes and phyla (top 30) 网络图按类别着色;节点大小与节点的度成正比;红边为正相关, 绿边为负相关 |
相关性分析表明土壤CO2释放速率与碳循环功能基因组成显著相关(R2=0.80;P < 0.01), 尤其是与一些碳水化合物代谢功能基因显著相关, 例如与碳水化合物代谢功能基因K05341、K00009、K01840、K00016、K01575、K00035和K03337显著正相关;而与K00052、K03780、K01653、K01578、K06151、K01692及K12454等碳水化合物代谢功能基因显著负相关(图 5)。
3 讨论农田管理措施(如耕作、覆盖方式等)会对土壤理化性质、土壤微生物群落多样性等产生影响[25—27], 进而影响由土壤微生物介导的碳循环过程[30]。已有许多关于保护性耕作下土壤微生物活性、多样性及群落组成的研究[31—32]。一些研究已经表明不同耕作处理下SOC的周转与特定土壤微生物群落特征的密切关系[14—15], 然而具体涉及土壤碳循环功能基因组成的研究仍极少。秸秆还田是农田土壤微生物的主要输入碳源, 土壤微生物对秸秆的分解利用影响土壤以及农田生态系统的碳循环过程。因此, 保护性耕作(免耕)中秸秆的输入量、还田方式以及种类都将是引起土壤微生物群落碳循环功能差异的主要原因。本研究利用宏基因测序技术, 探索了土壤碳循环功能基因对于秸秆还田和少耕的响应。基于宏基因组分析结果, 发现耕作处理之间土壤碳循环功能基因丰度和组成存在差异。一方面耕作处理之间一些糖苷水解酶(如GH102、GH5_38和GH13_17), 糖基转移酶(如GT39)和多糖裂解酶(PL17和PL5_1)在基因丰度上存在显著差异。碳水化合物活性酶(CAZy)可用于合成和降解碳水化合物和糖缀合物。糖基转移酶通常参与二糖、寡糖和多糖的生物合成[33];糖苷水解酶和多糖裂解酶在纤维素、半纤维素和木质素的水解中起重要作用[34—35]。因为底物性质会改变微生物群落的结构和功能基因丰度, 以确保它们可以获得足够的资源[36]。总体上, 免耕和秋翻土壤这些基因的相对丰度高于常规耕作(图 2), 说明来自免耕和秋翻土壤的微生物在分解残留物方面具有更强的活性, 释放简单的、易被植物吸收的小分子物质, 重新激活养分循环, 进而改善土壤质量[37—38]。已有研究表明, 秸秆还田(碳源输入)使土壤中积累了高浓度的碳[39], 由此推断免耕和秋翻中大量的以秸秆还田形式输入的碳源可以上调一些CAZy基因(相对于常规耕作), 因此, 有利于促成有机物含量高的土壤营养条件。
另一方面, 研究发现耕作处理间土壤碳循环功能基因组成具有显著差异, 秋翻与常规耕作具有类似的土壤碳固定, 甲烷代谢以及碳水化合物代谢的功能基因组成, 免耕与其他处理存在差异, 单独聚为一类(图 4)。土壤DOC及土壤容重是驱动土壤碳循环功能基因组成的主导因子(图 3), 绝大多数免耕土壤上调的碳循环功能基因与土壤DOC、容重或含水量显著正相关(图 5)。秸秆还田不仅增加土壤水溶性有机碳含量, 同时秸秆覆盖也有利于维持土壤湿度[40—42], 这些改善的物理化学环境显著改变土壤微生物丰度以及群落组成[42—43]。本研究结果表明, 秸秆还田(免耕和秋翻)可以增加土壤微生物所需要的碳源, 并且有利于提高土壤微生物活性, 比如碳水化合物活性酶(CAZy), 但是还需要保护性耕作(免耕)所形成的微环境为土壤微生物群落功能的发挥提供场所。另外, 本研究中奇古菌门(Thaumarchaeota)对土壤碳循环功能的贡献最大, 虽然土壤中古菌的相对丰度小于细菌, 但其在土壤碳循环中可能发挥着重要的作用, 深入探讨保护性耕作土壤古菌的功能有助于阐明农田生态系统土壤微生物的固碳机制。
多年野外监测数据表明免耕处理生长季节前期(5至7月)的土壤CO2释放速率比秋翻和常规耕作低(图 1)。与传统耕作相比, 免耕通过减少对土壤结构的破坏有利于有机物质的积累[40]。这与之前的研究结果一致, 即免耕(实施13年后)以0.80 Mg C hm-2 a-1)的速率显著增加了土壤有机碳储量(30 cm深度)[39]。生长季节后期(8至10月)各处理间土壤CO2释放速率无显著差异, 与土壤系统复杂性, 植物生长因素的影响有关。土壤CO2释放速率与碳循环功能基因组成显著相关, 主要与土壤碳水化合物代谢功能基因包括K05341、K00009、K01840、K00016、K01575、K00035和K03337显著正相关;而与K00052、K03780、K01653、K01578、K06151、K01692及K12454等碳水化合物代谢功能基因显著负相关(图 5)。免耕处理通过调节土壤碳水化合物代谢功能基因的相对丰度, 即下调与土壤CO2释放速率正相关的土壤碳水化合物代谢功能基因的相对丰度, 上调负相关的土壤碳水化合物代谢功能基因的相对丰度(图 4、5), 从而导致免耕处理生长季节前期的土壤CO2释放低于秋翻和常规耕作处理(图 1)。再者, 秸秆还田和减少扰动导致的土壤DOC及容重变化是引起土壤碳循环功能基因差异的主导因子。因此, 推断免耕的秸秆还田和减少耕作的共同效应通过影响土壤微生物碳循环功能基因组成来降低土壤CO2释放速率, 进而提高土壤固碳潜力。研究初步探索了耕作方式对土壤碳循环功能基因组成与土壤CO2排放的影响, 以土壤全基因组的测定和野外监测为主要手段, 并未涉及具体的碳循环途径, 只能通过相关分析来解释土壤CO2排放与土壤碳循环功能基因组成之间的关系, 因此还需要进行更加深入的调控试验来揭示其中的调控机制。
4 结论宏基因组数据分析结果表明耕作处理之间土壤碳循环功能基因组成存在差异。相对于常规处理, 免耕和秋翻处理的糖苷水解酶(GH102、GH5_38和GH13_17), 糖基转移酶(GT39)和多糖裂解酶(PL17和PL5_1)等碳水化合物活性酶(CAZy)上调。但是免耕土壤碳循环功能基因组成不同于秋翻和常规耕作, 单独聚为一类, 免耕土壤拥有较多上调丰度的碳固定以及甲烷代谢功能基因, 而碳水化合物代谢功能基因的丰度既有上调也有下调。这些显著差异的土壤碳循环功能基因组成与土壤CO2释放显著相关。免耕处理生长季节前期较低的土壤CO2释放速率与土壤碳水化合物代谢功能基因的调控作用相关, 即免耕处理下调与土壤CO2释放速率正相关的功能基因的相对丰度, 而上调与之负相关的功能基因的相对丰度。土壤水溶性有机碳和容重是土壤碳循环功能基因组成的主要驱动因子。推断免耕秸秆还田和减少耕作的叠加效应通过改变土壤碳循环功能基因组成来提高土壤固碳潜力。
| [1] |
Kuzyakov Y, Larionova A A. Root and rhizomicrobial respiration: a review of approaches to estimate respiration by autotrophic and heterotrophic organisms in soil. Journal of Plant Nutrition and Soil Science, 2005, 168(4): 503-520. DOI:10.1002/jpln.200421703 |
| [2] |
Kuzyakov Y. Sources of CO2 efflux from soil and review of partitioning methods. Soil Biology and Biochemistry, 2006, 38(3): 425-448. DOI:10.1016/j.soilbio.2005.08.020 |
| [3] |
夏龙龙, 颜晓元, 蔡祖聪. 我国农田土壤温室气体减排和有机碳固定的研究进展及展望. 农业环境科学学报, 2020, 39(4): 834-841. |
| [4] |
韩明会, 李保国, 张丹, 李颖. 再生农业——基于土地保护性利用的可持续农业. 中国农业科学, 2021, 54(5): 1003-1016. |
| [5] |
Abdalla K, Chivenge P, Ciais P, Chaplot V. No-tillage lessens soil CO2 emissions the most under arid and sandy soil conditions: results from a meta-analysis. Biogeosciences, 2016, 13(12): 3619-3633. DOI:10.5194/bg-13-3619-2016 |
| [6] |
张国, 王效科. 我国保护性耕作对农田温室气体排放影响研究进展. 农业环境科学学报, 2020, 39(4): 872-881. |
| [7] |
Fontaine S, Bardoux G, Abbadie L, Mariotti A. Carbon input to soil may decrease soil carbon content. Ecology Letters, 2004, 7(4): 314-320. DOI:10.1111/j.1461-0248.2004.00579.x |
| [8] |
薛建福, 赵鑫, Dikgwatlhe S B, 陈阜, 张海林. 保护性耕作对农田碳、氮效应的影响研究进展. 生态学报, 2013, 33(19): 6006-6013. |
| [9] |
Lugato E, Berti A, Giardini L. Soil organic carbon (SOC) dynamics with and without residue incorporation in relation to different nitrogen fertilisation rates. Geoderma, 2006, 135: 315-321. DOI:10.1016/j.geoderma.2006.01.012 |
| [10] |
Lu F, Wang X K, Han B, Ouyang Z Y, Duan X N, Zheng H, Miao H. Soil carbon sequestrations by nitrogen fertilizer application, straw return and no-tillage in China's cropland. Global Change Biology, 2009, 15(2): 281-305. DOI:10.1111/j.1365-2486.2008.01743.x |
| [11] |
Bardgett R D, Freeman C, Ostle N J. Microbial contributions to climate change through carbon cycle feedbacks. The ISME Journal, 2008, 2(8): 805-814. DOI:10.1038/ismej.2008.58 |
| [12] |
Gleixner G, Czimczik C, Kramer C, Lühker B, Schmidt M W I. Plant compounds and their turnover and stabilization as soil organic matter//Schulze E D, Heimann M, Harrison S, Holland E, Lloyd J, Prentice I C, Schimel D, eds. Global Biogeochemical Cycles in the Climate System. San Diego: Academic Press, 2001: 201-215.
|
| [13] |
Li Y, Chang S X, Tian L H, Zhang Q P. Conservation agriculture practices increase soil microbial biomass carbon and nitrogen in agricultural soils: a global meta-analysis. Soil Biology and Biochemistry, 2018, 121: 50-58. DOI:10.1016/j.soilbio.2018.02.024 |
| [14] |
孙冰洁. 不同耕作方式下土壤微生物在黑土有机碳固定中的作用研究[D]. 长春: 中国科学院研究生院(东北地理与农业生态研究所), 2016.
|
| [15] |
Li Y M, Duan Y, Wang G L, Wang A Q, Shao G Z, Meng X H, Hu H Y, Zhang D M. Straw alters the soil organic carbon composition and microbial community under different tillage practices in a meadow soil in Northeast China. Soil and Tillage Research, 2021, 208: 104879. DOI:10.1016/j.still.2020.104879 |
| [16] |
Zhang S X, McLaughlin N B, Cui S Y, Yang X M, Liu P, Wu D H, Liang A Z. Effects of long-term tillage on carbon partitioning of nematode metabolism in a Black soil of Northeast China. Applied Soil Ecology, 2019, 138: 207-212. DOI:10.1016/j.apsoil.2019.03.006 |
| [17] |
Zhang S X, Li Q, Lü Y, Zhang X P, Liang W J. Contributions of soil biota to C sequestration varied with aggregate fractions under different tillage systems. Soil Biology and Biochemistry, 2013, 62: 147-156. DOI:10.1016/j.soilbio.2013.03.023 |
| [18] |
Trivedi P, Delgado-Baquerizo M, Trivedi C, Hu H W, Anderson I C, Jeffries T C, Zhou J Z, Singh B K. Microbial regulation of the soil carbon cycle: evidence from gene-enzyme relationships. The ISME Journal, 2016, 10(11): 2593-2604. DOI:10.1038/ismej.2016.65 |
| [19] |
Ren C J, Zhang X Y, Zhang S H, Wang J Y, Xu M P, Guo Y X, Wang J, Han X H, Zhao F Z, Yang G H, Doughty R. Altered microbial CAZyme families indicated dead biomass decomposition following afforestation. Soil Biology and Biochemistry, 2021, 160: 108362. DOI:10.1016/j.soilbio.2021.108362 |
| [20] |
Liu Y X, Qin Y, Chen T, Lu M P, Qian X B, Guo X X, Bai Y. A practical guide to amplicon and metagenomic analysis of microbiome data. Protein & Cell, 2021, 12(5): 315-330. |
| [21] |
Bouchez T, Blieux A L, Dequiedt S, Domaizon I, Dufresne A, Ferreira S, Godon J J, Hellal J, Joulian C, Quaiser A, Martin-Laurent F, Mauffret A, Monier J M, Peyret P, Schmitt-Koplin P, Sibourg O, D'oiron E, Bispo A, Deportes I, Grand C, Cuny P, Maron P A, Ranjard L. Molecular microbiology methods for environmental diagnosis. Environmental Chemistry Letters, 2016, 14(4): 423-441. DOI:10.1007/s10311-016-0581-3 |
| [22] |
Souza R C, Hungria M, Cantão M E, Vasconcelos A T R, Nogueira M A, Vicente V A. Metagenomic analysis reveals microbial functional redundancies and specificities in a soil under different tillage and crop-management regimes. Applied Soil Ecology, 2015, 86: 106-112. DOI:10.1016/j.apsoil.2014.10.010 |
| [23] |
Yang J J, Li G H, Qian Y, Yang Y F, Zhang F. Microbial functional gene patterns related to soil greenhouse gas emissions in oil contaminated areas. Science of the Total Environment, 2018, 628-629: 94-102. DOI:10.1016/j.scitotenv.2018.02.007 |
| [24] |
Jing H, Li J J, Yan B S, Wei F R, Wang G L, Liu G B. The effects of nitrogen addition on soil organic carbon decomposition and microbial C-degradation functional genes abundance in a Pinus tabulaeformis forest. Forest Ecology and Management, 2021, 489: 119098. DOI:10.1016/j.foreco.2021.119098 |
| [25] |
Li D H, Liu C M, Luo R B, Sadakane K, Lam T W. MEGAHIT: an ultra-fast single-node solution for large and complex metagenomics assembly via succinct de Bruijn graph. Bioinformatics, 2015, 31(10): 1674-1676. DOI:10.1093/bioinformatics/btv033 |
| [26] |
Noguchi H, Park J, Takagi T. MetaGene: prokaryotic gene finding from environmental genome shotgun sequences. Nucleic Acids Research, 2006, 34(19): 5623-5630. DOI:10.1093/nar/gkl723 |
| [27] |
Li W Z, Godzik A. Cd-hit: a fast program for clustering and comparing large sets of protein or nucleotide sequences. Bioinformatics, 2006, 22(13): 1658-1659. DOI:10.1093/bioinformatics/btl158 |
| [28] |
Li R, Li Y, Kristiansen K, Wang J. SOAP: short oligonucleotide alignment program. Bioinformatics, 2008, 24(5): 713-714. DOI:10.1093/bioinformatics/btn025 |
| [29] |
Buchfink B, Xie C, Huson D H. Fast and sensitive protein alignment using DIAMOND. Nature Methods, 2015, 12(1): 59-60. DOI:10.1038/nmeth.3176 |
| [30] |
刘子涵, 黄方园, 黎景来, 张鹏, 杨宝平, 丁瑞霞, 聂俊峰, 贾志宽. 覆盖模式对旱作农田土壤微生物多样性及群落结构的影响. 生态学报, 2021, 41(7): 2750-2760. |
| [31] |
贾淑霞, 孙冰洁, 梁爱珍, 陈学文, 张士秀, 魏守才, 刘四义, 陈升龙, 张晓平. 耕作措施对东北黑土微生物呼吸的影响. 中国农业科学, 2015, 48(9): 1764-1773. |
| [32] |
李彤, 王梓廷, 刘露, 廖允成, 刘杨, 韩娟. 保护性耕作对西北旱区土壤微生物空间分布及土壤理化性质的影响. 中国农业科学, 2017, 50(5): 859-870. |
| [33] |
Schmid J, Heider D, Wendel N J, Sperl N, Sieber V. Bacterial glycosyltransferases: challenges and opportunities of a highly diverse enzyme class toward tailoring natural products. Frontiers in Microbiology, 2016, 7: 182. |
| [34] |
Rossi M F, Mello B, Schrago C G. Performance of hidden Markov models in recovering the standard classification of glycoside hydrolases. Evolutionary Bioinformatics, 2017, 13: 1-5. |
| [35] |
Kaushal G, Kumar J, Sangwan R S, Singh S P. Metagenomic analysis of geothermal water reservoir sites exploring carbohydrate-related thermozymes. International Journal of Biological Macromolecules, 2018, 119: 882-895. |
| [36] |
Schimel J P, Gulledge J. Microbial community structure and global trace gases. Global Change Biology, 1998, 4(7): 745-758. |
| [37] |
León J D, Osorio N W. Role of litter turnover in soil quality in tropical degraded lands of Colombia. The Scientific World Journal, 2014, 2014: 693981. |
| [38] |
Zhang L, Lv J P. Metagenomic analysis of microbial community and function reveals the response of soil respiration to the conversion of cropland to plantations in the Loess Plateau of China. Global Ecology and Conservation, 2020, 23: e01067. |
| [39] |
Zhang Y, Li X J, Gregorich E G, Mclaughlin N B, Zhang X P, Guo Y F, Liang A Z, Fan R Q, Sun B J. No-tillage with continuous maize cropping enhances soil aggregation and organic carbon storage in Northeast China. Geoderma, 2018, 330: 204-211. |
| [40] |
陈学文, 张晓平, 梁爱珍, 贾淑霞, 时秀焕, 范如芹, 魏守才. 耕作方式对黑土硬度和容重的影响. 应用生态学报, 2012, 23(2): 439-444. |
| [41] |
Page K L, Dang Y P, Dalal R C, Reeves S, Thomas G, Wang W J, Thompson J P. Changes in soil water storage with no-tillage and crop residue retention on a Vertisol: impact on productivity and profitability over a 50 year period. Soil and Tillage Research, 2019, 194: 104319. |
| [42] |
Hao M M, Hu H Y, Liu Z, Dong Q L, Sun K, Feng Y P, Li G, Ning T Y. Shifts in microbial community and carbon sequestration in farmland soil under long-term conservation tillage and straw returning. Applied Soil Ecology, 2019, 136: 43-54. |
| [43] |
Pinheiro E F M, PEreira M G, Anjos L H C. Aggregate distribution and soil organic matter under different tillage systems for vegetable crops in a Red Latosol from Brazil. Soil and Tillage Research, 2004, 77(1): 79-84. |
 2023, Vol. 43
2023, Vol. 43