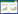文章信息
- 肖红, 刘玉玲, 刘忠宽, 李鹏珍, 戎郁萍
- XIAO Hong, LIU Yuling, LIU Zhongkuan, LI Pengzhen, RONG Yuping
- 氮磷添加对草甸草原土壤氮磷转化功能基因丰度的影响
- Responses of soil nitrogen and phosphorus transformation functional genes abundances to nitrogen and/or phosphorus additions in a meadow steppe
- 生态学报. 2023, 43(1): 313-326
- Acta Ecologica Sinica. 2023, 43(1): 313-326
- http://dx.doi.org/10.5846/stxb202112093495
-
文章历史
- 收稿日期: 2021-12-09
- 网络出版日期: 2022-08-29
2. 河北省农林科学院农业资源环境研究所, 石家庄 050051
2. Institute of Agricultural Resources and Environment Hebei Academy of Agriculture and Forestry Sciences, Shijiazhuang 050051, China
氮(N)和磷(P)作为主要的营养元素, 对调控陆地生态系统植物生产力和土壤微生物功能具有重要作用[1]。氮磷肥添加是提高过度放牧退化草地生产力和牧草品质的主要养分管理措施之一[2]。然而, 过量的氮输入会使土壤酸化、增加硝酸盐淋溶损失和温室气体排放等[3]。由土壤氮转化微生物驱动的土壤硝化和反硝化过程与硝酸盐淋溶损失和N2O气体排放紧密相关[4—6], 因此, 了解草原生态系统参与氮循环的主要土壤微生物功能群对养分添加的响应机理有助于草地养分调控, 避免养分过量输入引起的氮损失和温室气体排放[2]。
土壤硝化作用(NH4+向NO3-的转化)是由氨氧化细菌(AOB)和氨氧化古菌(AOA)分泌的氨单加氧酶催化的, 氨氧化是土壤硝化作用的关键限速步骤[7]。amoA基因编码氨单加氧酶的A亚基, 氨氧化细菌amoA基因(amoA-AOB)和氨氧化古菌amoA基因(amoA-AOA)目前被确定为土壤硝化作用过程的标记基因[8]。土壤反硝化作用(NO3-→NO2-→NO→N2O→N2的转化过程)是由一系列反硝化细菌功能群产生的还原酶催化的。反硝化的第一步(NO3-→NO2-)是由narG和napA基因编码的硝酸盐还原酶催化的, 而NO2-还原成NO的过程是由nirK或nirS编码的亚硝酸盐还原酶催化的, 最后一步N2O转化为N2的过程是由nosZ编码的氧化亚氮还原酶催化的[3, 9]。因此, amoA-AOB、amoA-AOA、narG、nirK、nirS和nosZ是目前研究较多的土壤氮转化功能基因, 通常用这些基因的拷贝数来表征土壤氨氧化和反硝化微生物的丰度[10—11]。有关氮添加对土壤氮转化功能基因丰度影响的研究较多, 但尚未有统一的结论。例如, 有研究表明氮添加可增加amoA-AOB的丰度, 而对amoA-AOA没有影响[2, 12]。然而, Carey等[7]通过meta分析得出, 氮添加对amoA-AOB和amoA-AOA丰度均有促进作用。Tang等[3]在森林生态系统的研究发现氮磷同时添加可显著增加amoA-AOA的丰度, 而降低了amoA-AOB的丰度。此外, 反硝化功能基因(narG、nirK、nirS和nosZ)丰度对氮、磷添加的响应也存在不一致的情况[3—4, 11]。这种响应的不一致性表明氨氧化和反硝化功能基因丰度对养分添加的响应会随着生态系统类型和施肥种类发生变化。同时氨氧化和反硝化功能基因丰度也表现出季节性变化[13—14], 这表明土壤氨氧化和反硝化功能基因丰度可能与环境因子(温度和湿度)有密切关系[15—17]。
相对于氮转化功能基因, 磷转化功能基因的研究较少。磷酸酶由碱性磷酸酶(ALP)和酸性磷酸酶(ACP)组成, 是催化磷酸酯(除了肌醇磷酸酯, 例如肌醇六磷酸盐)水解释放出磷酸盐的酶。前人研究表明, ALP来自土壤微生物, 而ACP主要由植物合成和分泌[18—19]。phoA、phoX和phoD是原核生物编码ALP基因, 这三个同源基因的分布随生态系统不同而变化。phoA、phoD和phoX广泛分布在水生生态系统, 而陆地生态系统中phoD基因的分布要高于pho A和phoX[20]。因此, phoD基因可作为土壤有机磷转化的标记基因[21]。Hu等[20]研究发现, 长期添加高剂量有机肥(秸秆和牛粪)可显著改变编码碱性磷酸酶基因的细菌群落多样性, phoD群落结构主要受土壤有机碳的调控, 而不是土壤磷含量。Chen等[22]研究发现, 长期添加磷肥增加了phoD基因的多样性, phoD基因丰度与土壤有效磷含量负相关, 而编码碱性磷酸酶基因的细菌群落结构与土壤pH和有效磷含量有关。目前关于phoD基因的研究主要集中在农田生态系统中, 且氮肥或氮磷肥同时添加对phoD基因丰度和多样性的影响相对较少, phoD基因对氮、磷添加的响应机理尚不清楚。
本研究对氮、磷添加后呼伦贝尔草甸草原不同生长时期土壤氨氧化(amoA-AOA和amoA-AOB)、反硝化(narG、nirK、nirS和nosZ)和磷转化(phoD)基因丰度进行分析, 探究土壤理化性质与氮、磷转化功能基因丰度的关系, 明确土壤氮、磷转化功能基因丰度及氮损失对氮、磷添加的响应规律, 为定向调控土壤氮、磷转化过程, 提高养分利用效率, 减少温室气体N2O排放提供科学依据。
1 材料与方法 1.1 试验地概况研究地点位于呼伦贝尔市海拉尔区特尼河9队(49°20′—49°26′N, 119°55′—120°9′E, 海拔628—649 m), 是大兴安岭西麓丘陵向内蒙古高原的过渡区。该地区属温带大陆性季风气候, 年均温-1.2 ℃, 1月最冷月平均气温-25.9 ℃, 7月最热月平均气温20.0℃, >10 ℃积温1780—1820℃, 无霜期90—115 d, 年均降水量354 mm, 全年降水的75%集中在6—9月份。2018—2020年总降水量分别为289.0 mm、279.0 mm、357.0 mm。草甸草原以羊草(Leymus chinensis)和贝加尔针茅(Stipa baicalensis)建群, 土壤为黑钙土或暗栗钙土, 土壤pH为6.50, 土层厚30—40 cm, 表层土壤(0—10 cm)有机碳含量51.0 g/kg, 全氮3.7 g/kg, 全磷0.5 g/kg。
1.2 试验设计2018年5月在试验区选取地势平坦、植被分布均匀的草地进行氮磷添加试验, 双因子裂区试验设计, 主区为氮添加, 副区为磷添加。设5个氮水平(N0:不施N;N1:1.55 g N m-2 a-1;N2:4.65 g N m-2 a-1;N3:13.95 g N m-2 a-1;N4:27.9 g N m-2 a-1), 3个磷水平(P0:不施P;P1:5.24 g P m-2 a-1;P2:10.48 g P m-2 a-1), 共15个处理, 4个区组, 总计60个小区。每个小区面积2 m×3 m, 主区间距2 m, 副区间距1 m。氮的添加量参考前人在内蒙古草地进行的氮添加梯度(0—30 g N m-2 a-1)[23], 1.55 g N m-2 a-1的添加量近似等于2000—2010年中国年平均氮沉降量[21], 13.95 g N m-2 a-1的添加量可达到内蒙古草地植物生长的氮饱和阈值[24]。磷的添加量也参考前人在内蒙古草地进行的磷添加梯度(0—13.9 g P m-2 a-1)[25], 本研究磷添加量能解除内蒙古草地植物生长磷限制[25]。氮肥使用硝酸铵(NH4NO3, 分析纯), 磷肥使用五氧化二磷(P2O5, 分析纯)。添加时间为2018—2020年, 每年6月6日进行氮磷添加。将肥料溶解在24 L水(相当于4 mm的降水)中, 以水溶液的形式用喷壶均匀喷施于试验小区, 不施肥的小区喷施等量的水。按照当地打草场的利用方式, 每年8月底对处理小区进行刈割处理, 留茬高度为8 cm。
1.3 土壤样品采集2020年5月下旬(施肥前, 5月28日)、7月和8月中旬(7月15日, 8月20日), 用直径5 cm的土钻在每个小区内随机取0—10 cm土样4个, 混合成1个土壤样品, 过2 mm筛后分成三份, 一份用于测定土壤含水量, 另一份储存于-80 ℃冰箱用于土壤DNA的提取, 剩余的土壤自然风干后测定土壤化学性质。
1.4 土壤理化性质测定土壤全碳(TC)和全氮(TN)用元素分析仪(Vario MAX CN; Elementar, Hanau, Germany)测定。
土壤全磷(TP)和速效磷(Olsen-P)采用钼锑抗比色法测定[26]。
土壤pH用酸度计(FE20-FiveEasyTM, Mettler Toledo, Germany)测定。
土壤无机氮含量的测定: 2020年5—8月测定各处理小区土壤无机氮含量。采用原位、非破坏性的离子交换膜(Ionics, Ringwood, NJ, USA)进行取样[27], 采用AA3流动分析仪(AutoAnalyser 3; Seal Analytical, Norderstedt, Germany)测定NH4+-N和NO3--N含量。NH4+-N和NO3--N含量的计算公式如下: [(浓度单位为μg N/mL)×(70 mL KCl)]/(50 cm2膜面积×放置天数)。
1.5 土壤DNA提取及实时荧光定量聚合酶链式反应分析(qRT-PCR)土壤DNA提取采用土壤基因组DNA提取试剂盒(DP336, 天根, 中国)。采用罗氏LightCycle 96的实时定量聚合酶链式反(PCR)仪对16S rRNA、phoD、amoA-AOB、amoA-AOA、narG、nirK、nirS和nosZ基因拷贝数进行分析。qRT-PCR分析采用荧光定量试剂盒SuperReal PreMix Plus (SYBR Green), 总反应体系为20 μL, 包括: 10 μL的2×SuperReal SYBR Green PreMix Plus, 0.5 μL上游引物(10 μM), 0.5 μL下游引物(10 μM), 5 μL的DNA模板和4 μL的ddH2O。所有基因引物合成序列信息和相应的PCR扩增程序参照之前的文献报道[12, 20]。
1.6 数据分析采用三因素方差分析检验氮添加(N), 磷添加(P)与不同取样时间(D)的主效应及其交互作用对土壤理化指标及氮、磷转化功能基因拷贝数的影响。采用裂区方差分析检验每个取样时间下氮、磷添加及其交互作用对土壤理化指标及氮、磷转化功能基因拷贝数的影响, 其中氮水平和磷水平作为固定因子, 区组为随机因子。采用邓肯(Duncan)多重比较(P<0.05)检验氮或磷添加水平间的差异, 方差分析均使用SPSS(version 19.0; IBM, Armonk, New York, USA)进行。采用冗余分析(RDA)评估不同取样时间下氮、磷转化功能基因拷贝数与土壤理化指标间的关系。数据作图采用GraphPad(version 8.0.1, GraphPad Software Inc, San Diego, CA, USA)进行。
2 结果与分析 2.1 氮磷添加对土壤理化性质的影响氮、磷添加、取样时间及其三者交互作用显著影响土壤NH4+-N和NO3--N含量(表 1, P<0.001), 氮添加显著增加5—8月份土壤NO3--N含量(表 2, P<0.01), 而磷添加显著降低了NO3--N含量(表 2, P<0.001), 7月份NO3--N含量最低。氮、磷添加交互作用显著影响土壤5月和8月的NH4+-N(表 2, P<0.01), 氮添加显著增加土壤NH4+-N含量(表 2, P<0.001), 高氮水平下添加磷可显著增加NH4+-N的含量(表 2, P<0.05)。氮添加显著增加7月份NH4+-N含量(表 2, P<0.001), 而磷添加对NH4+-N含量无显著影响(表 2, P>0.05)。磷添加显著增加了土壤Olsen-P含量(表 2, P<0.001), 高氮处理(N3和N4)显著降低了5月和8月土壤Olsen-P含量(表 2, P<0.05)。
| 土壤特性 Soil properties |
N | P | D | N×P | N×D | P×D | N×P×D |
| P | P | P | P | P | P | P | |
| NH4+-N | *** | *** | *** | *** | *** | *** | *** |
| NO3—N | *** | *** | *** | *** | *** | *** | *** |
| Olsen P | *** | *** | *** | 0.07 | 0.57 | ** | 0.94 |
| TC | 0.55 | 0.06 | 0.45 | 0.69 | 0.98 | 0.88 | 1.00 |
| TN | 0.06 | 0.24 | ** | 0.76 | 0.99 | 0.89 | 1.00 |
| TP | *** | *** | *** | 0.28 | 0.97 | 0.72 | 0.93 |
| TC: TN | 0.07 | 0.07 | *** | 0.86 | 0.94 | 0.90 | 0.96 |
| TC: TP | ** | *** | ** | 0.44 | 0.67 | 0.80 | 0.73 |
| TN: TP | *** | *** | ** | 0.29 | 0.70 | 0.75 | 0.78 |
| pH | *** | *** | *** | ** | * | 0.20 | 0.12 |
| SWC | *** | * | *** | 0.33 | *** | 0.60 | * |
| NH4+-N: 土壤铵态氮soil ammonium nitrogen;NO3--N: 土壤硝态氮soil nitrate;Olsen-P: 土壤速效磷soil available phosphorus;TC: 土壤总碳soil total carbon;TN: 土壤总氮soil total nitrogen;TP: 土壤全磷soil total phosphorus;TC: TN: 土壤全碳与全氮的比值the ratio of soil total carbon to soil total nitrogen;TC: TP: 土壤全碳与全磷的比值the ratio of soil total carbon to soil total phosphorus;TN: TP: 土壤全氮与全磷的比值the ratio of soil total nitrogen to soil total phosphorus;SWC: 土壤含水量soil water content;*, * *, * * *分别代表P<0.05, P<0.01, P<0.001 | |||||||
| 处理 Treatments |
NH4+-N/(μg cm-2 d-1) | NO3--N/(μg cm-2 d-1) | Olsen P/(mg/kg) | TC/(g/kg) | TN/(g/kg) | TP/(g/kg) | TC: TN | TC: TP | TN: TP | pH | SWC/% | |||||||||||||||||||||||||||||||||
| 5月 | 7月 | 8月 | 5月 | 7月 | 8月 | 5月 | 7月 | 8月 | 5月 | 7月 | 8月 | 5月 | 7月 | 8月 | 5月 | 7月 | 8月 | 5月 | 7月 | 8月 | 5月 | 7月 | 8月 | 5月 | 7月 | 8月 | 5月 | 7月 | 8月 | 5月 | 7月 | 8月 | ||||||||||||
| N0 | P0 | 0.04 | 0.01 | 0.02 | 0.16 | 0.05 | 0.35 | 4.11 | 3.64 | 4.85 | 44.19 | 46.51 | 45.54 | 3.97 | 3.99 | 4.10 | 0.58 | 0.58 | 0.61 | 11.15 | 11.66 | 11.14 | 76.49 | 80.69 | 76.18 | 6.86 | 6.92 | 6.83 | 6.46 | 6.50 | 6.30 | 15.53 | 9.99 | 28.17 | ||||||||||
| N0 | P1 | 0.04 | 0.01 | 0.03 | 0.13 | 0.05 | 0.28 | 25.50 | 27.73 | 32.29 | 47.21 | 46.52 | 47.28 | 4.20 | 3.98 | 4.23 | 0.66 | 0.67 | 0.73 | 11.24 | 11.70 | 11.19 | 72.16 | 69.29 | 65.25 | 6.42 | 5.92 | 5.83 | 6.30 | 6.39 | 6.25 | 16.38 | 10.59 | 28.62 | ||||||||||
| N0 | P2 | 0.04 | 0.02 | 0.02 | 0.12 | 0.05 | 0.29 | 48.16 | 46.20 | 59.90 | 46.64 | 47.34 | 47.15 | 4.14 | 4.00 | 4.19 | 0.76 | 0.74 | 0.83 | 11.27 | 11.83 | 11.25 | 61.78 | 63.81 | 56.82 | 5.48 | 5.40 | 5.05 | 6.20 | 6.28 | 6.09 | 17.80 | 10.33 | 27.46 | ||||||||||
| N1 | P0 | 0.05 | 0.01 | 0.02 | 0.17 | 0.06 | 0.43 | 2.60 | 5.82 | 5.83 | 42.37 | 44.83 | 42.84 | 3.85 | 3.85 | 3.89 | 0.53 | 0.53 | 0.53 | 11.01 | 11.65 | 11.01 | 79.55 | 84.88 | 81.50 | 7.23 | 7.29 | 7.40 | 6.36 | 6.49 | 6.37 | 15.65 | 10.17 | 26.70 | ||||||||||
| N1 | P1 | 0.05 | 0.02 | 0.03 | 0.15 | 0.05 | 0.36 | 22.82 | 27.83 | 32.92 | 43.57 | 45.12 | 43.78 | 3.89 | 3.85 | 3.91 | 0.63 | 0.65 | 0.69 | 11.19 | 11.72 | 11.22 | 69.43 | 69.88 | 64.98 | 6.21 | 5.96 | 5.78 | 6.32 | 6.18 | 6.35 | 15.69 | 9.75 | 26.02 | ||||||||||
| N1 | P2 | 0.05 | 0.01 | 0.03 | 0.12 | 0.05 | 0.36 | 50.01 | 54.76 | 69.81 | 44.60 | 44.81 | 44.75 | 4.01 | 3.86 | 4.00 | 0.76 | 0.72 | 0.79 | 11.15 | 11.63 | 11.21 | 58.79 | 62.90 | 56.44 | 5.28 | 5.42 | 5.04 | 6.28 | 6.29 | 6.10 | 15.06 | 9.95 | 28.77 | ||||||||||
| N2 | P0 | 0.05 | 0.02 | 0.03 | 0.15 | 0.06 | 0.85 | 1.89 | 4.17 | 3.81 | 44.80 | 43.94 | 45.76 | 4.06 | 3.80 | 4.12 | 0.54 | 0.58 | 0.55 | 11.04 | 11.59 | 11.12 | 83.18 | 76.25 | 84.37 | 7.54 | 6.59 | 7.58 | 6.33 | 6.36 | 6.34 | 14.31 | 9.72 | 28.59 | ||||||||||
| N2 | P1 | 0.04 | 0.01 | 0.03 | 0.11 | 0.05 | 0.64 | 19.36 | 29.16 | 29.27 | 43.64 | 43.55 | 44.20 | 3.92 | 3.74 | 4.01 | 0.58 | 0.61 | 0.61 | 11.14 | 11.65 | 11.03 | 75.64 | 72.49 | 72.46 | 6.79 | 6.23 | 6.57 | 6.29 | 6.31 | 6.39 | 13.41 | 9.12 | 27.14 | ||||||||||
| N2 | P2 | 0.04 | 0.02 | 0.06 | 0.11 | 0.05 | 0.37 | 44.08 | 48.78 | 55.30 | 43.88 | 42.64 | 43.75 | 3.92 | 3.66 | 3.91 | 0.64 | 0.67 | 0.77 | 11.20 | 11.68 | 11.20 | 69.42 | 63.61 | 57.07 | 6.19 | 5.46 | 5.10 | 6.16 | 6.12 | 6.13 | 13.84 | 9.43 | 27.14 | ||||||||||
| N3 | P0 | 0.08 | 0.08 | 0.29 | 0.16 | 0.09 | 3.52 | 1.89 | 4.34 | 4.18 | 45.84 | 46.53 | 45.48 | 4.14 | 4.04 | 4.12 | 0.50 | 0.52 | 0.58 | 11.09 | 11.54 | 11.05 | 92.45 | 89.18 | 79.53 | 8.35 | 7.73 | 7.19 | 6.21 | 6.26 | 6.08 | 13.65 | 9.72 | 27.11 | ||||||||||
| N3 | P1 | 0.05 | 0.07 | 0.39 | 0.13 | 0.07 | 1.95 | 16.19 | 24.46 | 23.74 | 45.10 | 45.25 | 45.36 | 4.05 | 3.84 | 4.06 | 0.62 | 0.60 | 0.64 | 11.15 | 11.80 | 11.17 | 73.03 | 75.75 | 72.13 | 6.55 | 6.43 | 6.44 | 6.19 | 6.21 | 6.14 | 12.96 | 9.29 | 28.68 | ||||||||||
| N3 | P2 | 0.08 | 0.05 | 0.42 | 0.12 | 0.06 | 1.30 | 35.31 | 47.64 | 54.98 | 44.14 | 44.39 | 43.57 | 3.93 | 3.84 | 3.92 | 0.63 | 0.63 | 0.72 | 11.24 | 11.57 | 11.16 | 70.11 | 71.21 | 60.75 | 6.23 | 6.15 | 5.45 | 6.11 | 6.21 | 6.15 | 12.68 | 9.46 | 26.30 | ||||||||||
| N4 | P0 | 0.14 | 0.18 | 0.97 | 0.59 | 0.15 | 7.26 | 3.78 | 3.80 | 4.18 | 44.07 | 45.36 | 44.95 | 4.07 | 3.92 | 4.14 | 0.55 | 0.49 | 0.56 | 10.83 | 11.59 | 10.89 | 80.30 | 92.38 | 81.12 | 7.42 | 7.99 | 7.45 | 6.03 | 6.11 | 5.74 | 14.48 | 9.76 | 26.90 | ||||||||||
| N4 | P1 | 0.25 | 0.20 | 1.35 | 0.24 | 0.10 | 5.39 | 17.53 | 23.77 | 30.30 | 46.12 | 47.16 | 45.93 | 4.10 | 4.14 | 4.21 | 0.60 | 0.62 | 0.69 | 11.26 | 11.39 | 10.93 | 76.49 | 77.05 | 66.96 | 6.80 | 6.77 | 6.12 | 6.11 | 6.02 | 5.74 | 13.03 | 9.88 | 27.83 | ||||||||||
| N4 | P2 | 0.29 | 0.20 | 1.77 | 0.22 | 0.10 | 4.94 | 40.36 | 47.89 | 49.03 | 45.12 | 45.77 | 45.74 | 4.06 | 3.98 | 4.11 | 0.68 | 0.72 | 0.70 | 11.10 | 11.50 | 11.14 | 66.38 | 63.93 | 65.69 | 5.97 | 5.56 | 5.90 | 6.10 | 5.94 | 6.02 | 12.38 | 9.33 | 25.59 | ||||||||||
| 因子Factor | P | P | P | P | P | P | P | P | P | P | P | P | P | P | P | P | P | P | P | P | P | P | P | P | P | P | P | P | P | P | P | P | P | |||||||||||
| N | *** | *** | *** | ** | *** | *** | ** | 0.49 | * | 0.15 | 0.15 | 0.13 | 0.26 | 0.08 | 0.13 | * | 0.10 | 0.10 | 0.45 | 0.19 | 0.25 | ** | * | 0.15 | ** | * | 0.06 | ** | ** | *** | *** | 0.10 | 0.38 | |||||||||||
| P | * | 0.98 | *** | *** | *** | *** | *** | *** | *** | 0.42 | 0.79 | 0.94 | 0.89 | 0.73 | 0.87 | *** | *** | *** | 0.07 | 0.71 | 0.06 | *** | *** | *** | *** | *** | *** | ** | ** | * | 0.26 | 0.44 | 0.49 | |||||||||||
| N×P | ** | 0.95 | *** | ** | *** | ** | 0.77 | 0.77 | * | 0.40 | 0.94 | 0.96 | 0.08 | 0.81 | 0.98 | 0.49 | 0.37 | 0.09 | 0.76 | 0.39 | 0.82 | 0.56 | 0.12 | 0.82 | 0.58 | 0.13 | 0.71 | * | 0.41 | *** | ** | 0.20 | 0.08 | |||||||||||
| N0:0 g N m-2 a-1;N1:1.55 g N m-2 a-1;N2:4.65 g N m-2 a-1;N3:13.95 g N m-2 a-1;N4:27.9 g N m-2 a-1;P0:0 g P2O5 m-2 a-1;P1:12 g P2O5 m-2 a-1;P2:24 g P2O5 m-2 a-1 | ||||||||||||||||||||||||||||||||||||||||||||
氮、磷添加对土壤TC、TN含量和TC: TN比的影响不显著(表 1, P>0.05), 显著影响土壤TP含量、TC: TP和TN: TP(表 1, P<0.01)。磷添加显著增加了TP含量(表 2, P<0.001), 降低了土壤TC: TP比和TN: TP比(表 2, P<0.001), 氮添加降低TP含量(表 2, P<0.05), 提高TC: TP比和TN: TP比(表 2, P<0.05)。
土壤pH受到氮、磷添加交互作用的显著影响(表 1, P<0.01), 氮、磷单独添加均可降低土壤pH(表 2, P<0.01), 高氮水平下添加磷对土壤pH的降低不明显。土壤水含量(SWC)受氮、磷添加和取样时间交互作用的显著影响(表 1, P<0.05), 氮添加可显著降低5月份SWC, 高氮水平下, 磷添加可降低SWC(表 2, P<0.01)。7月份SWC低于5月和8月份。
2.2 氮磷添加对土壤细菌和磷酸酶基因丰度的影响氮、磷添加及其交互作用对土壤细菌丰度(16S rRNA)的影响不显著(表 3, P>0.05), 但不同取样时间16S rRNA基因丰度差异显著(表 3, P<0.05), 5月份16S rRNA基因丰度整体高于7、8月份(图 1)。土壤磷酸酶基因(phoD)不受氮、磷添加及取样时间的影响(表 3, P>0.05)。
| 功能基因 Functional genes |
N | P | D | N×P | N×D | P×D | N×P×D |
| P | P | P | P | P | P | P | |
| 16S rRNA | 0.92 | 0.81 | * | 0.90 | 0.52 | 0.84 | 0.99 |
| phoD | 0.88 | 0.76 | 0.07 | 0.92 | 0.47 | 0.99 | 1.00 |
| amoA-AOA | 0.29 | 0.06 | *** | 0.34 | 0.17 | 0.68 | 0.95 |
| amoA-AOB | *** | * | 0.96 | * | *** | 0.11 | 0.20 |
| narG | * | 0.92 | *** | 0.37 | 0.89 | 0.91 | 0.82 |
| nirS | 0.05 | 0.79 | 0.06 | 0.11 | 0.20 | 0.53 | 0.40 |
| nirK | 0.22 | 0.68 | *** | 0.85 | 0.49 | 0.72 | 0.99 |
| nosZ | 0.85 | 0.94 | 0.74 | 0.25 | 0.08 | 0.95 | 0.94 |
| AOA: AOB | *** | 0.79 | * | 0.11 | *** | 0.20 | * |
| amoA: narG | *** | *** | *** | *** | 0.14 | 0.07 | * |
| nirK: nirS | ** | 0.70 | *** | ** | ** | 0.72 | 0.07 |
| nirK+nirS | 0.22 | 0.67 | *** | 0.85 | 0.49 | 0.72 | 0.99 |

|
| 图 1 氮磷添加对不同取样时间土壤细菌和磷酸酶基因丰度的影响 Fig. 1 Effects of nitrogen and phosphorus additions on the abundances of bacteria and phosphatase gene in different sampling dates N0:0 g N m-2 a-1;N1:1.55 g N m-2 a-1;N2:4.65 g N m-2 a-1;N3:13.95 g N m-2 a-1;N4:27.9 g N m-2 a-1;P0:0 g P2O5 m-2 a-1;P1:12 g P2O5 m-2 a-1;P2:24 g P2O5 m-2 a-1; 不同大写字母表示氮效应显著;不同小写字母表示磷效应显著; ns, *, * *, ***分别代表P>0.05, P<0.05, P<0.01, P<0.001 |
土壤amoA-AOA和amoA-AOB基因表达丰度对氮、磷添加及取样时间的响应不同, 不同取样时间下amoA-AOA基因拷贝数均未受到氮、磷添加及其交互作用显著影响(图 2, P>0.05), 但5月份的amoA-AOA基因拷贝数低于7月份和8月份。amoA-AOB基因拷贝数不受取样时间的显著影响(表 3, P>0.05)。氮添加可显著增加5—8月份amoA-AOB基因拷贝数(P<0.05), 且8月份高氮处理(N3和N4)下的作用效果更为显著(P<0.001)(图 2)。5月份N2和N3处理下, 磷添加(P2)可显著降低amoA-AOB基因拷贝数。氮、磷添加对8月份amoA-AOB基因拷贝数的影响存在交互作用, 高氮水平下, 磷添加可降低amoA-AOB基因拷贝数(图 2,P<0.05)。

|
| 图 2 氮磷添加对不同取样时间土壤氨氧化古菌和氨氧化细菌amoA基因丰度及比值的影响 Fig. 2 Effects of nitrogen and phosphorus additions on amoA abundance of ammonia oxidizing archaea and ammonia oxidizing bacteria and their ratios in different sampling dates AOA: AOB: 土壤氨氧化古菌与氨氧化细菌的比值the ratio of amoA-AOA to amoA-AOB |
土壤amoA-AOA和amoA-AOB基因表达丰度的比值(AOA: AOB)受到氮、磷添加及取样时间三者交互作用的显著影响(表 3, P<0.05)。氮添加对5—8月份AOA: AOB均有显著影响(图 2, P<0.01), 高氮处理(N4)可显著降低AOA: AOB(图 2, P<0.05)。氮、磷添加对7月和8月AOA: AOB的影响具有交互作用, 低氮处理下, 磷添加可降低AOA: AOB, 而高氮处理下添加磷可提高AOA: AOB(图 2, P<0.05)。磷添加对5月份的AOA: AOB无显著影响(图 2, P>0.05), 而氮添加对5月份AOA: AOB的影响呈先升高后降低的趋势。
2.4 氮磷添加对土壤反硝化功能基因丰度的影响取样时间显著影响反硝化基因nirK拷贝数(表 3, P<0.001), 8月份nirK拷贝数高于5月和7月份, 且高氮(N3)处理的nirK拷贝数显著高于N0处理(图 3, P<0.05), 氮、磷添加对5月和7月份nirK拷贝数的影响不显著(图 3, P>0.05)。nirS拷贝数不受氮、磷添加及取样时间的影响(图 3, P>0.05)。氮、磷添加及取样时间对7月和8月份nirK: nirS的影响也不显著(图 3, P>0.05), 但氮、磷交互作用对5月份nirK: nirS的影响显著, 高氮处理(N3)可显著降低nirK: nirS, 单独磷添加可显著增加nirK: nirS, 而N2处理下磷添加可显著降低nirK: nirS(图 3, P<0.05)。氮、磷添加对5—8月份narG拷贝数无显著影响(图 4, P>0.05), 氮添加对5月和7月份nosZ拷贝数的影响不显著, N2和N3处理可显著增加8月份nosZ的拷贝数(图 4, P<0.05)。

|
| 图 3 氮磷添加对不同取样时间土壤反硝化功能基因nirK、nirS丰度及其比值的影响 Fig. 3 Effects of nitrogen and phosphorus additions on the abundances of nirK and nirS involved in denitrification and their ratios in different sampling dates |
|
| 图 4 氮磷添加对取样时间土壤反硝化功能基因narG、nosZ丰度的影响 Fig. 4 Effects of nitrogen and phosphorus additions on the abundances of narG and nosZ involved in denitrification in different sampling dates |
土壤硝酸盐渗漏潜势(amoA: narG)受到氮、磷添加、取样时间及其交互作用的影响(表 3, P<0.05)。氮添加可增加5—8月份amoA: narG, 5月和7月amoA: narG受到氮磷添加交互作用的影响(图 5, P<0.05)。5月份, N2、N3和N4处理下添加磷可降低amoA: narG;7月份, N2处理下添加磷可降低amoA: narG(图 5, P<0.05)。不同时间土壤气态氮损失潜势(nirK+nirS)有差异(表 3, P<0.001), 8月份nirK+nirS高于5月和7月份, 且高氮(N3)处理的nirK+nirS显著高于N0处理(图 5, P<0.05), 氮、磷添加对5月和7月份的nirK+nirS影响不显著(图 5, P>0.05)。

|
| 图 5 氮磷添加对不同取样时间土壤氮损失潜势的影响 Fig. 5 Effects of nitrogen and phosphorus additions on the potential of soil N loss in different sampling dates |
为探究氮、磷添加下氨氧化和反硝化基因丰度与土壤理化性质的关系, 对不同取样时间(5月、7月和8月)土壤理化性质与氨氧化和反硝化基因丰度分别进行了RDA分析, 并将三个月份的数据作为重复值进行了综合分析(5—8月)。结果表明, 5月、7月和8月份土壤理化性质对氮转化功能基因丰度变化的解释度分别为30.4%、33.2%和61.1%(图 6), 其中NH4+-N和NO3--N与amoA-AOB基因丰度和amoA: narG正相关。5月和7月份, NH4+-N与amoA-AOB基因丰度的相关性最大(图 6), 此外, Olsen-P与AOA: AOB比正相关(图 6)。NH4+-N、NO3--N、SWC、pH和TC: TN比的解释度相对较大, NH4+-N和NO3--N与amoA-AOB和amoA: narG正相关, pH与AOA: AOB比正相关, SWC与nirK+nirS具有明显的正相关关系(图 6)。
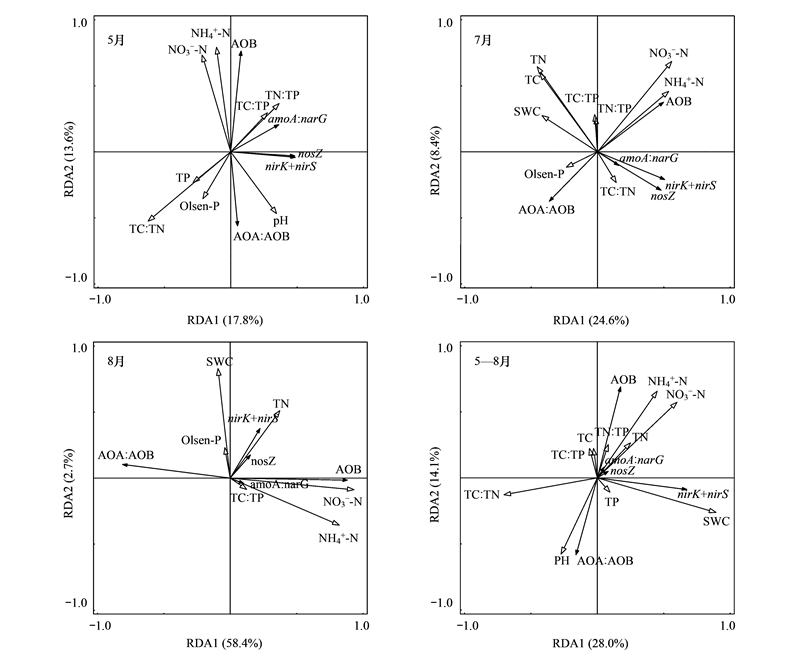
|
| 图 6 冗余分析(RDA) 土壤理化性质与氨氧化和反硝化基因丰度的关系 Fig. 6 The redundancy analysis (RDA) showing the relationship between the soil properties and the abundance of genes involved in ammonia oxidation and denitrification NH4+-N: 土壤铵态氮soil ammonium nitrogen;NO3--N: 土壤硝态氮soil nitrate;Olsen-P: 土壤速效磷soil available phosphorus;TC: 土壤总碳soil total carbon;TN: 土壤总氮soil total nitrogen;TP: 土壤全磷soil total phosphorus;TC: TN: 土壤全碳与全氮的比值the ratio of soil total carbon to soil total nitrogen;TC: TP: 土壤全碳与全磷的比值the ratio of soil total carbon to soil total phosphorus;TN: TP: 土壤全氮与全磷的比值the ratio of soil total nitrogen to soil total phosphorus;SWC: 土壤含水量soil water content |
本研究结果表明呼伦贝尔草甸草原土壤amoA-AOA基因丰度高于amoA-AOB基因丰度(AOA: AOB >1), 氮添加可显著增加5—8月份amoA-AOB基因拷贝数, 而对amoA-AOA基因拷贝数没有影响, 进而降低AOA: AOB比(图 2), 增强amoA-AOB的重要性。Sterngren等[28]认为, 氨氧化古菌是氮缺乏环境下硝化作用的主要驱动者, 而氮输入可增加氨氧化细菌的丰度及活性, 增强氨氧化细菌调控土壤总硝化速率的相对重要性, 与本研究结果相似, 说明土壤氨氧化细菌更青睐于高的土壤无机氮含量, 而氨氧化古菌对土壤氮可利用性的响应较小[29—30]。本研究中RDA分析结果也表明, 土壤NH4+-N含量对amoA-AOB基因拷贝数变异的贡献度最大, 且成正相关关系(图 6)。除土壤无机氮含量外, 土壤amoA-AOB基因丰度还受到pH的影响, 这与长期氮添加可降低土壤pH有关[3], 本研究中pH与amoA-AOB基因拷贝数呈负相关关系(图 6)。但也有研究表明, 氨氧化细菌更喜欢中性偏碱性的土壤环境, 而氨氧化古菌更喜欢酸性土壤环境[31—32]。此外, Nicol等[33]发现当土壤pH从4.9升高到5.9时, amoA-AOA和amoA-AOB基因丰度并未发生改变。因此, 氮添加对amoA-AOB基因丰度的影响主要与土壤无机氮含量有关, 土壤pH可能与氮添加引起的amoA-AOB基因丰度的变异并没有直接的相关关系。
土壤有效磷含量也对氨氧化微生物的丰度有一定影响。在酸性森林土壤环境中, 高的土壤有效磷含量可促进amoA-AOA基因丰度[3], 是磷添加通过促进氮矿化可为微生物提供更多的能量, 进而促进氨氧化过程[34]。然而磷添加后高的土壤有效磷含量不利于氨氧化细菌的生长, 抑制amoA-AOB基因丰度[3]。在高寒草甸氮添加的基础上, 添加磷可降低有效氮磷比值抑制amoA-AOA基因丰度, 而对amoA-AOB基因丰度无显著影响[2]。因此, 土壤磷的有效性对氨氧化基因丰度的影响是复杂的, 不同的生态系统土壤amoA-AOA和amoA-AOB基因丰度对有效磷含量的响应不同。本研究结果表明, 高氮水平下, 磷添加可降低amoA-AOB基因丰度, 而对amoA-AOA基因丰度没有显著影响(图 2), RDA结果表明, Olsen-P含量与amoA-AOB基因丰度呈负相关关系(图 6)。本研究发现高氮处理下磷添加可降低土壤NO3--N含量, 增加土壤NH4+-N含量, 这与磷添加抑制了amoA-AOB基因丰度, 减缓了氨氧化进程有关。
nirK或nirS编码的亚硝酸盐还原酶催化由亚硝酸盐还原成NO的过程, 是土壤中气态氮损失和N2O排放的重要途径[9]。不同的生态系统nirK和nirS基因的相对丰度不同, 一些农田生态系统中, nirS基因丰度显著高于nirK基因丰度, 亚硝酸盐还原过程主要是由nirS型细菌驱动[35—36]。然而, 本研究中nirK基因丰度显著高于nirS基因丰度(nirK: nirS >1), 这说明nirK型细菌是该生态系统亚硝酸盐还原过程的主要驱动者, 其他草原生态系统中也发现nirK基因丰度高于nirS基因丰度[2, 11]。但nirK和nirS基因丰度对氮添加的响应也存在不一致性[2—3, 35]。本研究发现nirS基因丰度比较稳定, 不受氮添加和取样时间的影响, nirK的丰度对氮添加的响应受到取样时间的影响, 氮添加只有在8月份对nirK基因丰度具有显著的促进作用, 且8月份nirK的丰度显著高于5和7月。Azziz等[37]研究表明, 植物生长时期是nirS和nirK基因丰度的主要影响因素。在冬小麦田的研究中也发现, nirS和nirK基因丰度在开花期对氮磷添加的响应更明显, 而在其他生长时期氮磷添加对nirS和nirK基因丰度的影响不显著[4]。土壤反硝化细菌受到许多环境因子的影响, 比如碳的可利用性、土壤湿度和pH等[38]。Tang等[3]认为矿质氮含量的增加引起的土壤酸化和盐分离子的富集可能是氮添加导致nirS和nirK基因丰度降低的原因。本研究RDA的结果表明, nirK+nirS基因丰度与SWC正相关, 与pH负相关(图 6)。同时, 8月份SWC和nirK的丰度最大, 这说明在土壤湿度高的情况下可促进土壤反硝化过程。
3.2 氮、磷添加对土壤氮损失潜势的影响土壤硝化和反硝化基因丰度也可以表征土壤硝酸盐淋溶损失和气态氮损失潜势。amoA: narG比值的高低可以表征硝酸盐淋溶损失潜势的大小[4]。前人研究发现, 氮和磷同时添加增加amoA: narG, 会提高硝酸盐淋溶损失潜势[3]。本研究也发现氮添加可提高amoA: narG, 增加硝酸盐淋溶损失潜势。相反, 高氮处理下添加磷可降低土壤氨氧化基因amoA-AOB的丰度, 减小amoA: narG, 进而降低硝酸盐淋溶损失潜势。这与高氮处理下添加磷可降低土壤NO3--N含量的结果相吻合, 但是土壤NO3--N含量的减低也有可能是高氮处理下添加磷增加了植物生物量, 促进了植物对NO3--N的吸收[39]。亚硝酸盐还原酶基因nirK或nirS催化亚硝酸盐转化为NO和气态氮的产生[40]。因此, nirK+nirS的值可表示气态氮损失潜势的大小, 草地生态系统N2O的排放与土壤矿质氮的有效性密切相关[41—42]。本研究发现, nirK+nirS与土壤NH4+-N和NO3--N含量虽呈正相关关系, 但相关性较弱, 而与SWC的相关性最强(图 6), 这说明土壤气态氮损失并非完全依赖于土壤有效氮含量, 主要受土壤湿度的调控。
3.3 土壤磷转化phoD基因丰度对氮、磷添加的响应长期施磷肥(28年)的玉米田, 土壤磷的有效性与碱性磷酸单酯酶活性、phoD基因丰度呈负相关关系, 即碱性磷酸单酯酶活性受土壤有效磷的调控[22]。因此, 在缺磷时碱性磷酸单酯酶活性和phoD基因丰度会增加[43—44]。然而, Ikoyi等[45]通过短期温室试验发现, phoD基因丰度与土壤有效磷之间没有显著的相关关系。本研究发现, 土壤有效磷含量的变化并没有导致phoD基因丰度的改变, phoD基因丰度不受氮、磷添加的影响。这种不一致性可能与这些研究中施磷的时间和数量有关, 长期施肥对土壤微生物群落结构的影响更为显著[46]。此外, Hu等[20]通过长期(10年)的有机肥处理发现, 土壤中phoD基因丰度明显增加, 然而, phoD与16S rRNA基因丰度的比值却在所有处理中无显著差异, 这表明长期施用有机肥促进了整个土壤细菌的生长, 而不仅仅是phoD型细菌的生长。本研究中氮、磷添加对16S rRNA基因的丰度也没有显著影响, 这说明短期的氮、磷添加试验并不会引起呼伦贝尔草甸草原土壤phoD与16S rRNA基因丰度的改变。
4 结论本研究发现, 呼伦贝尔草甸草原土壤细菌16S rRNA、磷酸酶基因phoD、氨氧化古菌amoA-AOA、反硝化基因nirS和narG丰度相对比较稳定, 而反硝化基因nirK和nosZ丰度对氮添加的响应受到生长季节的调控, 且与土壤含水量的变化紧密相关。土壤湿度较大时, 氮添加可增加nirK丰度, 提高土壤气态氮损失潜势。土壤氨氧化细菌amoA-AOB丰度受氮磷添加的共同调控, 氮添加通过增加amoA-AOB基因丰度增强了土壤硝酸盐淋溶损失潜势, 而高氮处理下添加磷可减小硝酸盐淋溶损失潜势。因此, 磷添加对提高土壤氮的利用效率具有重要作用, 但有关氮磷添加协同调控土壤氮损失的机制还需要深入探究。
| [1] |
Marklein A R, Houlton B Z. Nitrogen inputs accelerate phosphorus cycling rates across a wide variety of terrestrial ecosystems. The New Phytologist, 2012, 193(3): 696-704. DOI:10.1111/j.1469-8137.2011.03967.x |
| [2] |
Ma W B, Jiang S J, Assemien F, Qin M S, Ma B B, Xie Z, Liu Y J, Feng H Y, Du G Z, Ma X J, le Roux X. Response of microbial functional groups involved in soil N cycle to N, P and NP fertilization in Tibetan alpine meadows. Soil Biology and Biochemistry, 2016, 101: 195-206. DOI:10.1016/j.soilbio.2016.07.023 |
| [3] |
Tang Y Q, Zhang X Y, Li D D, Wang H M, Chen F S, Fu X L, Fang X M, Sun X M, Yu G R. Impacts of nitrogen and phosphorus additions on the abundance and community structure of ammonia oxidizers and denitrifying bacteria in Chinese fir plantations. Soil Biology and Biochemistry, 2016, 103: 284-293. DOI:10.1016/j.soilbio.2016.09.001 |
| [4] |
Wang Y, Ji H F, Wang R, Guo S L. Responses of nitrification and denitrification to nitrogen and phosphorus fertilization: does the intrinsic soil fertility matter?. Plant and Soil, 2019, 440(1/2): 443-456. |
| [5] |
Yang Y F, Wu L W, Lin Q Y, Yuan M T, Xu D P, Yu H, Hu Y G, Duan J C, Li X Z, He Z L, Xue K, van Nostrand J, Wang S P, Zhou J Z. Responses of the functional structure of soil microbial community to livestock grazing in the Tibetan alpine grassland. Global Change Biology, 2013, 19(2): 637-648. DOI:10.1111/gcb.12065 |
| [6] |
郑燕, 侯海军, 秦红灵, 朱亦君, 魏文学. 施氮对水稻土N2O释放及反硝化功能基因(narG/nosZ)丰度的影响. 生态学报, 2012, 32(11): 3386-3393. |
| [7] |
Carey C J, Dove N C, Beman J M, Hart S C, Aronson E L. Meta-analysis reveals ammonia-oxidizing bacteria respond more strongly to nitrogen addition than ammonia-oxidizing Archaea. Soil Biology and Biochemistry, 2016, 99: 158-166. DOI:10.1016/j.soilbio.2016.05.014 |
| [8] |
Hu H W, Chen D L, He J Z. Microbial regulation of terrestrial nitrous oxide formation: understanding the biological pathways for prediction of emission rates. FEMS Microbiology Reviews, 2015, 39(5): 729-749. DOI:10.1093/femsre/fuv021 |
| [9] |
Zumft W G. Cell biology and molecular basis of denitrification. Microbiology and Molecular Biology Reviews: MMBR, 1997, 61(4): 533-616. |
| [10] |
Cui Y X, Zhang Y L, Duan C J, Wang X, Zhang X C, Ju W L, Chen H S, Yue S C, Wang Y Q, Li S Q, Fang L C. Ecoenzymatic stoichiometry reveals microbial phosphorus limitation decreases the nitrogen cycling potential of soils in semi-arid agricultural ecosystems. Soil and Tillage Research, 2020, 197: 104463. DOI:10.1016/j.still.2019.104463 |
| [11] |
Che R X, Qin J L, Tahmasbian I, Wang F, Zhou S T, Xu Z H, Cui X Y. Litter amendment rather than phosphorus can dramatically change inorganic nitrogen pools in a degraded grassland soil by affecting nitrogen-cycling microbes. Soil Biology and Biochemistry, 2018, 120: 145-152. DOI:10.1016/j.soilbio.2018.02.006 |
| [12] |
Pereg L, Morugán-Coronado A, McMillan M, García-Orenes F. Restoration of nitrogen cycling community in grapevine soil by a decade of organic fertilization. Soil and Tillage Research, 2018, 179: 11-19. DOI:10.1016/j.still.2018.01.007 |
| [13] |
Isobe K, Oka H, Watanabe T, Tateno R, Urakawa R, Liang C, Senoo K, Shibata H. High soil microbial activity in the winter season enhances nitrogen cycling in a cool-temperate deciduous forest. Soil Biology and Biochemistry, 2018, 124: 90-100. DOI:10.1016/j.soilbio.2018.05.028 |
| [14] |
Jung J, Yeom J, Han J, Kim J, Park W. Seasonal changes in nitrogen-cycle gene abundances and in bacterial communities in acidic forest soils. Journal of Microbiology: Seoul, Korea, 2012, 50(3): 365-373. |
| [15] |
Chen J, Nie Y X, Liu W, Wang Z F, Shen W J. Ammonia-oxidizing Archaea are more resistant than denitrifiers to seasonal precipitation changes in an acidic subtropical forest soil. Frontiers in Microbiology, 2017, 8: 1384. DOI:10.3389/fmicb.2017.01384 |
| [16] |
Szukics U, Abell G C J, Hödl V, Mitter B, Sessitsch A, Hackl E, Zechmeister-Boltenstern S. Nitrifiers and denitrifiers respond rapidly to changed moisture and increasing temperature in a pristine forest soil. FEMS Microbiology Ecology, 2010, 72(3): 395-406. DOI:10.1111/j.1574-6941.2010.00853.x |
| [17] |
高思齐, 宋艳宇, 宋长春, 马秀艳, 蒋磊. 增温和外源碳输入对泥炭地土壤碳氮循环关键微生物功能基因丰度的影响. 生态学报, 2020, 40(13): 4617-4627. |
| [18] |
Chen X D, Jiang N, Condron L M, Dunfield K E, Chen Z H, Wang J K, Chen L J. Soil alkaline phosphatase activity and bacterial phoD gene abundance and diversity under long-term nitrogen and manure inputs. Geoderma, 2019, 349: 36-44. DOI:10.1016/j.geoderma.2019.04.039 |
| [19] |
Krämer S. Acid and alkaline phosphatase dynamics and their relationship to soil microclimate in a semiarid woodland. Soil Biology and Biochemistry, 2000, 32(2): 179-188. DOI:10.1016/S0038-0717(99)00140-6 |
| [20] |
Hu Y J, Xia Y H, Sun Q, Liu K P, Chen X B, Ge T D, Zhu B L, Zhu Z K, Zhang Z H, Su Y R. Effects of long-term fertilization on phoD-harboring bacterial community in Karst soils. Science of the Total Environment, 2018, 628/629: 53-63. DOI:10.1016/j.scitotenv.2018.01.314 |
| [21] |
Dai Z, Liu G, Chen H, Chen C, Wang J, Ai S, Wei D, Li D, Ma B, Tang C, Brookes P C, Xu J. Long-term nutrient inputs shift soil microbial functional profiles of phosphorus cycling in diverse agroecosystems. The ISME Journal, 2020, 14(3): 757-770. DOI:10.1038/s41396-019-0567-9 |
| [22] |
Chen X D, Jiang N, Condron L M, Dunfield K E, Chen Z H, Wang J K, Chen L J. Impact of long-term phosphorus fertilizer inputs on bacterial phoD gene community in a maize field, Northeast China. Science of the Total Environment, 2019, 669: 1011-1018. DOI:10.1016/j.scitotenv.2019.03.172 |
| [23] |
Bai Y F, Wu J G, Clark C M, Naeem S, Pan Q M, Huang J H, Zhang L X, Han X G. Tradeoffs and thresholds in the effects of nitrogen addition on biodiversity and ecosystem functioning: evidence from Inner Mongolia Grasslands. Global Change Biology, 2010, 16(1): 358-372. DOI:10.1111/j.1365-2486.2009.01950.x |
| [24] |
Liu X, Zhang Y, Han W, Tang A, Shen J, Cui Z, Vitousek P, Erisman J W, Goulding K, Christie P, Fangmeier A, Zhang F. Enhanced nitrogen deposition over China. Nature, 2013, 494(7438): 459-462. DOI:10.1038/nature11917 |
| [25] |
Zhan S X, Wang Y, Zhu Z C, Li W H, Bai Y F. Nitrogen enrichment alters plant N: P stoichiometry and intensifies phosphorus limitation in a steppe ecosystem. Environmental and Experimental Botany, 2017, 134: 21-32. DOI:10.1016/j.envexpbot.2016.10.014 |
| [26] |
鲍士旦. 土壤农化分析 (3版). 北京: 中国农业出版社, 2000.
|
| [27] |
Liu C, Wang L, Song X X, Chang Q, Frank D A, Wang D L, Li J, Lin H J, Du F Y. Towards a mechanistic understanding of the effect that different species of large grazers have on grassland soil N availability. Journal of Ecology, 2018, 106(1): 357-366. DOI:10.1111/1365-2745.12809 |
| [28] |
Sterngren A E, Hallin S, Bengtson P. Archaeal ammonia oxidizers dominate in numbers, but bacteria drive gross nitrification in N-amended grassland soil. Frontiers in Microbiology, 2015, 6: 1350. |
| [29] |
Simonin M, le Roux X, Poly F, Lerondelle C, Hungate B A, Nunan N, Niboyet A. Coupling between and among ammonia oxidizers and nitrite oxidizers in grassland mesocosms submitted to elevated CO2 and nitrogen supply. Microbial Ecology, 2015, 70(3): 809-818. DOI:10.1007/s00248-015-0604-9 |
| [30] |
Chen Y L, Hu H W, Han H Y, Du Y, Wan S Q, Xu Z W, Chen B D. Abundance and community structure of ammonia-oxidizing Archaea and Bacteria in response to fertilization and mowing in a temperate steppe in Inner Mongolia. FEMS Microbiology Ecology, 2014, 89(1): 67-79. DOI:10.1111/1574-6941.12336 |
| [31] |
Prosser J I, Nicol G W. Archaeal and bacterial ammonia-oxidisers in soil: the quest for niche specialisation and differentiation. Trends in Microbiology, 2012, 20(11): 523-531. DOI:10.1016/j.tim.2012.08.001 |
| [32] |
Yao H Y, Gao Y M, Nicol G W, Campbell C D, Prosser J I, Zhang L M, Han W Y, Singh B K. Links between ammonia oxidizer community structure, abundance, and nitrification potential in acidic soils. Applied and Environmental Microbiology, 2011, 77(13): 4618-4625. DOI:10.1128/AEM.00136-11 |
| [33] |
Nicol G W, Leininger S, Schleper C, Prosser J I. The influence of soil pH on the diversity, abundance and transcriptional activity of ammonia oxidizing Archaea and bacteria. Environmental Microbiology, 2008, 10(11): 2966-2978. DOI:10.1111/j.1462-2920.2008.01701.x |
| [34] |
Bauhus J, Khanna P K. Carbon and nitrogen turnover in two acid forest soils of southeast Australia as affected by phosphorus addition and drying and rewetting cycles. Biology and Fertility of Soils, 1994, 17(3): 212-218. DOI:10.1007/BF00336325 |
| [35] |
Yang Y D, Hu Y G, Wang Z M, Zeng Z H. Variations of the nirS-, nirK-, and nosZ-denitrifying bacterial communities in a northern Chinese soil as affected by different long-term irrigation regimes. Environmental Science and Pollution Research International, 2018, 25(14): 14057-14067. DOI:10.1007/s11356-018-1548-7 |
| [36] |
Yin C, Fan F L, Song A L, Cui P Y, Li T Q, Liang Y C. Denitrification potential under different fertilization regimes is closely coupled with changes in the denitrifying community in a black soil. Applied Microbiology and Biotechnology, 2015, 99(13): 5719-5729. DOI:10.1007/s00253-015-6461-0 |
| [37] |
Azziz G, Monza J, Etchebehere C, Irisarri P. nirS- and nirK-type denitrifier communities are differentially affected by soil type, rice cultivar and water management. European Journal of Soil Biology, 2017, 78: 20-28. DOI:10.1016/j.ejsobi.2016.11.003 |
| [38] |
Wallenstein M D, Myrold D D, Firestone M, Voytek M. Environmental controls on denitrifying communities and denitrification rates: insights from molecular methods. Ecological Applications, 2006, 16(6): 2143-2152. DOI:10.1890/1051-0761(2006)016[2143:ECODCA]2.0.CO;2 |
| [39] |
Jiang J, Wang Y P, Yang Y H, Yu M X, Wang C, Yan J H. Interactive effects of nitrogen and phosphorus additions on plant growth vary with ecosystem type. Plant and Soil, 2019, 440(1/2): 523-537. |
| [40] |
Levy-Booth D J, Prescott C E, Grayston S J. Microbial functional genes involved in nitrogen fixation, nitrification and denitrification in forest ecosystems. Soil Biology and Biochemistry, 2014, 75: 11-25. DOI:10.1016/j.soilbio.2014.03.021 |
| [41] |
van Kessel C, Venterea R, Six J, Adviento-Borbe M A, Linquist B, van Groenigen K J. Climate, duration, and N placement determine N2O emissions in reduced tillage systems: a meta-analysis. Global Change Biology, 2013, 19(1): 33-44. DOI:10.1111/j.1365-2486.2012.02779.x |
| [42] |
Geng F Z, Li K H, Liu X J, Gong Y M, Yue P, Li Y G, Han W X. Long-term effects of N deposition on N2O emission in an alpine grassland of Central Asia. CATENA, 2019, 182: 104100. DOI:10.1016/j.catena.2019.104100 |
| [43] |
Randall K, Brennan F, Clipson N, Creamer R, Griffiths B, Storey S, Doyle E. Soil bacterial community structure and functional responses across a long-term mineral phosphorus (Pi) fertilisation gradient differ in grazed and cut grasslands. Applied Soil Ecology, 2019, 138: 134-143. DOI:10.1016/j.apsoil.2019.02.002 |
| [44] |
Zhang G N, Chen Z H, Zhang A M, Chen L J, Wu Z J, Ma X Z. Phosphorus composition and phosphatase activities in soils affected by long-term application of pig manure and inorganic fertilizers. Communications in Soil Science and Plant Analysis, 2014, 45(14): 1866-1876. DOI:10.1080/00103624.2014.909831 |
| [45] |
Ikoyi I, Fowler A, Schmalenberger A. One-time phosphate fertilizer application to grassland columns modifies the soil microbiota and limits its role in ecosystem services. Science of the Total Environment, 2018, 630: 849-858. DOI:10.1016/j.scitotenv.2018.02.263 |
| [46] |
Liang Y T, Wu L Y, Clark I M, Xue K, Yang Y F, van Nostrand J D, Deng Y, He Z L, McGrath S, Storkey J, Hirsch P R, Sun B, Zhou J Z. Over 150 years of long-term fertilization alters spatial scaling of microbial biodiversity. mBio, 2015, 6(2): e00240-e00215. |
 2023, Vol. 43
2023, Vol. 43