文章信息
- 陈晓宇, 姚蒙, 李晟
- 山地景观遗传学研究文献综述
- 生态学报. 2022, 42(7): 3033-3043
- http://dx.doi.org/10.5846/stxb202106191631
-
文章历史
- 收稿日期: 2021-06-19
- 网络出版日期: 2021-12-15
2. 北京大学生态研究中心, 北京大学城市与环境学院, 北京 100871
随着人类土地利用等活动对环境的加速改造, 全球自然栖息地破碎化日益严重, 导致生物多样性急剧丧失[1-2]。环境变化与栖息地破碎化直接威胁野生生物个体生存与种群延续, 种群的持续缩减继而导致种内遗传多样性下降, 而遗传多样性的下降又进一步促使种群遗传衰退和存活力下降, 导致种群进一步缩减乃至物种濒危[3]。因此, 为了保护物种及其遗传多样性, 一方面需要了解决定生物多样性维持与丧失的微观机制, 另一方面需要关注栖息地景观格局的形成与改变将对生物多样性造成的影响[4-5]。为回答与探究这些问题, 景观遗传学(Landscape Genetics)应运而生, 它将影响生物多样性格局和种群生存的宏观(景观尺度)与微观(基因水平)机制有机地联系起来, 帮助我们更深入地理解与应对全球变化对生物多样性的效应。景观遗传学是一门交叉学科, 它结合了种群遗传学与景观生态学的原理与方法, 旨在揭示景观特征与微进化过程(如基因流、遗传漂变和自然选择)之间的相互作用[6-8]。Stéphanie Manel于2003年首次提出了景观遗传学的概念, 标志着景观遗传学学科的正式诞生[6]。随后, 景观遗传学快速发展。美国景观生态学会在2005年举办了首次以景观遗传学为主题的学术研讨会[7-8]。Landscape Ecology与Molecular Ecology杂志分别在2006年与2010年发表了以景观遗传学为主题的专刊。第一本以景观遗传学命名的专著《Landscape Genetics: Concepts, Methods, Applications》于2015由Niko Balkenhol等人撰写出版[9]。
许多山地生态系统是全球或区域尺度的生物多样性热点地区, 如中国西南山地、东喜马拉雅山地、中亚山地、东部赤道非洲山地、赤道安第斯山等[10]。山地因为其自身的海拔高差大, 温差明显, 形成了山地垂直自然带, 即在不同的海拔高度上形成了不同的气候、土壤环境, 使得适应不同环境的生物能够共同栖息于同一连续的生态系统中[11-12]。山地生态系统的独特性及其在生物多样性保护中的重要地位使其成为生态学研究的重点生态系统类型之一。同时, 山地生态系统对全球变化非常敏感, 气候变化、土地利用的改变、旅游业的发展、基础设施的建设、栖息地破碎化都能对山地生态系统产生直接或间接的影响[13-15]。因此, 我们需要了解物种在时空尺度上如何适应山地景观特征, 以及各种环境变化因素如何影响山地物种的遗传多样性分布模式。山地景观遗传学(Mountain Landscape Genetics)正是在景观遗传学的学科发展中, 针对山地生态系统和物种遗传格局的独特性和重要性发展形成的新兴分支。
山地景观遗传学通过综合景观生态学、空间统计学、地理学和种群遗传学等数据和分析方法来了解山地景观中物种遗传多样性的空间分布, 了解山地景观的形成过程对种群遗传空间格局的影响, 了解山地景观特征对种群基因交流和遗传分化的影响。为了全面了解目前山地景观遗传学的发展状况与重点研究问题, 为山地生态系统及其生物多样性保护管理提供科学参考, 本研究基于系统的文献检索与综述, 回顾了山地景观遗传学的发展历程, 探讨了目前该领域主要的研究方向与关键问题, 总结了常用的研究分析方法, 并对该领域内目前面临的挑战和未来发展趋势进行了讨论与展望。
1 发展情况 1.1 论文发表数量统计基于Web of Science核心数据库(Core Collection), 分别以landscape ecology和population genetics、landscape genetics和mountain为主题词, 对1900-2020年间学术期刊发表的英文研究论文与综述的标题、摘要、关键词进行检索。对检索到的结果逐一审核, 去除与该领域无关的文献, 共获得479篇景观遗传学文献, 其中192篇为山地景观遗传学相关文献。山地景观遗传学文献与其他景观遗传学文献的发表数量趋势基本保持一致(图 1)。第一篇山地景观遗传学方向的文章发表于1999年, 研究了美国西南部“山地岛屿(mountain islands)”松科黄松类乔木的分类与遗传结构[16]。其后, 2008-2013年该领域论文发表数量增长最为迅速, 2013-2016年发文数量经历短暂的下降, 2016年至今该领域研究又逐渐回升(图 1)。
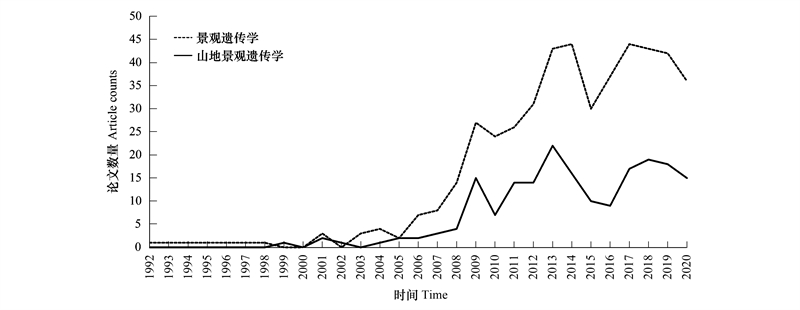
|
| 图 1 山地景观遗传学英文文献发表趋势 Fig. 1 Trends in publishing literature in English on mountain landscape genetics |
此外, 基于中国知网, 以“山”和“种群遗传”或“群体遗传”为主题词, 对1900-2020年间学术期刊发表的中文研究论文与综述的标题、摘要、关键词进行检索。对检索到的结果逐一审核, 去除与该领域无关的文献, 共获得31篇山地景观遗传学相关的中文文献。由于基于知网搜索的文献均为在中国进行的研究, 为避免造成统计偏差不加入后续的文献统计分析, 仅对中国所进行的相关研究的关键研究问题进行分析, 详见本文第2部分。
1.2 研究国家与地区山地景观遗传学是世界各地研究者广泛关注的研究方向, 截至2020年底, 来自46个国家的研究机构参与发表过山地景观遗传学相关英文论文。就国家而言, 美国是发表山地景观遗传学文章数量最多的国家(占所有发表文献数量的48.4%), 其次是德国(12.5%)、加拿大(12.0%)和中国(10.9%), 中国也是发表山地景观遗传学文章数量最多的亚洲国家(图 2)。从研究地点来看, 研究最多的是欧洲(38.4%)与北美洲(31.5%)的山地生态系统。落基山脉(Rocky Mountains, 27篇)、阿尔卑斯山脉(Alps, 19篇)、内华达山脉(Sierra Nevada, 11篇)、喜马拉雅-横断山脉(Himalaya-Hengduan Mountians, 6篇)、比利牛斯山脉(Pyrenees, 5篇)、阿巴拉契亚山脉(Appalachian Mountains, 5篇)是研究热点山地(图 3)。
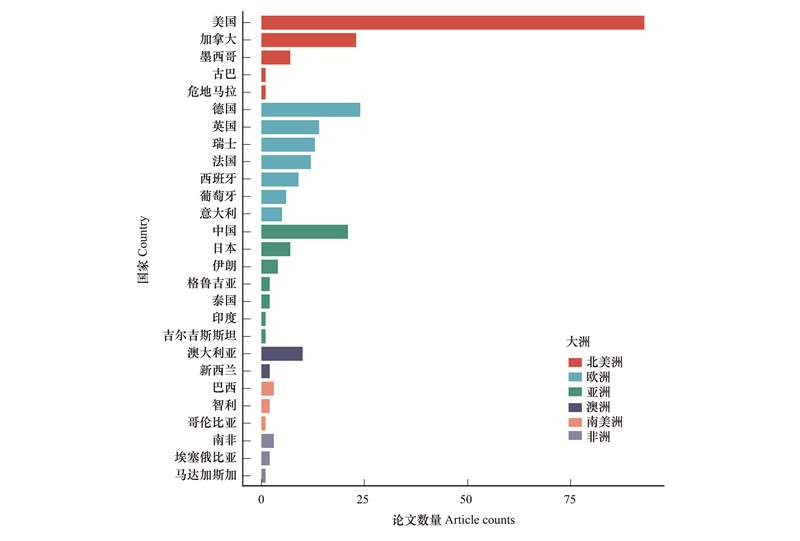
|
| 图 2 山地景观遗传学研究者单位从属国家与大洲 Fig. 2 The distribution and number of mountain landscape genetics research institutions |
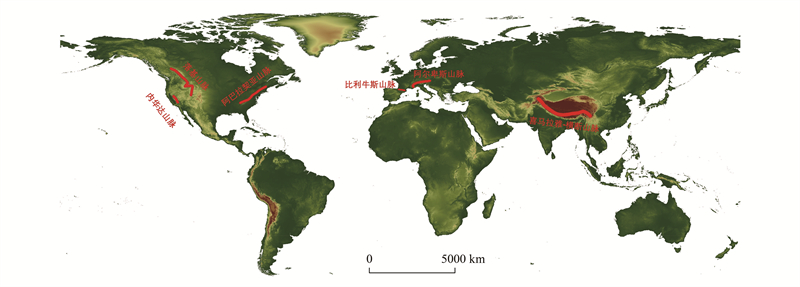
|
| 图 3 山地景观遗传学研究热点山地 Fig. 3 Mountain landscape genetics research hotspots |
利用HistCite软件[17]对系统文献检索获得的192篇山地景观遗传学英文研究论文进行分析。分别基于GCS(Global Citation Score, 即该文献在Web of Science数据库中的总被引次数)与LCS(Local Citation Score, 即该文献在所有山地景观遗传学研究中的被引次数)进行了文献网络图绘制(图 4和图 5)[17]。基于GCS与LCS做出的网络图, 除节点大小具有差别外, 网络关系相似, 说明该领域研究与其他领域交叉较少, 原因可能在于山地景观遗传学本就是交叉研究的产物。该领域引用次数最高的是2005年的一篇关于哥伦比亚斑蛙(Rana luteiventris)的研究, 截至2020年底已被引用超过250次。该研究的结果显示, 哥伦比亚斑蛙的基因流动受山脊和海拔的限制, 低海拔地区基因流动水平高, 种群内的遗传变异与海拔高度呈显著负相关[18]。

|
| 图 4 基于LCS绘制的山地景观遗传学文献网络图(N=192) Fig. 4 Literature network map of mountain landscape genetics based on LCS (N=192) LCS: 该文献在所有山地景观遗传学研究中的被引次数Local Citation Score |
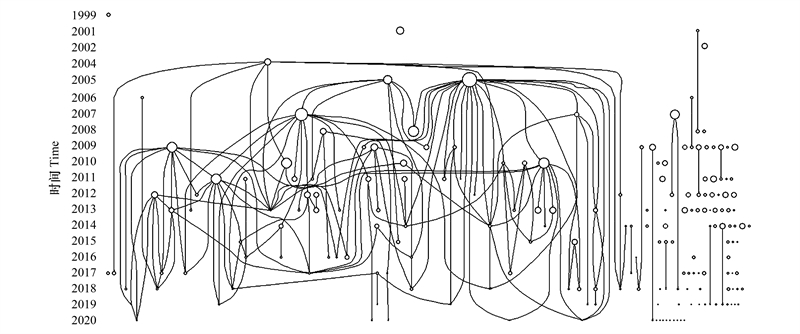
|
| 图 5 基于GCS绘制的山地景观遗传学文献网络图(N=192) Fig. 5 Literature network map of mountain landscape genetics based on GCS (N=192) GCS, 该文献在Web of Science数据库中的总被引次数Global Citation Score |
植物是山地景观遗传学研究最早关注的生物类群[16]。在所有研究类群中, 脊椎动物是研究发表最多的类群, 占发表文献总数的62.0%;脊椎动物中, 又以对哺乳类(占脊椎动物发表文献总数的52.9%)与两栖类(23.5%)的研究最多(图 6)。较早的山地景观遗传学研究集中于脊椎动物与植物, 这可能主要与其相对容易观察、样品易于获得等特点有关。近年来山地景观遗传学的研究类群更为广泛, 重要原因之一是DNA测序技术的快速发展, 使研究者能够以较高的成本效益获得大量的遗传数据[19-21]。

|
| 图 6 山地景观遗传学研究类群 Fig. 6 Research taxa of mountain landscape genetics |
景观遗传学重点关注的问题主要包括:识别基因流的路径与障碍; 量化景观特征对种群遗传结构时空变化的影响; 识别种群源汇动态和迁移廊道; 理解景观属性影响生态过程的时空尺度; 以及检验关于特定物种的生态学假说等[22]。山地景观遗传学是在山地景观尺度上进行的遗传学研究, 通过对Web of science检索出的192篇山地景观遗传学论文的研究内容进行逐一分类统计发现, 最受关注的是识别山地景观中的基因流路径或阻碍(占所有研究论文数量的82.8%), 以及量化评估山地景观特征如何影响种群遗传结构的时空变化(67.2%)。
山地景观中垂直高程差、地形复杂性等景观特征都可能对种群扩散或基因流动造成影响[18, 23-30]。海拔成为限制基因流动的原因主要有二:一是种群在低海拔和高海拔之间的扩散受到限制, 二是在低海拔种群和高海拔种群之间存在交配前隔离[18, 23]。低海拔种群与高海拔种群如果在扩散能力上存在较大差异, 则可能导致高、低海拔种群之间出现交配前隔离, 进而限制了基因在低海拔和高海拔区域之间的交流[18, 23]。海拔作为一个重要的生态限制因子, 除去它对生物的直接影响外, 也与它对环境条件中其他因素的影响密切相关, 比如温度、生长季节、森林覆盖等[18, 24-25]。由于海拔高差导致的气候差异可能是限制基因在景观中流动的关键因素[24]。山地景观中山脊的存在是另一影响种群遗传结构的重要因素:一方面可能阻碍种群间的基因流动, 另一方面山脊的存在也可能促进异域物种形成[26-28]。对于有潜力进行长距离迁徙的物种而言, 由于山脊的存在使其扩散速率很低, 阻碍了种群间基因流动, 从而导致山地两栖动物种群分化程度较高[18]。对于种群数量较大的物种而言, 山脊的存在可能促进异域物种形成[27-28]。要了解基因流动究竟受到哪些山地景观特征的限制, 需要获取实地山地景观特征的量化信息, 同时获取不同山地景观下的种群遗传数据, 才能验证真正阻碍或限制种群扩散与基因流动的原因。此外, 需要了解种群的主要基因库是低海拔种群还是高海拔种群。如果低海拔种群为一物种主要的基因库, 则该物种可能存在更大的濒危风险, 因为低海拔种群往往最先受到栖息地丧失、破碎化以及外来物种入侵的影响[19, 29-31]。
此外, 对中国山地景观遗传学的31篇研究论文进行分析发现, 其中“识别山地景观中的基因流路径或阻碍”(占所有研究论文数量的58.1%)与“量化评估山地景观特征如何影响种群遗传结构的时空变化”(45.2%)为中国山地景观遗传研究最为关注的问题。
近十年来, 中国山地景观遗传学快速发展, 截至2020年基于在中国进行的山地景观遗传学研究已发表论文53篇(Web of science, 22;知网, 31)。研究类群主要关注植物(占发文总数的62.3%)和脊椎动物(35.8%)。对脊椎动物的研究中以两栖动物为最多, 占所有脊椎动物发文数量的52.6%。相较于世界范围内其他区域的山地景观遗传研究而言, 中国相关研究在无脊椎动物与真菌等类群的研究上较少或存在空缺。中国的山地景观遗传学研究地点主要集中于喜马拉雅-横断山脉与秦岭, 其中喜马拉雅-横断山脉的研究主要关注植物。喜马拉雅-横断山脉的相关研究中藏川杨(Populus szechuanicavar)的研究最多, 研究发现对于青藏高原塞吉拉山的藏川杨而言, 海拔对于该物种的基因流动而言是一个重要的限制因子, 但并未造成高低海拔种群间的地理隔离, 高低海拔种群均处于较高的遗传多样性水平, 而且结合景观基因组分析发现了4个与海拔变化相关的基因[32-33]。此外, 还对喜马拉雅-横断山脉的山生柳(Salix oritrepha)、金露梅(Potentilla fruticosa)与喜马拉雅铁杉(Tsuga dumosa)等展开了研究[34-36]。在秦岭则主要开展了针对大熊猫(Ailuropoda melanoleuca)与两栖类的研究[37-38]。研究物种中有2种受威胁程度较高, 分别是大熊猫与宁陕齿突蟾(Scutiger ningshanensis)[37-38]。针对秦岭地区大熊猫种群的研究发现, 该种群的基因流强度主要与坡向和地形复杂度这两个因素有关, 东向坡向促进基因流动, 地形复杂的景观限制基因流动; 基于研究结果, 研究人员提出了能够促进栖息地斑块间大熊猫基因流的最佳廊道[37]。对宁陕齿突蟾的研究发现地形复杂度及秦岭山脉的阻隔是诱使该种群遗传结构形成的重要原因, 并根据局域种群的遗传结构将该物种划分为3个管理单元, 为该物种的保护与管理提供了科学信息[38]。
3 山地景观遗传学研究方法山地景观遗传学研究融合了景观生态学与种群遗传学的研究方法, 通过获取山地生态系统中生物种群的遗传信息, 量化山地景观中的特征变量, 同时结合空间统计分析方法, 分析山地遗传格局形成的原因[6, 39-40]。
3.1 遗传标记种群遗传学常用的标记包括等位酶(Allozyme)、限制片段长度多态性(Restriction Fragment Length Polymorphisms, RFLP)、扩增片段长度多态性(Amplified Fragment Length Polymorphisms, AFLP)、随机扩增多态性DNA标记(Random Amplified Polymorphic DNA, RAPD)、单核苷酸多态性(Single Nucleotide Polymorphism, SNP)、微卫星位点(Simple Sequence Repeats, SSR)和线粒体DNA(Mitochondrial DNA, mtDNA)、叶绿体DNA(Chloroplast DNA, cpDNA)等[41-44]。在山地景观遗传学的早期研究中, 特别是在植物研究中, 研究者常选择形态学或生化标记如等位酶[16, 41]。等位酶是最早用于种群遗传研究的遗传标记之一, 其表达呈共显性, 易于进行简便的亲本分析, 但不能反映非编码蛋白的基因差异[45]。RFLP、AFLP等基于位点或片段差异计算不同样本DNA之间的遗传距离, 但也存在工作量大、依赖放射性标记等缺点[46-47]。迄今在山地景观遗传学研究中最常使用的遗传标记是SSR, 有63.5%的研究均使用了SSR。SSR分子标记具多态性高、数量丰富、突变率高、分辨率高、共显性遗传、操作简单、开发成本较低、检测技术高效等诸多优点[48-49]; 且随着测序技术的普及和成本降低, SSR分子标记技术的使用日益经济简便[50-51]。近年来, 随着高通量测序技术与生物信息学的发展, 相关研究中SNP的使用频率逐渐上升, 占所有研究10.4%。SNP标记相较于其他标记具有性价比高、可大规模自动化检测、在基因组中广泛分布、稳定性好等特点[52-54]。
3.2 空间遗传格局分析在山地景观遗传学研究中, 常用遗传多样性指数来量化评估种群的遗传多样程度。常用的遗传多样性指数包括等位基因频率(Allele Frequency)、等位基因数(Na)、有效等位基因数(Ne)、观察杂合度(Observed Heterozygosity, Ho)、期望杂合度(Expected Heterozygosity, He)等[55-56]。种群间的遗传差异程度常用遗传分化系数如Fst或Rst衡量, 并可通过个体网络图、主坐标分析(Principal Co-ordinates Analysis, PCoA)等方法进行可视化[57-59]。对山地景观中不同地理种群进行遗传分化程度划分, 可使用分子变异方差分析(Analysis of Molecular Variance, AMOVA)、贝叶斯分配检验(Bayesian assignment test, 如STRUCTURE、Geneland分析)等[60-61]。探究遗传格局与山地景观空间属性的相关性常用Mantel检验(Mantel′s test)、回归分析(Regression Analysis)、距离隔离检验(Isolation By Distance, IBD)等[62-63]。
4 讨论与展望 4.1 研究挑战与其他类型的地理环境相比, 山地景观具有高度的异质性, 随之形成的种群遗传结构及过程也十分复杂, 对研究者提出了诸多挑战。首先, 许多经典的种群遗传模型和空间格局分析方法不考虑景观异质性, 且存在一些严格的前提假设。如果将这些常规模型和统计分析方法直接应用于山地种群遗传的分析, 可能导致遗传格局和空间的错误匹配, 难以合理解释实际的生态现象。其次, 物种种群遗传结构的形成是长期历史气候和环境因素综合作用的结果。如果山地景观在短时间内发生较大的改变, 其对种群遗传结构的影响将存在时间滞后性, 因此, 在相关研究中需要同时考虑物种的世代周期与景观变化时间尺度的关系[64]。此外, 由于不同的遗传标记进化速率不同, 可能反映不同的时空尺度种群遗传格局的变化, 因此需要对遗传标记的突变速率进行分析, 慎重选择遗传标记。不同的景观遗传学研究中如果选用不同的遗传标记, 则不同研究的结果之间可能缺乏可比性。另外, 不同生物类群具有不同的扩散方式和繁殖模式, 需要选择适用于目标类群行为特征的空间统计方法。
4.2 研究应用山地生物种群的结构与遗传变异模式是长期景观格局与历史气候的结果, 但是近年来, 气候变化和人为活动(如土地利用改变、大规模森林砍伐、过度放牧等)使自然栖息地迅速退化和破碎化, 山地物种的栖息地面积缩小、破碎化或丧失, 导致很多山地生物种群迅速缩减、遗传多样性下降[65-70]。对于长距离扩散能力弱、有特殊栖息地偏好的物种而言, 栖息地的破碎与丧失往往对物种或种群往往会造成严重影响[68-69]。栖息地破碎化将直接导致景观连通性变弱, 物种扩散阻力增加, 难以充分进行基因交流, 使得小种群遗传多样性下降[71-72]。栖息地破碎化还直接影响到山地物种依赖的食物资源、小气候条件等[71-72]。因此, 山地濒危物种保护策略不仅要考虑其栖息地形的复杂性, 同时需要了解栖息地隔离状况, 确定濒危物种受胁关键景观原因, 从而保护和恢复迁移通道以促进栖息地斑块间的基因流动, 保障小种群存活力[62, 73]。
山地景观遗传学的研究对于山地生态系统具有重要应用价值。山地景观遗传学研究不仅可以帮助我们理解山地景观中基因流动与种群扩散模式的形成与阻碍, 还可以帮助我们理解人类发展导致山地景观改变将如何影响遗传多样性、种群生存力、物种多样性、群落结构甚至疾病传播[74]。基因组学方法和分析手段的快速发展, 使我们可以更精确地量化评估山地物种中基因频率、基因流随着时空变化而发生的改变。可以通过整合景观遗传数据与气候数据进行建模, 预测物种可适应各种景观属性的边界, 阐明物种可能以何种方式适应环境与景观的变化, 评估物种适应性、迁地保护或灭绝的概率与风险。
4.3 未来展望景观遗传学是21世纪开始逐渐发展起来的新兴交叉学科, 山地景观遗传学是其中的重要分支, 相关研究对于山地生态系统和生物多样性保护具有重要理论价值和实际意义。目前我国的相关研究, 在研究区域、研究类群和研究方向上均存在部分空白(图 6), 在诸多亟需科学信息的山地物种的研究上具有广阔的发展前景, 需要大力培养相关人才推动领域发展。山地景观遗传学作为交叉学科的衍生分支, 对研究人员的知识背景提出更高的要求, 因此在高校相关专业的课程设置上, 除传统的生态学和种群遗传学课程外, 应增设景观遗传学等课程, 并配套相关实验或实践课程, 奠定未来科研人员的知识基础。目前, 山地景观遗传学与其他研究领域的进一步深度交叉仍存在很大潜力(图 4和图 5), 未来应考虑与生物信息学、人工智能等多种研究领域的交叉与融合。此外, 现代各科学领域研究的发展都离不开大数据的支持, 应积极推动建立全球景观遗传与山地景观遗传学的研究数据库, 以便相关研究人员跟进领域研究最新进展, 开展地区间的比较研究, 填补现有研究类群与研究区域的空缺。随着景观遗传学方法与技术的不断发展和完善, 我们可以更深入地理解山地景观对种群遗传过程的影响, 从而制定更科学合理的保护策略, 更有效地保护山地生态系统的生物多样性。
| [1] |
Betts M G, Wolf C, Ripple W J, Phalan B, Millers K A, Duarte A, Butchart S H M, Levi T. Global forest loss disproportionately erodes biodiversity in intact landscapes. Nature, 2017, 547(7664): 441-444. DOI:10.1038/nature23285 |
| [2] |
He C Y, Liu Z F, Tian J, Ma Q. Urban expansion dynamics and natural habitat loss in China: a multiscale landscape perspective. Global Change Biology, 2014, 20(9): 2886-2902. DOI:10.1111/gcb.12553 |
| [3] |
Arenas M, Ray N, Currat M, Excoffier L. Consequences of range contractions and range shifts on molecular diversity. Molecular Biology and Evolution, 2012, 29(1): 207-218. DOI:10.1093/molbev/msr187 |
| [4] |
Fenderson L E, Kovach A I, Llamas B. Spatiotemporal landscape genetics: investigating ecology and evolution through space and time. Molecular Ecology, 2020, 29(2): 218-246. DOI:10.1111/mec.15315 |
| [5] |
Paz A, Ibáñez R, Lips K R, Crawford A J. Testing the role of ecology and life history in structuring genetic variation across a landscape: a trait-based phylogeographic approach. Molecular Ecology, 2015, 24(14): 3723-3737. DOI:10.1111/mec.13275 |
| [6] |
Manel S, Holderegger R. Ten years of landscape genetics. Trends in Ecology & Evolution, 2013, 28(10): 614-621. |
| [7] |
Manel S, Schwartz M K, Luikart G, Taberlet P. Landscape genetics: combining landscape ecology and population genetics. Trends in Ecology & Evolution, 2003, 18(4): 189-197. |
| [8] |
Epps C W, Keyghobadi N. Landscape genetics in a changing world: disentangling historical and contemporary influences and inferring change. Molecular Ecology, 2015, 24(24): 6021-6040. DOI:10.1111/mec.13454 |
| [9] |
Balkenhol N, Cushman S A, Storfer A T, Waits L P. Landscape Genetics: Concepts, Methods, Applications. Chichester: John Wiley & Sons, Ltd, 2016.
|
| [10] |
Myers N, Mittermeier R A, Mittermeier C G, da Fonseca G A B, Kent J. Biodiversity hotspots for conservation priorities. Nature, 2000, 403(6772): 853-858. DOI:10.1038/35002501 |
| [11] |
Xing Y W, Ree R H. Uplift-driven diversification in the Hengduan Mountains, a temperate biodiversity hotspot. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(17): E3444-E3451. |
| [12] |
Huber U M, Bugmann H K M, Reasoner M A. Global Change and Mountain Regions: An Overview of Current Knowledge. Dordrecht: Springer, 2005.
|
| [13] |
Peters M K, Hemp A, Appelhans T, Becker J N, Behler C, Classen A, Detsch F, Ensslin A, Ferger S W, Frederiksen S B, Gebert F, Gerschlauer F, Gütlein A, Helbig-Bonitz M, Hemp C, Kindeketa W J, Kühnel A, Mayr A V, Mwangomo E, Ngereza C, Njovu H K, Otte I, Pabst H, Renner M, R der J, Rutten G, Schellenberger Costa D, Sierra-Cornejo N, Vollstädt M G R, Dulle H I, Eardley C D, Howell K M, Keller A, Peters R S, Ssymank A, Kakengi V, Zhang J, Bogner C, B hning-Gaese K, Brandl R, Hertel D, Huwe B, Kiese R, Kleyer M, Kuzyakov Y, Nauss T, Schleuning M, Tschapka M, Fischer M, Steffan-Dewenter I. Climate-land-use interactions shape tropical mountain biodiversity and ecosystem functions. Nature, 2019, 568(7750): 88-92. DOI:10.1038/s41586-019-1048-z |
| [14] |
Barros A, Monz C, Pickering C. Is tourism damaging ecosystems in the Andes? Current knowledge and an agenda for future research. AMBIO, 2015, 44(2): 82-98. DOI:10.1007/s13280-014-0550-7 |
| [15] |
Razgour O, Kasso M, Santos H, Juste J. Up in the air: threats to Afromontane biodiversity from climate change and habitat loss revealed by genetic monitoring of the Ethiopian Highlands bat. Evolutionary Applications, 2021, 14(3): 794-806. DOI:10.1111/eva.13161 |
| [16] |
Rehfeldt G E. Systematics and genetic structure of Ponderosae taxa (Pinaceae) inhabiting the mountain islands of the Southwest. American Journal of Botany, 1999, 86(5): 741-752. DOI:10.2307/2656584 |
| [17] |
Garfield E, Paris S W, Stock W G. HistCiteTM: a software tool for informetric analysis of citation linkage. NFD Information-Wissenschaft und Praxis, 2006, 57(8): 391-400. |
| [18] |
Funk W C, Blouin M S, Corn P S, Maxell B A, Pilliod D S, Amish S, Allendorf F W. Population structure of Columbia spotted frogs (Rana luteiventris) is strongly affected by the landscape. Molecular Ecology, 2005, 14(2): 483-496. DOI:10.1111/j.1365-294X.2005.02426.x |
| [19] |
Taberlet P, Coissac E, Hajibabaei M, Rieseberg L H. Environmental DNA. Molecular Ecology, 2012, 21(8): 1789-1793. DOI:10.1111/j.1365-294X.2012.05542.x |
| [20] |
Purahong W, Wubet T, Krüger D, Buscot F. Application of next-generation sequencing technologies to conservation of wood-inhabiting fungi. Conservation Biology, 2019, 33(3): 716-724. DOI:10.1111/cobi.13240 |
| [21] |
Zhou J Z, Deng Y, Shen L N, Wen C Q, Yan Q Y, Ning D L, Qin Y J, Xue K, Wu L Y, He Z L, Voordeckers J W, Van Nostrand J D, Buzzard V, Michaletz S T, Enquist B J, Weiser M D, Kaspari M, Waide R, Yang Y F, Brown J H. Temperature mediates continental-scale diversity of microbes in forest soils. Nature Communications, 2016, 7: 12083. DOI:10.1038/ncomms12083 |
| [22] |
沈泽昊, 吉成均. 景观遗传学原理及其在生境片断化遗传效应研究中的应用. 生态学报, 2010, 30(18): 5066-5076. |
| [23] |
Funk W C, Greene A E, Corn P S, Allendorf F W. High dispersal in a frog species suggests that it is vulnerable to habitat fragmentation. Biology Letters, 2005, 1(1): 13-16. DOI:10.1098/rsbl.2004.0270 |
| [24] |
Shafer A B A, Northrup J M, White K S, Boyce M S, Côté S D, Coltman D W. Habitat selection predicts genetic relatedness in an alpine ungulate. Ecology, 2012, 93(6): 1317-1329. DOI:10.1890/11-0815.1 |
| [25] |
Bull R A S, Cushman S A, Mace R, Chilton T, Kendall K C, Landguth E L, Schwartz M K, Mckelvey K, Allendorf F W, Luikart G. Why replication is important in landscape genetics: American black bear in the Rocky Mountains. Molecular Ecology, 2011, 20(6): 1092-1107. DOI:10.1111/j.1365-294X.2010.04944.x |
| [26] |
Olah G, Smith A L, Asner G P, Brightsmith D J, Heinsohn R G, Peakall R. Exploring dispersal barriers using landscape genetic resistance modelling in scarlet macaws of the Peruvian Amazon. Landscape Ecology, 2017, 32(2): 445-456. DOI:10.1007/s10980-016-0457-8 |
| [27] |
Lougheed S C, Gascon C, Jones D A, Bogart J P, Boag P T. Ridges and rivers: a test of competing hypotheses of Amazonian diversification using a dart-poison frog (Epipedobates femoralis). Proceedings of the Royal Society B: Biological Sciences, 1999, 266(1431): 1829-1835. DOI:10.1098/rspb.1999.0853 |
| [28] |
Monsen K J, Blouin M S. Genetic structure in a montane ranid frog: restricted gene flow and nuclear-mitochondrial discordance. Molecular Ecology, 2003, 12(12): 3275-3286. DOI:10.1046/j.1365-294X.2003.02001.x |
| [29] |
Leu M, Hanser S E, Knick S T. The human footprint in the west: a large-scale analysis of anthropogenic impacts. Ecological Applications, 2008, 18(5): 1119-1139. DOI:10.1890/07-0480.1 |
| [30] |
Lele A, Arasumani M, Vishnudas C K, Joshi V, Jathanna D, Robin V V. Elevation and landscape change drive the distribution of a montane, endemic grassland bird. Ecology and Evolution, 2020, 10(14): 7755-7767. DOI:10.1002/ece3.6500 |
| [31] |
Zancolli G, R del M O, Steffan-Dewenter I, Storfer A. Comparative landscape genetics of two river frog species occurring at different elevations on Mount Kilimanjaro. Molecular Ecology, 2014, 23(20): 4989-5002. DOI:10.1111/mec.12921 |
| [32] |
Zheng C F, Tan L Z, Sang M M, Ye M X, Wu R L. Genetic adaptation of Tibetan poplar (Populus szechuanica var. tibetica) to high altitudes on the Qinghai-Tibetan Plateau. Ecology and Evolution, 2020, 10(20): 10974-10985. DOI:10.1002/ece3.6508 |
| [33] |
沈登锋, 薄文浩, 徐放, 姜立波, 毛柯, 韩玉霞, 邬荣领. 色季拉山不同海拔高度的藏川杨种群遗传多样性研究. 植物遗传资源学报, 2014, 15(4): 692-698. |
| [34] |
吴锦, 杨茜, 王久利, 高庆波, 张发起, 陈世龙. 青藏高原地区山生柳遗传多样性研究. 广西植物, 2019, 39(9): 1180-1190. |
| [35] |
Shimono A, Ueno S, Gu S, Zhao X, Tsumura Y, Tang Y. Range shifts of Potentilla fruticosa on the Qinghai-Tibetan Plateau during glacial and interglacial periods revealed by chloroplast DNA sequence variation. Heredity, 2010, 104(6): 534-542. DOI:10.1038/hdy.2009.158 |
| [36] |
Yu H B, Zhang Y L, Liu L S, Qi W, Li S C, Hu Z J. Combining the least cost path method with population genetic data and species distribution models to identify landscape connectivity during the late Quaternary in Himalayan hemlock. Ecology and Evolution, 2015, 5(24): 5781-5791. DOI:10.1002/ece3.1840 |
| [37] |
Ma T X, Hu Y B, Russo I R M, Nie Y G, Yang T Y, Xiong L J, Ma S, Meng T, Han H, Zhang X M, Bruford M W, Wei F W. Walking in a heterogeneous landscape: dispersal, gene flow and conservation implications for the giant panda in the Qinling Mountains. Evolutionary Applications, 2018, 11(10): 1859-1872. DOI:10.1111/eva.12686 |
| [38] |
Meng H Z, Li X C, Qiao P H. Population structure, historical biogeography and demographic history of the alpine toad Scutiger ningshanensis in the Tsinling Mountains of Central China. PLoS One, 2014, 9(6): e100729. DOI:10.1371/journal.pone.0100729 |
| [39] |
Storfer A, Murphy M A, Spear S F, Holderegger R, Waits L P. Landscape genetics: where are we now?. Molecular Ecology, 2010, 19(17): 3496-3514. DOI:10.1111/j.1365-294X.2010.04691.x |
| [40] |
王根绪, 刘国华, 沈泽昊, 王文志. 山地景观生态学研究进展. 生态学报, 2017, 37(12): 3967-3981. |
| [41] |
Dufresne F, Stift M, Vergilino R, Mable B K. Recent progress and challenges in population genetics of polyploid organisms: an overview of current state-of-the-art molecular and statistical tools. Molecular Ecology, 2014, 23(1): 40-69. DOI:10.1111/mec.12581 |
| [42] |
Helyar S J, Hemmer-Hansen J, Bekkevold D, Taylor M I, Ogden R, Limborg M T, Cariani A, Maes G E, Diopere E, Carvalho G R, Nielsen E E. Application of SNPs for population genetics of nonmodel organisms: new opportunities and challenges. Molecular Ecology Resources, 2011, 11(S1): 123-136. |
| [43] |
Hindrikson M, Remm J, Pilot M, Godinho R, Stronen A V, Baltrūnaité L, Czarnomska S D, Leonard J A, Randi E, Nowak C, Åkesson M, López-Bao J V, Álvares F, Llaneza L, Echegaray J, Vilà C, Ozolins J, Rungis D, Aspi J, Paule L, Skrbinšek T, Saarma U. Wolf population genetics in Europe: a systematic review, meta-analysis and suggestions for conservation and management. Biological Reviews, 2017, 92(3): 1601-1629. DOI:10.1111/brv.12298 |
| [44] |
Blanco-Pastor J L, Fernández-Mazuecos M, Coello A J, Pastor J, Vargas P. Topography explains the distribution of genetic diversity in one of the most fragile European hotspots. Diversity and Distributions, 2019, 25(1): 74-89. DOI:10.1111/ddi.12836 |
| [45] |
Hirao A S, Kudo G. Landscape genetics of alpine-snowbed plants: comparisons along geographic and snowmelt gradients. Heredity, 2004, 93(3): 290-298. DOI:10.1038/sj.hdy.6800503 |
| [46] |
Manel S, Poncet B N, Legendre P, Gugerli F, Holderegger R. Common factors drive adaptive genetic variation at different spatial scales in Arabis alpina. Molecular Ecology, 2010, 19(17): 3824-3835. DOI:10.1111/j.1365-294X.2010.04716.x |
| [47] |
Jay F, Manel S, Alvarez N, Durand E Y, Thuiller W, Holderegger R, Taberlet P, François O. Forecasting changes in population genetic structure of alpine plants in response to global warming. Molecular Ecology, 2012, 21(10): 2354-2368. DOI:10.1111/j.1365-294X.2012.05541.x |
| [48] |
Schoville S D, Dalongeville A, Viennois G, Gugerli F, Taberlet P, Lequette B, Alvarez N, Manel S. Preserving genetic connectivity in the European Alps protected area network. Biological Conservation, 2018, 218: 99-109. DOI:10.1016/j.biocon.2017.12.017 |
| [49] |
Litt M, Luty J A. A hypervariable microsatellite revealed by in vitro amplification of a dinucleotide repeat within the cardiac muscle actin gene. American Journal of Human Genetics, 1989, 44(3): 397-401. |
| [50] |
Pourabed E, Jazayeri Noushabadi M R, Jamali S H, Moheb Alipour N, Zareyan A, Sadeghi L. Identification and DUS testing of rice varieties through microsatellite markers. International Journal of Plant Genomics, 2015, 2015: 965073. |
| [51] |
Kalia R K, Rai M K, Kalia S, Singh R, Dhawan A K. Microsatellite markers: an overview of the recent progress in plants. Euphytica, 2011, 177(3): 309-334. DOI:10.1007/s10681-010-0286-9 |
| [52] |
Keighley M V, Langmore N E, Peñalba J V, Heinsohn R. Modelling dispersal in a large parrot: a comparison of landscape resistance models with population genetics and vocal dialect patterns. Landscape Ecology, 2020, 35(1): 129-144. DOI:10.1007/s10980-019-00938-0 |
| [53] |
Chan K O, Brown R M. Elucidating the drivers of genetic differentiation in Malaysian torrent frogs (Anura: Ranidae: Amolops): a landscape genomics approach. Zoological Journal of the Linnean Society, 2020, 190(1): 65-78. DOI:10.1093/zoolinnean/zlz151 |
| [54] |
Mulder K P, Cortes-Rodriguez N, Grant E H C, Brand A, Fleischer R C. North-facing slopes and elevation shape asymmetric genetic structure in the range-restricted salamander Plethodon shenandoah. Ecology and Evolution, 2019, 9(9): 5094-5105. DOI:10.1002/ece3.5064 |
| [55] |
Barley A J, Monnahan P J, Thomson R C, Grismer L L, Brown R M. Sun skink landscape genomics: assessing the roles of micro-evolutionary processes in shaping genetic and phenotypic diversity across a heterogeneous and fragmented landscape. Molecular Ecology, 2015, 24(8): 1696-1712. DOI:10.1111/mec.13151 |
| [56] |
Bertorelle G, Bruford M W, Hauffe H C, Rizzoli A, Vernesi C. Population Genetics for Animal Conservation. Cambridge: Cambridge University Press, 2009.
|
| [57] |
Pérez-Espona S, Pérez-Barbería F J, McLeod J E, Jiggins C D, Gordon I J, Pemberton J M. Landscape features affect gene flow of Scottish Highland red deer (Cervus elaphus). Molecular Ecology, 2008, 17(4): 981-996. DOI:10.1111/j.1365-294X.2007.03629.x |
| [58] |
Finn D S, Theobald D M, Black W C, Poff N L. Spatial population genetic structure and limited dispersal in a Rocky Mountain alpine stream insect. Molecular Ecology, 2006, 15(12): 3553-3566. DOI:10.1111/j.1365-294X.2006.03034.x |
| [59] |
Byrne M, Millar M A, Coates D J, Macdonald B M, McArthur S M, Zhou M, van Leeuwen S. Refining expectations for environmental characteristics of refugia: two ranges of differing elevation and topographical complexity are mesic refugia in an arid landscape. Journal of Biogeography, 2017, 44(11): 2539-2550. DOI:10.1111/jbi.13057 |
| [60] |
Sork V L, Smouse P E. Genetic analysis of landscape connectivity in tree populations. Landscape Ecology, 2006, 21(6): 821-836. DOI:10.1007/s10980-005-5415-9 |
| [61] |
Zalewski A, Piertney S B, Zalewska H, Lambin X. Landscape barriers reduce gene flow in an invasive carnivore: geographical and local genetic structure of American mink in Scotland. Molecular Ecology, 2009, 18(8): 1601-1615. DOI:10.1111/j.1365-294X.2009.04131.x |
| [62] |
Tsui C K M, Roe A D, El-Kassaby Y A, Rice A V, Alamouti S M, Sperling F A H, Cooke J E K, Bohlmann J, Hamelin R C. Population structure and migration pattern of a conifer pathogen, Grosmannia clavigera, as influenced by its symbiont, the mountain pine beetle. Molecular Ecology, 2012, 21(1): 71-86. DOI:10.1111/j.1365-294X.2011.05366.x |
| [63] |
Murphy M A, Evans J S, Cushman S A, Storfer A. Representing genetic variation as continuous surfaces: an approach for identifying spatial dependency in landscape genetic studies. Ecography, 2008, 31(6): 685-697. DOI:10.1111/j.1600-0587.2008.05428.x |
| [64] |
Jaffé R, Veiga J C, Pope N S, Lanes É C M, Carvalho C S, Alves R, Andrade S C S, Arias M C, Bonatti V, Carvalho A T, de Castro M S, Contrera F A L, Francoy T M, Freitas B M, Giannini T C, Hrncir M, Martins C F, Oliveira G, Saraiva A M, Souza B A, Imperatriz-Fonseca V L. Landscape genomics to the rescue of a tropical bee threatened by habitat loss and climate change. Evolutionary Applications, 2019, 12(6): 1164-1177. DOI:10.1111/eva.12794 |
| [65] |
Castillo J A, Epps C W, Davis A R, Cushman S A. Landscape effects on gene flow for a climate-sensitive montane species, the American pika. Molecular Ecology, 2014, 23(4): 843-856. DOI:10.1111/mec.12650 |
| [66] |
Moritz C, Agudo R. The future of species under climate change: resilience or decline?. Science, 2013, 341(6145): 504-508. DOI:10.1126/science.1237190 |
| [67] |
Aavik T, Thetloff M, Träger S, Hernández-Agramonte I M, Pärtel M. Delayed and immediate effects of habitat loss on the genetic diversity of the grassland plant Trifolium montanum. Biodiversity and Conservation, 2019, 28(12): 3299-3319. DOI:10.1007/s10531-019-01822-8 |
| [68] |
Asefa A, Mengesha G, Shimelis A, Mamo Y. Livestock grazing in afromontane grasslands in the northern bale mountains, Ethiopia: implications for bird conservation. Science, Technology and Arts Research Journal, 2015, 4(2): 112-121. |
| [69] |
Kidane Y, Stahlmann R, Beierkuhnlein C. Vegetation dynamics, and land use and land cover change in the Bale Mountains, Ethiopia. Environmental Monitoring and Assessment, 2012, 184(12): 7473-7489. DOI:10.1007/s10661-011-2514-8 |
| [70] |
Razgour O, Rebelo H, Puechmaille S J, Juste J, Ibáñez C, Kiefer A, Burke T, Dawson D A, Jones G. Scale-dependent effects of landscape variables on gene flow and population structure in bats. Diversity and Distributions, 2014, 20(10): 1173-1185. DOI:10.1111/ddi.12200 |
| [71] |
Nyssen J, Frankl A, Haile M, Hurni H, Descheemaeker K, Crummey D, Ritler A, Portner B, Nievergelt B, Moeyersons J, Munro N, Deckers J, Billi P, Poesen J. Environmental conditions and human drivers for changes to north Ethiopian mountain landscapes over 145 years. Science of the Total Environment, 2014, 485-486: 164-179. DOI:10.1016/j.scitotenv.2014.03.052 |
| [72] |
Saremi N F, Supple M A, Byrne A, Cahill J A, Coutinho L L, Dalén L, Figueiró H V, Johnson W E, Milne H J, O'Brien S J, O'Connell B, Onorato D P, Riley S P D, Sikich J A, Stahler D R, Villela P M S, Vollmers C, Wayne R K, Eizirik E, Corbett-Detig R B, Green R E, Wilmers C C, Shapiro B. Puma genomes from North and South America provide insights into the genomic consequences of inbreeding. Nature Communications, 2019, 10: 4769. DOI:10.1038/s41467-019-12741-1 |
| [73] |
Nali R C, Becker C G, Zamudio K R, Prado C P A. Topography, more than land cover, explains genetic diversity in a Neotropical savanna tree frog. Diversity and Distributions, 2020, 26(12): 1798-1812. DOI:10.1111/ddi.13154 |
| [74] |
Kozakiewicz C P, Burridge C P, Funk W C, VandeWoude S, Craft M E, Crooks K R, Ernest H B, Fountain-Jones N M, Carver S. Pathogens in space: advancing understanding of pathogen dynamics and disease ecology through landscape genetics. Evolutionary Applications, 2018, 11(10): 1763-1778. DOI:10.1111/eva.12678 |
 2022, Vol. 42
2022, Vol. 42




