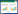文章信息
- 刘鹏, 毕江涛, 李文兵, 惠治兵, 肖国举, 孙权, 王静
- LIU Peng, BI Jiangtao, LI Wenbing, HUI Zhibing, XIAO Guoju, SUN Quan, WANG Jing
- 生物有机肥对盐碱地水稻叶片的影响转录组分析
- Transcriptome analysis of the effect of bio-organic fertilizer on rice leaves
- 生态学报. 2022, 42(6): 2342-2356
- Acta Ecologica Sinica. 2022, 42(6): 2342-2356
- http://dx.doi.org/10.5846/stxb202012103153
-
文章历史
- 收稿日期: 2020-12-10
- 网络出版日期: 2021-11-26
2. 宁夏大学生态环境学院, 银川 750021
2. School of Ecology and Environment, Ningxia University, Yinchuan 750021, China
盐渍土又称盐碱土是盐土和碱土以及不同程度盐化和碱化土壤的统称[1], 严重影响植物生长发育和农业生产力的发展[2-3]。盐胁迫会导致植物体内产生渗透胁迫和离子毒害作用, 间接影响植物的光合作用、能量代谢、膜透性和生长发育[4]。水稻是我国四大主粮之一, 种植水稻是生物改良盐碱地的一种有效途径[5], 但在盐碱胁迫下水稻整个生育周期性状都会受到影响, 叶片逐渐变短、变小, 颜色随之变浅[6], 植株生物量和分蘖能力下降, 产量降低[7-8]。随着人口不断增加, 耕地面积逐渐减少, 开发利用盐碱地对增加耕地资源, 改善生态环境具有重要意义[9-10]。
盐碱地修复改良方法主要为水利工程、化学、生物等措施[11], 生物修复包括植物和微生物修复, 具有投入低、修复效果好、生态效益高等优点[12-13]。生物有机肥在盐碱地修复改良中发挥着重要的作用, 既能作为传统有机肥增加土壤有机质和养分、改善物理结构[14-16], 又能外源输入有益微生物, 调节土壤微生物群落结构, 抑制病虫害[17-18], 改善作物水分状况、膜完整性和光合作用[19], 还可以减轻作物盐碱胁迫、提高作物产量及品质[20-22], 减轻化肥过量使用造成的环境污染[23]。
高通量RNA-Seq技术作为揭示植物响应盐胁迫分子机制的重要手段, 已取得了多项重要成果。张丽丽和张富春研究发现[24], 盐生植物盐穗木(Halostachys caspica)能够通过促进渗透调节和活性氧清除, 提高短期盐胁迫适应能力;董明等[25]研究结果表明, 高粱在盐胁迫下光合作用受到抑制, 激素信号和类黄酮的合成在响应盐胁迫中发挥着重要作用;在水稻响应盐胁迫中, 翻译调控[26]、脱落酸和油菜素甾醇等植物激素信号[27-29]、酚类和类黄酮物质合成[30]、Ca2+信号转导、丝裂原活化蛋白激酶(MAPK)级联、转录因子(TFs)、转录调节因子(TRs)、蛋白激酶(PKs)和其他关键功能蛋白起着重要作用[31]。
施用生物有机肥能有效修复改良盐碱地、改善水稻农艺性状[32], 但在基因表达水平上知之甚少。本研究采用转录组测序技术, 对生物有机肥不同处理下水稻叶片差异基因进行GO和KEGG富集分析, 功能和代谢通路注释, 为探究生物有机肥对盐碱地水稻生长影响的生物学机制提供理论基础。
1 材料与方法 1.1 试验地概况本试验地位于宁夏回族自治区银川市贺兰县金贵镇银光村(106.43°E, 38.49°N), 属于温带大陆性季风气候, 全年日照时数2851—3106 h, 有效积温1534.9℃, 年降水量200 mm左右, 时空分布不均, 多集中在6—9月。试验地土壤为淤灌土, pH 8.40、全盐1.83 g/kg、有机质9.9 g/kg、全氮1.18 g/kg、全磷0.59 g/kg、解碱氮42 mg/kg、有效磷3.38 mg/kg、速效钾320 mg/kg, 属于轻度盐碱土。
1.2 试验设计试验采用随机区组排列, 小区面积20 m2(5 m×4 m), 小区之间起垄覆膜, 单独排灌。水稻育苗插秧种植, 品种为宁粳48号。供试生物有机肥配方及(嗜)耐盐菌种均由课题组提供, 宁夏某生态肥业有限公司生产, 配料中加入硅元素制成盐碱地水稻专用生物有机肥[N+P2O5+K2O>5%, 有机质>45.0%, 有效活菌数(枯草芽孢杆菌、多黏类芽孢杆菌、盐单胞菌、乳酸菌、光合细菌等)>500亿/kg];供试化肥为市售磷酸二铵(含P2O5 46%, N 18%)。试验共设置4个处理, T1:生物有机肥+化肥, T2:生物有机肥灭活+化肥, T3:化肥, T4:空白对照(不施肥)。生物有机肥作为基肥, 施用量为1500 kg/hm2;化肥施用量为当地农户施肥习惯的1/2, 施用量为213.75 kg/hm2, 按照基肥、分蘖肥、穗肥5∶3∶2的量施用, 水肥管理与大田一致。
1.3 水稻农艺性状测定水稻灌浆期测定健康植株叶绿素含量、叶面积指数、株高、根长、有效分蘖数, 干物质量。叶绿素含量用SPAD-502便携式叶绿素仪测定;叶长、株高、根长用钢卷尺测量;叶宽用游标卡尺测量;用长宽矫正法来测定叶面积(叶面积指数=叶片长×宽×0.75)[33];干物质量采用烘干法测定, 每个处理取15个重复。
1.4 样品采集取水稻灌浆后期叶片, 用无菌水洗去灰尘后装入2 mL冻存管中, 随即放入液氮中速冻, 干冰送样, 委托北京诺禾致源科技股份有限公司完成RNA的提取、质控、建库及Illumina HiSeqTM测序, 每个处理取3次重复。
1.5 文库构建及测序提取样品RNA, 总量≥1 μg, 通过Oligo(dT)磁珠富集水稻叶片中带有poly A尾的mRNA, 随后在NEB Fragmentation Buffer中用二价阳离子将得到的mRNA随机打断。以片段化的mRNA为模版, 随机寡核苷酸为引物, 在M-MuLV逆转录酶体系中合成cDNA第一条链, 然后用R NaseH降解RNA链, 并在DNA polymerase I体系下, 以dNTPs为原料合成cDNA第二条链。纯化后的双链cDNA经过末端修复, 加A尾并连接测序接头, 用AMPure XP beads筛选250—300 bp左右的cDNA, 进行PCR扩增并再次使用AMPure XP beads纯化PCR产物, 最终获得文库, 然后进行转录组测序。
1.6 数据处理与分析利用clusterProfiler 3.4.4软件进行差异表达基因的GO和KEGG富集分析, P < 0.05作为显著性富集的阈值;利用SPSS软件对叶绿素相对含量、有效分蘖数、根长、叶面积和和干物质重量进行方差分析、多重比较, 株高进行非参数检验。
2 结果与分析 2.1 不同生物有机肥处理对水稻生长的影响由表 1可知, 施用生物有机肥+化肥后, 水稻叶片中叶绿素含量、有效分蘖数、根长、叶面积和干物质重量均显著高于化肥和空白对照(P < 0.05), 株高也大于其他处理, 说明施用生物有机肥能有效促进盐碱地水稻生长。
| 处理 Treatment |
叶绿素相对含量 Relative content of chlorophyll |
有效分蘖数 Number of effective tillers/(个/株) |
株高 Plant height/cm |
根长 Root length/cm |
叶面积 Leaf area/cm2 |
干物质重量 Dry matter weight/g |
| T1 | 43.95±3.61Aa | 2.67±0.72Aa | 98.89±4.99Aa | 18.14±2.45Aa | 22.23±5.95Aa | 15.65±4.08Aa |
| T2 | 41.33±3.37ABa | 2.07±0.70ABb | 98.33±7.76Aa | 16.50±2.34ABab | 25.50±5.41Aa | 14.08±4.06ABab |
| T3 | 38.19±3.54BCb | 1.67±0.62Bbc | 91.90±8.52Bb | 15.80±3.22ABb | 13.82±4.8Bb | 11.71±2.83BCbc |
| T4 | 36.63±4.37Cb | 1.53±0.64Bc | 81.33±8.12Cc | 14.73±2.12Bb | 12.21±4.53Bb | 10.51±2.37Cc |
| T1:生物有机肥+化肥Bio-organic fertilizer+chemical fertilizer;T2:生物有机肥灭活+化肥Bio-organic fertilizer inactivation+chemical fertilizer;T3:化肥Chemical fertilizer;T4:空白对照Blank control; 表中数据为平均值±标准差, 同列数据不同大、小写字母分别表示不同处理间差异达0.01、0.05水平; Q20:测序质量值≥20的碱基所占的百分比Percentage of bases with a sequencing quality value greater than or equal to 20;Q30:测序质量值≥30的碱基所占的百分比Percentage of bases with a sequencing quality value greater than or equal to 30;GC: G和C两种碱基所占总碱基的百分比Percentage of G and C bases in total bases | ||||||
为了保证数据分析的质量, 在分析前对原始数据进行处理, 去掉带接头、含无法确定碱基信息、低质量(Qphred≤20的碱基数占整个read长度的50%以上)的reads, 统计见表 2。整体上, 每个样品最终得到4000—7000万条Clean reads, Q20为96.86%—98.33%, Q30为91.87%—95.09%, GC含量为50.04%—55.22%, 数据整体测序错误率为0.03%, 小于1%, 表明测序质量良好, 数据可用于后续生物信息学分析。
| 样品名 Sample |
原始read Total raw reads |
过滤后read/% Total clean reads |
测序错误率/% error rate |
Q20比例 Q20% |
Q30比例 Q30% |
GC比例 GC% |
| T1-1 | 64088278 | 63393898(98.92%) | 0.03 | 96.92 | 91.91 | 51.23 |
| T1-2 | 50907448 | 50243748(98.7%) | 0.03 | 97.29 | 92.64 | 51.56 |
| T1-3 | 56069862 | 55243950(98.53%) | 0.03 | 97.45 | 92.97 | 51.32 |
| T2-1 | 57616424 | 56456528(97.99%) | 0.03 | 97.29 | 92.68 | 52.08 |
| T2-2 | 53123648 | 52425186(98.69%) | 0.03 | 97.54 | 93.18 | 50.04 |
| T2-3 | 42212280 | 40813840(96.69%) | 0.02 | 98.33 | 95.09 | 50.81 |
| T3-1 | 42482600 | 41989962(98.84%) | 0.03 | 96.95 | 91.89 | 52.86 |
| T3-2 | 47412764 | 46801644(98.71%) | 0.03 | 97.12 | 92.28 | 51.14 |
| T3-3 | 50198746 | 49472502(98.55%) | 0.03 | 97.30 | 92.60 | 50.51 |
| T4-1 | 45064262 | 44278554(98.26%) | 0.03 | 97.29 | 92.6 | 50.9 |
| T4-2 | 46393136 | 45339886(97.73%) | 0.03 | 96.86 | 91.87 | 51.91 |
| T4-3 | 58827290 | 57934848(98.48%) | 0.03 | 96.94 | 92.01 | 51.28 |
| T1-1、T1-2、T1-3、T2-1、T2-2、T2-3、T3-1、T3-2、T3-3、T4-1、T4-2、T4-3分别为不同处理的3个生物学重复. | ||||||
利用DESeq 2软件比较生物有机肥不同处理下水稻叶片的基因, 以P < 0.05且|log2FoldChange|>0.0作为筛选基准, 图 1为差异基因表达火山图, T2和T1之间检测到差异基因6593条, 说明生物有机肥中的微生物在参与调控水稻叶片基因表达中发挥了重要作用;T4和T3之间的1866条差异基因是由施用化肥引起的, T3和T1之间的4796条差异基因是由生物有机肥引起的, 说明生物有机肥对水稻叶片基因调控作用大于化肥;T4和T1之间检测到差异基因6976条, 高于T4vsT3与T3vsT1差异基因数量之和, 说明生物有机肥与化肥对水稻基因调控作用具有协同作用。生物有机肥对水稻基因表达具有重要意义, 因此对其进一步进行GO功能富集与KEGG富集分析。
|
| 图 1 基因火山图 Fig. 1 Volcano map of gene T1:生物有机肥+化肥Bio-organic fertilizer+chemical fertilizer;T2:生物有机肥灭活+化肥Bio-organic fertilizer inactivation+chemical fertilizer;T3:化肥Chemical fertilizer;T4:空白对照Blank control; vs: 两组样品中的基因表达比较, 将vs之前的定义为表达量为1; FC: 表示两样品(组)间表达量的比值Fold Change |
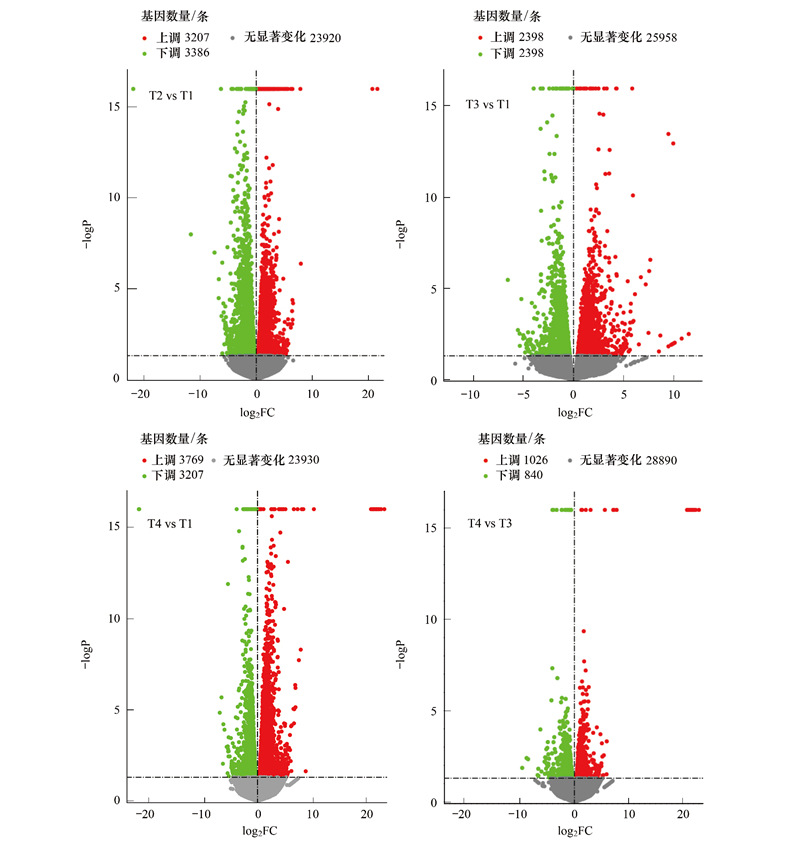
将得到的目标基因进行GO功能富集分析能够确定差异基因的主要生物学功能, 即生物学过程、细胞组分、分子功能三个主类。T2vsT1结果注释差异基因共4780条, 其中生物学过程1643条, 细胞组成635条, 分子功能2502条, 分别占比34.37%、13.28%、52.34%, 由此可以看出大多数差异基因与分子功能显著相关, 各组分最显著的10个Term见图 2, 生物学过程方面主要表现在翻译、肽生物合成和代谢、酰胺生物合成和代谢、有机氮化合物的生物合成、DNA构象变化等方面;在细胞组分中, 主要参与核糖体、细胞内核糖核蛋白复合物、核糖核蛋白复合体、非膜细胞器、细胞内非膜细胞器、细胞质部分、细胞质等方面;在分子功能中, 主要集中在核糖体的结构成分和结构分子活性等方面。
|
| 图 2 T2vsT1差异基因本体功能富集分析 Fig. 2 T2vsT1 differential gene ontology function enrichment analysis |
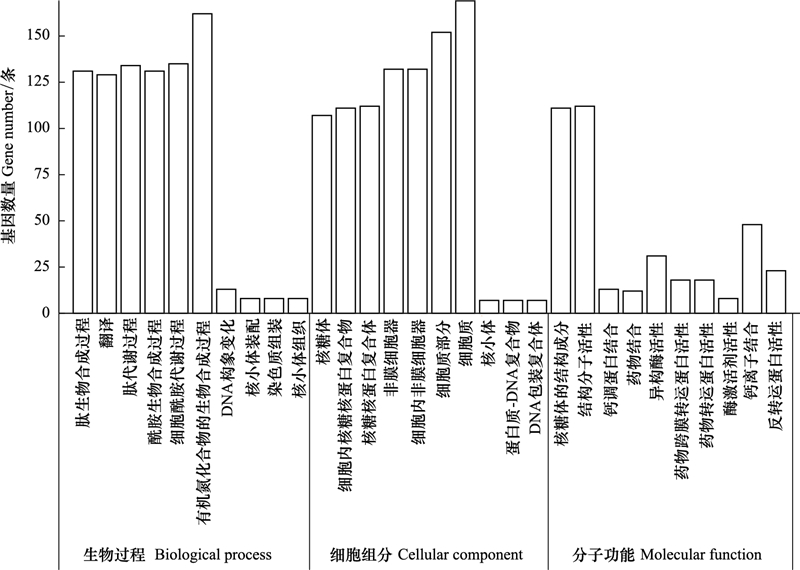
T3vsT1共注释到差异基因3829条, 生物学过程1335条, 细胞组成506条, 分子功能1988条, 分别占比34.87%、13.21%、51.92%, 可以看出施用生物有机肥影响水稻叶片的分子功能, 各组分最显著的10个Term见图 3, 生物学过程主要表现在光合作用、翻译、肽生物合成与代谢、酰胺生物合成与代谢;细胞组分中, 主要参与类囊体、类囊体部分、光系统、膜蛋白复合物、光合膜、核糖体、光系统II、高尔基体相关囊泡、高尔基体相关囊泡膜、细胞质部分、核糖核蛋白复合体、细胞质和细胞内核糖核蛋白复合物等方面;在分子功能中, 主要集中在结构分子活性、核糖体的结构成分、转录因子活性, 序列特异性DNA结合、核酸结合转录因子活性等方面。
|
| 图 3 T3vsT1差异基因本体功能富集分析 Fig. 3 T3vsT1 differential gene ontology function enrichment analysis |
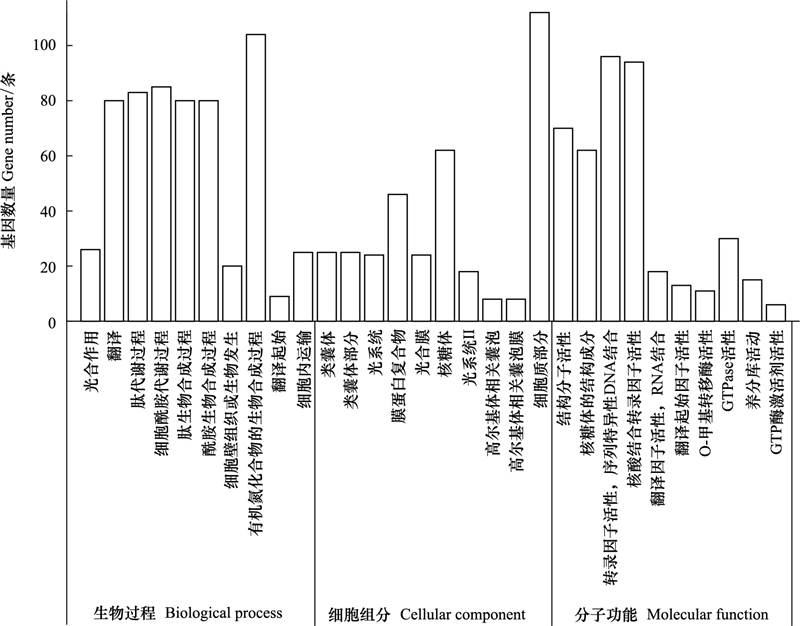
T4vsT1共注释到差异基因5252条, 生物学过程1840条, 细胞组成731条, 分子功能2681条, 分别占比35.03%、13.92%、51.05%, 说明生物有机肥与化肥联合作用能有效促进叶片基因差异表达, 各组分最显著的10个Term见图 4, 生物学过程主要表现在翻译、肽生物合成与代谢、细胞酰胺生物合成与代谢、有机氮化合物的生物合成、光合作用、蛋白质转运等方面;细胞组分中, 主要参与核糖体、核糖核蛋白复合体、细胞内核糖核蛋白复合物、细胞质部分、细胞质、非膜细胞器、细胞内非膜细胞器等方面;在分子功能中, 主要集中在结构分子活性、核糖体的结构成分、RNA结合、翻译因子活性, RNA结合、rRNA结合等方面。
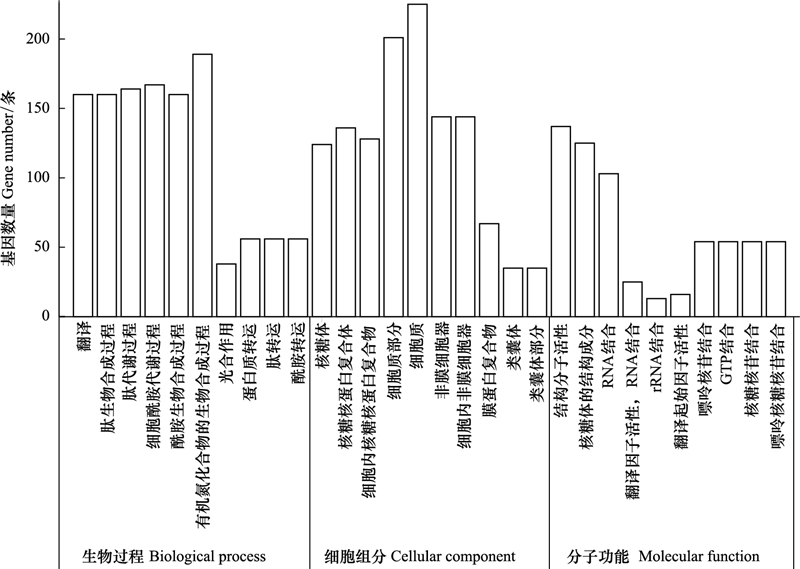
|
| 图 4 T4vsT1差异基因本体功能富集分析 Fig. 4 T4vsT1 differential gene ontology function enrichment analysis |
T4vsT3共注释到差异基因1396条, 生物过程523条, 细胞组成131条, 分子功能742条, 分别占比37.46%、9.38%、53.15%, 差异显著的基因集中在细胞组分和分子功能, 可以看出施用化肥主要对水稻叶片的生物学过程影响较小, 各组分最显著的10个Term见图 5。在细胞组分中, 差异基因主要参与的生物学过程包括类囊体、类囊体部分、光合膜等方面;在分子功能中, 差异基因主要集中在血红素结合、四吡咯结合、氧化还原酶活性作用于成对的供体上并结合或还原分子氧、铁离子结合等方面。
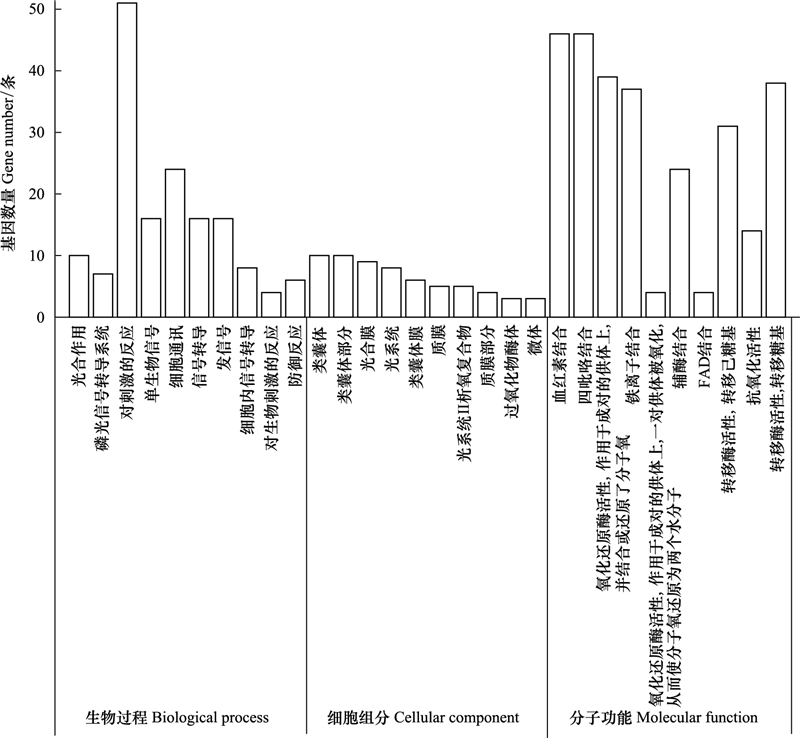
|
| 图 5 T4vsT3差异基因本体功能富集分析 Fig. 5 T4vsT3 differential gene ontology function enrichment analysis |
为了进一步表达差异基因的生物学行为, 根据KEGG数据库注释结果, 对生物有机肥不同处理下, 盐碱地水稻叶片差异基因进行分析。
T2vsT1共有965条差异基因注释到KEGG数据库中, 分布在112个分类代谢途径中, 最显著的20个通路见图 6, 涉及差异基因最多的代谢通路是核糖体(142条), 其次是植物激素信号转导(55条)、剪接体(47条)、植物-病原体相互作用(45条)、氨基酸生物合成(41条)、碳代谢(40条)、嘌呤代谢(38条)、MAPK信号传导途径-植物(35条)、内质网中蛋白质加工(34条)、胞吞作用(30条)等, 其中有2个代谢通路显著富集(P < 0.05), 分别是核糖体和光合作用(22条)。
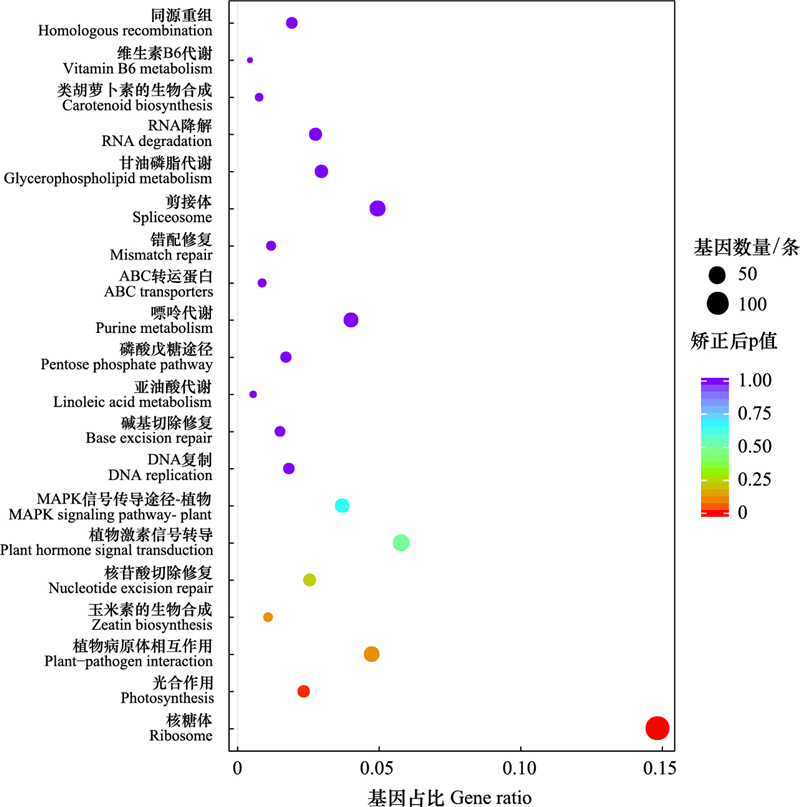
|
| 图 6 T2vsT1差异基因KEGG富集通路 Fig. 6 T2vsT1 differential gene KEGG enrichment pathway KEGG: 京都基因与基因组百科全书Kyoto Encyclopedia of Genes and Genomes; ABC: 转运蛋白的全称为腺苷三磷酸结合盒转运蛋白(ATP-Binding cassett)(ATP binding cassett transporters); MAPK: 丝裂原激活的蛋白激酶Mitogen-activated protein kinase |
T3vsT1共有732条差异基因得到注释, 分布在109个分类代谢途径中, 最显著的20个通路见图 7, 涉及差异基因最多的代谢通路是核糖体(81条), 其次是碳代谢(43条)、植物激素信号转导(40条)、内质网中蛋白质加工(32条)、氨基酸生物合成(31条)、谷胱甘肽代谢(30条)、氨基糖和核苷酸糖代谢(26条)、苯丙烷生物合成(26条)、MAPK信号传导途径-植物(25条)、半胱氨酸和蛋氨酸代谢(24条)等, 其中有4个代谢通路显著富集(P < 0.05), 分别是核糖体、光合作用、光合作用-天线蛋白和谷胱甘肽代谢。
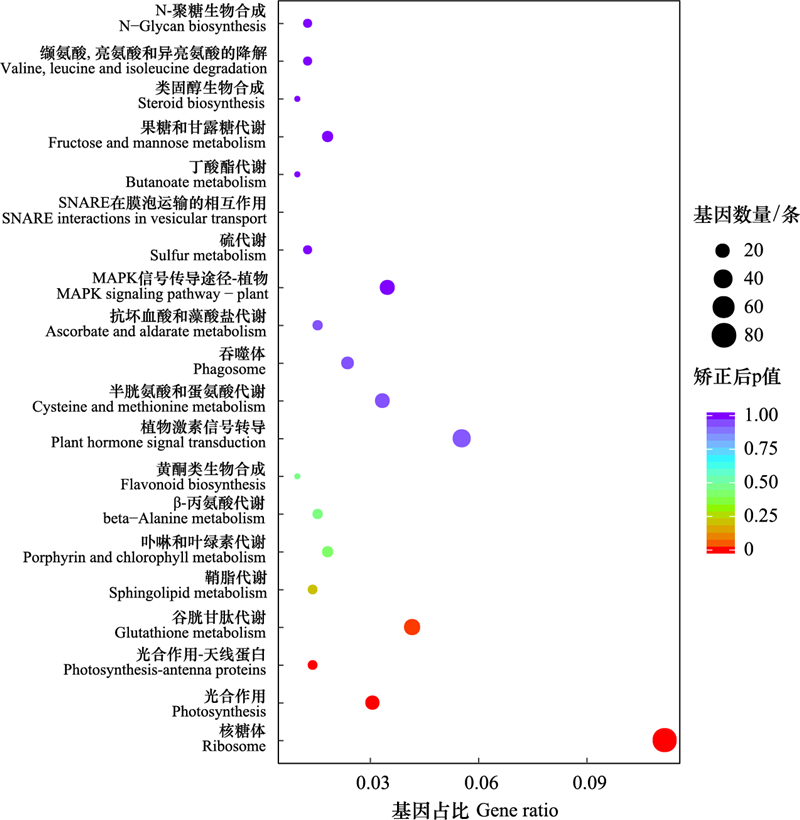
|
| 图 7 T3vsT1差异基因KEGG富集通路 Fig. 7 T3vsT1 differential gene KEGG enrichment pathway |
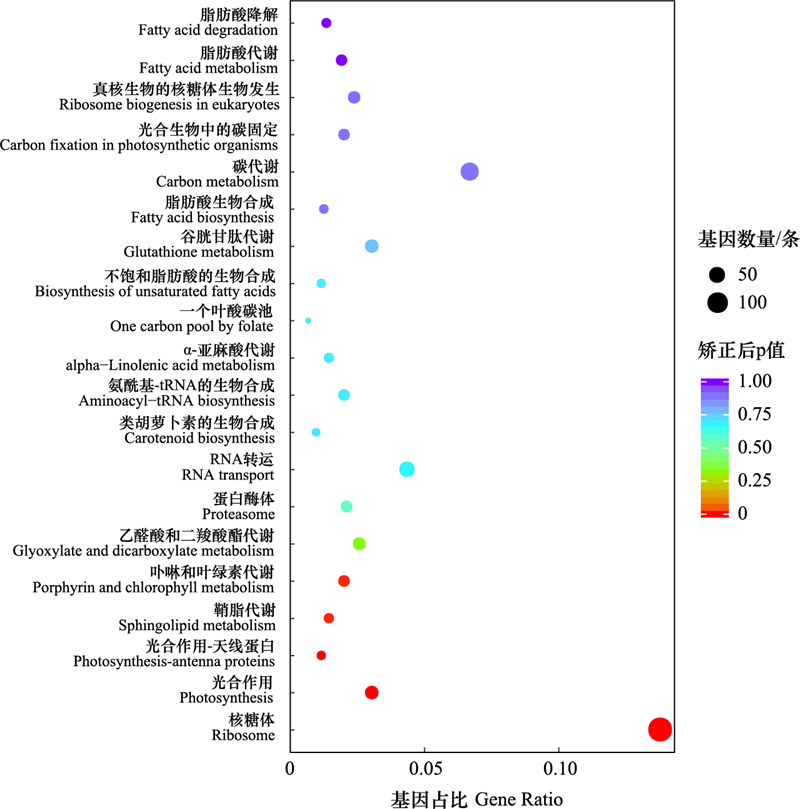
T4vsT1共有1076条差异基因得到注释, 分布在109个分类代谢途径中, 最显著的20个通路见图 8, 涉及差异基因最多的代谢通路是核糖体(147条)、碳代谢(71条)、氨基酸生物合成(48条)、RNA转运(46条)、内质网中蛋白质加工(45条)、嘌呤代谢(35条)、剪接体(35条)、植物激素信号转导(34条)、光合作用(32条)、谷胱甘肽代谢(32条)等, 有5个代谢通路显著富集(P < 0.05), 分别是核糖体、光合作用、光合作用-天线蛋白、鞘脂代谢、卟啉与叶绿素代谢。
|
| 图 8 T4vsT1差异基因KEGG富集通路 Fig. 8 T4vsT1 differential gene KEGG enrichment pathway |
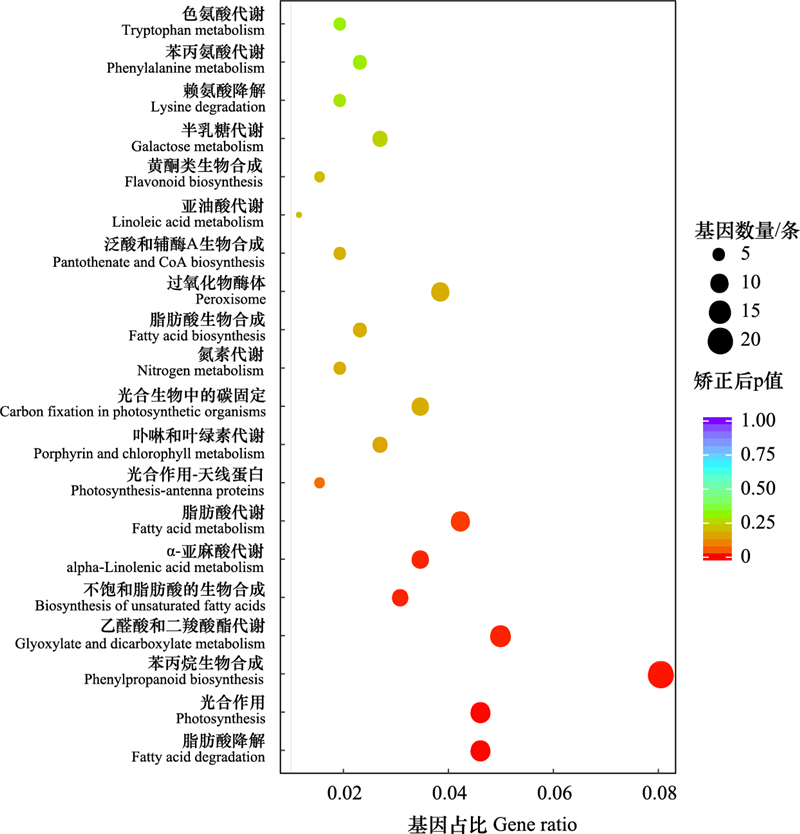
T4vsT3共有263条差异基因得到注释, 共100条代谢通路, 最显著的20个通路见图 9, 涉及差异基因最多的代谢通路是苯丙烷生物合成(21条)、碳代谢(21条)、植物激素信号转导(17条)、氨基酸生物合成(17条)、乙醛酸和二羧酸代谢(13条)、脂肪酸降解(12条)、光合作用(12条)、糖酵解/糖异生(12条)、脂肪酸代谢(11条)、MAPK信号传导途径-植物(11条)等, 有7个代谢通路显著富集(P < 0.05), 是脂肪酸降解、光合作用、苯丙烷生物合成、乙醛酸和二羧酸代谢、不饱和脂肪酸生物合成、α-亚麻酸代谢、脂肪酸代谢。
|
| 图 9 T4vsT3差异基因KEGG富集通路 Fig. 9 T4vsT3 differential gene KEGG enrichment pathway |
各组差异基因KEGG显著富集通路统计见表 3, 结果表明, 施用生物有机肥后, 核糖体和光合作用代谢通路显著富集(P < 0.05), 与前文GO分析中核糖体和光合作用相互佐证, 说明盐碱地施用生物有机肥诱导水稻叶片中的RNA合成蛋白质, 对其光合作用具有显著影响;谷胱甘肽是生物体内最主要的非蛋白巯基和含量最丰富的低分子量多肽, 具有很强的抗氧化性, 参与植物抗逆中的多项功能活动[34], 在本研究中, T3vsT1和T4vsT1中谷胱甘肽代谢通路基因上调23和27条, 下调7和5条, 说明生物有机肥能够提高盐碱地水稻抗逆性。
| 组别Group | 代谢通路Metabolic pathway | 数量Count | 上调Up | 下调Down |
| T2vsT1 | 核糖体 | 142 | 141 | 1 |
| 光合作用 | 22 | 19 | 3 | |
| T3vsT1 | 核糖体 | 81 | 15 | 66 |
| 光合作用 | 22 | 22 | 0 | |
| 光合作用-天线蛋白 | 10 | 10 | 0 | |
| 谷胱甘肽代谢 | 30 | 23 | 7 | |
| T4vsT1 | 核糖体 | 147 | 32 | 115 |
| 光合作用 | 32 | 31 | 1 | |
| 光合作用-天线蛋白 | 12 | 12 | 0 | |
| 鞘脂代谢 | 15 | 4 | 11 | |
| 卟啉与叶绿素代谢 | 21 | 18 | 3 | |
| T4vsT3 | 脂肪酸降解 | 12 | 3 | 9 |
| 光合作用 | 12 | 12 | 0 | |
| 苯丙烷生物合成 | 21 | 10 | 11 | |
| 乙醛酸和二羧酸的代谢 | 13 | 6 | 7 | |
| 不饱和脂肪酸的生物合成 | 8 | 5 | 3 | |
| α-亚麻酸代谢 | 9 | 2 | 7 | |
| 脂肪酸代谢 | 11 | 5 | 6 |
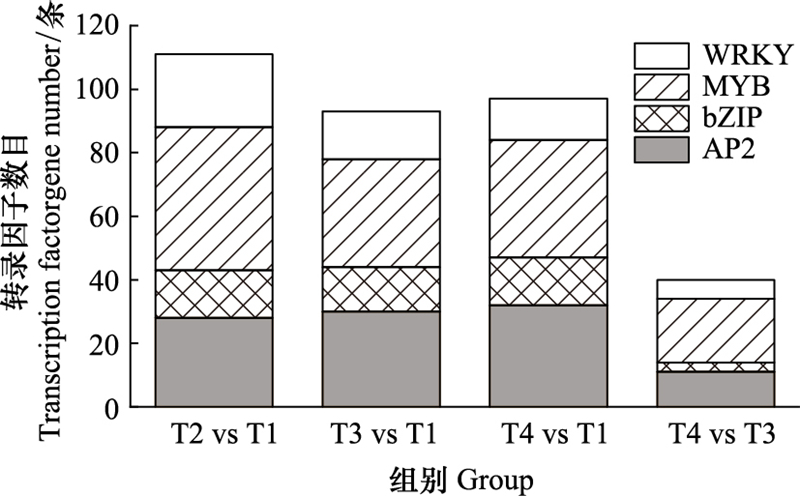
转录因子是细胞响应外界胁迫以后主要调控某些基因转录的调控因子[35]。目前, 已鉴定出参与植物盐胁迫应答反应的转录因子有AP2/EREBP、MYB、WRKY以及bZIP转录因子家族成员[36]。图 10为各组差异基因注释在转录因子家族的基因数量, T2vsT1中4个转录因子家族共有111条差异基因, 其中上调39条(MYB家族23条), 下调72条(AP2家族23条, MYB家族22条, WRKY家族21条);T3vsT1共有93条差异基因, 上调44条(AP2家族10条, bZIP家族10条, MYB家族15条), 下调49条(AP2家族20条, MYB家族15条, WRKY家族10条);T4vsT1共有97条差异基因, 上调53条(AP2家族19条, bZIP家族11条, MYB家族18条), 下调44条(AP2家族13条, MYB家族19条), T4vsT3共有40条差异基因, 上调24条(AP2家族11条), 下调18条(MYB家族13条)。
|
| 图 10 响应盐胁迫家族基因数量统计 Fig. 10 Number statistics of family genes in response to salt stress |
转录组学是功能基因组学的重要组成部分, 它是从整体水平上研究细胞中基因转录的情况及其转录调控规律[37]。植物对逆境胁迫的响应机制十分复杂, 当细胞感受到逆境胁迫时, 相应的受体将会收到信号, 控制相关的基因表达, 并产生生理反应, 同时在转录和翻译等水平做出应答[38]。本研究中, 从水稻农艺性状可以看出, 施用生物有机肥能有效减轻盐分对水稻的胁迫, 促进盐碱地水稻生长。对不同生物有机肥处理下盐碱地水稻叶片进行转录测序分析, Q30碱基百分比大于91.87及以上, 表明测序质量良好, 满足转录组分析的基本要求。
施肥不仅可以开启一些基因上调表达, 同时也可以关闭一些基因表达(基因沉默), 还可以调节一些基因下调表达[39]。王蓥燕[40]在NPK定位实验中发现, 化肥配施农家肥能明显促进16S rRNA和nosZ基因拷贝数。本研究GO功能分析中, T2vsT1中差异基因引起显著差异的功能有16个, T3vsT1中有36个, T4vsT1中有60个, 说明施用生物有机肥能有效刺激水稻叶片基因差异表达, 这些差异基因在生物学过程中细胞学过程、代谢过程相关基因分布较多;细胞组分中主要集中在细胞器、细胞器膜;在分子功能中差异显著表达基因与结合、催化活性有关。张敏瑜[41]接种AM真菌和Foc4能诱导巴西蕉的基因差异表达, 且2株菌同时接种时差异基因显著表达量大于单株菌的表达量。Chandra研究结果表明[42], 耐盐解淀粉芽孢杆菌能够调节水稻基因差异表达提高水稻耐盐性。本研究中, T2vsT1中差异显著上调基因均大于下调数量, 说明生物有机肥中的枯草芽孢杆菌等(嗜)耐盐菌株对盐碱地水稻生物学过程具有重要的影响。
核糖体是最古老的、精细复杂的细胞器, 其结构和组成从原核到真核保持着高度的保守性[43], 是细胞机制的一个重要部分, 负责蛋白质合成, 在控制细胞生长、分裂和发育中起着重要作用[44], 核糖体相关基因表达可能会影响生物体的一些机能。谢丽霞[45]从极端嗜盐曲霉核糖体中克隆出Sp RPS3ae和Sp RPL44两条蛋白基因导入植物和微生物中均能有效提高其抗盐性, 夏丽丹等[46]在分析盐肤木对铅胁迫的响应时发现, 核糖体相关基因是盐肤木应对铅胁迫的主要调节基因。在本研究中, 差异表达基因的GO与KEGG的分析中均发现, 核糖体相关基因在T2vsT1、T3vsT1、T4vsT1中均差异表达, 说明核糖体内可能是盐碱地水稻施用生物有机肥后应对盐碱环境的重要调节基因。
光合作用是植物产生能量的主要途径, 叶片是植物进行光合作用的重要器官, 对植物的生长发育至关重要[25]。但盐碱胁迫会造成植物细胞膜损伤、气孔关闭、CO2同化速率降低, 从而降低光合速率、蒸腾速率、气孔开放、细胞间CO2浓度和叶绿素含量影响水稻生长发育, 从而影响作物产量[47-48]。本研究中KEGG代谢途径的生物信息学分析发现, T3vsT1和T4vsT1中光合作用和光合作用-天线蛋白代谢通路基因均显著上调, 而光合作用-天线蛋白是光反应中原初反应中的一类补光蛋白复合体, 能够捕获光能并把能量传递至反应中心[25], 说明施用生物有机肥能有效促进盐碱地水稻叶片对光能的捕获, 从而增强水稻光合作用。
植物激素不但调控植物的生长发育, 而且还参与植物的非生物胁迫[25], 在T2vsT1、T3vsT1和T4vsT1中分别有55条、40条、34条差异基因注释在植物激素信号传导通路上, 涉及的植物激素有生长素、细胞分裂素、赤霉素、脱落酸、乙烯、油菜素甾醇、茉莉素和水杨酸, 意味着施用生物有机肥对植物激素具有一定影响, 脱落酸是植物利用类胡萝卜素参与合成的一种应激反应激素[49], 能激发质膜结合通道或从细胞内Ca2+储存库释放Ca2+与第二信使Ca2+整合, 帮助植物在盐胁迫下存活[50], 在植物应对环境胁迫中发挥着重要作用[51]。在本研究中类胡萝卜素生物合成通路基因在T3vsT1中上调5条、下调1条, 在T4vsT1中上调10条, 无下调基因, 说明盐碱地施用生物有机肥能促进类胡萝卜素生物合成, 促进水稻抗性提高。
植物经常受到病原性细菌、真菌、病毒、线虫等的攻击[52], 但是植物具有天生的免疫系统来抵御病原体[53], 当受到病原体攻击时, 每个细胞都能够自主识别和应对病原体[54]。枯草芽孢杆菌能有效防治水稻的纹枯病[55]、稻瘟病[56]等病害, 本研究中T3vsT1和T4vsT1中植物-病原体相互作用代谢通路上调基因数量大于下调基因数量, 说明施用生物有机肥在一定程度上能够激活盐碱地水稻叶片自身对病原体的抗性, 微生物在其中可能发挥了重要作用, 在T2中对生物有机肥进行了灭活处理, 但在T2vsT1中上调基因数量小于下调基因, 其原因需要进一步研究。
参与响应盐胁迫的转录因子在盐胁迫下呈现出不同的应答反应模式, 表明这些转录因子在盐胁迫应答途径中扮演着不同的角色[36]。AP2/EREBP是一个大的转录因子家族, 包含EREBP和AP2等2个亚族。研究证实, AP2/EREBP转录家族基因与植物逆境响应有关[57], MYB蛋白在包括盐胁迫在内的植物非生物胁迫调控中有重要作用[58], WRKY转录因子具有高度保守的WRKY结构域, 能够参与损伤、衰老、发育、抗病等抗逆反应[59]。不同生物有机肥处理诱导盐胁迫下水稻叶片响应盐胁迫的转录因子家族基因开始表达, 其中施生物有机肥能促使水稻叶片已知响应盐胁迫的家族基因差异表达, 主要参与盐胁迫响应的基因都属于AP2和MYB转录因子家族, 说明施用生物有机肥能有效促进植物响应盐胁迫基因表达, 增强作物耐受性。
4 结论以生物有机肥不同处理下的盐碱地水稻叶片为研究材料, 运用RNA-Seq技术进行转录组测序, 并对差异基因进行GO和KEGG代谢通路富集分析, 解析其参与的生物学功能和代谢途径。施用生物有机肥能够改变盐碱地水稻基因表达, 从而调控水稻的生物学功能和代谢过程, 影响水稻的生长发育。本研究初步揭示了生物有机肥施用对盐碱地水稻促生和抗逆作用的生物学机制。
| [1] |
王遵亲. 中国盐渍土. 北京: 科学出版社, 1993.
|
| [2] |
Ahmad H R, Aziz T, Hussain S, Akraam M, Sabir M, Kashif S U R, Hanafi M M. Zinc-enriched farm yard manure improves grain yield and grain zinc concentration in rice grown on a saline-sodic soil. International Journal of Agriculture & Biology, 2012, 14(5): 787-792. |
| [3] |
杨海霞, 郭绍霞, 刘润进. 盐碱地生境中丛枝菌根真菌多样性与功能变化特征. 应用生态学报, 2015, 26(1): 311-320. |
| [4] |
吉福桑, 杨振, 徐亚, 汪奇, 李新国, 乔飞. 盐胁迫下巴西蕉叶片的转录组和蛋白质组关联分析. 分子植物育种, 2020, 18(23): 7671-7678. |
| [5] |
罗成科, 肖国举, 张峰举, 李茜. 不同浓度复合盐胁迫对水稻产量和品质的影响. 干旱区资源与环境, 2017, 31(1): 137-141. |
| [6] |
陈丽珍, 叶剑秋, 王荣香. 水稻盐胁迫的研究进展. 热带农业科学, 2011, 31(3): 87-93. DOI:10.3969/j.issn.1009-2196.2011.03.019 |
| [7] |
吕海艳. 盐碱胁迫对水稻根系形态特征及产量的影响[D]. 长春: 中国科学院研究生院(东北地理与农业生态研究所), 2014.
|
| [8] |
梁正伟, 杨福, 王志春, 陈渊. 盐碱胁迫对水稻主要生育性状的影响. 生态环境, 2004, 13(1): 43-46. DOI:10.3969/j.issn.1674-5906.2004.01.014 |
| [9] |
尹志荣, 黄建成, 桂林国. 稻作条件下不同施肥模式对原土盐碱地的改良培肥效应. 土壤通报, 2016, 47(2): 414-418. |
| [10] |
王才林, 张亚东, 赵凌, 路凯, 朱镇, 陈涛, 赵庆勇, 姚姝, 周丽慧, 赵春芳, 梁文化, 孙明法, 严国红. 耐盐碱水稻研究现状、问题与建议. 中国稻米, 2019, 25(1): 1-6. DOI:10.3969/j.issn.1006-8082.2019.01.001 |
| [11] |
杨帆, 王志春, 马红媛, 杨福, 田春杰, 安丰华. 东北苏打盐碱地生态治理关键技术研发与集成示范. 生态学报, 2016, 36(22): 7054-7058. |
| [12] |
张涛, 李素艳, 孙向阳, 王振宇, 张洋, 张乐, 龚小强, 赵秀芳, 谢志远, 宋国香. 磷石膏、红糖等对蚯蚓改良滨海盐土的促进作用. 土壤学报, 2017, 54(1): 255-264. |
| [13] |
张永宏, 吴秀梅, 班乃荣, 雷金银. 盐碱地的生物修复研究. 农业科技通讯, 2009(7): 99-101. DOI:10.3969/j.issn.1000-6400.2009.07.043 |
| [14] |
Chavada N B, Patel R, Vanpuria S, Raval B P, Thakkar P V. A study on isolated diazotrophic (Non-Symbiotics) bacteria from saline desert soil as a Biofertilizer. International Journal of Pharmaceutical and Applied Sciences, 2010, 1(1): 52-54. |
| [15] |
李晓爽. 掺沙及施用生物有机肥对盐碱地水盐运移和冬小麦生长发育影响的研究[D]. 北京: 中国农业科学院, 2020.
|
| [16] |
张金柱, 郭春景, 张兴, 王金刚, 车代弟. 生物有机肥对中度盐碱土理化性质影响的研究. 湖北农业科学, 2008, 47(12): 1420-1422. DOI:10.3969/j.issn.0439-8114.2008.12.014 |
| [17] |
Zheng X F, Zhu Y J, Wang Z R, Zhang H F, Chen M C, Chen Y P, Wang J P, Liu B. Effects of a novel bio-organic fertilizer on the composition of rhizobacterial communities and bacterial wilt outbreak in a continuously mono-cropped tomato field. Applied Soil Ecology, 2020, 156: 103717. DOI:10.1016/j.apsoil.2020.103717 |
| [18] |
Luo J, Ran W, Hu J, Yang X M, Xu Y C, Shen Q R. Application of bio-organic fertilizer significantly affected fungal diversity of soils. Soil Science Society of America Journal, 2010, 74(6): 2039-2048. DOI:10.2136/sssaj2009.0437 |
| [19] |
Chauhan P S, Lata C, Tiwari S, Chauhan A S, Mishra S K, Agrawal L, Chakrabarty D, Nautiyal C S. Transcriptional alterations reveal Bacillus amyloliquefaciens-rice cooperation under salt stress. Scientific Reports, 2019, 9(1): 11912. DOI:10.1038/s41598-019-48309-8 |
| [20] |
Subowo Y B. Penambahan pupuk hayati jamur sebagai pendukung pertumbuhan tanaman padi (Oryza sativa) pada tanah salin. Prosiding Seminar Nasional Masyarakat Biodiversitas Indonesia, 2015, 1(1): 150-154. |
| [21] |
Fu R M, Xing W H, Zhang H, Wu X W, Xue T T, Gu Y N, Wang Y Y, Yu F, Du M L, Chen W L. Isolation and screening of halo-tolerant strains and their application in saline soil remediation. Journal of Simulation, 2015, 3(2): 24-28. |
| [22] |
李北齐, 王倡宪, 孟瑶, 刘杜金, 孟庆有, 李新达, 王贵强. 生物有机肥对盐碱土壤养分及玉米产量的影响. 中国农学通报, 2011, 27(21): 182-186. |
| [23] |
El-Shahat R M, Sherif A E A, Mohamed F M. Response of barley grown in saline soil to bio-fertilizer as a partial substitutive of mineral fertilizer. Global Journal of Scientific Researches, 2014, 2(5): 144-153. |
| [24] |
张丽丽, 张富春. 短期盐胁迫下盐穗木的转录组分析. 植物研究, 2018, 38(1): 91-99. |
| [25] |
董明, 再吐尼古丽·库尔班, 吕芃, 杜瑞恒, 叶凯, 侯升林, 刘国庆. 高粱苗期耐盐性转录组分析和基因挖掘. 中国农业科学, 2019, 52(22): 3987-4001. DOI:10.3864/j.issn.0578-1752.2019.22.005 |
| [26] |
Li Y F, Zheng Y, Vemireddy L R, Panda S K, Jose S, Ranjan A, Panda P, Govindan G, Cui J X, Wei K N, Yaish M W, Naidoo G C, Sunkar R. Comparative transcriptome and translatome analysis in contrasting rice genotypes reveals differential mRNA translation in salt-tolerant Pokkali under salt stress. BMC Genomics, 2018, 19(S10): 935. DOI:10.1186/s12864-018-5279-4 |
| [27] |
楚乐乐, 罗成科, 李芳兰, 路旭平, 马天利, 李培富. 盐胁迫下OsDSR2 RNAi转基因水稻的生理特性及转录组学分析. 植物遗传资源学报, 2020, 21(4): 954-965. |
| [28] |
栗露露, 殷文超, 牛梅, 孟文静, 张晓星, 童红宁. 油菜素甾醇调控水稻盐胁迫应答的作用研究. 植物学报, 2019, 54(2): 185-193. |
| [29] |
Wang W S, Zhao X Q, Li M, Huang L Y, Xu J L, Zhang F, Cui Y R, Fu B Y, Li Z K. Complex molecular mechanisms underlying seedling salt tolerance in rice revealed by comparative transcriptome and metabolomic profiling. Journal of Experimental Botany, 2016, 67(1): 405-419. DOI:10.1093/jxb/erv476 |
| [30] |
Chandran A K N, Kim J W, Yoo Y H, Park H L, Kim Y J, Cho M H, Jung K H. Transcriptome analysis of rice-seedling roots under soil-salt stress using RNA-Seq method. Plant Biotechnology Reports, 2019, 13(6): 567-578. DOI:10.1007/s11816-019-00550-3 |
| [31] |
Kong W L, Zhong H, Gong Z Y, Fang X Y, Sun T, Deng X X, Li Y S. Meta-analysis of salt stress transcriptome responses in different rice genotypes at the seedling stage. Plants, 2019, 8(3): 64. DOI:10.3390/plants8030064 |
| [32] |
曹力毅. 微生物肥料对盐碱地水稻生长及其土壤环境的影响[D]. 银川: 宁夏大学, 2019.
|
| [33] |
陶洪斌, 林杉. 打孔称重法与复印称重法和长宽校正法测定水稻叶面积的方法比较. 植物生理学通讯, 2006, 42(3): 496-498. |
| [34] |
麦维军, 王颖, 梁承邺, 张明永. 谷胱甘肽在植物抗逆中的作用. 广西植物, 2005, 25(6): 570-575. DOI:10.3969/j.issn.1000-3142.2005.06.013 |
| [35] |
马进, 郑钢. 利用转录组测序技术鉴定紫花苜蓿根系盐胁迫应答基因. 核农学报, 2016, 30(8): 1470-1479. |
| [36] |
朱冬梅, 贾媛, 崔继哲, 付畅. 植物对盐胁迫应答的转录因子及其生物学特性. 生物技术通报, 2010(4): 16-21. |
| [37] |
张贤, 王建红, 喻曼, 曹凯, 庄俐, 徐昌旭, 曹卫东. 基于RNA-seq的能源植物芒转录组分析. 生物工程学报, 2015, 31(10): 1437-1448. |
| [38] |
吴孚桂, 刘慧芳, 聂佳俊, 韦云飞, 马启林. 水稻幼穗响应盐胁迫的转录组分析. 热带作物学报. (2020-07-03)[2020-12-10]. http://kns.cnki.net/kcms/detail/46.1019.S.20200703.0855.002.html.
|
| [39] |
齐明, 何贵平, 王海蓉, 邱勇斌, 程亚平, 徐肇友. 杉木施肥引发基因表达响应机制的转录组分析. 中南林业科技大学学报, 2020, 40(4): 101-110. |
| [40] |
王蓥燕. 石灰性紫色水稻土中nirS和nosZ基因对不同施肥处理的响应特征及其垂直分布[D]. 成都: 四川农业大学, 2017.
|
| [41] |
张敏瑜. 丛枝菌根真菌与香蕉Foc4的互作效应及其抗枯分子机制[D]. 厦门: 华侨大学, 2019.
|
| [42] |
Nautiyal C S, Srivastava S, Chauhan P S, Seem K, Mishra A, Sopory S K. Plant growth-promoting bacteria Bacillus amyloliquefaciens NBRISN13 modulates gene expression profile of leaf and rhizosphere community in rice during salt stress. Plant Physiology and Biochemistry, 2013, 66: 1-9. DOI:10.1016/j.plaphy.2013.01.020 |
| [43] |
靳聪聪, 侯名语, 潘延云. 拟南芥核糖体蛋白生物学功能研究进展. 植物生理学报, 2018, 54(2): 203-212. |
| [44] |
Barakat A, Szick-Miranda K, Chang I F, Guyot R, Blanc G, Cooke R, Delseny M, Bailey-Serres J. The organization of cytoplasmic ribosomal protein genes in the Arabidopsis genome. Plant Physiology, 2001, 127(2): 398-415. DOI:10.1104/pp.010265 |
| [45] |
谢丽霞. 极端嗜盐曲霉核糖体蛋白基因SpRPS3ae和SpRPL44的克隆及其抗逆功能研究[D]. 长春: 吉林大学, 2013.
|
| [46] |
夏丽丹, 张虹, 胡华英, 曹升, 周垂帆. RNA-Seq定量分析盐肤木对铅胁迫的响应. 农业环境科学学报, 2019, 38(7): 1459-1467. |
| [47] |
Aminian R, Mohammadi S, Hoshmand S, Khodombashi M. Chromosomal analysis of photosynthesis rate and stomatal conductance and their relationships with grain yield in wheat (Triticum aestivum L.) under water-stressed and well-watered conditions. Acta Physiologiae Plantarum, 2011, 33(3): 755-764. DOI:10.1007/s11738-010-0600-0 |
| [48] |
程海涛, 姜华, 薛大伟, 郭龙彪, 曾大力, 张光恒, 钱前. 水稻芽期与幼苗前期耐碱性状QTL定位. 作物学报, 2008, 34(10): 1719-1727. DOI:10.3321/j.issn:0496-3490.2008.10.006 |
| [49] |
Yu Z P, Duan X B, Luo L, Dai S J, Ding Z J, Xia G M. How plant hormones mediate salt stress responses. Trends in Plant Science, 2020, 25(11): 1117-1130. DOI:10.1016/j.tplants.2020.06.008 |
| [50] |
Edel K H, Kudla J. Integration of calcium and ABA signaling. Current Opinion in Plant Biology, 2016, 33: 83-91. DOI:10.1016/j.pbi.2016.06.010 |
| [51] |
Yang L, Liu Q H, Liu Z B, Yang H, Wang J M, Li X F, Yang Y. Arabidopsis C3HC4-RING finger E3 ubiquitin ligase AtAIRP4 positively regulates stress-responsive abscisic acid signaling. Journal of Integrative Plant Biology, 2016, 58(1): 67-80. DOI:10.1111/jipb.12364 |
| [52] |
Hupp S, Rosenkranz M, Bonfig K, Pandey C, Roitsch T. Noninvasive phenotyping of plant-pathogen interaction: consecutive in situ imaging of fluorescing Pseudomonas syringae, plant phenolic fluorescence, and chlorophyll fluorescence in Arabidopsis leaves. Frontiers in Plant Science, 2019, 10: 1239. DOI:10.3389/fpls.2019.01239 |
| [53] |
de Wit P J G M. How plants recognize pathogens and defend themselves. Cellular and Molecular Life Sciences, 2007, 64(21): 2726-2732. DOI:10.1007/s00018-007-7284-7 |
| [54] |
Zipfel C. Pattern-recognition receptors in plant innate immunity. Current Opinion in Immunology, 2008, 20(1): 10-16. DOI:10.1016/j.coi.2007.11.003 |
| [55] |
Jamali H, Sharma A, Roohi, Srivastava A K. Biocontrol potential of Bacillus subtilis RH5 against sheath blight of rice caused by Rhizoctonia solani. Journal of Basic Microbiology, 2020, 60(3): 268-280. DOI:10.1002/jobm.201900347 |
| [56] |
朱华珺, 周瑚, 任佐华, 刘二明. 枯草芽孢杆菌JN005胞外抗菌物质及对水稻叶瘟防治效果. 中国水稻科学, 2020, 34(5): 470-478. |
| [57] |
马进, 郑钢, 裴翠明, 张振亚. 南方型紫花苜蓿根系盐胁迫应答转录因子鉴定与分析. 浙江农林大学学报, 2016, 33(2): 201-208. |
| [58] |
陈娜, 迟晓元, 潘丽娟, 陈明娜, 禹山林. MYB转录因子在植物盐胁迫调控中的研究进展. 植物生理学报, 2015, 51(9): 1395-1399. |
| [59] |
田林, 尹丹丹, 成铁龙, 夏新莉, 尹伟伦. 盐胁迫下比拉底白刺差异表达基因的转录组分析. 林业科学研究, 2020, 33(1): 1-10. |
 2022, Vol. 42
2022, Vol. 42