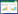文章信息
- 侯冠群, 来剑斌, 李静, 刘振, 公华锐, 王兵, 孙志刚, 欧阳竹, 侯瑞星
- HOU Guanqun, LAI Jianbin, LI Jing, LIU Zhen, GONG Huarui, WANG Bing, SUN Zhigang, OUYANG Zhu, HOU Ruixing
- 成土时间对黄河三角洲植物及微生物演替的影响
- Driving force of soil age on vegetation and microbial succession in the Yellow River Delta
- 生态学报. 2022, 42(21): 8839-8859
- Acta Ecologica Sinica. 2022, 42(21): 8839-8859
- http://dx.doi.org/10.5846/stxb202107211977
-
文章历史
- 收稿日期: 2021-07-21
- 网络出版日期: 2022-04-03
2. 中国科学院大学资源与环境学院, 北京 100049;
3. 中国地质调查局烟台海岸带地质调查中心, 烟台 264004
2. College of Resources and Environment, University of Chinese Academy of Sciences, Beijing 100049, China;
3. Yantai Coastal Zone Geological Survey Center, China Geological Survey, Yantai 264004, China
滨海新生土地演替的研究有助于湿地资源的保护, 为改善当地的生态环境提供科学依据[1—2]。了解新生土地生境更新、演替规律, 还有助于评估人类活动的影响[3], 使开发速度低于生境更新、演替的速度, 或者引导和控制新生土地生境更新、演替的速度和方向[4]。
演替的监测需要频繁连续的时间序列。但是, 初生演替的研究往往受到时空因素干扰或限制, 通常采用时空替代法拟合时间序列, 如冰川退缩迹地研究中通常采用样线法[5], 而土壤年龄的驱动力研究多采用样地法[6]。在滨海三角洲演替的研究中, 采样和分析方法通常为“空间换时间”[7—8]或针对植被演替阶段的样地法[9—11]。虽然前人很早关注到河口三角洲土壤年龄对生态系统演替阶段可能带来的影响[8, 11—12], 但缺少成土时间的驱动力的定量研究, 这可能造成演替时间和空间的不匹配, 进而形成对演替过程的描述产生偏差。
黄河三角洲自1855年黄河夺大清河流路入渤海以来, 黄河尾闾多次改道, 形成多个呈叠瓦状复式结构的亚三角洲堆积体[13]。其中有4个亚三角洲堆积体与其他洲体的重叠程度较低, 在地表形成相对独立的冲积扇区, 成土时期分别为1904—1929年, 1929—1934年, 1964—1976年和1976年至今[14—15]。亚三角洲堆积体重叠时, 表层土壤成土时期由晚近流路的冲淤决定[16], 故相同亚三角洲堆积体的土壤具有相对一致的发育年龄。各扇区均从内陆向滨海延伸, 均包含滨海典型植被群落和植被演替过程, 并且均在内陆深处形成了农田土壤。
黄河三角洲土壤含盐量较高且以裸地为起点[17]。随着演替进行, 植物种类增加, 物种多样性上升, 植被盖度、物种丰富度增加[3, 8], 土壤质量呈现逐渐改善的趋势[7—8, 18]。植被演替和土壤演替在滨海生态系统内交替发生, 且均受到成土时间和离海距离等因素影响。成土时间的不同带来生态系统年龄的差异, 且与离海距离共同影响土壤脱盐程度和熟化特征[8, 18]。盐度影响海岸和河口的碳源和汇功能[19], 进而影响演替过程。白茅是轻度耐盐植物, 可作为可垦殖的植被指标[8], 耕地主要由白茅草甸和芦苇-荻草甸开垦而来[3]。黄河三角洲土壤和植被随时间的演替过程也伴随着微生物的演替, 微生物群落的演替受土壤化学性质和植物种类影响[10, 12, 17], 而微生物群落作为生物地球化学循环的关键因素, 又塑造了环境特征和功能[20—22]。
黄河三角洲多个亚三角洲堆积体可提供对滨海土壤向农田土壤演替的时空要素开展研究的平台。本文探究成土时间是否为黄河三角洲植物和微生物群落演替的生态驱动力, 为提高评估滨海生态系统植物和微生物演替规律的准确性提供科学依据。
1 材料与方法 1.1 研究区概况本研究采样区域位于山东省东营市黄河三角洲地区(37°31.807′—38°07.664′N, 118°15.858′—119°13.384′ E), 地跨东营市河口区、垦利区、利津县。黄河三角洲属暖温带季风气候, 年平均气温12.3℃, 年平均降水量542.3mm。土壤以滨海盐土和滨海潮土为主, 土壤盐渍化严重, 主要是氯化物盐土和氯化物潮化盐土, 土壤组成以泥沙为主, 养分含量低[23]。自然植被以盐生植物为主。植被带垂直于河床分布, 沿河流由海向陆发展[9]。
1.2 样品采集2020年5月, 在各时期冲积扇区范围内进行土样和植被信息采集(图 1), 按成土历史分别命名为路线1扇区、路线2扇区、路线3扇区和路线4扇区(下文分别简称R1、R2、R3和R4)。分别由河口所在海岸沿河道或古河道方向设计采样路线, 每条采样路线由沿海到内陆对样地编号为样地1、样地2……样地6(下文分别简称S1、S2……R6)。采样标准为自成土以来受人类活动干扰较小的、有代表性的原生景观植被, 包括典型的盐滩、盐生草地、中生草地和灌丛草地, 并采集农田样地。基于实地调查, 选取样地21处, 如表 1所示。R1扇区受人类近海工业和养殖活动影响, 未发现原生盐滩;R4扇区成土时间较晚, 未发育形成灌丛草地。R1S4、R2S4、R3S5、R4S4为各冲积扇区发现的内陆顶级景观植被, 农田样地在以上样地5—10km外的内陆选取。
|
| 图 1 研究区域和采样地点示意图 Fig. 1 Location of the study area and sampling sites R1(Route 1, 即路线1, 下同)、R2、R3和R4分别采样的路线1、路线2、路线3和路线4所考察的冲积扇区, 成土年代分别为1904—1929年、1929—1934年、1964—1976年和1976年至今; 每条采样路线由沿海到内陆对样地编号为S1(Sample site 1, 即样地1, 下同)、S2、S3、S4、S5和S6; 例如, R2S1(Sample site 1 of route 2)意为采样路线2所在扇区的第1个样地; R1S1为潮间带淤泥对照 |
| 样地名 Names of sample sites |
地理位置 Geographical position |
纬度 Latitude |
经度 Longitude |
河口海岸距离/km Distance to estuary shoreline /km |
植被类型 Vegetation type |
| R1S2 | 新户镇郭局村南 | N38°01′ | E118°22′ | 10.3 | 柽柳盐生草地 |
| R1S3 | 义和镇北大屋子村 | N37°54′ | E118°21′ | 22.9 | 禾草中生草地 |
| R1S4 | 义和镇七顷村 | N37°50′ | E118°20′ | 30.2 | 柽柳灌丛草地 |
| R1S5 | 虎滩乡北 | N37°46′ | E118°22′ | 37.0 | 农田 |
| R2S1 | 垦利区永丰河口附近 | N37°30′ | E118°56′ | 0.0 | 无植被滩地 |
| R2S2 | 永安镇红光新村西 | N37°32′ | E118°49′ | 8.2 | 柽柳盐生草地 |
| R2S3 | 永安镇十一村南 | N37°32′ | E118°47′ | 10.6 | 禾草中生草地 |
| R2S4 | 永安镇后二十五村西 | N37°33′ | E118°44′ | 15.8 | 菊蒿灌丛草地 |
| R2S5 | 垦利区东羊村东 | N37°35′ | E118°38′ | 25.1 | 农田 |
| R2S6 | 垦利区虹吸沟 | N37°36′ | E118°37′ | 28.0 | 农田 |
| R3S1 | 河口区桩124路西口南 | N38°07′ | E118°51′ | 0.70 | 柽柳盐生滩地 |
| R3S2 | 河口区101探井路偏西南 | N38°05′ | E118°51′ | 3.7 | 芦苇盐生草地 |
| R3S3 | 河口区五一七队屋子南 | N38°02′ | E118°51′ | 8.8 | 碱蓬盐生草地 |
| R3S4 | 仙河镇东星村东南 | N37°57′ | E118°53′ | 18.9 | 菊蒿中生草地 |
| R3S5 | 孤岛镇丰收村东北 | N37°54′ | E118°52′ | 25.6 | 芦苇灌丛草地 |
| R3S6 | 孤岛镇三村屋子北 | N37°48′ | E118°48′ | 37.7 | 农田 |
| R4S1 | 黄河口保护区东端路口 | N37°43′ | E119°13′ | 2.60 | 碱蓬盐生滩地 |
| R4S2 | 黄河口保护区偏东 | N37°45′ | E119°06′ | 11.4 | 芦苇湿生草地 |
| R4S3 | 黄河口保护区西部 | N37°46′ | E119°00′ | 20.4 | 芦苇盐生草地 |
| R4S4 | 黄河口镇利林村东北 | N37°44′ | E118°52′ | 32.5 | 柽柳中生草地 |
| R4S5 | 黄河口镇护林管理河段 | N37°43′ | E118°46′ | 41.5 | 农田 |
| R1(Route 1, 即路线1, 下同)、R2、R3和R4分别采样的路线1、路线2、路线3和路线4所考察的冲积扇区, 成土年代分别为1904—1929年、1929—1934年、1964—1976年和1976年至今; 每条采样路线由沿海到内陆对样地编号为S1(是Sample site 1, 即样地1, 下同)、S2、S3、S4、S5和S6; 例如, R2S1(Sample site 1 of route 2)意为采样路线2所在扇区的第1个样地 | |||||
每处样地采用五点取样法, 样点间距5—10m。使用带刻度的土钻在每个样点采集20cm深度土样, 将5个样点各深度的土样分别于自封袋中混合均匀。在每个样地的中心样点划出1m×1m样方, 在自然群落采集所有草本植物地上部茎叶用于生物量测定, 并记录样方内物种信息;记录该处自然群落的各植物类群的盖度信息。
各样地的5个样点的土壤样品混合, 分成两部分。其中一部分风干并过20目标准筛, 再从其中取约20g过100目标准筛, 用于土壤理化性质测定。另一部分立即过20目筛, 填充5mL离心管并标号, 埋藏于干冰中冷冻, 每个样地设三个重复, 送至上海美吉生物医药科技有限公司测序。
1.3 土壤理化性质测定取过20目筛的干土, 使用去离子水按水土比5∶1制备浸提液, 以PHS—3E酸度计测定pH, 以Mettler Toledo电导仪(FiveEasy Plus FE20)测定电导率(EC)。土壤含水量(SWC)用铝盒烘干法测定;土壤有机质(SOM)依据NY/T1121.6—2006标准滴定;全氮(TN)使用浓硫酸消解—凯氏定氮法测定;全磷(TP)和全钾(TK)依据DZ/T0219.1—2006标准使用ARL 9900X射线荧光光谱仪测定;碱解氮(AN)依据LY/T 1228—2015标准滴定;速效磷(AP)依据LY/T1232—2015标准使用TU—1810紫外可见光分光光度计滴定;速效钾(AP)依据NY/T890—2004标准使用ICE3500原子吸收分光光度计测定。以上10个指标连同碳氮比(CN.ratio)作为本研究的土壤理化性质因子集合。
1.4 植被多样性和时空因子计算方法植被样品取回后, 于烘箱中105℃处理24h, 称得生物量干重, 记为plant.bioM。
植物物种丰富度为样地中所能识别的物种种类数, 记为plant.S。统计植物物种数时, 考虑到5月许多植物处于苗期或营养生长期而不易辨别, 故将同一科的相同生活型的部分物种进行了合并。样地区域植被总盖度记为plant.cov。
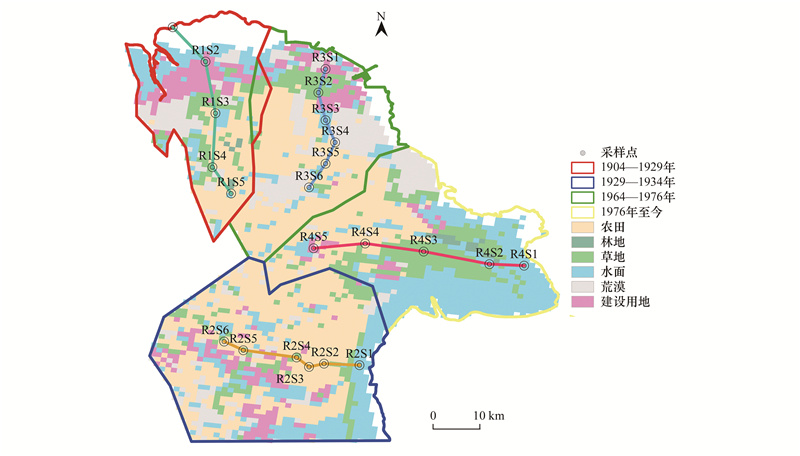
植物物种多样性计算方法为Shannon-Wiener指数[24], 计算公式为:
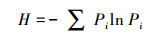
|
式中, Pi指第i个物种的出现频率。下文简称Shannon指数。
以样方内植被各物种的多度与植株个体总数的比值为Pi计算物种组成Shannon多样性, 记为Shannon.abun。考虑到作为环境因子时, 1m2的样方对样地植被信息代表性可能不足, 故另以样地植被各类群的盖度占比为Pi计算类群盖度Shannon多样性, 记为Shannon.cov。以上5个指标作为本研究的植物多样性因子集合。
各冲积扇区成土时间计算方式为亚三角洲体形成年份区间的中值与采样时间(2020年)的年代差;河口海岸距离计算方法为样地与黄河河口或古河口所在海岸方向的距离。
1.5 微生物群落组成测序方法根据E.Z.N.A. soil DNA kit (Omega Bio-tek, Norcross, GA, U.S.)说明书进行微生物群落总DNA抽提。使用1%的琼脂糖凝胶电泳检测DNA的提取质量, 使用NanoDrop2000测定DNA浓度和纯度;使用338F (5′-ACTCCTACGGGAGGCAGCAG-3′) 和806R (5′-GGACTACHVGGGTWTCTAAT-3′) 对16S rRNA基因V3-V4可变区进行PCR扩增, 使用SSU0817F (5′-TTAGCATGGAATAATRRAATAGGA-3′)和SSU1196R (5′-TCTGGACCTGGTGAGTTTCC-3′)对18S rRNA基因V4—V5可变区进行PCR扩增。随后基于Illumina MiSeq进行16S rRNA、18S rRNA扩增子测序。使用Fastp(https://github.com/OpenGene/fastp, version 0.20.0)软件对原始测序序列进行质控, 使用FLASH(http://www.cbcb.umd.edu/software/flash, version 1.2.7)软件进行拼接:(1)过滤reads尾部质量值20以下的碱基, 设置50bp的窗口, 如果窗口内的平均质量值低于20, 从窗口开始截去后端碱基, 过滤质控后50bp以下的reads, 去除含N碱基的reads;(2)根据PE reads之间的重叠关系, 将成对reads拼接成一条序列, 最小重叠长度为10bp;(3)拼接序列的overlap区允许的最大错配比率为0.2, 筛选不符合序列;(4)根据序列首尾两端的条形码和引物区分样品, 并调整序列方向, 条形码允许的错配数为0, 最大引物错配数为2。DNA抽提、荧光定量PCR、MiSeq测序、序列质控、拼接均由商业测序公司完成(美吉生物, 上海)。
1.6 数据分析以97%的相似度[25]对序列进行操作分类单元(OTU)聚类并剔除嵌合体, 以RDP classifier比对Silva 16S rRNA、18S rRNA数据库(v138.1)来对每条序列进行物种分类注释, 设置比对阈值为70%。获得OTU数据表后, 分别筛选原核生物域和真菌界, 分别筛得24380、1493个OTU。保留至少3个样本中reads数≥5的OTU, 并按最小样本数抽平。所得结果中, 每个重复组细菌的OTU频数合计为23698, 测序深度指数≥96.0%;真菌的OTU频数合计为29965, 测序深度指数≥99.8%;测序深度指数表明处理得到的数据集足以代表该区域真实的微生物群落。对细菌群落和真菌群落, 分别将其Sobs指数、Shannon多样性指数以及丰度占比前8的门类的各样点reads数等10个因子组合作为微生物群落组成因子(bacteria, fungi)。
细菌物种的功能属性的文献查阅以原核生物标准命名列表(LPSN)数据库的信息为主, 若一个细菌属下所有公开发表(validly published)的物种均有耐盐3%及以上或生长所需盐浓度的下限>0.5%, 则该属被识别为耐盐菌;若一个细菌属所有公开发表的物种均具备反硝化能力, 则该属被识别为反硝化菌;反硫化菌和自养菌等鉴定方法同上。
各扇区顶级景观植被群落的差异可能体现成土时间对演替阶段的限制。本文对各扇区顶级景观植被群落的微生物进行PICRUSt2(Phylogenetic Investigation of Communities by Reconstruction of Unobserved States 2)分析, 再将取得的基于KEGG(Kyoto Encyclopedia of Genes and Genomes)数据库的功能通路信息结果进行线性判别分析(LDA), 识别对各样地功能通路组成的差异效果影响较大的通路, 判别标准为LDA得分(log10转化)大于2, 且克鲁斯卡尔-沃利斯秩和检验(Kruskal-Wallis rank sum test)为统计学显著(LDA score>2, P<0.05)。
对成土时间的驱动力进行偏曼特尔检验(Mantel)时, 将本文中除成土时间外的其他环境因子(distance、vegetation、soil properties、bacteria、fungi)构建控制环境矩阵(算法为欧氏距离), 检验植被、细菌、真菌群落的物种矩阵(算法为布雷柯蒂斯距离的可变性能够被成土时间解释的部分。环境变量和生物变量相关分析(Bioenv)选取本研究除bacteria、fungi之外的18个环境因子。VPA分析时, 由于微生物OTU矩阵0值较多, 在计算Bray-Curtis距离时进行了海林格距离转化, 绘图方法如Delgado-Baquerizo等的研究[6]。
多样性统计、Bioenv分析、Mantel分析、方差分解分析使用R包vegan完成。相关性分析使用R软件包ggcor完成。LDA分析和LEfSe分析于Galaxy/Hutlab网页完成。物种Venn图和PICRUSt2在美吉生信云网页绘制。其他统计图使用ArcGIS 10.2、Adobe Illustrator 2021和R软件包ggplot2、reshape2绘制。
2 结果与分析 2.1 土壤理化性质和微生物OTU组成异质性检验在百年尺度上, 黄河三角洲流域不同时期扇区的土壤理化性质相近(图 2)。河口海岸距离与有机质(r=0.75)、全氮(r=0.65)、电导率(r=-0.82)、pH(r=-0.42)极显著相关(P<0.001), 且以上4个土壤理化指标相互间均有线性耦合(P<0.001)。成土时间仅与速效钾显著相关(r=-0.27, P<0.05), 但非影响速效钾的首要环境因素, 速效钾主要受到土壤有机质(r=0.40, P<0.01)和全氮(r=0.48, P<0.001)的影响。
|
| 图 2 成土时间、海陆距离与土壤理化指标的相关性特征 Fig. 2 Correlation characteristics of soil age, sea-land distance and soil properties soil.age:成土时间; distance:样地与黄河河口或古河口所在海岸方向的距离; SOM:土壤有机质Soil organic matter; TN:全氮Total nitrogen; EC:电导率Electroconductibility; pH:酸碱度Potential of hydrogen; SWC:土壤水含量Soil water content; TP:全磷Total phosphorus; TK:全钾Total potassium; AN:碱解氮Available nitrogen; AP:速效磷Available phosphorus; AK:速效钾Available potassium; CN.ratio:碳氮比Carbon nitrogen ratio; 左下部分红色圆形表示因子间显著正相关(P<0.05), 浅蓝色圆形表示显著负相关(P<0.05); 右上部分为简化的箱线图, *(P<0.05), **(P<0.01), ***(P<0.001)代表因子间相关性显著 |
各扇区微生物群落的OTU组成相近(图 3)。细菌群落共筛选出6374个OTU, 其中4381个(68.7%)为4个扇区共有OTU, 272个(4.3%)为单个扇区特有OTU(图 3)。真菌群落共筛选出391个OTU, 其中260个(66.5%)为4个扇区共有OTU, 15个(3.8%)为单个扇区特有OTU。1512个(23.7%)细菌OTU的丰度随成土时间的变化显著(P<0.05), 67个(17.1%)真菌OTU的丰度随成土时间的变化显著(P<0.05)。4个冲积扇区自然禀赋相似, 土壤微生物OTU组成相近, 生态系统的空间演替可充分体现成土时间对生物演替的驱动作用。

|
| 图 3 各冲积扇区细菌和真菌OTU数量分布特征 Fig. 3 Quantitative distribution characteristics of bacteria and fungi OTUs among alluvial sectors OTU:操作分类单元Operational taxonomic unit; 韦恩图表示OTU在4个冲积扇区的共现性, 数字表示OTU个数;条形图表示OTU在4个冲积扇区的总数(Sobs指数);饼图表示OTU丰度(样本内reads频数)与成土时间的Spearman相关性特征, 浅红色、浅绿色和灰色分别表示丰度随成土时间显著增加(r>0, P<0.05)、显著减少(r<0, P<0.05)和无显著相关(P>0.05)的个数 |
伴随成土时间的增加, 植物物种数和科类丰富度整体呈增加趋势(表 2)。研究区域内, 代表性的盐生植物有芦苇(phragmites communis Trin.)、柽柳(Tamarix chinensis Lour.) 及翅碱蓬(Suaeda heteropter Kitag)等, 优势植物类群为禾本科(Gramineae)、藜科(Chenopodiaceae)、菊科(Compositae)、柽柳科(Tamaricaceae)等。中生草地群落的指示种为白茅(Imperata cylindrica (L.) Beauv.)、罗布麻(Apocynum venetum L.)、野大豆(Glycine soja Sieb. et Zucc.)等。成土年代最近的R4扇区尚未发育出灌丛草地的植被类型, 而最早的R1扇区出现了自然生长的白蜡、洋槐等树木。
| 样地名 Names of sample sites |
建群种 Constructive species |
常见种 Common species |
科类数 Number of families |
种数 Number of species |
盖度 Coverage |
类群盖度Shannon指数 Shannon index by coverage |
物种组成Shannon指数 Shannon index by abundance |
| R1S2 | 芦苇、柽柳 | 狗牙根、鸦葱 | 5 | 9 | 0.90 | 1.05 | 0.89 |
| R1S3 | 碱茅、芦苇 | 白茅、鹅绒藤 | 10 | 20 | 0.95 | 1.45 | 0.68 |
| R1S4 | 柽柳、白茅 | 灰藜、碱蓬 | 7 | 19 | 1.00 | 1.72 | 0.86 |
| R1S5 | 小麦 | 播娘蒿、雀麦 | 6 | 10 | 0.90 | 0.36 | 0.03 |
| R2S2 | 芦苇、柽柳 | 狗牙根、薹草 | 6 | 9 | 0.60 | 1.51 | 0.41 |
| R2S3 | 白茅、芦苇 | 茵陈蒿、野大豆 | 7 | 12 | 0.85 | 1.60 | 1.60 |
| R2S4 | 茵陈蒿、柽柳 | 小獐茅、白茅 | 11 | 20 | 1.00 | 1.39 | 1.64 |
| R3S1 | 柽柳、鸦葱 | 芦苇、狗牙根 | 4 | 5 | 0.30 | 1.57 | 1.72 |
| R3S2 | 芦苇、柽柳 | 碱蓬、二色补血草 | 7 | 14 | 0.70 | 1.86 | 0.69 |
| R3S3 | 碱蓬、柽柳 | 翅碱蓬、芦苇 | 4 | 5 | 0.50 | 0.89 | 1.54 |
| R3S4 | 白茅、苣荬菜 | 茵陈蒿、芦苇 | 10 | 17 | 1.00 | 1.96 | 1.12 |
| R3S5 | 芦苇、柽柳 | 鹅绒藤、茵陈蒿 | 6 | 13 | 0.95 | 1.76 | 0.82 |
| R3S6 | (未播农田) | 芦苇、大车前 | 3 | 3 | 0.00 | 0.48 | 0.43 |
| R4S1 | 翅碱蓬 | 柽柳 | 2 | 2 | 0.10 | 0.30 | 0.04 |
| R4S2 | 芦苇、荻 | 盐角草 | 2 | 3 | 0.40 | 0.07 | 0.00 |
| R4S3 | 芦苇、柽柳 | 碱蓬、翅碱蓬 | 6 | 7 | 0.75 | 1.05 | 0.56 |
| R4S4 | 柽柳、狗牙根 | 芦苇、荩草 | 5 | 8 | 0.80 | 1.25 | 1.14 |
| R4S5 | 小麦 | 藜、芦苇 | 4 | 9 | 0.85 | 0.18 | 0.00 |
| 物种组成Shannon指数是以样方内植被各物种的平均多度作为1.4中公式的Pi, 计算得到的Shannon多样性指数;类群盖度Shannon指数是以植被各植物类群的盖度作为1.4中公式的Pi, 计算得到的Shannon多样性指数。无植被地带未列入 | |||||||
R4S4、R3S5、R2S4、R1S4为各冲积扇区发现的内陆顶级景观植被(表 1), 建群种除柽柳外, 从耐盐的狗牙根、芦苇向轻度耐盐[8]的茵陈蒿和白茅过渡(表 2)。R1S3、R2S4、R3S4和R4S3为各扇区中科类丰富度较高的样地, 且一致分布在距海20km附近。随成土时间增加, 距海20km附近依次呈现从盐生草地、中生草地向灌丛草地或高禾草地景观的过渡特征。
成土时间驱动植物生物量(r=0.53, P<0.001)和植物物种丰富度(r=0.29, P<0.05)增加, 且植被盖度、Shannon多样性均随成土时间增加而增大(图 4)。内陆区域受人类活动干扰的影响, 农田植物物种多样性较低, 表现为植物Shannon多样性与土壤有机质和全氮呈负相关(P<0.01)。
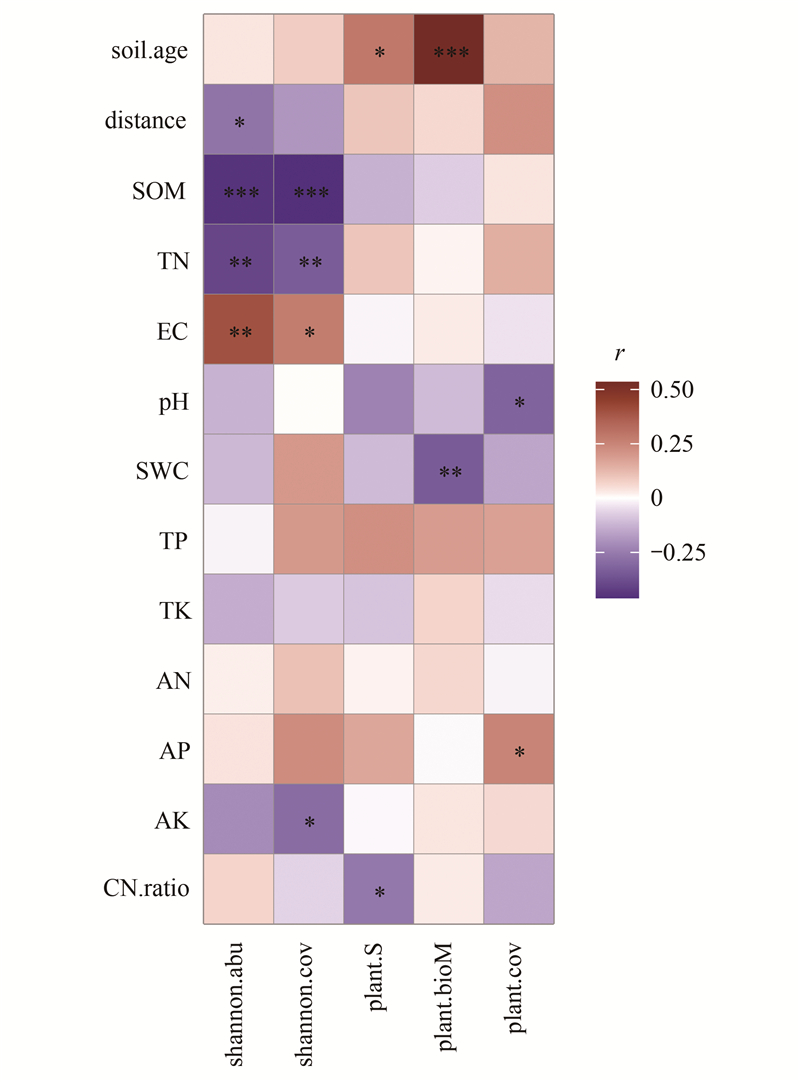
|
| 图 4 成土时间、海陆距离、土壤理化指标与植物多样性的相关性特征 Fig. 4 Correlation characteristics of soil age, sea-land distance, soil properties and plant species diversity shannon.abu:植物Shannon多样性指数(以样方内植物物种多度计算) Shannon index of plant communities calculated by abundance;shannon.cov:植被Shannon多样性指数(以样地内各类群的盖度替代多度计算) Shannon index of plant communities calculated by coverage;plant.S:植物物种丰富度Number of species of plant communities;plant.bioM:1m2样方内植被的地上部干生物量Aboveground biomass of plants in the 1 m2 sample site;plant.cov:样地内的植被盖度; *(P<0.05), **(P<0.01), ***(P<0.001)代表因子间相关性显著 |
研究区域内, 各扇区微生物物种门类组成相似, 但各门类相对优势度存在差异(表 3)。细菌群落3个主要优势门类为变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、绿弯菌门(Chloroflexi), 总占比为59.7%。真菌群落3个主要优势门类为子囊菌门(Ascomycota)、担子菌门(Basidiomycota)、毛霉门(Mucoromycota), 总占比为94.9%。
| 细菌门类名 Names of bacteria phyla |
相对丰度Relative abundance/% | ||||
| R1扇区 R1 sector |
R2扇区 R1 sector |
R3扇区 R1 sector |
R4扇区 R1 sector |
总计 In all |
|
| 变形菌门Proteobacteria | 23.44 | 25.80 | 28.83 | 24.40 | 26.18 |
| 放线菌门Actinobacteria | 23.63 | 19.30 | 19.75 | 19.62 | 19.70 |
| 绿弯菌门Chloroflexi | 13.81 | 15.35 | 12.85 | 13.36 | 13.78 |
| 酸杆菌门Acidobacteria | 8.89 | 10.10 | 7.05 | 7.56 | 8.26 |
| 拟杆菌门Bacteroidetes | 5.40 | 5.59 | 9.16 | 8.48 | 7.41 |
| 芽单胞菌门Gemmatimonadetes | 6.13 | 6.36 | 6.41 | 8.78 | 6.68 |
| 厚壁菌门Firmicutes | 7.35 | 3.38 | 3.36 | 4.54 | 4.38 |
| 脱硫杆菌门Desulfobacterota | 2.13 | 3.03 | 3.44 | 2.61 | 3.38 |
| 粘细菌门Myxococcota | 1.28 | 1.51 | 1.28 | 1.49 | 1.36 |
| 浮霉菌门Planctomycetota | 0.77 | 1.18 | 0.85 | 0.86 | 0.92 |
| 候选门级辐射类群*Patescibacteria | 0.63 | 0.68 | 1.03 | 1.04 | 0.85 |
| 奇异甲烷氧化菌门*Methylomirabilota | 1.50 | 0.84 | 0.23 | 0.45 | 0.67 |
| 其他Others | 5.04 | 6.88 | 5.75 | 6.81 | 6.43 |
| 真菌门类名 Names of fungi phyla |
相对丰度Relative abundance/% | ||||
| R1扇区 R1 sector |
R2扇区 R1 sector |
R3扇区 R1 sector |
R4扇区 R1 sector |
总计 In all |
|
| 子囊菌门Ascomycota | 82.53 | 82.60 | 83.07 | 81.09 | 81.60 |
| 担子菌门Basidiomycota | 10.64 | 7.64 | 9.58 | 13.07 | 10.33 |
| 毛霉门Mucoromycota | 3.56 | 3.83 | 3.39 | 1.00 | 2.98 |
| 壶菌门Chytridiomycota | 0.64 | 3.13 | 0.41 | 0.87 | 1.55 |
| 捕虫霉门Zoopagomycota | 1.03 | 0.96 | 0.31 | 0.21 | 0.59 |
| 芽枝霉门Blastocladiomycota | 0.30 | 0.36 | 0.72 | 0.01 | 0.36 |
| 未定名类群* LKM15 | 0.20 | 0.08 | 0.26 | 0.37 | 0.24 |
| 隐真菌门Cryptomycota | 0.05 | 0.04 | 0.04 | 0.05 | 0.08 |
| 新美鞭菌门Neocallimastigomycota | / | 0.01 | 0.00 | / | 0.00 |
| 其他Others | 1.05 | 1.35 | 2.22 | 3.33 | 2.27 |
| * 中文学名未确定:Patescibacteria拉丁语意为“代谢能力有限的细菌”;Methylomirabilota拉丁语意为“奇特的甲烷氧化菌”;LKM15为尚未获得培养的真菌分支; / 该采样扇区各样点18S rDNA克隆文库未检出该门类 | |||||
细菌群落的Shannon多样性受到土壤pH限制(r=-0.26, P<0.05, 图 5)。有机质含量显著促进真菌群落物种数(Sobs指数)增加(r=-0.62, P<0.001), 电导率和pH则是限制真菌物种数的限制因子(r=-0.69, P<0.001, 图 5)。植被盖度和植物Shannon多样性的提高促进真菌物种多样性增加(r>0.28, P<0.05)。
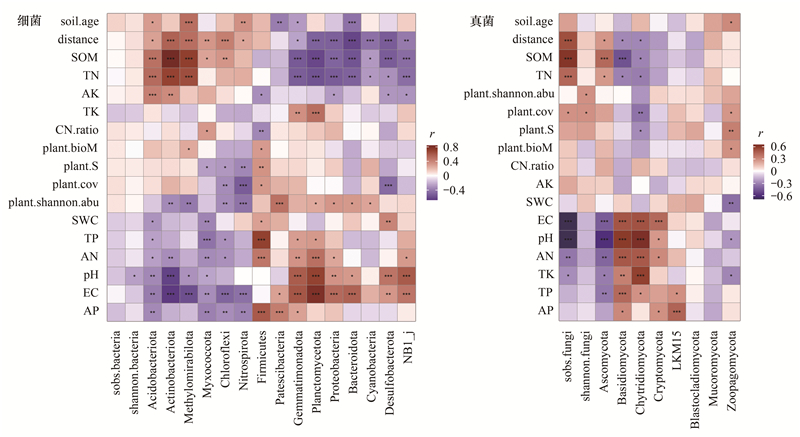
|
| 图 5 细菌和真菌多样性、门类相对丰度与环境因子的相关性热图 Fig. 5 Correlation heat maps between bacterial and fungal diversity, relative abundance of main phylum and environmental factors sobs.bacteria: 细菌sobs指数(OTU水平物种数目); sobs.fungi: 真菌sobs指数(OTU水平物种数目); shannon.bacteria: 细菌Shannon多样性指数(以OTU多度计算); shannon.fungi: 真菌Shannon多样性指数(以OTU多度计算); *(P<0.05), **(P<0.01), ***(P<0.001)代表因子间相关性显著 |
河口海岸距离、土壤有机质、全氮、pH和电导率是影响微生物门类组成的主要因子, 与多数主要门类的相对丰度有较强的相关性(图 5)。成土时间对微生物群落门类相对丰度影响则相对独立。
随着时间演替, 部分门类的占比变化呈现相反趋势。成土时间驱动硝化螺菌门(Nitrospirota)、酸杆菌门(Acidobacteriota)、Methylomirabilota相对丰度显著增加(r>0.29, P<0.05, 图 5), 同时拟杆菌门(Bacteroidota)、芽单胞菌门(Gemmatimonadetes)、Patescibacteria相对丰度显著降低(r<-0.27, P<0.05)。
成土时间还驱动Sumerlaeota、装甲菌门(Armatimonadota)、异常球菌门(Deinococcota)、Acetothermia、Hydrogenedentes、盐厌氧菌门(Halanaerobiaeota)、海微菌门(Marinimicrobia-SAR406-clade)、Calditrichota、Campilobacterota等次要门类相对丰度的降低(r<-0.26, P<0.05, 图 4未列出)。
丰度与成土时间相关性显著的真菌门类为捕虫霉门(Zoopagomycota)(r=0.30, P<0.05)。研究区域内, 子囊菌门(Ascomycota)和担子菌门(Basidiomycota)在真菌群落的丰度占比达91.9%(表 3)。对这2个门类的相对丰度影响最大的环境因子为pH(r=0.60, P<0.001, 图 5)。
2.4 细菌和真菌群落差异物种分析在从门到属的分类层级, 部分微生物群落物种类群富集特征显著(LDA score>2, P<0.05, 图 6)。如, 细菌厚壁菌门(Firmicutes)和真菌毛霉门(Mucoromycota)和捕虫霉门(Zoopagomycota)在较早形成的R1扇区中可作为真菌群落的生物标志物(biomarker), 而担子菌门(Basidiomycota)在R4扇区丰度最高(LDA score>2, P<0.05)。
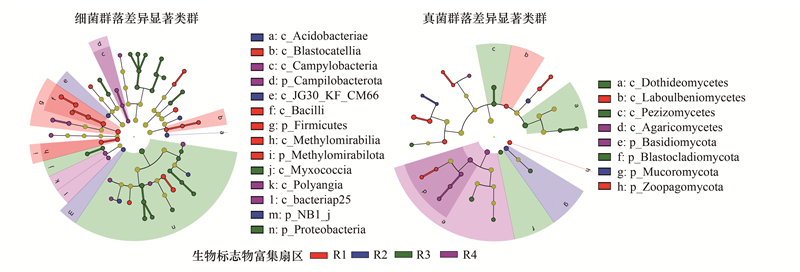
|
| 图 6 各冲积扇区细菌和真菌LEfSe生物标志物 Fig. 6 LEfSe biomakers of bacteria and fungi among alluvial sectors LEfSe:多级物种差异判别分析Linear discriminant analysis effect size; 图中从内之外辐射的圆圈代表了由门到属的分类级别, 不同级别的圆圈代表该水平下的一个分类单元, 小圆圈的直径大小代表相对丰度大小(log10转换)。树图中未展示无生物标志物的盲枝。黄色节点表示无显著差异的分类单元, 其他节点表示对冲积扇区间物种差异起重要作用的生物标志物(biomarker) |
富集于R3或R4扇区的细菌属, 包括Erythrobacter[26]、Fusibacter[27]、Gracilimonas[28]、Halanaerobium[29]、Limibaculum[30]、Marinobacter[31]、Palleronia-Pseudomaribius[32]、Pontibacter[33]、Salinimicrobium[34]、Zeaxanthinibacter[35]等, 经文献分析发现除Erythrobacter[26]外均为耐盐类群。富集于R4扇区的Campilobacterota为硫氧化化能自养细菌[36—38]。富集于R3或R4扇区的大型真菌为盘菌纲(Pezizomycetes)和烟管菌属(Bjerkandera), 而富集于R1的大型真菌为隶属于蘑菇科(Agaricaceae)的侧耳属(Pleurotus)。以上物种均对各冲积扇区的微生物群落结构组成的差异具有重要影响。
为控制海陆距离变量, 对距海20km附近的R1S3、R2S4、R3S4和R4S3等4个样地的细菌群落的属进行多级物种差异判别分析(Linear discriminant analysis Effect Size, LEfSe)。去除“norank”或“unclassified”开头的没有明确分类信息或分类名称的类群, 并去除平均丰度为0(Mean=0)的属, 最终得到554个细菌属。215个(38.8%)属对距海20km附近微生物群落结构组成的差异具有重要影响(LDA score>2, P<0.05), 其中73个属的显著富集区域为R4S3样地, 其他68、21、53个属分别为R3S4、R2S4和R1S3样地的生物标志物(biomarker)(表 4)。在这215个属中, 至少100个属为嗜盐细群, 其中有86个属(86.0%)的显著富集区域为成土较晚的R3和R4扇区;涉及反硝化、反硫化、光异养、嗜热等功能性状的细菌类群同样集中于R3和R4扇区;R4扇区显著富集的自养微生物多达13种, 其中10种可营化能自养。
| 功能表型 Function phenotypes |
样地名Names of sample sites | |||
| R1S3 | R2S4 | R3S4 | R4S3 | |
| 生物标志物总数Number of biomarkers | 53 | 21 | 68 | 73 |
| 嗜盐Halophilic | 6 | 8 | 41 | 45 |
| 可反硝化Denitrification | 3 | 1 | 10 | 10 |
| 可反硫化Desulphurization | 4 | 0 | 1 | 9 |
| 可自养Autotrophic | 4 | 5 | 1 | 13 |
| 可化能自养Chemoautotrophic | 0 | 4 | 1 | 10 |
| 可光异养Photoheterotrophic | 0 | 0 | 3 | 3 |
| 在LPSN(The List of Prokaryotic names with Standing in Nomenclature)数据库, 若一个细菌属下所有公开发表(validly published)的物种均有耐盐3%及以上或生长所需盐浓度的下限>0.5%, 则该属被识别为耐盐菌;若一个细菌属所有公开发表的物种均具备反硝化能力, 则该属被识别为反硝化菌;其他功能属的鉴定方法同上 | ||||
针对细菌通路, PICRUSt2分析了各扇区顶级景观群落功能通路的异质性。各样地细菌群落代谢通路相似, 但是代谢通路的丰度值不同, 其中有12个level 2代谢通路存在组间显著差异(图 7)。
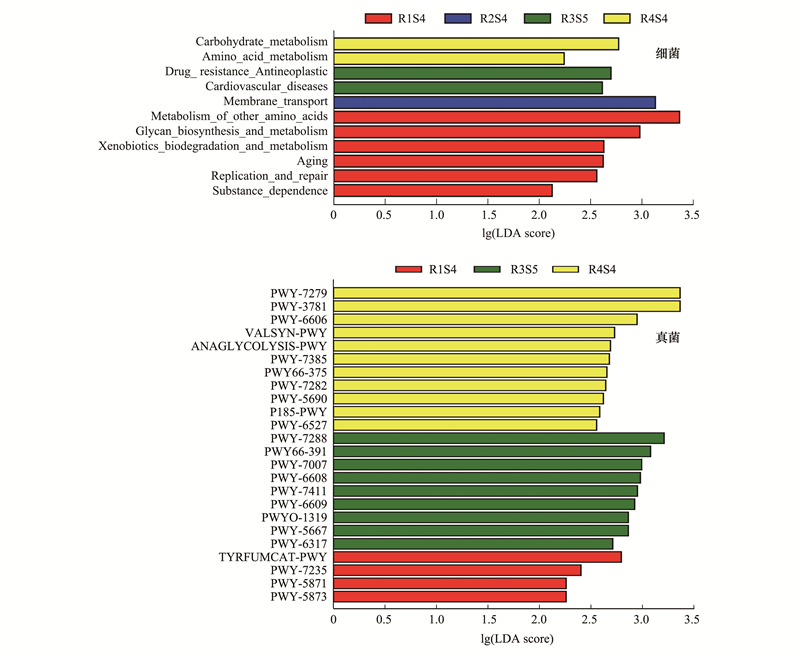
|
| 图 7 细菌和真菌群落KEGG途径丰度差异的线性判别分析(LDA)条形图 Fig. 7 Bar graphs of linear discrimination analysis (LDA) for the mean differences of KEGG pathway for bacteria and fungi communities LDA:线性判别分析Linear discriminant analysis; Carbohydrate metabolism:碳水化合物代谢;Amino acid metabolism:氨基酸代谢;Drug resistance_Antimicrobial:耐药性(抗菌药物);Cardiovascular diseases:心血管疾病相关;Membrane transport:跨膜运输;Metabolism of other amino acids:其他氨基酸代谢;Glycan biosynthesis and metabolism:甘氨酸的生物合成与代谢;Xenobiotics biodegradation and metabolism:外源物质的生物降解与代谢;Aging:细胞老化调控;Replication and repair:复制和修复;Substance dependence:物质依赖;PWY—7279:有氧呼吸Ⅰ(细胞色素c)(酵母类) aerobic respiration Ⅱ (cytochrome c) (yeast);PWY—3781:有氧呼吸Ⅰ(细胞色素c) aerobic respiration Ⅰ (cytochrome c);PWY—6606:鸟苷核苷酸降解Ⅱ guanosine nucleotides degradation Ⅱ;VALSYN—PWY:L-缬氨酸生物合成L-valine biosynthesis;PWY—7385:1, 3-丙二醇生物合成(基因改造的) 1, 3-propanediol biosynthesis (engineered);PWY66—375:leukotriene biosynthesis白三烯生物合成;PWY—7282:4-氨基-2-甲基-5-磷酸甲基嘧啶生物合成(酵母) 4-amino-2-methyl-5-phosphomethylpyrimidine biosynthesis (yeast);PWY—5690:三羧酸循环Ⅱ(植物和真菌) TCA cycle Ⅱ (plants and fungi);P185—PWY:甲醛同化Ⅲ(二羟丙酮循环) formaldehyde assimilation Ⅲ (dihydroxyacetone cycle), ;PWY—6527:水苏糖降解stachyose degradation;PWY—7288:脂肪酸-β-氧化(过氧化物酶体、酵母) fatty acid & beta;-oxidation (peroxisome, yeast);PWY66—391:脂肪酸-β-氧化Ⅵ(过氧化物酶体) fatty acid & beta;-oxidation Ⅵ (peroxisome);PWY—7007:甲基酮生物合成methyl ketone biosynthesis;PWY—6608:鸟苷核苷酸降解Ⅲ guanosine nucleotides degradation Ⅲ;PWY—7411:磷脂合成的超通道(酵母) superpathway of phosphatidate biosynthesis (yeast);PWY—6609:腺嘌呤和腺苷补救合成途径Ⅲ adenine and adenosine salvage Ⅲ;PWY0—1319:胞苷二磷酸-二酰甘油生物合成ⅡCDP-diacylglycerol biosynthesis Ⅱ;PWY—5667:胞苷二磷酸-二酰甘油生物合成Ⅰ CDP-diacylglycerol biosynthesis Ⅰ;PWY—6317:半乳糖降解Ⅰ(Leloir途径) galactose degradation Ⅰ (Leloir pathway);TYRFUMCAT—PWY:L-酪氨酸降解L-tyrosine degradation I;PWY—7235:泛醌-6生物合成的超途径(真核生物) superpathway of ubiquinol-6 biosynthesis (eukaryotic);PWY—5873:泛醌-7生物合成(真核生物) ubiquinol-7 biosynthesis (eukaryotic);PWY—5871:泛醌-9生物合成(真核生物) ubiquinol-9 biosynthesis (eukaryotic) |
R4S4样地显著富集的细菌群落功能通路较多涉及基础生命活动。在该样地的细菌群落, 碳水化合物代谢(Carbohydrate metabolism)和氨基酸代谢(Amino acid metabolism)两个level 2代谢通路的丰富度相对较高(LDA score>2, P<0.05, 图 7)。存在组间显著差异的level 3代谢通路中, 所有从属于遗传信息处理(level 1: Genetic Information Processing)的9个通路均富集于R4S4扇区, 涉及氨酰tRNA生物合成(ko00970)、核糖体(ko03010)、RNA降解(ko03018)、DNA复制(ko03030)、蛋白质运出(ko03060)、核苷酸切除修复(ko03420)、错配修复(ko03430)、同源重组(ko03440)和内质网蛋白质加工(ko04141)等生命活动必需的复制和表达过程(LDA score>2, P<0.05, 表 5)。此外, 涉及嘌呤代谢(ko00230)、嘧啶代谢(ko00240)、氨基酸生物合成(ko01230)等关键分子的通路的相对丰度均显著高于其他扇区(LDA score>2, P<0.05)。光合作用(ko00195)和光合生物固碳(ko00710)通路在R4S4样地细菌群落相对丰度较高(LDA score>2, P<0.05)。
| 代谢通路 Metabolism pathways |
KEGG编码 KEGG code |
分类 category |
LDA值 LDA score (log 10) |
差异通路富集样地 Sample site in which the pathway was enriched |
| DNA复制 DNA replication |
ko03030 | 基因信息处理 | 2.60 | R4S4 |
| 核苷酸切除修复 Nucleotide excision repair |
ko03420 | 基因信息处理 | 2.26 | R4S4 |
| 错配修复Mismatch repair | ko03430 | 基因信息处理 | 2.66 | R4S4 |
| 同源重组Homologous recombination | ko03440 | 基因信息处理 | 2.62 | R4S4 |
| 氨酰tRNA生物合成 Aminoacyl-tRNA biosynthesis |
ko00970 | 基因信息处理 | 2.71 | R4S4 |
| 核糖体Ribosome | ko03010 | 基因信息处理 | 3.33 | R4S4 |
| 糖酵解/糖异生 Glycolysis/Gluconeogenesis |
ko00010 | 碳水化合物代谢 | 2.61 | R1S4 |
| 戊糖和葡萄糖醛酸的相互转化 Pentose and glucuronate interconversions |
ko00040 | 碳水化合物代谢 | 2.21 | R1S4 |
| 果糖和甘露糖代谢 Fructose and mannose metabolism |
ko00051 | 碳水化合物代谢 | 2.46 | R1S4 |
| 半乳糖代谢 Galactose metabolism |
ko00052 | 碳水化合物代谢 | 2.35 | R1S4 |
| 淀粉和蔗糖代谢 Starch and sucrose metabolism |
ko00500 | 碳水化合物代谢 | 2.48 | R1S4 |
| 乙醛酸和二羧酸代谢 Glyoxylate and dicarboxylate metabolism |
ko00630 | 碳水化合物代谢 | 2.48 | R1S4 |
| 丙酸代谢 Propanoate metabolism |
ko00640 | 碳水化合物代谢 | 2.54 | R1S4 |
| 丁酸代谢 Butanoate metabolism |
ko00650 | 碳水化合物代谢 | 2.37 | R1S4 |
| C5支链二元酸代谢 C5-Branched dibasic acid metabolism |
ko00660 | 碳水化合物代谢 | 2.08 | R4S4 |
| 硫代谢Sulfur metabolism | ko00920 | 能量代谢 | 2.33 | R1S4 |
| 甲烷代谢Methane metabolism | ko00680 | 能量代谢 | 2.40 | R3S5 |
| 光合作用Photosynthesis | ko00195 | 能量代谢 | 2.51 | R4S4 |
| 光合生物的固碳作用 Carbon fixation in photosynthetic organisms |
ko00710 | 能量代谢 | 2.17 | R4S4 |
| 嘌呤代谢 Purine metabolism |
ko00230 | 核苷酸代谢 | 2.73 | R4S4 |
| 嘧啶代谢 Pyrimidine metabolism |
ko00240 | 核苷酸代谢 | 2.74 | R4S4 |
| 芳香族化合物的降解 Degradation of aromatic compounds |
ko01220 | 全局代谢 | 2.58 | R1S4 |
| 2-氧代羰基酸代谢 2-Oxocarboxylic acid metabolism |
ko01210 | 全局代谢 | 2.74 | R4S4 |
| 氨基酸的生物合成 Biosynthesis of amino acids |
ko01230 | 全局代谢 | 3.31 | R4S4 |
| 表中KEGG编号为细菌群落PICRUSt2(Phylogenetic Investigation of Communities by Reconstruction of Unobserved States 2)分析结果中存在组间显著差异存在组间显著差异(LDA score>2, P<0.05)的Level 3代谢通路的KEGG数据库编号, 分类指KEGG数据库的高级分类单元:基因信息处理:Genetic information processing (level 1); 碳水化合物代谢:Carbohydrate metabolism (level 2); 能量代谢:Energy metabolism (level 2); 核苷酸代谢:Nucleotide metabolism (level 2); 全局代谢:Global and overview maps (level 2); 表中列出的差异通路克鲁斯卡尔-沃利斯秩和检验(Kruskal-Wallis rank sum test)均为统计学显著(P<0.05) | ||||
R1S4样地细菌群落富集的细菌群落功能通路则涉及多样化的代谢过程。涉及细胞老化(Aging)、外源物质的生物降解与代谢(Xenobiotics biodegradation and metabolism)、复制与修复(Replication and repair)等level 2通路丰度高于其他样地(LDA score>2, p<0.05, 图 7)。在营养代谢方面, R1扇区有关单糖的通路丰度较高, 如糖酵解/糖异生(ko00010)、戊糖和葡萄糖醛酸转化(ko00040)、果糖和甘露糖代谢(ko00051)、半乳糖代谢(ko00052)、丁酸(ko00650)等(LDA score>2, P<0.05, 表 5)。芳香族化合物降解(ko01220)的代谢功能也富集于R1S4样地(LDA score>2, P<0.05)。
针对真菌通路, PICRUSt2分析得到了各扇区顶级景观群落MetaCyc pathway信息及其丰度, 共有25条MetaCyc代谢通路存在组间显著差异(图 7)。R4S4样地显著富集的真菌代谢通路亦较多涉及基础代谢, 如TCA循环(PWY-5690)、丙二醇生物合成(PWY-7385)、有关细胞色素c的有氧呼吸I(PWY-3781、PWY-7279)和葡萄糖酵解(ANAGLYCOLYSIS-PWY)通路(LDA score>2, p<0.05, 图 7)。R1S4样地显著富集的主要是有氧呼吸相关的泛醌类合成通路, 如泛醌-6(PWY-7235合成)、泛醌-7(PWY-7235)和泛醌-9合成(PWY-7235)(LDA score>2, P<0.05)。
2.6 成土时间驱动力的统计学检验成土时间对植物、细菌、真菌的群落组成变异的解释能力分别为4.76%、2.98%、3.68%(图 8), 均大于全球范围土壤年龄对生态系统指标的平均解释能力2.1%[6], 表明成土时间在黄河三角洲是重要的生态驱动力。成土时间对植物、细菌、真菌的群落组成变异的独特方差解释比例分别为3.41%、2.61%、1.12%, 独特解释比例大于0, 说明成土时间对生物群落组成变化的影响不能被其他状态因子完全代替, 因此成土时间是为黄河三角洲生物演替带来额外解释度的显著驱动因子。
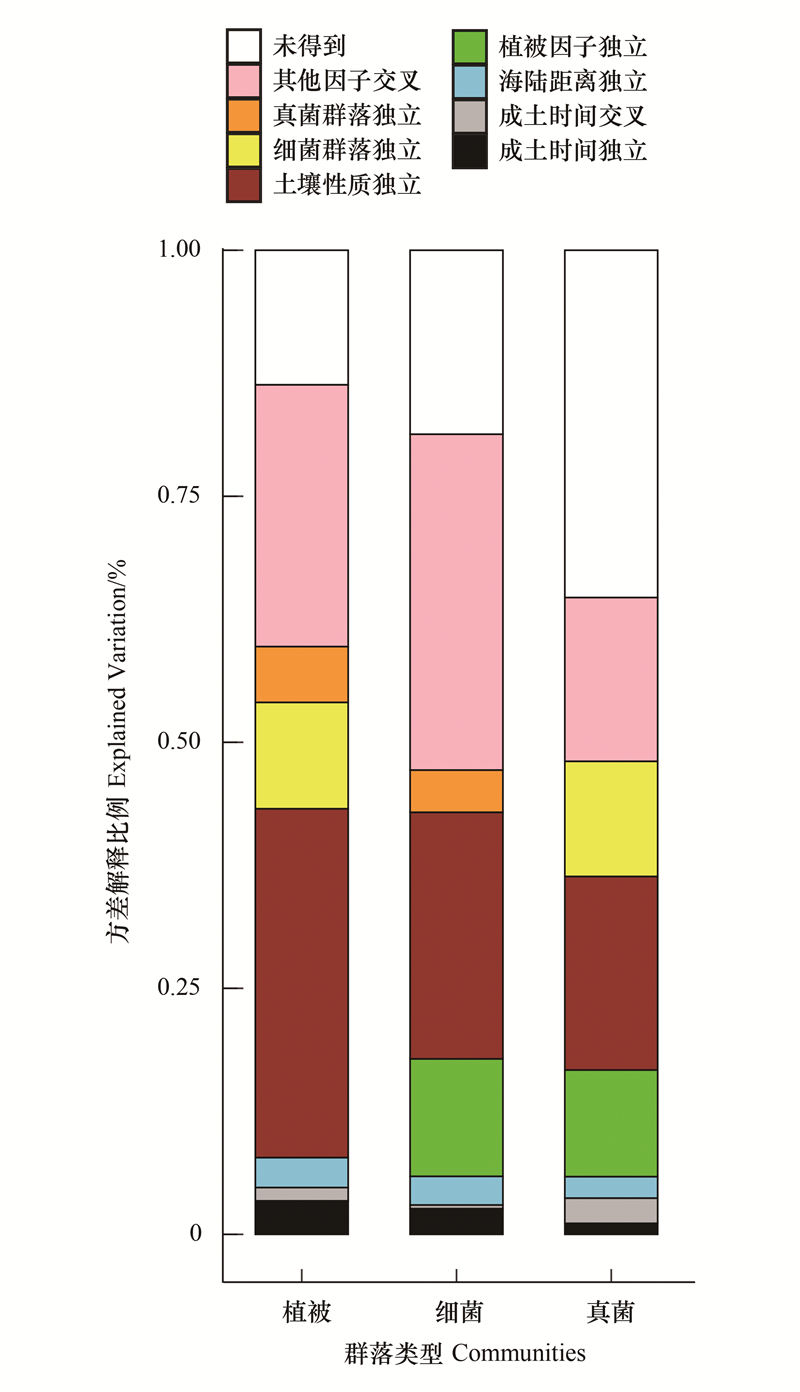
|
| 图 8 植被和微生物群落组成的方差分解分析 Fig. 8 Variance decomposition analysis (VPA) of vegetation and microbial community composition 色柱表示相关环境因子(或组合)对植被、细菌和真菌群落组成的独立方差解释度代表其对群落变化的贡献;植被因子:植物物种丰富度、Shannon多样性、植被盖度、地上部干生物量;土壤性质:土壤有机质、全氮、全磷、全钾、碱解氮、速效磷、速效钾、碳氮比、电导率、酸碱度、含水量;细菌群落:细菌OTU水平的Sobs指数、Shannon多样性、占比前8门类的相对丰度;真菌群落:细菌OTU水平的Sobs指数、Shannon多样性、占比前8门类的相对丰度;其他因子交叉:成土时间之外的环境因子对群落变化的方差解释度的重叠部分;未得到:以上因子均未能解释的残差 |
成土时间对细菌和真菌群落组成矩阵均显著偏相关(Partial Mantel P<0.05)(表 6), 表明成土时间单一因子对微生物演替过程中群落物种组成有极显著驱动力。若不加入成土时间, 细菌和真菌群落的Bioenv模型的相关系数相对最佳模型的拟合精度分别相对降低1.41%、5.78%(表 7), 表明在黄河三角洲微生物演替研究中将成土时间纳入考虑可提高拟合精度。
| 群落 Communities |
检验因子 Test factor |
控制因子数目 Number of variables controlled |
曼特尔相关系数 Mantel r |
曼特尔P值 Mantel P-value |
置换检验次数 Permutations |
| 植被Vegetation | soil.age | 37 | 0.0383 | 0.1761 | 9999 |
| 细菌Bacteria | soil.age | 27 | 0.1377 | 0.0005 | 9999 |
| 真菌Fungi | soil.age | 27 | 0.1490 | 0.0105 | 9999 |
| 以图 8中除成土时间之外的所有环境因子(distance、vegetation、soil properties、bacteria、fungi)构建控制环境矩阵(Euclidean距离算法), 检验植被、细菌、真菌群落的物种矩阵(Bray-Curtis距离算法)的可变性能够被成土时间解释的部分 | |||||
| 群落 Communities |
加入成土时间 Soil age was in consideration |
最佳环境因子组合 Optimal combination of environmental factors |
拟合系数 fitting coefficient |
| 细菌Bacteria | 是 | soil.age, distance, SOM, TN, pH, EC, plant.cov, shannon.cov | 85.00% |
| 否 | distance, SOM, TN, pH, EC, plant.cov, shannon.cov | 83.80% | |
| 真菌Fungi | 是 | soil.age, distance, pH, EC, AP, shannon.cov | 58.52% |
| 否 | distance, pH, EC, AP, shannon.cov | 53.25% | |
| 选取河口海岸距离、植被因子、土壤理化性质等17个环境因子为对照, 检验加入成土时间对Bioenv分析得到的最佳环境因子组合及其拟合系数的影响 | |||
由于成土时间带来演替时间的梯度, 各扇区内陆植物群落结构呈由简单向复杂的演变趋势。研究区域内, 植物分布和多样性在随成土时间的差异主要体现在生物量(r=0.53, P<0.001)和植物物种数(r=0.29, P<0.05)。距海约20km的自然植被样地呈现了植被类型由低级向高级的过渡特征。生态系统的空间演替体现了成土时间对自然植被由低级向高级发展的驱动作用。刘志杰[39]的样方调查同样发现, 成土时间在1964年后的黄河三角洲叶瓣植物种类较少, 植物种类不超过10科;灌草丛在成陆时期较长的区域成为主要植被类型之一。陈彭祯霓等[40]以冰川退缩区百年时间跨度的植被初生演替为研究对象, 发现不同演替时间的样地植物物种组成差异较大, 木本植物生物量随演替时间增加而增加;黄河三角洲植物群落结构演替规律与之相符, 丰富了百年尺度的植被初生演替规律的研究。
随成土历史的延长, 白茅的分布向海岸推进, 体现了成土时间对近海土壤生境条件改善的驱动作用。白茅的出现代表演替到达较高阶段, 种群结构趋于稳定[41]。本研究中, 在白茅分布地点, 植被的共同点为植被盖度在70%以上。在R1扇区, 白茅为常见种。在R4、R3和R2冲积扇区, 白茅分别在距入海口约20km、14km及9km进入群落并成为伴生种。R4冲积扇区未演替出白茅占优的草地群落, 白茅仅在人类聚落附近偶见;R3S4样地是在R3扇区发现的距海最近的白茅群落, 白茅盖度约25%;而在距海仅15.8km的R2S3样地, 白茅盖度已达40%。前人研究证实, 白茅是黄河三角洲盐土改良的指示植物, 如侯本栋等[2]发现, 白茅群落的土壤有机质、全氮含量显著高于柽柳群落、盐地碱蓬群落和裸地, 且含盐量相对较低。
3.2 成土时间对黄河三角洲微生物演替的影响成土时间的增加可驱动细菌和真菌群落结构(OTU的Bray-Curtis距离矩阵)的改变, 这种改变具有一定的方向性(Partial Mantel r≥0.1377, P<0.05, 表 6)。除去土壤理化性质、植物互作关系等的共线性干扰后, 成土时间对微生物群落变化依然具有独特影响;成土时间可为群落变化趋势提供额外的解释度(图 8, 表 7)。成土时间在黄河三角洲局地尺度是独特而显著的重要生态驱动力(图 8), 这与Delgado-Baquerizo等[6]对土壤年龄驱动力的整合分析相符, 即成土时间主要影响与生物活动相关的缓慢变化的生态指标。
微生物群落的功能特征也随成土时间增加而发生显著改变(图 7, 表 5)。在成土时期较近的区域, 微生物群落涉及基因信息处理等基础生命活动的功能通路相对丰度较高, 与Yang等[42]关于黄河三角洲氨基酸代谢通路在高盐土壤丰度更高的发现一致, 推测在演替初期环境胁迫下, 微生物群落依靠功能冗余提高抗逆性[43]及维持群落功能的稳定性[44—45];也可能是响应于频繁而幅度较大的环境变化, 微生物需要能够迅速利用周边资源生存、生长和繁殖[21], 故采取r-选择竞争策略[46]。在生态系统年龄较长的R1扇区, 涉及细胞老化调控等功能通路的丰度显著较高(LDA score>2, P<0.05)符合环境趋于稳定带来选择过程优势的增加[47]的理论;单糖代谢、芳香族化合物降解等碳代谢相关基因通路的丰度发生显著变化(表 5), 为前人关于微环境因素影响微生物碳源利用[42, 48]、土壤发育对生态位分化有促进作用[21]等论点提供了支撑。
功能微生物的富集特征是微生物群落组成随成土时间变化的原因之一。LEfSe分析表明, 在各扇区距海20km附近的各样地, 对细菌群落组成变异贡献显著的215个可识别属中, 嗜盐细菌、反硝化细菌、反硫化细菌等功能微生物的显著富集区域均为成土较晚的R3和R4扇区(表 4), 其优势度随演替时间有下降趋势。对不同扇区细菌群落组成的整体差异起关键作用的细菌属也以耐盐菌为主, 集中区域同样为成土较晚的R3和R4扇区(图 6)。范运梁[49]依据微生物培养实验发现, 伴随盐生植物的生长, 耐盐细菌的比例显著下降。苗菁等[11]还发现, 随着植被的覆盖以及植物源有机物的输入, 盐度对土壤细菌属的作用逐渐减弱。耐盐细菌类群的减少趋势与盐度随演替降低的趋势[8, 21]相适应。随盐土向农田土壤演替, 根系生长可改善土壤结构、降低土壤容重、增加土壤孔隙[50], 通气性的增加促进好氧型生物活性的增加[51], 而反硝化、反硫化均为厌氧过程[52—53], 导致反硝化菌与反硫化菌竞争力随成土时间增加的下降。
成土时间引起部分适应高营养环境的类群相对丰度的增加, 以及部分先锋类群相对丰度的降低。酸杆菌门(Acidobacteriota)在环境土壤中一般占细菌总量20%左右[54], 在土壤营养较高的黑土地或森林土壤, 酸杆菌门占比更高[55—57]。酸杆菌相对丰度平均占比仅为8.26%(表 3), 苗永君[12]在黄河三角洲细菌多样性的研究同样发现, 酸杆菌在农用地和自然植被土地类型的相对丰度占比均<10%。而且, 酸杆菌随土壤有机质增加(r=-0.47, P<0.001)和电导率下降(r=-0.39, p<0.01)而增加(图 5), 表明其在黄河三角洲属于随生境改良而增加的类群。拟杆菌门(Bacteroidota)、Sumerlaeota、Hydrogenedentes、海微菌门SAR406分支(Marinimicrobia\SAR406-clade)、异常球菌门(Deinococcota)和浮霉菌门(Gemmatimonadota)则随土壤有机质增加(r<-0.33, P<0.01)和电导率降低(r>0.33, P<0.01)而出现相对丰度的显著下降, 是优势度随土壤改良而降低的先锋类群。在成土早期相对贫瘠的极端生境, 适应寡营养生境的先锋微生物容易取得较高优势度[10, 21], 但是土壤营养限制的解除不利于这些种群的壮大[58], 如李辉等[59]发现, 在赤泥自然成土的过程中, 富营养细菌与贫营养细菌丰度比值随演替明显增加。
成土时间的增加驱动微生物生态位的分化。细菌群落功能通路的分化趋势(图 6)体现了微生物生态位的分化。营养方式的多样化同样是微生物生态位分化的体现, 如寄生土壤线虫、轮虫和变形虫的捕虫霉门[60](Zoopagomycota)真菌(r=0.30, P<0.05), 寄生昆虫的黑僵菌[61](Metarhizium anisopliae)真菌(r=0.32, P<0.05), 寄生变形虫的Neochlamydia[62]细菌(r=0.26, P<0.05), 与共生固氮的Ensifer[63](r=0.31, P<0.05)和Azoarcus[64](r=0.33, P<0.01)细菌均为优势度随成土时间增加而增大的类群, 演替初期此类物种受制于宿主资源的贫乏。在对距海20km附近样地细菌群落组成的对比中, 还发现与反刍动物共生的瘤胃球菌属[65](Ruminococcus)在R1S3样地的显著富集(LDA score=2.28, P<0.05), 说明随演替时间而增强[3]的人类活动能为微生物提供更多可选生态位。
3.3 成土时间驱动力的微生物因素植被是滨海生态系统发展其生态系统功能最关键的过程[10]。盐生植物盖度提高可减弱土壤的自然蒸腾, 抑制盐分在土壤表层的持续累积;翅碱蓬等真盐生植物能大量吸收盐分, 使土壤脱盐[66]。盐生植被改善土壤质量的作用为微生物提供良好的生存条件, 植物根系的分泌物及植物残体是土壤微生物的主要可利用的碳源[58, 67]。
但是, 成土初期盐滩光板地的存在表明先锋植物的定居需要等待土壤的形成和改良过程。在极端生境下, 高等植物无法存活, 但细菌群落物种数和多样性较少受环境因子影响(图 5, 表明某些适应力较强的菌种可定居。在微生物定居于原生裸地的同时, 即开始了成土进程[68], 成土初期的功能微生物促进了生境的改良。
自养微生物促进土壤碳氮积累。光合作用相关通路富集在成土相对较早的扇区, 这与Wang等[10]光合作用相关基因显著富集于新生湿地的发现一致。蓝藻细菌门能够进行产氧的光合作用, 部分物种能进行生物固氮, 还可以通过光合作用释放特异的蛋白质信号分子来调控植物的生长和发育[67]。但是, 本研究中蓝藻门(Cyanobacteria)相对丰度与成土时间无显著相关性(P>0.05, 图 4), 暗示蓝藻以外的光合生物贡献于细菌群落光合作用表型的富集。本研究在16S rDNA克隆文库中发现了Candidatus Chloroploca[69]、Rhodobacter[70]等光合硫氧化细菌以及光异养的Rubribacterium[71]、Rubellimicrobium[71]等紫色非硫细菌, 还发现了距海20km附近发现的光异养细菌在R3、R4扇区的富集(表 4), 此类光合生物很可能对成土早期的细菌光合作用起到了关键作用。在R4S3扇区中还发现了化能自养细菌的富集现象(表 4), 包括3种耐盐的自养硝化菌和6个可硫氧化自养的细菌属。前人强调成土早期以蓝藻为主的光合自养微生物对土壤碳氮的积累起到关键作用[59, 72], 本研究推测滨海盐土化能自养菌和蓝藻以外的光合生物在土壤碳氮积累方面的作用同样具有生态学意义, 这与近海河流沉积物硫元素的富集和活跃的元素化学循环[73]相适应, 有待实验验证。除蓝藻和自养硝化细菌外, 耐盐固氮菌也具有促进土壤氮积累的功能潜力。16S rDNA克隆文库中发现的耐盐的共生固氮菌Ensifer[63]和Azoarcus[64]分别可以与豆科植物和禾本科植物形成根瘤;耐碱的中慢生根瘤菌属[74—75](Mesorhizobium)也有为先锋植物提供了早期土壤所缺乏的氮源的潜力。
成土初期, 耐盐细菌能促进土壤养分循环和有效性的提升。耐盐细菌对成土初期的有机质矿化起到关键作用[1, 8, 11], 盐渍土中丰富的盐生细菌对碳代谢和循环至关重要[42]。在潮滩、湿地地区, 常年积水的水位条件会形成的厌氧环境, 造成较慢的碳库周转速率[18], 而这种环境下, 部分微生物还具有以无机离子为电子受体驱动有机质的矿化的潜力。在16S rDNA克隆文库中, 识别到了Dethiosulfatibacter[76]、Desulfotignum[77]等能够以SO42-为电子受体的耐盐厌氧菌, Calorithrix[78]、Thiohalorhabdus[79]等能够以NO3-为电子受体的耐盐厌氧菌, 以及Anaerosolibacter[80]、Deferrisoma[81]等能够以Fe3+为电子受体的耐盐厌氧菌, 以上微生物也有能力促进氮、硫、铁等元素的循环。
李辉等[59]还发现, 微生物可通过分泌有机酸、菌丝改良土壤结构等方式参与土壤的改良。微生物能够释放土壤酶, 催化氧化还原、有机质矿化、腐殖物质在土壤中的合成、以及生长活性物的释放等多种生物化学反应[22], 改善植物的生长环境。盐生植被与微生物循环反复作用[18], 最终实现滨海盐土向农田的转化。
4 结论成土时间对细菌和真菌群落变化具有显著的偏相关性(Mantel P<0.05), 对植物、细菌和真菌群落组成变化的解释能力高于全球尺度土壤年龄对生态系统指标的平均解释能力。成土时间驱动黄河三角洲植物、细菌和真菌群落组成变化, 也造成黄河三角洲微生物群落的功能特征的显著异质性。在百年尺度上, 成土时间是黄河三角洲植物、细菌和真菌群落演替的重要而显著的驱动力, 将成土时间纳入研究变量能够为生物群落演替的研究提供额外的解释度。该结论为提高评估滨海生态系统植物和微生物演替规律的准确性提供了科学依据。
| [1] |
郗金标, 宋玉民, 邢尚军, 马丙尧, 张建锋, 常丽华. 黄河三角洲生态系统特征与演替规律. 东北林业大学学报, 2002, 30(6): 111-114. DOI:10.3969/j.issn.1000-5382.2002.06.034 |
| [2] |
侯本栋, 马风云, 邢尚军, 宋玉民, 刘艳. 黄河三角洲不同演替阶段湿地群落的土壤和植被特征. 浙江林学院学报, 2007, 24(3): 313-318. |
| [3] |
叶庆华, 田国良, 刘高焕, 叶景敏, 娄维国. 黄河三角洲新生湿地土地覆被演替图谱. 地理研究, 2004, 23(2): 257-264, 282. DOI:10.3321/j.issn:1000-0585.2004.02.014 |
| [4] |
王树功. 珠江河口区典型湿地景观演变及调控研究[D]. 广州: 中山大学, 2005.
|
| [5] |
姚轶锋, 曹叔楠, 彭方, 李承森. 北极新奥尔松Austre Lovénbreen冰川退缩迹地不同演替阶段的植物组成与植被群落特征分析. 极地研究, 2014, 26(3): 362-368. |
| [6] |
Delgado-Baquerizo M, Reich P B, Bardgett R D, Eldridge D J, Lambers H, Wardle D A, Reed S C, Plaza C, Png G K, Neuhauser S, Berhe A A, Hart S C, Hu H W, He J Z, Bastida F, Abades S, Alfaro F D, Cutler N A, Gallardo A, García-Velázquez L, Hayes P E, Hseu Z Y, Pérez C A, Santos F, Siebe C, Trivedi P, Sullivan B W, Weber-Grullon L, Williams M A, Fierer N. The influence of soil age on ecosystem structure and function across biomes. Nature Communications, 2020, 11: 4721. DOI:10.1038/s41467-020-18451-3 |
| [7] |
Johnson W B, Sasser C E, Gosselink J G. Succession of vegetation in an evolving river delta, Atchafalaya Bay, Louisiana. The Journal of Ecology, 1985, 73(3): 973. DOI:10.2307/2260162 |
| [8] |
吴志芬, 赵善伦, 张学雷. 黄河三角洲盐生植被与土壤盐分的相关性研究. 植物生态学报, 1994, 18(2): 184-193. DOI:10.3321/j.issn:1005-264X.1994.02.010 |
| [9] |
余悦. 黄河三角洲原生演替中土壤微生物多样性及其与土壤理化性质关系[D]. 济南: 山东大学, 2012.
|
| [10] |
Wang J N, Wang J J, Zhang Z, Li Z F, Zhang Z G, Zhao D C, Wang L D, Lu F, Li Y Z. Shifts in the bacterial population and ecosystem functions in response to vegetation in the Yellow River Delta wetlands. mSystems, 2020, 5(3): e00412-e00420. |
| [11] |
苗菁, 宗万松, 吕敏, 丁晶, 王晓凤, 郭笃发. 基于高通量测序技术研究土壤细菌对盐生植被演替的响应. 土壤通报, 2018, 49(3): 602-608. DOI:10.19336/j.cnki.trtb.2018.03.15 |
| [12] |
苗永君. 黄河三角洲不同土地利用类型土壤细菌和氮循环功能菌群研究[D]. 济南: 山东大学, 2019.
|
| [13] |
陈述彭, 赵英时. 遥感地学分析. 北京: 遥感地学分析, 1990.
|
| [14] |
叶庆华, 陈沈良, 黄翀, 薛允传, 田国良, 陈述彭, 石亚男, 刘庆生, 刘高焕. 近、现代黄河尾闾摆动及其亚三角洲体发育的景观信息图谱特征. 中国科学: D辑: 地球科学, 2007, 37(6): 813-823. |
| [15] |
崔步礼, 常学礼, 陈雅琳, 董琼, 李维庆. 黄河水文特征对河口海岸变化的影响. 自然资源学报, 2006, 21(6): 957-964. DOI:10.3321/j.issn:1000-3037.2006.06.013 |
| [16] |
李栓科. 近代黄河三角洲的沉积特征. 地理研究, 1989, 8(4): 45-55. |
| [17] |
邢平平. 黄河三角洲土壤微生物群落结构对盐生植被演替的响应[D]. 济南: 山东师范大学, 2013.
|
| [18] |
刘兴华, 章海波, 李远, 代振飞, 付传城, 骆永明. 黄河三角洲滩涂——湿地——旱地土壤团聚体有机质组分变化规律. 土壤学报, 2019, 56(2): 374-385. |
| [19] |
Shi H Y, Li Q J, Sun J C, Gao G D, Sui Y, Qiao S W, You Z J. Variation of Yellow River runoff and its influence on salinity in Laizhou Bay. Journal of Ocean University of China, 2020, 19(6): 1235-1244. DOI:10.1007/s11802-020-4413-5 |
| [20] |
Brown M V, Philip G K, Bunge J A, Smith M C, Bissett A, Lauro F M, Fuhrman J A, Donachie S P. Microbial community structure in the North Pacific Ocean. The ISME Journal, 2009, 3(12): 1374-1386. DOI:10.1038/ismej.2009.86 |
| [21] |
周汉昌, 马安周, 刘国华, 庄国强. 冰川消退带微生物群落演替及生物地球化学循环. 生态学报, 2018, 38(24): 9021-9033. |
| [22] |
叶协锋, 张友杰, 鲁喜梅, 魏跃伟, 李琰琰, 刘国顺. 土壤微生物与土壤营养关系研究进展. 土壤通报, 2010, 41(1): 237-241. DOI:10.19336/j.cnki.trtb.2010.01.048 |
| [23] |
白世红, 马风云, 侯栋, 王迪. 黄河三角洲植被演替过程种群生态位变化研究. 中国生态农业学报, 2010, 18(3): 581-587. |
| [24] |
马克平, 刘玉明. 生物群落多样性的测度方法Ⅰα多样性的测度方法(下). 生物多样性, 1994, 2(4): 231-239. DOI:10.3321/j.issn:1005-0094.1994.04.009 |
| [25] |
Stackebrandt E, Goebel B M. Taxonomic note: a place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology. International Journal of Systematic and Evolutionary Microbiology, 1994, 44(4): 846-849. DOI:10.1099/00207713-44-4-846 |
| [26] |
Koch C, Kroppenstedt R M, Rainey F A, Stackebrandt E. 16S ribosomal DNA analysis of the genera Micromonospora, Actinoplanes, Catellatospora, Catenuloplanes, Couchioplanes, Dactylosporangium, and Pilimelia and emendation of the family Micromonosporaceae. International Journal of Systematic Bacteriology, 1996, 46(3): 765-768. DOI:10.1099/00207713-46-3-765 |
| [27] |
Ravot G, Magot M, Fardeau M L, Patel B K C, Thomas P, Garcia J L, Ollivier B. Fusibacter paucivorans gen. nov., sp. nov., an anaerobic, thiosulfate-reducing bacterium from an oil-producing well. International Journal of Systematic and Evolutionary Microbiology, 1999, 49(3): 1141-1147. DOI:10.1099/00207713-49-3-1141 |
| [28] |
Choi D H, Zhang G I, Noh J H, Kim W S, Cho B C. Gracilimonas tropica gen. nov., sp. nov., isolated from a Synechococcus culture. International Journal of Systematic and Evolutionary Microbiology, 2009, 59(Pt 5): 1167-1172. |
| [29] |
Zeikus J G, Hegge P W, Thompson T E, Phelps T J, Langworthy T A. Isolation and description of Haloanaerobium praevalens gen. nov. and sp. nov., an obligately anaerobic halophile common to Great Salt Lake sediments. Current Microbiology, 1983, 9(4): 225-233. DOI:10.1007/BF01567586 |
| [30] |
Shin Y H, Kim J H, Suckhoom A, Kantachote D, Kim W. Limibaculum halophilum gen. nov., sp. nov., a new member of the family Rhodobacteraceae. International Journal of Systematic and Evolutionary Microbiology, 2017, 67(10): 3812-3818. DOI:10.1099/ijsem.0.002200 |
| [31] |
Gauthier M J, Lafay B, Christen R, Fernandez L, Acquaviva M, Bonin P, Bertrand J C. Marinobacter hydrocarbonoclasticus gen. nov., sp. nov., a new, extremely halotolerant, hydrocarbon-degrading marine bacterium. International Journal of Systematic Bacteriology, 1992, 42(4): 568-576. DOI:10.1099/00207713-42-4-568 |
| [32] |
Martínez-Checa F, Quesada E, Martínez-Cánovas M J, Llamas I, Béjar V. Palleronia marisminoris gen. nov., sp. nov., a moderately halophilic, exopolysaccharide-producing bacterium belonging to the 'Alphaproteobacteria', isolated from a saline soil. International Journal of Systematic and Evolutionary Microbiology, 2005, 55(6): 2525-2530. DOI:10.1099/ijs.0.63906-0 |
| [33] |
Xu M B, Wang Y, Dai J, Jiang F, Rahman E, Peng F, Fang C X. Pontibacter populi sp. nov., isolated from the soil of a Euphrates poplar (Populus euphratica) forest. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(Pt 3): 665-670. |
| [34] |
Lim J M, Jeon C O, Lee S S, Park D J, Xu L H, Jiang C L, Kim C J. Reclassification of Salegentibacter catena Ying et al. 2007 as Salinimicrobium catena gen. nov., comb. nov. and description of Salinimicrobium xinjiangense sp. nov., a halophilic bacterium isolated from Xinjiang Province in China. International Journal of Systematic and Evolutionary Microbiology, 2008, 58(2): 438-442. DOI:10.1099/ijs.0.65297-0 |
| [35] |
Lee Y, Jeong H I, Jeong S E, Jeon C O. Zeaxanthinibacter aestuarii sp. nov., isolated from estuary sediment and emended description of the genus Zeaxanthinibacter Asker et al. 2007. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(9): 3264-3269. DOI:10.1099/ijsem.0.001185 |
| [36] |
Inagaki F, Ken T K, Nealson K H, Horikoshi K. Sulfurovum lithotrophicum gen. nov., sp. nov., a novel sulfur-oxidizing chemolithoautotroph within the ε-Proteobacteria isolated from Okinawa Trough hydrothermal sediments. International Journal of Systematic and Evolutionary Microbiology, 2004, 54(5): 1477-1482. DOI:10.1099/ijs.0.03042-0 |
| [37] |
Inagaki F. Sulfurimonas autotrophica gen. nov., sp. nov., a novel sulfur-oxidizing-proteobacterium isolated from hydrothermal sediments in the Mid-Okinawa Trough. International Journal of Systematic and Evolutionary Microbiology, 2003, 53(6): 1801-1805. |
| [38] |
Kodama Y, Watanabe K. Sulfuricurvum kujiense gen. nov., sp. nov., a facultatively anaerobic, chemolithoautotrophic, sulfur-oxidizing bacterium isolated from an underground crude-oil storage cavity. International Journal of Systematic and Evolutionary Microbiology, 2004, 54(Pt 6): 2297-2300. |
| [39] |
刘志杰. 黄河三角洲滨海湿地环境区域分异及演化研究[D]. 青岛: 中国海洋大学, 2013.
|
| [40] |
陈彭祯霓, 任亮晶, 罗辑, 孙守琴, 刘巧, 张伟, 戴巡. 海螺沟冰川退缩区原生演替序列植被物种多样性与生物量的关系. 应用与环境生物学报, 2021, 28(3): 1-10. |
| [41] |
房用, 王淑军, 刘月良, 上官秀玲, 王月海, 梁玉. 现代黄河三角洲的植被群落演替阶段. 东北林业大学学报, 2008, 36(9): 89-93. DOI:10.3969/j.issn.1000-5382.2008.09.031 |
| [42] |
Yang C, Lv D T, Jiang S Y, Lin H, Sun J Q, Li K J, Sun J. Soil salinity regulation of soil microbial carbon metabolic function in the Yellow River Delta, China. Science of the Total Environment, 2021, 790: 148258. DOI:10.1016/j.scitotenv.2021.148258 |
| [43] |
李晶, 刘玉荣, 贺纪正, 郑袁明. 土壤微生物对环境胁迫的响应机制. 环境科学学报, 2013, 33(4): 959-967. |
| [44] |
Wohl D L, Arora S, Gladstone J R. Functional redundancy supports biodiversity and ecosystem function in a closed and constant environment. Ecology, 2004, 85(6): 1534-1540. DOI:10.1890/03-3050 |
| [45] |
Chaer G, Fernandes M, Myrold D, Bottomley P. Comparative resistance and resilience of soil microbial communities and enzyme activities in adjacent native forest and agricultural soils. Microbial Ecology, 2009, 58(2): 414-424. |
| [46] |
Pianka E R. On r- and K-selection. The American Naturalist, 1970, 104(940): 592-597. DOI:10.1086/282697 |
| [47] |
Dini-Andreote F, Stegen J C, van Elsas J D, Salles J F. Disentangling mechanisms that mediate the balance between stochastic and deterministic processes in microbial succession. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(11): E1326-E1332. |
| [48] |
Chen L J, Li C S, Feng Q, Wei Y P, Zheng H, Zhao Y, Feng Y J, Li H Y. Shifts in soil microbial metabolic activities and community structures along a salinity gradient of irrigation water in a typical arid region of China. Science of the Total Environment, 2017, 598: 64-70. DOI:10.1016/j.scitotenv.2017.04.105 |
| [49] |
范运梁. 盐生植物对黄河三角洲微生物多样性的影响[D]. 济南: 山东师范大学, 2010.
|
| [50] |
朱奕豪, 刘晓丽, 陈为峰, 宋希亮, 林雪婷, 牛旭昌, 刘萍. 黄河三角洲废弃盐田复垦土壤碳氮磷生态化学计量学特征. 水土保持学报, 2020, 34(6): 352-360. |
| [51] |
Zhang X F, Xin X L, Zhu A N, Yang W L, Zhang J B, Ding S J, Mu L, Shao L L. Linking macroaggregation to soil microbial community and organic carbon accumulation under different tillage and residue managements. Soil and Tillage Research, 2018, 178: 99-107. |
| [52] |
王海涛, 郑天凌, 杨小茹. 土壤反硝化的分子生态学研究进展及其影响因素. 农业环境科学学报, 2013, 32(10): 1915-1924. |
| [53] |
徐永波. 废水生物脱氮除硫特性研究[D]. 哈尔滨: 哈尔滨工业大学, 2007.
|
| [54] |
王光华, 刘俊杰, 于镇华, 王新珍, 金剑, 刘晓冰. 土壤酸杆菌门细菌生态学研究进展. 生物技术通报, 2016, 32(2): 14-20. |
| [55] |
Liu J J, Sui Y Y, Yu Z H, Shi Y, Chu H Y, Jin J, Liu X B, Wang G H. Soil carbon content drives the biogeographical distribution of fungal communities in the black soil zone of northeast China. Soil Biology and Biochemistry, 2015, 83: 29-39. |
| [56] |
Fierer N, Bradford M A, Jackson R B. Toward an ecological classification of soil bacteria. Ecology, 2007, 88(6): 1354-1364. |
| [57] |
Navarrete A A, Kuramae E E, de Hollander M, Pijl A S, van Veen J A, Tsai S M. Acidobacterial community responses to agricultural management of soybean in Amazon forest soils. FEMS Microbiology Ecology, 2013, 83(3): 607-621. |
| [58] |
崔军. 河口湿地围垦后长期耕作下土壤理化性质演变、碳固定机制及细菌群落演替的研究[D]. 上海: 复旦大学, 2011.
|
| [59] |
李辉, 曲洋, 姚敏杰, 田文杰, 王小庆, 石犇, 曹丽娜, 岳凌帆, 曹凯琴. 赤泥自然成土过程及其微生物驱动机制. 应用生态学报, 2021, 32(4): 1452-1460. |
| [60] |
Drechsler C. Predaceous fungi. Biological Reviews, 1941, 16(4): 265-290. |
| [61] |
Zimmermann G. Review on safety of the entomopathogenic fungus Metarhizium anisopliae. Biocontrol Science and Technology, 2007, 17(9): 879-920. |
| [62] |
Horn M, Wagner M, Müller K D, Schmid E N, Fritsche T R, Schleifer K H, Michel R. Neochlamydia hartmannellae gen. nov., sp. nov. (Parachlamydiaceae), an endoparasite of the amoeba Hartmannella vermiformis The GenBank accession number for the sequence reported in this paper is AF177275. Microbiology, 2000, 146(5): 1231-1239. |
| [63] |
Young J M. The genus name Ensifer Casida 1982 takes priority over Sinorhizobium chen et al. 1988, and Sinorhizobium morelense Wang et al. 2002 is a later synonym of Ensifer adhaerens Casida 1982. Is the combination "Sinorhizobium adhaerens" (Casida 1982) Willems et al. 2003 legitimate? Request for an Opinion. International Journal of Systematic and Evolutionary Microbiology, 2003, 53(Pt 6): 2107-2110. |
| [64] |
Reinhold-Hurek B, Hurek T, Gillis M, Hoste B, Vancanneyt M, Kersters K, de Ley J. Azoarcus gen. nov., nitrogen-fixing proteobacteria associated with roots of kallar grass (Leptochloa fusca (L.) kunth), and description of two species, Azoarcus indigens sp. nov. and Azoarcus communis sp. nov. International Journal of Systematic Bacteriology, 1993, 43(3): 574-584. |
| [65] |
la Reau A J, Meier-Kolthoff J P, Suen G. Sequence-based analysis of the genus Ruminococcus resolves its phylogeny and reveals strong host association. Microbial Genomics, 2016, 2(12): e000099. |
| [66] |
张俊. 黄河三角洲自然保护区原生湿地生态系统演化规律研究. 山东林业科技, 2006, 36(3): 88-89. |
| [67] |
孙建平, 刘雅辉, 左永梅, 韩民利, 张虹伟, 吕晶晶. 盐地碱蓬根际土壤细菌群落结构及其功能. 中国生态农业学报: 中英文, 2020, 28(10): 1618-1629. |
| [68] |
Williams C H, Steinbergs A. Soil sulphur fractions as chemical indices of available sulphur in some Australian soils. Australian Journal of Agricultural Research, 1959, 10(3): 340. |
| [69] |
Gorlenko V M, Bryantseva I A, Kalashnikov A M, Gaisin V A, Sukhacheva M V, Gruzdev D S, Kuznetsov B B. Candidatus 'chloroploca asiatica' gen. nov., sp. nov., a new mesophilic filamentous anoxygenic phototrophic bacterium. Microbiology, 2014, 83(6): 838-848. |
| [70] |
Imhoff J F, TRuPER H G, Pfennig N. Rearrangement of the species and genera of the phototrophic "purple nonsulfur bacteria". International Journal of Systematic Bacteriology, 1984, 34(3): 340-343. |
| [71] |
Boldareva E N, Moskalenko A A, Makhneva Z K, Tourova T P, Kolganova T V, Gorlenko V M. Rubribacterium polymorphum gen. nov., sp. nov., a novel alkaliphilic nonsulfur purple bacterium from an Eastern Siberian soda lake. Microbiology, 2009, 78(6): 732-740. |
| [72] |
连宾. 碳酸盐岩风化成土过程中的微生物作用. 矿物岩石地球化学通报, 2010, 29(1): 52-56. |
| [73] |
Jiang M, Sheng Y Q, Liu Q Q, Wang W J, Liu X Z. Conversion mechanisms between organic sulfur and inorganic sulfur in surface sediments in coastal rivers. Science of the Total Environment, 2021, 752: 141829. |
| [74] |
Jarvis B D W, van Berkum P, Chen W X, Nour S M, Fernandez M P, Cleyet-Marel J C, Gillis M. Transfer of Rhizobium loti, Rhizobium huakuii, Rhizobium ciceri, Rhizobium mediterraneum, and Rhizobium tianshanense to Mesorhizobium gen. nov. International Journal of Systematic and Evolutionary Microbiology, 1997, 47(3): 895-898. |
| [75] |
Kabdullayeva T, Crosbie D B, Marín M. Mesorhizobium norvegicum sp. nov., a rhizobium isolated from a Lotus corniculatus root nodule in Norway. International Journal of Systematic and Evolutionary Microbiology, 2020, 70(1): 388-396. |
| [76] |
Takii S, Hanada S, Tamaki H, Ueno Y, Sekiguchi Y, Ibe A, Matsuura K. Dethiosulfatibacter aminovorans gen. nov., sp. nov., a novel thiosulfate-reducing bacterium isolated from coastal marine sediment via sulfate-reducing enrichment with Casamino acids. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(Pt 10): 2320-2326. |
| [77] |
Kuever J, Könneke M, Galushko A, Drzyzga O. Reclassification of Desulfobacterium phenolicum as Desulfobacula phenolica comb. nov. and description of strain SaxT as Desulfotignum balticum gen. nov., sp. nov. International Journal of Systematic and Evolutionary Microbiology, 2001, 51(Pt 1): 171-177. |
| [78] |
Kompantseva E I, Kublanov I V, Perevalova A A, Chernyh N A, Toshchakov S V, Litti Y V, Antipov A N, Bonch-Osmolovskaya E A, Miroshnichenko M L. Calorithrix insularis gen. nov., sp. nov., a novel representative of the Phylum Calditrichaeota. International Journal of Systematic and Evolutionary Microbiology, 2017, 67(5): 1486-1490. |
| [79] |
Sorokin D Y, Tourova T P, Galinski E A, Muyzer G, Kuenen J G. Thiohalorhabdus denitrificans gen. nov., sp. nov., an extremely halophilic, sulfur-oxidizing, deep-lineage gammaproteobacterium from hypersaline habitats. International Journal of Systematic and Evolutionary Microbiology, 2008, 58(12): 2890-2897. |
| [80] |
Hong H, Kim S J, Min U G, Lee Y J, Kim S G, Roh S W, Kim J G, Na J G, Rhee S K. Anaerosolibacter carboniphilus gen. nov., sp. nov., a strictly anaerobic iron-reducing bacterium isolated from coal-contaminated soil. International Journal of Systematic and Evolutionary Microbiology, 2015, 65(Pt_5): 1480-1485. |
| [81] |
Slobodkina G B, Reysenbach A L, Panteleeva A N, Kostrikina N A, Wagner I D, Bonch-Osmolovskaya E A, Slobodkin A I. Deferrisoma camini gen. nov., sp. nov., a moderately thermophilic, dissimilatory iron(Ⅲ)-reducing bacterium from a deep-sea hydrothermal vent that forms a distinct phylogenetic branch in the Deltaproteobacteria. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(Pt_10): 2463-2468. |
 2022, Vol. 42
2022, Vol. 42