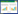文章信息
- 朱怡, 吴永波, 安玉亭
- ZHU Yi, WU Yongbo, AN Yuting
- 基于高通量测序的禁牧对土壤微生物群落结构的影响
- Effects of grazing prohibition on soil microbial community structure based on high-throughput sequencing
- 生态学报. 2022, 42(17): 7137-7146
- Acta Ecologica Sinica. 2022, 42(17): 7137-7146
- http://dx.doi.org/10.5846/stxb202104261095
-
文章历史
- 收稿日期: 2021-04-26
- 修订日期: 2022-05-02
2. 江苏省南方现代林业协同创新中心, 南京 210037;
3. 盐城市麋鹿研究所, 盐城 224136
2. Collaborative Innovation Center of Sustainable Forestry in Southern China of Jiangsu Province, Nanjing 210037, China;
3. Yancheng Milu Institute, Yancheng 224136, China
麋鹿(Elaphurus davidianus)是一种原产于我国的大型食草动物, 为国家一级保护动物[1]。江苏大丰麋鹿国家级自然保护区(以下称保护区)的麋鹿数量从1986年的39头发展到2020年的5681头, 每头麋鹿占有的生境面积远少于保护区制定的管理方案。麋鹿种群数量的急剧增加, 导致食物匮乏, 保护区内环境压力上升, 圈养区地表向裸地演变, 扩大野放规模成为保护区的解决方式。动物各项活动均依托于环境, 进而对环境产生一定影响[2]。土壤微生物是土壤生态系统的重要组成部分, 直接参与土壤中的物质分解和养分循环过程。Chen等[3]认为, 土壤微生物多样性与植物生长情况有关。高通量测序具有效率高, 结果精确和成本相对低廉的优点, 逐步成为微生物群落研究的主流技术。目前高通量测序已被广泛运用于退耕还林[4]、作物定植年限与种植方式[5—6]以及土地管理方式[7—8]等方面对土壤微生物群落的影响。多年放牧影响了土壤微生物多样性[9], 围栏封育是恢复放牧地土壤环境的措施之一。Fenetahun等[10]在博拉纳地区研究围封对草地环境的影响中发现, 短期围封就可实现草地承载力的大幅提升。张胜男等[11]分析了短期封育和自由放牧对科尔沁沙地榆树疏林土壤微生物群落结构的影响, 发现封育后土壤细菌群落转变为快速生长型细菌为主导。斯贵才等[12]在围栏封育对受损草地的修复研究中证明, 围栏封育有助于提高土壤水溶性有机碳水平, 利于土壤微生物生长, 以及土壤生态功能的恢复。
多年的麋鹿干扰为保护区环境带来压力, 土壤出现退化趋势[13], 保护区需制定相应的恢复方案以修复麋鹿生境。本文以江苏大丰麋鹿国家级自然保护区第一核心区内禁牧点和补饲点土壤为研究对象, 利用高通量测序, 结合土壤理化性质分析禁牧点和补饲点土壤细菌和真菌群落结构差异及其环境影响因子, 探究禁牧对土壤微生物群落结构的影响, 研究可为保护区制定麋鹿生境恢复方案提供参考。
1 研究区概况江苏大丰麋鹿国家级自然保护区地处北纬32°59′—33°03′, 东经120°47′—120°53′, 总面积为2677hm2, 分为核心区、缓冲区和实验区。核心区下包括第一核心区(479 hm2)、第二核心区(578 hm2)和第三核心区(1620 hm2)三部分。区内整体地势平坦, 平均海拔为2m[14], 土质为粉砂质。属于海洋和季风气候的过渡型气候, 夏季高温多雨, 冬季低温少雨, 夏秋季多台风, 为全年的降水高峰期。常年无霜期299d, 日照2325.4h, 平均气温13.7—14.5℃, 年平均降水量980-1100mm[15]。
2 研究方法 2.1 样品采集采样于2020年10月在保护区第一核心区内进行。第一核心区为圈养区, 部分区域地表已演变为裸地。利用蹄印密度和粪便遗留量划分麋鹿干扰强度[16], 蹄印密度和粪便遗留量越大, 代表麋鹿干扰强度越大。选取6个面积约为2m2的禁牧点(均位于第一核心区西南角)和距离禁牧点最近的1个全年使用的补饲点, 采集0-20cm土壤, 并记录每个采样点坐标及其优势植物种类(表 1)。选取的补饲点地表裸露, 无植被覆盖, 每日进行两次人工补饲;6个禁牧点均建于2016年, 外围以不锈钢围网呈三角形围绕, 建成前一直有麋鹿活动。在每个禁牧点内划定1个1 m×1 m的样方, 按五点取样法采集土壤并均匀混合, 标记为(Inactive area, IA);在补饲点划定6个1 m×1 m的样方(每个样方间隔10 m), 按五点取样法采集土壤并均匀混合, 标记为(Active area, AA), 共取得样品12份。将每份样品等分为2袋, 一袋带回实验室风干, 用于土壤理化性质测定;一袋保存于-80℃超低温冰箱, 用于土壤微生物群落高通量测序。
| 采样区 Sampling area |
样品 Sample |
纬度 Latitude |
经度 Longitude |
粪便遗留量 Fecal residue/ (颗/m2) |
蹄印密度 Hoofprint density/ (个/m2) |
优势植物 Dominant vegetation |
| IA | IA1 | 32°58′51.56″N | 120°48′37.43″E | 0 | 0 | 狼尾草(Pennisetum alopecuroides)、乌桕(Sapium sebiferum)、一年蓬(Erigeron annuus)、加拿大一枝黄花(Solidago canadensis) |
| IA2 | 32°58′54.64″N | 120°48′35.06″E | 0 | 0 | 乌桕、狼尾草 | |
| IA3 | 32°58′54.42″N | 120°48′32.62″E | 0 | 0 | 乌桕、狼尾草 | |
| IA4 | 32°58′54.89″N | 120°48′23.34″E | 0 | 0 | 乌桕、狼尾草、加拿大一枝黄花、一年蓬 | |
| IA5 | 32°58′54.05″N | 120°48′17.54″E | 0 | 0 | 乌桕、狼尾草、白茅(Imperata cylindrica) | |
| IA6 | 32°58′54.58″N | 120°48′20.31″E | 0 | 0 | 乌桕、狼尾草 | |
| AA | AA1 | 32°58′47.79″N | 120°48′38.28″E | 108 | 26 | — |
| AA2 | 32°58′47.95″N | 120°48′36.25″E | 76 | 35 | ||
| AA3 | 32°58′47.73″N | 120°48′34.49″E | 89 | 31 | ||
| AA4 | 32°58′47.94″N | 120°48′33.52″E | 125 | 59 | ||
| AA5 | 32°58′48.10″N | 120°48′31.29″E | 96 | 40 | ||
| AA6 | 32°58′47.98″N | 120°48′30.39″E | 86 | 46 | ||
| IA: 禁牧点Inactive area;AA:补饲点Active area | ||||||
采用FastDNA® SPIN Kit for Soil试剂盒(FastDNA® SPIN Kit for Soil, Norcross, MP, USA)抽提样品总脱氧核糖核酸(DNA), DNA浓度和纯度检测利用NanoDrop2000进行, 提取的DNA质量利用1%琼脂糖凝胶电泳检测;细菌和真菌的PCR引物分别为:338F (5′-ACTCCTACGGGAGGCAGCAG-3′)、806R (5′-GGACTA CHVGGGTWTCTAA T-3′)[17]和ITS1F(5′-CTTGGTCATTTAGAGGAAGTAA-3′)、ITS2R(5′-GCTGCGTTCT TCATCGATGC-3′)[18]。细菌的聚合酶链式反应(PCR)扩增体系为20 μL: 5×FastPfu Buffer 4 μL; 2.5 mM dNTPs 2 μL; 正向引物(5μM) 0.8 μL; 反向引物(5μM) 0.8 μL; Fast Pfu聚合酶0.4 μL; BSA 0.2 μL; Template DNA 10 ng;补ddH2O至20 μL。真菌的PCR扩增体系为20 μL: 10× Buffer 2 μL; 2.5 mM dNTPs 2 μL; 正向引物(5 μM) 0.8 μL; 反向引物(5 μM) 0.8 μL; rTaq聚合酶0.2 μL; BSA 0.2 μL; Template DNA 10 ng;补ddH2O至20 μL。扩增程序如下:95℃预变性3min, 27个(细菌)/35个(真菌)循环(95℃变性30s, 55℃退火30s, 72℃延伸45s), 最后72℃延伸10min, 10℃终止反应, 使用的PCR仪为ABI GeneAmp® 9700型。PCR产物用2%的琼脂糖凝胶电泳检测, 选择主带为500—750bp之间的条带, 使用AxyPrepDNA凝胶回收试剂盒(AXYGEN公司)回收PCR产物。回收后的产物交由上海美吉生物医药科技有限公司进行文库构建, 构建好的文库经检验合格后进行高通量测序。
2.3 土壤理化性质测定土壤理化性质测定方法参考《土壤农化分析》(第三版)[19]。土壤样品风干后研磨过筛, 土壤容重采用环刀法;土壤pH采用电极法(水土比为5∶1);土壤含水率采用烘干法;土壤有机碳采用重铬酸钾容量法-外加热法;土壤水解性氮采用碱解扩散法;土壤有效磷采用NaHCO3浸提-钼锑抗比色法;土壤速效钾采用NH4OAc浸提-火焰光度法。
2.4 数据处理土壤理化性质数据在Excel2016中汇总, SPSS 22.0进行显著性分析。高通量测序数据分析均在上海美吉生物医药科技有限公司提供的在线分析平台上完成。
3 结果分析 3.1 禁牧对土壤理化性质的影响土壤理化性质测定结果如表 2。禁牧点土壤容重低于补饲点, 土壤含水率为30.13%, 补饲点土壤含水率为19.06%。禁牧点土壤有机碳显著高于补饲点(P < 0.05), 补饲点土壤水解性氮、土壤有效磷和土壤速效钾含量均显著高于禁牧点(P < 0.05)。土壤pH在禁牧点和补饲点内差异不显著。
| 样品Sample | 容重 Bulk density BD/(g/cm3) |
pH | 含水率 Soil moisture content, SMC/% |
有机碳 Soil organic carbon, SOC/(g/kg) |
水解性氮 Available nitrogen, AN/(mg/kg) |
有效磷 Available phosphorus, AP/(mg/kg) |
速效钾 Available potassium, AK/(mg/kg) |
| 禁牧点 Inactive area, IA |
1.08±0.40b | 9.03±0.06a | 30.13±3.60a | 4.72±1.62a | 108.82±6.92b | 7.10±3.26b | 136.48±11.34b |
| 补饲点 Active area, AA |
1.51±0.03a | 9.06±0.02a | 19.06±2.05b | 3.27±0.29b | 318.58±15.61a | 7.67±2.82a | 220.58±20.00a |
| 表中数据为平均值±标准差, 不同小写字母代表差异显著(P<0.05) | |||||||
以97%序列相似度水平为条件, 对序列进行质控和过滤, 得到细菌和真菌的可操作分类单元(OUT)数分别为5792、955, 细菌多于真菌。如图 1所示, 细菌和真菌的稀释曲线随着取样数的增加已趋于平缓, 表明加大取样数出现新的类群可能性小, 测序结果可反映禁牧点和补饲点土壤微生物群落结构的实际情况。此外, 禁牧点中检出的细菌和真菌OTU数高于补饲点。
|
| 图 1 禁牧点与补饲点土壤细菌和真菌稀释曲线 Fig. 1 Dilution curves of soil bacteria and fungi for inactive areas and active areas IA1为1号禁牧点样品; AA1为1号补饲点样品, 以此类推; OTU: 可操作分类单元Operational taxonomic units |
表 3为禁牧点和补饲点土壤细菌和真菌群落的多样性指数。由表可知, 细菌和真菌文库的覆盖率均大于98%, 覆盖率较高。禁牧点Shannon指数、Ace指数和Chao指数均高于补饲点, Simpson指数低于补饲点, 但无显著差异。
| 样品 Sample |
Shannon指数 | Simpson指数 | Ace指数 | Chao指数 | 覆盖度Coverage/% | |||||||||
| 细菌 Bacteria |
真菌 Fungi |
细菌 Bacteria |
真菌 Fungi |
细菌 Bacteria |
真菌 Fungi |
细菌 Bacteria |
真菌 Fungi |
细菌 Bacteria |
真菌 Fungi |
|||||
| IA | 6.19a | 2.51a | 0.01a | 0.24a | 2672.66a | 232.18a | 2640.03a | 232.52a | 98.15a | 99.98a | ||||
| AA | 5.85a | 2.22a | 0.01a | 0.26a | 2475.56a | 112.98a | 2456.83a | 113.84a | 98.10a | 99.99a | ||||
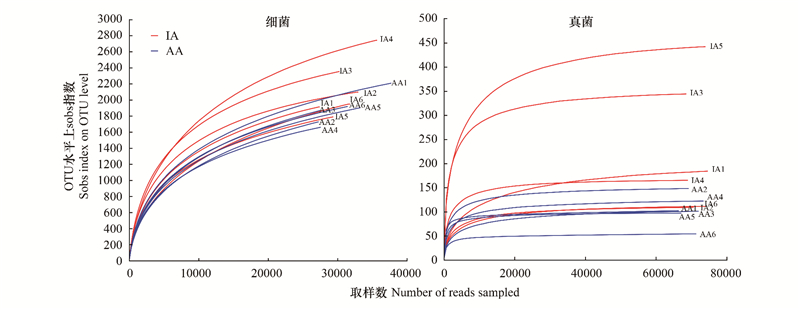
图 2为门水平土壤细菌群落组成, 除相对丰度小于1%的类群外, 共得到15个类群。其中相对丰度前5位的细菌门为变形菌门(Proteobacteria)、绿弯菌门(Chloroflexi)、酸杆菌门(Acidobacteriota)、放线菌门(Actinobacteriota)和厚壁菌门(Firmicutes)。变形菌门的相对丰度在禁牧点和补饲点内均大于25%, 为优势细菌门。禁牧点变形菌门的相对丰度为32%, 高于补饲点4.49%。禁牧点绿弯菌门的相对丰度比补饲点低4.81%, 酸杆菌门和放线菌门相对丰度高于补饲点, 差值为0.63%和6.67%。厚壁菌门的相对丰度在禁牧点内为6.12%, 低于补饲点4.48%。
|
| 图 2 禁牧点与补饲点土壤细菌门水平群落组成 Fig. 2 Composition of soil bacterial phylum level communities at inactive areas and active areas |
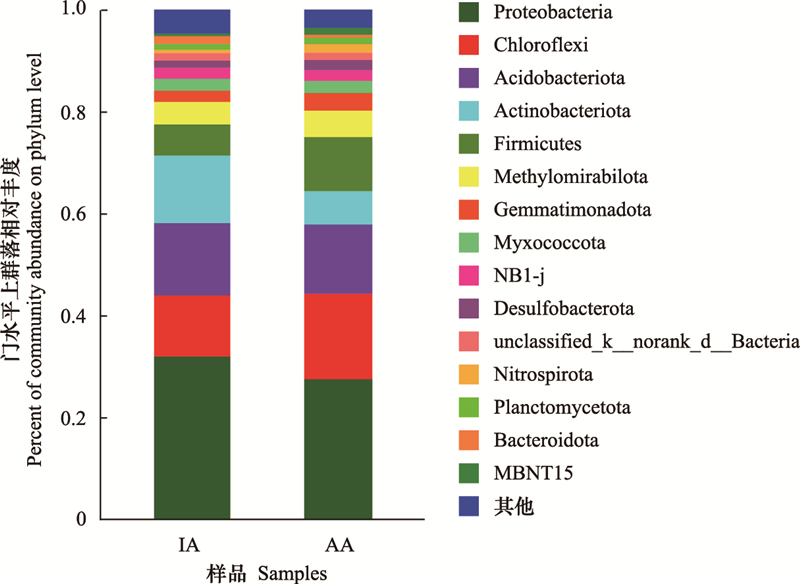
图 3为纲水平土壤细菌群落组成, 除相对丰度小于1%的类群外, 共得到23个类群。相对丰度前5位的细菌纲分别属于变形菌门、绿弯菌门、酸杆菌门和厚壁菌门, 以上类群在禁牧点和补饲点内的相对丰度之和均大于50%。α-变形菌纲(Alphaproteobacteria)在禁牧点和补饲点内的相对丰度均大于15%, 为优势细菌纲;禁牧点内γ-变形菌纲(Gammaproteobacteria)的相对丰度为14.88%, 高于补饲点7.91%;厌氧绳菌纲(Anaerolineae)、Vicinamibacteria纲和芽孢杆菌纲(Bacilli)的相对丰度在禁牧点内均低于补饲点。
|
| 图 3 禁牧点与补饲点土壤细菌纲水平群落组成 Fig. 3 Composition of soil bacterial class level communities at inactive areas and active areas |
图 4为门水平土壤真菌群落组成, 结果中检出未分类真菌, 禁牧点中未分类真菌检出比例高于补饲点。除未分类真菌外, 以子囊菌门(Ascomycota)的相对丰度最高, 且大于50%, 为真菌优势门。禁牧点内子囊菌门相对丰度为57.93%, 低于补饲点15%;禁牧点内担子菌门(Basidiomycota)相对丰度高于补饲点, 差值为7.25%;禁牧点和补饲点中被孢霉门(Mortierellomycota)相对丰度差值小于1%。
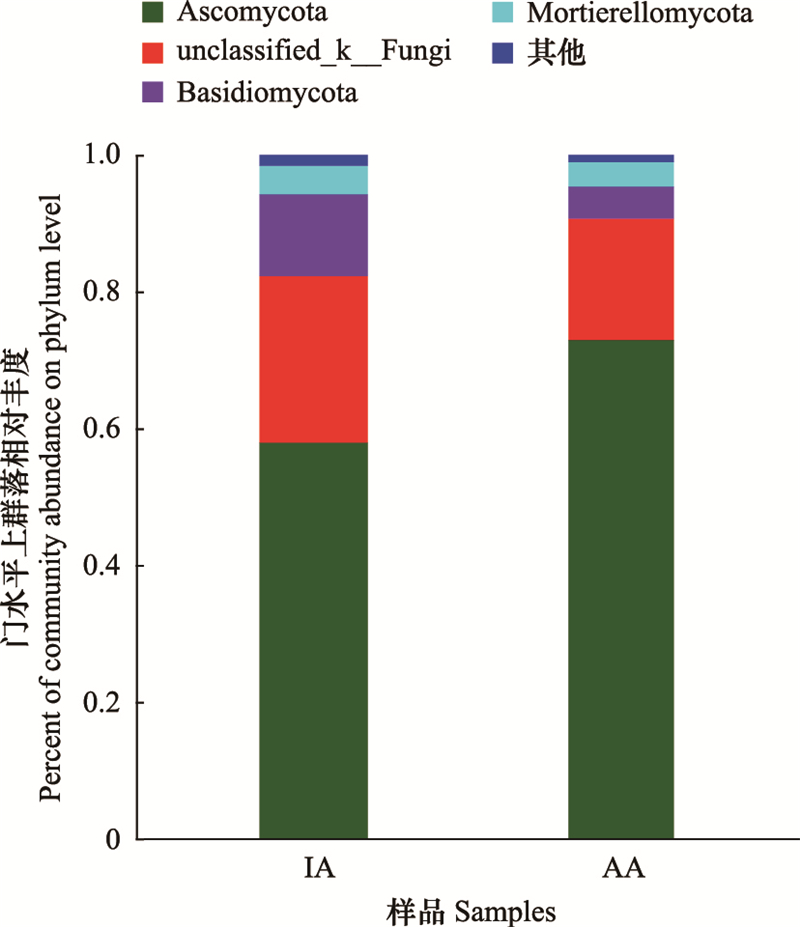
|
| 图 4 禁牧点与补饲点土壤真菌门水平群落组成 Fig. 4 Composition of soil fungal phylum level communities at inactive areas and active areas |
图 5为纲水平土壤真菌群落组成。除某未分类真菌外, 相对丰度前5位的真菌纲分别属于子囊菌门、担子菌门和被孢霉门。对比补饲点, 禁牧点粪壳菌纲(Sordariomycetes)相对丰度下降, 下降幅度为19.08%。伞菌纲(Agaricomycetes)在禁牧点内的相对丰度高于补饲点7.31%。被孢霉纲(Mortierellomycetes)相对丰度在禁牧点和补饲点内的差值小于1%。禁牧点中散囊菌纲(Eurotiomycetes)的相对丰度为6.77%, 高于补饲点6.60%。
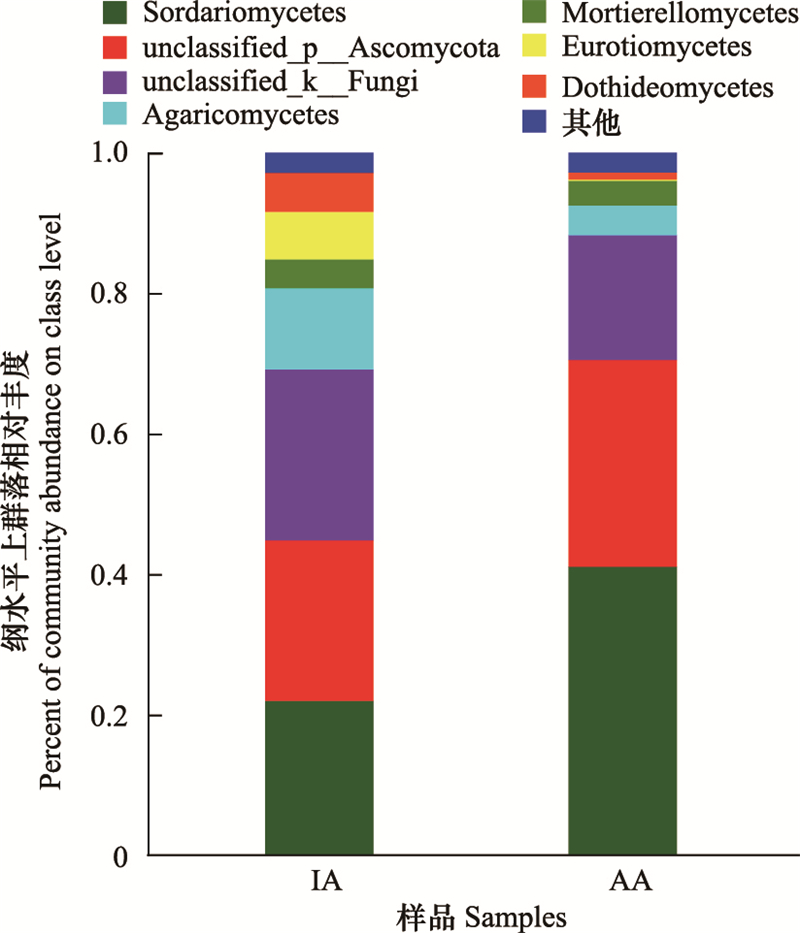
|
| 图 5 禁牧点与补饲点土壤真菌纲水平群落组成 Fig. 5 Composition of soil fungal class level communities at inactive areas and active areas |
采用Bray-curtis距离对禁牧点和补饲点土壤微生物群落多样性差异进行分析, 结果如图 6所示。细菌群落中, 各补饲点样品距离均小于0.50, 表明补饲点内部土壤细菌群落多样性差异较小;各禁牧点样品距离均大于0.50, 表明不同禁牧点土壤细菌群落多样性差异较大。对比禁牧点和补饲点, 除IA1外, 各样品距离均大于0.50, 表明禁牧点和补饲点土壤细菌群落多样性差异较大。真菌群落中, 任意两个样品的距离均大于0.60, 表明禁牧点和补饲点土壤真菌群落多样性相似性较小。
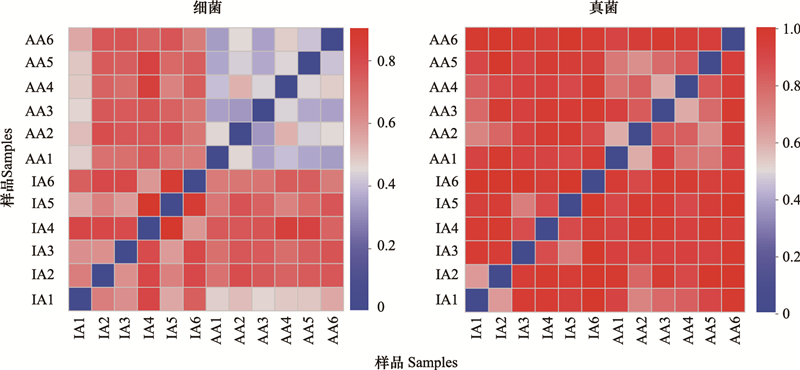
|
| 图 6 禁牧点与补饲点土壤细菌和真菌群落多样性差异分析热图 Fig. 6 Heat map of the difference in diversity of soil bacterial and fungal communities between inactive areas and active areas 图中不同颜色的色块表示样品距离, 蓝色越深表示样品距离越小, 相似性越大;红色越深表示样品距离越大, 相似性越小 |
图 7为细菌和真菌群落与土壤理化性质的关系。冗余分析(RDA)分别解释了细菌和真菌群落45.26%和52.90%的总方差。在测定的土壤理化性质中, 土壤pH(P=0.14)和水解性氮(P=0.09)对细菌群落影响较大, pH(P=0.10)和有效磷(P=0. 30)是影响真菌群落最主要的土壤环境因子。总体来说, 细菌受土壤环境因子的影响大于真菌, 土壤pH是影响细菌和真菌群落最主要的土壤环境因子。
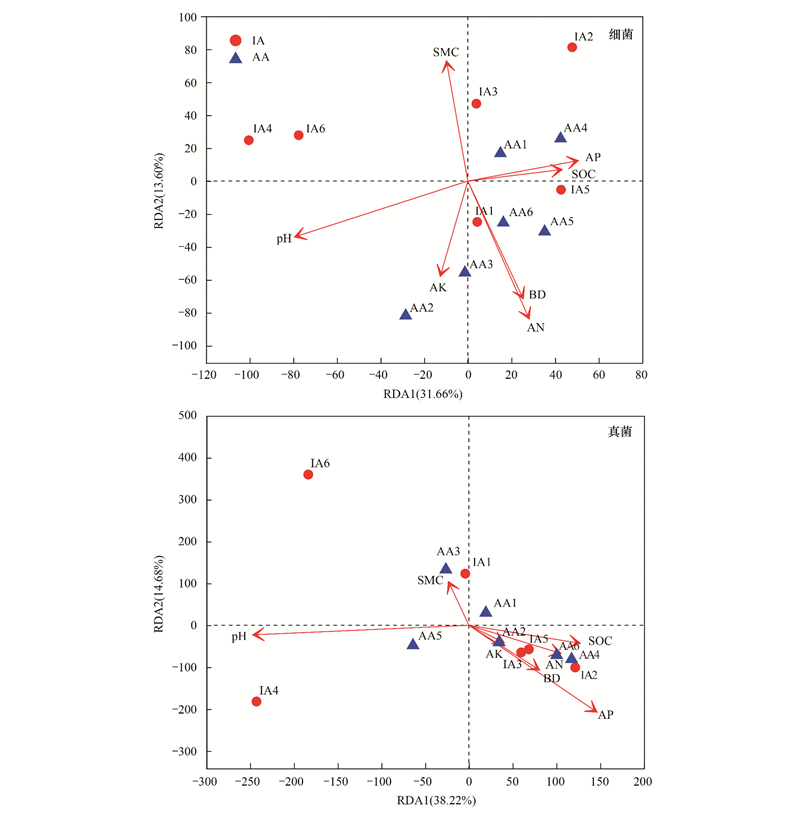
|
| 图 7 土壤理化性质与细菌和真菌群落的冗余分析 Fig. 7 Redundancy analysis of soil physicochemical properties with bacterial and fungal communities BD: 容重Bulk density;SMC:含水率Soil moisture content;pH:氢离子浓度指数Hydrogen ion concentration;SOC:有机碳Soil organic carbon;AN:水解性氮Available nitrogen;AP:有效磷Available phosphorus;AK:速效钾Available potassium |
本文研究发现, 禁牧点内细菌和真菌群落多样性和丰度均优于补饲点, 但无显著差异, 与郑佳华等[20]的研究结果一致。而Zhou等[21]发现, 未放牧地土壤细菌多样性显著低于中度放牧地和轻度放牧地, 这意味着禁牧可能并不是导致土壤微生物多样性变化的主要因素。
禁牧改变了土壤微生物群落结构, 与韦应莉等[22]的研究结果一致。禁牧点和补饲点中检出的细菌OTU数多于真菌, 细菌数量占优势, 这与黄明勇等[23]在天津滨海盐碱土地区的研究结果一致。细菌群落中相对丰度较高的门为变形菌门、绿弯菌门、酸杆菌门、放线菌门和厚壁菌门, 类似于已有土壤微生物群落组成的研究结果[24—26]。围网围封阻挡麋鹿进入禁牧点, 禁牧点内植被凋落物堆积, 植被覆盖率高于80%, 地表光照强度降低。绿弯菌门包含可利用光照的菌种[27], 光照强度的下降, 一定程度上使绿弯菌门相对丰度下降。本文研究结果中禁牧造成酸杆菌门和放线菌门相对丰度有不同程度的上升, 放线菌门相对丰度的上升幅度高于酸杆菌门。酸杆菌门为贫营养型细菌[28], 可适应较恶劣的土壤环境, 其中部分菌种和放线菌门的部分菌种可利用纤维素[29—30]。禁牧点内植物残体较多, 大量纤维素待分解, 为可利用纤维素的酸杆菌门和放线菌门细菌创造有利环境。
本文研究结果中禁牧点和补饲点子囊菌门的相对丰度均大于50%, 为优势真菌门。目前已知98%的陆生真菌属子囊菌门和担子菌门, 且子囊菌门下真菌种数多于担子菌门[31], 表明在一个样本中检出的真菌属子囊菌门的概率高于担子菌门。禁牧造成粪壳菌纲相对丰度下降, 散囊菌纲的相对丰度上升。散囊菌纲和粪壳菌纲同属子囊菌门, 包含多种腐生型真菌[32], 两者可能会存在竞争关系, 这有待进一步探讨。禁牧点真菌群落多样性的相似性低于补饲点, 考虑到不同禁牧点优势植物种类并不完全一致, 表明真菌受植被种类的影响较大, 与已有研究结果类似[33]。
4.2 土壤微生物群落与土壤理化性质的关系RDA分析中, 土壤pH对细菌和真菌群落的影响较大, 但未达到显著, 可能是因为禁牧点和补饲点土壤pH差异较小。土壤pH与多数禁牧点和补饲点的细菌和真菌群落呈负相关, 保护区土壤pH超出多数细菌和真菌生长的适宜pH范围, 不利于多数细菌和真菌的生长。水解性氮对土壤细菌群落的影响仅次于土壤pH, 且与多数补饲点土壤细菌群落呈正相关。补饲点内高强度麋鹿活动带来大量麋鹿粪便, 粪便中的氮素可为土壤细菌生长提供氮源[34], 从而促进细菌生长。与土壤pH相反, 土壤有效磷与多数样点的真菌群落呈正相关, 可能与真菌参与土壤磷转化, 增加土壤磷的有效性有关[35]。
有研究表明, 土壤微生物群落具有季节差异[36], 后续可以考虑分析土壤微生物群落结构的季节变化状况, 以全面了解禁牧对土壤微生物群落结构的影响。
5 结论研究表明, 禁牧显著影响土壤理化性质, 改变了土壤微生物群落多样性。土壤微生物群落结构中, 细菌的优势菌门为变形菌门, 真菌的优势菌门为子囊菌门。禁牧在门水平上提高了变形菌门、放线菌门和担子菌门的相对丰度, 降低了绿弯菌门、厚壁菌门和子囊菌门的相对丰度, 禁牧点与补饲点土壤微生物群落多样性的相似性较低。RDA分析中, 细菌受土壤环境因子的影响大于真菌, 其中土壤pH是影响细菌和真菌群落最大的环境因子。本文揭示了禁牧对土壤微生物群落结构的影响, 可为保护区制定麋鹿生境恢复方案提供参考。
| [1] |
白加德, 张渊媛, 钟震宇, 程志斌, 曹明, 孟玉萍. 中国麋鹿种群重建35年: 历程、成就与挑战. 生物多样性, 2021, 29(2): 160-166. |
| [2] |
Yang F, Niu K C, Collins C G, Yan X B, Ji Y G, Ling N, Zhou X H, Du G Z, Guo H, Hu S J. Grazing practices affect the soil microbial community composition in a Tibetan alpine meadow. Land Degradation & Development, 2019, 30(1): 49-59. |
| [3] |
Chen Q L, Cui H L, Su J Q, Penuelas J, Zhu Y G. Antibiotic resistomes in plant microbiomes. Trends in Plant Science, 2019, 24(6): 530-541. DOI:10.1016/j.tplants.2019.02.010 |
| [4] |
刘晓华, 魏天兴. 高通量测序分析黄土高原退耕还林区土壤细菌群落特征. 环境科学, 2021, 42(9): 4489-4499. |
| [5] |
顾美英, 徐万里, 马凯, 冯雷, 张志东, 欧提库尔·玛合木提, 朱静, 唐光木, 唐琦勇, 楚敏. 不同定植年限核桃园土壤细菌群落多样性及碳代谢功能特征. 生态学杂志, 2021, 40(7): 2045-2056. |
| [6] |
张杰雪, 王占青, 全小龙, 梁军, 史惠兰, 陈梦词, 乔有明. 高寒地区人工草地土壤微生物群落对不同种植方式和年限的响应. 草地学报, 2021, 29(2): 270-280. |
| [7] |
Chen L L, Saixi Y L, Yi R, Baoyin T. Characterization of soil microbes associated with a grazing-tolerant grass species, Stipa breviflora, in the Inner Mongolian desert steppe. Ecology and Evolution, 2020, 10(19): 10607-10618. DOI:10.1002/ece3.6715 |
| [8] |
Lienhard P, Terrat S, Prévost-Bouré N C, Nowak V, Régnier T, Sayphoummie S, Panyasiri K, Tivet F, Mathieu O, Levêque J, Maron P A, Ranjard L. Pyrosequencing evidences the impact of cropping on soil bacterial and fungal diversity in Laos tropical grassland. Agronomy for Sustainable Development, 2014, 34(2): 525-533. DOI:10.1007/s13593-013-0162-9 |
| [9] |
Yang Y F, Wu L W, Lin Q Y, Yuan M T, Xu D P, Yu H, Hu Y G, Duan J C, Li X Z, He Z L, Xue K, van Nostrand J, Wang S P, Zhou J Z. Responses of the functional structure of soil microbial community to livestock grazing in the Tibetan alpine grassland. Global Change Biology, 2013, 19(2): 637-648. DOI:10.1111/gcb.12065 |
| [10] |
Fenetahun Y, You Y, Xu X W, Wang Y D. Effects of grazing enclosures on species diversity, phenology, biomass, and carrying capacity in borana rangeland, southern Ethiopia. Frontiers in Ecology and Evolution, 2021, 8: 623627. DOI:10.3389/fevo.2020.623627 |
| [11] |
张胜男, 闫德仁, 黄海广, 胡小龙. 短期封育对科尔沁沙地榆树疏林土壤微生物群落结构的影响. 生态学杂志, 2020, 39(9): 2860-2867. |
| [12] |
斯贵才, 袁艳丽, 王建, 王光鹏, 雷天柱, 张更新. 围封对当雄县高寒草原土壤微生物和酶活性的影响. 草业科学, 2015, 32(1): 1-10. |
| [13] |
安玉亭, 刘彬, 王立波, 解生彬, 薛丹丹, 吴永波. 不同麋鹿干扰强度对栖息地土壤理化特性的影响. 生态学报, 2020, 40(11): 3571-3578. |
| [14] |
贾媛媛. 大丰麋鹿种群适合度及其生境现状调查分析[D]. 南京: 南京农业大学, 2017.
|
| [15] |
卢霞, 赵倩, 林雅丽, 吴亚楠, 顾杨. 大丰麋鹿自然保护区土地利用/覆盖变化监测研究. 淮海工学院学报(自然科学版), 2018, 27(3): 74-81. DOI:10.3969/j.issn.1672-6685.2018.03.016 |
| [16] |
黄旭, 张健, 杨万勤, 刘洋, 闫帮国, 胡方洁, 苏江峰. 川西亚高山林牧交错区土壤动物多样性. 生态学报, 2010, 30(19): 5161-5173. |
| [17] |
Xu N, Tan G C, Wang H Y, Gai X P. Effect of biochar additions to soil on nitrogen leaching, microbial biomass and bacterial community structure. European Journal of Soil Biology, 2016, 74: 1-8. DOI:10.1016/j.ejsobi.2016.02.004 |
| [18] |
Chen B S, Du K Q, Sun C, Vimalanathan A, Liang X L, Li Y, Wang B H, Lu X M, Li L J, Shao Y Q. Gut bacterial and fungal communities of the domesticated silkworm (Bombyx mori) and wild mulberry-feeding relatives. The ISME Journal, 2018, 12(9): 2252-2262. DOI:10.1038/s41396-018-0174-1 |
| [19] |
鲍士旦. 土壤农化分析(第三版). 北京: 中国农业出版社, 2000.
|
| [20] |
郑佳华, 赵萌莉, 王琪, 张峰, 张彬, 张军. 利用方式对大针茅草原土壤微生物群落结构及多样性的影响. 生态学报, 2022, 42(12). DOI:10.5846/stxb202103290818 |
| [21] |
Zhou X Q, Wang J Z, Hao Y B, Wang Y F. Intermediate grazing intensities by sheep increase soil bacterial diversities in an Inner Mongolian steppe. Biology and Fertility of Soils, 2010, 46(8): 817-824. DOI:10.1007/s00374-010-0487-3 |
| [22] |
韦应莉, 曹文侠, 李建宏, 张爱梅, 李小龙. 不同放牧与围封高寒灌丛草地土壤微生物群落结构PLFA分析. 生态学报, 2018, 38(13): 4897-4908. |
| [23] |
黄明勇, 杨剑芳, 王怀锋, 张小平. 天津滨海盐碱土地区城市绿地土壤微生物特性研究. 土壤通报, 2007, 38(6): 1131-1135. DOI:10.3321/j.issn:0564-3945.2007.06.020 |
| [24] |
于少鹏, 史传奇, 胡宝忠, 丁俊男, 孟博, 杨春雪. 古大湖湿地盐碱土壤微生物群落结构及多样性分析. 生态学报, 2020, 40(11): 3764-3775. |
| [25] |
邵颖, 曹四平, 刘长海, 罗梦娇. 基于高通量测序的南泥湾湿地土壤细菌多样性分析. 干旱区资源与环境, 2019, 33(2): 158-163. |
| [26] |
牛世全, 龙洋, 李海云, 达文燕, 胡山, 李渭娟, 朱学泰, 孔维宝. 应用Illumina MiSeq高通量测序技术分析河西走廊地区盐碱土壤微生物多样性. 微生物学通报, 2017, 44(9): 2067-2078. |
| [27] |
高志伟, 刘凡惠, 贾美清, 张国刚, 黄静, 吴光红, 杨英花. 基于Illumina高通量测序的天津北大港湿地沉积物细菌群落特征和多样性分析. 天津师范大学学报(自然科学版), 2021, 41(4): 45-52. |
| [28] |
耿德洲, 黄菁华, 霍娜, 王楠, 杨盼盼, 赵世伟. 黄土高原半干旱区不同种植年限紫花苜蓿人工草地土壤微生物和线虫群落特征. 应用生态学报, 2020, 31(04): 1365-1377. |
| [29] |
刘彩霞, 董玉红, 焦如珍. 森林土壤中酸杆菌门多样性研究进展. 世界林业研究, 2016, 29(06): 17-22. |
| [30] |
靳新影, 张肖冲, 金多, 陈韵, 李靖宇. 腾格里沙漠东南缘不同生物土壤结皮细菌多样性及其季节动态特征. 生物多样性, 2020, 28(6): 718-726. |
| [31] |
Wang H Y, Guo S Y, Huang M R, Thorsten L H, Wei J C. Ascomycota has a faster evolutionary rate and higher species diversity than Basidiomycota. Science China-Life sciences, 2010, 53(10): 1163-1169. DOI:10.1007/s11427-010-4063-8 |
| [32] |
闫茹, 冯薇, 王玺婧. 呼伦贝尔沙地不同固沙植物土壤真菌群落组成及多样性. 中国水土保持科学, 2021, 19(01): 60-68. |
| [33] |
纪垚, 王继华, 苑泽宁, 关健飞, 刘晨宇, 祝亚楠. 不同恢复方式对七星河湿地真菌群落多样性影响. 黑龙江大学工程学报, 2017, 8(1): 60-66. |
| [34] |
朱佳伟, 李俊芳, 陈颀, 杨峥, 刘艳菊. 半散放麋鹿的粪便成分分析. 黑龙江畜牧兽医, 2017(22): 213-214. |
| [35] |
吴金凤, 刘鞠善, 李梓萌, 王德利. 草地土壤磷循环及其对全球变化的响应. 中国草地学报, 2021, 43(06): 102-111. |
| [36] |
Lipson D A, Schadt C W, Schmidt S K. Changes in soil microbial community structure and function in an alpine dry meadow following spring snow melt. Microbial Ecology, 2002, 43(3): 307-314. DOI:10.1007/s00248-001-1057-x |
 2022, Vol. 42
2022, Vol. 42