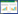文章信息
- 罗芳丽, 王沫竹, 董必成, 陈禹含, 于飞海
- LUO Fangli, WANG Mozhu, DONG Bicheng, CHEN Yuhan, YU Feihai
- 克隆和有性亲体效应及其调控机制
- Clonal and sexual parental effects and their mechanisms
- 生态学报. 2022, 42(16): 6474-6486
- Acta Ecologica Sinica. 2022, 42(16): 6474-6486
- http://dx.doi.org/10.5846/stxb202107211968
-
文章历史
- 收稿日期: 2021-07-21
- 网络出版日期: 2022-04-20
2. 中国科学院植物研究所系统与进化植物学国家重点实验室, 北京 100093;
3. 台州学院湿地生态学与克隆生态学研究所/植物进化生态学与保护浙江省重点实验室, 台州 318000
2. State Key Laboratory of Systematic and Evolutionary Botany, Institute of Botany, Chinese Academy of Sciences, Beijing 100093, China;
3. Institute of Wetland Ecology & Clone Ecology; Zhejiang Provincial Key Laboratory of Plant Evolutionary Ecology and Conservation, Taizhou University, Taizhou 318000, China
植物个体的表型不仅取决于该个体的基因型(遗传信息)和所处的环境, 同时也可能受其亲代个体(即亲体)所处环境的影响[1—4]。这种亲体环境对子代个体表型的修饰效应被称为亲体效应(也称亲代效应、亲系效应或亲本效应), 包括母体效应(也称母代效应、母系效应或母本效应)和父体效应(paternal effect, 也称父代效应、父系效应或父本效应)。由于亲体效应反映的是子代个体表型对亲体环境的响应模式, 因此, 亲体效应有时也被称为跨代可塑性[3, 5]。
生物因素(如采食、竞争、植物激素等)和非生物因素(如养分、水分、光照等)均可诱导亲体效应的产生[6—7]。早在1909年, 植物的亲体效应就被德国学者发现[4], 但这种效应最初被认为是母体的“环境噪音”, 会对子代个体造成不利影响[8]。近年来的进化生态学研究发现, 在很多情况下, 亲体效应是可遗传的, 并可诱导子代个体进行适应于母体环境的表型特征调整, 从而提高子代个体在与母体环境相似的环境下的行为、生态位宽度、进化潜势以及入侵能力等[9—11], 进而影响植物种群的结构、动态、进化方向和进化速率等, 因而具有重要的生态和进化意义[3, 5, 12—13]。亲体效应在非克隆植物中已被广泛报导[3, 13—16], 在克隆植物中也开始受到越来越多的关注[17—21]。
1.1 有性亲体效应母体植株可通过有性繁殖过程(如产生种子)将母体环境的影响传递给子代植株, 并影响子代植株的表型(图 1);这种通过有性繁殖传递的亲体效应, 称之为有性亲体效应。有性亲体效应中的子代是指通过两性细胞的融合形成的受精卵发育成的新个体[22]。由于种子通常与母体植株密切相关, 因此, 植物的这种亲体效应常常表现为母体效应[7]。自然界中所有的非克隆植物和大多数克隆植物都具有性繁殖能力[22—23]。因此, 有性亲体效应在非克隆植物和克隆植物中均广泛存在[2, 24]。

|
| 图 1 克隆和有性亲体效应在植物子代个体、种群和群落水平的作用 Fig. 1 Roles of clonal and sexual parental effects at individual, population and community levels of offspring plants |
越来越多的研究表明, 母体环境不仅可以影响子代个体的表型, 而且可以影响子代个体表型对环境变化的响应。例如, 母体植株所经历的土壤养分水平可以显著影响其种子的萌发率、子代个体的株高和叶生物量[24—25], 母体植株所经历的光照胁迫可以影响其种子的萌发率和萌发时间[1], 母体植株所经历的昆虫采食胁迫可以影响其种子重量、茎生物量和根冠比[17, 26]。此外, 母体效应引起的表型变异能显著提高子代植株对胁迫环境的适应性[8, 10]。例如, 母体效应在子代植株适应养分胁迫[27]、光照胁迫[28—29]、采食[26]、水分胁迫[30]、盐分胁迫[31]以及病原体感染[32]中均起着积极的作用。母体效应还能通过影响次生代谢产物(如茉莉酸和水杨酸等)的产生来提高入侵植物对本地天敌的抵御能力, 从而显著提高在入侵地的入侵性[33]。一些研究发现, 母体效应在低资源环境(如土壤养分贫瘠、干旱和低光照等)下对子代适合度的影响较高资源环境下更为显著[13—14]。也有研究表明, 母体效应会随着作用时间的延长而逐渐减弱, 或者被子代对其所在环境较强的表型响应而掩盖[34]。
1.2 克隆亲体效应克隆植物是指具有在自然条件下自发地产生遗传结构相同并具有潜在独立性新单元或个体的能力或习性的植物, 它包括克隆生长和克隆生殖两种主要繁殖方式[23]。克隆生长是高等植物最常见的无性繁殖方式[35]。克隆植物通过克隆生长所产生子代个体(即子代分株, 简称子株)的基因型与亲体(即亲代分株, 简称母株, mother ramet)完全一致, 且能够独立于亲体而生存[23]。子株直接来源于其亲体的营养繁殖体, 其在发育早期可能已对亲体环境产生了预适应, 同时, 亲体环境也可能是其发育后期所要经历的环境。因此, 子株生长发育与其亲体环境的关系密切[7, 17]。
克隆植物通过无性繁殖过程(如匍匐茎/根状茎产生新个体)将亲体环境的影响传递给子株, 并与子株的基因型和所处环境共同影响子株的表型(图 1)。通过无性繁殖传递的亲体效应, 称之为克隆亲体效应。亲体环境可影响母株营养繁殖体(如茎、根和根状茎等)的营养储备、代谢物质以及表观遗传变异等, 进而影响子代表型[5, 7]。与有性亲体效应不同, 由于营养生长的亲体无母体和父体之分, 因此克隆亲体效应也无母体效应与父体效应之分。此外, 由于营养生长形成的子代往往包含多个分株, 分株的数量和大小之间往往存在权衡[36]。因此, 克隆亲体效应在子株分株和整株水平上可能是不同的, 即当该效应对分株生长具有积极效应时, 并不能表明对子株整株的生长也具有促进作用, 反之亦然[37]。
已有研究表明, 当子株经历与亲体相似的环境时, 克隆亲体效应能显著提高子株的总生物量和平均生物量[12]。例如, 与来源于非干旱亲体的子株相比, 受干旱胁迫的亲体所产生的子株表现出更强的抗旱性[5]。然而, 也有研究发现克隆亲体效应对子株生长的影响不显著, 这可能与该亲代环境作用时间的长短、亲代植株发育阶段以及子代环境作用的方向和强度等有关[34]。因此, 克隆亲体效应对克隆植物种群, 尤其对有性繁殖受限和/或缺乏遗传变异的克隆植物种群的长期进化过程起着非常重要的作用[38—39]。尽管目前国内外已经开展了一些针对克隆植物亲体效应的研究, 但这些研究绝大多数是在个体水平进行的[17, 21], 而有关亲体效应在植物种群和群落水平作用的研究仍十分缺乏(图 1)。
1.3 亲体效应对后代表型和适合度的影响Dong等[37]认为, 亲体效应对后代表型和适合度的影响可能包括以下四种类型(图 2)。第一种类型为亲体效应不依赖于后代所处的环境, 即亲体在良好环境条件下产生的后代个体总是比在不良环境条件下产生的后代个体的适合度高, 或良好环境条件下亲体产生的后代个体的表型对后代个体的适合度提高总是有利的[40—42]。第二种类型为亲体效应在后代环境与母代环境相似时是有利的, 而在与母代环境不同时是不利的[1, 43]。第三种类型为亲体效应只有在后代处于良好环境条件下才有利, 而在后代处于不良环境条件下没有显著影响[10]。第四种类型为亲体效应只有在后代处于不良环境条件下才有利, 而在后代处于良好环境条件下没有影响[44]。
一些研究表明, 当子代个体经历与母体相似的环境条件时, 有性亲体效应往往能显著提高子代个体对这种环境的适应性, 如显著提高子代植株的个体大小、碳水化合物积累、生物量、繁殖产出和幼年植株的存活率[10, 25, 30], 提前植物的花期[45]以及提高种间竞争能力[25]等。相反, 当子代环境与母代环境不一致时, 亲体效应可能不显著, 甚至可能表现出负效应, 如延迟繁殖期、降低繁殖产出等[14]。因此, 亲体效应可以对子代适合度产生积极或消极的影响, 这与母代环境密切相关[14]。同时, 有研究发现有性亲体效应可以在代际间进行叠加, 例如两代(祖母代和母代)与一代(母代)处于干旱胁迫环境相比, 其子代具有更高的营养物质含量、更快的萌发过程、更高的存活率和更大的植株个体, 表明亲体效应在代际间的叠加能让子代提前适应相似的环境胁迫, 从而显著提高其适合度[30]。亲体效应对子代表型和适合度的影响可能进一步影响植物的种内[31]和种间关系[14, 25], 进而影响植物种群和群落的结构、功能和稳定性(图 1)。
1.3.2 克隆亲体效应对后代表型和适合度的影响克隆亲体效应可以通过亲体产生的营养繁殖体传递至子代, 在一定条件下有助于提高克隆植物对环境变化(如资源异质性)的适应能力(图 1)。与有性亲体效应一致, 在克隆亲体效应中子代适合度受亲代和子代环境的共同影响;不同的是, 克隆亲体效应同时也受营养繁殖体类型、营养物质水平和代谢物质水平等的影响[7, 17, 46—47]。在克隆亲体效应的研究中, 平均适合度和总适合度为常用指标, 它们分别以同一母株产生的所有分株的适合度指标(如分株数和/或生物量)的平均值或总和来衡量[37]。当亲代与子代均处于高养分环境时, 克隆亲体效应能显著提高克隆植物空心莲子草(Alternanthera philoxeroides)子代的平均适合度和总适合度[17, 37, 46]。同时, 克隆亲体效应也可显著提高克隆植物凤眼莲(Eichhornia crassipes)和空心莲子草等的入侵性[48—49]。已有研究表明, 不同克隆繁殖体类型的克隆亲体效应强度不同, 例如, 养分处理下空心莲子草通过母代茎形成子代的亲体效应更强, 而在采食胁迫下根形成子代的亲体效应更显著[17]。然而, 对该物种亲体效应的研究也发现其某些表观遗传变异可持续十个无性世代, 但并非长期稳定, 而是表现出中等水平的稳定性[49]。这种暂时的表观遗传稳定性可能是克隆植物(特别是遗传变异受限的植物)适应未知生境的重要机制[48, 50]。
1.4 亲体效应对后代竞争能力的影响 1.4.1 有性亲体效应对后代种内和种间竞争能力的影响母体可通过影响其种子的营养物质储备来改变子代个体的表型可塑性, 从而影响子代个体的竞争能力(图 1)。资源获取能力强的母体通常能产生高质量的种子, 使子代个体具有较强的竞争能力[2, 13, 51];同时, 在子代个体发育早期, 良好的资源条件也能提高其成株的表型性状、生物量积累和竞争能力[40, 52]。此外, 母代植株还可通过表观遗传变异(详见2.3)影响子代植株的表型可塑性, 从而改变子代的竞争力[3, 51, 53—54]。相反, 当种子的营养储备少、子代早期资源状况差或子代环境不可预测时, 母体效应可能不会显著影响甚至会抑制子代个体表型性状, 从而降低子代个体的种内和种间竞争能力[37]。由于母体环境、物种或基因型的差异, 种子的营养储备往往不同[8, 53]。因此, 相同或不同植株间可能会产生表型性状的分化, 改变对资源利用的有效性, 从而影响植物种群或群落内的种内和种间竞争强度。
已有研究较多地关注非生物因子胁迫下有性亲体效应对子代竞争能力的影响[13—14], 然而, 自然生态系统中往往同时存在非生物因子和生物因子胁迫的影响。比如, 母代植物种内/种间竞争(即生物因子胁迫)或者母代非生物与生物因子胁迫的交互效应(如竞争×土壤水分)所引起的表型可塑性也能进行跨代遗传[3, 51]。当母体经历较强的种内/种间竞争环境时, 其子代个体也具有较强的竞争能力, 表现在种子具有较快的萌发过程、植株具有较快的生长速率和较高的表型可塑性等[3, 51]。
1.4.2 克隆亲体效应对后代种内和种间竞争能力的潜在影响如果克隆亲体效应对后代个体表型的改变能提高其生长和适合度[17, 20, 37, 55], 那么克隆亲体效应可能增强植物后代个体的种内和种间竞争能力(图 1)。相反, 如果克隆亲体效应对植物后代个体表型的改变在某些情况下降低其生长和适合度[1, 10, 14], 那么克隆亲体效应将削弱后代个体的种内和种间竞争能力[25]。然而, 目前国内外有关克隆亲体效应对植物种内和种间关系影响的研究仍十分缺乏[14, 25], 已开展的少数几例研究均针对非克隆植物, 探讨其有性亲体效应的影响[14, 25, 31, 53, 56]。克隆亲体效应可能通过改变后代尤其是群落优势种的种内和种间竞争能力, 从而影响群落的组成和多样性。目前, 有关克隆亲体效应对植物种内和种间关系影响的研究还有待开展(图 1)。
1.5 亲体效应对种群结构、功能和稳定性的潜在影响植物亲代个体所经历环境的多样性可通过亲体效应使后代个体产生表型的多样性[17, 37, 41, 54], 而这种亲体效应介导的表型多样性对植物种群的结构、功能和稳定性将可能产生重要的影响(图 1)。大量研究表明, 生物多样性的提高对生态系统功能(如生产力)和稳定性(如抗干扰能力)具有重要的促进作用, 这种效应不仅体现在物种(多样性)水平[57—58], 而且也体现在种内(遗传多样性)水平[59—60]。物种多样性和遗传多样性对生态系统功能和稳定性的促进效应主要是由于物种多样性高的群落或遗传多样性高的种群包含更多对资源获取策略不同的物种或基因型, 即包含更多功能差异的表型, 从而能够更加充分的利用群落或种群中的资源(互补效应)[61], 或者由于这些群落或种群包含对其功能影响较大的优势物种或基因型, 即包含具有优势功能的表型(选择效应)[61]。物种多样性和遗传多样性介导的种间和种内表型多样性均涉及非常高的遗传变异, 而克隆和有性亲体效应介导的种内表型多样性可以完全不涉及或仅涉及少量的遗传变异[17, 37, 41]。与物种多样性和种群遗传多样性的效果类似, 如果亲体效应介导的表型多样性高的种群包含更多功能差异的表型或包含具有优势功能的表型, 那么通过互补效应和/或选择效应, 该种群的功能和稳定性可以得到显著提高。然而, 迄今为止, 国内外尚未开展有关亲体效应介导的表型多样性对种群结构、功能和稳定性影响的研究(图 1)。
1.6 亲体效应对群落结构、功能和稳定性的潜在影响对于植物群落中的优势物种, 有性亲体效应介导的后代表型多样性也可能进一步影响植物群落的组成、功能和稳定性[62—64]。与群落中优势种遗传多样性介导的表型多样性在群落水平的效应相似, 如果群落中优势种亲体效应介导的后代不同表型之间具有互补的资源利用能力, 那么后代表型多样性的增加可能提高该种群的资源利用效率和生产力, 从而使群落中其它物种由于难以获取到必需的资源而衰退[54]。在这种情况下, 群落中优势种亲体效应介导的后代表型多样性将降低群落的物种多样性和生产力。相反, 如果群落中优势种亲体效应介导的不同表型后代个体之间在竞争、促进或化感等能力上存在显著差异, 即具有功能差异, 使得群落中其它物种可以与该优势种不同表型的个体共存, 那么群落优势植物亲体效应介导的表型多样性可提高整个群落的物种多样性和生产力[54]。然而, 国内外尚缺乏有关亲体效应介导的表型多样性对群落结构、功能和稳定性影响的研究(图 1)。
2 亲体效应的潜在调控机制亲体效应受繁殖体营养储备、代谢物质水平、表观遗传变异等众多机制的调控[5]。不同调控机制下, 亲体效应作用的历时长短不同。与表观遗传机制相比, 供给机制和代谢物质调控机制对后代影响的历时相对较短[7]。在自然种群中, 这些调控机制并不能严格地区分开, 也不会相互排斥, 往往是多个机制共同影响后代表型[8]。
2.1 供给机制养分供给效应是指亲代植株将碳水化合物、脂质、蛋白质和矿质营养等分配给繁殖体, 通过繁殖体将营养物质提供给子代[8, 51]。养分供给效应在自然界普遍存在, 这在早期的研究中已被证实[4]。种子和营养繁殖体的营养储备被认为是关键的从亲体获取的性状, 通常与繁殖体数量存在权衡关系[8]。营养物质的供给量取决于亲体所处环境的资源状况和亲体的营养分配策略, 同时也受植物遗传因素和环境因素(如干旱、高温、遮阴、放牧等)的影响[51]。在相同环境条件下, 不同物种或同一物种的不同基因型个体的母体细胞质等也可影响种子的营养物质储备[8]。养分供给效应不仅影响营养繁殖芽的萌发、种子的萌发和子代幼苗存活率, 同时对子代生长、竞争能力和繁殖等也具有显著的影响[20, 37, 65—66]。
亲代较高的繁殖体分配还可能产生银勺效应(也称后续效应), 即营养储备较高的繁殖体形成的后代个体无论其生长环境条件如何, 总是具有较高的适合度[40, 67]。因此, 银勺效应能提高子代个体后期在胁迫环境下的表型可塑性、生物量积累、繁殖能力以及适合度[67—68]。近期关于克隆植物空心莲子草亲体效应的研究表明银勺效应显著提高了子代植株后期在低土壤养分条件下的生物量积累和营养繁殖能力[21]。由于该效应强调个体发育早期良好的环境条件对发育后期耐受性和适应性的影响, 有研究指出该效应对子代的影响往往具有滞后效应, 例如, 子代个体发育至成株后才表现出较强的竞争力[52, 69]。
子代个体的适合度也能反映亲体环境的营养状况:养分状况良好生境的亲体所产生的繁殖体具有较高的营养储备, 能为子代个体提供更多的营养物质, 产生的子代适合度较高;相反, 亲代植株养分供给不足时, 繁殖体营养储备较少, 供给效应减弱, 子代个体的适合度也相应降低[5, 70]。研究表明, 种子的养分供给效应对于幼苗早期适应胁迫环境具有重要作用。例如, 母体养分供给充足的子代植株能形成庞大的根系以适应干旱土壤, 或形成高大的枝干以适应遮阴环境[71]。然而, 在自然种群中, 养分供给效应也存在一定的局限性。首先, 养分供给效应对植物发育早期的表型可塑性影响较为显著, 随后逐渐减弱[3, 24]。其次, 养分供给不足时, 种子的产量和质量均不高, 供给效应较弱。最后, 由于较大的种子具有较强的竞争力和耐受能力, 而较小的种子在种子库中具有较长的存活率, 因此, 种子养分供给往往还与土壤种子库维持存在权衡关系[8]。
2.2 代谢物质调控机制植物繁殖体不仅能从亲体获取营养物质, 同时也可获得一些代谢物质, 如蛋白质、mRNAs、防御化合物、次生代谢产物和激素等[8]。其中, 蛋白质既可作为供给机制中的营养物质, 也可以作为调控分子起作用。研究表明, 蛋白质和mRNAs在调控种子休眠和萌发过程中起着重要的作用[72]。亲体环境能改变其基因表达, 繁殖体中的mRNAs能将遗传信息进行准确转录并合成蛋白, 有助于胁迫条件下种子的萌发[73—74]。母体环境胁迫还可以改变种子中激素的含量及胚对激素的敏感性;例如, 与非遮阴条件下相比, 长芒苋(Amaranthus palmeri)母体在遮阴条件下产生的种子中脱落酸的含量提高了44%[75]。种子萌发常常受多种调控因子如激素水平、蛋白质、mRNAs等的共同作用, 但这些调控因子对种子萌发和子代幼苗发育影响的相对重要性以及相关机制目前尚不明确[8, 72]。
2.3 表观遗传机制亲体的环境条件可以诱导其产生表观遗传变异, 并且这种表观遗传变异可能遗传给后代, 从而引起后代个体表型的变异, 因此, 表观遗传变异可能是亲体效应的潜在机制之一[3, 76]。
2.3.1 表观遗传变异表观遗传变异基于一系列可激活、降低或完全消除某些基因表达的分子过程, 包括DNA甲基化、染色质结构重塑、以及小RNA分子介导的调控过程等;这些分子机制可调控基因表达, 进而改变植物个体的表型[77—78]。其中, DNA甲基化广泛存在于植物体中, 它主要来源于遗传控制、环境诱导及自发产生的表观突变, 被认为是植物亲体效应的重要调控机制之一[9, 79]。
DNA甲基化是由DNA甲基转移酶(DNA methyltransferase, DNMT)催化S-腺苷甲硫氨酸(S-adenosyl-Lmethionine, SAM)作为甲基供体, 将甲基基团加在CpG岛上(CCGG), 从而将胞嘧啶转变为5-甲基胞嘧啶的一种反应[77, 80]。DNA甲基化广泛存在于细菌、真菌及动植物体中。植物基因组的甲基化常发生在对称的CG或CHG位点, 以及非对称CHH位点上(H = A, C, T), 并参与调控转录抑制[81]。首先, 基因调控区域的胞嘧啶甲基化可直接干扰转录因子与其识别位点的结合, 进而抑制基因表达[80—81]。其次, 一些结合蛋白也可特异性地结合到甲基化位点上, 并通过募集组蛋白去乙酰化酶形成竞争性转录阻遏物抑制转录因子的结合[80—81]。最后, DNA甲基化还可通过染色质重塑影响基因转录与表达[82—83]。因此, DNA甲基化可通过调控基因表达及与转录机制的相互作用影响植物表型。
已有研究表明, 环境诱导的亲代表观遗传变异至少在某些情况下可以直接遗传给子代个体, 并影响其对环境变化的表型响应[49, 84—85]。例如, 亲代经历土壤养分、水分、温度和盐分等处理介导的表观遗传变异可以显著影响植物种子的萌发以及后代个体的生长和抵御胁迫的能力[85—86];亲代经历的光照条件诱导的DNA甲基化可以显著影响空心莲子草后代个体的生活史性状[18];亲代遭受采食胁迫所诱导的表观遗传变异也可以影响野萝卜(Raphanus raphanistrum)有性后代个体对采食的防御能力[26]。因此, 表观遗传变异给植物提供一种新的快速应对环境变化的途径, 使得某些性状的快速进化成为可能[77, 87]。
2.3.2 遗传变异、表观遗传变异与表型变异遗传变异是指DNA序列的变化[59, 88], 而表观遗传变异是在没有DNA序列改变的情况下基因表达和基因功能的可遗传变化[89—90]。表观遗传变异受遗传变异的调控, 但很多情况下表观遗传变异可以部分的、甚至完全的独立于遗传变异[91—92]。一些植物种群的遗传变异与表观遗传变异之间存在显著相关性, 如拟南芥(Arabidopsis thaliana)、西班牙紫罗兰(Viola cazorlensis)、互花米草(Spartina alterniflora)、短芒大麦草(Hordeum brevisubulatum)和水稻等, 表明表观遗传变异可由遗传变异所驱动, 且两者共同影响植物的表型变异[93—98]。表观遗传变异和遗传变异两者间的相关性可能是由顺式(cis)或反式(trans)遗传信息促使特定位点表观遗传印记的获得或保留所引起的[99]。然而, 也有一些植物种群的表观遗传变异与遗传变异之间无显著相关性, 如香菇草和野生马铃薯(Solanum ruiz-lealii)种群可在遗传变异水平较低的情况下, 表现出较高的表观遗传变异[81, 92, 100—102]。表观遗传变异的独立性可能是由于减数分裂或突变过程中发生了表观遗传信息的随机漂变[78, 103]。种群表观遗传变异与遗传变异的关系对于研究种群进化具有重要意义[104];因此, 更多相关的研究亟待开展。
2.3.3 亲体效应与表观遗传变异(1) 有性亲体效应的表观遗传机制
DNA甲基化可在有性繁殖的减数分裂过程中进行保守复制并稳定传递给子代[5, 105]。在DNA复制过程中, 特异的甲基转移酶可识别新合成的半甲基化双链, 并基于链对称性将甲基加在新链的非甲基化胞嘧啶上, 从而复制甲基化模式, 实现DNA甲基化的跨代遗传[46, 106]。因此, 表观遗传可使植物“记住”所经历的环境, 预测并克服未来的环境压力, 并通过多代的可靠传递, 选择适应性信息[81, 107]。然而, 表观遗传信息在减数分裂过程中可能会发生DNA甲基化重置的现象, 阻碍表观遗传信息在世代间的传递, 从而削弱亲体效应[105, 108—109]。
有性亲体效应下DNA甲基化具有不同程度的遗传稳定性:短暂变异, 短期可遗传变异(两代间遗传), 以及长期可遗传变异(多代间遗传)[81, 110]。尽管可遗传的表观遗传变异被认为是亲体效应的重要调控机制, 但表观遗传的稳定性及其与亲体效应的关系尚不明确, 直接验证两者关系的研究仍非常缺乏[5, 46]。
(2) 克隆亲体效应的表观遗传机制
对于克隆植物的亲体效应, 由于营养繁殖缺少减数分裂重组及分离过程中的变异发生机制, 因此, 受母代环境影响的表观遗传信息能更有效地传递至新产生的子代, 并成为可遗传表型变异的重要来源[46, 49, 111]。例如, 当克隆植物香菇草的母代和子代均处于遮阴条件时, DNA甲基化变异的遗传率可达到94.44%[102]。环境胁迫的强度及持续时间可影响克隆亲体效应, 在多代间重复相同的环境胁迫有利于克隆亲体效应的维持[12, 109]。例如, 与来源于非干旱胁迫的子代相比, 受干旱胁迫的母代所产生的子代表现出更强的耐旱性[5]。因此, 表观遗传变异对克隆植物种群, 特别是有性繁殖受限、缺乏遗传变异的克隆植物种群尤为重要, 提高了这些植物对环境变化的适应能力, 在其长期进化过程中可能发挥着重要的作用[38—39]。然而, 目前关于表观遗传变异在克隆亲体效应中的作用尚不清楚[111]。
3 克隆植物、亲体效应与表观遗传机制克隆植物在自然界广泛存在, 是很多生态系统的优势物种[23, 112—113]。克隆植物特有的生活史性状包括营养繁殖、克隆整合、克隆可塑性和克隆内分工等, 这些性状可以增加克隆植物抗逆、抗干扰的能力以及对异质性资源的利用能力[35, 47]。研究表明, 很多克隆植物如空心莲子草、凤眼莲和虎杖(Fallopia japonica)等种群的遗传多样性极低, 仅由一个或几个优势基因型组成[23, 48, 50, 114], 但却存在较高的表观遗传变异(表现为较高的DNA甲基化水平)。通过克隆亲体效应, 受母代环境影响的表观遗传信息能有效地传递给后代, 并影响无性后代个体的表型[7, 111], 然而, 它是否会影响无性后代个体的克隆生活史性状和适合度目前尚不清楚。如果克隆亲体效应能通过表观遗传变异影响后代个体的克隆生活史性状, 那么它将可以极大地提高克隆植物适应环境胁迫和环境异质性的能力(图 1)。
在自然生境中, 克隆植物种群内和种群间表观遗传变异的大小及差异, 以及表观遗传变异、遗传变异、表型变异和环境变化之间的关系均是目前生态表观遗传学亟待解决的关键科学问题(图 1)。由于不同生境下的植物种群遗传变异程度不同, 自然植物种群遗传变异和表观遗传变异对表型变异的贡献程度较难区分开[91—92], 然而, 明确这些变异之间的关系是深入理解表观遗传变异影响克隆植物进化潜势的前提和基础[104]。在区分表观遗传变异和遗传变异效应的研究中, 通常选用同一种系的后代个体来控制遗传变异, 但不同个体间仍存在遗传变异[26, 115—116]。与非克隆植物相比, 克隆植物遗传变异程度相对较小, 来自同一基因型基株的后代个体可以严格控制遗传变异的潜在影响, 因而易于区分遗传变异与表观遗传变异的作用[9, 102]。因而, 克隆植物更有利于探究表观遗传变异在多种生态和进化过程中的功能意义[46, 117]。
4 展望亲体效应对植物尤其是克隆植物的长期适应进化具有重要意义[38—39, 47]。然而, 个体水平上克隆亲体效应对克隆后代特有生活史性状的影响及其表观遗传机制尚不明确, 种群和群落水平上克隆和有性亲体效应的作用及其表观遗传机制的相关研究就更为缺乏。亲体效应所介导的后代(尤其是优势物种或基因型)的表型多样性可能与物种或基因型多样性具有相似的生态学效应[7, 56, 118—121]。由于不同表型之间可能具有资源利用互补、生态位分化或形成优势表型的潜力, 种群表型多样性越高, 其生态功能(如生产力积累或抗干扰能力)可能也越强。
基于前期相关基础, 研究认为仍有以下几个重要问题需要进一步关注。在个体水平上:(1)克隆植物自发表观突变的发生频率及其遗传稳定性如何?(2)遗传变异与表观遗传变异调控克隆生活史性状变异的贡献程度如何?(3)克隆植物基因组特点、倍性水平、生活史特征、生境预测能力等是否会对表观遗传变异产生影响?在种群和群落水平上:(4)克隆和有性亲体效应介导的表型变异是否影响植物的种内和种间关系?(5)克隆和有性亲体效应介导的表型多样性是否影响植物种群的结构和生产力?(6)克隆和有性亲体效应介导的表型多样性是否影响群落的组成和生产力?(7)植物野外种群表观遗传变异与其种群和群落特征之间是否存在相关关系?通过阐明克隆和有性亲体效应在种群和群落水平的作用及表观遗传机制, 以期能深入理解克隆和非克隆植物种群的适应与进化、群落的结构与动态以及生态系统的结构、功能和稳定性, 进一步推动生态表观遗传学和植物生态学的发展。
| [1] |
Galloway L F. Maternal effects provide phenotypic adaptation to local environmental conditions. New Phytologist, 2005, 166(1): 93-100. DOI:10.1111/j.1469-8137.2004.01314.x |
| [2] |
Germain R M, Gilbert B. Hidden responses to environmental variation: maternal effects reveal species niche dimensions. Ecology Letters, 2014, 17(6): 662-669. DOI:10.1111/ele.12267 |
| [3] |
Puy J, de Bello F, Dvořáková H, Medina N G, Latzel V, Carmona C P. Competition-induced transgenerational plasticity influences competitive interactions and leaf decomposition of offspring. New Phytologist, 2021, 229(6): 3497-3507. DOI:10.1111/nph.17037 |
| [4] |
Roach D A, Wulff R D. Maternal effects in plants. Annual Review of Ecology and Systematics, 1987, 18: 209-235. DOI:10.1146/annurev.es.18.110187.001233 |
| [5] |
Herman J J, Sultan S E. DNA methylation mediates genetic variation for adaptive transgenerational plasticity. Proceedings of the Royal Society B: Biological Sciences, 2016, 283(1838): 20160988. DOI:10.1098/rspb.2016.0988 |
| [6] |
Boyko A, Kovalchuk I. Transgenerational response to stress in Arabidopsis thaliana. Plant Signaling & Behavior, 2010, 5(8): 995-998. |
| [7] |
Latzel V, Klimešová J. Transgenerational plasticity in clonal plants. Evolutionary Ecology, 2010, 24(6): 1537-1543. DOI:10.1007/s10682-010-9385-2 |
| [8] |
Herman J J, Sultan S E. Adaptive transgenerational plasticity in plants: case studies, mechanisms, and implications for natural populations. Frontiers in Plant Science, 2011, 2: 102. |
| [9] |
Hawes N A, Fidler A E, Tremblay L A, Pochon X, Dunphy B J, Smith K F. Understanding the role of DNA methylation in successful biological invasions: a review. Biological Invasions, 2018, 20(9): 2285-2300. DOI:10.1007/s10530-018-1703-6 |
| [10] |
Latzel V, Janeček Š, Doležal J, Klimeová J, Bossdorf O. Adaptive transgenerational plasticity in the perennial Plantago lanceolata. Oikos, 2014, 123(1): 41-46. DOI:10.1111/j.1600-0706.2013.00537.x |
| [11] |
Yang F, Yang X J, Baskin J M, Baskin C C, Cao D C, Huang Z Y. Transgenerational plasticity provides ecological diversity for a seed heteromorphic species in response to environmental heterogeneity. Perspectives in Plant Ecology Evolution and Systematics, 2015, 17(3): 201-208. DOI:10.1016/j.ppees.2015.03.003 |
| [12] |
González A P R, Chrtek J, Dobrev P I, Dumalasová V, Fehrer J, Mráz P, Latzel V. Stress-induced memory alters growth of clonal offspring of white clover (Trifolium repens). American Journal of Botany, 2016, 103(9): 1567-1574. DOI:10.3732/ajb.1500526 |
| [13] |
Li Y, Hou L Y, Yang L Y, Yue M. Transgenerational effect alters the interspecific competition between two dominant species in a temperate steppe. Ecology and Evolution, 2021, 11(3): 1175-1186. DOI:10.1002/ece3.7066 |
| [14] |
Baker B H, Sultan S E, Lopez-Ichikawa M, Waterman R. Transgenerational effects of parental light environment on progeny competitive performance and lifetime fitness. Philosophical Transactions of the Royal Society B: Biological Sciences, 2019, 374(1768): 20180182. DOI:10.1098/rstb.2018.0182 |
| [15] |
Fenesi A, Dyer A R, Geréd J, Sándor D, Ruprecht E. Can transgenerational plasticity contribute to the invasion success of annual plant species?. Oecologia, 2014, 176(1): 95-106. DOI:10.1007/s00442-014-2994-7 |
| [16] |
Groot M P, Kooke R, Knoben N, Vergeer P, Keurentjes J J B, Ouborg N J, Verhoeven K J F. Effects of multi-generational stress exposure and offspring environment on the expression and persistence of transgenerational effects in Arabidopsis thaliana. PLoS One, 2016, 11(3): e0151566. DOI:10.1371/journal.pone.0151566 |
| [17] |
Dong B C, Alpert P, Yu F H. Transgenerational effects of herbivory and soil nutrients transmitted via vegetative reproduction in the clonal plant Alternanthera philoxeroides. Perspectives in Plant Ecology, Evolution and Systematics, 2019, 41: 125498. DOI:10.1016/j.ppees.2019.125498 |
| [18] |
Dong B C, Meng J, Yu F H. Effects of parental light environment on growth and morphological responses of clonal offspring. Plant Biology, 2019, 21(6): 1083-1089. DOI:10.1111/plb.13001 |
| [19] |
González A P R, Preite V, Verhoeven K J F, Latzel V. Transgenerational effects and epigenetic memory in the clonal plant Trifolium repens. Frontiers in Plant Science, 2018, 9: 1677. DOI:10.3389/fpls.2018.01677 |
| [20] |
Latzel V, Münzbergová Z. Anticipatory behavior of the clonal plant Fragaria vesca. Frontiers in Plant Science, 2018, 9: 1847. DOI:10.3389/fpls.2018.01847 |
| [21] |
Portela R, Dong B C, Yu F H, Barreiro R, Roiloa S R, Matos D M S. Trans-generational effects in the clonal invader Alternanthera philoxeroides. Journal of Plant Ecology, 2020, 13(1): 122-129. DOI:10.1093/jpe/rtz043 |
| [22] |
周云龙, 刘全儒. 植物生物学 (第四版). 北京: 高等教育出版社, 2016.
|
| [23] |
董鸣. 克隆植物生态学. 北京: 科学出版社, 2011.
|
| [24] |
Latzel V, Klimešová J, Hájek T, Gómez S, Šmilauer P. Maternal effects alter progeny's response to disturbance and nutrients in two Plantago species. Oikos, 2010, 119(11): 1700-1710. DOI:10.1111/j.1600-0706.2010.18737.x |
| [25] |
Bossdorf O, Shuja Z, Banta J A. Genotype and maternal environment affect belowground interactions between Arabidopsis thaliana and its competitors. Oikos, 2009, 118(10): 1541-1551. DOI:10.1111/j.1600-0706.2009.17559.x |
| [26] |
Agrawal A A. Herbivory and maternal effects: mechanisms and consequences of transgenerational induced plant resistance. Ecology, 2002, 83(12): 3408-3415. DOI:10.1890/0012-9658(2002)083[3408:HAMEMA]2.0.CO;2 |
| [27] |
Latzel V, Hájek T, Klimešová J, Gómez S. Nutrients and disturbance history in two Plantago species: maternal effects as a clue for observed dichotomy between resprouting and seeding strategies. Oikos, 2009, 118(11): 1669-1678. DOI:10.1111/j.1600-0706.2009.17767.x |
| [28] |
Galloway L F, Etterson J R. Transgenerational plasticity is adaptive in the wild. Science, 2007, 318(5853): 1134-1136. DOI:10.1126/science.1148766 |
| [29] |
McIntyre P J, Strauss S Y. Phenotypic and transgenerational plasticity promote local adaptation to sun and shade environments. Evolutionary Ecology, 2014, 28(2): 229-246. DOI:10.1007/s10682-013-9670-y |
| [30] |
Herman J J, Sultan S E, Horgan-Kobelski T, Riggs C. Adaptive transgenerational plasticity in an annual plant: grandparental and parental drought stress enhance performance of seedlings in dry soil. Integrative and Comparative Biology, 2012, 52(1): 77-88. DOI:10.1093/icb/ics041 |
| [31] |
Castro B M, Moriuchi K S, Friesen M L, Badri M, Nuzhdin S V, Strauss S Y, Cook D R, von Wettberg E. Parental environments and interactions with conspecifics alter salinity tolerance of offspring in the annual Medicago truncatula. Journal of Ecology, 2013, 101(5): 1281-1287. DOI:10.1111/1365-2745.12125 |
| [32] |
Vivas M, Zas R, Sampedro L, Solla A. Environmental maternal effects mediate the resistance of maritime pine to biotic stress. PLoS One, 2013, 8(7): e70148. DOI:10.1371/journal.pone.0070148 |
| [33] |
Mounger J, Ainouche M L, Bossdorf O, Cavé-Radet A, Li B, Parepa M, Salmon A, Yang J, Richards C L. Epigenetics and the success of invasive plants. Philosophical Transactions of the Royal Society B: Biological Sciences, 2021, 376(1826): 20200117. DOI:10.1098/rstb.2020.0117 |
| [34] |
Auge G A, Leverett L D, Edwards B R, Donohue K. Adjusting phenotypes via within- and across-generational plasticity. New Phytologist, 2017, 216(2): 343-349. DOI:10.1111/nph.14495 |
| [35] |
董鸣, 于飞海. 克隆植物生态学术语和概念. 植物生态学报, 2007, 31(4): 689-694. DOI:10.3321/j.issn:1005-264X.2007.04.020 |
| [36] |
Wang P, Xu Y S, Dong B C, Xue W, Yu F H. Effects of clonal fragmentation on intraspecific competition of a stoloniferous floating plant. Plant Biology, 2014, 16(6): 1121-1126. |
| [37] |
Dong B C, van Kleunen M, Yu F H. Context-dependent parental effects on clonal offspring performance. Frontiers in Plant Science, 2018, 9: 1824. DOI:10.3389/fpls.2018.01824 |
| [38] |
McKey D, Elias M, Pujol B, Duputié A. The evolutionary ecology of clonally propagated domesticated plants. New Phytologist, 2010, 186(2): 318-332. DOI:10.1111/j.1469-8137.2010.03210.x |
| [39] |
Kalisz S, Purugganan M D. Epialleles via DNA methylation: consequences for plant evolution. Trends in Ecology & Evolution, 2004, 19(6): 309-314. |
| [40] |
Engqvist L, Reinhold K. Adaptive trans-generational phenotypic plasticity and the lack of an experimental control in reciprocal match/mismatch experiments. Methods in Ecology and Evolution, 2016, 7(12): 1482-1488. DOI:10.1111/2041-210X.12618 |
| [41] |
Dong B C, Fu T, Luo F L, Yu F H. Herbivory-induced maternal effects on growth and defense traits in the clonal species Alternanthera philoxeroides. Science of the Total Environment, 2017, 605-606: 114-123. DOI:10.1016/j.scitotenv.2017.06.141 |
| [42] |
Dong B C, Wang M Z, Liu R H, Luo F L, Li H L, Yu F H. Direct and legacy effects of herbivory on growth and physiology of a clonal plant. Biological Invasions, 2018, 20(12): 3631-3645. DOI:10.1007/s10530-018-1801-5 |
| [43] |
Dyer A R, Brown C S, Espeland E K, McKay J K, Meimberg H, Rice K J. SYNTHESIS: the role of adaptive trans-generational plasticity in biological invasions of plants. Evolutionary Applications, 2010, 3(2): 179-192. DOI:10.1111/j.1752-4571.2010.00118.x |
| [44] |
Dziminski M A, Roberts J D. Fitness consequences of variable maternal provisioning in quacking frogs (Crinia georgiana). Journal of Evolutionary Biology, 2006, 19(1): 144-155. DOI:10.1111/j.1420-9101.2005.00978.x |
| [45] |
Suter L, Widmer A. Environmental heat and salt stress induce transgenerational phenotypic changes in Arabidopsis thaliana. PLoS One, 2013, 8(4): e60364. DOI:10.1371/journal.pone.0060364 |
| [46] |
Verhoeven K J F, Preite V. Epigenetic variation in asexually reproducing organisms. Evolution, 2014, 68(3): 644-655. DOI:10.1111/evo.12320 |
| [47] |
Adomako M O, Alpert P, Du D L, Yu F H. Effects of fragmentation of clones compound over vegetative generations in the floating plant Pistia stratiotes. Annals of Botany, 2021, 127(1): 123-133. DOI:10.1093/aob/mcaa150 |
| [48] |
Zhang Y Y, Zhang D Y, Barrett S C H. Genetic uniformity characterizes the invasive spread of water hyacinth (Eichhornia crassipes), a clonal aquatic plant. Molecular Ecology, 2010, 19(9): 1774-1786. DOI:10.1111/j.1365-294X.2010.04609.x |
| [49] |
Shi W, Chen X J, Gao L X, Xu C Y, Ou X K, Bossdorf O, Yang J, Geng Y P. Transient stability of epigenetic population differentiation in a clonal invader. Frontiers in Plant Science, 2019, 9: 1851. DOI:10.3389/fpls.2018.01851 |
| [50] |
Ye W H, Li J, Cao H L, Ge X J. Genetic uniformity of Alternanthera philoxeroides in South China. Weed Research, 2003, 43(4): 297-302. DOI:10.1046/j.1365-3180.2003.00346.x |
| [51] |
Germain R M, Grainger T N, Jones N T, Gilbert B. Maternal provisioning is structured by species' competitive neighborhoods. Oikos, 2019, 128(1): 45-53. DOI:10.1111/oik.05530 |
| [52] |
Van Allen B G, Rudolf V H W. Habitat-mediated carry-over effects lead to context-dependent outcomes of species interactions. Journal of Animal Ecology, 2015, 84(6): 1646-1656. DOI:10.1111/1365-2656.12408 |
| [53] |
Puy J, Carmona C P, Dvořáková H, Latzel V, de Bello F. Diversity of parental environments increases phenotypic variation in Arabidopsis populations more than genetic diversity but similarly affects productivity. Annals of Botany, 2021, 127(4): 425-436. DOI:10.1093/aob/mcaa100 |
| [54] |
Westerband A C, Funk J L, Barton K E. Intraspecific trait variation in plants: a renewed focus on its role in ecological processes. Annals of Botany, 2021, 127(4): 397-410. DOI:10.1093/aob/mcab011 |
| [55] |
Münzbergová Z, Latzel V, Šurinová M, Hadincová V. DNA methylation as a possible mechanism affecting ability of natural populations to adapt to changing climate. Oikos, 2019, 128(1): 124-134. DOI:10.1111/oik.05591 |
| [56] |
Dechaine J M, Brock M T, Weinig C. Maternal environmental effects of competition influence evolutionary potential in rapeseed (Brassica rapa). Evolutionary Ecology, 2015, 29(1): 77-91. DOI:10.1007/s10682-014-9735-6 |
| [57] |
Adomako M O, Ning L, Tang M, Du D L, van Kleunen M, Yu F H. Diversity- and density-mediated allelopathic effects of resident plant communities on invasion by an exotic plant. Plant and Soil, 2019, 440(1/2): 581-592. |
| [58] |
Isbell F, Cowles J, Dee L E, Loreau M, Reich P B, Gonzalez A, Hector A, Schmid B. Quantifying effects of biodiversity on ecosystem functioning across times and places. Ecology Letters, 2018, 21(6): 763-778. DOI:10.1111/ele.12928 |
| [59] |
Hughes A R, Inouye B D, Johnson M T J, Underwood N, Vellend M. Ecological consequences of genetic diversity. Ecology Letters, 2008, 11(6): 609-623. DOI:10.1111/j.1461-0248.2008.01179.x |
| [60] |
Zhu Y Y, Chen H R, Fan J H, Wang Y Y, Li Y, Chen J B, Fan J X, Yang S S, Hu L P, Leung H, Mew T W, Teng P S, Wang Z H, Mundt C C. Genetic diversity and disease control in rice. Nature, 2000, 406(6797): 718-722. DOI:10.1038/35021046 |
| [61] |
Loreau M, Hector A. Partitioning selection and complementarity in biodiversity experiments. Nature, 2001, 412(6842): 72-76. DOI:10.1038/35083573 |
| [62] |
Agashe D. The stabilizing effect of intraspecific genetic variation on population dynamics in novel and ancestral habitats. The American Naturalist, 2009, 174(2): 255-267. DOI:10.1086/600085 |
| [63] |
Fridley J D, Grime J P. Community and ecosystem effects of intraspecific genetic diversity in grassland microcosms of varying species diversity. Ecology, 2010, 91(8): 2272-2283. DOI:10.1890/09-1240.1 |
| [64] |
Struebig M J, Kingston T, Petit E J, Le Comber S C, Zubaid A, Mohd-Adnan A, Rossiter S J. Parallel declines in species and genetic diversity in tropical forest fragments. Ecology Letters, 2011, 14(6): 582-590. DOI:10.1111/j.1461-0248.2011.01623.x |
| [65] |
Gómez J M. Bigger is not always better: conflicting selective pressures on seed size in Quercus ilex. Evolution, 2004, 58(1): 71-80. DOI:10.1111/j.0014-3820.2004.tb01574.x |
| [66] |
Fenner M, Thompson K. The Ecology of Seeds. Cambridge: Cambridge University Press, 2005.
|
| [67] |
Hopwood P E, Moore A J, Royle N J. Effects of resource variation during early life and adult social environment on contest outcomes in burying beetles: a context-dependent silver spoon strategy?. Proceedings of the Royal Society B: Biological Sciences, 2014, 281(1785): 20133102. DOI:10.1098/rspb.2013.3102 |
| [68] |
Barrett E L B, Moore A J, Moore P J. Diet and social conditions during sexual maturation have unpredictable influences on female life history trade-offs. Journal of Evolutionary Biology, 2009, 22(3): 571-581. DOI:10.1111/j.1420-9101.2008.01671.x |
| [69] |
Levine J M, Rees M. Effects of temporal variability on rare plant persistence in annual systems. The American Naturalist, 2004, 164(3): 350-363. DOI:10.1086/422859 |
| [70] |
Moles A T, Westoby M. Seed size and plant strategy across the whole life cycle. Oikos, 2006, 113(1): 91-105. DOI:10.1111/j.0030-1299.2006.14194.x |
| [71] |
Baker B H, Berg L J, Sultan S E. Context-dependent developmental effects of parental shade versus sun are mediated by DNA methylation. Frontiers in Plant Science, 2018, 9: 1251. DOI:10.3389/fpls.2018.01251 |
| [72] |
Donohue K. Completing the cycle: maternal effects as the missing link in plant life histories. Philosophical Transactions of the Royal Society B: Biological Sciences, 2009, 364(1520): 1059-1074. DOI:10.1098/rstb.2008.0291 |
| [73] |
Rajjou L, Gallardo K, Debeaujon I, Vandekerckhove J, Job C, Job D. The effect of alpha-amanitin on the Arabidopsis seed proteome highlights the distinct roles of stored and neosynthesized mRNAs during germination. Plant Physiology, 2004, 134(4): 1598-1613. DOI:10.1104/pp.103.036293 |
| [74] |
Peleg Z, Blumwald E. Hormone balance and abiotic stress tolerance in crop plants. Current Opinion in Plant Biology, 2011, 14(3): 290-295. DOI:10.1016/j.pbi.2011.02.001 |
| [75] |
Jha P, Norsworthy J K, Riley M B, Bridges W Jr. Shade and plant location effects on germination and hormone content of Palmer amaranth (Amaranthus palmeri) seed. Weed Science, 2010, 58(1): 16-21. DOI:10.1614/WS-09-059.1 |
| [76] |
Hauser M T, Aufsatz W, Jonak C, Luschnig C. Transgenerational epigenetic inheritance in plants. Biochimica et Biophysica Acta (BBA)-Gene Regulatory Mechanisms, 2011, 1809(8): 459-468. DOI:10.1016/j.bbagrm.2011.03.007 |
| [77] |
Bossdorf O, Richards C L, Pigliucci M. Epigenetics for ecologists. Ecology Letters, 2008, 11(2): 106-115. |
| [78] |
Richards C L, Bossdorf O, Verhoeven K J F. Understanding natural epigenetic variation. New Phytologist, 2010, 187(3): 562-564. DOI:10.1111/j.1469-8137.2010.03369.x |
| [79] |
Huang X N, Li S G, Ni P, Gao Y C, Jiang B, Zhou Z C, Zhan A B. Rapid response to changing environments during biological invasions: DNA methylation perspectives. Molecular Ecology, 2017, 26(23): 6621-6633. DOI:10.1111/mec.14382 |
| [80] |
Cervera M T, Ruiz-García L, Martínez-Zapater J M. Analysis of DNA methylation in Arabidopsis thaliana based on methylation-sensitive AFLP markers. Molecular Genetics and Genomics, 2003, 268(6): 832-833. DOI:10.1007/s00438-003-0829-z |
| [81] |
Schulz B, Eckstein R L, Durka W. Epigenetic variation reflects dynamic habitat conditions in a rare floodplain herb. Molecular Ecology, 2014, 23(14): 3523-3537. DOI:10.1111/mec.12835 |
| [82] |
Busconi M, Colli L, Sánchez R A, Santaella M, De-Los-Mozos Pascual M, Santana O, Roldán M, Fernández J A. AFLP and MS-AFLP analysis of the variation within saffron crocus (Crocus sativus L.) germplasm. PLoS One, 2015, 10(4): e0123434. DOI:10.1371/journal.pone.0123434 |
| [83] |
Grativol C, Hemerly A S, Ferreira P C G. Genetic and epigenetic regulation of stress responses in natural plant populations. Biochimica et Biophysica Acta (BBA)-Gene Regulatory Mechanisms, 2012, 1819(2): 176-185. DOI:10.1016/j.bbagrm.2011.08.010 |
| [84] |
Banta J A, Richards C L. Quantitative epigenetics and evolution. Heredity, 2018, 121(3): 210-224. DOI:10.1038/s41437-018-0114-x |
| [85] |
Chang Y N, Zhu C, Jiang J, Zhang H M, Zhu J K, Duan C G. Epigenetic regulation in plant abiotic stress responses. Journal of Integrative Plant Biology, 2020, 62(5): 563-580. DOI:10.1111/jipb.12901 |
| [86] |
Akhter Z, Bi Z Z, Ali K, Sun C, Fiaz S, Haider F U, Bai J P. In response to abiotic stress, DNA methylation confers epigenetic changes in plants. Plants, 2021, 10(6): 1096. DOI:10.3390/plants10061096 |
| [87] |
Bossdorf O, Zhang Y Y. A truly ecological epigenetics study. Molecular Ecology, 2011, 20(8): 1572-1574. DOI:10.1111/j.1365-294X.2011.05044.x |
| [88] |
Avramidou E V, Ganopoulos I V, Doulis A G, Tsaftaris A S, Aravanopoulos F A. Beyond population genetics: natural epigenetic variation in wild cherry (Prunus avium). Tree Genetics & Genomes, 2015, 11(5): 95. |
| [89] |
Busconi M, Soffritti G, Stagnati L, Marocco A, Martínez J M, De Los Mozos Pascual M, Fernandez J A. Epigenetic stability in Saffron (Crocus sativus L.) accessions during four consecutive years of cultivation and vegetative propagation under open field conditions. Plant Science, 2018, 277: 1-10. DOI:10.1016/j.plantsci.2018.09.005 |
| [90] |
Jablonka E, Lamb M J. The changing concept of epigenetics. Annals of the New York Academy of Sciences, 2002, 981(1): 82-96. |
| [91] |
Massicotte R, Whitelaw E, Angers B. DNA methylation: a source of random variation in natural populations. Epigenetics, 2011, 6(4): 421-427. DOI:10.4161/epi.6.4.14532 |
| [92] |
Foust C M, Preite V, Schrey A W, Alvarez M, Robertson M H, Verhoeven K J F, Richards C L. Genetic and epigenetic differences associated with environmental gradients in replicate populations of two salt marsh perennials. Molecular Ecology, 2016, 25(8): 1639-1652. DOI:10.1111/mec.13522 |
| [93] |
Becker C, Hagmann J, Müller J, Koenig D, Stegle O, Borgwardt K, Weigel D. Spontaneous epigenetic variation in the Arabidopsis thaliana methylome. Nature, 2011, 480(7376): 245-249. DOI:10.1038/nature10555 |
| [94] |
Dubin M J, Zhang P, Meng D Z, Remigereau M S, Osborne E J, Casale F P, Drewe P, Kahles A, Jean G, Vilhjálmsson B, Jagoda J, Irez S, Voronin V, Song Q, Long Q, Rätsch G, Stegle O, Clark R M, Nordborg M. DNA methylation in Arabidopsis has a genetic basis and shows evidence of local adaptation. eLife, 2015, 4: e05255. DOI:10.7554/eLife.05255 |
| [95] |
Herrera C M, Bazaga P. Epigenetic differentiation and relationship to adaptive genetic divergence in discrete populations of the violet Viola cazorlensis. New Phytologist, 2010, 187(3): 867-876. DOI:10.1111/j.1469-8137.2010.03298.x |
| [96] |
Robertson M, Schrey A, Shayter A, Moss C J, Richards C. Genetic and epigenetic variation in Spartina alterniflora following the Deepwater Horizon oil spill. Evolutionary Applications, 2017, 10(8): 792-801. DOI:10.1111/eva.12482 |
| [97] |
Shan X H, Li Y D, Liu X M, Wu Y, Zhang M Z, Guo W L, Liu B, Yuan Y P. Comparative analyses of genetic/epigenetic diversities and structures in a wild barley species (Hordeum brevisubulatum) using MSAP, SSAP and AFLP. Genetics and Molecular Research, 2012, 11(3): 2749-2759. DOI:10.4238/2012.August.17.2 |
| [98] |
Garg R, Chevala V V S N, Shankar R, Jain M. Divergent DNA methylation patterns associated with gene expression in rice cultivars with contrasting drought and salinity stress response. Scientific Reports, 2015, 5: 14922. DOI:10.1038/srep14922 |
| [99] |
Herrera C M, Bazaga P. Untangling individual variation in natural populations: ecological, genetic and epigenetic correlates of long-term inequality in herbivory. Molecular Ecology, 2011, 20(8): 1675-1688. DOI:10.1111/j.1365-294X.2011.05026.x |
| [100] |
Medrano M, Herrera C M, Bazaga P. Epigenetic variation predicts regional and local intraspecific functional diversity in a perennial herb. Molecular Ecology, 2014, 23(20): 4926-4938. DOI:10.1111/mec.12911 |
| [101] |
Marfil C F, Camadro E L, Masuelli R W. Phenotypic instability and epigenetic variability in a diploid potato of hybrid origin, Solanum ruiz-lealii. BMC Plant Biology, 2009, 9(1): 21. DOI:10.1186/1471-2229-9-21 |
| [102] |
Wang M Z, Li H L, Li J M, Yu F H. Correlations between genetic, epigenetic and phenotypic variation of an introduced clonal herb. Heredity, 2020, 124(4): 146-155. |
| [103] |
Riddle N C, Richards E J. The control of natural variation in cytosine methylation in Arabidopsis. Genetics, 2002, 162(1): 355-363. DOI:10.1093/genetics/162.1.355 |
| [104] |
Richards C L, Alonso C, Becker C, Bossdorf O, Bucher E, Colomé-Tatché M, Durka W, Engelhardt J, Gaspar B, Gogol‐Döring A, Grosse I, van Gurp T P, Heer K, Kronholm I, Lampei C, Latzel V, Mirouze M, Opgenoorth L, Paun O, Prohaska S J, Rensing S A, Stadler P F, Trucchi E, Ullrich K, Verhoeven K J F. Ecological plant epigenetics: evidence from model and non-model species, and the way forward. Ecology Letters, 2017, 20(12): 1576-1590. DOI:10.1111/ele.12858 |
| [105] |
González A P R, Dumalasová V, Rosenthal J, Skuhrovec J, Latzel V. The role of transgenerational effects in adaptation of clonal offspring of white clover (Trifolium repens) to drought and herbivory. Evolutionary Ecology, 2017, 31(3): 345-361. DOI:10.1007/s10682-016-9844-5 |
| [106] |
Miryeganeh M, Saze H. Epigenetic inheritance and plant evolution. Population Ecology, 2020, 62(1): 17-27. DOI:10.1002/1438-390X.12018 |
| [107] |
Paun O, Verhoeven K J F, Richards C L. Opportunities and limitations of reduced representation bisulfite sequencing in plant ecological epigenomics. New Phytologist, 2019, 221(2): 738-742. DOI:10.1111/nph.15388 |
| [108] |
Paszkowski J, Grossniklaus U. Selected aspects of transgenerational epigenetic inheritance and resetting in plants. Current Opinion in Plant Biology, 2011, 14(2): 195-203. DOI:10.1016/j.pbi.2011.01.002 |
| [109] |
Boyko A, Blevins T, Yao Y L, Golubov A, Bilichak A, Ilnytskyy Y, Hollunder J, Meins F Jr, Kovalchuk I. Transgenerational adaptation of Arabidopsis to stress requires DNA methylation and the function of dicer-like proteins. PLoS One, 2010, 5(3): e9514. DOI:10.1371/journal.pone.0009514 |
| [110] |
Herman J J, Spencer H G, Donohue K, Sultan S E. How stable 'should' epigenetic modifications be? Insights from adaptive plasticity and bet hedging. Evolution, 2014, 68(3): 632-643. DOI:10.1111/evo.12324 |
| [111] |
Latzel V, González A P R, Rosenthal J. Epigenetic memory as a basis for intelligent behavior in clonal plants. Frontiers in Plant Science, 2016, 7: 1354. |
| [112] |
Gross K L, Herben T, Klimešová J. Introduction to special issue on the ecology of clonal plants. Folia Geobotanica, 2017, 52(3-4): 265-267. DOI:10.1007/s12224-017-9309-9 |
| [113] |
Wang Y J, Müller-Schärer H, van Kleunen M, Cai A M, Zhang P, Yan R, Dong B C, Yu F H. Invasive alien plants benefit more from clonal integration in heterogeneous environments than natives. New Phytologist, 2017, 216(4): 1072-1078. DOI:10.1111/nph.14820 |
| [114] |
Richards C L, Schrey A W, Pigliucci M. Invasion of diverse habitats by few Japanese knotweed genotypes is correlated with epigenetic differentiation. Ecology Letters, 2012, 15(9): 1016-1025. DOI:10.1111/j.1461-0248.2012.01824.x |
| [115] |
Mastan S G, Rathore M S, Bhatt V D, Chikara J, Ghosh A. DNA methylation and methylation polymorphism in ecotypes of Jatropha curcas L. using methylation-sensitive AFLP markers. Molecular Biology Reports, 2014, 41(12): 8261-8271. DOI:10.1007/s11033-014-3734-0 |
| [116] |
Zhang Y Y, Latzel V, Fischer M, Bossdorf O. Understanding the evolutionary potential of epigenetic variation: a comparison of heritable phenotypic variation in epiRILs, RILs, and natural ecotypes of Arabidopsis thaliana. Heredity, 2018, 121(3): 257-265. DOI:10.1038/s41437-018-0095-9 |
| [117] |
Rapp R A, Wendel J F. Epigenetics and plant evolution. New Phytologist, 2005, 168(1): 81-91. DOI:10.1111/j.1469-8137.2005.01491.x |
| [118] |
Badyaev A V, Uller T. Parental effects in ecology and evolution: mechanisms, processes and implications. Philosophical Transactions of the Royal Society B: Biological Sciences, 2009, 364(1520): 1169-1177. DOI:10.1098/rstb.2008.0302 |
| [119] |
Hereford J, Moriuchi K S. Variation among populations of Diodia teres (Rubiaceae) in environmental maternal effects. Journal of Evolutionary Biology, 2005, 18(1): 124-131. DOI:10.1111/j.1420-9101.2004.00797.x |
| [120] |
Plaistow S J, Lapsley C T, Benton T G. Context‐dependent intergenerational effects: the interaction between past and present environments and its effect on population dynamics. The American Naturalist, 2006, 167(2): 206-215. DOI:10.1086/499380 |
| [121] |
Li C, Wang T, Zhang M, Xu J. Maternal environment effect of warming and eutrophication on the emergence of curled pondweed, Potamogeton crispus L. Water, 2018, 10(9): 1285. DOI:10.3390/w10091285 |
 2022, Vol. 42
2022, Vol. 42