文章信息
- 李玉龙, 鲍相渤, 李轶平, 周遵春, 付杰, 高祥刚, 陈百灵, 李云峰
- LI Yulong, BAO Xiangbo, LI Yiping, ZHOU Zunchun, FU Jie, GAO Xianggang, CHEN Bailing, LI Yunfeng
- 基于环境DNA宏条形码技术的辽东湾典型围海养殖池塘内水母多样性研究
- Research on jellyfish diversity of marine aquaculture pond in Liaodong Bay using environmental DNA metabarcoding technique
- 生态学报. 2022, 42(13): 5303-5313
- Acta Ecologica Sinica. 2022, 42(13): 5303-5313
- http://dx.doi.org/10.5846/stxb202102170448
-
文章历史
- 收稿日期: 2021-02-17
- 网络出版日期: 2022-03-18
水母是一种海洋胶质类浮游动物, 是海洋生态系统中的重要组成部分, 在海洋生态系统物质循环和能量传递过程中具有重要作用[1-3]。另一方面, 水母是海洋生态系统中的重要捕食者, 其食物组成以浮游动物为主, 也能捕食鱼卵、仔稚鱼以及各种动物的浮游幼体等, 水母的过量繁殖或暴发往往对海洋渔业造成巨大负面影响并破坏海洋生态系统平衡[4-6]。随着全球化进程的加快和经济的快速发展, 环境污染、气候变化及过度捕捞等因素使海洋生态系统发生了有利于水母类的变化, 全球多处海域发生水母暴发的现象日益增多, 引发一系列的经济和社会问题[7-13];再加上更加频繁的航运、海洋工程建设的增多等有利于水母的扩散和定殖, 使水母成为海洋生态系统中易发生生物入侵的重要种类, 给海洋生态系统的健康和稳定带来威胁。
针对水母暴发引起的一系列问题, 开发快速、便捷的水母种类调查和检测技术对查清在自然海域中水母暴发的种类、监测及预警具有重要意义, 也是了解不同海域水母种类生物入侵的前提。然而, 水母大多具有世代交替的生活史, 水螅体世代营固着生活, 水母体世代营浮游生活, 由于水母复杂的生活史以及不同时期形态变异较大等原因, 再加上很多水母水螅体阶段个体微小以及不同种属间水螅型及水母型形态差异大, 且样品保存过程中易碎、易变形致使形态学特征容易丢失, 依据传统方法对水母进行分类鉴定和检测往往耗时长、成本高, 且较为繁琐和困难, 阻碍了对其进行早期监测和预警。鉴于水母种类鉴定及检测在水母暴发及防治中的重要性, 已有学者利用DNA条形码标记如线粒体分子标记COⅠ、16S[14-15]和核基因分子标记如18S、ITS等开展了水母种类的DNA条形码分析以及一些灾害水母种类如白斑水母(Phyllorhiza spp.)、金黄水母(Chrysaora sp.)、海月水母(Aurelia sp.)和白色霞水母(Cyanea nozakii)等[16-19]的种类鉴定与检测工作并取得较好的效果, 证明了DNA条形码技术在水母种类鉴定中的应用潜能。但前期的研究工作大多采用传统测序技术, 受限于当时技术手段的局限性, 往往只能对单一水母种类进行检测或鉴定, 且操作步骤繁琐、工作量大, 限制了分子DNA条形码技术的进一步应用。
建立针对水母种类的快速有效的检测技术, 既是对水母进行早期监测、预警的需要, 也可为水母类的生物多样性调查、生物入侵等提供基础数据。基于高通量测序技术获得生物特异性基因识别DNA条形码序列的扩增子测序方法, 称为DNA宏条形码技术。近年新兴的环境DNA宏条形码(Environmental DNA metabarcoding)技术能够通过直接提取环境样品(如水、土壤等)中的DNA, 利用针对目标类群的通用引物扩增后再进行高通量测序, 进而对取样环境中存在的目标物种进行识别。整个过程无需采集目标生物, 具有简便、快速、检测灵敏度高和信息量大等特点, 弥补了传统形态学监测的不足, 目前已开始应用于生物多样性评估及检测、生物入侵、动物食性分析等方面的研究[20-25], 并展现出较大的应用潜能。
利用环境DNA宏条形码技术对水生生物进行监测已成为目前的研究热点, 并在鱼类、海洋哺乳类、底栖生物、贝类、浮游动、植物等[24-31]生物类群的调查监测中进行了应用并取得较好的效果, 但目前尚无针对水母类的相关报道。营口现代渔业科技产业园海蜇养殖池塘为辽东湾近岸围海养殖区, 其前身为营口盐业有限责任公司为制盐修建的人工纳潮构筑物, 近似长方形, 面积约9000hm2, 通过自然纳潮进行海蜇围海养殖, 是较具代表性的辽东湾典型围海养殖池塘。其位置位于辽东湾东北近岸大清河入海口附近, 由于濒临河口区, 且离港口、码头距离较近, 各种水母种类丰富, 而且修建的堤坝等人工建筑可以为各种水母的定殖提供良好的栖息环境, 很多水母种类的水螅体可以很好地附着底栖生活, 往往能够引起水母种类的富集, 使其成为研究水母种类物种多样性和检测的合适材料。因此, 本研究首次运用环境DNA宏条形码技术对这一典型围海养殖池塘水母种类多样性进行调查评价, 不但能进一步加深对我国北方海域水母种类及暴发的了解, 并且可以验证环境DNA宏条形码技术在自然海域水母监测应用中的可行性, 为水母类的物种鉴定、监测以及早期预警等提供新的技术手段。
1 材料与方法 1.1 水样采集本次采样于2019年8月28日进行, 分别选取营口现代科技产业园围海养殖池塘沿岸7个采样点, 各采样点定位见图 1。每个采样点使用容量为1L的取水器采集表层及底层水样, 每个采样点在同一位置的同一水深进行多次采样, 以增加物种的检出率, 然后混合保存至已消毒的5L塑料桶中。为了提高目标种的检出率, 所有样品均在24h内使用0.45μm(MCE; Whatman公司)和3μm混合纤维素滤膜进行真空抽滤, 每个滤膜滤水量为2.5L。为评估是否存在外源DNA污染, 每次过滤设置1个阴性对照。在每份样品过滤后, 滤膜立即冷冻保存, 并对滤膜接触面进行消毒和冲洗, 防止样品间的交叉污染。冷冻滤膜置低温保温箱保存带回实验室备用。
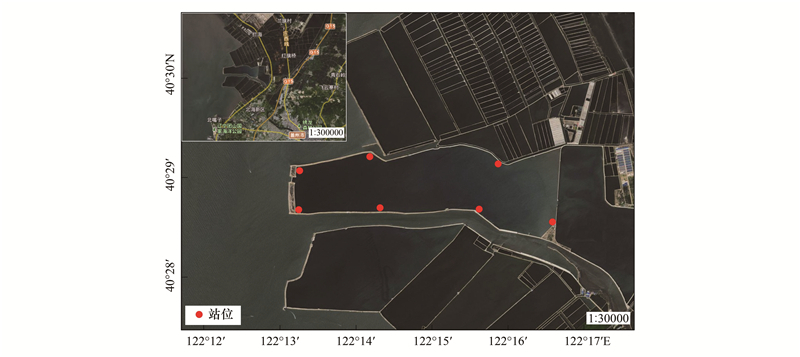
|
| 图 1 水样采样点示意图 Fig. 1 Information of sampling locations |
采用CTAB法提取滤膜DNA, 并使用空白滤膜设置为阴性对照。样品总DNA提取后, 使用DNeasy tissue kit (Qiagen, Valencia, CA)纯化试剂盒对DNA样品进行纯化, 纯化后的eDNA样品于-20℃冰箱保存备用。分别使用带Barcode特异引物的18S V4区528F-706R、线粒体COⅠ通用引物mlCOIintF-jgHCO2198对环境DNA模板进行PCR扩增。引物序列分别为: 528F: 5′-GCCTCCCTCGCGCCATCAGGCGGTAATTCCAGCTCCAA-3′, 706R: 5′-GCCTTGCCAGCCCGATCAGAATCCRAGAATTTCACCTCT-3′;mlCOIintF: 5′- GGWACWGGWTGA ACWGTWTAYCCYCC-3′, jgHCO2198: 5′-TAIACYTCIGGRTGICCRAARAAYCA-3′[20]。反应体系25μL, 包括:0.2mmol/L每种dNTPs, 0.2μmol/L每种引物, 1μLDNA模板, 1U Taq, 2.0mmol/L MgCl2, 2.5μL 10×缓冲液, 灭菌超纯水补足剩余体系。在Bio-rad T100梯度PCR仪上进行PCR扩增, 反应程序:98℃预变性1 min, 98℃变性10s, 50-55℃退火30 s, 72℃延伸30s, 30个循环;最后72℃保温5min。每次PCR反应使用ddH2O为模板作PCR阴性对照。每份样品重复扩增2次, 将同一样品的PCR产物混合后, 使用2%的琼脂凝胶电泳检测。所有阴性对照均无目的条带, 表明从采样、过滤、DNA提取至PCR扩增均无外源DNA污染。使用ThermoScientific公司GeneJET纯化PCR产物, 根据PCR产物浓度进行等质量混样操作, 充分混匀后用2%的琼脂糖凝胶电泳纯化PCR产物, 18S V4区选择主带大小在400-450bp的序列, 割胶回收目标条带;线粒体COⅠ片段选择主带大小在300-350bp的序列, 割胶回收目标条带。使用Illumina公司Truseq DNA PCR-Free Library Preparation Kit建库试剂盒进行文库的构建, 构建好的文库经Qubit定量和文库检测, 每个样品的PCR产物单独建库, 然后使用Illumina NovaSeq 6000测序平台进行第二代高通量测序(天津诺禾致源生物信息科技有限公司)。
1.3 数据分析各样品的原始测序reads用OBITools1.01.22程序包(http://metabarcoding.org/obitools/doc)经质控、过滤、拼接后得到有效数据, 建立参考数据库、序列比对及分类等步骤, 基于有效数据以97%的一致性进行OTUs (Operational taxonomic units)聚类和物种分类分析。鉴于当某一序列在数据库中最佳匹配序列多于1条时, OBITools程序会自动将物种鉴定结果标记为能够涵盖所有匹配物种的最低分类单元, 但不能加入研究地物种分布信息的参考[32], 因此分别将所得刺胞动物门(不包括珊瑚虫纲)物种18S rDNA和线粒体COⅠ的OTU代表序列经BLAST程序再次检索后在GeneBank上找到对应的物种。由于条形码分辨率及数据库的局限, 不是每条序列的识别都能达到物种水平, 因此将水母类OTU代表序列作为分子可操作分类单元(MOTU)来对不同水母类群进行区分。序列的物种鉴定原则如下:
(1) 18S rDNA序列以在相同的基因序列区域内, 当覆盖度(coverage)不低于95%且一致度达到99.5%及以上时, 判定样品来自匹配序列对应的物种。若存在不止一种物种的匹配序列, 则结合水母的分布特征及可能的同种异名等综合判断排除不符合的物种, 并推测可能的近缘物种或认为物种鉴定失败;最高一致度 < 99.5%且≥95%, 一致度最高序列仅有1条, 且与得分次之的序列一致度差距≥2%时, 记为一致度最高序列的上一级分类单元, 最高一致度对应有多个物种时, 则记为能够涵盖所有一致度最高的当地物种的最低分类单元。
(2) COⅠ基因序列在相同的基因序列区域内, 当覆盖度不低于95%、与单一物种比对的一致度≥99%, 且符合物种分布记录, 记为该物种;如发生与单一物种比对一致度≥99%但该物种非当地物种时, 则记为该物种在当地有分布的最近缘物种或认为物种鉴定失败;由于COI基因在不同种间差异较大[14-15], 最高一致度 < 99%且≥85%, 一致度最高序列仅有1条, 且与得分次之的序列一致度差距≥5%时, 记为一致度最高序列的上一级分类单元, 最高一致度对应有多个物种时, 则记为能够涵盖所有一致度最高的当地物种的最低分类单元。
2 结果与分析 2.1 基于18S rDNA宏条形码的水母种类组成除1个采样点因3μm混合纤维素滤膜丢失未进行检测外其余所有13个样品均获得可检测的PCR产物。7个采样点基于18S rDNA宏条形码标记检测出的水母物种种类序列信息见表 1, 物种鉴定信息见表 2。7个采样点13个滤膜样品共检测出水母种类8种, 包括2种钵水母纲大型水母海蜇(Rhopilema esculentum)、白色霞水母;1种筐水母亚纲水母两手筐水母(Solmundella bitentaculata), 1种未鉴定到种的硬水母亚纲棍手水母科水母, 1种花水母亚纲高手水母科鳞茎高手水母(Bougainvillia muscus)、2种软水母亚纲水母黑疣真瘤水母(Eutima krampi)和曲膝薮枝螅(Obelia geniculata), 以及1种未鉴定到种的管水母亚纲盛装水母科水母。其中仅养殖水母种类海蜇在各采样点展现出相对较高的序列丰度, 其余水母种类序列丰度较低(表 1)。
| 纲 Class |
科 Family |
种名 Species |
A | B | C | D | E | F | G | ||||||||||||
| 0.45 | 3 | 0.45 | 3 | 0.45 | 3 | 0.45 | 3 | 0.45 | 3 | 0.45 | 0.45 | 3 | |||||||||
| 钵水母纲 Scyphomedusae |
根口水母科 | 海蜇 Rhopilema esculentum | 1815 | 799 | 804 | 306 | 26 | 31 | 25 | 50 | 20 | 1 | 38 | 597 | 20565 | ||||||
| 霞水母科, | 白色霞水母 Cyanea nozakii | 11 | 3 | 4 | 2 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 104 | |||||||
| 筐水母亚纲 Narcomedusae |
间囊水母科 | 两手筐水母 Solmundella bitentaculata |
3 | 2 | 3 | 8 | 30 | 1 | 59 | 5 | 0 | 1 | 7 | 0 | 4 | ||||||
| 硬水母亚纲 Trachymedusae |
棍手水母科 | 未知 | 0 | 0 | 1 | 0 | 1 | 0 | 6 | 0 | 0 | 0 | 2 | 0 | 0 | ||||||
| 花水母亚纲 Anthomedusae |
高手水母科 | 鳞茎高手水母 Bougainvillia muscus |
1 | 0 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | ||||||
| 软水母亚纲 Leptomedusae |
和平水母科 | 黑疣真瘤水母 Eutima krampi | 3 | 2 | 2 | 4 | 1 | 2 | 0 | 66 | 2 | 0 | 3 | 0 | 0 | ||||||
| 钟螅水母科 | 曲膝薮枝螅 Obelia geniculata | 1 | 0 | 0 | 0 | 1 | 1 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | |||||||
| 管水母亚纲 Siphonophorae |
盛装水母科 | 未知 | 1 | 0 | 3 | 1 | 4 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | ||||||
| 水母最低分类阶元 Lowest taxon unit of Jellyfish |
GenBank最佳匹配物种 Best match species in GenBank |
||
| 物种拉丁名 Scientific name |
最高一致度 Best identity (%) |
序列号 Accession no. |
|
| 海蜇 Rhopilema esculentum |
Rhopilema esculentum | 100% | JX845352.1 |
| 白色霞水母 Cyanea nozakii |
Cyanea nozakii | 100% | KT445894.1 |
| 两手筐水母 Solmundella bitentaculata |
Solmundella bitentaculata|Aeginopsis laurentii | 100%|100% | EU247812.1 KY007604.1| MK968443.1 |
| 棍手水母科 Rhopalonematidae |
Sminthea arctica |Aglantha digitale | 97.71%|97.71% | MG979354.1 |MG979364.1 |
| 鳞茎高手水母 Bougainvillia muscus |
Bougainvillia muscus| Pachycordyle michaeli| Bougainvillia sp. | 100%|100%|100% | KT722388.1| MG788990.1| KF962203.1 |
| 黑疣真瘤水母 Eutima krampi |
Eutima krampi | 100% | FJ418675.1 |
| 曲膝薮枝螅 Obelia geniculata |
Obelia geniculata | 100% | KX665385.1 |
| 盛装水母科 Agalmidae |
Nanomia bijuga| Athorybia rosacea| Agalma elegans| Halistemma rubrum | 99.67%|99.67%|99.67%|99.67% | AF358071.1| AY937316.1 | AY937313.1|AY937323.1 |
7个采样点基于COⅠ宏条形码标记检测出的水母物种种类序列信息见表 3, 物种鉴定信息见表 4。虽然COⅠ基因在水母种间的遗传变异较大, 如基于28种水螅水母的线粒体COⅠ遗传标记进行的研究表明, 同属不同种间遗传距离为0.092-0.215(平均0.171), 同科不同属间遗传距离为0.127-0.351(平均0.189), 但其种内差异并不大, 遗传距离为0-0.033(平均0.008), COⅠ条形码标记在水母类群中有明显的条形码间隔, 同属种间的COⅠ变异速率约是同种个体间的30倍[15]。因此本研究在进行水母物种判别时参考这一标准, 按序列一致度≥99%归为同种, 序列一致度 < 99%且≥85%归为同属或同科。7个采样点检测出至少19种水母种类, 其中10种鉴定到种, 包括钵水母纲4种海蜇、沙蜇(Nemopilema nomurai)、白色霞水母、海月水母, 水螅水母6种, 包括软水母亚纲钟螅水母科半球美螅水母(Clytia hemisphaerica)、美螅水母(Clytia gracilis)、双叉薮枝螅(Obelia dichotoma)、和平水母科锡兰和平水母(Eirene ceylonensis)、塔形和平水母(Eirene pyramidalis)和短柄和平水母(Eirene brevistylus);9种仅鉴定到属或科, 包括钵水母纲游水母科1种(Pelagia sp.), 花水母亚纲棍螅水母科萨氏水母属1种(Sarsia sp.), 软水母亚纲钟螅水母科美螅水母属2种(Clytia sp.)、薮枝螅属2种(Obelia sp.)、钟螅水母科1种(Bonneviella sp.), 感棒水母科1种(Laodicea sp.)和羽螅水母科1种(Aglaophenia sp.)。此外, 由于GeneBank数据库的缺乏还存在一些OUT代表序列仅能鉴定到科及以上分类阶元(< 85%且≥80%) 的水母种类, 如花水母亚纲棍螅水母科、Ptilocodiidae科、软水母亚纲钟螅水母科、高手水母科以及管水母亚纲盛装水母科、离翼水母科种类等, 实际鉴定出的水母种类数可能更多(表 4)。同18S rDNA结果一致, 海蜇在各采样点均展现出较高的序列丰度, 此外渤海常见小型水母种类如萨氏水母属、美螅水母属、薮枝螅等在各采样点展现出的序列丰度也较为丰富(表 3)。
| 纲 Class |
科 Family |
种名 Species |
A | B | C | D | E | F | G | ||||||||||||
| 0.45 | 3 | 0.45 | 3 | 0.45 | 3 | 0.45 | 3 | 0.45 | 3 | 0.45 | 0.45 | 3 | |||||||||
| 钵水母纲 | 根口水母科 | 海蜇 Rhopilema esculentum | 3180 | 1635 | 1272 | 883 | 168 | 151 | 31 | 98 | 413 | 28 | 142 | 13151 | 2448 | ||||||
| Scyphomedusae | 霞水母科 | 白色霞水母 Cyanea nozakii | 8 | 0 | 1 | 1 | 0 | 0 | 3 | 0 | 23 | 0 | 1 | 2 | 6 | ||||||
| 根口水母科 | 沙蜇 Nemopilema nomurai | 0 | 1 | 1 | 2 | 0 | 1 | 0 | 3 | 19 | 0 | 3 | 0 | 0 | |||||||
| 洋须水母科 | 海月水母 Aurelia coerulea | 0 | 0 | 15 | 9 | 0 | 6 | 0 | 0 | 0 | 7 | 0 | 0 | 0 | |||||||
| 游水母科 | Pelagia sp. | 0 | 1 | 17 | 13 | 3 | 0 | 8 | 4 | 0 | 8 | 1 | 0 | 6 | |||||||
| 花水母亚纲 | 棍螅水母科 | 萨氏水母属 | 2 | 2 | 200 | 1240 | 37 | 124 | 6 | 450 | 76 | 506 | 1 | 1 | 0 | ||||||
| Anthomedusae | 未知 | 0 | 4 | 93 | 279 | 31 | 32 | 0 | 83 | 6 | 113 | 7 | 0 | 5 | |||||||
| Ptilocodiidae | 未知 | 0 | 0 | 6 | 0 | 5 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | |||||||
| 软水母亚纲 | 钟螅水母科 | 半球美螅水母 Clytia hemisphaerica | 0 | 1 | 124 | 421 | 91 | 1 | 0 | 7 | 12 | 34 | 1 | 0 | 0 | ||||||
| Leptomedusae | Clytia gracilis | 26 | 252 | 48 | 129 | 0 | 24 | 4 | 24 | 3 | 100 | 0 | 2 | 9 | |||||||
| 美螅水母属 Clytia sp. | 2 | 0 | 3 | 5 | 8 | 0 | 0 | 5 | 0 | 0 | 1 | 0 | 0 | ||||||||
| 双叉薮枝螅水母 Obelia dichotoma | 19 | 12 | 52 | 21 | 49 | 4 | 64 | 8 | 5 | 9 | 89 | 2 | 1 | ||||||||
| 薮枝螅属 Obelia sp. | 0 | 1 | 0 | 0 | 0 | 143 | 1 | 0 | 14 | 56 | 14 | 0 | 0 | ||||||||
| 感棒水母科 | 未知 | 0 | 0 | 164 | 367 | 15 | 6 | 0 | 28 | 23 | 31 | 0 | 0 | 0 | |||||||
| 和平水母科 | Laodicea sp. | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 | 0 | 0 | 0 | |||||||
| 锡兰和平水母 Eirene ceylonensis | 0 | 1 | 0 | 0 | 0 | 11 | 0 | 311 | 2 | 0 | 0 | 0 | 0 | ||||||||
| 塔形和平水母 Eirene pyramidalis | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 5 | 0 | 12 | 0 | 0 | 0 | ||||||||
| 短柄和平水母 Eirene brevistylus | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 | 0 | 0 | 0 | 0 | 0 | ||||||||
| 羽螅科 | 未知 | 0 | 0 | 0 | 7 | 0 | 0 | 0 | 1 | 0 | 2 | 0 | 0 | 0 | |||||||
| 管水母亚纲 | 盛装水母科 | 未知 | 2 | 0 | 2 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ||||||
| Siphonophorae | 离翼水母科 | 未知 | 0 | 0 | 14 | 6 | 0 | 0 | 35 | 2 | 2 | 9 | 0 | 0 | 0 | ||||||
| 水母最低分类阶元 Lowest taxon unit of Jellyfish |
GenBank最佳匹配物种 Best match species in GenBank | ||
| 物种拉丁名 Scientific name |
最高一致度 Best identity/% |
序列号 Accession no. |
|
| 海蜇 Rhopilema esculentum | Rhopilema esculentum | 100% | JX845352.1 |
| 白色霞水母 Cyanea nozakii | Cyanea nozakii | 99% | JX845350.1 |
| 沙蜇 Nemopilema nomurai | Nemopilema nomurai | 99% | HF930549.1 |
| 海月水母 Aurelia coerulea | Aurelia coerulea | 100% | KX691598.1 |
| 游水母科 Pelagiidae | Pelagia sp. | 86% | KY655788.1 |
| 萨氏水母属 Sarsia | Sarsia tubulosa | 89% | KC440087.1 |
| 棍螅水母科 Corynidae | Corynidae sp. | 84% | MG421474.1 |
| Ptilocodiidae | Thecocodium quadratum | 85% | KT981908.1 |
| 筒螅科 Tubularidae | Ectopleura viridis | 83% | MG811649.1 |
| 半球美螅水母 Clytia hemisphaerica | Clytia hemisphaerica | 100% | KX665199.1 |
| 美螅水母 Clytia gracilis | Clytia gracilis | 99% | KX665195.1 |
| 美螅水母属 Clytia | Clytia folleata | 85% | KF962082.1 |
| 美螅水母属 Clytia | Clytia sp. | 85% | KF962101.1 |
| 双叉薮枝螅 Obelia dichotoma | Obelia dichotoma | 99% | KX665256.1 |
| 曲膝薮枝螅 Obelia geniculata | Obelia geniculata | 95% | KX665162.1 |
| 薮枝螅属 Obelia | Obelia sp. | 85% | MG421474.1 |
| 薮枝螅属 Obelia | Obelia sp.3 | 86% | KC440003.1 |
| 钟螅水母科 Campanulariidae | Bonneviella sp.4 | 85% | AY789893.1 |
| 钟螅水母科 Campanulariidae | Eucheilota maculata | 84% | KC440070.1 |
| 感棒水母科 Laodiceidae | Laodicea sp. | 90% | MW278609.1 |
| 锡兰和平水母 Eirene ceylonensis | Eirene ceylonensis | 99% | HM053525.1 |
| 塔形和平水母 Eirene pyramidalis | Eirene pyramidalis | 100% | JQ716144.1 |
| 短柄和平水母 Eirene brevistylus | Eirene brevistylus | 99% | KF962118.1 |
| 羽螅科 Plumulariidae | Aglaophenia struthionides | 85% | MH282629.1 |
| 高手水母科 Bougainvillidae | Nemopsis bachei | 82% | MK308284.1 |
| 盛装水母科 Agalmidae | Agalma elegans | 82% | GQ119942.1 |
| 离翼水母科 Apolemiidae | Apolemia sp. | 84% | GQ119954.1 |
水母是刺胞动物门在浮游生物中的主要代表, 也是海洋浮游生物的主要类群之一, 除终生浮游种类(如自育水母纲、管水母亚纲)之外, 大多数水母具有世代交替的生活史[3]。对大多数水母而言, 由于对整个生活史缺乏了解, 且其形态性状易随发育阶段或生境的改变而产生较大变化, 因此对水母尤其是小型水母的物种鉴定一直是分类学工作的难点[15]。常规形态学鉴定步骤繁琐、专业性强, 易造成同种异名或异种同名等错误, 必须由有经验的专业人员借助显微镜才能进行准确区分。再加上一些水母种类存在隐种(cryptic species)现象[33-34], 形态多样性可能无法完全反映其真正的物种多样性水平。上述因素导致水母尤其是小型水母调查难以量化和监测, 与其它海洋生物类群相比水母生态学调查基础数据缺乏, 水母已成为海洋生态调查中的“短板”之一。与此同时, 近年水母暴发现象日益增多, 由于隐蔽性强、所造成的灾害易扩散、治理手段缺乏等原因, 对海洋生态系统具有很强的破坏性, 正严重威胁着海洋生态系统的健康和稳定[4-11], 因此亟需建立高效、快速、准确的水母监测方法。
环境DNA宏条形码技术是近几年发展起来的新一代DNA条形码鉴定技术, 具有高通量、成本低、检测灵敏度高等优点, 在水生动物的物种鉴定、生物多样性检测及外来入侵物种监测等方面具有明显优势。与传统调查方法相比eDNA宏条形码技术不仅可以节省调查作业和物种鉴定所需的时间、费用等, 而且对水生生物无附带伤害、具有更高的检测成功率, eDNA宏条形码技术有望成为集高效、精准和标准化为一体的新一代生物多样性监测工具, 具有对环境样品中生物物种进行有效监测的潜力[22-25, 35-37], 这在本文对水母类进行的研究中也得到了佐证。本研究使用两种DNA宏条形码标记对辽东湾东北部围海养殖池塘水母种类进行物种鉴定, 基于18S rDNA分子标记鉴定出2纲8种水母种类, 而基于线粒体COⅠ标记则鉴定出2纲19种水母种类, 所鉴定出的水母种类不仅涵盖了辽东湾常见水母种类如钵水母纲海蜇、沙蜇、白色霞水母、海月水母, 水螅水母总纲半球美螅水母、美螅水母、双叉薮枝螅、曲膝薮枝螅、锡兰和平水母、塔形和平水母、短柄和平水母;还包括一些传统调查中的少见及偶见水母种类如筐水母亚纲、硬水母亚纲、管水母亚纲、花水母亚纲以及软水母亚纲种类[38-40], 研究结果表明环境DNA宏条形码技术可以有效应用于水母种类的物种监测。
3.2 DNA条形码标记的选择及其对水母物种识别能力的比较目前很多学者采用基于DNA条形码的分子生物学分类方法作为水母类物种鉴定的必要补充[14-15, 33-34, 41]。如程方平等[14]对中国北方海域习见水母种类DNA条形码进行了分析, Laakmann和Holst[41]在大西洋北海水螅水母鉴定以及张铛妮等[15]对中国南海北部湾海域水螅水母DNA条形码进行的分析工作均证实了DNA条形码标记在疑难水母种类鉴定中的作用。一般而言, 不同分子标记基因的适用范围并不完全一致, 不同的分子标记对不同物种的识别能力不同从而影响物种鉴定的准确性, 因此需根据待检测生物类群以及研究目的综合选择合适的分子标记[24-25, 42]。核糖体18S rRNA基因和线粒体细胞色素C氧化酶亚基I (COⅠ)基因是应用最广泛的两种真核生物分子标记基因, 18S rDNA序列是适用于进化较慢物种的种类鉴定和系统分析的合适分子标记, 但由于其基因序列较为保守, 在解决较小分类阶元如属的种间关系尤其是在进行某些近缘种的物种鉴定时具有较大的局限性, 适用于研究亲缘关系较远的生物[43]。与之相反, COⅠ基因具有进化速度快和功能保守性强的特点, 具有较高的物种识别能力, 常用来分析浮游动物、甲壳动物、微型后生动物等[20, 44-46]。
本研究中, 基于18S V4区分子标记的宏条形码仅鉴定出2纲8种水母种类, 而以COⅠ为标记的宏条形码鉴定出2纲19种水母种类, 表明基于COⅠ宏条形码的eDNA技术的水母检出率明显高于基于18S rDNA宏条形码的eDNA技术。这与Fernández等[47]将室内群落模拟实验、基于eDNA技术和基于传统监测方法的野外实验对河流内大型无脊椎动物监测进行的模拟实验结果一致, 其结果也表明基于COⅠ宏条形码的eDNA技术的物种检出率明显高于传统监测方法和基于18S rDNA宏条形码的eDNA技术。因此在监测不同的生物类群时, 标记基因的恰当选择对eDNA技术在生物多样性监测中的应用具有重要作用, 对水母这一生物类群而言, COⅠ宏条形码标记可能具有更高的物种识别能力。此外, 利用两种分子标记对水母物种的识别结果并不完全一致, 两者共同检测出的水母种类仅有3种, 这一方面可能归因于两种分子标记的识别能力不一致所导致, 另一方面水母DNA尤其是低丰度种类在水体中具有复杂的存在机制, 其产生、降解及移动过程受多种环境因素的影响, 不同的采样方案和引物选择也可导致不同的检测结果[48-49]。因此, 在利用环境DNA宏条形码技术对水母类进行物种鉴定时, 联合采用多个分子标记可能得出更为全面准确的结论。
3.3 环境DNA宏条形码技术的优势与不足及其在水母监测中的应用潜力关于环境DNA宏条形码技术的优势与不足, 目前已有多篇文献进行了论述[22-25], 如相比传统监测方法, 环境DNA宏条形码技术具有非破坏性采样;便捷高效、耗时少、成本低;灵敏度高、快速和高效检测群落多样性等优势;但也存在一些不足, 如针对不同生物类群尚无通用的标准化操作流程;时间和空间的不一致性;种类鉴定高度依赖分子数据库的完整性;只能进行定性分析无法进行定量分析以及可能存在的污染问题等, 此处不再进行详细赘述。虽然本研究未利用传统调查方法对围海养殖池塘的水母种类进行调查, 但基于传统监测[38-40]结果显示, 大型水母种类如海月水母、海蜇、沙蜇、白色霞水母以及小型水母种类如和平水母属、薮枝螅水母、美螅水母属等为辽东湾及渤海海域常见水母种类。本研究基于2种DNA宏条形码标记共鉴定出2纲22种水母种类, 不仅包括海蜇、沙蜇、白色霞水母、海月水母和小型水母种类如美螅水母属、和平水母属等常见种类, 而且还检测出已报道的多种黄渤海常见水母种类如薮枝螅水母、黑疣真瘤水母、鳞茎高手水母、两手筐水母等[39-40]。此外, 由于数据库中水母种类的局限性, 还存在一些仅能鉴定到科及以上分类阶元的水母种类, 实际鉴定出的水母种类数量可能更多, 证实了环境DNA宏条形码技术在水母种类物种鉴定和监测中的优势及可行性。
与此同时, 本研究利用环境DNA宏条形码技术对水母进行的分析也存在上述不足, 如由于分子数据库的缺乏导致一些水母种类无法鉴定;同一采样点的样品利用不同滤膜进行扩增的种类鉴定结果不一致;只能进行定性分析无法进行定量分析等。此外, 目前的环境DNA宏条形码技术还存在一些技术层面无法解决的问题, 如引物选择、PCR扩增偏向性、测序误差等。但结合了新一代高通量测序技术的eDNA宏条形码方法可以在短时间内对水环境样品中的水母物种进行识别和检测, 避免了传统方法受样品条件、操作者实践经验和专业知识等限制的不利影响, 具有快速和高效检测水母多样性的潜力, 可以作为一种重要的补充工具用于水母的快速调查、监测及预警。
由此可见, 环境DNA宏条形码技术具有高通量、高灵敏度、高分辨率的技术优势, 在解决包括水母类在内的水生生物种类鉴定和生物群落多样性监测等方面尚有较大的应用潜能, 随着物种分子数据库的不断完善和测序技术的进一步发展, 在解决了环境DNA宏条形码技术定量分析准确性方面的短板之后, 环境DNA宏条形码技术有望成为物种识别和多样性分析、生物入侵以及生态环境监测等领域的常规分析手段。
| [1] |
张芳, 孙松, 李超伦. 海洋水母类生态学研究进展. 自然科学进展, 2009, 19(2): 121-130. DOI:10.3321/j.issn:1002-008X.2009.02.001 |
| [2] |
马喜平, 高尚武. 渤海水母类生态的初步研究--种类组成、数量分布与季节变化. 生态学报, 2000, 20(4): 533-540. DOI:10.3321/j.issn:1000-0933.2000.04.001 |
| [3] |
许振祖, 黄加祺, 林茂, 郭东晖, 王春光. 中国刺胞动物门水螅虫总纲. 北京: 海洋出版社, 2014.
|
| [4] |
Uye S, Ueta U. Recent increase of jellyfish populations and their nuisance to fisheries in the Inland Sea of Japan. Bulletin of the Japanese Society of Fisheries Oceanography, 2004, 68(1): 9-19. |
| [5] |
Hay S. Marine ecology: gelatinous bells may ring change in marine ecosystems. Current Biology, 2006, 16(17): R679-R682. DOI:10.1016/j.cub.2006.08.010 |
| [6] |
Purcell J E, Uye S I, Lo W T. Anthropogenic causes of jellyfish blooms and their direct consequences for humans: a review. Marine Ecology Progress Series, 2007, 350: 153-174. DOI:10.3354/meps07093 |
| [7] |
孙松. 水母暴发研究所面临的挑战. 地球科学进展, 2012, 27(3): 257-261. |
| [8] |
Sun S, Sun X X, Jenkinson I R. Preface: giant jellyfish blooms in Chinese waters. Hydrobiologia, 2015, 754(1): 1-11. DOI:10.1007/s10750-015-2320-3 |
| [9] |
Uye S I. Human forcing of the copepod-fish-jellyfish triangular trophic relationship. Hydrobiologia, 2011, 666(1): 71-83. DOI:10.1007/s10750-010-0208-9 |
| [10] |
Pauly D, Graham W, Libralato S, Morissette L, Palomares M L D. Jellyfish in ecosystems, online databases, and ecosystem models. Hydrobiologia, 2009, 616(1): 67-85. DOI:10.1007/s10750-008-9583-x |
| [11] |
Frolova A, Miglietta M P. Insights on bloom forming jellyfish (Class: Scyphozoa) in the gulf of Mexico: environmental tolerance ranges and limits suggest differences in habitat preference and resistance to climate change among congeners. Frontiers in Marine Science, 2020, 7: 93. DOI:10.3389/fmars.2020.00093 |
| [12] |
Suzuki K S, Suzuki K W, Kumakura E, Sato K, Oe Y, Sato T, Sawada H, Masuda R, Nogata Y. Seasonal alternation of the ontogenetic development of the moon jellyfish Aurelia coerulea in Maizuru Bay, Japan. PLoS One, 2019, 14(11): e0225513. DOI:10.1371/journal.pone.0225513 |
| [13] |
Dong J, Wang B, Duan Y, Yoon W D, Wang A Y, Liu X Z, Li Y L, Sun M, Chai Y. Initial occurrence, ontogenic distribution-shifts and advection of Nemopilema nomurai (Scyphozoa: Rhizostomeae) in Liaodong Bay, China, from 2005-2015. Marine Ecology Progress Series, 2018, 591: 185-197. DOI:10.3354/meps12272 |
| [14] |
程方平, 王敏晓, 王彦涛, 张芳, 李超伦, 孙松. 中国北方习见水母类的DNA条形码分析. 海洋与湖沼, 2012, 43(3): 451-459. |
| [15] |
张珰妮, 郑连明, 何劲儒, 张文静, 林元烧, 李阳. 基于线粒体COⅠ和16S片段序列的北部湾北部水螅水母DNA条形码分析. 生物多样性, 2015, 23(1): 50-60. |
| [16] |
Bayha K M, Graham W M. A new Taqman PCR-based method for the detection and identification of scyphozoan jellyfish polyps. Hydrobiologia, 2009, 616(1): 217-228. DOI:10.1007/s10750-008-9590-y |
| [17] |
Ki J S, Hwang D S, Lee J S. Simultaneous detection of Aurelia and Chrysaora scyphozoan jellyfish on a DNA microarray. Journal of the Marine Biological Association of the United Kingdom, 2010, 90(6): 1111-1117. DOI:10.1017/S0025315409990373 |
| [18] |
王建艳, 甄毓, 王国善, 米铁柱, 于志刚. 基于mt-16S rDNA和mt-COⅠ基因的海月水母分子生物学鉴定方法和检测技术. 应用生态学报, 2013, 24(3): 847-852. |
| [19] |
李玉龙, 董婧, 王彬, 孙明, 王爱勇, 王文波. 基于18S和ITS-5.8S rDNA基因序列的白色霞水母(Cyanea nozakii)的分子鉴定与检测. 海洋与湖沼, 2016, 47(1): 158-165. |
| [20] |
Leray M, Yang J Y, Meyer C P, Mills S C, Agudelo N, Ranwez V, Boehm J T, Machida R J. A new versatile primer set targeting a short fragment of the mitochondrial COⅠ region for metabarcoding metazoan diversity: application for characterizing coral reef fish gut contents. Frontiers in Zoology, 2013, 10(1): 34. DOI:10.1186/1742-9994-10-34 |
| [21] |
Egan S P, Grey E, Olds B, Feder J L, Ruggiero S T, Tanner C E, Lodge D M. Rapid molecular detection of invasive species in ballast and harbor water by integrating environmental DNA and light transmission spectroscopy. Environmental Science & Technology, 2015, 49(7): 4113-4121. |
| [22] |
赵明, 赵梦迪, 马春艳, 张凤英, 蒋科技, 王田, 马凌波. 环境DNA在水域生态中的研究进展. 中国水产科学, 2018, 25(4): 714-720. |
| [23] |
单秀娟, 李苗, 王伟继. 环境DNA(eDNA)技术在水生生态系统中的应用研究进展. 渔业科学进展, 2018, 39(3): 23-29. |
| [24] |
李晗溪, 黄雪娜, 李世国, 战爱斌. 基于环境DNA-宏条形码技术的水生生态系统入侵生物的早期监测与预警. 生物多样性, 2019, 27(5): 21-34. |
| [25] |
李萌, 尉婷婷, 史博洋, 郝希阳, 徐海根, 孙红英. 环境DNA技术在淡水底栖大型无脊椎动物多样性监测中的应用. 生物多样性, 2019, 27(5): 480-490. |
| [26] |
舒璐, 林佳艳, 徐源, 曹特, 封吉猛, 彭作刚. 基于环境DNA宏条形码的洱海鱼类多样性研究. 水生生物学报, 2020, 44(5): 1080-1086. |
| [27] |
张宛宛, 谢玉为, 杨江华, 杨雅楠, 李娣, 张咏, 于红霞, 张效伟. DNA宏条形码(metabarcoding)技术在浮游植物群落监测研究中的应用. 生态毒理学报, 2017, 12(1): 15-24. |
| [28] |
孙晶莹, 杨江华, 张效伟. 环境DNA(eDNA)宏条形码技术对枝角类浮游动物物种鉴定及其生物量监测研究. 生态毒理学报, 2018, 13(5): 76-86. |
| [29] |
高养春, 李海涛, 王孝程, 孙艺, 战爱斌, Aileen TAN Shau-Hwai, 李宏俊. 利用宏DNA条形码研究浮游动物多样性-以鸭绿江口为例. 生态学报, 2020, 40(11): 3822-3832. |
| [30] |
宋伦, 吴景, 宋广军, 董少杰, 王志松. 基于环境DNA技术的辽东湾真核微藻群落结构特征. 生态学报, 2020, 40(17): 6243-6257. |
| [31] |
吴昀晟, 唐永凯, 李建林, 刘凯, 李红霞, 王钦, 俞菊华, 徐跑. 环境DNA在长江江豚监测中的应用. 中国水产科学, 2019, 26(1): 124-132. |
| [32] |
邵昕宁, 宋大昭, 黄巧雯, 李晟, 姚蒙. 基于粪便DNA及宏条形码技术的食肉动物快速调查及食性分析. 生物多样性, 2019, 27(5): 543-556. |
| [33] |
Lindner A, Govindarajan A F, Migotto A E. Cryptic species, life cycles, and the phylogeny of Clytia (Cnidaria: Hydrozoa: Campanulariidae). Zootaxa, 2011, 2980(1): 23-36. DOI:10.11646/zootaxa.2980.1.2 |
| [34] |
Zhou K L, Zheng L M, He J R, Lin Y S, Cao W Q, Zhang W J. Detection of a new Clytia species (Cnidaria: Hydrozoa: Campanulariidae) with DNA barcoding and life cycle analyses. Journal of the Marine Biological Association of the United Kingdom, 2013, 93(8): 2075-2088. DOI:10.1017/S0025315413000969 |
| [35] |
马克平. DNA条形码: 从物种到生物群区. 生物多样性, 2015, 23(3): 279-280. |
| [36] |
周欣. 运用基因组学方法探究生物多样性: 大数据时代的"胡克显微镜". 生物多样性, 2019, 27(5): 475-479. |
| [37] |
刘山林. DNA条形码参考数据集构建和序列分析相关的新兴技术. 生物多样性, 2019, 27(5): 526-533. |
| [38] |
蒋双, 陈介康. 黄渤海水螅水母、管水母和栉水母的地理分布. 海洋通报, 1994, 13(3): 17-23. |
| [39] |
孙松, 张芳, 李超伦, 杨波, 吉鹏. 黄海小型水母的分布特征. 海洋与湖沼, 2012, 43(3): 429-437. |
| [40] |
左涛, 吴强, 王俊, 李忠义. 莱州湾水母种类多样性及群集结构的季节变化. 生态学报, 2016, 36(18): 5646-5656. |
| [41] |
Laakmann S, Holst S. Emphasizing the diversity of North Sea hydromedusae by combined morphological and molecular methods. Journal of Plankton Research, 2014, 36(1): 64-76. DOI:10.1093/plankt/fbt078 |
| [42] |
高旭, 杨江华, 张效伟. 浮游动物DNA宏条形码标志基因比较研究. 生态毒理学报, 2020, 15(2): 61-70. |
| [43] |
Brannock P M, Ortmann A C, Moss A G, Halanych K M. Metabarcoding reveals environmental factors influencing spatio-temporal variation in pelagic micro-eukaryotes. Molecular Ecology, 2016, 25(15): 3593-3604. DOI:10.1111/mec.13709 |
| [44] |
Zhou X, Li Y Y, Liu S L, Yang Q, Su X, Zhou L L, Tang M, Fu R B, Li J G, Huang Q F. Ultra-deep sequencing enables high-fidelity recovery of biodiversity for bulk arthropod samples without PCR amplification. GigaScience, 2013, 2(1): 4. DOI:10.1186/2047-217X-2-4 |
| [45] |
Bourlat S J, Borja A, Gilbert J, Taylor M I, Davies N, Weisberg S B, Griffith J F, Lettieri T, Field D, Benzie J, Glöckner F O, Rodríguez-Ezpeleta N, Faith D P, Bean T P, Obst M. Genomics in marine monitoring: new opportunities for assessing marine health status. Marine pollution bulletin, 2013, 74(1): 19-31. DOI:10.1016/j.marpolbul.2013.05.042 |
| [46] |
Avó A P, Daniell T J, Neilson R, Oliveira S, Branco J, Adão H. DNA barcoding and morphological identification of benthic nematodes assemblages of estuarine intertidal sediments: advances in molecular tools for biodiversity assessment. Frontiers in Marine Science, 2017, 4: 66. |
| [47] |
Fernández S, Rodríguez S, Martínez J L, Borrell Y J, Ardura A, García-Vázquez E. Evaluating freshwater macroinvertebrates from eDNA metabarcoding: a river Nalón case study. PLoS One, 2018, 13(8): e0201741. DOI:10.1371/journal.pone.0201741 |
| [48] |
Barnes M A, Turner C R. The ecology of environmental DNA and implications for conservation genetics. Conservation Genetics, 2016, 17(1): 1-17. DOI:10.1007/s10592-015-0775-4 |
| [49] |
Deiner K, Walser J C, Mächler E, Altermatt F. Choice of capture and extraction methods affect detection of freshwater biodiversity from environmental DNA. Biological Conservation, 2015, 183: 53-63. DOI:10.1016/j.biocon.2014.11.018 |
 2022, Vol. 42
2022, Vol. 42