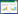文章信息
- 张可, 王林玲, 党晓群, 李旭东, 李月, 陆欢欢, 牛泽清, 袁峰, 朱朝东, 黄敦元
- ZHANG Ke, WANG Linling, DANG Xiaoqun, LI Xudong, LI Yue, LU Huanhuan, NIU Zeqing, YUAN Feng, ZHU Chaodong, HUANG Dunyuan
- 基于DNA宏条形码技术的蓝彩带蜂粉源植物多样性研究
- Analysis of species and diversities in the pollen plants of Nomia chalybeata (Hymenoptera: Halictidae) using DNA metabarcoding technique
- 生态学报. 2022, 42(11): 4504-4514
- Acta Ecologica Sinica. 2022, 42(11): 4504-4514
- http://dx.doi.org/10.5846/stxb202108102204
-
文章历史
- 收稿日期: 2021-08-10
- 网络出版日期: 2021-11-01
2. 中国科学院动物研究所动物进化与系统学院重点实验室, 北京 100101
2. Key Laboratory of Zoological Systematics and Evolution, Institute of Zoology, Chinese Academy of Sciences, Beijing 100101, China
昆虫授粉是一种重要的生态交互作用, 不仅涉及超过80%的被子植物繁殖还参与约75%的农作物生产过程[1-3]。近年来由于农业扩张、传粉昆虫栖息地的丧失和破碎、杀虫剂和除草剂的使用、微生物病原体感染等原因, 传粉者数量呈大幅降低趋势[4-6]。膜翅目蜜蜂总科作为生态系统重要的传粉类群, 主要依靠植物的花粉和花蜜作为食物来源, 其中花蜜主要提供碳水化合物, 花粉主要提供蛋白质和脂质[7], 因此, 蜜蜂在特定植物上采食花粉和花蜜以满足自身营养需求的同时帮助这些植物实现了授粉功能[8-11]。研究表明粉源植物是生态系统中重要的组成部分, 是蜜蜂等传粉者赖以生存的物质基础, 它们的数量和质量会影响蜜蜂物种多样性[12-13]。
野生蜜蜂与家养蜜蜂是相对而言的, 野生蜜蜂具有种类多、分布广、适应性强等特性, 诸多研究表明:特定生态系统中, 野生蜜蜂提供的授粉服务功能是家养蜜蜂无法取代的, 如在苹果、蓝莓、覆盆子、油茶等经济作物的授粉中, 野生蜜蜂扮演非常重要的角色[14-18]。Angelella[19]等人对美国中大西洋东岸和弗吉尼亚海滩地区21个农场的开花植物系统研究表明, 家养蜜蜂和野生蜜蜂之间存在粉源植物资源的竞争。该竞争不仅会抑制野生蜜蜂的数量, 而且会降低植物的座果率和结实率。尤其在开花植物资源有限的情况下, 这会增加家养蜜蜂和野生传粉者之间的生态位重叠, 并加剧对野生传粉者种群的负面影响。
野生蜜蜂类群是野生植物和农作物的重要传粉昆虫, 如壁蜂属多数物种因具有易管理、低成本和高传粉效率等特点, 所以常作为人工放养野生蜜蜂被广泛应用于苹果、樱桃、梨等果树的授粉[20];大分舌蜂(Colletes gigas)和油茶地蜂(Andrena camellia)等野生蜜蜂是湖南、江西等南方地区油茶优势传粉昆虫[21];白斑切叶蜂(Megachile strupigera)广泛分布于中国南方地区, 是野生植物及农林作物的重要传粉昆虫之一, 它的主要粉源植物为黄荆和山牡荆, 这对维持白斑切叶蜂种群的稳定具有重要作用[22]。野生蜜蜂的大部分服务和益处与食物、药物和授粉等有关[23], 但到目前为止对野生蜜蜂的研究多为形态学、线粒体基因、生活方式等方面, 对粉源植物研究甚少, 而野生蜜蜂群落的丰富度和多样性与授粉有关[24], 因此, 了解野生蜜蜂的主要粉源植物的种类组成及其分布情况对促进它们的种群繁衍有着重要意义。
DNA宏条形码技术是利用高通量测序手段快速获取混合样本中所有物种的混合条形码扩增序列, 并通过生物信息学分析手段来鉴定混合样本中物种组成及相对丰度的方法[25-26]。目前, 该方法在植物研究方面常用的序列有:核基因ITS/ITS2片段、叶绿体基因MatK、psbA-trnH、rbcL、trnL等以及不同序列组合[27]。相比传统的蜂蜜孢粉学(Melissopalynology), DNA宏条形码技术在花粉鉴定方面具有鉴定速度快、分类分辨率高等优势[27-29]。Jones[30]等人使用DNA宏条形码技术鉴定来自英格兰和威尔士441个意大利蜜蜂蜂蜜样本中的植物类群, 将2017年与1952年(蜂蜜孢粉学)的样本植物类群进行比较, 推断出蜜蜂主要觅食植物的变化是由植物在景观中的可用性变化所驱动的, 同时也证明了DNA宏条形码技术相比显微鉴定技术具有更高的植物分类分辨率。
蓝彩带蜂(Nomia chalybeata Smith)属隧蜂科(Halictidae)彩带蜂属(Nomia), 广泛分布于安徽、山东、江西、福建、广西等我国东部地区, 以及日本、韩国、印度、缅甸等国家[31-32]。该蜂是一种典型的广访花性(Polylectic)野生蜜蜂, 主要在腐木及土中筑巢, 巢室为一到多丛[33]。本研究基于蓝彩带蜂在国内的主要分布区, 在我国东部18个不同样地该蜂巢穴中获取20份蓝彩带蜂蜂粮样本, 利用DNA宏条形码技术研究该蜂粉源植物的多样性, 为全面掌握该蜂的粉源植物及保护策略提供理论依据。
1 材料和方法 1.1 样品采集根据蓝彩带蜂成虫活动期, 于2020年6月至8月在我国东部地区9个省份共采集18个样地20份蓝彩带蜂的蜂粮样品(在江西省吉安市的井冈山市和青原区分别于6月和8月进行1次样品采集)(表 1, 附图 1)。由于蓝彩带蜂的独栖性, 因此同一样地的蓝彩带蜂蜂粮采取混合多个巢穴内蜂粮样本至1-2g, 置于2mL无菌离心管内密封保存, 每个样品为3个重复, 所有样品带回实验室后-20℃留存并用于后续实验。
| 样本 Sample |
采集地点 Collecting sites |
经纬度Latitude and Longitude | 海拔高度/m Elevation |
采集日期 Date of collection |
|
| N° | E° | ||||
| AHGZ | 安徽省蚌埠市固镇县 | 33.25 | 117.37 | 47.5 | 2020.8.6 |
| AHJZ | 安徽省六安市金寨县 | 31.36 | 115.56 | 189.41 | 2020.8.4 |
| CQSMS | 重庆市四面山自然保护区 | 28.58 | 106.37 | 1133.74 | 2020.8.30 |
| CQWS | 重庆市巫山县当阳乡 | 31.40 | 109.98 | 774.05 | 2020.8.29 |
| FJSM | 福建省三明市胡访镇 | 26.18 | 117.23 | 413.94 | 2020.8.23 |
| GXHP | 广西省北海市合浦县闸口镇 | 21.71 | 109.49 | -11.24 | 2020.7.25 |
| GXLZ | 广西省柳州市雒容镇 | 24.38 | 109.68 | 100.67 | 2020.7.24 |
| HNCS | 湖南省长沙市跳马镇 | 28.25 | 113.17 | 41.27 | 2020.8.24 |
| HNHY | 湖南省衡阳市衡阳县 | 26.98 | 112.43 | 83.24 | 2020.8.19 |
| HNLS | 海南省陵水黎族自治县 | 18.40 | 109.99 | -5.66 | 2020.7.27 |
| HNQY | 湖南省永州市祁阳县 | 26.39 | 111.85 | 93.52 | 2020.7.22 |
| HNWC | 海南省文昌市龙楼镇 | 19.68 | 111.01 | -0.66 | 2020.7.26 |
| JXGX | 江西省赣州市赣县区 | 25.99 | 114.83 | 158.02 | 2020.8.11 |
| JXJG6 | 江西省吉安市井冈山市 | 26.47 | 114.21 | 365.73 | 2020.6.25 |
| JXJG8 | 江西省吉安市井冈山市 | 26.47 | 114.21 | 365.73 | 2020.8.13 |
| JXQY6 | 江西省吉安市青原区 | 27.07 | 115.10 | 116.25 | 2020.6.26 |
| JXQY8 | 江西省吉安市青原区 | 27.07 | 115.10 | 116.25 | 2020.8.14 |
| JXYZ | 江西省宜春市袁州区 | 27.76 | 114.21 | 108.07 | 2020.6.27 |
| SDLL | 山东省临沂市兰陵县 | 34.99 | 118.12 | 145.00 | 2020.8.18 |
| ZJCS | 浙江省衢州市常山县 | 28.98 | 118.64 | 104.90 | 2020.8.25 |

|
| 附图 1 蓝彩带蜂雄性、雌性蜂及样品采集 Male and female of Nomia chalybeata and sample collection A雄性蓝彩带蜂;B雌性蓝彩带蜂;C蓝彩带蜂的卵;D蓝彩带蜂的幼虫及蜂粮;E蓝彩带蜂的巢穴 |
将混合样本加入液氮充分研磨后, 使用E.Z.N.A.® Soil DNA Kit(Omega Bio-tek, Norcross, GA, USA)按照试剂盒说明书进行DNA抽提, 利用1%琼脂糖凝胶电泳检测。
1.3 PCR扩增和测序对花粉核基因组的ITS2区基因序列进行扩增, 使用引物ITSS2F:5′-ATGCGATACTTGGTGTGAAT-3′;ITS4R:5′-TCCTCCGCTTATTGATATGC-3′。PCR反应体系为20 μL, 其中:2 μL DNA模板(5 ng/μL), 4 μL FastPfu Buffer (5×), 2.5 μL dNTPs (2.5mmol/L), 上下游引物各0.8 μL (5 μmol/L), 0.4 μL FastPfu Polymerase, 添加ddH2O至20 μL。PCR反应条件:95℃预变性5 min;95℃变性30s, 55℃退火30s, 72℃延伸45s, 共29个循环;72℃延伸10 min;12℃保存。PCR产物经2%琼脂糖凝胶电泳检测后, 使用AxyPrep DNA Gel Extraction Kit(Axygen Biosciences, Union City, CA, U.S.)将其纯化。纯化后产物送至上海凌恩生物科技有限公司基于Illumina MiSeq平台进行双端测序(2×250 bp)。
1.4 数据处理 1.4.1 数据质控与优化测序原始数据使用Trimmomatic[34]过滤read尾部低质量碱基(Q<20);利用FLASH v1.2.11 [35]对短片段进行拼接(重叠区长度≥10bp, 重叠区错配率≤0.2), 根据引物方向校正拼接序列方向;用Usearch v10.0(http://drive5.com/uparse/)去除嵌合体[36]。最终得到每个样本的有效序列并用于后续类别注释。
1.4.2 OTU聚类及注释使用Usearch v10.0(http://drive5.com/uparse/)软件按照97%相似性对非重复序列(不含单序列)进行可操作性分类单元(OTU)聚类, 得到OTU的代表序列[36]。基于Unite v8.2 (http://unite.ut.ee/index.php)数据库采用RDP classifier v2.2贝叶斯算法(http://rdp.cme.msu.edu/)对OTU代表序列进行物种分类注释分析[37]。由于引物具有通用性, 注释为真菌的OTU序列在本研究中被舍弃, 同时删除样本测序中小于10的序列物种。
1.4.3 分析数据数据处理在Excel 2016软件进行统计, 运用R软件计算ACE指数、Shannon指数, 利用SPSS 22.0进行单因素方差分析及多重比较, 利用Origin 2019b进行柱状图绘制。基于OTU利用R软件计算各样本Bray-Curtis距离并采用factoextra包进行层次聚类(Hierarchical clustering)绘图。利用R软件vegan包利用非度量多维尺度法(NMDS)对聚类进行Beta多样性分析, 进一步进行相似性分析(ANOSIM)检验组间是否存在显著差异。
2 结果 2.1 蓝彩带蜂蜂粮测序数据及物种注释本研究中共获得2 722 294条有效序列, 平均长度为(338.5±5.6) bp, 序列按照97%的相似水平进行聚类获得491个OTUs。采用RDP classifier贝叶斯算法对OTU代表序列进行物种分类分析, 注释结果为47科, 99属, 124种粉源植物(表 2)。
| 样本 Sample |
有效序列数量 Number of valid sequences |
平均长度 AvgLen/bp |
不同分类阶元的归类数量 Number of different taxonomic categories |
||
| 科Family | 属Genus | 种Species | |||
| AHGZ | 43344 | 337.4 | 12 | 18 | 18 |
| AHJZ | 50320 | 336.8 | 12 | 15 | 16 |
| CQSMS | 53286 | 333.2 | 5 | 5 | 5 |
| CQWS | 45565 | 329.0 | 13 | 16 | 19 |
| FJSM | 44681 | 337.0 | 7 | 10 | 10 |
| GXHP | 38797 | 337.0 | 14 | 19 | 21 |
| GXLZ | 53809 | 337.9 | 8 | 9 | 13 |
| HNCS | 46687 | 337.0 | 5 | 5 | 7 |
| HNHY | 43209 | 337.9 | 9 | 12 | 12 |
| HNLS | 44522 | 356.6 | 11 | 13 | 14 |
| HNQY | 39098 | 336.7 | 5 | 5 | 5 |
| HNWC | 45587 | 338.2 | 9 | 12 | 12 |
| JXGX | 43802 | 331.4 | 6 | 8 | 11 |
| JXJG6 | 43149 | 337.7 | 5 | 6 | 7 |
| JXJG8 | 41032 | 344.3 | 7 | 8 | 8 |
| JXQY6 | 46409 | 336.5 | 6 | 8 | 9 |
| JXQY8 | 47604 | 342.2 | 5 | 7 | 8 |
| JXYZ | 35595 | 339.6 | 4 | 5 | 5 |
| SDLL | 49214 | 346.7 | 23 | 42 | 49 |
| ZJCS | 50867 | 336.8 | 11 | 15 | 16 |
| 总计Total | 906577 | - | 47 | 99 | 124 |
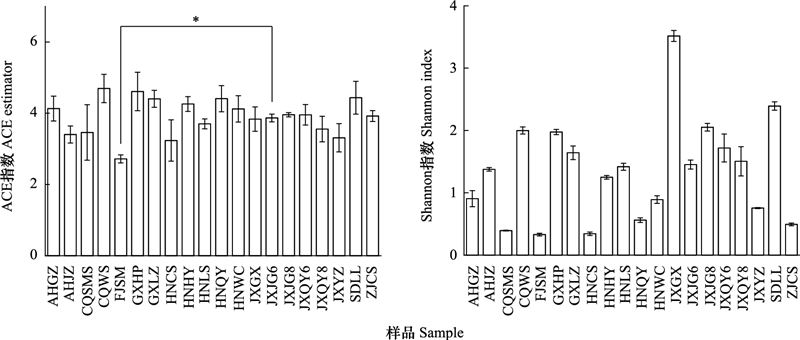
基于ITS扩增子获得的分类结果, 获得蓝彩带蜂蜂粮样本粉源植物共47科99属124种, 统计分析粉源植物物种组成和相对丰度(图 1)。在属分类阶元上, 粉源植物相对丰度较高的属为牡荆属(Vitex)、紫金牛属(Ardisia)、悬钩子属(Rubus)、耳草属(Hedyotis)、破布叶属(Microcos)、柏拉木属(Blastus)、稻属(Oryza)、凤仙花属(Impatiens)、银莲花属(Anemone)、珍珠菜属(Lysimachia)等, 所占比例分别为43.47%、17.07%、8.35%、5.50%、5.00%、4.03%、2.83%、2.21%、2.12%、1.73%。在种分类阶元上, 粉源植物主要包括黄荆(Vitex negundo)、罗伞树(Ardisia quinquegona)、山莓(Rubus corchorifolius)、疏花耳草(Hedyotis matthewii)、破布叶(Microcos paniculata)、少花柏拉木(Blastus pauciflorus)、野生稻(Oryza rufipogon)、野棉花(Anemone vitifolia)、水金凤(Impatiens noli-tangere)、藜状珍珠菜(Lysimachia chenopodioides)等, 所占比例分别为43.46%、17.06%、8.35%、5.50%、5.00%、4.03%、2.83%、2.12%、2.09%、1.57%。
|
| 图 1 蓝彩带蜂粉源植物在属、种水平的组成及相对丰度 Fig. 1 Composition and relative abundance of pollen plants of Nomia chalybeata at genus and species level |
基于OTU水平进行Alpha多样性指数分析, 利用ACE指数、Shannon指数来反映粉源植物物种丰富度及多样性。CQWS、GXHP、SDLL的ACE指数(分别为4.69±0.40、4.61±0.54、4.44±0.46)较高, FJSM的ACE指数(2.72±0.11)最低(图 2)。JXGX、SDLL的Shannon指数(分别为3.52±0.09、2.39±0.07)较高, FJSM的Shannon指数(0.33±0.02)最低, 不同样地间具有显著差异(附表 1, 图 2)。综上, SDLL样地的丰富度和多样性最高, FJSM样地的丰富度和多样性最低。
| P | AHGZ | AHJZ | CQSMS | CQWS | FJSM | GXHP | GXLZ | HNCS | HNHY | HNLS | HNQY | HNWC | JXGX | JXJG6 | JXJG8 | JXQY6 | JXQY8 | JXYZ | SDLL |
| AHJZ | 0.98 | ||||||||||||||||||
| CQSMS | 0.98 | 0.03 | |||||||||||||||||
| CQWS | 0.23 | 0.09 | 0.07 | ||||||||||||||||
| FJSM | 0.94 | 0.00 | 1.00 | 0.01 | |||||||||||||||
| GXHP | 0.37 | 0.02 | 0.03 | 1.00 | 0.00 | ||||||||||||||
| GXLZ | 0.30 | 1.00 | 0.37 | 0.94 | 0.27 | 0.99 | |||||||||||||
| HNCS | 0.93 | 0.00 | 1.00 | 0.01 | 1.00 | 0.00 | 0.23 | ||||||||||||
| HNHY | 1.00 | 0.69 | 0.04 | 0.05 | 0.00 | 0.01 | 0.97 | 0.00 | |||||||||||
| HNLS | 0.87 | 1.00 | 0.16 | 0.04 | 0.04 | 0.05 | 1.00 | 0.02 | 0.98 | ||||||||||
| HNQY | 1.00 | 0.00 | 0.93 | 0.00 | 0.32 | 0.00 | 0.24 | 0.29 | 0.00 | 0.02 | |||||||||
| HNWC | 1.00 | 0.25 | 0.58 | 0.00 | 0.27 | 0.01 | 0.25 | 0.20 | 0.46 | 0.08 | 0.42 | ||||||||
| JXGX | 0.01 | 0.04 | 0.05 | 0.01 | 0.02 | 0.03 | 0.01 | 0.02 | 0.03 | 0.00 | 0.01 | 0.00 | |||||||
| JXJG6 | 0.73 | 1.00 | 0.23 | 0.11 | 0.11 | 0.21 | 1.00 | 0.07 | 0.99 | 1.00 | 0.06 | 0.10 | 0.00 | ||||||
| JXJG8 | 0.18 | 0.11 | 0.07 | 1.00 | 0.02 | 1.00 | 0.83 | 0.01 | 0.06 | 0.04 | 0.00 | 0.00 | 0.01 | 0.08 | |||||
| JXQY6 | 0.86 | 1.00 | 0.83 | 1.00 | 0.79 | 1.00 | 1.00 | 0.78 | 1.00 | 1.00 | 0.87 | 0.97 | 0.31 | 1.00 | 1.00 | ||||
| JXQY8 | 1.00 | 1.00 | 0.94 | 1.00 | 0.91 | 1.00 | 1.00 | 0.91 | 1.00 | 1.00 | 0.97 | 1.00 | 0.29 | 1.00 | 1.00 | 1.00 | |||
| JXYZ | 1.00 | 0.05 | 0.00 | 0.10 | 0.04 | 0.04 | 0.60 | 0.10 | 0.10 | 0.32 | 0.84 | 1.00 | 0.06 | 0.45 | 0.11 | 0.96 | 1.00 | ||
| SDLL | 0.07 | 0.06 | 0.06 | 0.28 | 0.02 | 0.29 | 0.22 | 0.01 | 0.04 | 0.01 | 0.00 | 0.00 | 0.02 | 0.02 | 0.44 | 0.99 | 0.96 | 0.09 | |
| ZJCS | 1.00 | 0.00 | 0.88 | 0.02 | 0.14 | 0.00 | 0.34 | 0.32 | 0.00 | 0.06 | 1.00 | 0.51 | 0.03 | 0.14 | 0.02 | 0.86 | 0.96 | 0.13 | 0.02 |
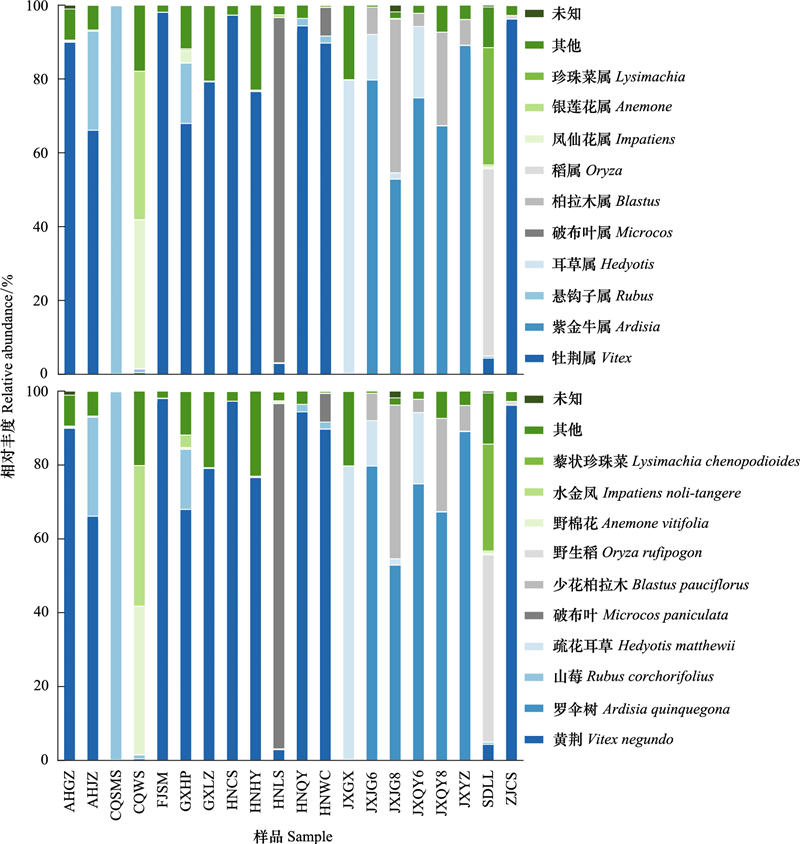
不同样地蓝彩带蜂粉源植物种类及相对丰度不同, 基于此对不同样地蓝彩带蜂粉源植物进行层次聚类分析, 采用Bray-curtis距离计算各样本间距离。当类距离为27时, 聚类结果可分为七类(图 3), 其中第一聚类(A聚类, 下同)共10个样地分别为AHGZ、AHJZ、FJSM、GXHP、GXLZ、HNCS、HNHY、HNQY、HNWC、ZJCS;第二聚类(B聚类)共5个样地分别为JXJG6、JXJG8、JXQY6、JXQY8、JXYZ;剩余样地各为一个聚类。各聚类的优势种都不同, A聚类优势种是黄荆, B聚类优势种是罗伞树, C聚类(JXGX)优势种是疏花耳草, D聚类(CQWS)优势种是野棉花, E聚类(CQSMS)优势种是山莓, F聚类(HNLS)优势种是破布叶, G聚类(SDLL)优势种是野生稻。
|
| 图 3 蓝彩带蜂粉源植物层次聚类图 Fig. 3 Hierarchical cluatering of pollen plants of N. chalybeata |
利用非度量多维尺度法(NMDS)对A聚类进行Beta多样性分析结果显示十个样地距离都不远, 进一步对A聚类的组间距离进行相似性分析, 结果显示A聚类两两对比不存在显著差异(P>0.05)。对B聚类进行Beta多样性分析, 结果显示B聚类五个样地相聚很近(图 4), 对B聚类的组间距离进行相似性分析, 结果显示B聚类粉源植物结构两两对比不存在显著差异(P>0.05)。其中相同地点不同时期的JXJG6和JXJG8粉源植物结构不存在显著差异(R2= 1, P =0.1);JXQY6和JXQY8粉源植物结构也不存在显著差异(R2= 0.593, P=0.1)。因此在6月和8月两个时期蓝彩带蜂的粉源植物结构无显著差异。
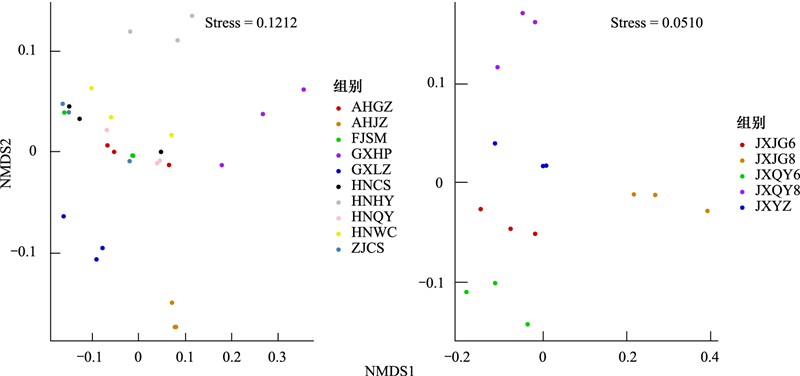
|
| 图 4 利用非度量多维尺度法(NMDS) 对蓝彩带蜂粉源植物A、B聚类进行Beta多样性分析 Fig. 4 The Beta diversity of A and B cluster of pollen plants of Nomia chalybeata analyzed by Non-metric multidimensional scaling (NMDS) Nomia chalybeata |
目前DNA宏条形码作为一种新型鉴定手段在蜂蜜溯源、昆虫-植物传粉网络、水生态等研究领域中应用广泛[38-40]。对于植物鉴定而言, 基于通用引物的可得性、高水平的分类分辨率, 生命条形码联盟推荐rbcL和matK的质体DNA (ptDNA)基因区作为标准DNA条形码标记[41]。因为它们是植物核心DNA条形码之一, 而且一些国家对本地植物区系建立了相应的rbcL DNA条形码参考库, 可大大提升对物种的鉴别能力[42]。ITS2作为条形码序列是基于通用引物保守区域的可用性、扩增的方便性以及足够的可变性, 甚至可以区分密切相关的物种[43-45]。近年来在植物类群的定性分析方面, Richardson[46]等人利用蜂蜜孢粉学和ITS2 metabarcoding对美国俄亥俄州麦迪逊县蜂群采集的花粉进行鉴定, 结果显示ITS2 metabarcoding鉴定到19个植物科, 蜂蜜孢粉学仅鉴定到8个科, ITS2序列具有更高的灵敏度和分类分辨率, 而且对于花粉的定量分析, 使用ITS2 metabarcoding和蜂蜜孢粉学共同鉴定优于单独的方法。本研究虽未利用蜂蜜孢粉学进行对比, 但研究结果与文献记载基本吻合, 进一步补充蓝彩带蜂等野生蜜蜂粉源植物DNA宏条形码技术研究方面的空白。
本研究在数据处理方面对样本数据进行一个阈值设置:去除每个样本最大计数数小于10的序列, 这是由于DNA宏条形码会产生假阳性结果[47-48]。假阳性通常是由于污染、嵌合序列和/或生物信息学管道的错误识别驱动的、缺少条形码空白或样品的稀缺性等原因造成, 因此尤其是在濒危物种和入侵物种的监测中, 物种的误判对生态系统的影响是非常严重的[40], DNA宏条形码在鉴定物种方面仍需与传统鉴定方法(蜂蜜孢粉学)相结合才能更精确、快速的鉴定物种。
基于OTU水平, 对不同样地的ACE指数、Shannon指数进行分析发现不同地区的Alpha多样性是有差异的, 这可能是因为蓝彩带蜂的栖息地周围粉源植物不同造成物种丰富度和多样性均有一定程度差异。在山东省临沂市兰陵县采集的蜂粮显示此地蓝彩带蜂粉源植物的物种丰富度和多样性最高, 物种丰富度和多样性最低的是福建省三明市胡访镇采集的蜂粮。在江西省吉安市井冈山市和江西省吉安市青原区的两次采集样品(JXJG6、JXJG8、JXQY6、JXQY8)的ACE指数、Shannon指数均无差异, Beta多样性分析结果也显示距离很近, 且结构两两对比不存在显著差异, 因此在6月和8月两个时期蓝彩带蜂的粉源植物多样性和结构无显著差异。六月这两个样地主要粉源植物罗伞树的相对丰度分别为79.7%和74.9%, 而八月相对丰度分别为52.8%和67.3%, 表明该蜂成虫活动期主要粉源植物变化不大, 优势粉源植物罗伞树花期约为五月, 果期约十一月[49], 从六月至八月的主要粉源植物花期过时, 蓝彩带蜂逐渐开始利用其他粉源植物。
早期研究者根据中国科学院馆藏各地送检标本的信息总结蓝彩带蜂到访植物主要有:黄荆、草木犀(Melilotus officinalis)、醉鱼草属(Buddleja)、水柳(Homonoia riparia)、紫苜蓿(Medicago sativa)、槐(Styphnolobium japonicum)、益母草(Leonurus japonicus)、菊科(Compositae)等[31]。本研究鉴定到的粉源植物中黄荆、草木犀、菊科、醉鱼草属、紫苜蓿、槐均有包含;益母草未被检测, 但是本研究中检测到与益母草同属的细叶益母草(Leonurus sibiricus)及假鬃尾草(Leonurus chaituroides), 可能是送检标本采集地有益母草物种分布而没有同属细叶益母草和假鬃尾草的分布, 也有可能形态学鉴定有误;水柳在本研究中也未被检测, 水柳花期一般为3至5月[50], 而蓝彩带蜂成虫活动期为6至8月[31], 所以标本采集者在水柳上采集到的只是栖息状态下的蓝彩带蜂。
Biesmeijer[51]等人在英国及荷兰境内的调查表明:蜜蜂和蜜(粉)源植物的多样性呈下降趋势, 并对当地的经济作物和野生物种产生不利影响。在群落范围内植物-传粉者相互作用对维持生物多样性十分重要[52], 且粉源植物和传粉昆虫二者间具有密切关联; 邓惠等[53]的研究发现:忍冬属的4种植物花色发生变化则传粉者的访花频率随之改变, 花变色的特性、范围差异会导致该物种由不同种类的传粉昆虫进行授粉, 这是开花植物与昆虫长期相互适应并共同进化的结果; Goulnik等[54]对2017-2019年关于植物-传粉者互作网络等关键词的文章进行分析发现土地利用集约化可能会导致花色的改变、花蜜管平均深度的降低以及花粉质量的降低, 花的性状可能与传粉者匹配性状的改变有关, 传粉者与其传粉植物间有着十分的密切关系。因此调查研究蓝彩带蜂的主要粉源植物对于维持生物多样性具有重要作用, 为研究野生蜜蜂-植物间的相互作用奠定基础。
| [1] |
Klein A M, Vaissière B E, Cane J H, Steffan-Dewenter I, Cunningham S A, Kremen C, Tscharntke T. Importance of pollinators in changing landscapes for world crops. Proceedings of the Royal Society B: Biological Sciences, 2007, 274(1608): 303-313. DOI:10.1098/rspb.2006.3721 |
| [2] |
Lefebvre V, Daugeron C, Villemant C, Fontaine C. Empidine dance flies pollinate the woodland geranium as effectively as bees. Biology Letters, 2019, 15(7): 20190230. DOI:10.1098/rsbl.2019.0230 |
| [3] |
Ollerton J, Winfree R, Tarrant S. How many flowering plants are pollinated by animals?. Oikos, 2011, 120(3): 321-326. DOI:10.1111/j.1600-0706.2010.18644.x |
| [4] |
Aizen M A, Aguiar S, Biesmeijer J C, Garibaldi L A, Inouye D W, Jung C, Martins D J, Medel R, Morales C L, Ngo H, Pauw A, Paxton R J, Sáez A, Seymour C L. Global agricultural productivity is threatened by increasing pollinator dependence without a parallel increase in crop diversification. Global Change Biology, 2019, 25(10): 3516-3527. DOI:10.1111/gcb.14736 |
| [5] |
Barroso-Arévalo S, Vicente-Rubiano M, Puerta F, Molero F, Sánchez-Vizcaíno J M. Immune related genes as markers for monitoring health status of honey bee colonies. BMC Veterinary Research, 2019, 15(1): 72. DOI:10.1186/s12917-019-1823-y |
| [6] |
Potts S G, Biesmeijer J C, Kremen C, Neumann P, Schweiger O, Kunin W E. Global pollinator declines: trends, impacts and drivers. Trends in Ecology & Evolution, 2010, 25(6): 345-353. |
| [7] |
Seitz N, Vanengelsdorp D, Leonhardt S D. Are native and non-native pollinator friendly plants equally valuable for native wild bee communities?. Ecology and Evolution, 2020, 10(23): 12838-12850. DOI:10.1002/ece3.6826 |
| [8] |
赵盼盼, 周嘉聪, 林开淼, 张秋芳, 袁萍, 曾晓敏, 苏莹, 徐建国, 陈岳民, 杨玉盛. 海拔梯度变化对中亚热带黄山松土壤微生物生物量和群落结构的影响. 生态学报, 2019, 39(6): 2215-2225. |
| [9] |
Di Pasquale G, Salignon M, Le Conte Y, Belzunces L P, Decourtye A, Kretzschmar A, Suchail S, Brunet J L, Alaux C. Influence of pollen nutrition on honey bee health: do pollen quality and diversity matter?. PLoS One, 2013, 8(8): e72016. DOI:10.1371/journal.pone.0072016 |
| [10] |
Scheiner R, Page R E, Erber J. Sucrose responsiveness and behavioral plasticity in honey bees (Apis mellifera). Apidologie, 2004, 35(2): 133-142. DOI:10.1051/apido:2004001 |
| [11] |
Williams N M, Ward K L, Pope N, Isaacs R, Wilson J, May E A, Ellis J, Daniels J, Pence A, Ullmann K, Peters J. Native wildflower plantings support wild bee abundance and diversity in agricultural landscapes across the United States. Ecological Applications, 2015, 25(8): 2119-2131. DOI:10.1890/14-1748.1 |
| [12] |
黄敦元, 黄思程, 李红英, 窦飞越, 寇若玫, 李月, 罗安才. 传粉昆虫蜜(粉)源植物的鉴定方法探讨. 重庆师范大学学报: 自然科学版, 2020, 37(3): 1-8. |
| [13] |
林翰哲, 华伟平. 武夷山国家级自然保护区野生蜜源植物资源分布研究. 福建农业学报, 2020, 35(8): 876-882. |
| [14] |
李捷, 朱朝东, 王凤鹤, 黄敦元, 张彦周, 丁亮, 黄海荣. 野生蜜蜂及其传粉作用的研究现状. 生物多样性, 2007, 15(6): 687-692. DOI:10.3321/j.issn:1005-0094.2007.06.017 |
| [15] |
Fijen T P M, Scheper J A, Boom T M, Janssen N, Raemakers I, Kleijn D. Insect pollination is at least as important for marketable crop yield as plant quality in a seed crop. Ecology Letters, 2018, 21(11): 1704-1713. DOI:10.1111/ele.13150 |
| [16] |
Garibaldi L A, Steffan-Dewenter I, Winfree R, Aizen M A, Bommarco R, Cunningham S A, Kremen C, Carvalheiro L G, Harder L D, Afik O, Bartomeus I, Benjamin F, Boreux V, Cariveau D, Chacoff N P, Dudenhöffer J H, Freitas B M, Ghazoul J, Greenleaf S, Hipólito J, Holzschuh A, Howlett B, Isaacs R, Javorek S K, Kennedy C M, Krewenka K M, Krishnan S, Mandelik Y, Mayfield M M, Motzke I, Munyuli T, Nault B A, Otieno M, Petersen J, Pisanty G, Potts S G, Rader R, Ricketts T H, Rundlöf M, Seymour C L, Schüepp C, Szentgyörgyi H, Taki H, Tscharntke T, Vergara C H, Viana B F, Wanger T C, Westphal C, Williams N, Klein A M. Wild pollinators enhance fruit set of crops regardless of honey bee abundance. Science, 2013, 339(6127): 1608-1611. DOI:10.1126/science.1230200 |
| [17] |
Li H Y, Orr M C, Luo A C, Dou F Y, Kou R M, Hu F, Zhu C D, Huang D Y. Relationships between wild bee abundance and fruit set of Camellia oleifera Abel. Journal of Applied Entomology, 2021, 145(4): 277-285. DOI:10.1111/jen.12849 |
| [18] |
Mallinger R E, Gratton C. Species richness of wild bees, but not the use of managed honeybees, increases fruit set of a pollinator-dependent crop. Journal of Applied Ecology, 2015, 52(2): 323-330. DOI:10.1111/1365-2664.12377 |
| [19] |
Angelella G M, McCullough C T, O'Rourke M E. Honey bee hives decrease wild bee abundance, species richness, and fruit count on farms regardless of wildflower strips. Scientific Reports, 2021, 11(1): 3202. DOI:10.1038/s41598-021-81967-1 |
| [20] |
窦飞越, 李红英, 宋海涛, 寇若玫, 周泽扬, 罗阿蓉, 黄敦元. 凹唇壁蜂的营巢生物学研究. 环境昆虫学报, 2021: 1-14. (2021-03-12). https://kns.cnki.net/kcms/detail/detail.aspx?dbcode=CAPJ&dbname=CAPJLAST&filename=KCTD20210311002&uniplatform=NZKPT&v=L9va4xRwie6x%25mmd2F6eEI7PHYNE9PmjVx1NhPF2LJt6mBC34kuk6CURvt4GrTWC9LkUJ.
|
| [21] |
黄敦元, 谷平, 苏田娟, 余江帆, 何波, 牛泽清, 朱朝东. 大分舌蜂主要生物学特性研究. 环境昆虫学报, 2015, 37(1): 133-138. |
| [22] |
何波, 谷战英, 李红英, 黄敦元. 基于DNA条形码的白斑切叶蜂粉源植物种类及多样性分析. 生态学报, 2020, 40(6): 2122-2129. |
| [23] |
Matias D M S, Leventon J, Rau A L, Borgemeister C, Von Wehrden H. A review of ecosystem service benefits from wild bees across social contexts. Ambio, 2017, 46(4): 456-467. DOI:10.1007/s13280-016-0844-z |
| [24] |
Greenleaf S S, Kremen C. Wild bees enhance honey bees' pollination of hybrid sunflower. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(37): 13890-13895. DOI:10.1073/pnas.0600929103 |
| [25] |
Deagle B E, Kirkwood R, Jarman S N. Analysis of Australian fur seal diet by pyrosequencing prey DNA in faeces. Molecular Ecology, 2009, 18(9): 2022-2038. DOI:10.1111/j.1365-294X.2009.04158.x |
| [26] |
Glenn T C. Field guide to next-generation DNA sequencers. Molecular Ecology Resources, 2011, 11(5): 759-769. DOI:10.1111/j.1755-0998.2011.03024.x |
| [27] |
李磊, 蒋敬, 陈云霞. DNA宏条形码技术在动植物法医鉴定中的应用进展. 南京林业大学学报: 自然科学版, 2021, 45(1): 235-241. |
| [28] |
Bell K L, Fowler J, Burgess K S, Dobbs E K, Gruenewald D, Lawley B, Morozumi C, Brosi B J. Applying pollen DNA metabarcoding to the study of plant-pollinator interactions. Applications in Plant Sciences, 2017, 5(6): 1600124. DOI:10.3732/apps.1600124 |
| [29] |
CBOL Plant Working Group. A DNA barcode for land plants. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(31): 12794-12797. DOI:10.1073/pnas.0905845106 |
| [30] |
Jones L, Brennan G L, Lowe A, Creer S, Ford C R, De Vere N. Shifts in honeybee foraging reveal historical changes in floral resources. Communications Biology, 2021, 4(1): 37. DOI:10.1038/s42003-020-01562-4 |
| [31] |
黄海荣. 中国彩带蜂亚科(膜翅目: 蜜蜂总科: 隧蜂科)系统分类研究[D]. 北京: 北京林业大学, 2008.
|
| [32] |
Li H Y, Lu H H, Huang S G, Fan X D, Luo A C, Huang D Y. The complete mitogenome of Nomia chalybeata (Hymenoptera: Halictidae) and phylogenetic analysis. Mitochondrial DNA Part B, 2020, 5(3): 2844-2845. DOI:10.1080/23802359.2020.1790320 |
| [33] |
Hannan A, Maeta Y, Miyanaga R. Nesting biology and life cycle of Nomia (Acunomia) chalybeata Smith on Iriomote Island, southernmost Archipelago of Japan, with notes on the simultaneous occurrence of diapausing and non-diapausing prepupae within the same nests (Hymenoptera: Halictidae). Journal of the Saudi Society of Agricultural Sciences, 2013, 12(2): 91-99. DOI:10.1016/j.jssas.2012.08.002 |
| [34] |
Bolger A M, Lohse M, Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics, 2014, 30(15): 2114-2120. DOI:10.1093/bioinformatics/btu170 |
| [35] |
Mago c ˇ T, Salzberg S L. FLASH: fast length adjustment of short reads to improve genome assemblies. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507 |
| [36] |
Edgar R C. UPARSE: highly accurate OTU sequences from microbial amplicon reads. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [37] |
Kõljalg U, Nilsson R H, Abarenkov K, Tedersoo L, Taylor A F S, Bahram M, Bates S T, Bruns T D, Bengtsson-Palme J, Callaghan T M, Douglas B, Drenkhan T, Eberhardt U, Dueñas M, Grebenc T, Griffith G W, Hartmann M, Kirk P M, Kohout P, Larsson E, Lindahl B D, Lücking R, Martín M P, Matheny P B, Nguyen N H, Niskanen T, Oja J, Peay K G, Peintner U, Peterson M, Põldmaa K, Saag L, Saar I, Schüßler A, Scott J A, Senés C, Smith M E, Suija A, Taylor D L, Telleria M T, Weiss M, Larsson K H. Towards a unified paradigm for sequence-based identification of fungi. Molecular Ecology, 2013, 22(21): 5271-5277. DOI:10.1111/mec.12481 |
| [38] |
Tanaka K, Nozaki A, Nakadai H, Shiwa Y, Shimizu-Kadota M. Using pollen DNA metabarcoding to profile nectar sources of urban beekeeping in Kōtō-ku, Tokyo. BMC Research Notes, 2020, 13(1): 515. DOI:10.1186/s13104-020-05361-2 |
| [39] |
Lucas A, Bodger O, Brosi B J, Ford C R, Forman D W, Greig C, Hegarty M, Neyland P J, De Vere N. Generalisation and specialisation in hoverfly (Syrphidae) grassland pollen transport networks revealed by DNA metabarcoding. Journal of Animal Ecology, 2018, 87(4): 1008-1021. DOI:10.1111/1365-2656.12828 |
| [40] |
赵彦伟, 陈家琪, 董丽, 麻晓梅, 白洁, 田凯. 环境DNA技术在水生态领域应用研究进展. 农业环境科学学报, 1-15. (2021-09-07). https://kns.cnki.net/kcms/detail/detail.aspx?dbcode=CAPJ&dbname=CAPJLAST&filename=NHBH20210907000&uniplatform=NZKPT&v=9ez7O3Csmh7hfFJ5oNfy21rfWsEGpYZFBZVS0TL2h0gqHB9U5MHAnpnslhmuJD%25mmd2B0.
|
| [41] |
Hollingsworth P M, Graham S W, Little D P. Choosing and using a plant DNA barcode. PLoS One, 2011, 6(5): e19254. DOI:10.1371/journal.pone.0019254 |
| [42] |
Hawkins J, De Vere N, Griffith A, Ford C R, Allainguillaume J, Hegarty M J, Baillie L, Adams-Groom B. Using DNA Metabarcoding to identify the floral composition of honey: a new tool for investigating honey bee foraging preferences. PLoS One, 2015, 10(8): e0134735. DOI:10.1371/journal.pone.0134735 |
| [43] |
Chen S L, Yao H, Han J P, Liu C, Song J Y, Shi L C, Zhu Y J, Ma X Y, Gao T, Pang X H, Luo K, Li Y, Li X W, Jia X C, Lin Y L, Leon C. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species. PLoS One, 2010, 5(1): e8613. DOI:10.1371/journal.pone.0008613 |
| [44] |
Han J P, Zhu Y J, Chen X C, Liao B S, Yao H, Song J Y, Chen S L, Meng F Y. The short ITS2 sequence serves as an efficient taxonomic sequence tag in comparison with the full-length ITS. BioMed Research International, 2013, 2013(5): 741476. |
| [45] |
Pang X H, Shi L C, Song J Y, Chen X C, Chen S L. Use of the potential DNA barcode ITS2 to identify herbal materials. Journal of Natural Medicines, 2013, 67(3): 571-575. DOI:10.1007/s11418-012-0715-2 |
| [46] |
Richardson R T, Lin C H, Sponsler D B, Quijia J O, Goodell K, Johnson R M. Application of ITS2 Metabarcoding to determine the provenance of pollen collected by honey bees in an agroecosystem. Applications in Plant Sciences, 2015, 3(1): 1400066. DOI:10.3732/apps.1400066 |
| [47] |
Potter C, De Vere N, Jones L E, Ford C R, Hegarty M J, Hodder K H, Diaz A, Franklin E L. Pollen metabarcoding reveals broad and species-specific resource use by urban bees. PeerJ, 2019, 7(5): e5999. |
| [48] |
Pornon A, Escaravage N, Burrus M, Holota H, Khimoun A, Mariette J, Pellizzari C, Iribar A, Etienne R, Taberlet P, Vidal M, Winterton P, Zinger L, Andalo C. Using metabarcoding to reveal and quantify plant-pollinator interactions. Scientific Reports, 2016, 6(1): 27282. DOI:10.1038/srep27282 |
| [49] |
中国科学院中国植物志编辑委员会. 中国植物志. 第五十八卷. 北京: 科学出版社, 1979.
|
| [50] |
中国科学院中国植物志编辑委员会. 中国植物志. (第四十四卷第二分册). 北京: 科学出版社, 1996: 87.
|
| [51] |
Biesmeijer J C, Roberts S P M, Reemer M, Ohlemüller R, Edwards M, Peeters T, Schaffers A P, Potts S G, Kleukers R, Thomas C D, Settele J, Kunin W E. Parallel declines in pollinators and insect-pollinated plants in Britain and the Netherlands. Science, 2006, 313(5785): 351-354. DOI:10.1126/science.1127863 |
| [52] |
Wei N, Kaczorowski R L, Arceo-Gómez G, O'Neill E M, Hayes R A, Ashman T. Pollinators contribute to the maintenance of flowering plant diversity. Nature, 2021, 597(7878): 688-692. DOI:10.1038/s41586-021-03890-9 |
| [53] |
邓惠, 向甘驹, 郭友好, 杨春锋. 秦岭忍冬属4种植物的繁育系统及花色变化的研究. 植物科学学报, 2017, 35(1): 1-12. |
| [54] |
Goulnik J, Plantureux S, Dajoz I, Michelot-Antalik A. Using matching traits to study the impacts of land-use intensification on plant-pollinator interactions in European grasslands: a review. Insects, 2021, 12(8): 680. DOI:10.3390/insects12080680 |
 2022, Vol. 42
2022, Vol. 42