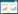文章信息
- 杨海乐, 杜浩, 祁洪芳, 俞录贤, 危起伟
- YANG Haile, DU Hao, QI Hongfang, YU Luxian, WEI Qiwei
- 青藏高原沙柳河流域自然径流驱动的流域生物信息流量化特征——以环境微生物为指标
- Watershed biological information flow driven by natural runoff in Shaliu River Basin on Qinghai-Tibet Plateau indicated by environmental microbes
- 生态学报. 2021, 41(9): 3475-3487
- Acta Ecologica Sinica. 2021, 41(9): 3475-3487
- http://dx.doi.org/10.5846/stxb202001010001
-
文章历史
- 收稿日期: 2020-01-01
- 网络出版日期: 2021-03-09
2. 青海湖裸鲤救护中心, 青海省青海湖裸鲤繁育与保护重点实验室, 西宁 810016
2. Qinghai Key Laboratory of Qinghai-Lake Naked Carps Breeding and Conservation, Rescue and Rehabilitation Center of Naked Carps of Qinghai Lake, Xining 810016, China
流域是生态系统的一个基本单元[1-2], 对于生态学研究具有非常重要的意义[3-4]。流域生态学(watershed ecology)是研究流域范围内陆地和水体生态系统相互关系的学科(全国科学技术名词审定委员会), 其主体在于以流域生态系统过程为核心将所关联起的流域内各相关子系统整合起来进行研究[5]。流域生态系统过程研究的核心工作是对流域生态系统中依托于水循环过程的各个子系统之间及之内的物质流、能量流、信息流进行研究[6]。在近百年来的流域生态相关研究中, 物质流和能量流的问题都得到了广泛的关注和深入的研究[7-10], 相比较而言, 信息流虽然曾有研究涉及[11-14], 但还很少, 也没有建立起相应的研究范式、概念模型和框架体系, 是整个流域生态系统过程研究中的明显短板。为推动流域生态学研究格局的进一步完善, 并促进其进一步发展, 有必要探索流域信息流的研究, 尤其是流域生物信息流的研究。
生物信息流有三个层次: 1)不同生命物质间的信息传递, 即分子生物学层面的转录、翻译等过程所承载的生物信息传递[15-16];2)生物物种间及生物个体间相互作用的信息传递, 即生态学层面的生物相互作用过程所承载的生物信息传递[17-18];3)生物空间迁移的信息传递, 即生物体或生物物质的扩散与迁移过程所承载的生物信息传递[19-20]。结合流域生态系统过程和生物信息流的研究内容, 本研究提出“流域生物信息流”的概念, 并将其定义为“生物信息依托于流域生态系统过程在不同空间和系统之间进行传递、交流、作用、反馈的路径、过程与控制”, 主要关注生物体及生物质所承载的生物信息在时空上的迁移扩散, 以及与生物体及生物质间相互作用相伴随的生物信息作用和反馈。流域生态系统过程研究主要关注流域生态系统的水陆间、干支流间、上下游间、不同生态斑块间、不同要素间的相互作用过程、周期性节律和趋势性变迁[21]。相应的, 流域生物信息流研究主要关注于水陆间、干支流间、上下游间、不同生态斑块间的生物信息流, 这些生物信息流的周期性节律和趋势性变迁, 以及地貌、水文、人类活动对这些生物信息流的影响等。
本研究以青藏高原区域生态系统组成相对简单、人类干扰相对较弱的青海湖重要入湖河流——沙柳河流域为研究区域, 以河流水体微生物和岸带土壤微生物为研究对象, 用环境DNA技术对沙柳河流域由陆地到水体、由上游到下游、由支流到干流的流域生物信息流(即自然径流驱动的微生物种类的空间输移)进行定量研究, (1)量化由陆地到水体的流域生物信息流的输移效率并探讨其驱动因素, (2)量化由上游到下游、由支流到干流的流域生物信息流的输移效率并探讨其驱动因素, (3)量化降雨对流域生物信息流输移效率的影响并探讨其影响机制。以期抛砖引玉, 为流域生态学研究者的流域生态系统过程相关研究和探索提供借鉴。
1 研究区域与方法 1.1 研究区域青海湖位于中国西部青藏高原上, 是中国最大的高原半咸水内陆湖泊, 对于生态保护来讲具有重要的战略意义, 同时其流域生态系统相对简单, 对于流域生态学研究来讲具有重要的模式意义。沙柳河是青海湖五条主要入湖河流之一, 发源于青海省海北藏族自治州刚察县境内桑斯扎山南麓, 全长106 km, 流经刚察县城后注入青海湖, 流域面积1320 km2。流域内以山间草原-草甸牧区为主, 生态系统类型在空间分布上同质性较高, 人类活动干扰也较弱, 对于流域生物信息流研究来讲, 是一个较为理想的简化模型式的研究区域。
本研究在沙柳河干支流上共设置9个样点(如图 1), 沿沙柳河干流由下而上依次是SL1、SL2、SL3、SL4、SL5m。SL4b是位于SL3和SL4之间的一个支流上的一个样点, SL4b处的河流径流量小于SL4处的河流径流量, SL5、SL6、SL6b是位于SL4和SL5m之间的一个支流上的3个样点, SL5处的河流径流量小于SL5m处的河流径流量, SL6b处的河流径流量小于SL6处的河流径流量。
|
| 图 1 样点设置 Fig. 1 Sampling sites SL1, 即沙柳河1号采样点;采样点下括号内的距离, 指该采样点到河流入湖口的径流距离 |
2019年6月25日和26日, 自沙柳河入湖口向上游依次采样, 每个样点采河流水样和岸带土样, 其中岸带土壤采样处离河流水体直线距离不超过5 m。6月25日, 白天天气晴好, 河水清澈, 共采了河流入湖口SL1和河流下游SL2共2个样点的样品;6月25日晚, 开始下雨, 26日白天继续下零星小雨, 河水较浊, 自下而上依次采了剩下的7个样点的样品。
河流水样采集, 用新的纯净水水瓶在河中流水区域用河水涮洗3次后取1.5 L河水, 在冰袋泡沫箱中保持低温, 带回刚察青海湖裸鲤放流站实验室。用0.2 μm水系微孔滤膜抽滤, 将抽滤后的滤膜装入灭菌的50 mL离心管, -20℃保存带回武汉农业农村部淡水生物多样性保护重点实验室。用DNeasy PowerWater Kit试剂盒提取微生物DNA, 送测序公司基于Illumina HiSeq测序平台针对细菌16S rRNA扩增子(V3—V4区)进行高通量测序。
岸带土样采集, 用灭菌的50 mL离心管在岸带离水体5 m距离内的区域采取草皮下的土壤样品, 在冰袋泡沫箱中保持低温, 带回刚察青海湖裸鲤放流站实验室, -20℃冷冻处理并保存带回武汉农业农村部淡水生物多样性保护重点实验室。用DNeasy PowerSoil Kit试剂盒提取微生物DNA, 送测序公司基于Illumina HiSeq测序平台针对细菌16S rRNA扩增子(V3—V4区)进行高通量测序。
对测序所得序列(reads)进行拼接过滤, 获得优化序列(clean tags), 并进行聚类划分(0.97的相似度), 整理出OTUs(Operational Taxonomic Units), 以silva132/16s_bacteria为比对物种分类数据库进行物种注释(分类置信度0.7), 然后进行样品间OTUs种类组成的对比分析等。
1.3 流域生物信息流分析流域生物信息流可通过两个量化层面而分为两类: 第一类是数量定量, 即基于流域生态系统过程中所输移的生物信息的碱基数来定量分析, 该类(层面)的流域生物信息流的量化用信息论的量化方式来量化;第二类是种类定量, 即基于流域生态系统过程中所输移的生物信息的种类(比如OTUs、物种等)来定量分析, 该类(层面)的流域生物信息流的量化用基于种类的平行对比来量化。种类定量的流域生物信息流的量化, 用以描述流域生物信息流输出生态系统的生物组成特征及其对输入生态系统的生物组成的影响方式, 监测技术和计算方案相对简单。数量定量的流域生物信息流的量化, 用以描述流域生物信息流输出生态系统的群落结构特征及其对输入生态系统的各相关种群结构的影响能力, 对监测技术要求较高。本文的流域生物信息流探索性研究, 从相对较简单的种类定量着手。
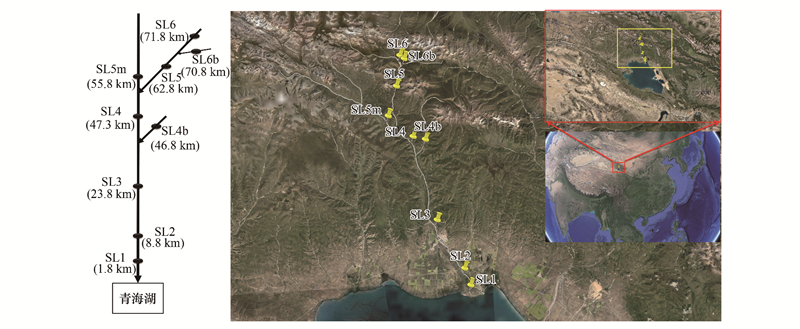
根据流域生物信息流由陆地到水体、由上游到下游的过程, 比较相关样品间的所检出生物种类组成(OTU)数据, 用覆盖度(coverage)和有效度(efficiency)来种类定量衡量流域生物信息流的基本特征。覆盖度用输入的OTU种类数(源-汇共有部分)占所输入生态系统总的OTU种类数(汇的整体部分)的比例来表示, 衡量流域生物信息流输入对汇生态系统的生物多样性检出状况的贡献和影响(公式1);有效度用输入的OTU种类数(源-汇共有部分)占输出生态系统总的可输出的OTU种类数(源的整体部分)的比例来表示, 衡量流域生物信息流所承载的生物多样性信息对汇生态系统的输入能力(公式2)。
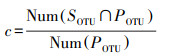
|
(1) |
|
(2) |
其中, c指流域生物信息流的覆盖度, e指流域生物信息流的有效度, SOTU指源样品(上下游流域生物信息流中的上游样品、陆水流域生物信息流中的陆地样品)的OTU种类组成, POTU指汇样品(上下游流域生物信息流中的下游样品、陆水流域生物信息流中的水体样品)的OTU种类组成, SOTU∩POTU指源-汇共有的OTU种类组成, Num(SOTU∩POTU)指源-汇共有的OTU种类数, Num(POTU)指汇的OTU种类数, Num(SOTU)指源的OTU种类数。
根据影响流域生物信息流由陆地到水体、由上游到下游过程的要素, 比较相关样品间的所检出生物种类组成(OTU)数据, 用输移能力(transport capacity)、环境过滤效应(filtration)、衰减效应(attenuation)来量化描述流域生物信息流的控制性过程。对于由陆地到水体的流域生物信息流来讲, 以同一样点的土壤样品与水体样品共有OTU种类数占该土壤样品所拥有的OTU种类总数的比例, 表征该样点岸带生物信息向水体的输移效率(即有效度, 公式2);以同一样点的土壤样品与水体样品共有OTU种类数占该土壤样品与所有水体样品共有OTU类型数的比例, 表征该样点岸带生物信息向水体的输移能力(公式3);以各土壤样品与所有水体样品共有OTU类型数占该土壤样品OTU类型总数的比例, 从对立面反映该样点岸带生物信息在输入水体过程中的环境过滤效应(公式4);流域生物信息流当中既包括有效流域生物信息流(标记有生命活性物质的生物信息的输移和传递), 也包括无效流域生物信息流(标记无生命活性物质的生物信息的输移和传递), 无效流域生物信息流在输移过程中存在自然降解衰减的过程, 这种衰减一定程度上可以反映到生物信息种类的减少, 因为水陆样点间距不超过5 m, 所以忽略衰减效应。
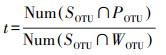
|
(3) |
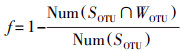
|
(4) |
其中, t指输运流域生物信息流的输移能力, f指流域生物信息流所经历的环境过滤效应, SOTU指源样品(在此指土壤样品)的OTU种类组成, POTU指汇样品的OTU种类组成, WOTU指所有水体样品整体的OTU种类组成, SOTU∩POTU指源-汇共有的OTU种类组成, SOTU∩WOTU指源-水共有的OTU种类组成Num(SOTU∩POTU)指源-汇共有的OTU种类数, Num(SOTU∩WOTU)指源-水共有的OTU种类数, Num(SOTU)指源的OTU种类数。
因为河流上游到下游的流域生物信息流输移距离较长, 无效流域生物信息流在输移过程中的自然降解导致了一定的无效流域生物信息流衰减效应, 所以需要予以考虑。由河川径流驱动的流域生物信息流其输移能力在上下游间几近一致, 因此本研究设定单位距离内的输移能力为一个常数;河流上下游之间的生态环境具有一定的一致性, 因此本研究设定基于河流水体生物信息的流域生物信息流中的无效流域生物信息流为一个常数;衰减效应随着输移过程中无效流域生物信息流的淘汰出清而由上到下逐渐减弱, 因而本研究引入半衰距离概念;样点间物理化学环境发生明显变化时会形成环境过滤效应, 环境过滤效应主要作用于有效流域生物信息流, 环境过滤效应的强弱主要依赖于物理化学环境变化的程度, 所以本研究设定特定样点间的环境过滤效应为一个常数;基于以上, 可得计算公式5。
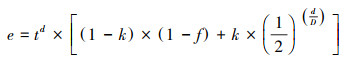
|
(5) |
其中, e指流域生物信息流累计输移效率, t指单位距离内的输移能力, k指流域生物信息流中的无效流域生物信息流占比, d指流域生物信息流的输移距离, D指无效流域生物信息流的半衰距离, f指环境过滤效应。
本研究中, 由于由样点SL3到SL2的流域生物信息流涉及采样天气状况不一致的问题, 由样点SL2到SL1的流域生物信息流涉及入湖口咸淡水交汇产生的环境过滤效应的问题, 所以以样点SL3以上的各样点间流域生物信息流数据进行基础的流域生物信息流输移能力、无效流域生物信息流占比和无效流域生物信息流半衰距离的估算, 然后与样点SL2处的流域生物信息流数据进行对比, 估算采样天气条件切换带来的影响, 与样点SL1处的流域生物信息流数据进行对比, 估算环境过滤效应。用演化算法进行规划求解, 以随机数为初始值, 进行30次计算, 然后对所得参数组进行统计。
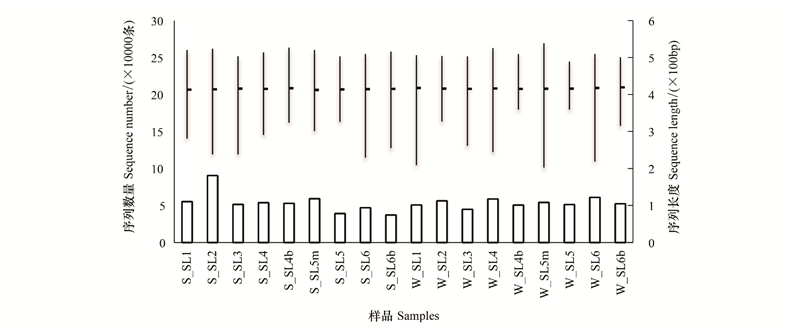
2 结果与分析 2.1 各样品的序列状况和OTU状况本实验包括9个水体微生物样品(W系列)和9个土壤微生物样品(S系列), 测序共获得优化序列968122条, 优化碱基402800844bp, 优化序列平均长度为416.06bp。各样品的序列数量和长度分布如图 2。
|
| 图 2 各样品的序列数量和长度 Fig. 2 The number and length of DNA sequences in each sample 样品以样品类型(S、W)加样点编号(SL1、SL2、SL3、SL4、SL4b、SL5m、SL5、SL6、SL6b)的形式来表示, 其中S指土壤样品, W指水体样品。比如S_SL1, 即样点SL1处的土壤样品 |
对所有样品的数据进行OTU物种注释分析, 共获得7436个OTU, 隶属于2515种、1154属、569科、328目、109纲、42门, 其中水体微生物样品中共有6356个OTU, 土壤微生物样品中共有5297个OTU。具体到各个样品, 水体微生物样品的OTU数量呈基本稳定态势, 土壤微生物样品的OTU数量则呈自下游到上游逐渐递减的态势;水体微生物样品的OTU种类整体上比土壤微生物样品的要多, 除了样点SL1和SL2(图 3)。
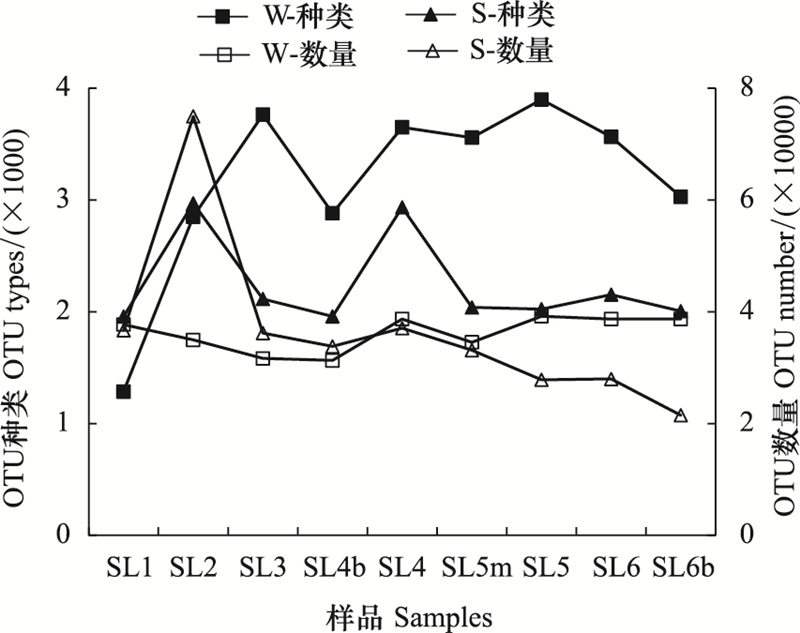
|
| 图 3 各样品的OTU数量和种类 Fig. 3 The numbers and types of OTU in each sample S指土壤样品, W指水体样品, 种类指OTU的种类数, 数量指OTU的数量总数 |
分析土壤微生物样品的OTU种类的空间差异, 即比较上下游相邻样点的土壤微生物样品OTU种类, 统计上游样点所独有的OTU种类数。结果显示(1)样点SL5m、SL5与相邻下游样点SL4之间的OTU种类差异最小, (2)下游样点与上游(流量相对较大的)干流样点之间的OTU种类差异相比于与上游(流量相对较小的)支流样点之间的OTU种类差异更小(图 4)。
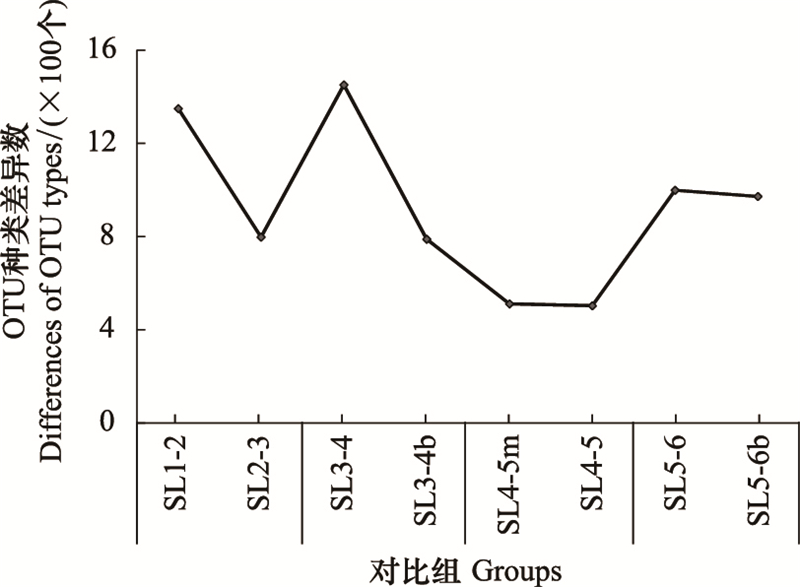
|
| 图 4 上下游相邻样点的土壤微生物样品OTU种类差异统计 Fig. 4 The differences of OTU types in the soil samples between adjacent sampling sites along the river network 上下游相邻样点间的差异, 其差异对用点位对比表示, 例如SL1-2指样点SL1和样点SL2之间的对比 |
覆盖度用以衡量流域生物信息流输入对汇生态系统的生物种类组成信息的贡献和影响;有效度用以衡量流域生物信息流所承载的生物种类组成信息对汇生态系统的有效输入能力。由土壤到水体、由上游到下游的流域生物信息流的覆盖度大都小于有效度(除了W_SL5-W_SL4这对关系和涉及SL1和SL2的2个位点的4对关系);由土壤到水体的流域生物信息流的覆盖度均小于相邻位点的由水体上游到下游的流域生物信息流的覆盖度;样点SL1和SL2(降雨前采样)的由土壤到水体的流域生物信息流的有效度小于其他7个样点(降雨期间采样)的(表 1)。
| 覆盖度 Coverage |
有效度 Effectivity |
覆盖度 Coverage |
有效度 Effectivity |
|||
| S_SL6-W_SL6 | 0.434783 | 0.719591 | S_SL4b-W_SL4b | 0.404927 | 0.595712 | |
| S_SL6b-W_SL6b | 0.312644 | 0.472084 | S_SL3-W_SL3 | 0.329702 | 0.586761 | |
| S_SL5-W_SL5 | 0.303155 | 0.583992 | W_SL4-W_SL3 | 0.667109 | 0.687757 | |
| W_SL6-W_SL5 | 0.697102 | 0.762412 | W_SL4b-W_SL3 | 0.565090 | 0.738029 | |
| W_SL6b-W_SL5 | 0.620159 | 0.798283 | S_SL2-W_SL2 | 0.460189 | 0.441602 | |
| S_SL5m-W_SL5m | 0.409219 | 0.713725 | W_SL3-W_SL2 | 0.753069 | 0.570404 | |
| S_SL4-W_SL4 | 0.580115 | 0.721635 | S_SL1-W_SL1 | 0.306915 | 0.201736 | |
| W_SL5-W_SL4 | 0.703095 | 0.658374 | W_SL2-W_SL1 | 0.689200 | 0.311119 | |
| W_SL5m-W_SL4 | 0.746371 | 0.765880 | ||||
| 流域生物信息流用样品间的对比来表示。例如S_SL6-W_SL6指在样点SL6处由岸带土壤到河流水体的流域生物信息流;W_SL6-W_SL5指从样点SL6到样点SL5的由上游到下游的水体中的流域生物信息流 | ||||||
降雨天(中上游7个样点), 由陆地到水体的流域生物信息流的输移效率为62.76%左右(0.627643±0.087327), 输移能力为68.49%左右(0.684876±0.091302), 环境过滤效应为8.38%左右(0.083816±0.020574), 因为水陆样点间距不超过5m, 所以忽略其衰减效应。各样点由陆地到水体的流域生物信息流的环境过滤效应、输移能力、输移效率具体见表 2。
| 环境过滤效应 Filtration |
输移能力 Capacity |
输移效率 Efficiency |
环境过滤效应 Filtration |
输移能力 Capacity |
输移效率 Efficiency |
|||
| S_SL6-W_SL6 | 0.048747 | 0.756467 | 0.719591 | S_SL4b-W_SL4b | 0.107198 | 0.667238 | 0.595712 | |
| S_SL6b-W_SL6b | 0.064307 | 0.504529 | 0.472084 | S_SL3-W_SL3 | 0.095981 | 0.649059 | 0.586761 | |
| S_SL5-W_SL5 | 0.109190 | 0.655574 | 0.583992 | S_SL2-W_SL2 | 0.222821 | 0.568211 | 0.441602 | |
| S_SL5m-W_SL5m | 0.080882 | 0.776533 | 0.713725 | S_SL1-W_SL1 | 0.216037 | 0.257329 | 0.201736 | |
| S_SL4-W_SL4 | 0.080409 | 0.784735 | 0.721635 |
由上到下的流域生物信息流, 以各干支流最上游样点为起点, 沿河川径流方向由上到下, 共4条流域生物信息流连续输移线。计算各河流上游到下游的流域生物信息流路径的点间输移效率和累计输移效率(表 3), 该效率是上下游样点区域间输移能力、衰减效应、环境过滤效应共同作用的结果。
| 点间输移效率 Efficiency |
点间径流距离 Distance/km |
累计输移效率 Efficiency_cum |
累计径流距离 Distance_cum/km |
||
| A | SL6-SL5 | 0.762412 | 9 | 0.762412 | 9 |
| ***-SL4 | 0.774467 | 15.5 | 0.590463 | 24.5 | |
| ***-SL3 | 0.876010 | 23.5 | 0.517251 | 48 | |
| ***-SL2 | 0.800434 | 15 | 0.414025 | 63 | |
| ***-SL1 | 0.464092 | 7 | 0.192146 | 70 | |
| B | SL6b-SL5 | 0.798283 | 8 | 0.798283 | 8 |
| ***-SL4 | 0.669975 | 15.5 | 0.534830 | 23.5 | |
| ***-SL3 | 0.910494 | 23.5 | 0.486959 | 47 | |
| ***-SL2 | 0.787119 | 15 | 0.383295 | 62 | |
| ***-SL1 | 0.515073 | 7 | 0.197425 | 69 | |
| C | SL5m-SL4 | 0.765880 | 8.5 | 0.765880 | 8.5 |
| ***-SL3 | 0.757064 | 23.5 | 0.579820 | 32 | |
| ***-SL2 | 0.772661 | 15 | 0.448004 | 47 | |
| ***-SL1 | 0.439147 | 7 | 0.196740 | 54 | |
| D | SL4b-SL3 | 0.738029 | 23 | 0.738029 | 23 |
| ***-SL2 | 0.753173 | 15 | 0.555864 | 38 | |
| ***-SL1 | 0.426966 | 7 | 0.237335 | 45 | |
| 4条流域生物信息流输移链分别用A、B、C、D来排号, 输移链用样点的自上而下顺序排列来表示; 比如A, 表示第一条流域生物信息流输移链;SL6b-SL5, 表示从样点SL6b到样点SL5的流域生物信息流;***-SL4, 表示前一样点/环节到样点SL4的流域生物信息流 | |||||
由上到下的流域生物信息流, 其输移能力约为99.42%(0.994245±0.000941) km, 其无效流域生物信息流占比约为43.46%(0.434635±0.041681), 无效流域生物信息流的半衰距离约为14.52(14.523377±1.440539)km, 样点SL3和SL2的采样天气条件切换(上游雨下游晴)带来的环境过滤效应约为0.57%(0.005687±0.005450), 河口样点SL1处的半咸水所产生的环境过滤效应约为54.42%(0.544164±0.010042)。根据由上游到下游的流域生物信息流计算公式, 不考虑样点SL2、SL1的环境过滤效应, 基础综合流域生物信息流输移有效度约为97.41%(0.974105±0.000926) km。
比较干支流交汇处上下游的3个样点中的4个样品的OTU种类, 共3组, 结果显示, W_SL6对W_SL5的覆盖度比W_SL6b对W_SL5的覆盖度更大, W_SL5m对W_SL4的覆盖度比W_SL5对W_SL4的覆盖度更大, W_SL4对W_SL3的覆盖度比W_SL4b对W_SL3的覆盖度更大;在3组样品的OTU种类比较中, W_SL4、W_SL5、W_SL3三个样品中的独有OTU种类占比依次增加, 即来自上游2个样点到本样点(W_SL4、W_SL5、W_SL3)间汇水区的流域生物信息流特异性依次增强(表 4)。
| 覆盖度 Coverage |
河流径流距离 Distance |
流量比较 Compare of fluxes |
覆盖度 Coverage |
河流径流距离 Distance |
流量比较 Compare of fluxes |
|||
| W_SL6-W_SL5 | 0.697102 | 9 km | SL6 > SL6b | S_SL4-W_SL4 | 0.580115 | |||
| W_SL6b-W_SL5 | 0.620159 | 8 km | W_SL4_Only | 0.080526 | ||||
| S_SL5-W_SL5 | 0.303155 | W_SL4-W_SL3 | 0.667109 | 23.5 km | SL4 > SL4b | |||
| W_SL5_Only | 0.132855 | W_SL4b-W_SL3 | 0.565090 | 23 km | ||||
| W_SL5-W_SL4 | 0.703095 | 15.5 km | SL5 < SL5m | S_SL3-W_SL3 | 0.329702 | |||
| W_SL5m-W_SL4 | 0.746371 | 8.5 km | W_SL3_Only | 0.229277 | ||||
| W_SL5_Only指不来自于样品W_SL6、W_SL6b和S_SL5的OTU, 其主要来自支流样点SL6、SL6b至样点SL5河段的汇水区向样点SL5的水体输入的流域生物信息流 | ||||||||
流域生物信息流是流域生态系统过程的重要组成部分, 包括有效流域生物信息流和无效流域生物信息流(图 5)。在自然径流驱动的流域生态系统过程中, 物质、能量、信息都沿着由陆地到水体、由支流到干流、由上游到下游的方向进行流动[22-23]。对下游样点的水体环境DNA进行检测, 可以了解上游区域的生物群落组成情况[12, 24];对水体样点的环境DNA进行检验, 可以了解流域内陆地上生物群落组成情况[11, 25]。对于宏体生物来讲, 以环境DNA形式从陆地到水体、从支流到干流、从上游到下游的流域生物信息流往往只是其组织碎片或者游离细胞, 甚至裸露的DNA[24, 26-28]。对于微生物来讲, 以环境DNA形式从陆地到水体、从支流到干流、从上游到下游的流域生物信息流往往既包含活的生物个体或细胞, 也有死的生物个体或细胞[29-30]。宏体生物的组织碎片、游离细胞和裸露DNA, 以及微生物的死亡个体和细胞, 因为不再具有生命活性, 相应地其承载的流域生物信息流都是无效流域生物信息流, 因而对输入地生态系统的生物种类组成不会产生明显的影响。宏体生物个体甚至种群, 以及微生物的存活个体和细胞, 因为具有生命活性, 相应地其承载的流域生物信息流(比如鱼类洄游[31-32], 水生生物入侵[33-34]等)属于有效流域生物信息流, 因而对输入地生态系统的生物种类组成及群落结构会产生较明显的影响。
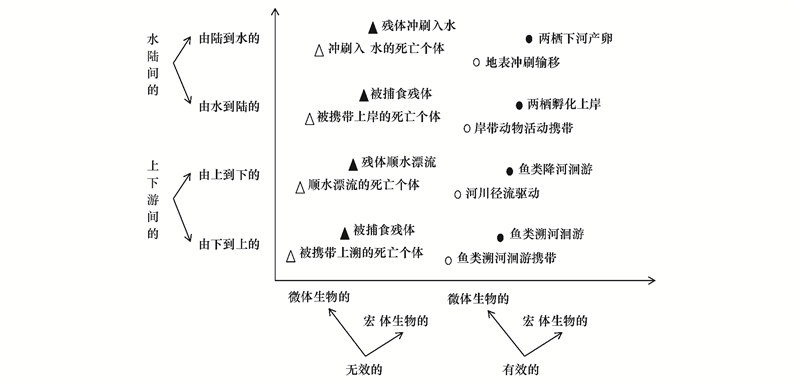
|
| 图 5 流域生物信息流的类型 Fig. 5 Types of watershed biological information flows 在环境DNA技术应用中微体生物指可以将整个个体甚至群落作为环境DNA样品进行采集、处理、提取、测序、分析的生物, 宏体生物指只能将个体身上脱落的微小的组织碎片、少量细胞、游离DNA作为环境DNA样品进行采集、处理、提取、测序、分析的生物 |
有效流域生物信息流在一定程度上增加了输入地的生物多样性。对于宏体生物来讲, 周期性的生物迁入能够增加迁入地的生物多样性, 比如一些山间河流和季节性河流, 洄游产卵亲鱼和季节性迁移水生生物的到来, 在一定程度上增加了该河流的生物多样性[35]。对于微生物来讲, 由上游和由陆地输入的有效流域生物信息流能够在一定程度上增加输入地的微生物生物多样性。本研究中, 同一样点的岸带土壤微生物和河流水体微生物在种类组成上有一定程度的重合(表 1), 可以认为绝大多数是陆地土壤对河流水体的微生物输入, 虽然不能排除由人类、家畜、陆生野生动物等的取水、饮水、涉河等活动将一些水体微生物带到岸带土壤中, 但因为其在整个流域生态系统过程中所占份额远不及自然径流所驱动的由陆到水的流域生态系统过程所占份额, 所以其绝对不是主要的部分。上下游相邻样点间的水体微生物在种类组成上有一定程度的重合(表 1), 可以认为绝大多数是上游对下游的微生物输入, 虽然不能排除由水生野生动物的溯河迁移等活动将一些下游水体微生物带到上游, 但因为其在整个流域生态系统过程中所占份额远不及自然径流所驱动的由上游到下游的流域生态系统过程所占份额, 所以其绝对不是主要的部分。
流域生物信息流对输入地的生物多样性的增加是非累积的。本研究中, 由土壤到水体、由上游到下游的微生物群落组成覆盖度普遍小于有效度(除了W_SL5-W_SL4这对关系和涉及SL1和SL2的2个位点的4对关系)(表 1), 也就是说, 多来源的流域生物信息流的输入使得汇生态系统的可检出生物多样性获得增长, 且大于任一源生态系统的生物多样性。Deiner等曾提出一个描述生物多样性、环境DNA与河流等级之间关系的概念模型[11], 涉及这一问题。其假设环境DNA所反映的物种丰富度随着河流的Strahler等级的增大而不断升高最终达到稳定, 但对空间尺度适用范围并没有给出一个说明和限定[11]。本研究中, 虽然从上游到下游每个样点水体微生物生物多样性都受流域生物信息流的输入影响而增长, 但从这一系列样点的OTU种类数来看, 并没有出现Deiner等的模型所预期的持续增长最终达到稳定的情况, 而是呈现为OTU种类基本稳定(除了样点SL1和SL2), 且从上游到下游的流域生物信息流在输移过程中存在相当程度的丢失(图 3), 每个样点的水体微生物多样性相当程度上依赖于其上游紧邻的有限长度河段汇水区的微生物多样性。而决定这一河段长度的是流域生物信息流的输移效率。这一规律对于水体环境DNA技术应用来讲, 尤其重要。
流域生物信息流对输入地的生物多样性影响的大小, 依赖于各个源生态系统的总生物信息量(在本研究中指种类)和流域生物信息流的输移效率。因为土壤微生物样品的OTU种类整体上比水体微生物样品的要少(图 3), 由土壤到水体(同一样点的两种样品间比较)的流域生物信息流输移效率普遍小于由上游到下游(相邻样点间比较)的流域生物信息流输移效率(表 1), 导致由土壤到水体的流域生物信息流普遍小于由上游到下游的流域生物信息流, 所以由土壤到水体的流域生物信息流对输入地生态系统的影响也普遍小于由上游到下游的流域生物信息流对输入地生态系统的影响, 反映在指标上即, 由土壤到水体的微生物组成覆盖度普遍小于由水体上游到下游的相邻位点的微生物组成覆盖度(表 1)。
3.2 流域生物信息流的输移效率及其影响因素输移效率和输移距离是流域生物信息流过程的两个关键指标。由上游到下游、由支流到干流的流域生物信息流在环境DNA监测的视角被探讨得比较多[12, 24], 但其输移效率究竟有多大?输移距离究竟有多远?目前并尚没有系统的研究, 输移效率的估算只有少量研究涉及[12, 36];输移距离的估算, 只有一些针对宏体生物的环境DNA可监测距离的研究, 估算出的距离从小几十米[37-38], 到几百米、数千米[36, 39], 再到上百千米[40]都有。当然, 已有的这些都是无效流域生物信息流的输移距离和输移效率, 而有效流域生物信息流的输移距离和输移效率则未见相关研究。由陆地到水体的流域生物信息流也在环境DNA监测的视角有所探讨[11, 25], 其输移效率有多大?输移距离有多远?相关研究更为稀少, 虽然Deiner等提出了一个描述生物多样性、环境DNA与河流等级之间关系的概念模型, 以及一个只用几个水体环境DNA样品就能评估流域层面的生物多样性状况的愿景[11], 但是, 并没有量化分析输移效率和输移距离的问题。
本研究中, 由土壤到水体的流域生物信息流, 在雨天, 其输移效率为62.76%左右(中上游7个样点, 降雨第二天采样, 且采样时伴有零星小雨);在晴天, 其输移效率约为44.16%(样点SL2, 降雨前采样), 而河流入湖口样点SL1由于受青海湖咸水胁迫约束, 其输移效率约为20.17%(降雨前采样)(表 1)。需要强调说明的是, 这里的输移效率是河流水体5米距离内岸带的样点到邻近水体的流域生物信息流输移效率。在雨天, 由河流上游到下游的流域生物信息流, 其基础综合输移效率为97.41%/km(中上游7个样点之间, 降雨第二天采样, 且采样时伴有零星小雨);虽然样点SL2是在降雨前采样, SL3是在降雨天采样, SL3到SL2的流域生物信息流涉及到晴/雨条件转换的问题, 但其环境过滤效应仅为约0.57%, 因而其输移效率约为96.32%/km, 也就是说晴雨条件并不对由河流上游到下游的流域生物信息流输移产生明显;由于样点SL2和上游样点一样是淡水环境, 河流入湖口样点SL1则是咸淡水交汇区, 并且样点SL1和SL2都是在晴天采的样, 所以由SL2输入到SL1的有效流域生物信息流主要受半咸水环境胁迫问题的影响(环境过滤效应约为54.42%), 其输移效率约为84.63%/km, 也就是说水体理化条件对由河流上游到下游的流域生物信息流输移产生了明显影响。
由陆地到水体的流域生物信息流主要受地表表面流和地下潜流驱动, 同时还受由陆到水的环境转变所产生的环境过滤效应的影响;由河流上游到下游的流域生物信息流主要受河川径流驱动, 同时还受远距离输移过程中的衰减效应的影响(图 6)。由陆地到水体、由上游到下游、由支流到干流的流域生态系统过程(包括流域生物信息流)主要由自然径流(包括地表表面流、地下潜流、河川径流)所驱动[22-23]。因为(1)地表表面流和地下潜流远不及河道径流对颗粒物和碎屑的携带能力大, 其所驱动的由土壤到水体的流域生物信息流输移能力也小于由上游到下游的流域生物信息流输移能力, (2)陆-水环境转变所产生的环境过滤效应比上-下游输移过程中的衰减效应强, 所以导致了由土壤到水体的流域生物信息流输移效率也小于由上游到下游的流域生物信息流输移效率, 即由土壤到水体的微生物组成有效度普遍小于由水体上游到下游的相邻位点的微生物组成有效度(表 1)。
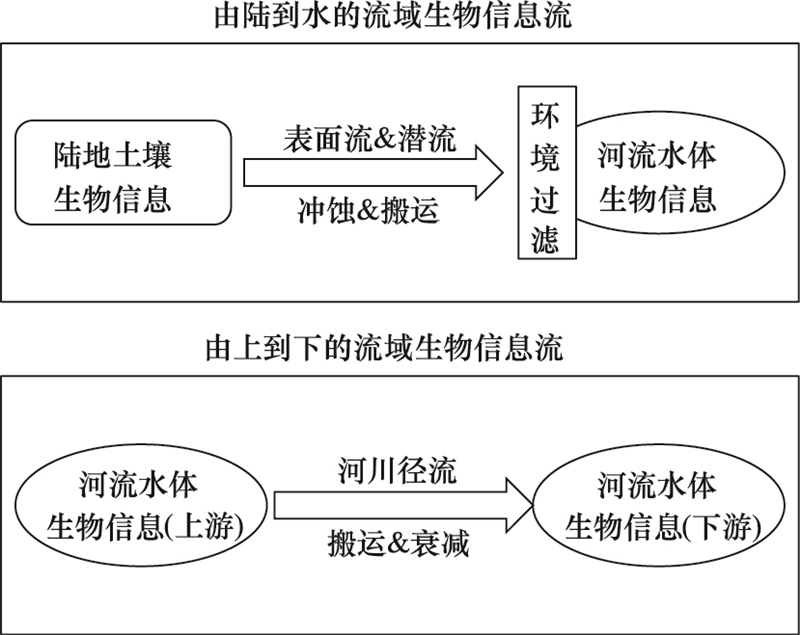
|
| 图 6 自然径流驱动下的两种流域生物信息流的驱动模式和输移过程 Fig. 6 The diagram of two types of watershed biological information flow driven by runoff |
本研究中, 由土壤到水体的流域生物信息流, 在雨天(大量地表碎屑被侵蚀、搬运而冲入河道, 河水浑浊), 输移能力为68.49%左右(即约68.49%的生物信息能从陆地上输移到水体里), 环境过滤效应为8.38%左右(即约8.38%的生物信息流在陆水界面切换时被过滤掉), 进而其输移效率为62.76%左右;而在晴天, 输移能力则降为约56.82%, 环境过滤效应升为约22.28%, 进而其输移效率降为约44.16%(表 2)。由河流上游到下游的流域生物信息流, 输移能力约为99.42%/km, 其中无效流域生物信息流比例约为43.46%, 无效流域生物信息流的半衰距离约为14.52 km, 其基础综合输移效率为97.41%/km左右。
降雨促使地表表面流、地下潜流和河道径流增加, 增加了对颗粒物和碎屑的输移能力, 进而驱动了由陆地到水体的流域生物信息流输移能力的增大, 同时也通过颗粒物营造的微环境减弱了陆-水环境切换带来的环境过滤效应[41], 进而增大了流域生物信息流的输移效率。本研究中, 由岸带土壤到河流水体的流域生物信息流, 在雨天, 输移能力为68.49%左右, 环境过滤效应为8.38%左右, 其输移效率为62.76%左右;在晴天, 输移能力约为56.82%, 环境过滤效应约为22.28%, 其输移效率约为44.16%(表 2)。雨天的输移能力显著大于晴天的输移能力, 雨天的环境过滤效应显著小于晴天的环境过滤效应, 进而导致雨天的输移效率显著大于晴天的输移效率。
有效流域生物信息流的影响力主要与径流强度相关, 而非与河流径流距离相关。在由上游到下游的流域生物信息流中, 径流强度越大, 流域生物信息流输移能力往往也越大, 对汇生态系统影响的程度往往也越大。本研究中, 对3组干支流交汇处上下游的3个样点的水体微生物样品的OTU种类的对比统计分析显示, 河川径流量大的样点输出的流域生物信息流对下游生态系统的影响(覆盖度)更大, 而非距离近的样点(表 4)。对于无效流域生物信息流来讲, 因为受环境DNA的降解、沉淀、被吸附等的限制, 其往往受河流径流距离的影响[12, 36, 42]。对于有效流域生物信息流来讲则不受这些因子的影响, 因而有效流域生物信息流对汇生态系统影响的程度也就与河流径流距离没有明确的相关关系。但这个判断只在一定空间尺度内(比如本研究中河流径流距离在几千米至小几十千米范围内)可检验, 尺度太小就会受无效流域生物信息流的影响, 尺度太大就会受水环境状况异质性和区间汇水区生物组成异质性的影响, 进而都会与河流径流距离存在一定的相关性。在本研究的3组干支流交汇处上下游的3个样点中的4个样品OTU种类的比较中, 3个下游样点的水体微生物样品中的独有OTU种类占比的大小(表 4), 反映了来自相邻2个上游样点到下游样点间汇水区的流域生物信息流的特异性强弱, 而流域生物信息流的特异性强弱基本依赖于汇水区内微生物群落相比于前后样点的特异性大小(图 4)。在大空间尺度上来讲, 越远的空间距离往往也意味着汇水区内越大的群落差异, 进而也就意味着上游样点对下游样点越小的流域生物信息流影响力。
4 结论本文根据流域生态学发展的需要和流域生态系统过程研究的需要, 提出了流域生物信息流概念, 并对青藏高原沙柳河流域的一系列水体微生物和土壤微生物样品进行了分析, 从生物信息种类组成层面定量研究了陆地到水体、支流到干流、上游到下游的流域生物信息流。研究结果显示: ①流域生物信息流在一定程度上增加了输入地的可检出生物多样性, 但这种增加是非累积的, 依赖于样点上游紧邻的有限长度河段汇水区的生物多样性和流域生物信息流的输移效率;②降雨导致了地表表面流的产生、地下潜流和河道径流的增加, 增强了对颗粒物和碎屑的冲蚀和搬运能力, 进而导致了流域生物信息流的输移能力增大、环境过滤效应减弱、输移效率增大。
目前迅速发展的环境DNA在水生态中的应用[11-12, 24], 其理论基础之一就是流域生物信息流。在目前的水体环境DNA研究中, 既涉及由上游到下游、由支流到干流的流域生物信息流[12, 24], 也涉及由陆地到水体的流域生物信息流[11, 25], 但目前做的最多的、关注最多的是由上游到下游、由支流到干流的流域生物信息流, 既涉及无效流域生物信息流[11], 也涉及有效流域生物信息流[25], 但关注最多研究最多的是无效流域生物信息流。本文中对这几类流域生物信息流均做了相关的厘定和探讨。另外, 本文还初步提及了由下游到上游、由水体到岸上的流域生物信息流。但在本案例研究中, 暂未实质性区分有效生物信息流和无效生物信息流, 暂未区分鱼类(青海湖裸鲤)洄游所驱动的流域生物信息流, 暂未区分放牧(人、羊、牦牛、马、狗等)和野生动物(鼠兔、鸟类、狐狸等)活动对流域生物信息流的影响, 暂未区分沙尘输移(春季的大风沙)对流域生物信息流的影响, 这些都需要下一步进行更为深入的研究。
| [1] |
Lotspeich F B. Watersheds as the basic ecosystem: this conceptual framework provides a basis for a natural classification system. Journal of the American Water Resources Association, 1980, 16(4): 581-586. DOI:10.1111/j.1752-1688.1980.tb02434.x |
| [2] |
Schindler D W, Newbury R W, Beaty K G, Campbell P. Natural water and chemical budgets for a small Precambrian lake basin in central Canada. Journal of the Fisheries Research Board of Canada, 1976, 33(11): 2526-2543. DOI:10.1139/f76-297 |
| [3] |
Hornbeck J W, Swank W T. Watershed ecosystem analysis as a basis for multiple-use management of eastern forests. Ecological Applications, 1992, 2(3): 238-247. DOI:10.2307/1941858 |
| [4] |
赵斌. 流域是生态学研究的最佳自然分割单元. 科技导报, 2014, 32(1): 12-12. |
| [5] |
杨海乐, 陈家宽. 流域生态学的发展困境——来自河流景观的启示. 生态学报, 2016, 36(10): 3084-3095. |
| [6] |
蔡庆华, 吴刚, 刘建康. 流域生态学: 水生态系统多样性研究和保护的一个新途径. 科技导报, 1997, 15(9705): 24-26. |
| [7] |
Jacob Kalff, 古滨河, 刘文正, 李宽意. 湖沼学: 内陆水生态系统. 北京: 高等教育出版社, 2011.
|
| [8] |
Smits A P, Schindler D E, Brett M T. Geomorphology controls the trophic base of stream food webs in a boreal watershed. Ecology, 2015, 96(7): 1775-1782. DOI:10.1890/14-2247.1 |
| [9] |
Childress E S, McIntyre P B. Life history traits and spawning behavior modulate ecosystem-level effects of nutrient subsidies from fish migrations. Ecosphere, 2016, 7(6): e1301. |
| [10] |
Holtgrieve G W, Schindler D E. Marine-derived nutrients, bioturbation, and ecosystem metabolism: reconsidering the role of salmon in streams. Ecology, 2011, 92(2): 373-385. DOI:10.1890/09-1694.1 |
| [11] |
Deiner K, Fronhofer E A, Mächler E, Walser J, Altermatt F. Environmental DNA reveals that rivers are conveyer belts of biodiversity information. Nature Communications, 2016, 7: 12544. DOI:10.1038/ncomms12544 |
| [12] |
Carraro L, Hartikainen H, Jokela J, Bertuzzo E, Rinaldo A. Estimating species distribution and abundance in river networks using environmental DNA. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(46): 11724-11729. DOI:10.1073/pnas.1813843115 |
| [13] |
Liu T, Zhang A N, Wang J W, Liu S F, Jiang X T, Dang C Y, Ma T, Liu S T, Chen Q, Xie S Q, Zhang T, Ni J R. Integrated biogeography of planktonic and sedimentary bacterial communities in the Yangtze River. Microbiome, 2018, 6(1): 16. DOI:10.1186/s40168-017-0388-x |
| [14] |
Chen J, Wang P F, Wang C, Wang X, Miao L Z, Liu S, Yuan Q S. Bacterial communities in riparian sediments: a large-scale longitudinal distribution pattern and response to dam construction. Frontiers in Microbiology, 2018, 9: 999. DOI:10.3389/fmicb.2018.00999 |
| [15] |
Zhou Y, Deng X F, Zang N, Li H T, Li G, Li C P, He M. Transcriptomic and proteomic investigation of HSP90A as a potential biomarker for HCC. Medical Science Monitor, 2015, 21: 4039-4049. DOI:10.12659/MSM.896712 |
| [16] |
Zhao Q Y. Nonalgorithmicity and algorithmicity of protein science. Advances in Bioscience and Biotechnology, 2011, 2(5): 340-346. DOI:10.4236/abb.2011.25050 |
| [17] |
钱韦, 陈晓亚, 方荣祥, 康乐. 生物信息流的人工操纵——作物病虫害导向性防控的新科学. 中国科学: 生命科学, 2017, 47(9): 889-892. |
| [18] |
钱韦, 曲静, 康乐. 生物信息流操纵: 作物病虫害导向性防控的新科学. 中国科学院院刊, 2017, 32(8): 805-813. |
| [19] |
Tero N, Aspi J, Siikamäki P, Jäkäläniemi A, Tuomi J. Genetic structure and gene flow in a metapopulation of an endangered plant species, Silene tatarica. Molecular Ecology, 2003, 12(8): 2073-2085. DOI:10.1046/j.1365-294X.2003.01898.x |
| [20] |
Farkas T E, Mononen T, Comeault A A, Nosil P. Observational evidence that maladaptive gene flow reduces patch occupancy in a wild insect metapopulation. Evolution, 2016, 70(12): 2879-2888. DOI:10.1111/evo.13076 |
| [21] |
杨海乐. 流域生态学的理论研究和案例分析[D]. 上海: 复旦大学, 2018: 1-216.
|
| [22] |
Willett S D, McCoy S W, Perron J T, Goren L, Chen C Y. Dynamic reorganization of river basins. Science, 2014, 343(6175): 1248765. DOI:10.1126/science.1248765 |
| [23] |
Ward N D, Keil R G, Medeiros P M, Brito D C, Cunha A C, Dittmar T, Yager P L, Krusche A V, Richey J E. Degradation of terrestrially derived macromolecules in the Amazon River. Nature Geoscience, 2013, 6(7): 530-533. DOI:10.1038/ngeo1817 |
| [24] |
Shogren A J, Tank J L, Egan S P, Bolster D, Riis T. Riverine distribution of mussel environmental DNA reflects a balance among density, transport, and removal processes. Freshwater Biology, 2019, 64(8): 1467-1479. DOI:10.1111/fwb.13319 |
| [25] |
Matsuoka S, Sugiyama Y, Sato H, Katano I, Harada K, Doi H. Spatial structure of fungal DNA assemblages revealed with eDNA metabarcoding in a forest river network in western Japan. Metabarcoding and Metagenomics, 2019, 3: e36335. DOI:10.3897/mbmg.3.36335 |
| [26] |
Valentini A, Taberlet P, Miaud C, Civade R, Herder J, Thomsen P F, Bellemain E, Besnard A, Coissac E, Boyer F, Gaboriaud C, Jean P, Poulet N, Roset N, Copp G H, Geniez P, Pont D, Argillier C, Baudoin J M, Peroux T, Crivelli A J, Olivier A, Acqueberge M, Le Brun M, Møller P R, Willerslev E, Dejean T. Next-generation monitoring of aquatic biodiversity using environmental DNA metabarcoding. Molecular Ecology, 2016, 25(4): 929-942. DOI:10.1111/mec.13428 |
| [27] |
Thomsen P F, Willerslev E. Environmental DNA-An emerging tool in conservation for monitoring past and present biodiversity. Biological Conservation, 2015, 183: 4-18. DOI:10.1016/j.biocon.2014.11.019 |
| [28] |
Taberlet P, Coissac E, Hajibabaei M, Rieseberg L H. Environmental DNA. Molecular Ecology, 2012, 21(8): 1789-1793. DOI:10.1111/j.1365-294X.2012.05542.x |
| [29] |
Bass D, Stentiford G D, Littlewood D T J, Hartikainen H. Diverse applications of environmental DNA methods in parasitology. Trends in Parasitology, 2015, 31(10): 499-513. DOI:10.1016/j.pt.2015.06.013 |
| [30] |
Sengupta M E, Hellström M, Kariuki H C, Olsen A, Thomsen P F, Mejer H, Willerslev E, Mwanje M T, Madsen H, Kristensen T K, Stensgaard A S, Vennervald B J. Environmental DNA for improved detection and environmental surveillance of schistosomiasis. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(18): 8931-8940. DOI:10.1073/pnas.1815046116 |
| [31] |
Schindler D E. The phenology of migration in an unpredictable world. Journal of Animal Ecology, 2019, 88(1): 8-10. DOI:10.1111/1365-2656.12937 |
| [32] |
Lemopoulos A, Uusi-Heikkilä S, Huusko A, Vasemägi A, Vainikka A. Comparison of migratory and resident populations of brown trout reveals candidate genes for migration tendency. Genome Biology and Evolution, 2018, 10(6): 1493-1503. DOI:10.1093/gbe/evy102 |
| [33] |
Havel J E, Kovalenko K E, Thomaz S M, Amalfitano S, Kats L B. Aquatic invasive species: challenges for the future. Hydrobiologia, 2015, 750(1): 147-170. DOI:10.1007/s10750-014-2166-0 |
| [34] |
Gallardo B, Aldridge D C. Inter-basin water transfers and the expansion of aquatic invasive species. Water Research, 2018, 143: 282-291. DOI:10.1016/j.watres.2018.06.056 |
| [35] |
Tolonen K E, Picazo F, Vilmi A, Datry T, Stubbington R, Pařil P, Rocha M P, Heino J. Parallels and contrasts between intermittently freezing and drying streams: from individual adaptations to biodiversity variation. Freshwater Biology, 2019, 64(10): 1679-1691. DOI:10.1111/fwb.13373 |
| [36] |
Tillotson M D, Kelly R P, Duda J J, Hoy M, Kralj J, Quinn T P. Concentrations of environmental DNA (eDNA) reflect spawning salmon abundance at fine spatial and temporal scales. Biological Conservation, 2018, 220: 1-11. DOI:10.1016/j.biocon.2018.01.030 |
| [37] |
Pilliod D S, Goldberg C S, Arkle R S, Waits L P. Factors influencing detection of eDNA from a stream-dwelling amphibian. Molecular Ecology Resources, 2014, 14(1): 109-116. DOI:10.1111/1755-0998.12159 |
| [38] |
Stoeckle B C, Kuehn R, Geist J. Environmental DNA as a monitoring tool for the endangered freshwater pearl mussel (Margaritifera margaritifera L.): a substitute for classical monitoring approaches?. Aquatic Conservation: Marine and Freshwater Ecosystems, 2016, 26(6): 1120-1129. DOI:10.1002/aqc.2611 |
| [39] |
Deiner K, Altermatt F. Transport distance of invertebrate environmental DNA in a natural river. PLoS One, 2014, 9(2): e88786. DOI:10.1371/journal.pone.0088786 |
| [40] |
Pont D, Rocle M, Valentini A, Civade R, Jean P, Maire A, Roset N, Schabuss M, Zornig H, Dejean T. Environmental DNA reveals quantitative patterns of fish biodiversity in large rivers despite its downstream transportation. Scientific Reports, 2018, 8: 10361. DOI:10.1038/s41598-018-28424-8 |
| [41] |
Wilpiszeski R L, Aufrecht J A, Retterer S T, Sullivan M B, Graham D E, Pierce E M, Zablocki O D, Palumbo A V, Elias D A. Soil aggregate microbial communities: towards understanding microbiome interactions at biologically relevant scales. Applied and Environmental Microbiology, 2019, 85(14): e00324-19. |
| [42] |
Shogren A J, Tank J L, Andruszkiewicz E, Olds B, Mahon A R, Jerde C L, Bolster D. Controls on eDNA movement in streams: transport, retention, and resuspension. Scientific Reports, 2017, 7: 5065. DOI:10.1038/s41598-017-05223-1 |
 2021, Vol. 41
2021, Vol. 41