文章信息
- 孙雨, 常晶晶, 田春杰
- SUN Yu, CHANG Jingjing, TIAN Chunjie
- 根际微生物组中细菌趋化系统的生态功能
- Ecological functions of the bacterial chemotaxis systems in rhizosphere microbiome
- 生态学报. 2021, 41(24): 9963-9969
- Acta Ecologica Sinica. 2021, 41(24): 9963-9969
- http://dx.doi.org/10.5846/stxb202009292520
-
文章历史
- 收稿日期: 2020-09-29
- 网络出版日期: 2021-08-04
根际作为土壤与植物间物质交换的活跃界面, 其物理、化学以及生物学特性与周围土壤都存在着显著差异[1]。在根际这一特殊的生态环境中, 与植物根系相关的微生物种类和数量巨大[2]。根际微生物在植物的生长发育以及抵抗逆境胁迫方面都扮演着重要的角色[3], 包括促进植物对营养元素的获取及利用[4-5]、调控植物的生长和环境适应性[6-7]等。根际微生物组与非根际土壤微生物组在物种多样性和丰富度方面均存在显著差异[8], 其中植物根系的调控作用是决定根际微生物组群落结构的重要因素之一[9]。因此, 深入揭示植物对根际微生物组的选择机制不仅可以在理论方面增进对植物-微生物互作关系的认识, 还能在实际应用中指导根际微生物组的改良与重组, 对未来农业生产具有重大意义。
植物调控根际微生物组的重要策略之一是通过根系分泌化合物, 选择性招募特定根际微生物[10-11]。在这一过程中, 土壤微生物通过何种机制来感应根系分泌物, 响应植物的招募?细菌的趋化反应给出了一种答案。土壤中存在一类趋化细菌, 能够利用自身独特的趋化系统感应其生存环境中存在的化学物质浓度梯度, 通过一系列信号转导过程调控细菌的运动方向, 使细菌趋近吸引物而趋离排斥物[12-13]。趋化细菌在没有化合物浓度梯度的环境中, 其运动方向是随机的, 向前直线运动数秒后停下“翻筋斗”, 然后再以不同的方向继续直线运动, 以“直线运动-转变方向-直线运动”这一过程循环往复[14];当周围环境中存在吸引物(碳源等营养物)的浓度梯度时, 趋化细菌转变方向的频率降低, 能够延长向高浓度区直线运动的时间, 总体表现为细菌趋近吸引物[15]。
细菌的趋化反应是其对碳源、能源竞争的一种表现, 体现了细菌的主动适应性。Wuichet和Zhulin[16]基于全基因组数据, 对近400株细菌的趋化系统进行了分析, 推测绝大部分的细菌可能都具有趋化性。但是, 目前针对细菌趋化系统的研究主要集中在分子层面对其信号转导和作用机理的解析, 缺少从生态学的角度揭示其在植物根际微生物群落结构和功能中的表现和作用。本文以根际微生物组中的细菌趋化系统为核心, 针对国内外最新研究进展, 从以下三个方面进行综述, 提出了研究根际细菌趋化系统生态功能的具体方向, 旨在阐明根际细菌趋化系统的生态功能, 增进对根际微生物组的募集组装过程的认知, 以及为根际微生物组的重组构建提供理论依据。
1 细菌趋化系统的组成和信号转导机制细菌的趋化反应是在长期进化过程中形成的“趋利避害”的本能, 研究人员以大肠杆菌(Escherichia coli)[17]、芽孢杆菌(Bacillus spp.)[18]、假单胞菌(Pseudomonas spp.)[19]等为对象, 对细菌的趋化反应进行了系统深入的研究。细菌的趋化系统是原核生物中最为复杂的信号转导系统之一, 虽然不同细菌中趋化系统的组成有一定差异, 但是其信号转导及调控机制在所有细菌中是相对保守的[15]。
细菌趋化系统由以下一系列不同功能的蛋白组成:趋化受体蛋白Mcps(Methyl-accepting Chemotaxis Proteins)、支架蛋白CheW/CheV、组氨酸激酶CheA、反应调节蛋白CheY、甲基酯酶CheB、甲基转移酶CheR、磷酸酶CheC/CheX/CheZ、以及脱酰胺酶CheD[16]。趋化系统的信号转导及调控机制如下(图 1):Mcps负责结合胞外趋化信号, 并通过支架蛋白CheW/V与CheA相连;Mcps结合信号物质后调控CheA的组氨酸激酶活性, 进而将信号向下游的CheY传递;被CheA磷酸化的CheY(CheY-P)通过鞭毛蛋白FliM与鞭毛-马达复合体相互作用, 调控细菌的运动方向。共同构成“适应系统”的CheB和CheR互相竞争, 调控Mcps的甲基化状态从而调节其对信号物质的结合亲和力, 不断“重置”细菌趋化系统的灵敏度。磷酸酶CheC/CheX/CheZ能够将CheY-P去磷酸化, 使CheY持续接受来自CheA的磷酸基团。脱酰胺酶CheD则能够调节CheC的磷酸酶活性, 并作用于Mcps。这一套精密运作的趋化系统使细菌能够灵敏感应胞外信号物质的浓度梯度, 适应周围环境的变化。
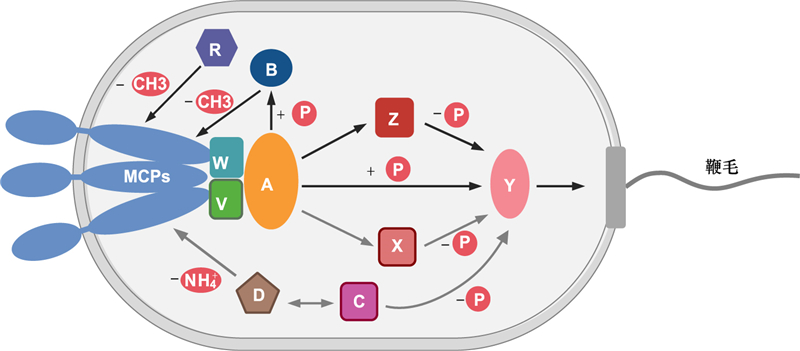
|
| 图 1 细菌趋化系统信号转导模式图 Fig. 1 A summary model illustrates the organization of bacterial chemotaxis systems Mcps: 趋化受体蛋白; V/W: 支架蛋白CheV和CheW; A: 组氨酸激酶CheA; B: 甲基酯酶CheB; R: 甲基转移酶CheR; D: 脱酰胺酶CheD; C/X/Z: 磷酸酶CheC/CheX/CheZ; Y: 反应调节蛋白CheY; P: 磷酸基团; CH3: 甲基; NH4+: 铵根; 本模式图在大肠杆菌趋化系统的基础上, 集中了当前细菌趋化系统研究的模式菌(包括芽孢杆菌、假单胞菌等)中已鉴定的所有蛋白组分, 带有轮廓线的组分与灰色箭头标注的反应不存在于大肠杆菌中 |
近年来, 大量的研究证明了细菌趋化系统能够介导植物与微生物相互作用。在根瘤菌和豆科植物共生体系的建立过程中, 豆科植物根系分泌类黄酮等化合物, 根瘤菌则能够利用趋化系统感应这一类信号分子从而靠近植物根系表面, Cooper[20]因此总结:根瘤菌对豆科植物根系分泌的信号分子的趋化反应是二者建立共生关系的第一步。此外, 在根际促生菌与植物的互作中细菌趋化系统也发挥重要作用。Allard-Massicotte等[21]发现, 模式植物拟南芥的根系分泌物能够诱导芽孢杆菌B. subtilis产生趋化反应进而吸引其达到根系, 并且B. subtilis对拟南芥根系的成功定殖依赖于其完整的趋化系统;芽孢杆菌B. velezensis对作物玉米、黄瓜根系分泌物的趋化反应调控了其对宿主的根系定殖[22-23]。此外, 另外一类典型的根际促生菌假单胞菌如P. fluorescens、P. putida等的趋化系统在其对宿主植物玉米、番茄根系定殖中的重要作用也得到了验证[24-25]。除了根瘤菌和根际促生菌等有益菌, 一些植物病原菌也能够利用自身的趋化系统感应植物根系分泌物, 通过趋化反应靠近植物根系并起始侵染致病过程。Hida等[26]研究发现, 番茄根系分泌物能够诱导青枯雷尔氏菌Ralstonia pseudosolanacearum产生趋化反应, 促进其向番茄根际的运动及对番茄根系的定殖。
上述的研究主要针对单一菌株与植物的互作, 在群落水平上, 也有研究证明了细菌趋化系统在介导植物与微生物互作中的重要作用。Kamutando等[27]对入侵树种银荆的研究发现, 其根际土壤中细菌趋化系统相关基因的丰度显著高于非根际土壤;Xu等[28]对全球范围内六个大洲的柑橘根际微生物组的研究表明, 细菌趋化系统相关基因在柑橘根际核心微生物组中显著富集。但是, 当前在群落水平上对细菌趋化系统的认识仍有局限, 将趋化系统作为一个整体考虑是可以理解的, 然而趋化系统作为一个复杂的信号转导系统, 不同组分的进化速率和变异程度可能并不完全同步, 所以针对信号系统各组分的细致研究对深入揭示其生态功能至关重要。
3 植物对根际细菌趋化系统的选择及其调控机制研究表明, 不同植物根际细菌趋化系统的分布存在一定差异, 主要体现在丰度和多样性方面。在趋化基因丰度方面, Mendes等[29]对大豆根际微生物组的研究发现, 抗镰刀菌根腐病大豆的根际趋化基因丰度显著高于易染病大豆, 并且趋化基因的丰度与根际细菌群落结构的复杂度呈正相关。在趋化基因多样性方面, Buchan等[30]采用了“T-RFLP”(Terminal restriction fragment length polymorphism, 末端限制性片段长度多态性)技术对小麦和豇豆根际细菌趋化系统中核心基因cheA的序列多样性进行了分析, 发现根际趋化细菌中cheA序列的多样性低于非根际细菌, 且在两种植物根际间差异显著。但是上述研究受当时技术所限, 缺少对全部趋化基因的系统分析, 尤其是趋化系统中受外界信号调控的组分, 包括结合外界信号物质的趋化受体mcp, 以及调控趋化受体结合物质灵敏度的cheB和cheR。由于趋化系统介导了植物与微生物间的互作, 根际微生物组中趋化基因的丰度能够一定程度上体现植物与微生物间互作关系的强弱, 趋化基因的多样性则体现了植物对细菌趋化系统的特异性选择。当前, 对根际趋化相关基因的丰度和多样性, 以及其体现的植物对趋化系统的选择缺少认识, 仍需进行深入探索。随着高通量测序技术的发展, 当前利用宏基因组分析结合传统荧光定量PCR手段已能够实现对特定信号通路中相关基因的丰度和多样性进行分析。
细菌趋化系统能够感应植物的根系分泌物, 已有的研究结果表明, 多种不同类型的根系分泌物包括苹果酸、草酸等有机酸[31-32], 组氨酸、精氨酸等氨基酸[33-34], 甘露糖、半乳糖等糖类[23, 35], 以及酚酸[36]、苯并恶唑嗪酮[37]等次级代谢产物都能够诱导根际趋化细菌产生趋化反应。Zhang等[38]通过HPLC鉴定了黄瓜和香蕉根系分泌物中的有机酸, 并分别检测了芽孢杆菌属细菌B. amyloliquefaciens和B. subtilis对两种植物根系分泌物的趋化反应, 发现黄瓜根系分泌物中特有的柠檬酸能够诱导B. amyloliquefaciens和Bacillus subtilis产生趋化反应, 而香蕉根系分泌物中特有的延胡索酸只能诱导B. subtilis产生趋化反应。该研究表明, 植物能够通过分泌特定的化合物对趋化细菌进行特异性选择, 而这一选择实现的前提则是细菌的趋化系统决定了其对何种化合物会产生趋化反应。从群落水平的角度来说, 植物的根系分泌谱与根际细菌趋化系统的组成之间一定存在特定关系, 这正是植物实现对细菌趋化系统特异性选择的基础。此外, 之前的研究主要是在实验室条件下利用特定的单一化合物或多种化合物进行细菌趋化反应的诱导, 缺少针对根系分泌物与细菌趋化反应间的全面系统性研究, 以及更深层次上与环境生态因子的复合影响效果。
植物根系代谢组学技术可以实现对植物根系分泌物中初级、次级代谢产物的定性及定量分析[39], 已成为植物代谢研究的常用手段。比如, Mönchgesang等[40]通过根系代谢组学技术对19种不同基因型拟南芥的根系分泌物谱进行了解析, 建立了拟南芥基因型与其根系分泌物谱之间的联系;Zhalnina等[41]通过比较基因组学和代谢组学的联合分析, 发现特定细菌对根系分泌物中芳香类有机酸的代谢偏好性决定了其在燕麦根际的特异性富集。因此, 未来可利用植物代谢组学技术深入探索植物对细菌趋化系统特异性选择的调控机制。
4 趋化细菌类群在根际微生物群落结构调控中的功能在自然生态系统中, 微生物之间通过营养代谢、信号交流、生存空间的竞争与共享等进行竞争或者协作[42]。在根际微生物组群落结构的调控因素中, 除宿主植物外, 微生物间相互作用也是重要因素之一[43]。细菌趋化系统不仅能够介导植物-微生物间相互作用, 还能够介导微生物间相互作用。Garbeva和de Boer[44]研究发现, 假单胞菌属细菌Pseudomonas sp.和土地杆菌属细菌Pedobacter sp.在碳源限制的条件下共同生长时, 表现出竞争性相互作用, 且二者的趋化基因表达水平都显著提高, 证明了趋化系统在介导细菌种间竞争中的功能。Lu等[45]对通过种间协作提高产氢效率的梭菌属细菌Clostridium cellulovorans和红假单胞菌属细菌Rhodopseudomonas palustris进行了转录组分析, 发现C. cellulovorans分泌的挥发性脂肪酸可能作为R. palustris的趋化吸引物, 导致其趋化基因表达水平上调, 说明趋化系统同样能够介导细菌的种间协作。此外, Haq等[46]的研究表明, 趋化系统还能够介导伯克霍尔德菌属细菌Burkholderia terrae与离褶伞属Lyophyllum和木霉属Trichoderma真菌间的相互作用。以上研究主要针对单一菌株间的相互作用, 在群落水平上, 细菌趋化系统在调控微生物间相互作用进而影响群落结构方面同样起到重要作用, 但是其生态功能仍缺少深入研究。
近年来, 生物信息学家开始使用共现性网络分析来探索微生物间相互作用并预测微生物群落中的关键核心类群[47-48]。比如, Agler等[49]研究发现, 在拟南芥叶际微生物组共现性网络中处于关键核心位置的特定微生物类群, 能够通过微生物间相互作用传递环境和宿主对微生物群落的影响效应, 明确了关键核心类群的调控功能。Ye等[50]最新的研究表明, 一株捕食性粘细菌Corallococcus sp. EGB能够通过趋化反应感应黄瓜的根系分泌物进而向根部迁移, 在迁移过程中, 捕食病原菌尖孢镰刀菌Fusarium oxysporum f. sp. cucumerinum;共现性网络分析的结果表明, 这一捕食性粘细菌还能够调控土壤微生物群落结构。该研究结果揭示了趋化细菌类群在土壤微生物群落结构调控中的生态功能, 为后续的研究提供了重要借鉴。
5 展望综上, 研究总结提出根际微生物组中细菌趋化系统的作用机制, 细菌趋化系统感应植物根系分泌物, 介导趋化细菌与植物间的相互作用;进一步, 趋化细菌类群利用趋化系统与其他微生物类群发生相互作用, 作为关键类群在根际微生物富集中发挥重要的调控作用。因此, 细菌趋化系统在根际微生物组募集组装过程中发挥着关键的生态功能, 这一功能的解析将是揭示植物对根际微生物组选择机制的重要突破口之一(图 2)。
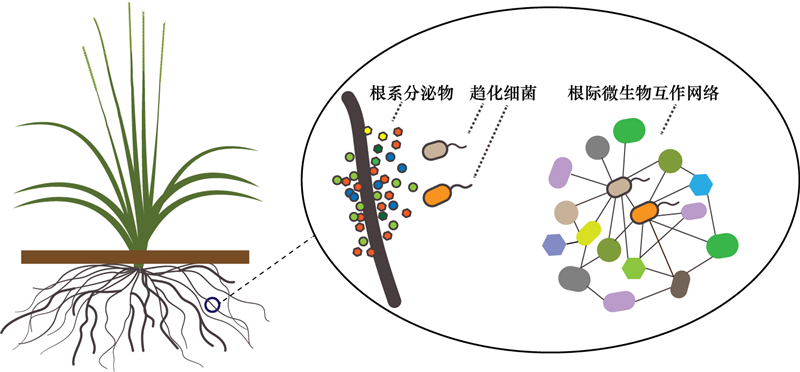
|
| 图 2 根际微生物组中的趋化细菌类群 Fig. 2 A model illustrates the function of chemotactic bacteria in rhizosphere microbiome |
针对根际微生物组中细菌趋化系统的生态功能, 建议未来研究方向从解析宿主植物对细菌趋化系统的选择规律入手, 明确趋化基因在根际的多样性及分布规律;然后建立根系分泌物与趋化系统的联系, 阐明宿主植物对趋化系统特异性选择的调控机制;并进一步探索关键趋化细菌类群在根际微生物群落结构中的调控功能。研究方法上, 在扩增子高通量测序和宏基因组测序的基础上, 进行代谢组学检测, 并结合菌株分离回接验证等, 将分子生物学、生态学和微生物学等相关知识有效融合, 最终揭示细菌趋化系统在植物对根际微生物组选择中的作用机制, 增进对根际微生物组募集组装过程的认识, 为根际微生物组的改良重组提供理论依据。
| [1] |
Hinsinger P, Bengough A G, Vetterlein D, Young I M. Rhizosphere: biophysics, biogeochemistry and ecological relevance. Plant and Soil, 2009, 321(1/2): 117-152. |
| [2] |
Berendsen R L, Pieterse C M J, Bakker P A H M. The rhizosphere microbiome and plant health. Trends in Plant Science, 2012, 17(8): 478-486. DOI:10.1016/j.tplants.2012.04.001 |
| [3] |
陆雅海, 张福锁. 根际微生物研究进展. 土壤, 2006, 38(2): 113-121. DOI:10.3321/j.issn:0253-9829.2006.02.001 |
| [4] |
Oldroyd G E D. Speak, friend, and enter: signalling systems that promote beneficial symbiotic associations in plants. Nature Reviews Microbiology, 2013, 11(4): 252-263. DOI:10.1038/nrmicro2990 |
| [5] |
Mbodj D, Effa-Effa B, Kane A, Manneh B, Gantet P, Laplaze L, Diedhiou A G, Grondin A. Arbuscular mycorrhizal symbiosis in rice: establishment, environmental control and impact on plant growth and resistance to abiotic stresses. Rhizosphere, 2018, 8: 12-26. DOI:10.1016/j.rhisph.2018.08.003 |
| [6] |
Zhang R F, Vivanco J M, Shen Q R. The unseen rhizosphere root-soil-microbe interactions for crop production. Current Opinion in Microbiology, 2017, 37: 8-14. DOI:10.1016/j.mib.2017.03.008 |
| [7] |
Santhanam R, Luu V T, Weinhold A, Goldberg J, Oh Y, Baldwin I T. Native root-associated bacteria rescue a plant from a sudden-wilt disease that emerged during continuous cropping. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(36): E5013-5020. DOI:10.1073/pnas.1505765112 |
| [8] |
Bulgarelli D, Rott M, Schlaeppi K, Ver Loren van Themaat E, Ahmadinejad N, Assenza F, Rauf P, Huettel B, Reinhardt R, Schmelzer E, Peplies J, Gloeckner F O, Amann R, Eickhorst T, Schulze-Lefert P. Revealing structure and assembly cues for Arabidopsis root-inhabiting bacterial microbiota. Nature, 2012, 488(7409): 91-95. DOI:10.1038/nature11336 |
| [9] |
Philippot L, Raaijmakers J M, Lemanceau P, van der Putten W H. Going back to the roots: the microbial ecology of the rhizosphere. Nature Reviews Microbiology, 2013, 11(11): 789-799. DOI:10.1038/nrmicro3109 |
| [10] |
Badri D V, Chaparro J M, Zhang R F, Shen Q R, Vivanco J M. Application of natural blends of phytochemicals derived from the root exudates of Arabidopsis to the soil reveal that phenolic-related compounds predominantly modulate the soil microbiome. Journal of Biological Chemistry, 2013, 288(7): 4502-4512. DOI:10.1074/jbc.M112.433300 |
| [11] |
Hu L F, Robert C A M, Cadot S, Zhang X, Ye M, Li B B, Manzo D, Chervet N, Steinger T, van der Heijden M G A, Schlaeppi K, Erb M. Root exudate metabolites drive plant-soil feedbacks on growth and defense by shaping the rhizosphere microbiota. Nature Communications, 2018, 9: 2738. DOI:10.1038/s41467-018-05122-7 |
| [12] |
朱晓艳, 沈重阳, 陈国炜, 张伟, 李保国, 王钢. 土壤细菌趋化性研究进展. 土壤学报, 2019, 56(2): 259-275. |
| [13] |
Raina J B, Fernandez V, Lambert B, Stocker R, Seymour J R. The role of microbial motility and chemotaxis in symbiosis. Nature Reviews Microbiology, 2019, 17(5): 284-294. DOI:10.1038/s41579-019-0182-9 |
| [14] |
Scharf B E, Hynes M F, Alexandre G M. Chemotaxis signaling systems in model beneficial plant-bacteria associations. Plant Molecular Biology, 2016, 90(6): 549-559. DOI:10.1007/s11103-016-0432-4 |
| [15] |
Tindall M J, Gaffney E A, Maini P K, Armitage J P. Theoretical insights into bacterial chemotaxis. WIREs Systems Biology and Medicine, 2012, 4(3): 247-259. DOI:10.1002/wsbm.1168 |
| [16] |
Wuichet K, Zhulin I B. Origins and diversification of a complex signal transduction system in prokaryotes. Science Signaling, 2010, 3(128): ra50. |
| [17] |
Parkinson J S, Hazelbauer G L, Falke J J. Signaling and sensory adaptation in Escherichia coli chemoreceptors: 2015 update. Trends in Microbiology, 2015, 23(5): 257-266. DOI:10.1016/j.tim.2015.03.003 |
| [18] |
Rao C V, Glekas G D, Ordal G W. The three adaptation systems of Bacillus subtilis chemotaxis. Trends in Microbiology, 2008, 16(10): 480-487. DOI:10.1016/j.tim.2008.07.003 |
| [19] |
Sampedro I, Parales R E, Krell T, Hill J E. Pseudomonas chemotaxis. FEMS Microbiology Reviews, 2015, 39(1): 17-46. |
| [20] |
Cooper J E. Early interactions between legumes and rhizobia: disclosing complexity in a molecular dialogue. Journal of Applied Microbiology, 2007, 103(5): 1355-1365. DOI:10.1111/j.1365-2672.2007.03366.x |
| [21] |
Allard-Massicotte R, Tessier L, Lécuyer F, Lakshmanan V, Lucier J F, Garneau D, Caudwell L, Vlamakis H, Bais H P, Beauregard P B. Bacillus subtilis early colonization of Arabidopsis thaliana roots involves multiple chemotaxis receptors. mBio, 2016, 7(6): e01664-16. |
| [22] |
Jin Y Q, Zhu H F, Luo S, Yang W W, Zhang L, Li S S, Jin Q, Cao Q, Sun S R, Xiao M. Role of maize root exudates in promotion of colonization of Bacillus velezensis strain S3-1 in rhizosphere soil and root tissue. Current Microbiology, 2019, 76(7): 855-862. DOI:10.1007/s00284-019-01699-4 |
| [23] |
Feng H C, Zhang N, Fu R X, Liu Y P, Krell T, Du W B, Shao J H, Shen Q R, Zhang R F. Recognition of dominant attractants by key chemoreceptors mediates recruitment of plant growth-promoting rhizobacteria. Environmental Microbiology, 2019, 21(1): 402-415. DOI:10.1111/1462-2920.14472 |
| [24] |
Oku S, Komatsu A, Nakashimada Y, Tajima T, Kato J. Identification of Pseudomonas fluorescens chemotaxis sensory proteins for malate, succinate, and fumarate, and their involvement in root colonization. Microbes and Environments, 2014, 29(4): 413-419. DOI:10.1264/jsme2.ME14128 |
| [25] |
Li T, Zhang J, Shen C H, Li H R, Qiu L Y. 1-aminocyclopropane-1-carboxylate: a novel and strong chemoattractant for the plant beneficial rhizobacterium Pseudomonas putida UW4. Molecular Plant-Microbe Interactions, 2019, 32(6): 750-759. DOI:10.1094/MPMI-11-18-0317-R |
| [26] |
Hida A, Oku S, Kawasaki T, Nakashimada Y, Tajima T, Kato J. Identification of the mcpA and mcpM genes, encoding methyl-accepting proteins involved in amino acid and L-malate chemotaxis, and involvement of McpM-mediated chemotaxis in plant infection by Ralstonia pseudosolanacearum (formerly Ralstonia solanacearum phylotypes Ⅰ and Ⅲ). Applied and Environmental Microbiology, 2015, 81(21): 7420-7430. DOI:10.1128/AEM.01870-15 |
| [27] |
Kamutando C N, Vikram S, Kamgan-Nkuekam G, Makhalanyane T P, Greve M, Le Roux J J, Richardson D M, Cowan D A, Valverde A. The functional potential of the rhizospheric microbiome of an invasive tree species, Acacia dealbata. Microbial Ecology, 2019, 77(1): 191-200. DOI:10.1007/s00248-018-1214-0 |
| [28] |
Xu J, Zhang Y Z, Zhang P F, Trivedi P, Riera N, Wang Y Y, Liu X, Fan G Y, Tang J L, Coletta-Filho H D, Cubero J, Deng X L, Ancona V, Lu Z J, Zhong B L, Roper M C, Capote N, Catara V, Pietersen G, Vernière C, Al-Sadi A M, Li L, Yang F, Xu X, Wang J, Yang H M, Jin T, Wang N. The structure and function of the global citrus rhizosphere microbiome. Nature Communications, 2018, 9(1): 4894. DOI:10.1038/s41467-018-07343-2 |
| [29] |
Mendes L W, Mendes R, Raaijmakers J M, Tsai S M. Breeding for soil-borne pathogen resistance impacts active rhizosphere microbiome of common bean. The ISME Journal, 2018, 12(12): 3038-3042. DOI:10.1038/s41396-018-0234-6 |
| [30] |
Buchan A, Crombie B, Alexandre G M. Temporal dynamics and genetic diversity of chemotactic-competent microbial populations in the rhizosphere. Environmental Microbiology, 2010, 12(12): 3171-3184. DOI:10.1111/j.1462-2920.2010.02290.x |
| [31] |
Tan S Y, Yang C L, Mei X L, Shen S Y, Raza W, Shen Q R, Xu Y C. The effect of organic acids from tomato root exudates on rhizosphere colonization of Bacillus amyloliquefaciens T-5. Applied Soil Ecology, 2013, 64: 15-22. DOI:10.1016/j.apsoil.2012.10.011 |
| [32] |
Yuan J, Zhang N, Huang Q W, Raza W, Li R, Vivanco J M, Shen Q R. Organic acids from root exudates of banana help root colonization of PGPR strain Bacillus amyloliquefaciens NJN-6. Scientific Reports, 2015, 5: 13438. DOI:10.1038/srep13438 |
| [33] |
Liu X L, Xie Z H, Wang Y X, Sun Y, Dang X X, Sun H S. A dual role of amino acids from Sesbania rostrata seed exudates in the chemotaxis response of Azorhizobium caulinodans ORS571. Molecular Plant-Microbe Interactions, 2019, 32(9): 1134-1147. DOI:10.1094/MPMI-03-19-0059-R |
| [34] |
Webb B A, Compton K K, del Campo J S M, Taylor D, Sobrado P, Scharf B E. Sinorhizobium meliloti chemotaxis to multiple amino acids Is mediated by the chemoreceptor McpU. Molecular Plant-Microbe Interactions, 2017, 30(10): 770-777. DOI:10.1094/MPMI-04-17-0096-R |
| [35] |
Liu Y P, Feng H C, Fu R X, Zhang N, Du W B, Shen Q R, Zhang R F. Induced root-secreted D-galactose functions as a chemoattractant and enhances the biofilm formation of Bacillus velezensis SQR9 in an McpA-dependent manner. Applied Microbiology and Biotechnology, 2020, 104(2): 785-797. DOI:10.1007/s00253-019-10265-8 |
| [36] |
Ray S, Mishra S, Bisen K, Singh S, Sarma B K, Singh H B. Modulation in phenolic root exudate profile of Abelmoschus esculentus expressing activation of defense pathway. Microbiological Research, 2018, 207: 100-107. DOI:10.1016/j.micres.2017.11.011 |
| [37] |
Neal A L, Ahmad S, Gordon-Weeks R, Ton J. Benzoxazinoids in root exudates of maize attract Pseudomonas putida to the rhizosphere. PLoS One, 2012, 7(4): e35498. DOI:10.1371/journal.pone.0035498 |
| [38] |
Zhang N, Wang D D, Liu Y P, Li S Q, Shen Q R, Zhang R F. Effects of different plant root exudates and their organic acid components on chemotaxis, biofilm formation and colonization by beneficial rhizosphere-associated bacterial strains. Plant and Soil, 2014, 374(1/2): 689-700. DOI:10.1007/s11104-013-1915-6 |
| [39] |
van Dam N M, Bouwmeester H J. Metabolomics in the rhizosphere: tapping into belowground chemical communication. Trends in Plant Science, 2016, 21(3): 256-265. DOI:10.1016/j.tplants.2016.01.008 |
| [40] |
Mönchgesang S, Strehmel N, Schmidt S, Westphal L, Taruttis F, Müller E, Herklotz S, Neumann S, Scheel D. Natural variation of root exudates in Arabidopsis thaliana-linking metabolomic and genomic data. Scientific Reports, 2016, 6: 29033. DOI:10.1038/srep29033 |
| [41] |
Zhalnina K, Louie K B, Hao Z, Mansoori N, da Rocha U N, Shi S J, Cho H, Karaoz U, Loqué D, Bowen B P, Firestone M K, Northen T R, Brodie E L. Dynamic root exudate chemistry and microbial substrate preferences drive patterns in rhizosphere microbial community assembly. Nature Microbiology, 2018, 3(4): 470-480. DOI:10.1038/s41564-018-0129-3 |
| [42] |
Faust K, Raes J. Microbial interactions: from networks to models. Nature Reviews Microbiology, 2012, 10(8): 538-550. DOI:10.1038/nrmicro2832 |
| [43] |
Hassani M A, Durán P, Hacquard S. Microbial interactions within the plant holobiont. Microbiome, 2018, 6(1): 58. DOI:10.1186/s40168-018-0445-0 |
| [44] |
Garbeva P, de Boer W. Inter-specific interactions between carbon-limited soil bacteria affect behavior and gene expression. Microbial Ecology, 2009, 58(1): 36-46. DOI:10.1007/s00248-009-9502-3 |
| [45] |
Lu H Y, Chen J H, Jia Y Y, Cai M W, Lee P K H. Transcriptomic responses of the interactions between Clostridium cellulovorans 743B and Rhodopseudomonas palustris CGA009 in a cellulose-grown coculture for enhanced hydrogen production. Applied and Environmental Microbiology, 2016, 82(15): 4546-4559. DOI:10.1128/AEM.00789-16 |
| [46] |
Haq I U, da Rocha Calixto R O, Yang P, dos Santos G M P, Barreto-Bergter E, van Elsas J D. Chemotaxis and adherence to fungal surfaces are key components of the behavioral response of Burkholderia terrae BS001 to two selected soil fungi. FEMS Microbiology Ecology, 2016, 92(11): fiw164. DOI:10.1093/femsec/fiw164 |
| [47] |
Barberán A, Bates S T, Casamayor E O, Fierer N. Using network analysis to explore co-occurrence patterns in soil microbial communities. The ISME Journal, 2012, 6(2): 343-351. DOI:10.1038/ismej.2011.119 |
| [48] |
Berry D, Widder S. Deciphering microbial interactions and detecting keystone species with co-occurrence networks. Frontiers in Microbiology, 2014, 5: 219. |
| [49] |
Agler M T, Ruhe J, Kroll S, Morhenn C, Kim S T, Weigel D, Kemen E M. Microbial hub taxa link host and abiotic factors to plant microbiome variation. PLoS Biology, 2016, 14(1): e1002352. DOI:10.1371/journal.pbio.1002352 |
| [50] |
Ye X F, Li Z K, Luo X, Wang W H, Li Y K, Li R, Zhang B, Qiao Y, Zhou J, Fan J Q, Wang H, Huang Y, Cao H, Cui Z L, Zhang R F. A predatory myxobacterium controls cucumber Fusarium wilt by regulating the soil microbial community. Microbiome, 2020, 8(1): 49. DOI:10.1186/s40168-020-00824-x |
 2021, Vol. 41
2021, Vol. 41