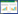文章信息
- 焦克, 张旭博, 徐梦, 刘晓洁, 安前东, 张崇玉
- JIAO Ke, ZHANG Xubo, XU Meng, LIU Xiaojie, AN Qiandong, ZHANG Chongyu
- 藏东南典型暗针叶林不同土壤剖面微生物群落特征
- Depth-related characteristics of soil microbial community along the soil profile of typical dark coniferous forest in southeast Tibet
- 生态学报. 2021, 41(12): 4864-4875
- Acta Ecologica Sinica. 2021, 41(12): 4864-4875
- http://dx.doi.org/10.5846/stxb202004291047
-
文章历史
- 收稿日期: 2020-04-29
- 网络出版日期: 2021-04-21
2. 中国科学院地理科学与资源研究所, 生态网络观测与模拟重点实验室, 北京 100101
2. Key Laboratory of Ecosystem Network Observation and Modeling/Institute of Geographic Sciences and Natural Resources Research, Chinese Academy of Sciences, Beijing 100101, China
土壤微生物是土壤的重要组成部分, 在有机质形成与分解、养分循环与转化和能量流动等方面起着重要作用。土壤微生物多样性和群落组成是微生物生态学研究的重要指标[1], 对认识各种生态系统下土壤生物地球化学循环过程及其对环境变化的响应有重要意义。土壤微生物多样性和群落结构的变化主要受土壤营养状况、质地、温度、水分、通气性、根系分泌物、植物残体等众多因子的综合影响[2], 而这些因子会随着土壤深度发生变化, 进而导致土壤微生物群落在土壤垂直空间上的差异性。已有研究表明, 随着深度的增加, 土壤微生物的物种多样性和功能多样性都下降[3-4], 并且土壤微生物群落的数量和组成也发生变化[5]。通常认为, 微生物活性在土壤表层(0—20 cm)最高, 但是更深层土壤中的微生物仍然在养分代谢、能量循环及土壤形成等过程中发挥重要作用[6-7]。因此, 对深层土壤微生物群落组成和功能的研究, 有助于深入认识土壤微生物在驱动陆地生态系统关键过程中的核心作用。
土壤微生物不同物种(或种群)之间存在着异常复杂的相互作用关系, 既包括捕食和寄生等对抗关系, 也包括共生和共栖等互惠互利关系, 而这些相互作用维系着微生物群落的稳定性, 进而有效保障生态系统过程和功能的稳定性[8]。生态网络分析技术最先应用于大型生物物种(或群落)之间的相互作用研究[9-10], 近年来随着微生物分子生物学技术的发展, 也开始应用于探索土壤微生物之间的微观相互作用关系(即微生物分子生态网络)[11]。Deng等[12]研发了一种新的基于随机矩阵理论的构建分子生态网络的方法, 能提高传统方法的可靠性和敏感性, 可用于高通量数据的模块化网络构建和功能预测[12-13]。对微生物分子生态网络的研究能够深入了解土壤微生物群落构建的多样性和复杂性, 并有助于更加准确的预测微生物群落构建和功能对环境变化的响应。例如, Lu等[14]通过构建健康和发病连作土壤的真菌分子生态网络, 发现健康土壤中的真菌网络比发病土壤更加高效和组织有序化。Zhou等[15]对我国西北毛乌素沙地生物结皮的研究揭示, 随着生物结皮的发展, 土壤微生物网络中的关键种群发生显著变化, 进而导致微生物群落生态系统功能随之发生改变。
青藏高原作为主要的高寒生态系统区域之一, 其生态系统对于气候变暖以及降水格局的改变十分敏感, 因此研究该区域不同生态类型下土壤微生物群落特征对深入了解和预测全球变化的影响有重要意义。目前针对青藏高原土壤微生物的研究多关注在高寒草地草甸生态系统类型下土壤微生物群落组成、功能及相互作用关系[16-18], 对其他生态系统类型的研究相对有限。西藏东南部地区植物类型十分丰富, 其中暗针叶林带是西藏东南部及横断山脉区山地植被垂直带谱中的优势带。藏东南暗针叶林带具有较高的生物量和生产力[19], 并且由于高海拔的低温显著抑制分解作用, 是青藏高原东南部重要的碳库, 对该区域乃至全球陆地生态系统碳循环和收支平衡至关重要。西藏色季拉山是藏东南部典型的山地生态系统, 其暗针叶林带主要分布在海拔3500—4200 m, 主要建群种为急尖长苞冷杉(Abies georgei var. smithii)和林芝云杉(Picea likiangensis var. linzhiensis), 其土壤有机碳储量和密度均处于较高水平[20]。近年来对色季拉山暗针叶林带土壤微生物有一些报道, 然而多集中于微生物量和表层土壤微生物群落组成特征[20-21], 对深层土壤的微生物群落结构及种群之间的相互作用关系认识有限。本研究在西藏色季拉山暗针叶林带采集表层土壤(0—20 cm)和底层土壤(40—60 cm), 利用Illumina Miseq高通量测序分析和微生物分子生态网络构建, 系统揭示土壤真菌和细菌多样性、群落组成及微生物生态网络结构在土壤垂直剖面的变化特征, 为深入了解高寒地区土壤微生物群落构建特征及预测未来环境变化的影响提供科学依据。
1 研究方法 1.1 研究地区概况色季拉山坐落于西藏自治区林芝市境内(29°35′—29°57′N, 94°25′—94°45′E), 属于念青唐古拉山脉, 是尼洋河流域和帕隆藏布江的分水岭, 其主峰海拔高度为5200 m。色季拉山位于藏东南湿润气候区与半湿润气候区的过渡地带, 受印度洋暖湿季风影响, 降雨量主要集中在夏秋两季。据西藏林芝高山森林生态系统国家野外科学观测研究站(海拔3900 m)观测, 色季拉山年平均气温为-0.73℃, 最高月为7月, 其平均气温为9.23℃, 最低月为1月, 其平均气温为-13.98℃。年平均降水量为1134 mm, 6—9月为雨季, 占全年降水的75%—82%。年平均蒸发量为544 mm, 年平均相对湿度为78.83%[22]。
1.2 样品采集与处理土壤样品的采集于2013年6月, 在色季拉山西坡典型暗针叶林分布区域(海拔3500—4200 m)随机选取9个采样样方(10 m×10 m), 每个样方之间间隔不小于100 m。在每个样方中随机挖取3个土壤剖面(0—60 cm), 先除去土壤表面的凋落物, 之后分别采集剖面的表层(0—20 cm)和底层(40—60 cm)土壤样品。每个样方中三个剖面的表层土样均匀混合为一个表层土样, 三个剖面的底层土样均匀混合为一个底层土样, 即每个样方获得两个样品, 9个样方共获得18个样品。采集的土壤样品装入封口袋中, 保存在冰盒里运输回实验室进行后续分析。将土壤样品过筛(2 mm), 挑出可见的石块、土壤动物和植物残体。提取DNA的土壤样品于-20℃下保存, 剩余测定土壤其他指标的样品风干于室温下保存。
1.3 土壤基本理化性质测定土壤pH用1 mol/L KCl浸提(土水比为1∶2.5)并用电位法测定。土壤有机碳(SOC)用重铬酸钾容量法-外加热法测定。土壤全氮含量使用元素分析仪测定(EA1108, Carlo Erba, Turin, Italy)。土壤含水量使用重量含水量表征, 即将新鲜土样于105℃烘干, 根据烘干前后的质量差计算土壤含水量。
1.4 高通量测序使用PowerSoil DNA提取试剂盒(MoBio Laboratories Inc., Carlsbad, CA, USA)进行土壤DNA的提取。使用NanoDrop ND-2000分光光度计(ThermoScientific, Wilmington, DE, USA)测定提取DNA的质量。使用引物515F(5′-GTGCCAGCMGCCGCGG-3′)和907R(5′-CCGTCAATTCMTTTRAGTTT-3′)扩增细菌16S rRNA基因, 使用引物1737F(5′-GGAAGTAAAAGTCGTAACAAGG-3′)和2043R(5′-GCTGCGTTCTTCATCGATGC-3′)扩增真菌ITS基因。扩增产物送至上海美吉生物医药有限公司进行高通量测序(Illumina MiSeq platform)。测序获得的原始序列保存为FASTQ格式, 使用Quantitative Insights Into Microbial Ecology(QIIME)对序列进行优化和数据质控。之后使用Usearch软件平台(version 7.0, http://drive5.com/uparse/)对所有序列进行OTU(operational taxonomic units)在97%的相似度水平下进行划分, 生成OTU表格。为了得到每个OTU对应的物种分类信息, 采用RDP classifier贝叶斯算法对97%相似水平的OTU代表序列进行分类学分析, 对比数据分别为SILVA细菌16S rRNA数据库(Release 119, http://www.arb-silva.de), 和UNITE真菌ITS数据库(Release 6.0, http://unite.ut.ee/index.php)。使用Mothur软件(version v.1.30.1, http://www.mothur.org/wiki/Schloss_SOP#Alpha_diversity)计算细菌和真菌群落的α-多样性指数, 包括丰富度(Chao1指数)和Shannon多样性指数。
1.5 分子生态网络构建与分析基于高通量测序获得的OTU数据进行微生物分子生态学网络的构建[12]。首先在MENA平台(http://ieg4.rccc.ou.edu/MENA/)中通过原始OUT标准化筛选、OTU丰度计算各个OTU之间的相关性, 之后基于随机矩阵理论(Random matrix theory)构建微生物网络, 分析得到的网络数据文件使用Cytoscape 3.7.0进行可视化, 具体操作步骤和参数的选择参照Deng等[12]提供的方法。微生物网络分析包括网络特征分析、模块检测、网络拓扑结构及特性分析等, 不同网络的拓扑结构特征使用不同的指数如平均连接度(Average connectivity)、平均聚类系数(Average clustering coefficient)、平均距离(Average path distance)等进行表征。微生物生态学网络中每个节点的功能通过模块的内连接度(Zi)和模块间连接度(Pi)的值来表征。根据简化网络的阈值[23], 网络节点可划分为四类:外围节点(Peripherals nodes, Zi < 2.5且Pi < 0.62)、模块枢纽(Module hubs, Zi ≥ 2.5且Pi < 0.62)、连接节点(Connectors, Zi < 2.5且Pi ≥ 0.62)和网络枢纽(Network hubs, Zi ≥ 2.5且Pi ≥ 0.62)。
1.6 统计分析数据表示为每个采样深度的平均值±标准误差。使用配对样本T检验比较表层和底层下土壤理化性质、细菌和真菌群落的α-多样性指数以及主要种群相对丰度的差异。使用Pearson相关性分析分析细菌、真菌群落的α-多样性指数及各种群相对丰度与土壤理化指标的关系。上述统计分析利用SPSS 22.0进行。不同剖面深度细菌和真菌的群落结构的差异(即β-多样性)在使用主坐标分析(PCoA)表征, 基于Bray-Curtis距离构建群落组成相似性矩阵, 表层和底层土壤微生物群落结构的差异用PERMANOVA进行检验, PCoA分析使用R软件包vegan进行。微生物分子生态网络使用MENA分析和Cytoscape 3.7.0软件进行可视化。
2 结果 2.1 土壤理化性质特征在色季拉山暗针叶林带, 土壤剖面深度显著影响了土壤碳、氮含量。与表层土壤相比, 底层土壤的SOC和TN含量显著下降, 分别降低了53.7%和53.3%, 但是土壤碳氮比在表层和底层土壤无显著性差异(表 1)。此外, 相比于表层土壤, 底层土壤的pH上升了0.44, 土壤含水量下降了30.5%, 但是都没有达到显著水平。
| 土壤深度Soil depth | pH | SOC/% | TN/% | C∶N | SWC/% |
| 表层土壤Topsoil | 5.28±0.16a | 4.82±0.48a | 0.30±0.04a | 16.02±0.94a | 36.79±3.42a |
| 底层土壤Subsoil | 5.72±0.11a | 2.23±0.22b | 0.14±0.01b | 15.93±1.06a | 25.57±3.79a |
| SOC: 土壤有机碳Soil organic carbon; TN: 总氮Total nitrogen; SWC: 土壤水分Soil water content; 表中数据表示平均值±标准误差, 不同小写字母表示环境因子在表层和底层土壤具有显著差异(P<0.05) | |||||
通过高通量测序, 本试验共获得了2879个真菌OTU和3013个细菌OTU。真菌和细菌的丰富度和Shannon指数的平均值在底层土壤均低于表层土壤, 但是仅真菌丰富度和细菌Shannon多样性指数在表层和底层的差异达到显著(表 2)。PCoA排序图表明, 细菌和真菌群落的结构在表层和底层土壤有明显区分, 且细菌群落结构在表层和底层土壤的差异明显高于真菌(图 1)。PERMAOVA检验表明, 土壤剖面深度显著影响了细菌(P < 0.01)和真菌(P < 0.01)的群落结构。
| 土壤深度 Soil depth |
细菌Bacteria | 真菌Fungi | |||
| Chao指数 | Shannon指数 | Chao指数 | Shannon指数 | ||
| 表层土壤Topsoil | 1704.83±46.93a | 5.92±0.02a | 591.36±31.29a | 3.65±0.18a | |
| 底层土壤Subsoil | 1633.76±75.37a | 5.66±0.07b | 376.47±29.21b | 3.38±0.13a | |
| 表中数据表示平均值±标准误差, 不同小写字母表示多样性指数在表层和底层土壤具有显著差异(在P < 0.05水平下) | |||||
|
| 图 1 色季拉山暗针叶林土壤真菌和细菌群落结构随剖面深度的变化特征(β-多样性) Fig. 1 Characteristics of Fungal and Bacterial Community Structure in dark coniferous forest in Mount Segrila with soil depth (β-diversity) |
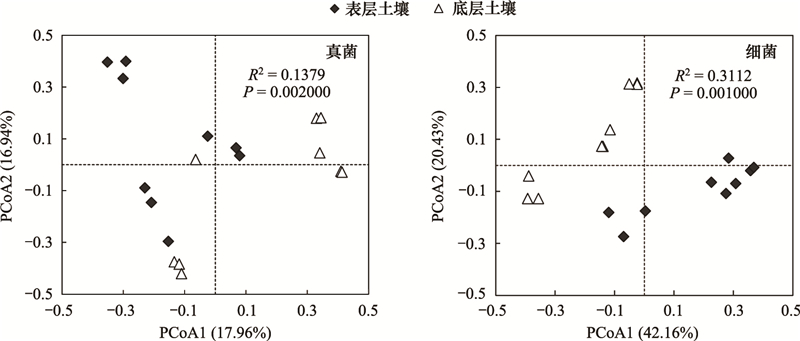
在本研究采集的土壤样品中, 子囊菌门(Ascomycota)、担子菌门(Basidiomycota)和接合菌门(Zygomycota) 是色季拉山暗针叶林土壤真菌的主要门类, 其平均相对丰度分别为40.41%、34.67%和18.52%。在纲水平下, 伞菌纲(Agaricomycetes, 平均相对丰度23.60%)、接合菌门未分类纲(Zygomycota_incertae_sedis, 18.52%)、座囊菌纲(Dothideomycetes, 9.82%)和银耳纲(Tremellomycetes, 8.35%)等是土壤真菌群落的优势种群。不同真菌种群对剖面深度的响应有明显异同(图 2), 例如子囊菌门未分类纲(Ascomycota_unclassified)、Dothideomycetes和Tremellomycetes表层土壤的相对丰度(4.10±0.98、15.90±2.47、14.58±4.61)均显著高于底层土壤(2.59±0.52、3.75±1.08、2.12±0.72), 而古菌根菌纲(Archaeorhizomycetes)和Zygomycota_incertae_sedis的相对丰度则在底层土壤(6.20±2.23、28.41±9.29)显著高于表层土壤(1.21±0.40、8.64±2.24)。
|
| 图 2 土壤真菌(纲水平)和细菌(门水平)群落优势种群(平均相对丰度大于1%)的相对丰度随剖面深度的变化特征 Fig. 2 Variations in the relative abundance of major taxonomic groups (> 1%) for fungal (at the class level) and bacterial (at the phylum level) community with profile depth 图中标注的*表示在不同土壤深度有显著差异(* P < 0.05; ** P < 0.01; *** P < 0.001) |
在细菌群落中, 变形菌门(Proteobacteria, 平均相对丰度32.71%)、酸杆菌门(Acidobacteria, 平均相对丰度31.91%)、绿弯菌门(Chloroflexi, 平均相对丰度10.25%)和放线菌门(Actinobacteria, 平均相对丰度9.0%)是主要的优势种群。土壤深度对不同细菌种群丰度的影响也存在差异, 例如拟杆菌门(Bacteroidetes)和Proteobacteria表层土壤的相对丰度(5.11±0.94、36.31±1.98)显著高于底层土壤(1.29±0.45、29.11±3.11), 而Chloroflexi的相对丰度则在底层土壤(14.09±2.36)显著高于表层土壤(6.08±1.38)。
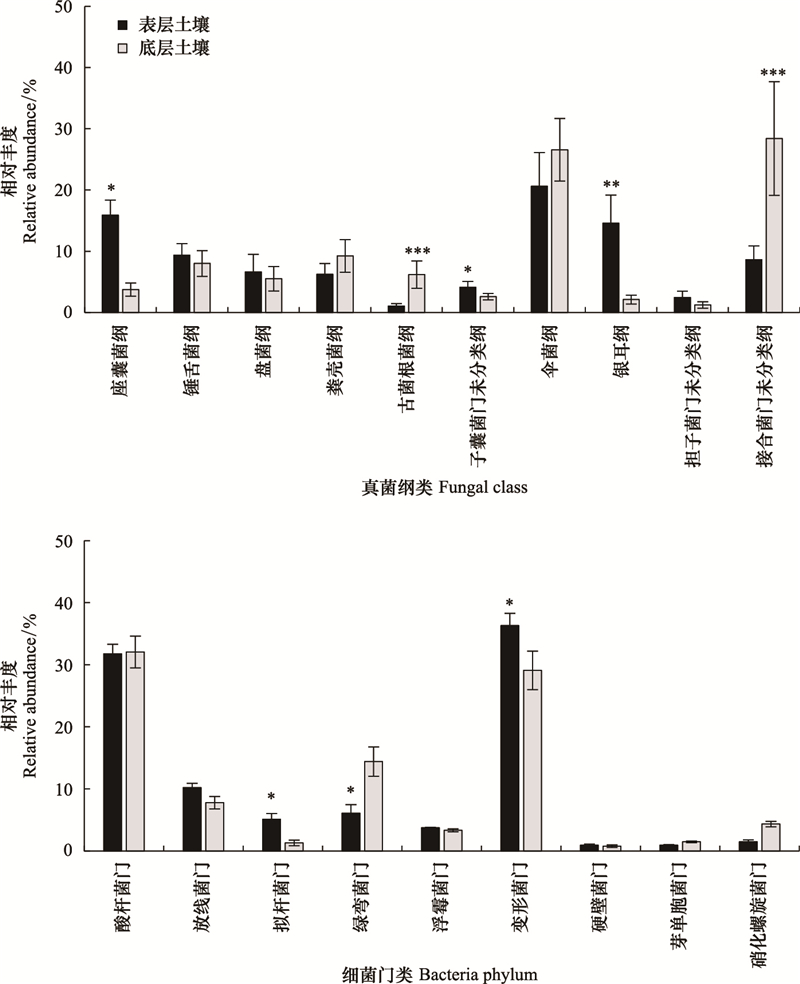
2.4 真菌和细菌分子生态网络结构特征基于高通量测序数据通过随机矩阵理论分别构建了表层和底层土壤微生物分子生态网络(图 3)。随土壤深度增加, 真菌网络的模块数减少, 而节点和连线增加(表 3)。表层和底层土壤真菌网络中的连接均以负相关为主, 并且表层土壤负相关的程度(222条, 占所有连线的98.23%)高于底层土壤(195条、占底层土壤总连线的65.22%)。对表层和底层土壤真菌网络的拓扑性质分析表明, 随土壤深度增加, 真菌网络的复杂程度(平均连接度)、群落聚类程度(平均聚类系数)、物种(节点)之间的距离(平均路径距离)以及模块性(表征微生物群落组织的有序性)均升高。相比于真菌, 细菌的分子生态网络更为复杂。随土壤深度增加, 细菌网络的模块和节点减少, 而连线增加。与真菌网落相反, 表层和底层土壤细菌网络中的连接都以正相关为主, 并且随土壤深度增加而升高:表层正相关连接占总连线的69.48%, 而底层为75.21%。相比于表层土壤, 底层土壤细菌网络的平均连接度和平均聚类系数升高, 而平均路径距离和模块性降低。
|
| 图 3 不同剖面深度下土壤真菌和细菌群落的共现性网络 Fig. 3 Co-occurrence network of fungal and bacterial communities at different soil profile depths 每个节点代表一个OUT, 节点的颜色代表细菌(门水平)和真菌(纲水平)分类。紫色连线表示负相关, 蓝色连线表示正相关 |
| 网络指数 Network Indexes |
真菌Fungi | 细菌Bacteria | |||
| 表层土壤 Topsoil |
底层土壤 Subsoil |
表层土壤 Topsoil |
底层土壤 Subsoil |
||
| 节点Node | 67 | 86 | 665 | 602 | |
| 连线Link | 226 | 299 | 1445 | 1916 | |
| 平均连接度Average connectivity | 6.746 | 6.953 | 4.346 | 6.365 | |
| 平均聚类系数Average clustering coefficient | 0.032 | 0.211 | 0.128 | 0.225 | |
| 平均路径距离Average path distance | 2.757 | 2.972 | 5.588 | 4.83 | |
| 模块性(模块数)Modularity(No. of module) | 0.223(6) | 0.397(4) | 0.669(83) | 0.620(56) | |
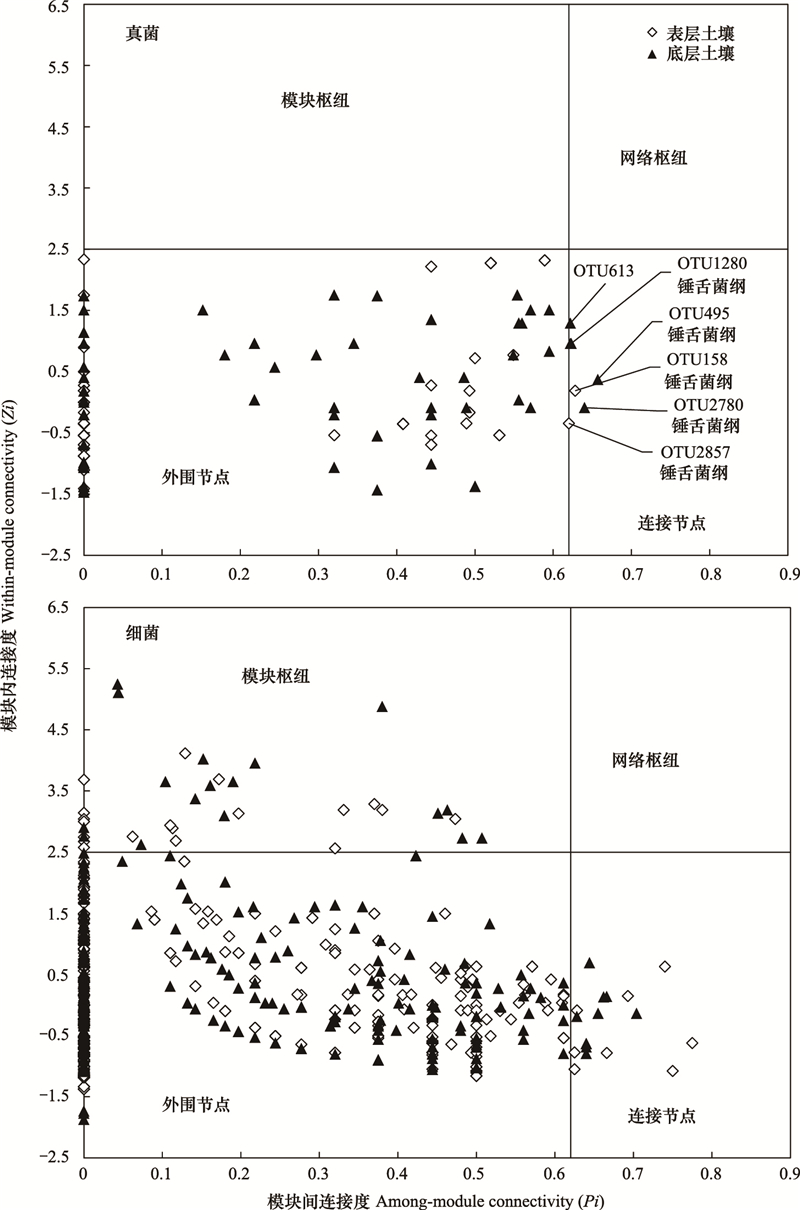
模块枢纽和连接节点是微生物群落中的关键基因, 能将各自模块内或其他模块中的许多节点连接起来。从基于网络拓扑性质的Zi-Pi分布图(图 4)可以看出, 底层土壤的真菌网络含有4个连接节点, 其中三个属于锤舌菌纲(Leotiomycetes)。表层土壤的真菌网络含有2个连接节点, 同样属于Leotiomycetes。真菌网络中余下96%的OTU都是外围节点, 没有出现模块枢纽和网络枢纽。相比于真菌, 细菌网络的拓扑结构更为复杂。表层土壤的细菌网络含有20个模块枢纽, 多数属于Acidobacteria(6个OTU)和Proteobacteria(7个OTU), 而底层土壤含有17个模块枢纽, 多数属于Acidobacteria(6个OTU)和Actinobacteria(4个OTU)(表 4)。表层土壤细菌网络含有9个连接节点, 多数属于Actinobacteria(5个OTU), 而底层土壤含有8个连接节点, 多数属于Proteobacteria(5个OTU)。此外, 属于Chloroflexi、Planctomycetes和SHA-109门下的某些OTU仅在表层土壤细菌网络中作为模块枢纽或者连接节点, 而Firmicutes、Gemmatimonadetes、WCHB1-60和BRC1门下的某些OTU仅在底层土壤细菌网络中作为模块枢纽或者连接节点(表 4)。
|
| 图 4 基于土壤真菌和细菌网络拓扑性质的Zi-Pi散点图 Fig. 4 Zi-Pi scatter plot based on topological properties of fungal and bacterial networks |
| 细菌分类(门) Bacteria taxa(phylum) |
模块枢纽Module hubs | 连接节点Connectors | |||
| 表层土壤Topsoil | 底层土壤Subsoil | 表层土壤Topsoil | 底层土壤Subsoil | ||
| 酸杆菌门Acidobacteria | OTU34, OTU181, OTU1054, OTU2907, OTU1885, OTU1228 |
OTU115, OTU126, OTU565, OTU1410, OTU1953, OTU2794 |
|||
| 放线菌门Actinobacteria | OTU2091 | OTU1287, OTU2410, OTU2537, OTU2872 |
OTU454, OTU530, OTU610, OTU1121, OTU1676 |
||
| 变形菌门Proteobacteria | OTU710, OTU1094, OTU2168, OTU2344, OTU2597, OTU2597, OTU1925 |
OTU1694, OTU1463, OTU974 |
OTU2862 | OTU16, OTU1126, OTU1471, OTU1781, OTU2216, |
|
| 拟杆菌门Bacteroidetes | OTU2171, OTU2315 | OTU1259, OTU1829 | OTU2738 | ||
| 绿弯菌门Chloroflexi | OTU1667 | OTU1517 | |||
| 浮霉菌门Planctomycetes | OTU2534, OTU2614 | ||||
| SHA-109 | OTU1403 | ||||
| 硬壁菌门Firmicutes | OTU2024 | ||||
| 芽单胞菌Gemmatimonadetes | OTU1944, OTU866 | OTU1388 | |||
| WCHB1-60 | OTU198 | ||||
| Candidate_division_BRC1 | OTU784 | ||||
土壤环境因子(例如土壤pH值、有机质、土壤含水量等)的变化显著影响土壤微生物群落的组成和活性, 因此不同深度下土壤环境因子的差异能够导致土壤微生物群落发生显著的变化[5]。本研究结果显示, 在色季拉山暗针叶林带, 土壤有机碳含量随土壤深度增加而显著下降(表 1), 而土壤真菌和细菌的多样性也随土壤深度增加而显著降低(表 2)。土壤有机碳是土壤微生物主要的能量来源和营养物质, 其数量和质量对土壤微生物多样性有显著影响[24]。同时, 土壤细菌和真菌群落结构在表层和底层土壤有显著差异(图 1), 并且不同深度下土壤有机碳含量的变化与多种微生物种群具有显著相关性, 例如Bacteroidetes(r=0.641, P < 0.01)、Chloroflexi(r=-0.705, P < 0.01)、Dothideomycetes(r=0.609, P < 0.01)和Tremellomycetes(r=0.596, P < 0.01)等。在藏西北高寒区域的研究结果也表明, 土壤有机碳含量的变化是调控不同剖面深度下土壤微生物群落组成和结构差异的关键因子[17]。不过, 尽管土壤微生物群落在表层和底层土壤有显著差异, 群落构建优势种群的种类没有发生明显变化。本研究结果显示, 真菌Agaricomycetes、Dothideomycetes、Tremellomycetes等纲以及细菌Acidobacteria、Proteobacteria、Actinobacteria、Chloroflexi等门在表层和底层土壤均为群落中的优势种群。与本研究相似, 孙怀博[3]在青藏高原阿里地区的研究也发现, 放线菌(41.3%)、变形菌(9.3%)、酸杆菌(7%)、芽单胞菌门(7.2%)、绿弯菌门(5.9%)等在表层土壤(0—15 cm)和底层土壤(16—30 cm)都是土壤细菌群落中的优势种群。这种土壤微生物优势菌群在剖面垂直分布的相似性可能是由于这些微生物种群中的微生物扩散作用强、分布广泛或者对环境变化的适应能力较强, 因而容易形成随机性和广泛性的分布特征[25]。
微生物分子生态网络结构具有无边界、小世界、模块化和层次结构等特点, 并且微生物网络拓扑结构的特征有助于揭示不同微生物种群之间的复杂相互作用关系[26]。本研究发现, 随着土壤深度的增加, 真菌和细菌网络的平均连接度和平均聚类系数均升高(表 3), 说明底层土壤微生物网络变得更复杂, 群落组织有序性更高。土壤养分和资源分布的异质性是影响微生物群落构建的重要原因, 土壤养分(尤其是碳氮)含量的增加能够提高土壤微生物的数量, 从而促进更复杂的相互作用关系。例如, Zhou等[15]发现生物结皮发展过程中土壤微生物网络的复杂性(平均聚类系数)随土壤养分状况的改善而逐渐增加。然而, 本研究中土壤碳氮养分均随剖面加深而降低, 而微生物网络却在底层土壤更为复杂。Morriën等的研究表明[27], 相比于较为简单的网络, 具有更加复杂网络的微生物群落具有更高效的资源利用循环和信息传输过程。因此, 造成色季拉山深层土壤微生物网络更加复杂的原因, 很可能是底层土壤有限的营养物质促使微生物群落演替发展出更加高效的养分利用效率[27-28]。本研究还发现, 真菌网络的平均路径距离随土壤深度而增加, 而细菌网络的平均路径距离则随土壤深度增加而降低。平均路径距离能够表征网络中物种(节点)之间连接的紧密性, 平均路径距离越小意味着该网络中物种之间相互作用越紧密。不过, 连接更加紧密的群落同时也意味着外界干扰在网络中扩散的速度更快, 对干扰的响应可能更加敏感[12], 因此这意味着深层真菌网络在应对干扰时更加稳定, 而细菌正相反。另外, 随土壤深度的增加, 真菌的网络模块性增加, 细菌网络模块性降低。网络模块性质通常用表指征系统对外界干扰的抵抗能力, 网络模块性越高, 其抵抗外界干扰能力越强[29], 因此色季拉山暗针叶林土壤中真菌网络在底层土壤更加稳定, 而细菌网络则在表层土壤更加稳定。此外, 土壤真菌和细菌网络结构的差异还反映在正负连接(即物种相互作用)的差异上:真菌网络以负相互作用为主, 而细菌网络以正相互作用为主(图 3)。微生物网络中的正相关连接可能反映了物种之间存在协同合作或者生态位重叠, 而负相关连接可能意味着竞争作用或者生态位分化[30-31]。真菌的个体通常相对较大, 生长速度相对细菌较慢, 并且许多真菌能够与针叶林树种形成外生菌根, 各自占据一定生态位, 可能因此在分子生态网络中表现出负连接为主。而细菌由于个体小、生长速度快、随机扩散作用强, 很容易出现生态位重叠, 因此其分子网络具有更多的正连接。但是, 理论上生态位重叠会导致竞争, 表现出物种间的负相互作用。尽管微生物分子生态网络分析近年来在研究物种间相互作用中的应用逐渐增加, 但是网络拓扑学特征对实际群落构建的解释还需要更多研究和验证, 因此获得的分子生态网络与真实情况可能存在一定的差异。不过, 分子生态网络中正负连接比例的变化可能更加有助于理解微生物相互作用关系对环境变化的响应。例如在Zhou等[15]的研究中, 细菌分子生态网络也以正连接为主, 但是随着土壤养分和水分的增加, 细菌网络中负相关连接的比例增加, 这与以往养分/水分增加促进细菌群落物种竞争的研究结果[32]一致。本研究中, 相比于表层土壤, 底层土壤真菌和细菌网络的正连接的比例都增加, 说明微生物种群正相互作用随着土壤深度而增加。这暗示着在更深层的土壤中, 微生物可能通过更高程度的协同合作获取有限的养分资源或者抵抗底层土壤中的环境胁迫[33]。
微生物网络结构中的每个模块象征着微生物群落的一个集合, 在这个集合中的物种彼此联系紧密, 但是和其他模块中的物种联系较少。模块内部的关键连接者(模块枢纽)和模块之间的连接者(连接节点)是群落结构的关键物种, 对维持群落结构稳定性起着重要的作用[23]。本研究发现, 相比于表层土壤, 底层土壤细菌和真菌网络的模块数、模块枢纽和连接节点都下降, 这说明土壤微生物群落的稳定性程度可能随剖面深度增加而降低。真菌网络中的连接节点在表层和底层土壤均属于Leotiomycetes(图 4), 说明这类真菌对色季拉山暗针叶林土壤真菌网络构建有重要意义。Leotiomycetes是土壤真菌群落中的的优势物种(平均相对丰度8.5%), 属于子囊菌门。子囊菌门是寒带和温带森林生态系统重要的土壤微生物类群, 尤其对凋落物分解过程有重要意义。例如, 在欧洲针阔混交林的研究发现, Leotiomycetes以及子囊菌门下Sordariomycetes和Eurotiomycetes的真菌种群是土壤胞外酶分泌的主要种群[34]。在真菌网络中没有出现模块枢纽(Generalists), 其他研究也报道过有些网络中不存在Generalists[35]。不过, Montoya等[9]通过蛋白互作网络证明Generalists中的模块枢纽相对更重要, 因为模块枢纽数量的增加能增加网络的稳定性。
相比于真菌, 土壤细菌网络的功能节点物种较为丰富, 并且这些节点所属生物学分类以及功能在表层与底层土壤网络中存在明显异同。土壤细菌网络功能节点中的大多数属于Acidobacteria、Actinobacteria及Proteobacteria这三个门(表 4), 而这三个门也是色季拉山暗针叶林土壤细菌群落中的优势种群。Acidobacteria在土壤中数量庞大、分布广泛, 在各种有机物质的降解过程以及碳、氢和铁元素的生物地球循环过程中有重要作用。本研究发现, Acidobacteria门下的OTU在表层和底层土壤细菌网络中均为模块枢纽, 说明其主要起着维持细菌网络稳定的作用。Actinobacteria也是土壤细菌中的重要种群, 能够分解较为复杂的有机碳源物质, 例如多糖和酚类化合物[36], 并且一些放线菌具有菌丝体结构。本研究中, Actinobacteria门下的OTU在表层土壤网络中均为连接节点, 主要起连接微生物网络的作用, 而在底层土壤网络中多发挥着稳定模块内部种群(模块枢纽)的作用。Proteobacteria是细菌中种类和代谢多样性最大的一门, 是土壤碳、氮、硫等生物地球化学循环过程中的核心参与者。本研究中Proteobacteria门下的OTU均在表层和底层土壤细菌网络中有关键作用, 并且在表层土壤主要是维持群落稳定(模块枢纽), 而在底层土壤则主要起连接微生物网络的作用(连接节点)。除了优势种群, 本研究还发现, 某些相对丰度较低的种群, 例如硬壁菌门(Firmicutes)、芽单胞菌门(Gemmatimonadetes)、SHA-109等门下的物种也在土壤中起着维持网络稳定或连接网络模块的作用(表 4)。此类微生物在群落中的数量虽不多, 但在微生物共生网络中起着非常关键的作用, 但是目前对其中复杂的相互作用关系尚未研究清楚。未来对这些细菌种群生理生态学的研究有助于揭示藏东南暗针叶林土壤微生物网络构建和稳定性机制, 并促进对高寒地区土壤微生物生态系统功能及其对环境变化的响应与反馈的认识。
4 结论在藏东南色季拉山暗针叶林带, 随着土壤剖面深度的增加, 土壤真菌和细菌的多样性显著降低, 同时群落组成和结构也发生了显著的改变。土壤真菌和细菌的分子生态网络都随着土壤深度的增加而变得更复杂, 但是深层土壤中真菌网络对环境变化可能更加敏感, 而细菌网络则在面对环境变化时可能具有更高的稳定性。锤舌菌纲是维持土壤真菌网络构建和稳定的关键种群, 而细菌网络中关键种群的数量及种类则包括变形菌门、酸杆菌、放线菌、硬壁菌等十余种门类, 且其网络功能在不同土壤深度有明显区别。研究结果有助于加深对青藏高原独特的微生物地理区系的认识, 并为深入理解及预测全球变化对高寒区生态系统的影响提供理论依据。
| [1] |
林先贵, 胡君利. 土壤微生物多样性的科学内涵及其生态服务功能. 土壤学报, 2008, 45(5): 892-900. DOI:10.3321/j.issn:0564-3929.2008.05.016 |
| [2] |
胡婵娟, 傅伯杰, 刘国华, 靳甜甜, 刘宇. 黄土丘陵沟壑区典型人工林下土壤微生物功能多样性. 生态学报, 2009, 29(2): 727-733. DOI:10.3321/j.issn:1000-0933.2009.02.022 |
| [3] |
孙怀博. 青藏高原阿里地区土壤细菌群落多样性及其分布的研究[D]. 南京: 南京农业大学, 2013.
|
| [4] |
邵元元, 王志英, 邹莉, 吴韶平. 百菌清对落叶松人工防护林土壤微生物群落的影响. 生态学报, 2011, 31(3): 819-829. |
| [5] |
Fierer N, Schimel J P, Holden P A. Variations in microbial community composition through two soil depth profiles. Soil Biology and Biochemistry, 2003, 35(1): 167-176. DOI:10.1016/S0038-0717(02)00251-1 |
| [6] |
Van Der Heijden M G A, Bardgett R D, Van Straalen N M. The unseen majority: soil microbes as drivers of plant diversity and productivity in terrestrial ecosystems. Ecology Letters, 2008, 11(3): 296-310. DOI:10.1111/j.1461-0248.2007.01139.x |
| [7] |
Wagg C, Bender S F, Widmer F, Van Der Heijden M G A. Soil biodiversity and soil community composition determine ecosystem multifunctionality. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(14): 5266-5270. DOI:10.1073/pnas.1320054111 |
| [8] |
McCann K S. The diversity-stability debate. Nature, 2000, 405(6783): 228-233. DOI:10.1038/35012234 |
| [9] |
Montoya J M, Pimm S L, Solé R V. Ecological networks and their fragility. Nature, 2006, 442(7100): 259-264. DOI:10.1038/nature04927 |
| [10] |
Holland M D, Hastings A. Strong effect of dispersal network structure on ecological dynamics. Nature, 2008, 456(7223): 792-794. DOI:10.1038/nature07395 |
| [11] |
Fuhrman J A. Microbial community structure and its functional implications. Nature, 2009, 459(7244): 193-199. DOI:10.1038/nature08058 |
| [12] |
Deng Y, Jiang Y H, Yang Y F, He Z L, Luo F, Zhou J Z. Molecular ecological network analyses. BMC Bioinformatics, 2012, 13(1): 113. DOI:10.1186/1471-2105-13-113 |
| [13] |
Luo F, Zhong J X, Yang Y F, Scheuermann R H, Zhou J Z. Application of random matrix theory to biological networks. Physics Letters A, 2006, 357(6): 420-423. DOI:10.1016/j.physleta.2006.04.076 |
| [14] |
Lu L H, Yin S X, Liu X, Zhang W M, Gu T Y, Shen Q R, Qiu H Z. Fungal networks in yield-invigorating and -debilitating soils induced by prolonged potato monoculture. Soil Biology and Biochemistry, 2013, 65: 186-194. DOI:10.1016/j.soilbio.2013.05.025 |
| [15] |
Zhou H, Gao Y, Jia X H, Wang M M, Ding J J, Cheng L, Bao F, Wu B. Network analysis reveals the strengthening of microbial interaction in biological soil crust development in the Mu Us Sandy Land, northwestern China. Soil Biology and Biochemistry, 2020, 144: 107782. DOI:10.1016/j.soilbio.2020.107782 |
| [16] |
Xu M, Li X L, Cai X B, Gai J P, Li X L, Christie P, Zhang J L. Soil microbial community structure and activity along a montane elevational gradient on the Tibetan Plateau. European Journal of Soil Biology, 2014, 64: 6-14. DOI:10.1016/j.ejsobi.2014.06.002 |
| [17] |
Chu H Y, Sun H B, Tripathi B M, Adams J M, Huang R, Zhang Y J, Shi Y. Bacterial community dissimilarity between the surface and subsurface soils equals horizontal differences over several kilometers in the western Tibetan Plateau. Environmental Microbiology, 2016, 18(5): 1523-1533. DOI:10.1111/1462-2920.13236 |
| [18] |
卢慧, 赵珩, 盛玉钰, 丛微, 王秀磊, 李迪强, 张于光. 基于高通量测序的两种高寒草甸土壤原核生物群落特征研究. 生态学报, 2018, 38(22): 8080-8087. |
| [19] |
Luo T X, Li W H, Zhu H Z. Estimated biomass and productivity of natural vegetation on the Tibetan Plateau. Ecological Applications, 2002, 12(4): 980-997. DOI:10.1890/1051-0761(2002)012[0980:EBAPON]2.0.CO;2 |
| [20] |
马和平, 郭其强, 刘合满, 钱登锋. 藏东南色季拉山西坡土壤有机碳库研究. 生态学报, 2013, 33(10): 3122-3128. |
| [21] |
Wang J T, Cao P, Hu H W, Li J, Han L L, Zhang L M, Zheng Y M, He J Z. Altitudinal distribution patterns of soil bacterial and archaeal communities along Mt. Shegyla on the Tibetan Plateau. Microbial Ecology, 2014, 69(1): 135-145. |
| [22] |
杜军, 高荣, 马鹏飞, 刘远明, 周刊社. 西藏色齐拉山地区立体气候特征初步分析. 高原山地气象研究, 2009, 29(1): 14-18. DOI:10.3969/j.issn.1674-2184.2009.01.003 |
| [23] |
Olesen J M, Bascompte J, Dupont Y L, Jordano P. The modularity of pollination networks. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(50): 19891-19896. DOI:10.1073/pnas.0706375104 |
| [24] |
Øvreås L, Torsvik V. Microbial diversity and community structure in two different agricultural soil communities. Microbial Ecology, 1998, 36(3/4): 303-315. |
| [25] |
贺纪正, 葛源. 土壤微生物生物地理学研究进展. 生态学报, 2008, 28(11): 5571-5582. DOI:10.3321/j.issn:1000-0933.2008.11.041 |
| [26] |
Faust K, Raes J. Microbial interactions: from networks to models. Nature Reviews Microbiology, 2012, 10(8): 538-550. DOI:10.1038/nrmicro2832 |
| [27] |
Morriën E, Hannula S E, Snoek L B, Helmsing N R, Zweers H, De Hollander M, Soto R L, Bouffaud M L, Buée M, Dimmers W, Duyts H, Geisen S, Girlanda M, Griffiths R I, Jørgensen H B, Jensen J, Plassart P, Redecker D, Schmelz R M, Schmidt O, Thomson B C, Tisserant E, Uroz S, Winding A, Bailey M J, Bonkowski M, Faber J H, Martin F, Lemanceau P, De Boer W, Van Veen Johannes A, Van Der Putten W H. Soil networks become more connected and take up more carbon as nature restoration progresses. Nature Communications, 2017, 8: 14349. DOI:10.1038/ncomms14349 |
| [28] |
Wagg C, Schlaeppi K, Banerjee S, Kuramae E E, Van Der Heijden M G A. Fungal-bacterial diversity and microbiome complexity predict ecosystem functioning. Nature Communications, 2019, 10: 4841. DOI:10.1038/s41467-019-12798-y |
| [29] |
Carpenter S R, Arrow K J, Barrett S, Biggs R, Brock W A, Crépin A S, Engstr m G, Folke C, Hughes T P, Kautsky N, Li C Z, McCarney G, Meng K, Mäler K G, Polasky S, Scheffer M, Shogren J, Sterner T, Vincent J R, Walker B, Xepapadeas A, Zeeuw A D. General resilience to cope with extreme events. Sustainability, 2012, 4(12): 3248-3259. DOI:10.3390/su4123248 |
| [30] |
Deng Y, Zhang P, Qin Y J, Tu Q C, Yang Y F, He Z L, Schadt C W, Zhou J Z. Network succession reveals the importance of competition in response to emulsified vegetable oil amendment for uranium bioremediation. Environmental Microbiology, 2016, 18(1): 205-218. DOI:10.1111/1462-2920.12981 |
| [31] |
Ghoul M, Mitri S. The ecology and evolution of microbial competition. Trends in Microbiology, 2016, 24(10): 833-845. DOI:10.1016/j.tim.2016.06.011 |
| [32] |
Wang S, Wang X B, Han X G, Deng Y. Higher precipitation strengthens the microbial interactions in semi-arid grassland soils. Global Ecology and Biogeography, 2018, 27(5): 570-580. DOI:10.1111/geb.12718 |
| [33] |
Hoek T A, Axelrod K, Biancalani T, Yurtsev E A, Liu J H, Gore J. Resource availability modulates the cooperative and competitive nature of a microbial cross-feeding mutualism. PLoS Biology, 2016, 14(8): e1002540. DOI:10.1371/journal.pbio.1002540 |
| [34] |
Schneider T, Keiblinger K M, Schmid E, Sterflinger-Gleixner K, Ellersdorfer G, Roschitzki Bernd, Richter A, Eberl L, Zechmeister-Boltenstern S, Riedel K. Who is who in litter decomposition? Metaproteomics reveals major microbial players and their biogeochemical functions. The ISME Journal, 2012, 6(9): 1749-1762. DOI:10.1038/ismej.2012.11 |
| [35] |
荀卫兵. 长期不同施肥下旱地红壤细菌群落结构特征及驱动因子的探究[D]. 南京: 南京农业大学, 2015.
|
| [36] |
Větrovský T, Steffen K T, Baldrian P. Potential of cometabolic transformation of polysaccharides and lignin in lignocellulose by soil Actinobacteria. PLoS One, 2014, 9(2): e89108. DOI:10.1371/journal.pone.0089108 |
 2021, Vol. 41
2021, Vol. 41