文章信息
- 李靖宇, 张肖冲, 陈韵, 靳新影, 马志山, 金多, 苏亮文
- LI Jingyu, ZHANG Xiaochong, CHEN Yun, JIN Xinying, MA Zhishan, JI Duo, SU Liangwen
- 腾格里沙漠东南缘藻结皮与藓结皮放线菌多样性及其潜在代谢功能
- Potential functions of actinobacteria diversity in cyanobacteria and moss crusts in the southeastern Tengger Desert
- 生态学报. 2020, 40(5): 1590-1601
- Acta Ecologica Sinica. 2020, 40(5): 1590-1601
- http://dx.doi.org/10.5846/stxb201901070055
-
文章历史
- 收稿日期: 2019-01-07
- 网络出版日期: 2019-12-17
生物土壤结皮(Biological Soil Crusts, BSCs)广泛分布于全球干旱、半干旱地区, 主要由蓝藻、绿藻、地衣、藓类和微生物以及其他生物体通过菌丝体、假根和分泌物等与土壤表层颗粒胶结形成十分复杂的地表覆盖体, 在防风固沙, 改善表层土壤生态功能方面发挥着重要作用[1-3]。土壤微生物作为BSCs的重要组成部分, 可通过分解植物残体并将其转化成土壤有机质和土壤腐殖质来改善土壤质量, 通过胞外多糖、金属螯合剂及其分泌的肽将钠、钾、镁、钙、锰、铁、镍、铜和锌等土壤养分进行保持和浓缩, 造成BSCs和BSCs下层化学性质的巨大差异[4-5]。自养和异养微生物参与甚至主导BSCs中的生物地球化学循环和能量流动[6]。不同发育演替阶段BSCs的微生物组成差异会导致固碳、固氮效率等方面存在明显不同[6-8]。Liu等[9]研究了不同恢复年限的BSCs对腾格里沙漠土壤微生物生物量的影响, 结果表明人工植被固沙区不同类型BSCs均可显著提高土壤微生物生物量碳和氮, 随着固沙年限的推移微生物生物量碳和氮含量也在增加, 表明BSCs的定殖和发展有利于土壤微生物特性和质量的改善[10]。
放线菌广泛分布于不同生态系统中, 其中干旱生境是发现放线菌新菌种最丰富的生态系统之一[11]。宏基因组学研究结果表明腾格里沙漠东南缘藓结皮土壤微生物组主要类群为放线菌[12]。BSCs作为沙漠极端环境中的生命存在形式之一, 可以通过生物化学反应路径固定大气中的氮和二氧化碳等物质, 进而推动形成复杂的微生物组[12]。微生物组的每一个成员都是复杂代谢网络的载体, 输入到其中的物质与能量经过代谢网络的转化形成复杂的代谢物库。放线菌是临床有用天然产物化学多样性的最重要来源之一, 也是新化学结构发掘的重要宝库[13]。在自然环境中, 放线菌生活的环境条件和生态相互作用可能影响天然产物的生物合成[14]。目前, 我们对这些环境条件的理解以及天然产物的生态作用尚处于起步阶段[14]。栖息在沙漠中的放线菌能够在极度干燥的条件下生长, 并且包含着能够合成独特抗菌活性化合物的基因簇[11]。通过对文献数据的分析, Mohammadipanah等人提出了开发适应干旱环境的放线菌以及探索干旱生态系统放线菌分布的必要性[11]。Sun等[15]人通过高通量测序研究表明, 放线菌是巴丹吉林沙漠和腾格里沙漠主要的微生物类群, 在属水平上Arthrobacter spp.和Kocuria spp.是腾格里沙漠的优势类群;通过分离培养的方法发现Streptomyces spp.是腾格里沙漠可培养放线菌的主要类群。
基于已报道文献, 关于腾格里沙漠东南缘广泛分布的藻结皮与藓结皮的放线菌群落组成及其潜在代谢功能的比较研究还较少。本文利用宏基因组学技术通过对藻结皮和藓结皮土壤微生物组的测序, 较为深入的分析了两种不同类型的BSCs中放线菌种群的分布特征、组成及其潜在代谢功能。可为开发利用极端环境条件下放线菌种资源、挖掘特定放线菌株的基因资源提供重要线索, 也为深入评估放线菌资源的生物学特性、潜在功能和应用奠定基础。
1 材料与方法 1.1 研究区域与样品采集研究区域设在宁夏回族自治区中卫市沙坡头地区腾格里沙漠东南缘。该地区年均气温10.0℃, 低温极值-25.1℃, 高温极值38.1℃, 全年日照时数3264 h, 年均降水量186.2 mm(1956—2012年), 年潜在蒸发量3000 mm, 年均风速2.9 m/s, 年均沙暴天数59 d[16]。1956年, 为确保包头-兰州铁路顺利通过腾格里沙漠东南缘, 通过设置沙障以及草方格, 在此建立了长16 km, 宽0.7 km(铁路北侧约0.5 km, 南侧约0.2 km)的植被保护带。随着沙子表面稳定后, 在草方格中种植旱生灌木, 如油蒿(Artemisia ordosica)、柠条锦鸡儿(Caragana koshinskii)和细枝岩黄芪(Hedysarum scoparium)等。到目前为止, 该地区的局部环境得到改善, 大量的BSCs覆盖了80%以上的土壤表面, 包括蓝藻结皮约占10%, 地衣结皮约占50%—60%以及藓结皮约占20%—30%[17]。
取样之前对腾格里沙漠东南缘进行现场考察后结合卫星图像, 实验预设了藻结皮6个重复, 藓结皮6个重复, 并按照空间交叉布局, 即沿着样带按照藻结皮-藓结皮交替预设取样点, 相邻两样点之间的距离大约为1 km。2017年9月17日选取沙坡头地区BSCs形成带藻结皮与藓结皮预设样点附近发育良好的位点进行取样, 每个样点按照五点取样法进行取样, 取样深度为各种结皮形成的自然厚度(藻结皮厚度范围约1—6 mm, 藓结皮厚度范围约11—22 mm), 将土样带回实验室, 过2 mm筛子, 将处理后的12个样品冷冻保存, 用于后续宏基因组DNA提取分析。藻结皮样品总有机碳含量范围为4.42—7.90 g/kg, 总氮含量范围为0.31—0.71 g/kg, 总磷含量范围为0.20—0.32 g/kg, 总钾含量范围为13.58—15.22 g/kg;藓结皮样品总有机碳含量范围为8.17—11.91 g/kg, 总氮含量范围为0.57—0.77 g/kg, 总磷含量范围为0.25—0.43 g/kg, 总钾含量范围为12.99—15.35 g/kg。取样点见图 1。
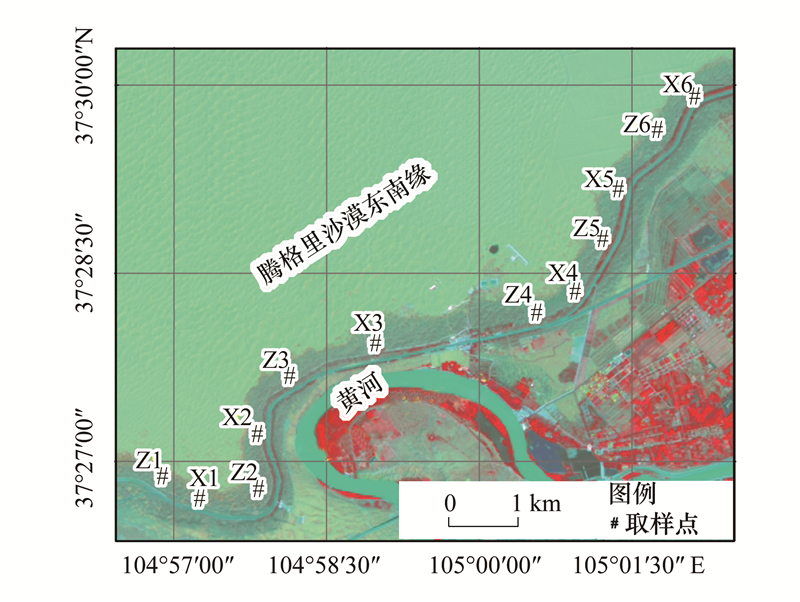
|
| 图 1 采样点位置图 Fig. 1 Sampling sites X1—X6代表藓结皮土壤样品, X1—X6 represent the moss crust samples;Z1—Z6代表藻结皮土壤样品, Z1—Z6 represent the cyanobacteria crust samples |
按照FastDNA Spin Kit for Soil (MP Biomedicals, Santa Ana, CA, USA)提取试剂盒说明书提取土壤宏基因组DNA, 利用1%琼脂糖凝胶电泳检测抽提的基因组DNA。将纯化后的宏基因组DNA破碎成片段, 按照TruSeqTM DNA Sample Prep Kit试剂盒说明书构建PE(paired-end)文库。然后根据HiSeq 3000/4000 PE Cluster Kit试剂盒说明书进行桥式PCR扩增。随后按照Illumina公司提供的标准实验流程, 通过HiSeq 2000高通量测序平台(Illumina)进行测序。
1.3 放线菌多样性及其功能分析首先对原始测序数据进行质量控制, 使用软件Seqprep(https://github.com/jstjohn/SeqPrep)剪切序列3′端和5′端的adapter序列, 使用软件Sickle(https://github.com/najoshi/sickle)去除质量剪切后长度小于50 bp、平均质量值低于20以及含N碱基的reads, 保留高质量的pair-end reads。质控后得到的短片段序列, 利用IDBA-UD(http://i.cs.hku.hk/~alse/hkubrg/projects/idba_ud/)软件针对不同测序深度短reads进行从头拼接组装, 从小k-mer开始到大k-mer不断迭代处理, 每次迭代根据设定阈值从低到高依次删掉短的和低深度的contigs, 逐步完成低深度和高深度短reads的拼接组装。使用MetaGene (http://metagene.cb.k.u-tokyo.ac.jp/)对拼接结果中的contigs进行ORFs(open reading frame)预测。将所有样品预测出来的基因序列, 用CD-HIT软件(http://www.bioinformatics.org/cd-hit/)进行聚类(默认参数为:95% identity、90% coverage), 每个类取最长的基因作为代表序列, 构建非冗余基因集。使用SOAPaligner软件(http://soap.genomics.org.cn/), 分别将每个样品的高质量reads与非冗余基因集进行比对(默认参数为:95% identity), 统计基因在对应样品中的丰度信息。使用BLASTP(BLAST Version 2.2.28 +, http://blast.ncbi.nlm.nih.gov/Blast.cgi)将非冗余基因集与NR数据库进行比对(BLAST比对参数设置期望值e-value为1e-5), 并通过NR库对应的分类学信息数据库获得物种注释结果。使用BLASTP(BLAST Version 2.2.28+, http://blast.ncbi.nlm.nih.gov/Blast.cgi)将非冗余基因集序列与KEGG的基因数据库(GENES)进行比对, BLAST比对参数设置期望值e-value为1e-5。根据比对结果使用KOBAS 2.0(KEGG Orthology Based Annotation System)进行功能注释。使用KO、Pathway、EC、Module对应的基因丰度总和计算该功能类别的丰度。最后通过上海美吉生物医药科技有限公司开发的I-Sanger云平台(www.i-sanger.com)挑选放线菌的物种注释信息和KEGG注释信息, 并进行Circos作图分析、PCA分析(Principal Component Analysis)、相似性分析(ANOSIM)、组间显著性差异检验分析以及物种与功能贡献度分析。
2 结果与分析 2.1 藻结皮与藓结皮放线菌群落组成腾格里沙漠东南缘藻结皮与藓结皮土壤微生物组主要形成以地嗜皮菌属(Geodermatophilus)、红色杆菌属(Rubrobacter)、类诺卡氏菌属(Nocardioides)、游动放线菌属(Actinoplanes)、芽生球菌属(Blastococcus)、链霉菌属(Streptomyces)、贫养杆菌属(Modestobacter)、糖丝菌属(Saccharothrix)、土壤红杆菌属(Solirubrobacter)、假诺卡氏菌属(Pseudonocardia)、小单孢子菌属(Micromonospora)、康奈斯氏杆菌属(Conexibacter)、大理石雕菌属(Marmoricola)、小月菌属(Microlunatus)以及弗兰克氏菌属(Frankia)等为主要类群的放线菌群落结构特征(图 2)。主成分分析结果表明主成分1可以解释藻结皮与藓结皮放线菌群落差异的36.01%, 主成分2可以解释27.86%, 主成分3可以解释9.45%。ANOSIM分析结果表明放线菌群落在藻结皮与藓结皮之间存在显著差异(图 2)。
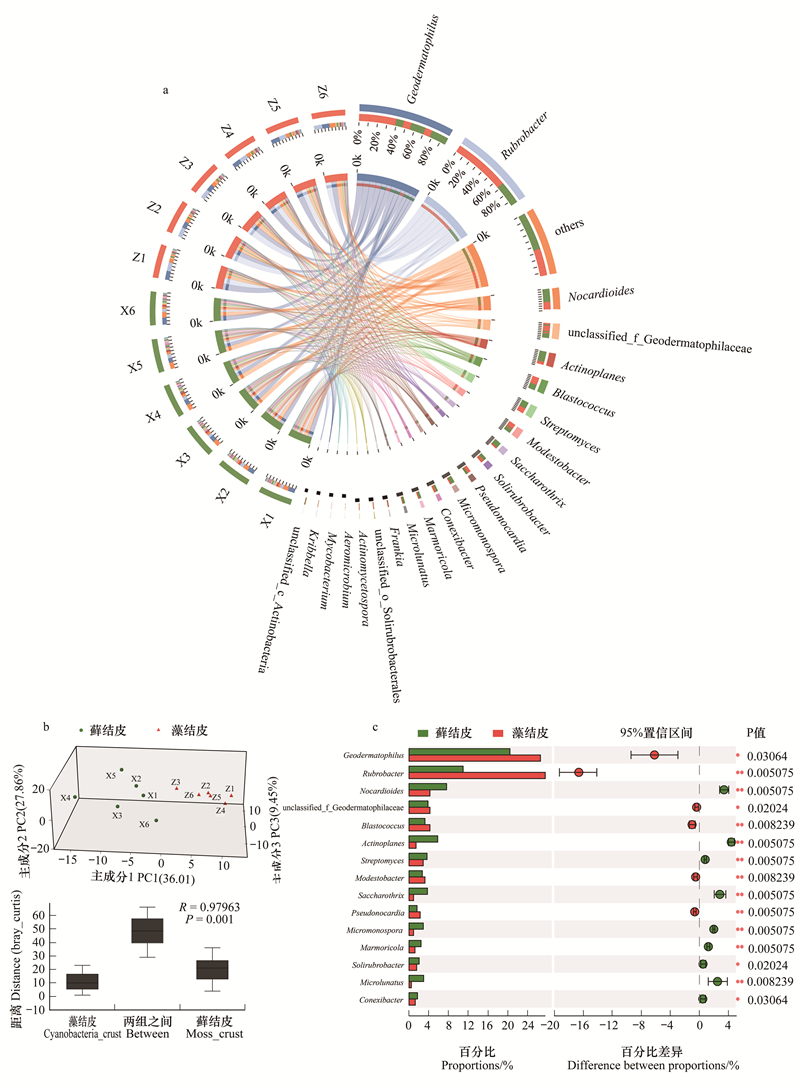
|
| 图 2 藻结皮与藓结皮放线菌门各属的Circos图, 主成分分析, ANOSIM比较分析和显著性差异分析 Fig. 2 Circos diagram, principal component analysis, ANOSIM comparative analysis and significant difference analysis for different genera in Actinobacteria phylum between cyanobacteria crust and moss crust Geodermatophilus地嗜皮菌属;Rubrobacter红色杆菌属;Nocardioides类诺卡氏菌属;unclassified_f_Geodermatophilaceae未分类的地嗜皮菌科属;Actinoplanes游动放线菌属;Blastococcus芽生球菌属;Streptomyces链霉菌属;Modestobacter贫养杆菌属;Saccharothrix糖丝菌属;Solirubrobacter土壤红杆菌属;Pseudonocardia假诺卡氏菌属;Micromonospora小单孢子菌属;Conexibacter康奈斯氏杆菌属;Marmoricola大理石雕菌属;Microlunatus小月菌属;Frankia弗兰克氏菌属;unclassified_o__Solirubrobacterales未分类的土壤红杆菌属;Actinomycetospora放线产孢菌属;Aeromicrobium气微菌属;Mycobacterium分支杆菌;Kribbella韩国生工菌属 |
地嗜皮菌属作为该地区藻结皮与藓结皮的优势放线菌类群之一, 在藻结皮中其相对丰度达到26.60%, 而藓结皮中其相对丰度为20.41%, 二者之间存在显著差异。红色杆菌属是藻结皮中相对丰度最高的放线菌类群, 其值达到27.55%;而该类群在藓结皮中的相对丰度为10.94%, 显著低于藻结皮中的相对丰度。类诺卡氏菌属在藓结皮(7.61%)中的相对丰度要显著高于藻结皮(4.23%);游动放线菌属在藓结皮(5.81%)中的相对丰度要显著高于藻结皮(1.42%);链霉菌属在藓结皮(3.66%)中的相对丰度要显著高于藻结皮(2.87%);糖丝菌属在藓结皮(3.73%)中的相对丰度要显著高于藻结皮(0.93%);小单孢子菌属在藓结皮(2.89%)中的相对丰度要显著高于藻结皮(0.92%);大理石雕菌属在藓结皮(2.43%)中的相对丰度要显著高于藻结皮(1.21%);土壤红杆菌属在藓结皮(2.04%)中的相对丰度要显著高于藻结皮(1.56%);小月菌属在藓结皮(2.96%)中的相对丰度要显著高于藻结皮(0.47%)(图 2)。
2.2 藻结皮与藓结皮放线菌群落代谢特征基于KEGG数据库对腾格里沙漠东南缘藻结皮与藓结皮土壤微生物组中放线菌群落的宏基因组数据进行了代谢功能注释, 结果表明相对丰度平均值(n=12)高于1%的代谢功能分类主要包括碳代谢(6.11%)、氨基酸生物合成(5.21%)、氧化磷酸化(4.32%)、嘌呤代谢(3.50%)、嘧啶代谢(3.60%)、氨基糖与核苷酸糖代谢(2.49%)、糖酵解/糖异生(2.04%)、原核生物中的碳固定途径(2.07%)、丙酮酸代谢(1.93%)、三羧酸循环(1.80%)、脂肪酸代谢(1.89%)、甘氨酸/丝氨酸和苏氨酸代谢(1.87%)、丙酸代谢(1.48%)、乙醛酸和二羧酸盐代谢(1.67%)、肽聚糖生物合成(1.27%)、丙氨酸/天门冬氨酸和谷氨酸代谢(1.46%)、丁酸代谢(1.51%)、淀粉与蔗糖代谢(1.18%)、甲烷代谢(1.35%)、2-羰基羧酸代谢(1.28%)、果糖与甘露糖代谢(1.52%)、磷酸戊糖途径(1.48%)、半乳糖代谢(1.37%)、半胱氨酸和蛋氨酸代谢(1.45%)、脂肪酸生物合成(1.44%)、缬氨酸/亮氨酸与异亮氨酸降解(1.06%)、精氨酸与脯氨酸代谢(1.07%)、脂多糖生物合成(1.08%)、甘油磷脂代谢(1.08%)和叶酸合成(1.02%)等(图 3)。
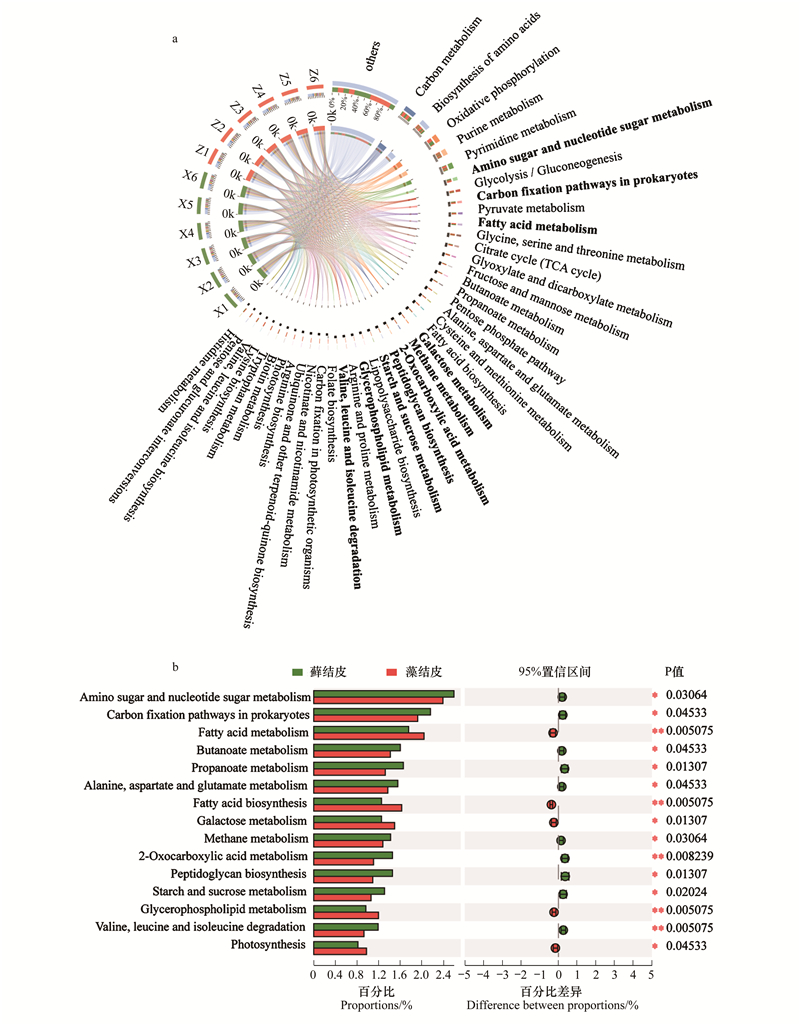
|
| 图 3 藻结皮与藓结皮放线菌门各属代谢功能分类Circos图和组间显著性差异分析 Fig. 3 Circos diagram and significant difference analysis for metabolism functional classification of different genera in Actinobacteria phylum between cyanobacteria crust and moss crust Carbon metabolism碳代谢;Biosynthesis of amino acids氨基酸生物合成;Oxidative phosphorylation氧化磷酸化;Purine metabolism嘌呤代谢;Pyrimidine metabolism嘧啶代谢;Amino sugar and nucleotide sugar metabolism氨基糖与核苷酸糖代谢;Glycolysis/Gluconeogenesis糖酵解/糖异生;Carbon fixation pathways in prokaryote原核生物中的碳固定途径;Pyruvate metabolism丙酮酸代谢;Fatty acid metabolism脂肪酸代谢;Glycine, serine and threonine metabolism甘氨酸、丝氨酸和苏氨酸代谢;Citrate cycle (TCA cycle)三羧酸循环;Glyoxylate and dicarboxylate metabolism乙醛酸和二羧酸盐代谢;Fructose and mannose metabolism果糖与甘露糖代谢;Butanoate metabolism丁酸代谢;Propanoate metabolism丙酸代谢;Pentose phosphate pathway磷酸戊糖途径;Alanine, aspartate and glutamate metabolism丙氨酸、天门冬氨酸和谷氨酸代谢;Cysteine and methionine metabolism半胱氨酸和蛋氨酸代谢;Fatty acid biosynthesis脂肪酸生物合成;Galactose metabolism半乳糖代谢;Methane metabolism甲烷代谢;2-Oxocarboxylic acid metabolism 2-羰基羧酸代谢;Peptidoglycan biosynthesis肽聚糖生物合成;Starch and sucrose metabolism淀粉与蔗糖代谢;Lipopolysaccharide biosynthesis脂多糖生物合成;Glycerophospholipid metabolism甘油磷脂代谢;Arginine and proline metabolism精氨酸与脯氨酸代谢;Valine, leucine and isoleucine degradation缬氨酸、亮氨酸与异亮氨酸降解;Folate biosynthesis叶酸合成;Carbon fixation in photosynthetic organisms光合生物中的固碳;Nicotinate and nicotinamide metabolism烟酸和烟酰胺代谢;Ubiquinone and other terpenoid-quinone biosynthesis泛醌及其他萜类醌生物合成;Arginine biosynthesis精氨酸生物合成;Photosynthesis光合作用;Biotin metabolism生物素代谢;Tryptophan metabolism色氨酸代谢;Lysine biosynthesis赖氨酸生物合成;Valine, leucine and isoleucine biosynthesis缬氨酸、亮氨酸与异亮氨酸生物合成;Pentose and glucuronate interconversions戊糖和葡糖醛酸盐相互转化;Histidine metabolism组氨酸代谢 |
相对丰度前5的代谢功能, 包括碳代谢、氨基酸生物合成、氧化磷酸化、嘌呤代谢以及嘧啶代谢, 在藻结皮和藓结皮之间没有显著差异。氨基糖与核苷酸糖代谢、原核生物中的碳固定途径、丁酸代谢、丙酸代谢、丙氨酸/天门冬氨酸和谷氨酸代谢、甲烷代谢、2-羰基羧酸代谢、肽聚糖生物合成、淀粉与蔗糖代谢以及缬氨酸/亮氨酸与异亮氨酸降解在藓结皮中显著高于藻结皮(图 3)。
2.3 藻结皮与藓结皮放线菌群落KEGG模块功能分析KEGG模块是手动定义的功能单元的集合, 用于测序基因组的注释和生物学解释。基于KEGG数据库对腾格里沙漠东南缘藻结皮与藓结皮土壤微生物组中放线菌群落的宏基因组数据进行了模块功能注释, 结果表明细菌核糖体模块的相对丰度最高, 藓结皮中相对丰度为14.63%, 藻结皮中相对丰度为10.51%, 且两种结皮类型存在显著差异。其次丰度最大的为原核生物NADH-醌氧化还原酶功能模块, 藓结皮中相对丰度为4.70%, 藻结皮中相对丰度为4.44%, 两种结皮类型之间不存在显著差异。相对丰度平均值(n=12)高于1%的其他功能模块主要包括三羧酸循环、氨酰-tRNA生物合成(真核生物)、ABC转运系统、ABC-2型转运系统、肌苷一磷酸生物合成、F型ATP酶(原核生物和叶绿体)、细胞色素C氧化酶(原核生物)、脂肪酸生物合成(延伸)、脂多糖转运系统、尿苷一磷酸生物合成、核黄素生物合成、磷酸戊糖途径、赖氨酸生物合成、分泌系统、磷酸盐饥饿反应双组分调节系统、糖酵解、脂多糖生物合成、肽/镍转运系统、烟酰胺腺嘌呤二核苷酸生物合成、异亮氨酸生物合成、三酰甘油生物合成、dTDP-L-鼠李糖生物合成、Ⅱ型分泌途径、RNA聚合酶(细菌)、DNA聚合酶Ⅲ复合物(细菌)等。其中, 三羧酸循环、氨酰-tRNA生物合成(真核生物)、ABC-2型转运系统、dTDP-L-鼠李糖生物合成以及DNA聚合酶Ⅲ复合物(细菌)等功能模块在藓结皮中显著高于藻结皮(图 4)。
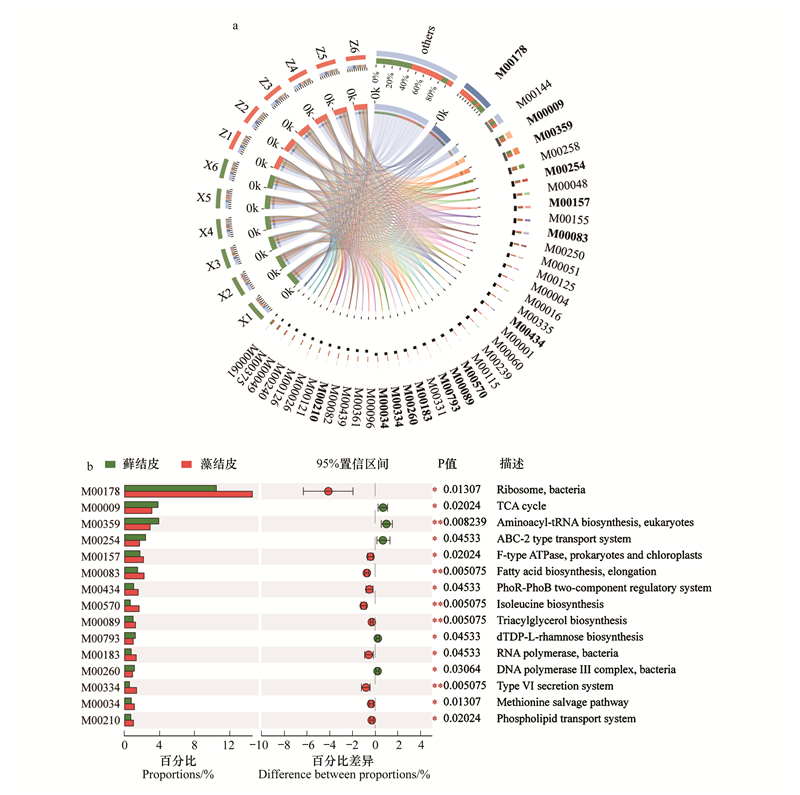
|
| 图 4 藻结皮与藓结皮放线菌门各属的Module功能分类Circos图和组间显著性差异分析 Fig. 4 Circos diagram and significant difference analysis for Module functional classification of different genera in Actinobacteria phylum between cyanobacteria crust and moss crust M00178:核糖体(细菌) Ribosome(bacteria);M00144:NADH:醌氧化还原酶(原核生物) NADH:quinone oxidoreductase(prokaryotes);M00009:三羧酸循环Citrate cycle;M00359:氨酰-tRNA生物合成(真核生物) Aminoacyl-tRNA biosynthesis(eukaryotes);M00258:公认的ABC转运系统Putative ABC transport system;M00254:ABC-2型转运系统ABC-2 type transport system;M00048:肌苷一磷酸生物合成Inosine monophosphate biosynthesis;M00157:F型ATP酶(原核生物和叶绿体) F-type ATPase(prokaryotes and chloroplasts);M00155:细胞色素C氧化酶(原核生物) Cytochrome c oxidase(prokaryotes);M00083:脂肪酸生物合成(延伸) Fatty acid biosynthesis(elongation);M00250:脂多糖转运系统Lipopolysaccharide transport system;M00051:尿苷一磷酸生物合成Uridine monophosphate biosynthesis;M00125:核黄素生物合成Riboflavin biosynthesis;M00004:磷酸戊糖途径Pentose phosphate pathway;M00016:赖氨酸生物合成Lysine biosynthesis;M00335:分泌系统secretion system;M00434:磷酸盐饥饿反应双组分调节系统PhoR-PhoB two-component regulatory system;M00001:糖酵解Glycolysis;M00060:脂多糖生物合成Lipopolysaccharide biosynthesis;M00239:肽/镍转运系统Peptides/nickel transport system;M00115:烟酰胺腺嘌呤二核苷酸生物合成NAD biosynthesis;M00570:异亮氨酸生物合成Isoleucine biosynthesis;M00089:三酰甘油生物合成Triacylglycerol biosynthesis;M00793:dTDP-L-鼠李糖生物合成dTDP-L-rhamnose biosynthesis;M00331:Ⅱ型分泌途径Type II general secretion pathway;M00183:RNA聚合酶(细菌) RNA polymerase(bacteria);M00260:DNA聚合酶Ⅲ复合物(细菌) DNA polymerase III complex(bacteria);M00334:Ⅵ型分泌系统Type VI secretion system;M00096:C5类异戊二烯生物合成C5 isoprenoid biosynthesis;M00361:核苷酸糖生物合成(真核生物) Nucleotide sugar biosynthesis(eukaryotes);M00439:寡肽转运系统Oligopeptide transport system;M00082:脂肪酸生物合成(起始) Fatty acid biosynthesis(initiation);M00210:磷脂转运系统Phospholipid transport system;M00121:血红素生物合成Heme biosynthesis;M00026:组氨酸生物合成Histidine biosynthesis;M00126:四氢叶酸生物合成Tetrahydrofolate biosynthesis;M00240:铁配合物转运系统Iron complex transport system;M00049:腺嘌呤核糖核酸生物合成Adenine ribonucleotide biosynthesis;M00375:羟基丙酸酯-羟基丁酸酯循环Hydroxypropionate-hydroxybutylate cycle;M00061:葡萄糖醛酸降解D-Glucuronate degradation |
基于样本中物种和功能的对应关系, 进行物种与功能相对丰度之间的关联分析, 找出特定物种的功能贡献度。选择了物种相对丰度前10和功能丰度前10的KEGG功能分类水平-3进行了物种和功能对应分析。结果表明, 在碳代谢功能分类中, 地嗜皮菌属和红色杆菌属的相对贡献在藻结皮中达到61.45%, 而在藓结皮中为38.69%;在氨基酸生物合成功能分类中, 地嗜皮菌属和红色杆菌属的相对贡献在藻结皮中达到61.37%, 而在藓结皮中为36.43%;在ABC转运体功能分类中, 地嗜皮菌属和红色杆菌属的相对贡献在藻结皮中达到46.36%, 而在藓结皮中为15.38%;在嘌呤代谢功能分类中, 地嗜皮菌属和红色杆菌属的相对贡献在藻结皮中达到60.51%, 而在藓结皮中为36.79%;在群体感应功能分类中, 地嗜皮菌属和红色杆菌属的相对贡献在藻结皮中达到51.11%, 而在藓结皮中为29.92%;在嘧啶代谢功能分类中, 地嗜皮菌属和红色杆菌属的相对贡献在藻结皮中达到60.67%, 而在藓结皮中为37.11%;在氧化磷酸化功能分类中, 地嗜皮菌属和红色杆菌属的相对贡献在藻结皮中达到56.71%, 而在藓结皮中为33.74%;在乙醛酸和二羧酸盐代谢功能分类中, 地嗜皮菌属和红色杆菌属的相对贡献在藻结皮中达到60.78%, 而在藓结皮中为38.01%;在丙酮酸代谢功能分类中, 地嗜皮菌属和红色杆菌属的相对贡献在藻结皮中达到61.08%, 而在藓结皮中为40.14%;在原核生物的碳固定途径功能分类中, 地嗜皮菌属和红色杆菌属的相对贡献在藻结皮中达到61.04%, 而在藓结皮中为39.15%(图 5)。从藻结皮和藓结皮这两种类型来看, 地嗜皮菌属和红色杆菌属对以上10中功能分类的贡献度在藓结皮中显著低于藻结皮, 而在藓结皮中, 类诺卡氏菌属、芽生球菌属、贫养杆菌属、游动放线菌属、链霉菌属、假诺卡氏菌属和糖丝菌属等对这些功能的相对贡献具有重要作用(见图 5)。
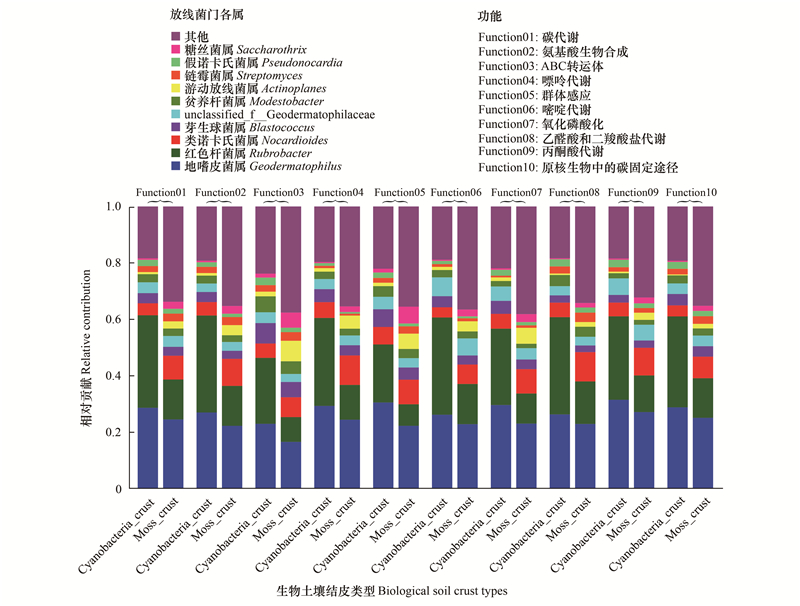
|
| 图 5 藻结皮与藓结皮放线菌门各属的物种与功能贡献度分析 Fig. 5 Species and functional contribution analysis for different genera in Actinobacteria phylum between cyanobacteria crust and moss crust |
放线菌多样性是腾格里沙漠东南缘藻结皮和藓结皮土壤微生物组的重要组成部分, 是BSCs进行生物地球化学循环的重要参与者。放线菌各属在藻结皮和藓结皮中的分布情况对于全面、深入认识BSCs的发育、演替提供了物种多样性数据。物种多样性以生命的形式将众多指导生物化学反应的基因多样性整合在一起, 构建起了复杂的代谢网络, 为放线菌适应极端环境、推动物质循环和能量流动提供了保障, 而这些复杂的代谢网络成为了临床有用天然产物化学多样性挖掘、开发、利用的潜在重要“生产线”。这可能有助于对BSCs中放线菌生态功能的深入理解和认识。从藻结皮到藓结皮的演替过程中, 放线菌多样性及其群落结构变化明显, 优势类群差异明显, 特别是在碳代谢等具体生态功能方面, 优势类群地嗜皮菌属和红色杆菌属的贡献较大, 这对于深入理解放线菌在生物土壤结皮演替过程中的生态功能具有重要的科学意义。
地嗜皮菌属作为藻结皮和藓结皮的优势属之一。该属微生物广泛分布于干旱极端环境条件下, 对于能够产生活性氧的辐射、紫外线以及干燥等环境胁迫具有很强的抗性[18]。通过比较发现, 藻结皮土壤微生物组中地嗜皮菌属相对丰度显著高于藓结皮。随着BSCs从藻结皮向藓结皮演替的过程中, 放线菌的优势属发生了显著变化, 局部环境的改善减少了地嗜皮菌属的相对丰度。这也间接说明了地嗜皮菌属在藻结皮适应极端干旱环境过程中具有更加重要的生态功能。同属于地嗜皮菌科的属主要包括地嗜皮菌属(模式属)、芽生球菌属和贫养杆菌属, 这三个属都呈现出藻结皮土壤微生物组中的相对丰度显著高于藓结皮。地嗜皮菌科不仅与石灰岩退化过程密切相关, 甚至能够腐蚀岩石, 而且对干旱、高温、离子辐射和低营养等恶劣环境具有较强的耐受性[19]。地嗜皮菌属的模式种Geodermatophilus obscurus Luedemann 1968全基因组测序以及生理特性研究结果表明, 其基因组(G+C)含量为74%, 基因多拷贝, 可大量产生细胞外功能性细菌淀粉样蛋白, 菌落呈现黑色[20]。有研究者从突尼斯和埃及的沙漠石头和纪念碑上分离到地嗜皮菌属菌株12株、芽生球菌属菌株5株以及贫养杆菌属菌株3株, 对这些菌株的酯酶酶学性质研究表明, 地嗜皮菌科具有丰富且独特的酯酶活性, 这些酯酶的活性与极度干旱、高温、强电离辐射及营养供给匮乏等为特征的严酷生态环境直接相关[21]。
红色杆菌属作为藻结皮相对丰度最大的类群, 在藓结皮中相对丰度显著下降。红杆菌属最早由Suzuki等人于1988年建立, 他们从日本的一个放射性温泉中分离出一种高度抗伽马辐射的细菌, 即Rubrobacter radiotolerans[22]。从此以后, 科学家们相继从工业排污热水、温泉、地热流以及火山口周围土壤等高温环境或非高温环境(如教堂墙壁生物膜、海绵)中分离出嗜热、耐盐或对γ射线具有高度抗性的红杆菌属菌株, 命名为Rubrobacter xylanophilus、Rubrobacter taiwanensis、Rubrobacter calidiflumis、Rubrobacter naidicus、Rubrobacter bracarensis、Rubrobacter aplysinae、Rubrobacter bracarensis[23]。红色杆菌属放线菌广泛分布于阿塔卡马沙漠、哈萨克斯坦极端干旱沙漠土壤、澳大利亚干旱土壤、中国腾格里沙漠以及巴丹吉林沙漠等干旱环境中[15, 24-26]。Rubrobacter radiotolerans菌株RSPS-4对电离辐射有很强的抵抗力, 全基因组测序结果表明其基因组(G+C)含量为66.91%, 含有3个环状质粒, 分别为190889 bp, 149806 bp和51047 bp, 含有3214个预测的蛋白质编码基因, 46个tRNA基因和一个rRNA操纵子, 该基因组拥有参与DNA修复的RecFOR途径的所有基因[27]。基于以上分析, 腾格里沙漠东南缘藻结皮和藓结皮中广泛分布的红色杆菌属对于适应沙漠干旱、强辐射等极端环境具有重要作用, 在以后的研究中需要加强对该类微生物的分离、纯化, 获得纯培养, 以便进一步深入探究该类微生物适应极端环境的分子机理。
随着BSCs的演替, 藓结皮土壤微生物组中类诺卡氏菌属、游动放线菌属、链霉菌属、糖丝菌属、小单孢子菌属、大理石雕菌属、土壤红杆菌属、小月菌属、康奈斯氏杆菌属相对丰度显著高于藻结皮。类诺卡氏菌属成员广泛分布于土壤、海水、火山灰、海胆、沙漠、地下水、沙滩、冰川、山地土、森林土、沉积物、药用植物(麻疯树属)以及银杏和玉米的根部等环境中[28]。游动放线菌属具有球形、圆柱形、指状、浅裂、瓶状或烧瓶状或非常不规则的孢子囊特征, 其成员已逐渐被认为是次生代谢产物的重要来源[29]。链霉菌属普遍存在于土壤中, 它们可以形成耐干燥和饥饿的孢子, 可以降解纤维素和几丁质等复杂有机分子, 在陆地碳循环中起着重要作用[30]。该属最显著的特性是其产生次生代谢物的能力, 其中许多具有重要的工业和药用价值, 这些次生代谢物之间还存在着相当大的结构多样性, 能够大量产生微生物来源的抗生素、抗真菌剂以及肿瘤抑制化合物[31]。当饥饿或环境变化导致营养生长结束时, 伴随着基因表达的一系列复杂变化, 抗生素的产生通常会被激活, 导致次生代谢产物的产生[32]。糖丝菌属的成员主要来源于土壤, 包括沙漠土壤, 是新抗生素的潜在生产者, 因能够产生对细菌和真菌具有活性的新型特殊代谢物而备受关注[33-34]。小单孢子菌属的成员主要来源于土壤、昆虫、海洋沉积物、植物、固氮根瘤、叶和根等环境中, 该属逐渐被认为是次生代谢产物的重要来源, 已从该属中分离出许多抗生素[35-36]。小单孢菌属中的内生放线菌作为潜在拮抗剂的来源, 为发现新型生物活性天然产物带来了巨大希望[36]。
4 结论腾格里沙漠东南缘藻结皮和藓结皮土壤微生物组中放线菌多样性高, 是两种BSCs的优势类群。主要形成以地嗜皮菌属和红色杆菌属为主要类群的放线菌群落结构特征。其中, 藻结皮中以红色杆菌属为优势类群, 而在藓结皮中该类群放线菌相对丰度显著下降;而藓结皮以地嗜皮菌属为优势类群, 而在藻结皮中该类群放线菌显著下降。在藻结皮向藓结皮演替的过程中, 放线菌群落结构发生了明显的变化。特别是在藓结皮中, 一些具有潜在产生重要次生代谢产物的放线菌类群的相对丰度显著高于藻结皮, 这可能会为开发、利用放线菌资源多样性提供重要参考。
| [1] |
Li X R, Tan H J, Hui R, Zhao Y, Huang L, Jia R L, Song G. Researches in biological soil crust of China:a review. Chinese Science Bulletin, 2018, 63(23): 2320-2334. DOI:10.1360/N972018-00390 |
| [2] |
Pointing S B, Belnap J. Microbial colonization and controls in dryland systems. Nature Reviews Microbiology, 2012, 10(8): 551-562. DOI:10.1038/nrmicro2831 |
| [3] |
李新荣, 张元明, 赵允格. 生物土壤结皮研究:进展、前沿与展望. 地球科学进展, 2009, 24(1): 11-24. DOI:10.3321/j.issn:1001-8166.2009.01.002 |
| [4] |
Qi Y B, Chen T, Pu J, Yang F Q, Shukla M K, Chang Q R. Response of soil physical, chemical and microbial biomass properties to land use changes in fixed desertified land. CATENA, 2018, 160: 339-344. DOI:10.1016/j.catena.2017.10.007 |
| [5] |
Yu J, Glazer N, Steinberger Y. Carbon utilization, microbial biomass, and respiration in biological soil crusts in the Negev Desert. Biology and Fertility of Soils, 2014, 50(2): 285-293. DOI:10.1007/s00374-013-0856-9 |
| [6] |
Zhang Q Y, Wang Q, Ouyang H L, Lan S L, Hu C X. Pyrosequencing reveals significant changes in microbial communities along the ecological succession of biological soil crusts in the tengger desert of China. Pedosphere, 2018, 28(2): 350-362. DOI:10.1016/S1002-0160(17)60477-6 |
| [7] |
Maier S, Tamm A, Wu D M, Caesar J, Grube M, Weber B. Photoautotrophic organisms control microbial abundance, diversity, and physiology in different types of biological soil crusts. The ISME Journal, 2018, 12(4): 1032-1046. DOI:10.1038/s41396-018-0062-8 |
| [8] |
韩炳宏, 牛得草, 贺磊, 任运涛, 吴让, 傅华. 生物土壤结皮发育及其影响因素研究进展. 草业科学, 2017, 34(9): 1793-1801. |
| [9] |
Liu Y M, Li X R, Xing Z S, Zhao X, Pan Y X. Responses of soil microbial biomass and community composition to biological soil crusts in the revegetated areas of the Tengger Desert. Applied Soil Ecology, 2013, 65: 52-59. DOI:10.1016/j.apsoil.2013.01.005 |
| [10] |
刘艳梅, 杨航宇, 李新荣. 生物土壤结皮对荒漠区土壤微生物生物量的影响. 土壤学报, 2014, 51(2): 394-401. |
| [11] |
Mohammadipanah F, Wink J. Actinobacteria from arid and desert habitats:diversity and biological activity. Frontiers in Microbiology, 2016, 6: 1541. |
| [12] |
李靖宇, 刘建利, 张琇, 王猛, 杨洲, 靳新影, 苟琪, 张翼飞. 腾格里沙漠东南缘藓结皮微生物组基因多样性及功能. 生物多样性, 2018, 26(7): 727-737. |
| [13] |
Genilloud O. Actinomycetes:still a source of novel antibiotics. Natural Product Reports, 2017, 34(10): 1203-1232. DOI:10.1039/C7NP00026J |
| [14] |
Behie S W, Bonet B, Zacharia V M, McClung D J, Traxler M F. Molecules to ecosystems:actinomycete natural products in situ. Frontiers in Microbiology, 2017, 7: 2149. |
| [15] |
Sun Y, Shi Y L, Wang H, Zhang T, Yu L Y, Sun H, Zhang Y Q. Diversity of bacteria and the characteristics of actinobacteria community structure in Badain Jaran desert and tengger desert of China. Frontiers in Microbiology, 2018, 9: 1068. DOI:10.3389/fmicb.2018.01068 |
| [16] |
李靖宇, 张琇. 腾格里沙漠不同生物土壤结皮微生物多样性分析. 生态科学, 2017, 36(3): 36-42. |
| [17] |
Lan S B, Wu L, Zhang D L, Hu C X. Analysis of environmental factors determining development and succession in biological soil crusts. Science of the Total Environment, 2015, 538: 492-499. DOI:10.1016/j.scitotenv.2015.08.066 |
| [18] |
Hezbri K, Ghodhbane-Gtari F, del Carmen Montero-Calasanz M, Nouioui I, Rohde M, Spröer C, Schumann P, Klenk H P, Gtari M. Geodermatophilus pulveris sp. nov., a gamma-radiation-resistant actinobacterium isolated from the Sahara desert. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(10): 3828-3834. DOI:10.1099/ijsem.0.001272 |
| [19] |
孙红敏, 余利岩, 张玉琴. 地嗜皮菌科放线菌的研究进展. 微生物学报, 2015, 55(12): 1521-1529. |
| [20] |
Ivanova N, Sikorski J, Jando M, Munk C, Lapidus A, del Rio T G, Copeland A, Tice H, Cheng J F, Lucas S, Chen F, Nolan M, Bruce D, Goodwin L, Pitluck S, Mavromatis K, Mikhailova N, Pati A, Chen A, Palaniappan K, Land M, Hauser L, Chang Y J, Jeffries C D, Meincke L, Brettin T, Detter J C, Rohde M, Göker M, Bristow J, Eisen J A, Markowitz V, Hugenholtz P, Kyrpides N C, Klenk H P. Complete genome sequence of Geodermatophilus obscurus type strain (G-20T). Standards in Genomic Sciences, 2010, 2(2): 158-167. DOI:10.4056/sigs.711311 |
| [21] |
Essoussi I, Ghodhbane-Gtari F, Amairi H, Sghaier H, Jaouani A, Brusetti L, Daffonchio D, Boudabous A, Gtari M. Esterase as an enzymatic signature of Geodermatophilaceae adaptability to Sahara desert stones and monuments. Journal of Applied Microbiology, 2010, 108(5): 1723-1732. DOI:10.1111/j.1365-2672.2009.04580.x |
| [22] |
Jurado V, Miller A Z, Alias-Villegas C, Laiz L, Saiz-Jimenez C. Rubrobacter bracarensis sp. nov., a novel member of the genus Rubrobacter isolated from a biodeteriorated monument. Systematic and Applied Microbiology, 2012, 35(5): 306-309. DOI:10.1016/j.syapm.2012.04.007 |
| [23] |
Norman J S, King G M, Friesen M L. Rubrobacter spartanus sp. nov., a moderately thermophilic oligotrophic bacterium isolated from volcanic soil. International Journal of Systematic and Evolutionary Microbiology, 2017, 67(9): 3597-3602. DOI:10.1099/ijsem.0.002175 |
| [24] |
Crits-Christoph A, Robinson C K, Barnum T, Fricke W F, Davila A F, Jedynak B, McKay C P, DiRuggiero J. Colonization patterns of soil microbial communities in the Atacama Desert. Microbiome, 2013, 1: 28. DOI:10.1186/2049-2618-1-28 |
| [25] |
Holmes A J, Bowyer J, Holley M P, O'Donoghue M, Montgomery M, Gillings M R. Diverse, yet-to-be-cultured members of the Rubrobacter subdivision of the Actinobacteria are widespread in Australian arid soils. FEMS Microbiology Ecology, 2000, 33(2): 111-120. DOI:10.1111/j.1574-6941.2000.tb00733.x |
| [26] |
Kutovaya O V, Lebedeva M P, Tkhakakhova A K, Ivanova E A, Andronov E E. Metagenomic characterization of biodiversity in the extremely arid desert soils of Kazakhstan. Eurasian Soil Science, 2015, 48(5): 493-500. DOI:10.1134/S106422931505004X |
| [27] |
Egas C, Barroso C, Froufe H J C, Pacheco J, Albuquerque L, da Costa M S. Complete genome sequence of the Radiation-Resistant bacterium Rubrobacter radiotolerans RSPS-4. Standards in Genomic Sciences, 2014, 9(3): 1062-1075. DOI:10.4056/sigs.5661021 |
| [28] |
Khan I U, Hussain F, Habib N, Xiao M, Ahmed I, Amin A, Zhi X Y, Li W J. Nocardioides thalensis sp. nov., isolated from a desert. International Journal of Systematic and Evolutionary Microbiology, 2017, 67(8): 2848-2852. DOI:10.1099/ijsem.0.002032 |
| [29] |
Qu Z, Bao X D, Xie Q Y, Zhao Y X, Yan B, Dai H F, Chen H Q. Actinoplanes sediminis sp. nov., isolated from marine sediment. International Journal of Systematic and Evolutionary Microbiology, 2018, 68(1): 71-75. DOI:10.1099/ijsem.0.002451 |
| [30] |
Choudoir M J, Buckley D H. Phylogenetic conservatism of thermal traits explains dispersal limitation and genomic differentiation of Streptomyces sister-taxa. The ISME Journal, 2018, 12(9): 2176-2186. DOI:10.1038/s41396-018-0180-3 |
| [31] |
Liu R, Deng Z X, Liu T G. Streptomyces species:Ideal chassis for natural product discovery and overproduction. Metabolic Engineering, 2018, 50: 74-84. DOI:10.1016/j.ymben.2018.05.015 |
| [32] |
Romero-Rodriguez A, Maldonado-Carmona N, Ruiz-Villafán B, Koirala N, Rocha D, Sánchez S. Interplay between carbon, nitrogen and phosphate utilization in the control of secondary metabolite production in Streptomyces. Antonie van Leeuwenhoek, 2018, 111(5): 761-781. DOI:10.1007/s10482-018-1073-1 |
| [33] |
Bouznada K, Bouras N, Mokrane S, Chaabane Chaouch F, Zitouni A, Pötter G, Spröer C, Klenk H P, Sabaou N. Saccharothrix isguenensis sp. nov., an actinobacterium isolated from desert soil. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(11): 4785-4790. DOI:10.1099/ijsem.0.001430 |
| [34] |
Ibeyaima A, Singh A K, Lal R, Gupta S, Goodfellow M, Sarethy I P. Saccharothrix tharensis sp. nov., an actinobacterium isolated from the Thar Desert, India. Antonie van Leeuwenhoek, 2018, 111(11): 2141--2147. DOI:10.1007/s10482-018-1106-9 |
| [35] |
Carro L, Veyisoglu A, Riesco R, Spröer C, Klenk H P, Sahin N, Trujillo M E. Micromonospora phytophila sp. nov. and Micromonospora luteiviridis sp. nov., isolated as natural inhabitants of plant nodules. International Journal of Systematic and Evolutionary Microbiology, 2018, 68(1): 248-253. DOI:10.1099/ijsem.0.002490 |
| [36] |
Zhao S S, Liu C X, Zheng W W, Ma Z X, Cao T T, Zhao J W, Yan K, Xiang W S, Wang X J. Micromonospora parathelypteridis sp. nov., an endophytic actinomycete with antifungal activity isolated from the root of Parathelypteris beddomei (Bak.) Ching. International Journal of Systematic and Evolutionary Microbiology, 2017, 67(2): 268-274. DOI:10.1099/ijsem.0.001614 |
 2020, Vol. 40
2020, Vol. 40