文章信息
- 李聪聪, 朱秉坚, 徐琳, 李香真, 姚敏杰
- LI Congcong, Zhu Bingjian, Xu Lin, LI Xiangzhen, YAO Minjie
- 高寒草甸优势植物叶内、根内与土壤原核微生物群落的分异
- Differentiations of prokaryotic communities in leaf and root endosphere of dominant plants and bulk soils in alpine meadows
- 生态学报. 2020, 40(14): 4942-4953
- Acta Ecologica Sinica. 2020, 40(14): 4942-4953
- http://dx.doi.org/10.5846/stxb201908221746
-
文章历史
- 收稿日期: 2019-08-22
- 修订日期: 2019-12-20
2. 中国科学院大学, 北京 100049;
3. 福建农林大学资源与环境学院土壤修复福建省高校工程研究中心, 福州 350002
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Engineering Research Center of Soil Remediation of Fujian Province University, College of Resources and Environment, Fujian Agriculture and Forestry University, Fuzhou 350002, China
在植物的内部(内生菌)和表面(附生菌)都存在着大量的微生物, 构成植物微生物组(plant microbiome)[1]。植物内生微生物长期与植物共同进化, 与宿主植物建立了共生关系[2]。植物内生菌参与了植物的众多生命过程, 如通过分泌生长激素(例如吲哚乙酸IAA)来促进植物生长[3], 促进植物对营养元素氮、磷的获取[4-5], 调节植物免疫系统, 增强对病原菌的抵抗力[6]等。内生微生物通常受宿主植物类型、植物不同部位、植物生长环境和土壤类型等多种因素的影响[7-8], 但关键因子随环境条件的变化而变化, 目前还没有统一的定论。
通常来说, 不同植物相同部位微生物群落存在一定的差异, 对杨树Populus, 槲皮树Quercus和松树Pinus三种植物根内微生物进行454扩增子测序, 结果表明植物类型是影响木本植物根内细菌群落差异的主要因子[9];进一步, 利用16 S rRNA基因Illumina测序技术对18种草根内和根际微生物的研究中同样发现植物类型是影响草根部微生物群落分化的主要因子[10];然而利用焦磷酸测序技术分析发现同一生长环境下的拟南芥(Arabidopsis thaliana)与其近缘种碎米荠(Cardamine hirsuta)根内微生物群落没有显著差异[11];类似的, 对仙人掌科两种植物Myrtillocactus geometrizans和Opuntia robusta采用16 S rRNA Illumina测序技术对其根内、根际微生物分析表明, 植物类型对两种植物之间根部微生物群落的分化的影响很小[12], 说明不同宿主对其微生物组的选择作用不一样。同种植物不同部位微生物组存在一定差异, Gottel等[13]利用16S rRNA基因454测序方法比较了成年杨树根内和根际细菌群落的组成, 发现二者在组成和群落结构上存在显著差异。同时, 植物的各部位又存在紧密的联系, Bai等[14]通过分离培养技术和无菌植物微生物定殖实验, 发现拟南芥根际和叶际微生物群落结构明显不同, 但微生物群落组成部分相同, 而且拥有共同的功能。土壤是植物内生菌的“种子库”[15], 影响着植物内生菌的组成, 土壤中微生物多样性显著高于植物内生微生物群落。在微生物组成上, 土壤与植物内生菌存在一定差异, 从门水平上看植物内变形菌门相对丰度较高, 而土壤中放线菌门更高[16];从更低级的分类水平上看, 例如属、种水平, 则因宿主和外界环境而异, 例如有研究发现, 干旱条件下多种草的根内链霉菌属Streptomyces显著增加, 与土壤存在显著差异[10]。尽管对不同植物地上地下不同部位微生物研究的很多, 但对自然状态下生长的植物, 尤其是严苛环境下的高寒草甸优势植物研究较少。不同优势植物根内、叶内原核微生物群落的构成、关键控制因子及其与非根际土壤微生物群落在组成和结构上的差异和联系还有待进一步探索。
青藏高原高寒草甸是一类重要的生态系统和高山植物基因库, 也是我国主要的天然牧场, 而草甸植物尤其是优势植物对草地畜牧业发展和高寒生态系统功能的维持具有重要意义。有研究发现植物内存在宿主特异性内生菌, 这些内生菌在寒冷条件仍然具有溶解矿物磷, 分解蛋白酶等功能, 在寒冷条件下表现出冷适应性[2, 17], 可能通过协助植物营养吸收等作用来帮助植物在寒冷条件下健康生长。青藏高原特殊的高寒缺氧环境, 属于全球变化的敏感区和生态系统的脆弱地区, 阐明高寒草甸优势植物与微生物的相互作用, 对系统认知高寒生态系统的结构、功能和保护有着重要意义。青藏苔草(Carex moorcroftii)、火绒草(Leontopodium jacotianum)、高山嵩草(Carex parvula)是青藏高寒草原、高寒草甸的优势植物。我们以这三种植物为对象, 研究了植物叶内、根内和非根际土壤的原核微生物群落的组成和多样性, 旨在揭示以下三个科学问题:1)叶内、根内和非根际土壤的原核微生物群落的结构和多样性的差异;2)原核微生物群落与样品来源类型(植物叶内、根内以及土壤)、不同植物类型之间的关系;3)植物叶内、根内特有的内生菌及其潜在的功能。
1 材料与方法 1.1 研究区概况本研究采样点在中国科学院青藏高原研究所那曲生态环境综合观测研究站(E 91°12′, N 30°31′)。该实验站位于藏北高原的腹地那曲县境内, 海拔均在4450 m以上, 属于高原亚寒带季风半湿润气候区, 年均降水量为406.2mm, 多集中在6—9月间, 相对湿度只有51%[18]。青藏高原上分布着各类草地面积约1.4亿hm2亩, 其中高寒草甸约占50%, 是那曲草原畜牧业的主要草地利用类型, 植被群落以高寒小嵩草草甸、藏嵩草草甸群落为主, 也是我国第五大放牧区。考虑到植物类型对植物微生物的影响, 本研究在那曲实验站的高寒草甸上选取了两种单子叶植物青藏苔草(Carex moorcroftii)、高山嵩草(Carex parvula)和一种双子叶植物火绒草(Leontopodium jacotianum)为研究对象。青藏苔草和高山嵩草属于苔属植物, 火绒草属于火绒属植物, 这三种植物是高寒草甸三种典型的草本植物, 分布广泛[19]。
1.2 采集方法于2018年7月份生长季采集植物。将整株植物挖出, 用无菌剪刀分别剪部分植物嫩叶和嫩根, 分别装在50 mL无菌离心管里, 取每一个植物样品时对应地取植物周边0—10 cm深的非根际土壤装入50 mL无菌离心管中。每种植物及其对应的土壤均采集6个重复样品。采集的样品放在冰块上运回实验室-20℃冰箱保存, 进行后续分子实验。
1.3 DNA提取、PCR扩增及高通量测序将土壤直接称量到带有无菌玻璃珠的离心管中, 用Mini-bead beater(Biospec MiniBeadbeater-16, USA)破碎;根和叶分别用灭菌后的PBS缓冲液(PBS:130 mM NaCl, 7 mM Na2HPO4, 3 mM NaH2PO4, pH 7.4)清洗6次, 充分洗掉表面附着物, 液氮磨碎后放入带有无菌玻璃珠的离心管中。土壤、叶内、根内总DNA根据MO BIO的DNA提取试剂盒(Power Soil DNA Isolation kit)的流程提取, 对提取的DNA利用NanoDrop 2000分光光度计进行质量检测, 将高质量的DNA模板统一稀释到10 ng/μl用于后续的16S rRNA基因的PCR反应。本实验中用到的扩增引物为:515F(5′-GTGYCAGCMGCCGCGGTA-3′), 909R (5′-CCCCGYCAATTCMTTTRAGT-3′)[20]。反应体系为25 μl:包括2 ×Es Taq Master Mix (康为世纪, 中国) 12.5 μl、引物各1 μl(浓度为1 μM)、DNA模板10 ng、超纯水(ddH2O) 9.5 μl。PCR反应程序为:预变性(94℃, 4 min), 变性(94 ℃, 30 s), 退火(55 ℃, 30 s), 延伸(72 ℃, 45 s)共30个循环, 最后72 ℃再延伸10 min。扩增产物利用胶回收试剂盒AxyPrep DNA Gel Extraction Kit(Axygen Biosciences, Union City, CA, USA)纯化后, 利用NanoDrop分光光度计进行质量检测, 合格的样品进行等摩尔质量混合, 使用TruSeqⓇ DNA PCR-Free Sample Preparation Kit建库试剂盒建库并利用Illumina HiSeq测序仪进行PE250测序。
1.4 生物信息处理双端测序下机数据用Flash-1.2.8软件[21]合并, 不同样品根据barcode的唯一性借助Fastx Toolkit软件(hannonlab.cshl.edu/fastxtoolkit/ind3ex.html)进行拆分。序列的质控、过滤、嵌合体的去除利用QIIME[22]平台上进行:低质量的序列(Q < 30)和长度不足300 bp的序列去掉, 嵌合体采用UCHIME[23]算法过滤, 进而得到高质量的测序数据。利用QIIME平台的CD-HIT算法将具有至少97%相似性的序列聚类为一个操作分类单元(Operational Taxonomic Units, OTU)。采用RDP classifier[24]算法对97%水平OTU进行分类学划分, 16S rRNA基因比对数据库为Greengenes(V13.8), 去除分类为叶绿体以及未分类的OTU。为了计算α多样性以及β多样性, 将所有样品随机重抽样到1000条序列, 最后总共得到3769个OTU。
1.5 统计分析用Kruskal-Wallis和Dunn′s Kruskal-Wallis Multiple Comparisons[25]检验门水平微生物类群的丰度在植物不同部位和不同植物之间的差异。不同部位相同OTUs比例的venn图由线上服务器工具构建(http://bioinfogcnb.csic.es/tools/venny/index.html)。α多样性指数包括Shannon多样性指数、物种均匀度(Pielou′s evenness index)以及观察到的物种数(Observed species)借助R中vegan包中的“diversity”函数, “specnumber”函数, “estimate”函数完成计算。利用R中的vegan包的“vegdist”函数计算Bray-Curtis相异矩阵, 然后利用vegan包中“metaMDS”函数进行非度量多维尺度分析(NMDS)。置换多元方差分析(PERMANOVAs)[26]用来检验微生物群落结构组成在不同部位和不同植物类型间的差异。利用STAMP[27](v2.1.3)软件的Kruskal-Wallis H-test和Welch′s多重检验方法找出与土壤有显著性差异的内生菌OTU。进一步, 利用PICRUSt, 基于KEGG数据库来预测叶内和根内高丰度差异OTU(相对丰度>1%)的潜在功能。
2 结果 2.1 三种植物不同部位微生物组成青藏苔草、火绒草、高山嵩草叶内、根内以及非根际土壤原核微生物在门分类水平上的组成如图 1所示。其中, 变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)在三种植物叶内和根内占据主导地位, 平均相对丰度之和高达70%以上;而土壤中则以变形菌门、放线菌门、拟杆菌门和酸杆菌门作为优势门类, 平均相对丰度之和高达60%以上。各门类在各部位和不同植物中的相对丰度以及差异性见表 1。结果表明, 放线菌门、拟杆菌门、绿弯菌门(Chloroflexi)、泉古菌门(Crenarchaeota)、浮霉菌门(Planctomycetes)、芽单胞菌(Gemmatimonadetes)、蓝细菌(Cyanobacteria)在土壤中相对丰度较高, 与根内和叶内有显著性差异(P<0.001);而叶内富含变形菌门和厚壁菌门, 与根内和土壤相比具有显著差异(P < 0.001);根内大部分门类相对丰度介于土壤和叶内之间。叶内, 根内以及土壤之间的venn如图 2, 根内与土壤有27.7%的共有OTUs, 而叶内与土壤有14%的共有OTUs, 说明植物根系微生物组装受土壤微生物群落的影响更大, 而叶微生物群落的组装则较少依赖于土壤微生物群落。另外, 土壤中存在大量特有的微生物, 比例达到42.5%。
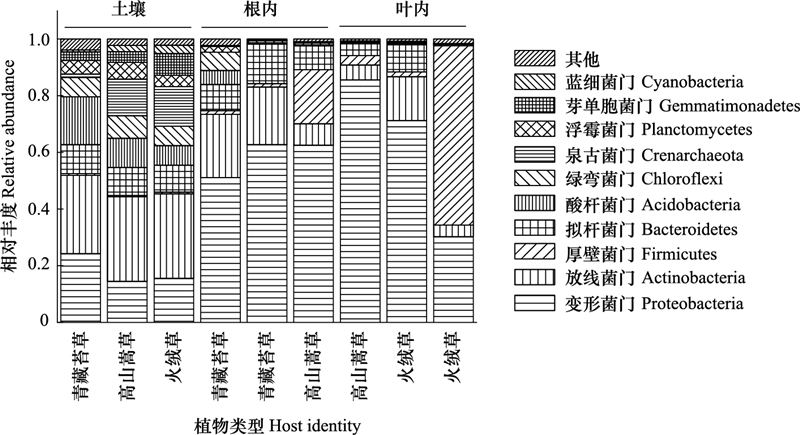
|
| 图 1 三种植物根内、叶内以及土壤原核微生物在门水平上优势群组成 Fig. 1 The relative abundances of top ten dominant (>0.1%) phyla |
| 门Phylum | 土壤Soil | 叶内Leaf endosphere | 根内Root endosphere | ||||||||
| 青藏苔草 | 高山嵩草 | 火绒草 | 青藏苔草 | 高山嵩草 | 火绒草 | 青藏苔草 | 高山嵩草 | 火绒草 | |||
| Proteobacteria | 24.28cd | 15.54d | 14.51d | 85.53a* | 30.24bcd* | 71.21ab* | 51.08abcd | 62.56abc | 62.71abc | ||
| Actinobacteria | 27.57a | 29.86a | 29.81a | 5.36bc | 4.10c | 15.49abc | 22.31ab | 7.59bc | 20.40abc | ||
| Firmicutes | 0.71c | 0.60c | 0.47c | 3.22abc* | 63.32a* | 1.67bc* | 1.37c* | 19.02ab* | 1.17c* | ||
| Bacteroidetes | 10.16a | 9.50a | 9.89a | 4.20a* | 0.45b* | 9.58a* | 9.15a | 8.49a | 14.00a | ||
| Acidobacteria | 16.91a | 6.94ab | 10.34a | 0.38bcd | 0.07d | 0.24cd | 4.94abc | 0.32cd | 0.38bcd | ||
| Chloroflexi | 6.71abc | 6.82ab | 7.92a | 0.27d | 0.07d | 0.36d | 6.43ab* | 0.63cd* | 0.75bcd* | ||
| Crenarchaeota | 1.28ab | 13.96a | 12.90a | 0.14b | 0.02b | 0.06b | 0.05b | 0.09b | 0.05b | ||
| Planctomycetes | 4.82a | 3.94ab | 5.93a | 0.23cd | 0.07d | 0.14cd | 2.06abc | 0.32bcd | 0.23cd | ||
| Gemmatimonadetes | 3.07ab | 7.71a | 3.75ab | 0.03c | 0.02c | 0.17bc | 0.27bc | 0.03c | 0.02c | ||
| Cyanobacteria | 0.66ab | 2.98a | 2.17a | 0.03bc | 0.04bc | 0.29ab | 0.08bc | 0.03bc | 0.00c | ||
| Thermi. | 0.02 | 0.18 | 0.05 | 0.35 | 1.52 | 0.09 | 1.10 | 0.47 | 0.00 | ||
| FBP | 0.24abc | 0.57ab | 1.04a | 0.08bc* | 0.00c* | 0.50ab* | 0.09bc | 0.00c | 0.03c | ||
| Armatimonadetes | 0.35a | 0.14ab | 0.20ab | 0.02b | 0.02b | 0.09ab | 0.26ab | 0.02b | 0.05ab | ||
| Nitrospirae | 0.74a | 0.14ab | 0.18a | 0.00b | 0.00b | 0.00b | 0.02b | 0.00b | 0.00b | ||
| Kruskal-Wallis和Dunn′s Kruskal-Wallis多重比较检验三种植物不同部位组间差异, 同一行不同小写字母标记的数值表示差异显著(P<0.05), 表中数据为平均相对丰度(n=6);*表示相同部位不同植物之间有差异的门 | |||||||||||
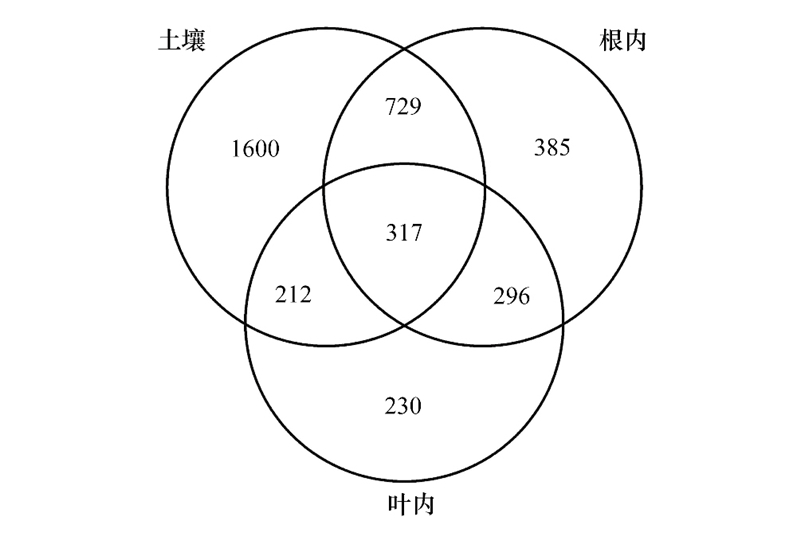
|
| 图 2 植物叶内、根内以及土壤之间特有和共有的OTUs Fig. 2 Venn diagrams showing unique and shared OTUs among different parts of plants |
从不同植物来看, 只有变形菌门、厚壁菌门、拟杆菌门、疣微菌门(Verrucomicrobia)以及FBP门在相同部位不同植物之间有显著性差异(表 1)。两种单子叶植物青藏苔草与高山嵩草叶内在变形菌门、拟杆菌门相对丰度上有显著性差异;根内在厚壁菌门、疣微菌门有显著性差异。双子叶植物火绒草与单子叶植物高山嵩草叶内在厚壁菌门、拟杆菌门以及FBP相对丰度上有显著性差异;根内在厚壁菌门上也有显著性差异。而双子叶植物火绒草与另一种单子叶植物青藏苔草各优势微生物门无显著性差异。三种植物对应的土壤间在所有门类上都无显著性差异, 说明植物对内生菌有更强选择性。总体来看, 大多数原核生物门类(13个门)在叶内、根内和土壤样品间具有显著性差异, 而少数门类(5个门)在不同植物相同部位或土壤之间具有显著性差异。说明了样品来源类型, 亦可说叶内、根内以及土壤的空间生态位分异是造成微生物组成差异的主要原因, 而植物种类对植物微生物的选择性相对较小。
2.2 植物内生原核微生物群落的α多样性我们计算了三种植物不同部位内生原核微生物群落的Shannon多样性指数、物种均匀度(Pielou′s evenness index)以及观察到的物种数(Observed species)(表 2、图 3)。相同植物不同部位叶内、根内以及土壤α多样性均具有显著性差异(P<0.001), 并且从叶内、根内再到土壤, α多样性逐渐升高。此外, 土壤样品间α多样性差异较小, 而叶内和根内样品间α多样性差异较大。从不同植物类型来看, 三种植物在土壤和根内α多样性上均具显著性差异(P<0.05), 但在叶内α多样性上没有显著性差异。另外, 青藏苔草在叶内和根内Shannon多样性、均匀度和观察到的物种数上都要高于高山嵩草和火绒草。单子叶植物青藏苔草与火绒草α多样性在土壤和根内均没有显著性差异, 而两种单子叶植物青藏苔草和高山嵩草α多样性在土壤和根内有显著性差异, 这种结果说明植物类型对微生物的选择性较弱, 与前面物种组成的分析结果是一致的。
| 样品类型 Sample Type |
植物类型 Host identity |
香浓多样性 Shannon diversity |
均匀度指数 Pielou′s evenness |
观察到的物种数 Observed species |
| 土壤Soil | 青藏苔草 | 5.78±0.21a | 0.93±0.02a | 483.83±39.52a |
| 高山嵩草 | 5.42±0.15b | 0.90±0.02b | 408.00±25.35b | |
| 火绒草 | 5.74±0.12a | 0.93±0.01a | 482.00±24.62a | |
| 根内Root endosphere | 青藏苔草 | 4.79±0.43c | 0.83±0.05c | 332.17±60.41c |
| 高山嵩草 | 3.6d±0.71e | 0.70±0.10d | 173.00±51.22d | |
| 火绒草 | 3.97±0.39d | 0.73±0.06d | 236.67±23.95d | |
| 叶内Leaf endosphere | 青藏苔草 | 3.1d±0.95e | 0.61±0.14d | 161.83±58.94d |
| 高山嵩草 | 2.27±0.76e | 0.51±0.13d | 82.00±29.86d | |
| 火绒草 | 3.28±1.02de | 0.62±0.16d | 199.50±61.66d | |
| Kruskal-Wallis和Dunn′s Kruskal-Wallis多重比较检验三种植物不同部位组间差异, 同一列不同小写字母标记的数值表示差异显著(P<0.05), 表中数据为平均值±标准差(n=6) | ||||
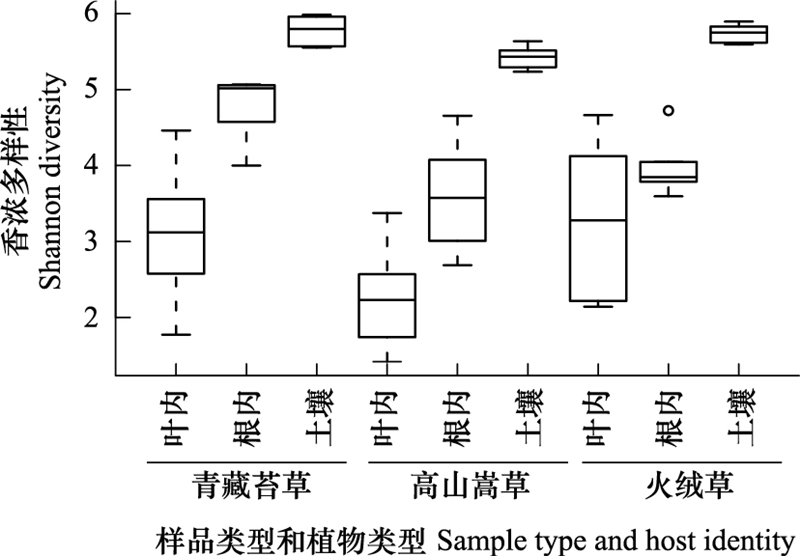
|
| 图 3 三种植物叶内、根内以及非根际土壤的香浓多样性 Fig. 3 Shannon diversity of leaf endosphere, root endosphere and soil for three plants |
基于Bray-Curtis距离的非度量多维尺度分析(NMDS)用来展示微生物群落间的差异(图 4)。在NMDS图中, 首先可以看到所有样品按照不同部位在NMDS1维度分开, 说明植物部位对植物微生物群落结构的分化有影响;其次, 在NMDS2维度, 相同部位按照植物类型聚集形成不同的集团, 表明不同植物类型对微生物群落结构差异也有一定的影响。另外, 根内微生物群落处于叶内和土壤样品之间, 而叶内和土壤微生物群落相隔较远, 说明了微生物在植物根内的定殖受土壤微生物群落影响更大, 而植物对叶内微生物群落的选择受土壤微生物群落影响较小。

|
| 图 4 三种植物根内、叶内以及土壤原核微生物群落基于Bray-Curtis距离的非度量多维尺度分析(NMDS) Fig. 4 Nonmetric multidimensional scaling analysis (NMDS) based on Bray-Curtis distances of prokaryotic communities associated with three plants |
基于Bray-Curtis距离置换多元方差分析(PERMANOVA)检验微生物群落结构组成在不同因子间的差异(表 3)。结果表明, 样品类型(叶内、根内、土壤)、植物类型以及二者的交互作用都对微生物群落差异有显著性影响, 总共解释了微生物群落51.94%的变异。其中样品类型对微生物群落间差异贡献度最大(R2=20.13%, P<0.001), 其次是植物类型和样品类型的交互作用(R2=17.40%, P<0.05), 而植物类型对微生物间变异的贡献度为14.41%(P<0.001)。
| 影响因素Factors | SS | F | R2 | P |
| 样品类型Sample Type | 4.244 | 9.214 | 20.13% | 0.001 |
| 植物类型Host identity | 3.039 | 6.598 | 14.41% | 0.001 |
| 植物类型:植物部位Sample Type: Host identity | 3.669 | 3.982 | 17.40% | 0.001 |
部分微生物可以从土壤和根际转移到植物内部, 但不同部位如叶内和根内也存在特定的与土壤有显著性差异的微生物类群。在OTU水平上, 我们将不同植物不同部位分别进行差异比较, 将与土壤有显著性差异并且相对丰度高于土壤的内生OTU定义为植物富集OTUs(enriched OTUs)。青藏苔草、火绒草和高山嵩草叶内分别发现了16个、27个和3个富集OTUs, 相对丰度之和为39.41%、15.01%、42.10%, 这些OTUs主要来自变形菌门和厚壁菌门, 且在土壤中相对丰度很低, 大部分内生富集OTUs在土壤中相对丰度小于0.01%, 在青藏苔草、火绒草和高山嵩草周边土壤中对应的相对丰度之和分别为为0.72%、0.51%、0.03%, 与在叶内的高丰度形成显著差异。部分叶内富集OTUs能分类到属水平上, 包括假单胞菌属(Pseudomonas)、不动杆菌属(Acinetobacter)、微小杆菌属(Exiguobacterium)、农杆菌属(Agrobacterium)、鞘氨醇单胞菌(Sphingomonas)等。根内富集OTUs在青藏苔草、火绒草和高山嵩草中分别为16个、48个和11个, 这些OTUs主要来自变形菌和放线菌门, 相对丰度之和为17.90%、33.97%、9.62%, 对应在土壤中的相对丰度为3.72%, 0.79%, 0.03%。类似的, 部分根内富集OTUs能分类到属水平上, 包括假单胞菌属、不动杆菌属以及德沃斯氏菌属(Devosia)。为了进一步确认哪些属在叶内或根内属于优势属, 我们从属水平上进行差异分析发现, 假单胞菌属, 不动杆菌属, 鞘氨醇单胞菌属以及德沃斯氏菌在叶内或根内属于优势属且与土壤有显著差异, 四种属在三种植物不同样品类型的相对丰度及差异分析如图 5所示, 假单胞菌在青藏苔草叶内、高山嵩草根内以及火绒草的叶内和根内均显著富集, 在火绒草中相对丰度高达30%, 且与土壤有显著性差异;不动杆菌属在三种植物叶内和根内丰度显著高于土壤;鞘氨醇单胞菌在三种植物叶内相对丰度高于根内和土壤;德沃斯氏菌在三种植物根内相对丰度较高, 特别是在高山嵩草中显著地富集。这些差异体现了不同部位和不同植物对不同属的选择性。
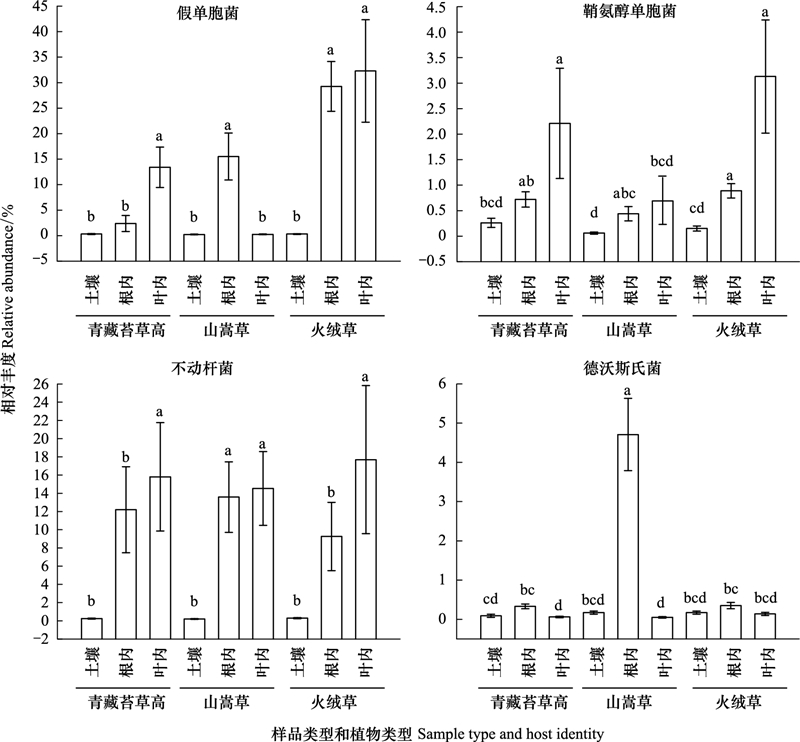
|
| 图 5 四种优势属在三种植物根内、叶内以及非根际土壤的相对丰度 Fig. 5 The relative abundance of four dominant genera in different parts of three plants |
在这些富集OTU当中有一部分OTU出现在80%以上的样品中(总共18个样品, 出现频率大于或等于15), 我们将这部分OTU定义为核心OTU(表 4)。在叶内共有6个核心OTU, 总的相对丰度为25.62%, 其中, 一个属于Bacillales, 三个OTU属于Pseudomonadales, 另外两个OTU分别属于Enterobacteriales和Rhizobiales;根内发现4个核心OTU, 总的相对丰度为14.13%, 其中, 两个属于Pseudomonadales, 另外两个分别属于Rhizobiales和Actinomycetales。利用KEGG数据库对相对丰度高于1%的富集OTUs进行功能预测, 我们在level 2水平上共发现了37个基因功能, 包括多种代谢过程如氨基酸代谢、碳水化合物代谢、次生代谢产物生物合成等, 环境信息处理过程如信号分子相互作用、信号转导以及膜转运;细胞过程如细胞运动、细胞生长和死亡以及细胞通讯, 这些潜在的生物功能, 为植物-微生物的相互作用奠定了基础。
| OTU ID | 相对丰度 Relative abundance/% |
样品类型 Sample type |
门 Phylum |
纲 Class |
目 Order |
科 Family |
属 Genus |
| OTU_324 | 10.693 | 叶内 | Firmicutes | Bacilli | Bacillales | Bacillaceae | |
| OTU_130 | 8.367 | 叶内 | Proteobacteria | Gammaproteobacteria | Pseudomonadales | Moraxellaceae | Acinetobacter |
| OTU_913 | 2.517 | 叶内 | Proteobacteria | Gammaproteobacteria | Pseudomonadales | Moraxellaceae | Acinetobacter |
| OTU_812 | 1.700 | 叶内 | Proteobacteria | Gammaproteobacteria | Enterobacteriales | Enterobacteriaceae | |
| OTU_196 | 1.308 | 叶内 | Proteobacteria | Alphaproteobacteria | Rhizobiales | Rhizobiaceae | Agrobacterium |
| OTU_682 | 1.029 | 叶内 | Proteobacteria | Gammaproteobacteria | Pseudomonadales | Moraxellaceae | Acinetobacter |
| OTU_106 | 6.171 | 根内 | Proteobacteria | Gammaproteobacteria | Pseudomonadales | Pseudomonadaceae | Pseudomonas |
| OTU_112 | 4.348 | 根内 | Proteobacteria | Gammaproteobacteria | Pseudomonadales | Pseudomonadaceae | Pseudomonas |
| OTU_2292 | 3.259 | 根内 | Proteobacteria | Alphaproteobacteria | Rhizobiales | Bradyrhizobiaceae | |
| OTU_810 | 0.356 | 根内 | Actinobacteria | Actinobacteria | Actinomycetales |
通过对高寒草甸三种优势植物青藏苔草、火绒草、高山嵩草叶内生、根内生以及土壤原核微生物组的研究, 发现样品类型是影响植物原核微生物组差异的最主要因子, 体现了叶内, 根内, 非根际土壤三者在空间上生态位的差异, 而植物类型对不同部位微生物的选择性相对较弱。从门水平上看, 植物内生原核微生物组都是以变形菌门、放线菌门、酸杆菌门、拟杆菌门和厚壁菌门为优势菌群[16, 28], 但不同部位主要门的相对丰度有显著性差异。变形菌门的相对丰度从土壤到根内和叶内不断升高, 在根内和叶内平均相对丰度占比都超过了50%;而放线菌门在土壤中相对丰度最高, 在根内和叶内相对丰度降低。在对杨树不同部位(土壤, 根内, 茎, 叶内)的研究中也发现相似的结论[29];水稻的根内微生物与土壤有显著性的差异, 并且变形菌门的变化趋势和本研究一致[30]。在OTU组成上, 根内与土壤共有28%的共有OTUs, 而叶内与土壤共有OTUs为16.2%, 说明只有部分土壤微生物能够进入植物的根内和叶内, 植物内生部位对微生物的选择作用更强[31]。相比于样品类型, 植物类型对微生物的选择性较弱。本研究发现在门分水平上绝大部分原核微生物在叶内, 根内, 土壤之间有显著性差异, 而在不同类型植物之间没有显著性差异。类似的, 在α多样性上叶内, 根内, 土壤间都表现出显著性差异, 而三种植物叶内α多样性之间无显著差异。并且无论是多样性、均匀度还是观察到的物种数, 土壤微生物α多样性远高于根内和叶内, 其中, 叶内的α多样性最低。土壤养分相对含量较低, 同时受到植物分泌物、枯枝落叶的影响较大, 原核微生物多样性高;而植物的地上部分特别是植物的叶, 长期暴露在环境严苛的空气当中[32], 高温暴晒、紫外线的强烈照射、叶内的特殊化学环境等因素使得只有部分微生物能定殖在叶表和叶内, α多样性较低。另外, 我们发现土壤微生物群落的α多样性变异程度最小, 说明土壤样本之间的差异较小;而叶内和根内变化较大, 特别是叶内微生物群落变异最大。在对拟南芥及其近缘种的研究中发现根内微生物群落在去掉小于20条序列的OTU后, 样品之间的微生物群落趋于稳定[11], 说明稀有物种是造成样品之间植物内生菌差异的主要原因。在群落结构上, 本研究发现不同样品类型和不同植物间微生物群落结构都表现出显著性差异, 样品类型对微生物群落结构差异的贡献度为20.13%, 而植物类型对微生物群落结构差异的贡献度为14.41%, 植物部位和非根际土壤对微生物的强烈选择作用在其他植物中也得到证明。研究发现同属三种龙舌兰叶内和根内原核微生物群落结构存在显著差异, 其中植物部位对原核微生物群落结构差异贡献度高达47.9%, 而植物类型的贡献度为10.8%[16]。另一个研究发现相对于植物基因型, 杨树不同样品类型包括根内、叶内、茎以及非根际土壤之间的差异更大[29]。而本研究以差异更大的三种植物(不同纲不同属)作为研究对象, 进一步证明了植物部位以及非根际土壤的对微生物的强烈选择性。表明植物叶内, 根内, 非根际土壤存在独特的生态位, 是微生物群落构建主要的驱动因子。叶内、根内以及非根际土壤对植物微生物的选择归结到底是由于微生物在不同部位生活历史的差异, 植物可以通过一系列手段来控制和选择微生物, 如, 根际分泌物[15]、养分有效性[32]、地上部分与地下部分环境差异[14]等都可成为不同器官对生活在其中的微生物的选择;而对于非根际土壤, 首先受到植物根际分泌物的影响, 其次也会受到气候和理化性质等因素较大的影响[9], 这些差异造成了叶内、根内以及土壤微生物选择的差异。尽管植物类型对微生物的选择较弱, 但植物物种的选择作用在什么条件下比较强, 对哪些微生物类群起决定性的选择作用, 我们了解的还有限, 尤其是对自然生长的高寒草甸植物。人们对丛枝菌根与植物之间的互利共生关系[33]研究较多, 但大部分其他微生物在植物中的作用和功能还有待探索。综上所述, 植物不同部位和非根际微生物既有统一性又有特异性, 并且样品类型对微生物的选择作用大于植物类型的选择作用。
3.2 青藏高原优势植物叶内和根内高丰度OTU的的富集及其潜在功能对植物的营养吸收及生态适应性有重要意义本研究在对植物内生菌和土壤原核微生物对比分析中发现, 不同植物叶内和根内存在与土壤原核微生物有显著差异的菌群(OTU)。对比拟南芥根内和对应土壤微生物组成发现70个OTU在拟南芥根部显著富集, 与土壤存在显著性差异, 并且这些微生物类群对植物部位和植物类型之间微生物群落的差异有显著的贡献[11]。这些OTU尽管数量不多, 但相对丰度较高, 在群落中属于优势物种。本研究还发现, 无论是在叶内还是在根内, 假单胞菌Pseudomonas都显著富集, 在根内总相对丰度为10.93%, 在叶内总相对丰度为13.43%, 并且在根内作为核心微生物出现在绝大部分样品中(表 4)。以往的研究发现, 在不同寒冷地带的不同植物内生菌中都分离出了假单胞菌及其相关的类群。Sheng et al(2001)报道, 从天山山脉超过20种高寒植物中分离出46%的内生菌与从其他寒冷地区分离出的内生菌相似, 其中假单胞菌、鞘氨醇单胞菌Sphingomonas、红球菌属Rhodococcus分布最广泛[34], 这些类群同样存在于本研究的样品中。在高寒林区采集的番红花Crocus albifloru中分离出数种与假单胞菌相关的高丰度内生菌, 如Pseudomonas s E3 AY745742, Pseudomonas s K94.23 AY456705, Pseudomonas trivialis AJ492831[35]。另一个研究发现, 从三种极地植物Oxyria digyna、Diapensia lapponica、Juncus trifidus中分离纯培养的内生菌株绝大部分在低温实验控制下(4℃)生长良好并且具有活性, 包括丰度较高的假单胞菌假、根瘤菌Rhizobium、慢生根瘤菌Bradyrhizobium、伯克氏菌属Burkholderia、红球菌属, 另外还发现有58%的菌株能够溶解磷矿物, 降解几丁质、纤维素或蛋白, 包括伯克霍尔德菌、叶居菌Frondihabitans、假单胞菌、鞘氨醇单胞菌和贪噬菌属Variovorax[17]。以上大部分微生物类群如假单胞菌, 根瘤菌, 慢生根瘤菌, 鞘氨醇单胞菌, 红球菌属在本研究的植物内生菌(根内和叶内)中同样聚集。同时我们也在叶内和根内, 特别是根内发现了多种具有潜在固氮功能的菌群, 如根瘤菌[36]、叶杆菌Phyllobacterium[37]、氨基杆菌属Aminobacter、德沃斯氏菌Devosia[38]、慢生根瘤菌[39], 这些菌群可能为非豆科植物提供氮营养。这些微生物的耐冷机制有待进一步探索, 有研究发现假单胞菌Pseudomonas sp. ID1通过产生胞外多糖作为低温防护剂, 使其在-20℃到-80℃之间仍具有活性[40], 另外有些微生物能够分泌冷休克蛋白维持细胞膜的流动性[41], 这些耐冷机制有助于寒冷条件下内生菌的生存。与此研究一致, 在对本研究中植物内生高丰度富集OTUs进行功能预测中, 我们发现了包括氨基酸代谢, 碳水化合物代谢, 膜转运, 信号转导等多种功能基因的存在。微生物可能通过这些潜在的功能参与植物的众多生命过程, 帮助植物在逆境中生长。以上结果表明, 植物内生菌中存在的特定菌群, 可能在植物营养获取方面起着重要的作用。另外, 这些菌群具有多种潜在的功能, 可能参与协同植物在高寒环境中生存, 并且这些内生菌跨越了不同植物和不同地域广泛存在, 可能与植物共同进化, 对植物抗逆性、生态适应性都具有重要的意义。
4 结论对青藏高原高寒草甸三种优势植物的研究发现, 不同植物部位存在着显著差异的原核微生物类群, 并且植物部位对微生物的选择性强于植物类型。高寒草甸植物内部存在着与土壤有显著差异的内生菌, 这些内生菌可能对协助植物在条件严苛的寒冷环境中正常生长有重要作用。但这些微生物与其宿主植物的互作机制还有待进一步探索, 可以通过实验室实验, 如无菌苗定殖实验, 探索这些微生物的具体作用。
| [1] |
Turner T R, James E K, Poole P S. The plant microbiome. Genome Biology, 2013, 14(6): 209. DOI:10.1186/gb-2013-14-6-209 |
| [2] |
Margesin R, Neuner G, Storey K B. Cold-loving microbes, plants, and animals-fundamental and applied aspects. Naturwissenschaften, 2007, 94(2): 77-99. DOI:10.1007/s00114-006-0162-6 |
| [3] |
Khan Z, Doty S L. Characterization of bacterial endophytes of sweet potato plants. Plant and Soil, 2009, 322(1/2): 197-207. |
| [4] |
Lanfranco L, Young J P W. Genetic and genomic glimpses of the elusive arbuscular mycorrhizal fungi. Current Opinion in Plant Biology, 2012, 15(4): 454-461. DOI:10.1016/j.pbi.2012.04.003 |
| [5] |
Smith S E, Smith F A, Jakobsen I. Mycorrhizal fungi can dominate phosphate supply to plants irrespective of growth responses. Plant Physiology, 2003, 133(1): 16-20. |
| [6] |
Monteiro R A, Balsanelli E, Wassem R, Marin A M, Brusamarello-Santos L C C, Schmidt M A, Tadra-Sfeir M Z, Pankievicz V C S, Cruz L M, Chubatsu L S, Pedrosa F O, Souza E M. Herbaspirillum-plant interactions:microscopical, histological and molecular aspects. Plant and Soil, 2012, 356(1/2): 175-196. |
| [7] |
Laforest-Lapointe I, Messier C, Kembel S W. Host species identity, site and time drive temperate tree phyllosphere bacterial community structure. Microbiome, 2016, 4: 27. DOI:10.1186/s40168-016-0174-1 |
| [8] |
Edwards J, Johnson C, Santos-Medellín C, Lurie E, Podishetty N K, Bhatnagar S, Eisen J A, Sundaresan V, Jeffery L D. Structure, variation, and assembly of the root-associated microbiomes of rice. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(8): E911-E920. DOI:10.1073/pnas.1414592112 |
| [9] |
Bonito G, Reynolds H, Robeson Ⅱ M S, Nelson J, Hodkinson B P, Tuskan G, Schadt C W, Vilgalys R. Plant host and soil origin influence fungal and bacterial assemblages in the roots of woody plants. Molecular Ecology, 2014, 23(13): 3356-3370. DOI:10.1111/mec.12821 |
| [10] |
Naylor D, DeGraaf S, Purdom E, Coleman-Derr D. Drought and host selection influence bacterial community dynamics in the grass root microbiome. The ISME Journal, 2017, 11(12): 2691-2704. DOI:10.1038/ismej.2017.118 |
| [11] |
Schlaeppi K, Dombrowski N, Oter R G, Van Themaat E V L, Schulze-Lefert P. Quantitative divergence of the bacterial root microbiota in Arabidopsis thaliana relatives. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(2): 585-592. DOI:10.1073/pnas.1321597111 |
| [12] |
Fonseca-Garcia C, Coleman-Derr D, Garrido E, Visel A, Tringe S G, Partida-Martínez L P. The cacti microbiome:interplay between habitat-filtering and host-specificity. Frontiers in Microbiology, 2016, 7: 150. |
| [13] |
Gottel N R, Castro H F, Kerley M, Yang Z M, Pelletier D A, Podar M, Karpinets T, Uberbacher E, Tuskan G A, Vilgalys R, Doktycz M J, Schadt C W. Distinct microbial communities within the endosphere and rhizosphere of Populus deltoides roots across contrasting soil types. Applied and Environmental Microbiology, 2011, 77(17): 5934-5944. DOI:10.1128/AEM.05255-11 |
| [14] |
Bai Y, Müller D B, Srinivas G, Garrido-Oter R, Potthoff E, Rott M, Dombrowski N, Münch P C, Spaepen S, Remus-Emsermann M, Hüttel B, McHardy A C, Vorholt J A, Schulze-Lefert P. Functional overlap of the Arabidopsis leaf and root microbiota. Nature, 2015, 528(7582): 364-369. DOI:10.1038/nature16192 |
| [15] |
Berendsen R L, Pieterse C M J, Bakker P A H M. The rhizosphere microbiome and plant health. Trends in Plant Science, 2012, 17(8): 478-486. DOI:10.1016/j.tplants.2012.04.001 |
| [16] |
Coleman-Derr D, Desgarennes D, Fonseca-Garcia C, Gross S, Clingenpeel S, Woyke T, North G, Visel A, Partida-Martinez L P, Tringe S G. Plant compartment and biogeography affect microbiome composition in cultivated and native Agave species. New Phytologist, 2016, 209(2): 798-811. DOI:10.1111/nph.13697 |
| [17] |
Nissinen R M, Männistö M K, Van Elsas J D. Endophytic bacterial communities in three arctic plants from low arctic fell tundra are cold-adapted and host-plant specific. Fems Microbiology Ecology, 2012, 82(2): 510-522. DOI:10.1111/j.1574-6941.2012.01464.x |
| [18] |
Guo Y J, Guo N, He Y J, Gao J H. Cuticular waxes in alpine meadow plants:climate effect inferred from latitude gradient in Qinghai-Tibetan Plateau. Ecology and Evolution, 2015, 5(18): 3954-3968. DOI:10.1002/ece3.1677 |
| [19] |
Wang C T, Cao G M, Wang Q L, Jing Z C, Ding L M, Long R J. Changes in plant biomass and species composition of alpine Kobresia meadows along altitudinal gradient on the Qinghai-Tibetan Plateau. Science in China Series C:Life Sciences, 2008, 51(1): 86-94. DOI:10.1007/s11427-008-0011-2 |
| [20] |
Tamaki H, Wright C L, Li X Z, Lin Q Y, Hwang C, Wang S P, Thimmapuram J, Kamagata Y, Liu W T. Analysis of 16S rRNA amplicon sequencing options on the Roche/454 next-generation titanium sequencing platform. PLoS One, 2011, 6(9): e25263. DOI:10.1371/journal.pone.0025263 |
| [21] |
Magoč T, Salzberg S L. FLASH:fast length adjustment of short reads to improve genome assemblies. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507 |
| [22] |
Caporaso J G, Kuczynski J, Stombaugh J, Bittinger K, Bushman F D, Costello E K, Fierer N, Peña A G, Goodrich J K, Gordon J I, Huttley G A, Kelley S T, Knights D, Koenig J E, Ley R E, Lozupone C A, McDonald D, Muegge B D, Pirrung M, Reeder J, Sevinsky J R, Turnbaugh P J, Walters W A, Widmann J, Yatsunenko T, Zaneveld J, Knight R. QⅡME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [23] |
Edgar R C, Notes A. Search and clustering orders of magnitude faster than BLAST. Bioinformatics, 2010, 26(19): 2460-2461. DOI:10.1093/bioinformatics/btq461 |
| [24] |
Wang Q, Garrity G M, Tiedje J M, Cole J R. Naïve Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy. Applied and Environmental Microbiology, 2007, 73(16): 5261-5267. DOI:10.1128/AEM.00062-07 |
| [25] |
Benjamini Y, Yekutieli D. The control of the false discovery rate in multiple testing under dependency. The Annals of Statistics, 2001, 29(4): 1165-1188. |
| [26] |
Warton D I, Wright S T, Wang Y. Distance-based multivariate analyses confound location and dispersion effects. Methods in Ecology and Evolution, 2012, 3(1): 89-101. DOI:10.1111/j.2041-210X.2011.00127.x |
| [27] |
Parks D H, Tyson G W, Hugenholtz P, Beiko R G. STAMP:statistical analysis of taxonomic and functional profiles. Bioinformatics, 2014, 30(21): 3123-3124. DOI:10.1093/bioinformatics/btu494 |
| [28] |
Beckers B, De Beeck M O, Weyens N, Boerjan W, Vangronsveld J. Structural variability and niche differentiation in the rhizosphere and endosphere bacterial microbiome of field-grown poplar trees. Microbiome, 2017, 5: 25. DOI:10.1186/s40168-017-0241-2 |
| [29] |
Cregger M A, Veach A M, Yang Z K, Crouch M J, Vilgalys R, Tuskan G A, Schadt C W. The Populus holobiont:dissecting the effects of plant niches and genotype on the microbiome. Microbiome, 2018, 6: 31. DOI:10.1186/s40168-018-0413-8 |
| [30] |
Bulgarelli D, Schlaeppi K, Spaepen S, Van Themaat E V L, Schulze-Lefert P. Structure and functions of the bacterial microbiota of plants. Annual Review of Plant Biology, 2013, 64: 807-838. DOI:10.1146/annurev-arplant-050312-120106 |
| [31] |
Redford A J, Bowers R M, Knight R, Linhart Y, Fierer N. The ecology of the phyllosphere:geographic and phylogenetic variability in the distribution of bacteria on tree leaves. Environmental Microbiology, 2010, 12(11): 2885-2893. DOI:10.1111/j.1462-2920.2010.02258.x |
| [32] |
Hacquard S, Garrido-Oter R, González A, Spaepen S, Ackermann G, Lebeis S, McHardy A C, Dangl J L, Knight R, Ley R, Schulze-Lefert P. Microbiota and host nutrition across plant and animal kingdoms. Cell Host & Microbe, 2015, 17(5): 603-616. |
| [33] |
Sikes B A. When do arbuscular mycorrhizal fungi protect plant roots from pathogens?. Plant Signaling & Behavior, 2010, 5(6): 763-765. |
| [34] |
Sheng H M, Gao H S, Xue L G, Ding S, Song C L, Feng H Y, An L Z. Analysis of the composition and characteristics of culturable endophytic bacteria within subnival plants of the Tianshan Mountains, Northwestern China. Current Microbiology, 2011, 62(3): 923-932. |
| [35] |
Reiter B, Sessitsch A. Bacterial endophytes of the wildflower Crocus albiflorus analyzed by characterization of isolates and by a cultivation-independent approach. Canadian Journal of Microbiology, 2006, 52(2): 140-149. |
| [36] |
Masson-Boivin C, Sachs J L. Symbiotic nitrogen fixation by rhizobia-the roots of a success story. Current Opinion in Plant Biology, 2018, 44: 7-15. DOI:10.1016/j.pbi.2017.12.001 |
| [37] |
Rojas A, Holguin G, Glick B R, Bashan Y. Synergism between Phyllobacterium sp (N2-fixer) and Bacillus licheniformis (P-solubilizer), both from a semiarid mangrove rhizosphere. Fems Microbiology Ecology, 2001, 35(2): 181-187. DOI:10.1111/j.1574-6941.2001.tb00802.x |
| [38] |
Rivas R, Velázquez E, Willems A, Vizcaíno N, Subba-Rao N S, Mateos P F, Gillis M, Dazzo F B, Martínez-Molina E. A new species of Devosia that forms a unique nitrogen-fixing root-nodule symbiosis with the aquatic legume Neptunia natans (L.f.) Druce. Applied and Environmental Microbiology, 2002, 68(11): 5217-5222. DOI:10.1128/AEM.68.11.5217-5222.2002 |
| [39] |
Hara S, Morikawa T, Wasai S, Kasahara Y, Koshiba T, Yamazaki K, Fujiwara T, Tokunaga T, Minamisawa K. Identification of nitrogen-fixing Bradyrhizobium associated with roots of field-grown sorghum by metagenome and proteome analyses. Frontiers in Microbiology, 2019, 10: 407. DOI:10.3389/fmicb.2019.00407 |
| [40] |
Carrión O, Delgado L, Mercade E. New emulsifying and cryoprotective exopolysaccharide from Antarctic Pseudomonas sp. ID1. Carbohydrate Polymers, 2015, 117: 1028-1034. DOI:10.1016/j.carbpol.2014.08.060 |
| [41] |
Barria C, Malecki M, Arraiano C M. Bacterial adaptation to cold. Microbiology, 2013, 159(12): 2437-2443. |
 2020, Vol. 40
2020, Vol. 40




