文章信息
- 黄峰, 王玮韧, 饶鑫, 郝珖存, 何聃, 王俊, 简曙光, 申卫军, 任海
- HUANG Feng, WANG Weireng, RAO Xin, HAO Guangcun, HE Dan, WANG Jun, JIAN Shuguang, SHEN Weijun, REN Hai
- 热带珊瑚岛植物种植对土壤改良及其微生物群落形成的影响
- Soil improvements and microbial community development following establishment of plant communities in a tropical coral island
- 生态学报. 2019, 39(17): 6227-6237
- Acta Ecologica Sinica. 2019, 39(17): 6227-6237
- http://dx.doi.org/10.5846/stxb201804240928
-
文章历史
- 收稿日期: 2018-04-24
- 网络出版日期: 2019-05-31
南沙群岛及其周边海域是我国重要的领土, 具有丰富的自然资源和重要的战略地位[1-2]。热带珊瑚岛是这些领土的重要组成部分, 在其上构建植被生态系统, 建设珊瑚岛宜居的生态环境是维持这些珊瑚岛持续发展的基础。植被生态系统的恢复是一项系统工程, 不仅涉及到恢复系统的各个组分, 如生产者、消费者和分解者, 以及它们赖以生存的非生物环境如气候和土壤, 也涉及到恢复系统的发展、演化阶段。
在恢复初期或先锋群落阶段, 通常小气候和土壤条件比较严酷。这个阶段一般采用一些适生、耐瘠的植物种类, 辅以相应的土壤改良和栽培管理措施以达到改善生境条件的目的。新建的热带珊瑚岛所在海域水热条件比较丰富, 但是土壤非常贫瘠, 主要的土壤介质为珊瑚和贝壳组成的珊瑚砂, 有机质和营养元素(如氮)含量极低[3]。因此, 针对新建珊瑚岛特殊的生境条件, 先锋群落建植阶段所采取的主要措施是选择适生植物种, 以及生境营造如土壤改良[1]。通过对珊瑚岛附近海域的岛屿进行调查, 前人的研究发现我国南海海岛的植物物种十分贫乏, 自然植物群落的结构也相对简单[1, 4-5]。在植物选择的过程中, 主要考虑那些对珊瑚岛环境适应性较强, 具有耐旱、耐盐、耐瘠薄、抗风等特性, 比如草海桐(Terminalia catappa)、木麻黄(Casuarina equisetifolia)、榄仁(Terminalia catappa)、海刀豆(Canavalia maritima)、厚藤(Ipomoea pes-caprae)等。其次, 为创造珊瑚岛更好的人居环境, 也要考虑一些具有一定食用和观赏价值的植物, 如花卉、蔬菜和水果[1]。目前所采取的土壤改良措施主要是在栽植时施用有机肥[1]。
相比于在退化森林土壤、沙漠土壤等开展植被新建和生态恢复来说, 热带珊瑚岛的生态恢复具有其更大的难度。热带珊瑚岛虽然年降水量不低(约2000 mm), 但降水季节分配非常不均, 干季长达6个月;新建的珊瑚岛地下水短期内还没有形成或水位较低, 土壤保水能力差, 为植物干季生长带来了严酷的考验[1-2]。然而, 通过一定的人工辅助土壤改良措施, 选择适生的植物种类, 长途运输种苗栽植, 以及施用一定的水肥管理措施, 目前在南沙新形成的几个珊瑚岛上已构建了多种类型的先锋植物群落, 包括防风林、固沙绿地、公园绿地、行道树、四旁绿化等。这些先锋群落是在频繁的人为干预下新建形成的, 后期阶段更多需要通过其自身的物质和能量循环来维系和发展。因此, 对这些先锋群落的结构和功能进行观测、分析, 对将来进行群落结构和功能的优化改良具有重要实践意义。同时, 新形成的珊瑚岛类似一张“白纸”, 在其上进行植被新建与恢复也对发展和检验岛屿生态学、恢复生态学的相关理论(如阈值理论)具有重要学术价值。
植物群落的恢复可预见性和其恢复效果的直观性。但是, 植物栽植后珊瑚岛上土壤微生物群落如何恢复和发展, 以及最终如何形成一个稳定的群落结构较难量化和评价。在一个健康的生态系统中, 土壤微生物对于整个生态系统的物质和能量循环起着重要的作用, 是判断一个生态系统健康与否的重要环节[6-8]。比如, 生态系统中物质与能量循环重要组成部分的土壤氮循环, 其主要包括的固氮作用、硝化作用、反硝化作用和氨化作用, 均是由微生物驱动实现的[9-12]。另外, 土壤微生物对于植物的生长也有非常切实的作用, 比如促进植物对氮、磷、铁元素的吸收[13-14], 刺激植物生长素合成并加快植物生长[15], 以及诱导植物的非系统性抗性并提高植物对病虫害的抵御能力[16]。因此, 在研究生态系统的重建和恢复过程中, 关注以真菌和细菌为代表的土壤微生物群落的重建和恢复同样具有重要的科学和实践意义。
本研究利用一个热带珊瑚岛上开展的植被新建试验, 通过对植物种植后的土壤进行取样, 研究随植物栽植带来的土壤微生物群落如何形成、适应和变化, 目的是为珊瑚岛的长期生态建设和保护提供本底的调查数据, 并为生态恢复实践和理论、土壤改良措施、海岛植被生态系统养分循环等提供关于土壤微生物方面的研究基础。
1 材料和方法 1.1 土壤样品收集热带珊瑚岛的成土基质主要为珊瑚和贝壳砂。珊瑚砂养分少、含水量低、高盐、强碱, 并不适宜微生物的生长, 因此在珊瑚岛植被的新建和恢复过程中, 外来微生物可能是其土壤微生物的主要来源。为具体研究这一过程, 本实验中采集的土壤样品包括移栽植物幼苗基质中的根际土和非根际土、珊瑚岛上人工配制的种植土, 以及植物栽植一段时间后的根围土;此后分别称为苗根际土、苗非根际土、种植土和珊瑚砂土, 共计有土样81个。移栽植物幼苗的根际土和非根际土样品采集自海南省文昌市凌叶园林绿化工程有限公司的苗圃, 采集时间为2017年8月(表 1)。每种植物选择3—5株幼苗, 连根拔出, 将其根系上附着的土壤抖落下来定为根际土, 离根系较远的土壤定为非根际土[17]。对来自同一种植物的根际土和非根际土分别进行混合, 最终分别获得22个分析样品(表 1)。
| 样品编号 Sample numbers |
植物种类 Plant species |
土样类型 Soil types |
| B1—B33号土样 | 为2017年6月6—12日采自珊瑚岛 | |
| B1, B2, B5, B6, B9, B10 | 草海桐 | 非根际土 |
| B3, B4, B11, B12 | 木麻黄 | 非根际土 |
| B19, B20 | 龙爪茅 | 非根际土 |
| B13, B14 | 海杧果、龙爪茅、四生臂形草、醴肠、狗牙根 | 非根际土 |
| B28—B33 | 椰子、龙爪茅, 椰子、酸豆、番木瓜等, 朱槿、夹竹桃、龙船花、海南龙血树、灰莉、榄仁树、椰子等 | 非根际土 |
| B15—B18, B21—B27 | 龙爪茅、牛筋草、醴肠、四生臂形草、狗牙根 | 根际土 |
| B34—B39号土样 | 为2017年6月15日采自珊瑚岛 | |
| B34—B39 | 未种植植物 | 种植土 |
| B40—B83号土样 | 为2017年8月14日采自海南文昌凌叶苗圃 | |
| B40—B83 | 草海桐、翠芦莉、朱槿、大叶相思、过江藤、海岸桐、海滨木巴戟、海刀豆、海杧果、红厚壳、花叶鸭脚木、黄金香柳、黄槿、灰莉、抗风桐、榄仁树、莲叶桐、木麻黄、水黄皮、杨叶肖槿、椰子、银叶树 | 双数编号为根际土, 单数编号为非根际土 |
| 注:植物种及对应拉丁学名:草海桐(Terminalia catappa), 木麻黄(Casuarina equisetifolia), 海杧果(Cerbera manghas), 龙爪茅(Dactyloctenium aegyptium), 四生臂形草(Brachiaria Subquadripara), 醴肠(Eclipta prostrata), 狗牙根(Cynodon dactylon), 牛筋草(Eleusine indica), 椰子(Cocos nucifera), 酸豆(Tamarindus indica), 番木瓜(Chaenomeles sinensis), 朱槿(Hibiscus rosa-sinensis), 夹竹桃(Nerium indicum), 龙船花(Ixora chinensis), 海南龙血树(Dracaena angustifolia), 灰莉(Fagraea ceilanica), 榄仁树(Terminalia catappa), 翠芦莉(Ruellia brittoniana), 大叶相思(Acacia auriculiformis), 过江藤(Phyla nodiflora), 海岸桐(Guettarda speciosa), 海滨木巴戟(Morinda citrifolia), 海刀豆(Canavalia maritima), 红厚壳(Calophyllum inophyllum), 花叶鸭脚木(Schefflera odorata), 黄金香柳(Melaleuca bracteata), 黄槿(Hibiscus tiliaceus), 抗风桐(Pisonia grandis), 莲叶桐(Hernandia nymphaeifolia), 水黄皮(Pongamia pinnata), 杨叶肖槿(Thespesia populnea), 银叶树(Heritiera littoralis) | ||
珊瑚岛采集的土壤包括两大类, 一类为种植土, 有6个样品, 为植物幼苗种植前珊瑚岛原有珊瑚砂、客土和营养土的混合土壤;另一类为植物幼苗种植6—12个月后, 植物群落的根围土(也即珊瑚砂土), 总共31个样品(表 1)。按照珊瑚岛植被新建过程中的主要植物群落类型, 我们选取植物群落11个, 在每个植物群落上划出3个10 m × 10 m的样方, 并在样方内用直径7 cm的土钻随机采集0—10 cm土样5个。采自同一个样方的土壤混合成1个混合样用于分析。所有土样采集后, 尽快运回实验室, 过2 mm土筛后保存于-80 ℃条件下备用。
1.2 土壤微生物总DNA的提取、PCR扩增和建库从每份采集的样品中, 称取土壤0.25 g, 并使用MOBIO PowerSoil® DNA Isolation Kit提取样品中的微生物基因组总DNA。利用琼脂糖凝胶电泳检测DNA的纯度和浓度。然后, 取适量的样品于离心管中, 使用无菌水稀释样品至1 ng/μL。以稀释后的基因组DNA为模板, 扩增细菌DNA的16S V4区(引物515F和806R)和真菌DNA的ITS1区(引物ITS5—1737F和ITS2—2043R)序列, 序列扩增的体系和方法按照文献进行[18-19]。获得的PCR产物使用2%浓度的琼脂糖凝胶进行电泳检测, 合格样品按照Qiagen公司提供的胶回收试剂盒回收产物。使用TruSeq® DNA PCR-Free Sample Preparation Kit建库试剂盒进行文库构建, 构建好的文库经过Qubit(Life Technologies, Carlsbad, CA, USA)和qPCR定量, 文库合格后, 使用HiSeq2500 PE250进行上机测序(Illumina, San Diego, CA, USA)。
1.3 扩增子测序和数据分析下机数据中拆分出各样品数据, 截去Barcode和引物序列后使用FLASH(V1.2.7, http://ccb.jhu.edu/software/FLASH/)对每个样品的reads进行拼接, 得到原始的Tags数据, 按照文献提供的方法进行过滤处理并得到高质量的Tags数据[20]。然后, 参照Qiime(V1.9.1, http://qiime.org/scripts/split_libraries_fastq.html)的Tags质量控制流程, 进行Tags截取和长度过滤[20]。经过以上处理后得到的Tags进行去除嵌合体序列(http://www.drive5.com/usearch/manual/chimera_formation.html)的处理, Tags序列通过(UCHIME Algorithm, http://www.drive5.com/usearch/manual/uchime_algo.html)与数据库(Gold database, http://drive5.com/uchime/uchime_download.html)进行比对检测嵌合体序列, 并最终去除其中的嵌合体序列[21], 得到最终的有效数据(Effective Tags)。
利用Uparse软件(Uparse v7.0.1001, http://drive5.com/uparse/)对所有样品的全部Effective Tags进行聚类, 默认以97%的一致性(Identity)将序列聚类成为OTUs(Operational Taxonomic Units), 去除单条序列代表的OTU, 同时筛选OTUs中出现频数最高的序列作为OTUs的代表序列[22]。对OTUs代表序列进行物种注释, 细菌使用Mothur方法与SILVA(http://www.arb-silva.de/)进行物种注释分析(设定阈值为0.8—1), 真菌使用Qiime软件(Version 1.9.1)中的blast方法(http://qiime.org/scripts/assign_taxonomy.html)与UNITE数据库(https://unite.ut.ee/)进行物种注释分析, 获得分类学信息并分别在各个分类水平上(界、门、纲、目、科、属、种)统计各样本的群落组成。最后, 以样品中数据量最少的一组为标准进行均一化处理, 结果用于后续的Alpha多样性分析和Beta多样性分析[20]。
使用Qiime软件(Version 1.9.1)计算微生物群落丰度指数(Chao、ACE), 多样性指数(Shannon、Simpson), 测度深度指数(Good′s coverage)和系统发育多样性指数(PD whole tree[20]。同时, 用Qiime软件计算Unifrac距离、构建UPGMA样品聚类树[20]。PCoA分析使用R软件(Version 3.4.1)的WGCNA, stats和ggplot2软件包, 使用R软件绘制PCoA图[23]。使用R软件的vegan包(Adonis, permutations=999)进行Beta多样性指数组间差异分析, 组组之间进行比较, 选用的是Tukey检验或者agricolae包的wilcox检验。共有微生物分析基于各组样品中平均相对丰度高于0.01%的OTU, 使用软件包VennDiagram进行四组样品韦恩图的绘制。共有微生物在四组样品中发生的相对丰度情况使用pheatmap进行分析和绘制。
2 结果 2.1 测序结果本实验共计获得真菌和细菌的OTU数目分别为11858和23124个(图 1)。在进行共有微生物的分析中, 先去除各组相对丰度平均值低于0.01%的OTU, 最后苗木非根际土、苗根际土、种植土和珊瑚砂土中剩余的真菌OTU数目分别为695、710、397个和560个;同时, 剩余的细菌OTU数目分别为140、141、148个和219个。
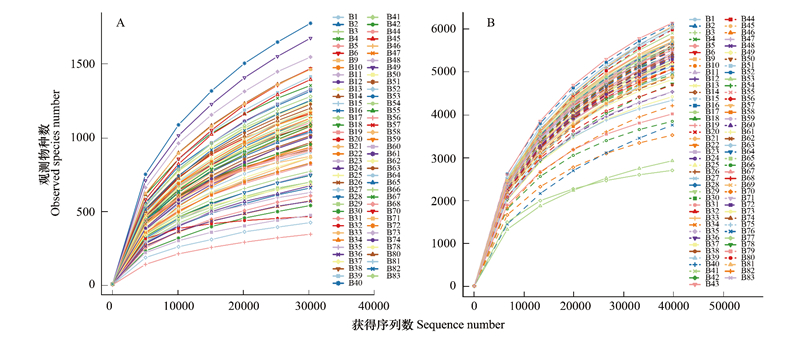
|
| 图 1 本实验样品真菌和细菌的稀释曲线 Fig. 1 The rarefaction curves of sequenced samples in this study A, 稀释曲线(30274)绘制样品获得真菌的序列数目和观察到物种数的情况;B, 稀释曲线(39730)绘制样品获得细菌的序列数目和观察到物种数的情况; B1—B33号土样, 为2017年6月6—12日采自珊瑚岛的样品;B34—B39号土样, 为2017年6月15日采自珊瑚岛的种植土样品;B40—B83号土样, 为2017年8月14日采自海南文昌凌叶苗圃样品 |
珊瑚岛本身的土壤为珊瑚砂, 其本身的微生物含量非常低, 本实验从珊瑚砂中提取的DNA总量并不能满足公司的测序要求。虽然珊瑚岛的土壤中汇集来自不同土壤来源的微生物, 但其中的真菌和细菌的丰度和多样性较苗非根际土、根际土和种植土都没有显著的升高(图 2)。相反, 珊瑚砂土中的真菌丰度和多样性比苗根际土和非根际土较低。具体表现为, 珊瑚砂土的真菌丰度为864.2±41.4, 显著性低于苗根际土的1086.1±64.3(df=3, F=10.73, P=0.014), 以及极显著低于苗非根际土的1251.4±48.1(df= 3, F=10.73, P < 0.001)。同时, 珊瑚砂土真菌的多样性(香农指数)为5.1±0.2, 相比于苗非根际土5.9±0.1(df=3, F=3.17, P=0.018)、根际土5.5±0.2和种植土5.4±0.2都有所降低。珊瑚砂土的细菌丰度为5342.8±61.6介于苗土和种植土之间, 但是并没有表现出显著性差异;而其香农指数为10.6±0.04, 要显著的高于苗根际土的10.2±0.1(df=3, F=3.49, P=0.024)。
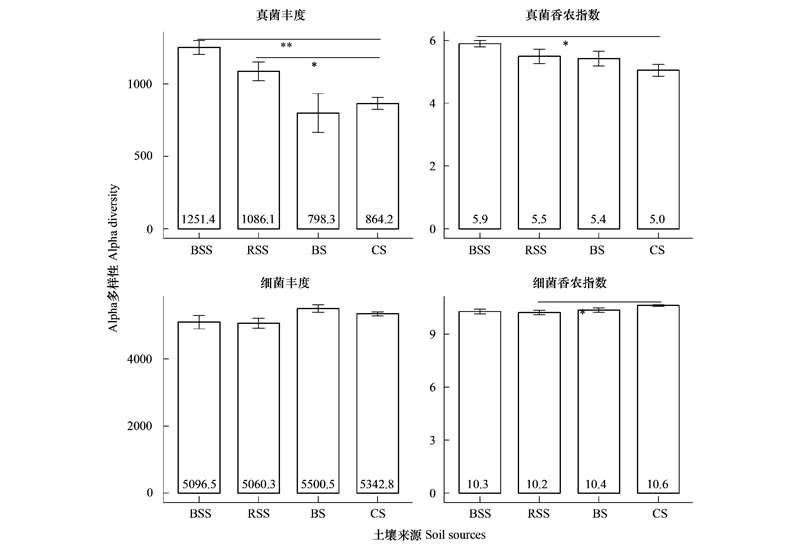
|
| 图 2 土壤样品中真菌和细菌群落的Alpha多样性比较 Fig. 2 Comparison of fungal and bacterial Alpha-diversities among the four soil sources BSS, 苗非根际土bulk soil of seedling;RSS, 苗根际土rhizosphere soil of seedling;BS, 种植土bulk soil;CS, 珊瑚砂土coral sand |
从微生物的群落组成和结构上来看, 样品间的真菌(为苗根际土、非根际土与珊瑚砂土的比较, df=1, F=7.59, R2=0.094, P=0.001)和细菌(df=3, F=8.6, R2=0.25, P=0.001)都存在极显著性差异。珊瑚砂土与来自文昌的苗非根际土和根际土相比, 其真菌和细菌群落在第一主成分上(分别是9.3%和26.8%)存在明显的差异(Tukey:真菌Pa珊瑚岛=0.024;细菌苗非根际土和珊瑚砂土Pa珊瑚岛=0.029, 苗根际土与珊瑚砂土Pa珊瑚岛=0.071)。而在植物种植之后, 珊瑚砂土壤的真菌和细菌群落相比种植土沿着第二主成分(分别是5.9%和7.1%)发生偏离(Tukey:真菌未检测显著性差异;细菌种植土和珊瑚砂土Pa珊瑚岛=0.048), 说明植物的种植可能开始对珊瑚砂土中的微生物群落产生着影响(图 3)。
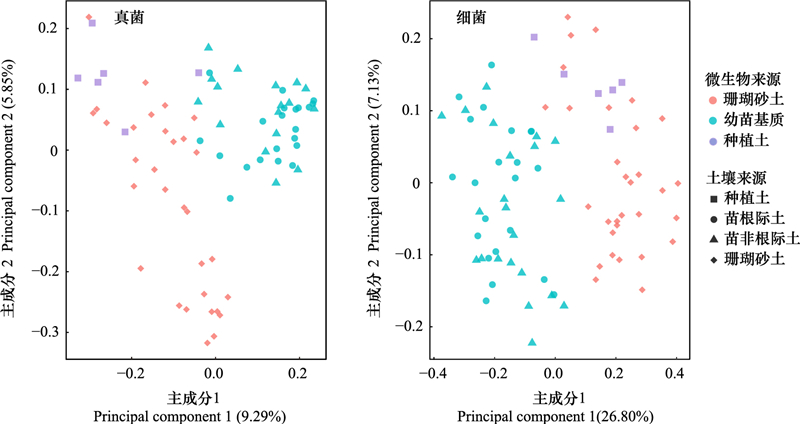
|
| 图 3 基于Bray-Curtis距离的PCoA作图比较样品间的Beta多样性 Fig. 3 Comparison of Beta-diversities among four soil sources using PCoA plot based on Bray-Curtis distances |
从以上结果可以看出, 在植物群落恢复过程中, 珊瑚砂土壤中的真菌和细菌类群并非是对来自不同土壤的微生物进行简单的叠加, 而是经历了一个特定的选择过程。植物种植后, 珊瑚砂土壤相对于种植土, 其真菌和细菌类群都发生了变化, 主要类群中粪壳菌纲(从21.5%到31%)、接合菌门(0.2%到17.4%)的相对丰度上升;相反, 伞菌纲(20.8%到0.9%)、座囊菌纲(16.3%到13.8%)、单毛壶菌纲(0.05%到0.01%)、β-变形菌(17.7%到0.1%)的相对丰度都有所下降(图 4)。
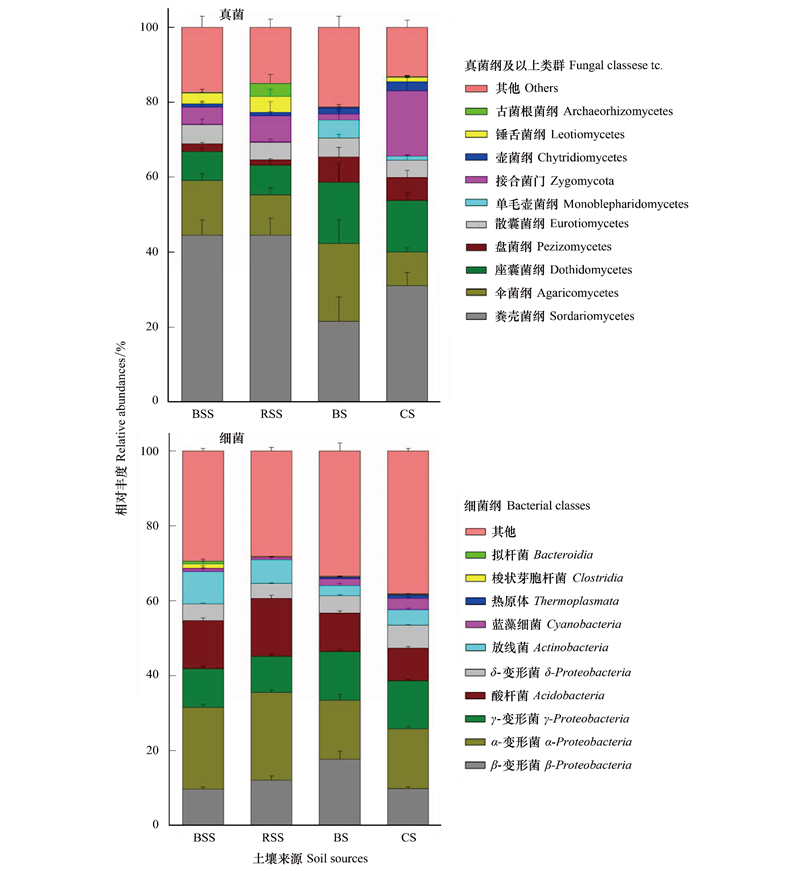
|
| 图 4 土壤样品中真菌和细菌的组成分布 Fig. 4 The composition of fungal and bacterial relative abundance among the four soil sources A, 真菌主要类群在相对丰度值上的分布情况;B, 细菌主要类群在相对丰度值上的分布情况 |
针对共有真菌的韦恩图分析, 可以看出, 珊瑚砂土(560个OTU)中保留着来自苗非根际土、苗根际土和种植土的OTU数目分别为210、212和188个, 去除三组中的重叠OTU后剩余323, 占珊瑚砂土的57.8%。同时, 珊瑚砂土中特有的OTU数目为237个, 占42.3%。四组样品共有的真菌OTU累计数为88个, 总共的相对丰度值约占40%左右, 以肉座菌目、微囊菌目、接合菌纲等8个真菌类群为主, 其中主要包括的真菌物种为35个。在植物引入之后, 珊瑚砂土中主要的真菌种群变化明显, 苗非根际土和根际土主要种群的发生更为相似, 而珊瑚砂土与种植土种群的发生更为相似, 但是已经能观察到珊瑚砂土相对于种植土发生的偏移。来自苗根际和非根际的16个优势种群(枝Ⅰ、Ⅲ、Ⅳ), 在珊瑚砂土中相对丰度值下降;相反, 其中的9个弱势种群(枝Ⅵ、Ⅶ), 在珊瑚砂土中相对丰度值上升。同时, 种植土的8个优势种群(枝Ⅴ)和7个弱势种群(枝Ⅶ)在珊瑚砂土中分别表现为下降和上升(图 5)。
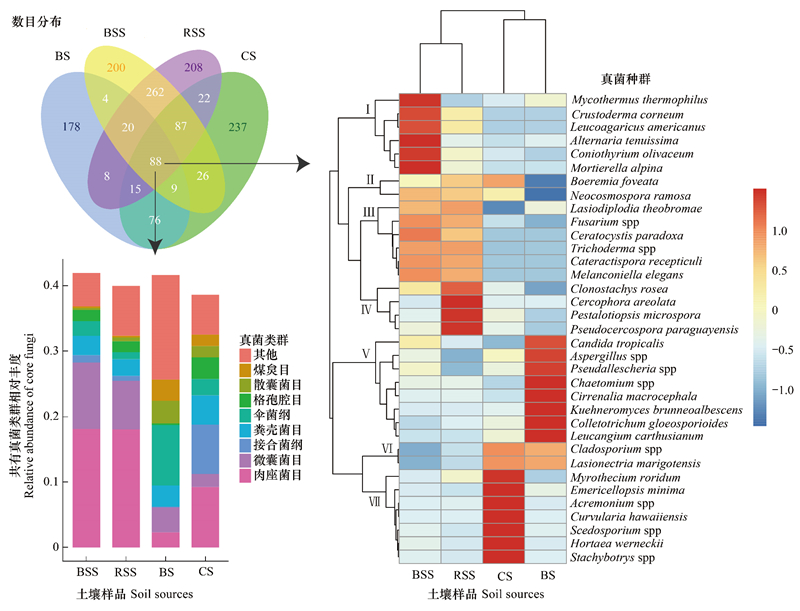
|
| 图 5 不同土壤样品间真菌共有OTU分析 Fig. 5 Fungal core OTU analysis among four soil sources |
针对共有细菌的韦恩图分析, 可以看出, 珊瑚砂土(219个OTU)中保留着来自苗非根际土、苗根际土和种植土的OTU数目分别为65、60和112个, 去除三组中的重叠OTU后剩余121, 占珊瑚砂土的55.3%。同时, 珊瑚砂土中特有的OTU数目为98个, 占44.7%。四组样品中共有的细菌OTU累计数目为51个, 总共相对丰度值约占17%左右, 以伯克霍尔德氏菌目、根瘤菌目、黄色单胞菌目、放线菌目、鞘脂单胞菌目、假单胞菌目和绿弯菌门的细菌类群为主, 主要包括的细菌物种为23个。在植物引入之后, 珊瑚砂土中主要的细菌类群同样变化明显, 苗非根际土和根际土主要类群的发生更为相似, 而珊瑚砂土与种植土类群的发生更为相似。来自苗根际和非根际的16个优势类群(枝Ⅳ、Ⅴ), 在珊瑚砂土中并未取得优势地位。同时, 种植土的3个优势类群(枝Ⅰ)在珊瑚砂土中表现为下降(图 6)。
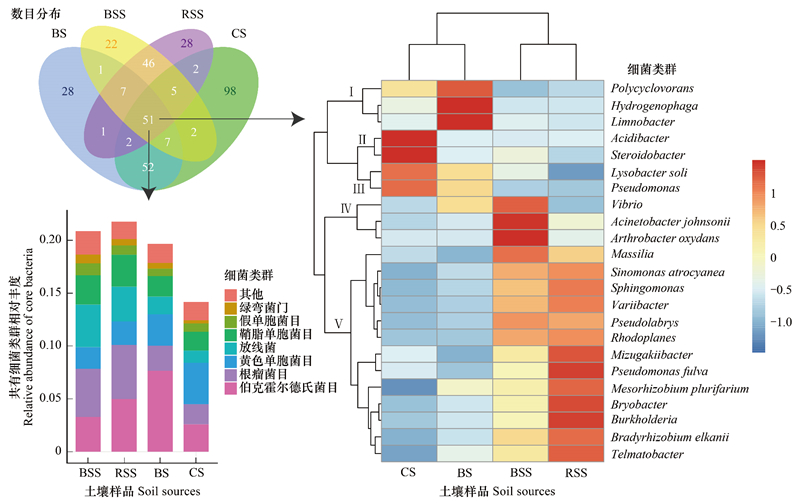
|
| 图 6 不同土壤样品间细菌共有OTU分析 Fig. 6 Bacterial core OTU analysis among four soil sources |
从中可以看出, 珊瑚砂土在植物引入之后, 主要的微生物类群在原有种植土的基础上发生变化, 同时也并未和植物根际土和非根际土的趋同。在植被恢复的过程中, 珊瑚砂土壤也在建成自己独特的微生物群落, 其中真菌群落以Boeremia foveata、Neocosmospora ramosa、Cladosporium spp、Lasionectria marigotensis、Myrothecium roridum、Emericellopsis minima、Acremonium spp、Curvularia hawaiiensis、Scedosporium spp、Hortaea werneckii和Stachybotrys spp为主(枝Ⅱ、Ⅵ、Ⅶ, 图 5), 而细菌群落以Acidibacter、Steroidobacter、Lysobacter soli和Pseudomonas为主(枝Ⅱ、Ⅲ, 图 6)。
3 讨论本研究利用ITS和16S扩增子测序的方法, 调查在珊瑚岛植物种植早期土壤微生物群落的形成和发育过程。从分析结果中可以看出, 改良过的种植土和引入植物基质中的非根际土、根际土很有可能是珊瑚岛土壤的主要微生物来源, 也即珊瑚岛早期土壤微生物群主要是外源;这一点从我们对珊瑚砂土微生物群落的分析也可以得到印证, 从珊瑚砂中提取的土壤微生物DNA总量不能满足公司的测序要求。然而, 随着时间的推移, 代表珊瑚岛的珊瑚砂土壤中的微生物组成, 以及群落结构并没有趋同于植物根际土和非根际土, 而是表现出对珊瑚岛特定环境的适应性, 也即随改良土和种植土引入的微生物群落在新的珊瑚岛环境下开始演化、超适应新环境的方向发展。因此, 持续长期监测并与本研究测定的初期状况进行比较, 将是一个非常有价值的关于土壤微生物如何与植物群落互作, 适应新环境, 演化发育的研究案例。
相比于其他研究中的土壤样品[24-26], 本研究从不同类型的土壤样品中所获得的平均真菌物种数(798—1251)和细菌物种数(5060—5501)较低。这可能与土壤类型, 环境条件和土壤DNA的提取方法有关。比如, 珊瑚砂土具有营养贫瘠、高盐等特点, 而且当地环境条件恶劣[3]。另一方面, 由于土壤样品量有限, 我们研究中只称取了0.25 g土壤进行DNA提取, 因此可能限制了所观察到的微生物物种数。
通常来说, 植物非根际土和根际土的微生物群落受植物的影响较大, 比如植物根际产生的分泌物, 能够吸引腐生型真菌和细菌的生长[27-29]。这可能是造成本实验中发现的非根际土和根际土细菌的主要类群要比珊瑚岛的土壤更丰富。而在真菌方面, 具有腐生生活史的真菌如粪壳菌纲[30-32], 以及能够分解植物根系凋落物的伞菌纲的真菌相对丰度也要高于珊瑚砂土[33-34];特别是粪壳菌纲的真菌, 可以看出在植物种植后, 其相对丰度值从种植土到珊瑚砂土中有所提升, 说明珊瑚砂土微生物群落处在恢复过程中, 而这种恢复受到植物种植的明显影响。同时, 珊瑚岛因为具有高温等特殊的气候条件, 这可能是造成其土壤中接合菌门真菌相对丰度较高的原因之一。
另外, 通过对核心微生物(相对丰度大于0.01%)的分析, 可以看出植物的非根际、根际土和种植土中携带有不少植物病害相关的真菌和细菌。植物相关土壤中携带的重要的病原真菌种类包括链格孢属、镰刀菌属、可可毛色二孢菌等[35-36], 而其细菌不仅包括某些病原物, 还包括一些像根瘤菌目的固氮菌, 说明其土壤中的微生物与植物的关系更为密切[9-10]。相比之下, 这些类群的真菌和细菌, 目前在珊瑚砂土壤中虽然同样存在, 但是它们的相对丰度值较植物的非根际土壤和根际土壤来说, 都要低很多。由此可见, 在植物栽植的早期, 与其密切相关的微生物也处在恢复过程中;而随着珊瑚岛植物群落的进一步发育, 特别是植物根系逐渐发达, 这部分微生物会如何发生变化, 也值得进一步的研究。另外, 珊瑚砂岛是一个独立的生态系统, 植被单一, 目前树木生长状况相对较弱, 某些病原菌可能在短时间内流行并危害植物。因此建议:(1)对植物引种地进行病害调查, 记录常见的植物病害, 并据此编辑可能发生病害的识别手册;(2)定期对岛上植被进行病害调查, 储备环境友好型杀菌剂, 尽量在病害发生早期对其进行有效的控制;(3)对出现的死亡植株残体、落叶进行清理, 集中焚烧和掩埋, 避免病原菌的滋生和蔓延。
总体上来说, 本研究追踪采集植物种植不同阶段根系所接触的土壤介质, 从苗圃里幼苗根际土、种植土到栽植后珊瑚砂土, 分析了土壤微生物群落的组成变化情况, 发现目前珊瑚岛土壤的主要微生物来源是随栽植苗木和改良土;明确了目前珊瑚岛上土壤微生物群落的主要真菌和细菌类群;识别出了一些重要的功能类群, 比如病原菌、固氮菌。这为将来进一步研究岛上土壤微生物群落的发展演化提供了基础资料。在土壤微生物群落进行发育、恢复的过程中, 如何对土壤理化性质进行进一步的改良以增加其蓄水保肥功能, 也是保障群落幼苗更新、群落自我发育的关键。初步的一些措施建议有:(1)可把现有群落中的凋落物收集形成堆肥腐化后再施回, 从而增加土壤肥力;(2)也可在现有群落枯落物表面唝施一些无机氮、磷、钾复合肥, 以促进枯落物早期阶段的分解;(3)后期分解阶段可增施一些锰、镁含量比较高的无机肥, 以促进枯落物后期阶段的分解。这些具体的土壤改良措施都需要进一步试验验证后推广。
| [1] |
刘东明, 陈红锋, 王发国, 易绮斐, 邢福武. 我国南沙群岛岛礁引种植物调查. 热带亚热带植物学报, 2015, 23(2): 167-175. |
| [2] |
任海, 简曙光, 张倩媚, 王发国, 沈彤, 王俊. 中国南海诸岛的植物和植被现状. 生态环境学报, 2017, 26(10): 1639-1648. |
| [3] |
王玮韧, 郝珖存, 王俊, 张炜, 黄峰, 陆宏芳, 简曙光, 何聃, 申卫军. 珊瑚岛礁表层土壤的主要化学性质分析. 热带亚热带植物学报, 2018, 26(5): 465-472. |
| [4] |
邢福武, 吴德邻, 李泽贤, 赵焕庭, 陈史坚. 我国南沙群岛的植物与植被概况. 广西植物, 1994, 14(2): 151-156. |
| [5] |
童毅, 简曙光, 陈权, 李玉玲, 邢福武. 中国西沙群岛植物多样性. 生物多样性, 2013, 21(3): 364-374. |
| [6] |
翁伯琦, 郑祥洲, 丁洪, 王煌平. 植被恢复对土壤碳氮循环的影响研究进展. 应用生态学报, 2013, 24(12): 3610-3616. |
| [7] |
李刚, 修伟明, 王杰, 于雯超, 吴元凤, 赵建宁, 宋晓龙, 杨殿林. 呼伦贝尔沙地不同植被恢复模式土壤氨氧化细菌群落结构及多样性. 农业环境科学学报, 2014, 33(1): 111-120. |
| [8] |
钟芳, 柴晓虹, 王国基, 段争虎. 植被恢复方式对黄土丘陵区土壤理化性质及微生物特性的影响. 中国沙漠, 2014, 34(4): 1064-1072. |
| [9] |
Jung J, Yeom J, Han J, Kim J, Park W. Seasonal changes in nitrogen-cycle gene abundances and in bacterial communities in acidic forest soils. Journal of Microbiology, 2012, 50(3): 365-373. DOI:10.1007/s12275-012-1465-2 |
| [10] |
Desai M S, Assig K, Dattagupta S. Nitrogen fixation in distinct microbial niches within a chemoautotrophy-driven cave ecosystem. The ISME Journal, 2013, 7(12): 2411-2423. DOI:10.1038/ismej.2013.126 |
| [11] |
Inoue J I, Oshima K, Suda W, Sakamoto M, Iino T, Noda S, Hongoh Y, Hattori M, Ohkuma M. Distribution and evolution of nitrogen fixation genes in the phylum Bacteroidetes. Microbes and Environments, 2015, 30(1): 44-50. DOI:10.1264/jsme2.ME14142 |
| [12] |
Stein L Y, Klotz M G. The nitrogen cycle. Current Biology, 2016, 26(3): R94-R98. DOI:10.1016/j.cub.2015.12.021 |
| [13] |
Yadav V, Kumar M, Deep D K, Kumar H, Sharma R, Tripathi T, Tuteja N, Saxena A K, Johri A K. A phosphate transporter root endophytic fungus Piriformospora indica plays a role in phosphate transport to the host plant. The Journal of Biological Chemistry, 2010, 285(34): 26532-26544. DOI:10.1074/jbc.M110.111021 |
| [14] |
Rosconi F, Trovero M F, de Souza E M, Fabiano E. Serobactins-mediated iron acquisition systems optimize competitive fitness of Herbaspirillum seropedicae inside rice plants. Environmental Microbiology, 2016, 18(8): 2523-2533. DOI:10.1111/1462-2920.13202 |
| [15] |
van der Heijden M G A, Bardgett R D, van Straalen N M. The unseen majority: soil microbes as drivers of plant diversity and productivity in terrestrial ecosystems. Ecology Letters, 2008, 11(3): 296-310. DOI:10.1111/j.1461-0248.2007.01139.x |
| [16] |
Chowdhury S P, Uhl J, Grosch R, Alquéres S, Pittroff S, Dietel K, Schmitt-Kopplin P, Borriss R, Hartmann A. Cyclic Lipopeptides of Bacillus amyloliquefaciens subsp. plantarum colonizing the lettuce rhizosphere enhance plant defense responses toward the bottom rot pathogen Rhizoctonia solani. Molecular Plant-Microbe Interactions, 2015, 28(9): 984-995. DOI:10.1094/MPMI-03-15-0066-R |
| [17] |
张守仕, 谢克英, 乔宝营, 张娜. 果树根际土壤取样分析技术研究. 落叶果树, 2015, 47(6): 25-27. |
| [18] |
Martin K J, Rygiewicz P T. Fungal-specific PCR primers developed for analysis of the ITS region of environmental DNA extracts. BMC Microbiology, 2005, 5: 28. DOI:10.1186/1471-2180-5-28 |
| [19] |
Purahong W, Wubet T, Lentendu G, Schloter M, Pecyna M J, Kapturska D, Hofrichter M, Krüger D, Buscot F. Life in leaf litter: novel insights into community dynamics of bacteria and fungi during litter decomposition. Molecular Ecology, 2016, 25(16): 4059-4074. DOI:10.1111/mec.13739 |
| [20] |
Caporaso J G, Kuczynski J, Stombaugh J, Bittinger K, Bushman F D, Costello E K, Fierer N, Peña A G, Goodrich J K, Gordon J I, Huttley G A, Kelley S T, Knights D, Koenig J E, Ley R E, Lozupone C A, McDonald D, Muegge B D, Pirrung M, Reeder J, Sevinsky J R, Turnbaugh P J, Walters W A, Widmann J, Yatsunenko T, Zaneveld J, Knight R. QⅡME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [21] |
Edgar R C, Haas B J, Clemente J C, Quince C, Knight R. UCHIME improves sensitivity and speed of chimera detection. Bioinformatics, 2011, 27(16): 2194-2200. DOI:10.1093/bioinformatics/btr381 |
| [22] |
Edgar R C. UPARSE: highly accurate OTU sequences from microbial amplicon reads. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [23] |
R Core Team. R: A language and environment for statistical computing. R Foundation for Statistical Computing, Vienna, Austria, 2013, URL: http://www.R-project.org/.
|
| [24] |
Lanzén A, Epelde L, Blanco F, Martín I, Artetxe U, Garbisu C. Multi-targeted metagenetic analysis of the influence of climate and environmental parameters on soil microbial communities along an elevational gradient. Scientific Reports, 2016, 6: 28257. DOI:10.1038/srep28257 |
| [25] |
Štursová M, Bárta J, Šantrčková H, Baldrian P. Small-scale spatial heterogeneity of ecosystem properties, microbial community composition and microbial activities in a temperate mountain forest soil. FEMS Microbiology Ecology, 2016, 92(12). DOI:10.1093/femsec/fiw185 |
| [26] |
Žifčáková L, Větrovsky' T, Howe A, Baldrian P. Microbial activity in forest soil reflects the changes in ecosystem properties between summer and winter. Environmental Microbiology, 2016, 18(1): 288-301. DOI:10.1111/1462-2920.13026 |
| [27] |
Nguema-Ona E, Vicré-gibouin M, Cannesan M A, Driouich A. Arabinogalactan proteins in root-microbe interactions. Trends in Plant Science, 2013, 18(8): 440-449. DOI:10.1016/j.tplants.2013.03.006 |
| [28] |
Mao Y J, Li X Z, Smyth E M, Yannarell A C, Mackie R I. Enrichment of specific bacterial and eukaryotic microbes in the rhizosphere of switchgrass (Panicum virgatum L.) through root exudates. Environmental Microbiology Reports, 2014, 6(3): 293-306. DOI:10.1111/1758-2229.12152 |
| [29] |
Wu L K, Wang J Y, Huang W M, Wu H M, Chen J, Yang Y Q, Zhang Z Y, Lin W X. Plant-microbe rhizosphere interactions mediated by Rehmannia glutinosa root exudates under consecutive monoculture. Scientific Reports, 2015, 5: 15871. DOI:10.1038/srep15871 |
| [30] |
Perdomo H, García D, Gené J, Cano J, Sutton D A, Summerbell R, Guarro J. Phialemoniopsis, a new genus of Sordariomycetes, and new species of Phialemonium and Lecythophora. Mycologia, 2013, 105(2): 398-421. DOI:10.3852/12-137 |
| [31] |
Chen H, Ma Y, Zhang W F, Ma T, Wu H X. Molecular phylogeny of Colletotrichum (Sordariomycetes: Glomerellaceae) inferred from multiple gene sequences. Genetics and Molecular Research, 2015, 14(4): 13649-13662. DOI:10.4238/2015.October.28.27 |
| [32] |
Réblová M, Réblová K, Štěpánek V. Molecular systematics of Barbatosphaeria (Sordariomycetes): multigene phylogeny and secondary ITS structure. Persoonia-Molecular Phylogeny and Evolution of Fungi, 2015, 35: 21-38. DOI:10.3767/003158515X687434 |
| [33] |
Liers C, Pecyna M J, Kellner H, Worrich A, Zorn H, Steffen K T, Hofrichter M, Ullrich R. Substrate oxidation by dye-decolorizing peroxidases (DyPs) from wood- and litter-degrading agaricomycetes compared to other fungal and plant heme-peroxidases. Applied Microbiology and Biotechnology, 2013, 97(13): 5839-5849. DOI:10.1007/s00253-012-4521-2 |
| [34] |
Mali T, Kuuskeri J, Shah F, Lundell T K. Interactions affect hyphal growth and enzyme profiles in combinations of coniferous wood-decaying fungi of Agaricomycetes. PLoS One, 2017, 12(9): e0185171. DOI:10.1371/journal.pone.0185171 |
| [35] |
Lysøe E, Harris L J, Walkowiak S, Subramaniam R, Divon H H, Riiser E S, Llorens C, Gabaldón T, Kistler H C, Jonkers W, Kolseth A K, Nielsen K F, Thrane U, Frandsen R J N. The genome of the generalist plant pathogen Fusarium avenaceum is enriched with genes involved in redox, signaling and secondary metabolism. PLoS One, 2014, 9(11): e112703. DOI:10.1371/journal.pone.0112703 |
| [36] |
Dang H X, Pryor B, Peever T, Lawrence C B. The Alternaria genomes database: a comprehensive resource for a fungal genus comprised of saprophytes, plant pathogens, and allergenic species. BMC Genomics, 2015, 16: 239. DOI:10.1186/s12864-015-1430-7 |
 2019, Vol. 39
2019, Vol. 39




