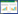文章信息
- 马超, 龚鑫, 郜红建, 吴婧, 李大明, 陈小云, 李辉信, 刘满强.
- MA Chao, GONG Xin, GAO Hongjian, WU Jing, LI Daming, CHEN Xiaoyun, LI Huixin, LIU Manqiang.
- 历史因素对土壤微生物群落与外来细菌入侵间关系的影响
- Legacy impacts on the relationships between soil microbial community and the invasion potential of non-indigenous bacteria
- 生态学报. 2018, 38(22): 7933-7941
- Acta Ecologica Sinica. 2018, 38(22): 7933-7941
- http://dx.doi.org/10.5846/stxb201804120832
-
文章历史
- 收稿日期: 2018-04-12
- 修订日期: 2018-10-08
2. 农田生态保育与污染防控安徽省重点实验室, 安徽农业大学资源与环境学院, 合肥 230036;
3. 江西省红壤研究所, 南昌 331717
2. Anhui Province Key Laboratory of Farmland Ecological Conservation and Pollution Prevention, School of Resources and Environment, Anhui Agricultural University, Hefei 230036, China;
3. Jiangxi Institute of Red Soil, Nanchang 331717, China
鉴于对指导生态保护实践和验证生态学经典理论发展均有重要作用, 土著生物群落特征对外来种入侵的影响(即群落可入侵性)一直都是入侵生态学的研究热点[1-3]。保护和基础生态学家曾分别根据群落可入侵性研究成果明确优先保护的生态区域和探究生物群落构建的机理[4-5]。但当前群落可入侵性和群落特征间关系的报道却存在诸多分歧, 既有认为物种多样性会增加群落稳定性的生物多样性阻抗假说[1], 也有与之对立的反多样性阻抗假说——生物多样性高的群落反而更易被入侵[6]。郑景明和马克平[7]提出研究尺度不同是造成上述分歧的主要原因:局域尺度上的生物入侵过程由生物相互作用主导, 生物多样性高的群落空余生态位少, 因而外来种入侵和本地物种多样性呈负相关[8];区域尺度上环境筛控制外来种的入侵, 生物多样性高则意味着资源多样性化程度高、生态位类型丰富, 因而外来入侵种表现与土著生物多样性成正比[9]。但现有关于土壤微域尺度生物群落特征对群落可入侵性影响的研究却比较少。
前人研究指出, 调控生物入侵的关键因素在入侵的不同阶段可能会存在差异, 从而使外来物种在入侵状态在不同阶段表现异同[10-13]。例如, Nawrot等[12]研究得出双壳类动物在红海地区的入侵到达和建立分别由环境筛和生物相互作用主导;Ricciardi和Cohen[13]调查发现外来物种在新栖息地的定殖能力与其对土著群落的危害程度无关。但不论外来种入侵处于何种阶段, 影响生物入侵的关键因素为何, 均不可能会影响被入侵地初始生物群落特征[14]。因此, 土著生物群落特征和群落可入侵性之间的关系也会因入侵阶段改变而变化。此外, 由于群落形成实际上是由生物相互作用和环境筛选共同调控的[15], 且群落的组成和结构与其经历的历史过程密切相关[16]。由此一个群落当下的特征不仅会反映系统的历史生物因素状况, 还可能反映系统的历史非生物因素的影响[17], 甚至全由历史非生物因素决定[18]。这意味着即使外来种入侵过程由生物相互作用主导, 但如果群落的生物多样性本身是由系统历史非生物因素控制的, 那么它最终也可能不会阻抗外来种的入侵(即不符合生物多样性阻抗假说)。所以, 历史影响因素不同也会引起土著群落特征和外来种入侵之间关系出现差异。综上可知, 探明土壤微域尺度不同入侵阶段的外来种状况与土著群落特征之间关系, 及其随土著群落特征决定因素变化状况尤为迫切。
本研究拟借助江西红壤研究所布设于1981年的长期施肥定位试验, 选择长期化肥和有机肥处理作用下的土壤, 并通过将土壤灭菌并回接已方和对方的土壤悬液重新构建土壤系统, 从而探究重建土壤系统生物群落特征与系统历史影响因素以及外源青枯菌接入重建土壤后存活状况之间的关系, 实现明确历史因素对土壤微生物群落特征与外来细菌入侵潜力间关系的影响及机制的目标。具体研究假说包括:1)不同土壤微生物群落特征对历史生物因素和非生物因素作用的响应不同;2)在任何入侵阶段, 外来细菌入侵潜力仅和与其受相同历史因素影响的生物群落特征相关, 而与其受不同历史因素影响的生物群落特征无关。
1 材料与方法 1.1 试验材料供试土壤分别采自江西省红壤研究所(28°15′30′E和116°20′24′′N)红壤性水稻土长期定位试验田的化肥(每季作物施N 90 kg/hm2、P2O5 45 kg/hm2、K2O 75 kg/hm2)和有机肥(早稻施22500 kg/hm2紫云英、晚稻施22500 kg/hm2猪粪)施肥处理, 土壤类型为美国制土壤分类中的强淋溶土和铁铝土[19]。从每处理的三个重复小区采集耕层土壤(0—15 cm), 混合后进行大中型土壤动物及根茬等残体剔除、过筛(5 mm)和冷藏(4℃), 基本理化性质如表 1。供试外来菌株为红色荧光蛋白标记的青枯菌Ralstonia solanacearum QL-Rs1115-RFP(R. solanacearum), 由南京农业大学江苏省有机固体废弃物利用重点实验室提供, RFP (红色荧光蛋白)标记后的菌株除了具Gmr (庆大霉素抗性)外, 其余性状同母株[20]。
| 土壤 Soil |
颗粒组成(砂%:粉%:黏%) Texture(sand%:clay%:silt%) |
酸碱度 pH(H2O) |
总有机碳 Total organic carbon/(g/kg) |
可溶性有机碳 Dissolved organic carbon/(mg/kg) |
全氮 Total nitrogen/(g/kg) |
速效氮 Available nitrogen/(mg/kg) |
微生物量碳 Microbial biomass/(mg/kg) |
| 化肥处理 Chemical fertilizer |
66:19:15 | 5.24±0.00 | 21.1±0.2 | 113.4±2.1 | 2.11±0.03 | 18.7±0.2 | 301.1±7.6 |
| 有机肥处理 Organic fertilizer |
84:12:4 | 5.31±0.00a | 26.6±0.8** | 142.5±4.5** | 2.60±0.06* | 17.6±0.7 | 586.0±9.1*** |
| 表中数字为平均值, *、**和***分别代表P < 0.05, < 0.01和0.001, a因为两组数据的标准差均为0, 故无法进行t检验 | |||||||
采用交叉互换设计以便区分土壤历史生物和非生物因素的作用[18, 21], 设置4个处理(图 1):1)化肥处理灭菌土壤+化肥处理土壤微生物群落(CsCm);2)化肥处理灭菌土壤+有机肥处理微生物群落(CsOm);3)有机肥处理灭菌土壤+化肥处理微生物群落(OsCm);4)有机肥处理灭菌土壤+有机肥处理土壤微生物群落(OsOm)。试验过程中进行5次破坏性采样, 每次每处理取3个重复。
|
| 图 1 两种施肥处理土壤生物和非生物因素交叉互换操作示意图 Fig. 1 The schematic diagram of reciprocal transplanted soil biotic and abiotic factors after sterilized two different fertilization soil CC和OO:初始化肥处理和有机肥处理土壤, initial soil of chemical fertilizer and organic fertilizer treatment;Cm, Cs, Os, 和Om:化肥处理土壤微生物群落, 化肥处理灭菌土壤, 有机肥处理土壤微生物群落和有机肥处理灭菌土壤, soil microbial inoculum and sterilized soil of chemical fertilizer treatment, as well as soil microbial inoculum and sterilized soil of organic fertilizer treatment;CsCm, CsOm, OsCm, 和OsOm:化肥处理灭菌土壤+化肥处理土壤微生物群落, 化肥处理灭菌土壤+有机肥处理微生物群落, 有机肥处理灭菌土壤+化肥处理微生物群落以及有机肥处理灭菌土壤+有机肥处理土壤微生物群落处理, microbial inoculum from organic amended soils was introduced into sterilized soils with either chemical or organic groups, as well as the microbial inoculum from chemically fertilized soils was introduced into sterilized soils with either chemical or organic groups |
根据试验设计对两种肥力土壤进行灭菌并回接已方和对方生物群落处理。首先, 称取100.0 g经γ-射线(50 KGy)辐射灭菌的两种施肥处理土壤, 分别放入250 mL带盖的玻璃瓶中。其次, 称取未灭菌土壤60.0 g, 加入盛180 mL无菌水和若干玻璃珠的三角烧瓶中并振摇30 min, 制成土壤生物接种悬液。最后, 将装有灭菌土壤的玻璃瓶等分成两份后, 分别均匀滴加已方和对方的土壤悬液3 mL即可。重建完成后, 将60个玻璃瓶一同放在25℃黑暗下培养180 d (使所构建系统稳定)再接种外来菌。接种外来细菌前, 随机破坏性取不同处理土壤各3份进行土壤微生物群落分析。
R. solanacearum接种前需在含30 μg/mL庆大霉素的50 mL LB培养基中进行富集培养(220 r/min, 30℃)。将培养至指数生长期的细菌用无菌水离心(8000 g, 5 min)重悬3次后稀释成108 CFU/mL的菌液。分别移取3 mL稀释过的R. solanacearum菌液至前述置换处理后的土壤系统中, 使最终接种量达107 CFU/g干土。25℃黑暗条件下继续培养56天, 于接种后第7、14、28天和56天进行破坏性采样。
1.3 指标测定土壤原生动物数量采用最大或然率法进行计数[22]。土壤微生物磷脂脂肪酸分析参照Frostegard等[23]方法进行。土壤细菌物种组成采用末端限制性片段长度多态性技术(T-RLFP)进行测定, 土壤总DNA提取使用FastDNA® SPIN Kit for Soil试剂盒(MP Biomedicals, Solon, OH, USA), 16S rRNA基因扩增引物为:5′端带荧光物质FAM标记的27f (6-FAM-5′-AGAGTTTGATCCTGGCTCAG-3′)和引物1492r (5′-GGTTACCTTGTTACGACTT-3′), 限制性内切酶HhaI, MspI和RSaI, 并以T-RFs的相对组成计算物种多样性[24]。土壤细菌代谢指纹分析通过Biolog-Eco板(Biolog, Hay-ward, CA, USA)培养测定实现, 由于供试土壤微生物碳源利用效率在72 h时最高, 因而以该时的试验结果计算功能多样性[25]。土壤微生物呼吸、土壤有机碳和总氮等的测定分别采用室内密闭培养法、高温外热重铬酸钾氧化-容量法和开氏消煮法[26]。
外来细菌接入土壤后的存活量采用修改过的稀释涂布计数法测定[27]。外来细菌接入土壤后的存活时间通过拟合Weibull单指数模型的方法获得[28], 即根据Weibull单指数模型的相关系数计算出土壤中外来细菌R. solanacearum达到检测线100 CFU/g所需的时间(Time needed to reach detection limit, ttd)。Weibull单指数模型:log(Nt)=log10(N0)-log10(t)p, 其中Nt为采样点土壤中外来细菌R. solanacearum的浓度, N0为R. solanacearum的初始土壤接种浓度, t为取样时间, δ为尺度参数, p为弧度参数。
1.4 数据分析数据分析前分别利用Kolmogorov-Smirnov和Levene方法检验数据的正态分布及方差齐性, 并在必要时进行转换。采用t检验分析土壤灭菌和交换接种前后土壤各微生物群落特征的差异。采用两因素方差分析探究灭菌土壤环境和接种土壤微生物群落单独和交互作用对重建土壤微生物群落特征以及外来细菌在其中存活的影响。冗于分析被用于揭示外来细菌存活量或存活时间与土壤各微生物群落特征的关系。线性回归分析进一步定量调查各土壤微生物群落特征与外来细菌存活状况的关系。数据统计和图形制作分别在R 3.4.3和Origin 9.0上完成。
2 结果和分析 2.1 灭菌土壤环境和接种生物群落对重建土壤微生物群落的影响重建土壤的微生物呼吸速率、细菌物种和功能多样性主要受灭菌后土壤环境因素影响(表 2)。仅接种生物群落不同时, 重建土壤的微生物呼吸速率、细菌物种多样性和功能多样性均无差异(P>0.05);当接种生物群落同为有机肥处理, 相比接入化肥处理的灭菌土壤, 接入有机肥的灭菌土壤微生物呼吸速率、细菌物种多样性和功能多样性分别显著下降了29.5%, 9.1%和6.5% (P < 0.05) (表 2)。
| 指标 Indices |
平均值和标准差 Mean ± s.e. under each treatment |
方差分析 F- and P-value from two-way ANOVA |
||||||
| CsCm | CsOm | OsCm | OsOm | SE | SM | SE× SM | ||
| 微生物呼吸 Microbial respiration/(mg kg-1 d-1) |
6.75±0.27 | 9.35±099B | 10.02±1.52 | 13.22±1.66A | 6.25* | 4.13 | 0.05 | |
| 原生动物数量Protozoan/(log个/g) | 11.28±0.29a | 9.28±0.21b | 11.20±0.48 | 9.77±0.05 | 0.35 | 24.37** | 0.66 | |
| 真菌生物量Fungi biomass/(nmol/g) | 2.08±0.45 | 1.81±0.24 | 1.14±0.16 | 1.61±0.17 | 3.11 | 1.00 | 1.35 | |
| 革兰氏阴阳细菌比 Gram-/Gram+ |
0.63±0.02b | 0.81±0.04a | 0.74±0.06 | 0.86±0.07 | 2.02 | 6.32* | 0.30 | |
| 细菌物种多样性 Bacterial species diversity |
2.07±0.06 | 2.02±0.07B | 2.29±0.10 | 2.24±0.01A | 8.25* | 0.41 | 0.00 | |
| 细菌功能多样性 Bacterial functional diversity |
2.84±0.07 | 2.89±0.04B | 2.91±0.09 | 3.10±0.01A | 4.20* | 2.82 | 0.88 | |
| t检验被用于分析土壤灭菌和交换接种前后土壤各微生物群落特征的差异, 小写字母表示同一施肥处理土壤灭菌后接种不同施肥影响下的土壤生物群落时t差异显著, 大写字母表示不同施肥处理土壤灭菌后接种相同处理土壤生物群落时t差异显著, 不同字母表示差异达到显著水平(P < 0.05)。两因素方差分析被用于探究灭菌土壤环境和接种土壤微生物群落单独和交互作用对重建土壤微生物群落特征的影响, *和**代表P < 0.05和P < 0.01; SE:灭菌土壤环境, sterilized soil;SM:接种微生物群落, soil microbial inoculum | ||||||||
重建土壤的原生动物数量、革兰氏阴性与阳性细菌比(G/G+)主要受接种土壤生物群落来源的影响(表 2)。在接种生物群落来源相同而土壤环境不同时, 原生动物数量和G/G+均无差异(P>0.05);当灭菌土壤同为化肥处理, 与接种化肥处理的生物群落相比, 接种有机肥处理土壤微生物群落后, 重建土壤原生动物数量和G/G+分别显著下降和上升了17.7%和22.2% (P < 0.05) (表 2)。重建后土壤的真菌生物量不受灭菌后土壤环境和接种生物群落来源的影响(P>0.05) (表 2)。
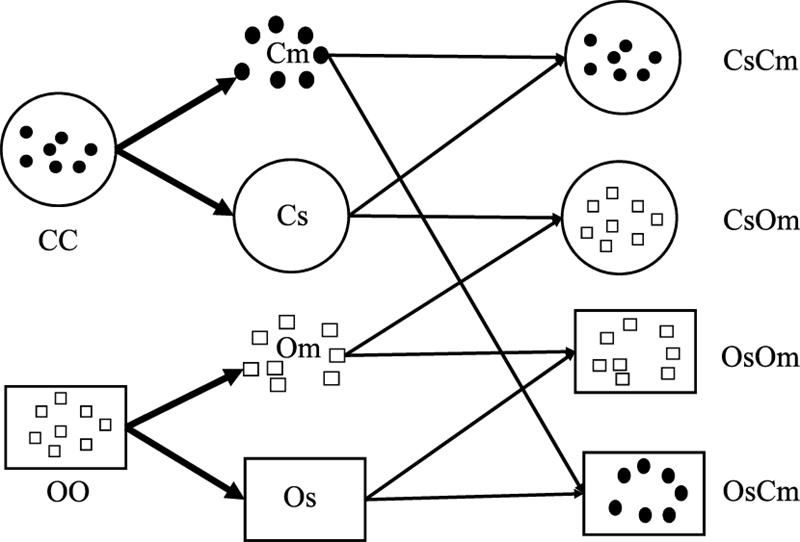
2.2 灭菌土壤环境和接种生物群落对外来细菌存活的影响R. solanacearum进入重建土壤后56天的存活量受主要微生物群落影响(F=10.78, P < 0.05) (图 2)。灭菌化肥处理土壤接种有机肥处理的土壤生物较接种自身生物会显著促进R. solanacearum的存活;灭菌有肥土壤接种化肥处理生物群落呈相反的趋势(图 2)。与存活量不同, R. solanacearum的存活时间却是由灭菌土壤环境决定的(F=2.35, P < 0.01) (图 2)。不论是接种有机肥还是化肥处理的生物群落, 灭菌有机肥处理土壤中外来R. solanacearum的存活时间始终显著高于灭菌化肥处理土壤的(图 2)。
|
| 图 2 不同灭菌土壤环境和微生物群落组合土壤中外来细菌的存活量和总存活时间 Fig. 2 Abundance and time needed to reach detection limit (ttd) of R. solanacearum after inoculating into different reconstructed soils 图中展示了两因素方差分析的F值和P值以及单因素方差分析和Duncan检验结果 |
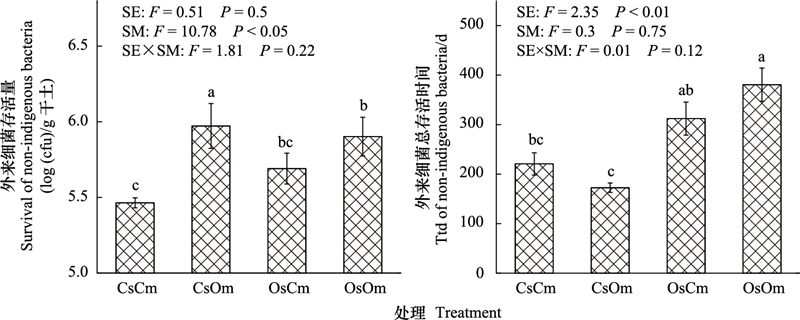
冗于分析显示, 不同土壤生物群落特征与R. solanacearum在土壤中存活量或存活时间的关系存在显著差异(图 3)。其中, R. solanacearum在56天的存活量与土壤原生动物数量和G/G+关系较为密切, 但R. solanacearum在土壤中存活时间则主要受土壤微生物呼吸速率、细菌物种和功能多样性影响。
|
| 图 3 外来R. solanacearum接入土壤后的存活状况与土壤生物群落特征关系的冗于分析 Fig. 3 Redundancy analysis (RDA) of soil microbial community characteristics and the survival of R. solanacearum in soil |
回归分析表明, 原生动物数量和G/G+同R. solanacearum在土壤的存活量明显相关, 且前者为负相关、后者为正相关(P < 0.05);而土壤微生物呼吸速率、细菌物种多样性和功能多样性则同R. solanacearum的存活时间均呈显著正相关(P < 0.05);土壤真菌生物量同上述二者均不相关(P>0.05) (图 4)。
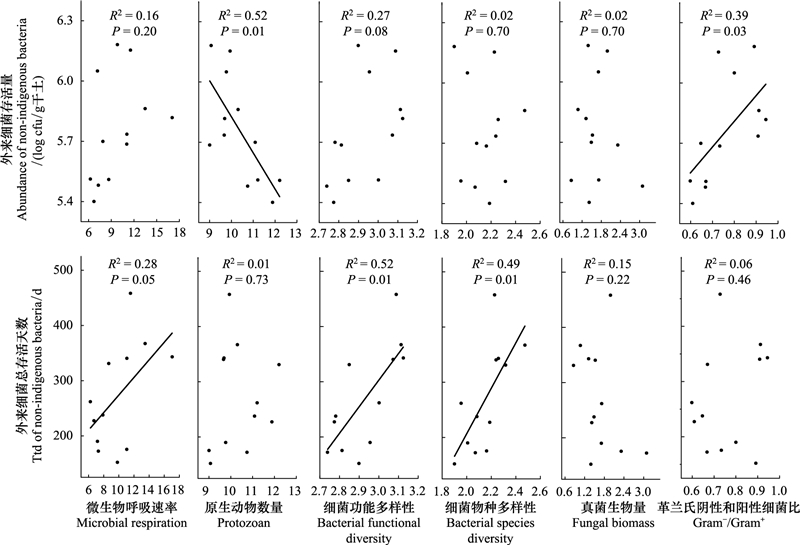
|
| 图 4 外来菌在土壤中存活状况与土壤各生物特征之间的关系 Fig. 4 Relationships between the survival of R. solanacearum in soil and resident soil microbial community characteristics 图中展示了相关性分析的R值和P值 |
重建土壤生物群落的原生动物数量、革兰氏阴性与阳性细菌比(G/G+)等群落特征主要由历史生物因素决定, 而微生物呼吸速率、细菌物种和功能多样等则受历史非生物因素控制(表 2), 这与Xun等[18]的发现类似。通过群落构建过程的研究, 前人发现群落的组成和结构是生物和非生物因素共同作用的结果[29]。因而重建土壤系统的微生物群落特征也会反映系统非生物因素状况。通过生物相互作用和环境筛选对外来种入侵相对贡献研究, 研究者发现系统生物群落特征不代表生物相互作用影响的情况[17, 30]。例如, Naeem等[17]指出当群落可入侵性被外界环境因素(如气候和资源)主导时, 生物多样性和群落可入侵性呈正相关, 不能反映土著物种的竞争作用对外来种存活的影响。土壤微生物活性、细菌的物种多样性和功能多样性等对土壤资源有效性的响应较为敏感, 而重建土壤生物群落的资源有效性又主要受灭菌土壤环境调控, 这使得上述微生物群落特征被灭菌的土壤环境特征所主导[1-33]。土壤G/G+会随灭菌土壤环境变化而出现一定波动, 但仅在接种悬液不同时才出现显著差异。这可能是因为系统中物种的出现与消失需较长的时间才能趋于平衡, 虽然8个月的时间跨度对周转速率较快的微生物群落来说较长, 但是从群落整体的组成和结构来说, 室内培养过程仍无法改变先前30多年田间管理措施所形成的影响格局[18, 29]。例如, 长期不同施肥措施不仅可通过调控土壤的养分供应来直接影响土壤微生物群落, 还能改变土壤pH、团聚体等理化性状而对土壤微生物产生间接作用[34]。土壤原生动物多以细菌为食, 自然也就会与土壤细菌的物种组成(G/G+)一样, 主要由接种土壤微生物群落所调控[35]。
已有的研究表明, 在不同入侵阶段, 决定外来种入侵成功与否的因素会有所差异[12-13]。与前人研究发现一致, 本研究显示历史生物因素主导外来细菌前期进入土壤系统的数量, 历史非生物因素控制外来细菌在土壤中的存活时间(图 2)。Levine等[2]也认为外来物种入侵初期受生物相互的影响强于环境因素的, 而后期则主要受到环境因素的影响。外来细菌接种后能够在56天后依然存在, 说明环境过滤作用(如pH)不会是其数量的限制因素。外来细菌进入土壤56天后数量没有爆增, 此时土壤有效资源相对丰富, 不会限制外来细菌的发展;但有限数量和尚未广泛分布的外来细菌会对捕食和拮抗作用等生物相互作用异常敏感[6-37]。反之, 随着进入土壤系统的时间延长, 外来细菌进入对数生长期, 其受捕食和拮抗作用的影响会减弱, 受土壤有效资源的影响会增强[38]。本研究还发现, 外来细菌进入土壤初期的存活数量与原生动物数量和G/G+相关;而外来细菌在土壤中存活天数与微生物活性、细菌物种多样性和功能多样性相关(图 3和图 4)。结合前段所述, 外来细菌的入侵状况是否可通过土壤微生物群落特征预测, 依赖于不同入侵阶段二者的历史影响因素是否一致。外来细菌接入土壤初期的入侵潜力会因原生动物数量和革兰氏阳性菌比例增加而降低。这符合天敌逃逸假说的论断, 因为原生动物和部分革兰氏阳性菌(如放线菌)可分别通过捕食和分泌杀菌性代谢产物抑制外来细菌的活动[39]。但是Yao等[28]研究却指出外来细菌进入土壤后所受的阻抗程度会随革兰氏阴性菌比例的增加而变强, 这或归因于不同研究所选择的外来菌种类和性状是不同的。Yao等[28]研究关注的是大肠杆菌(土壤常见细菌)存活时间和G/G+的关系, 而本研究分析的是青枯菌(一种土传病菌)接入土壤初期的存活量。外来细菌进入土壤后期的入侵潜力与土壤微生物活性、细菌物种和功能多样性呈正相关。一般认为小尺度上群落可入侵性与生物多样性会呈反比, 且物种多样性和功能多样性的表现应有差异[1, 10, 40]。上述分歧主要是因本研究中细菌物种和功能多样性以及土壤微生物呼吸均由历史非生物因素决定, 三者反映的均是土壤有效资源状况而非生物相互作用。
本研究的供试土壤是来自同一长期定位试验田块的不同施肥处理, 这两种化肥和有机肥影响的土壤在开展施肥处理前的生物和非生物性状是相同的。结合供试土壤基本性状和入侵试验结果可知, 长期不同施肥管理措施能够显著改变农田土壤的生物和非生物性状, 改变土壤的可入侵性。例如, 有机肥处理土壤中外来细菌的入侵潜力明显提升。这或归因于:1)有机肥可利用自身携带的微生物直接改变土著微生物群落结构, 可能改善外来细菌进入初期的存活状况;2)有机肥中丰富的有效资源可为外来细菌持续提供养分延长存活时间, 有机肥对土壤理化性质的改善也会进一步促进外来菌的生存[18, 27, 37]。
4 结论重建土壤系统的生物群落活性、组成和结构特征反映了历史生物和非生物因素的遗产效应。外来细菌不论是在入侵土壤前期还是后期, 其入侵潜力与生物群落特征相关与否均取决于二者的历史影响因素是否相同。长期不同施肥措施可通过改变农田土壤性质而实现对群落可入侵性的影响。本研究有助于不仅缓解群落多样性与可入侵性关系悖论, 还能指导有益菌剂田间使用和土传病害的防控。
致谢: 美国Georgia Institute of Technology黎邵鹏博士和Kianpoor Kalkhajeh Yusef博士写作,特此致谢。| [1] | Elton C. The Ecology of Invasions by Plants and Animals. London: Methuen, 1958: 50-50. |
| [2] | Levine J M, Adler P B, Yelenik S G. A meta-analysis of biotic resistance to exotic plant invasions. Ecology Letters, 2004, 7(10): 975–989. DOI:10.1111/ele.2004.7.issue-10 |
| [3] | Mallon C A, van Elsas J D, Salles J F. Microbial invasions:the process, patterns, and mechanisms. Trends in Microbiology, 2015, 23(11): 719–729. DOI:10.1016/j.tim.2015.07.013 |
| [4] | Sax D F, Stachowicz J J, Brown J H, Bruno J F, Dawson M N, Gaines S D, Grosberg R K, Hastings A, Holt R D, Mayfield M M, O'Connor M I, Rice W R. Ecological and evolutionary insights from species invasions. Trends in Ecology & Evolution, 2007, 22(9): 465–471. |
| [5] | 万方浩, 刘万学, 郭建英, 强胜, 李保平, 王进军, 杨国庆, 牛红榜, 桂富荣, 黄文坤, 蒋智林, 王文琪. 外来植物紫茎泽兰的入侵机理与控制策略研究进展. 中国科学:生命科学, 2011, 41(1): 13–21. |
| [6] | Robinson G R, Quinn J F, Stanton M L. Invasibility of experimental habitat islands in a California winter annual grassland. Ecology, 1995, 76(3): 786–794. DOI:10.2307/1939344 |
| [7] | 郑景明, 马克平. 植物群落多样性与可入侵性关系研究进展. 应用生态学报, 2006, 17(7): 1338–1343. DOI:10.3321/j.issn:1001-9332.2006.07.036 |
| [8] | Kennedy T A, Naeem S, Howe K M, Knops J M H, Tilman D, Reich P. Biodiversity as a barrier to ecological invasion. Nature, 2002, 417(6889): 636–638. DOI:10.1038/nature00776 |
| [9] | Jiang L, Morin P J. Productivity gradients cause positive diversity-invasibility relationships in microbial communities. Ecology Letters, 2004, 7(11): 1047–1057. DOI:10.1111/ele.2004.7.issue-11 |
| [10] | Kolar C S, Lodge D M. Progress in invasion biology:predicting invaders. Trends in Ecology & Evolution, 2001, 16(4): 199–204. |
| [11] | 李博, 陈家宽. 生物入侵生态学:成就与挑战. 世界科技研究与发展, 2002, 24(2): 26–36. DOI:10.3969/j.issn.1006-6055.2002.02.005 |
| [12] | Nawrot R, Chattopadhyay D, Zuschin M. What guides invasion success? Ecological correlates of arrival, establishment and spread of Red Sea bivalves in the Mediterranean Sea. Diversity & Distributions, 2015, 21(9): 1075–1086. |
| [13] | Ricciardi A, Cohen J. The invasiveness of an introduced species does not predict its impact. Biological Invasions, 2007, 9(3): 309–315. DOI:10.1007/s10530-006-9034-4 |
| [14] | Wei Z, Yang T J, Friman V P, Xu Y C, Shen Q R, Jousset A. Trophic network architecture of root-associated bacterial communities determines pathogen invasion and plant health. Nature Communications, 2015, 6: 8413. DOI:10.1038/ncomms9413 |
| [15] | Keddy P A. Assembly and response rules:two goals for predictive community ecology. Journal of Vegetation Science, 1992, 3(2): 157–164. DOI:10.2307/3235676 |
| [16] | 唐志尧, 王志恒, 方精云. 生物多样性分布格局的地史成因假说. 生物多样性, 2009, 17(6): 635–643. |
| [17] | Naeem S, Knops J M H, Tilman D, Howe K M, Kennedy T, Gale S. Plant diversity increases resistance to invasion in the absence of covarying extrinsic factors. Oikos, 2000, 91(1): 97–108. DOI:10.1034/j.1600-0706.2000.910108.x |
| [18] | Xun W B, Huang T, Zhao J, Ran W, Wang B R, Shen Q R, Zhang R F. Environmental conditions rather than microbial inoculum composition determine the bacterial composition, microbial biomass and enzymatic activity of reconstructed soil microbial communities. Soil Biology & Biochemistry, 2015, 90: 10–18. |
| [19] | FAO-UNESCO. Soil Map of the World 1: 5000000. Paris: UNESCO, 1974: 1-1. |
| [20] | Tan S Y, Yang C L, Mei X L, Shen S Y, Raza W, Shen Q R, Xu Y C. The effect of organic acids from tomato root exudates on rhizosphere colonization of Bacillus amyloliquefaciens T-5. Applied Soil Ecology, 2003, 64: 15–22. |
| [21] | Allison S D, Lu Y, Weihe C, Goulden M L, Martiny A C, Treseder K K, Martiny J B H. Microbial abundance and composition influence litter decomposition response to environmental change. Ecology, 2013, 94(3): 714–725. DOI:10.1890/12-1243.1 |
| [22] | Rønn R, Ekelund F, Christensen S. Optimizing soil extract and broth media for MPN enumeration of naked amoebae and heterotrophic flagellates in soil. Pedobiologia, 1995, 39(1): 10–19. |
| [23] | Frostegård Å, Tunlid A, Bååth E. Use and misuse of PLFA measurements in soils. Soil Biology and Biochemistry, 2011, 43(8): 1621–1625. DOI:10.1016/j.soilbio.2010.11.021 |
| [24] | Widmer F, Fließbach A, Laczkó E, Schulze-Aurich J, Zeyer J. Assessing soil biological characteristics:a comparison of bulk soil community DNA-, PLFA-, and BiologTM-analyses. Soil Biology & Biochemistry, 2001, 33(7/8): 1029–1036. |
| [25] | 王光华, 刘俊杰, 齐晓宁, 金剑, 王洋, 刘晓冰. Biolog和PCR-DGGE技术解析施肥对德惠黑土细菌群落结构和功能的影响. 生态学报, 2008, 28(1): 220–226. |
| [26] | 鲁如坤. 土壤农业化学分析方法. 北京: 中国农业科技出版社, 2000. |
| [27] | Ma C, Liu M Q, Wang H, Chen C Y, Fan W Q, Griffiths B, Li H X. Resource utilization capability of bacteria predicts their invasion potential in soil. Soil Biology & Biochemistry, 2015, 81: 287–290. |
| [28] | Yao Z Y, Wang H Z, Wu L S, Wu J J, Brookes P C, Xu J M. Interaction between the microbial community and Invading Escherichia coli O157:H7 in soils from vegetable fields. Applied & Environmental Microbiology, 2014, 80(1): 70–76. |
| [29] | 贺纪正, 王军涛. 土壤微生物群落构建理论与时空演变特征. 生态学报, 2015, 35(20): 6575–6583. |
| [30] | Gerhardt F, Collinge S K. Abiotic constraints eclipse biotic resistance in determining invasibility along experimental vernal pool gradients. Ecological Applications, 2007, 17(3): 922–933. DOI:10.1890/05-1146 |
| [31] | 郝瑞军, 李忠佩, 车玉萍. 好气和淹水处理间苏南水稻土有机碳矿化量差异的变化特征. 中国农业科学, 2010, 43(6): 1164–1172. |
| [32] | 朱平, 陈仁升, 宋耀选, 韩春坛, 刘光琇, 陈拓, 张威. 祁连山中部4种典型植被类型土壤细菌群落结构差异. 生态学报, 2017, 37(10): 3505–3514. |
| [33] | 郑学博, 樊剑波, 崔键, 徐磊, 祝振球, 周静, 何园球. 沼液还田对旱地红壤微生物群落代谢与多样性的影响. 生态学报, 2016, 36(18): 5865–5875. |
| [34] | Hartmann M, Frey B, Mayer J, Mäder P, Widmer F. Distinct soil microbial diversity under long-term organic and conventional farming. The ISME Journal, 2015, 9(5): 1177–1194. DOI:10.1038/ismej.2014.210 |
| [35] | 陈小云, 刘满强, 胡锋, 毛小芳, 李辉信. 根际微型土壤动物——原生动物和线虫的生态功能. 生态学报, 2007, 27(8): 3132–3143. DOI:10.3321/j.issn:1000-0933.2007.08.006 |
| [36] | Wilson M, Lindow S E. Inoculum density-dependent mortality and colonization of the phyllosphere by Pseudomonas syringae. Applied & Environmental Microbiology, 1994, 60(7): 2232–2237. |
| [37] | Liu M Q, Bjørnlund L, Rønn R, Christensen S, Ekelund F. Disturbance promotes non-indigenous bacterial invasion in soil microcosms:analysis of the roles of resource availability and community structure. PLoS One, 2012, 7(10): e45306. DOI:10.1371/journal.pone.0045306 |
| [38] | Mallon C A, Poly F, Roux X L, Marring I, van Elsas J D, Falles Salles J F. Resource pulses can alleviate the biodiversity-invasion relationship in soil microbial communities. Ecology, 2015, 96(4): 915–926. DOI:10.1890/14-1001.1 |
| [39] | Jousset A, Scheu S, Bonkowski M. Secondary metabolite production facilitates establishment of rhizobacteria by reducing both protozoan predation and the competitive effects of indigenous bacteria. Functional Ecology, 2008, 22(4): 714–719. DOI:10.1111/fec.2008.22.issue-4 |
| [40] | Eisenhauer N, Schulz W, Scheu S, Jousset A. Niche dimensionality links biodiversity and invasibility of microbial communities. Functional Ecology, 2013, 27(1): 282–288. DOI:10.1111/fec.2013.27.issue-1 |
 2018, Vol. 38
2018, Vol. 38