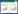文章信息
- 邓晓娟, 闫兴富, 刘建利, 刘培贵.
- DENG Xiaojuan, YAN Xingfu, LIU Jianli, LIU Peigui.
- 印度块菌-云南松菌根际土壤细菌的种群组成和群落结构
- Study of bacterial population composition and structure of Tuber indicum×Pinus yunnanensis ectomycorrhizosphere
- 生态学报. 2018, 38(19): 7066-7074
- Acta Ecologica Sinica. 2018, 38(19): 7066-7074
- http://dx.doi.org/10.5846/stxb201707261351
-
文章历史
- 收稿日期: 2017-07-26
- 网络出版日期: 2018-06-20
2. 中国科学院昆明植物研究所生物多样性与生物地理学重点实验室, 昆明 650201
2. Key Laboratory of Biodiversity and Biogeography, Kunming Institute of Botany, Chinese Academy of Sciences, Kunming 650201, China
德国微生物学家Lorenz Hiltner于1904年首次提出了根际(rhizosphere)的概念, 根系的表面以及受根系直接影响的土壤区域称为根际, 根际内的微生物数量和种类远远高于根际外的微生物数量和种类[1]。植物根系分泌的有机酸、碳水化合物等代谢产物, 为微生物的生长和繁殖提供了有益条件, 因此在植物的根际里定殖有很多生命活动旺盛的土壤微生物, 使根际成为植物、土壤和微生物及其环境相互作用的中心, 在植物和土壤之间构成了一个活跃的物质和能量交换界面[2]。根际里定殖的微生物称为根际微生物, 包括细菌、真菌、放线菌、藻类和原生动物, 根际微生物的种类和数量远远高于非根际微生物[3-4]。
大多数陆生植物的根系都会和菌根菌形成菌根, 这种互惠共生关系在自然界中十分普遍[5]。植物的根和菌根菌在土壤中形成了一个完整统一的生态系统, 因此在原有根际概念的基础上, 菌根际(mycorrhizosphere)的概念就普遍使用, 包括了菌根菌和菌根共生体[6]。菌根菌将其从共生植物处获得的碳源转化成海藻糖(trehalose), 这种双糖能够选择菌根根际中特定的细菌类群, 在菌根菌和根际细菌相互作用的过程中扮演了很重要的角色[7-8]。同时, 菌根改变了植物根的代谢功能, 菌根菌的存在使菌根根际的微生物组成和根际的微生物组成具有很大的差别[9-10]。菌根对其周围微生物群落进行选择和调节, 使其更有利于菌根本身的形成和发展[11]。真菌能否顺利的侵染植物的根并形成菌根, 不仅取决于其周围的非生物因素, 如土壤pH值、肥力、湿度和温度, 也取决于包括根际微生物在内的生物因素[12]。
块菌在商业贸易领域被称为松露, 是一种地下菌根真菌, 因含有丰富的氨基酸和具有独特的香味儿而享誉世界。块菌在欧洲是一种奢侈顶级食材, 被称为“世界珍味之王”, 同鱼子酱、鹅肝酱并称为法式三大美食。块菌在市场上最为常见的种类有三种, 包括产于欧洲的黑孢块菌(Tuber melanosporum)和意大利白块菌(T. magnatum), 以及产于亚洲的印度块菌(T. indicum)。印度块菌主产于我国的云南、四川和西藏, 每年以出口创汇为主, 是我国西南地区农民收入的重要来源。一方面, 印度块菌产区农民在经济利益的驱使下, 在印度块菌成熟前开始采挖, 且采挖方式落后粗暴, 导致自然块菌资源迅速耗竭, 生境受到严重的破坏, 印度块菌资源面临着濒危, 部分商业化采集区域已濒临绝迹。另一方面, 印度块菌的纯培养困难, 加剧了印度块菌自然资源所受到的压力[13-14]。为了缓解这种压力, 相关研究人员一直在尝试人工合成块菌菌根进而实现人工栽培。中国科学院昆明植物研究所科研人员经过十多年的努力, 块菌种植园已成功产出了印度块菌子实体(http://www.cas.cn/ky/kyjz/201212/t20121220_3725060.shtml)。在块菌菌根人工合成过程中, pH值、基质配比等非生物因素的影响已经有很多研究报道[15-17], 而微生物对于块菌菌根形成的影响鲜有报道。在菌根际的细菌类群中, 有些类群可以促进菌根的形成, 被称之为菌根促生细菌(plant grow-promoting rhizobacteria, PGPR), 研究根际细菌种群组成和群落结构是探索块菌菌根促生细菌的前提条件[18-21]。到目前为止, 关于印度块菌菌根际细菌的种群特征还未有报道。
在分子生物学技术被运用于环境微生物的研究之前, 人们对微生物的认识基本靠传统的分离和纯培养技术。尽管对于复杂环境中微生物的纯培养技术已经有了很大的发展和改进[22], 可培养的微生物还是不能很好的反映土壤微生物群落的多样性。通过传统分离方法可以获得并进行过研究的微生物种群数量只占自然界微生物总数的0.1%—1%[23]。高通量测序技术(high-throughput sequencing)又称为“下一代”测序技术, 能以一次并行对几万条至几百万条DNA分子进行序列测定和一般读长较短为标志, 能够有效探测土壤微生物多样性[24-25]。本文通过传统培养的方法, 以及高通量测序技术, 对印度块菌-云南松菌根根际细菌的种群组成和群落结构进行了研究, 对其根际细菌多样性及优势类群进行了探讨。
1 研究材料和方法 1.1 样品位置和采集云南省楚雄市是印度块菌主要的出产地, 本研究选取云南省楚雄州姚安县左门乡后山的纯云南松林为样品采集地, 面积约1000 m2, 海拔2221—2330 m, 土壤为偏碱性的石灰质土壤(pH介于6—8范围), 含碳量22.12—89.22 g/kg, 平均含钙量17.3 g/kg。
在采样地发现块菌后, 取出子囊果, 编号后放入自封袋中保存。用一把锋利且较长的小刀轻轻拨开块菌子囊果下方的泥土, 发现菌根后, 以其为中心, 取半径1 cm以内的所有土样, 放入15 mL离心管中保存, 注明采集号, 带回实验室处理。共采集到6份印度块菌×云南松菌根际土壤, 选取其中3份为本次实验样品。
1.2 块菌菌根际土壤可培养细菌的分离纯化和鉴定 1.2.1 样品的处理称取印度块菌×云南松菌根际土壤样品0.5 g, 加入5 mL灭菌的生理液(NaCl 0.85%)稀释成10-1菌液。同时制备10-2、10-3、10-4、10-5和10-6稀释液, 用以细菌的分离培养。
1.2.2 细菌的分离纯化分别在10-4、10-5和10-6的稀释浓度下培养细菌, TSA培养基生化培养箱28℃条件下培养。每个浓度的平板设置3个重复。
1.2.3 可培养细菌的鉴定和分析对分离培养的单菌落进行扩大培养, 挑取菌落进行鉴定。每份样品挑取的菌落数绘制物种累积曲线, 当挑选的菌落数增加而种类不变时, 不在增加挑选样本的数量。对细菌的单菌落提取总DNA, 扩增16S rDNA(通用引物27f/1378r)序列(扩增程序常规)。将扩增片段送至生工生物工程(上海)股份有限公司进行测序。测序结果通过NCBI上的Blast功能进行比对, 当序列的相似度大于97%时被归属为同一个分类操作单元(OTU)。使用SeqMan(DNASTAR Package)对序列进行编辑, 采用Clustal 3 version 1.81[26]进行多序列自动比对, 并通过BioEdit Version 5.0.9[27]手动调整矩阵。系统发育分析采用Tamura [28]的方法。
1.3 菌根际土壤细菌基因组总DNA的提取和分析 1.3.1 基因组总DNA的提取取0.5g左右的菌根际土壤, 使用MP公司的FastDNA Spin Kit for Soil(116560-200)试剂盒, 按照其说明书完成对菌根际土壤微生物基因组总DNA提取。
1.3.2 16S rRNA V1—V3区扩增和高通量测序扩增体系:5 μL 5×FastPfu缓冲液, 2.5 μL 2.5×10-3 mol/L dNTPs, 1.0 μL 0.5×10-5mol/L 27F引物(5′-AGAGTTTGATCCTGGCTCAG-3′), 1.0 μL 0.5×10-5mol/L 533R引物(5′-TTACCGCGGCTGCTGGCAC-3′), 10 ng模板以及0.5 μL Fast Pfu Polymerase, 最后加ddH2O到25 μL。获得的PCR产物进行1.5%琼脂糖凝胶电泳检测, 将扩增产物送至上海美吉生物医药科技有限公司, 利用罗氏454Titanium测序仪完成高通量测序序列分析。
1.3.3 测序数据分析在对序列进行分析中, 去除不含样品信息的序列。过长序列末端的质量会降低, 在分析时去除序列尾部的低质量序列。序列中含有模糊碱基(ambiguous)、单碱基高重复区(homologous)以及长度过短的序列, 将这些序列纳入分析会降低分析质量, 因此修剪、去除此部分序列, 得到供精准分析的优化序列, 并将序列提交至NCBI数据库进行分类操作单元(Operational Taxonomic Units, OTU)分析。生物信息分析中, 测序得到的每一条序列来自一个菌。要了解一个样品测序结果中的菌种、种属等信息, 需要对序列进行归类操作。通过归类操作, 将序列相似性达到97%归为一个OTU, 使用最大似然法聚类分析OTU。
2 研究结果和分析 2.1 菌根际可培养细菌多样性在10-5和10-6的稀释浓度下, 细菌在TSA培养基能够形成清晰独立的菌落(图 1), 选择菌落数在20—200的培养皿, 挑选细菌纯化培养, 总计得到菌根际土壤细菌796株。
|
| 图 1 印度块菌×云南松菌根际可培养细菌 Fig. 1 Culturable bacteria from Tuber indicum × Pinus yunnanensis mycorrhizosphere |
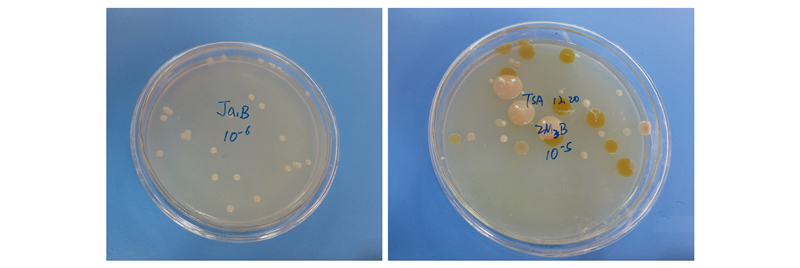
对可培养细菌菌落进行测序分析, 根据物种累积曲线(图 2), 126株细菌被测序, 其中76条序列(代表了61个OTUs)被用于本实验分析。通过比对, 在NCBI上下载39条序列, 总计115条细菌16S rDNA序列用于此次分析。排序后得到了长度为1611 bp的序列矩阵, 其中580个为信息位点(parsimony-informative), 129个非信息位点(parsimony-informative)。启发式搜索的最大简约性分析产生了31棵同等简约树, 步长为2035, 一致性指数(CI)为0.650, 留存指数(RI)为0.812。

|
| 图 2 可培养细菌菌落数量和OTUs之间的物种累积曲线 Fig. 2 Species accumulation curves between OTUs and numbers of culturable bacterial clone in mycorrhizosphere |
基于根际可培养细菌16S rDNA序列构建的最大简约数表明(图 3), 以2条大肠杆菌(Escherichia coli)的16S rDNA序列为外类群(Clade 4), 印度块菌×云南松菌根际细菌的76条序列很好的聚在了3个Clades里。Clade 1包含了下载的27条隶属于丙型变形菌门假單胞菌属(Pseudomonas)序列, 66条(代表了51个OTUs)本次实验获得的序列。Clade 2包含了下载的6条隶属于乙型变形菌门不动细菌属(Acinetobacter)序列, 6条(代表了6个OTUs)本次实验获得的序列。Clade 3包含了下载的4条隶属于放线菌门链霉菌属(Streptomyces)序列, 4条(代表了4个OTUs)本次实验获得的序列。
研究结果表明, 印度块菌×云南松菌根际可培养细菌分属于3个属的61个OTUs, 其中假单胞菌属(Pseudomonas)序列占83.6%, 不动杆菌属(Acinetobacter)序列占9.8%, 链霉菌属(Streptomyces)序列占6.6%。印度块菌×云南松菌根际可培养细菌具有较低的多样性, 其中假单胞菌是其绝对优势类群。

|
| 图 3 基于根际细菌16S rDNA序列构建的最大简约树中的一棵分支上的数字为≥50%的靴带值 Fig. 3 One of most parsimonious trees based on the analysis of 16S rDNA sequences in mycorrhizosphere. Numbers above branches indicate bootstrap support above 50% |
提取印度块菌×云南松菌根际土壤细菌总DNA, 扩增其16S rRNA V1-V3区并高通量测序, 总计得到8937条序列2073个OTUs。根据对其测序数量和OTUs数量之间的关系进行Rarefaction曲线分析(图 4), 当测序数量在8937条, 相似度设为97%时, OTUs的数量还在增加, 但曲线趋于缓和, 测序数量可以满足分析要求。
|
| 图 4 菌根际土壤细菌可变区序列的数量和OTUs之间的Rarefaction曲线分析 Fig. 4 Rarefaction curve between OTUs and sequences of bacteria in mycorrhizosphere |
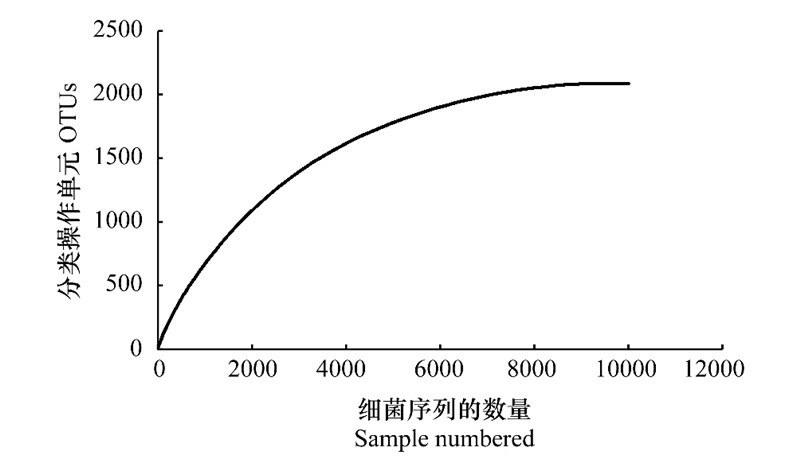
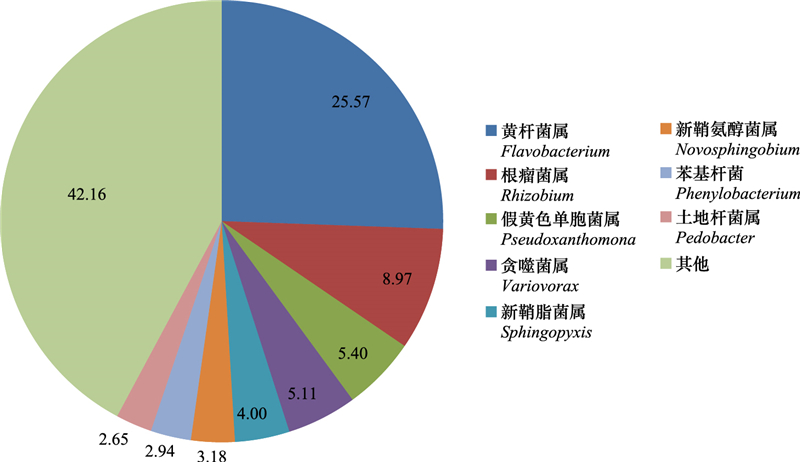
根际细菌的2073个OTUs分属于细菌的20门198属。在门的级别上, 变形菌门(Proteobacteria)细菌序列占全部序列的33.82%, 放线菌门(Actinobacteria)细菌序列占全部序列的21.95%, 酸杆菌门(Acidobacteria)细菌序列占全部序列的10.13%, 隶属于该3门的细菌OTUs占总OTUs的65.9%(图 5)。在属的级别上, 黄杆菌属(Flavobacterium)细菌序列占全部序列的25.57%, 根瘤菌属(Rhizobium)细菌序列占全部序列的8.97%, 假黄色单胞菌属(Pseudoxanthomona)细菌序列占全部序列的5.4%, 贪噬菌属(Variovorax)细菌序列占全部序列的5.11%, 新鞘脂菌属(Sphingopyxis)细菌序列占全部序列的4%, 新鞘氨醇菌属(Novosphingobium)序列占3.18%, 苯基杆菌属(Phenylobacterium)序列占2.94%, 土地杆菌属(Pedobacter)序列占2.65%, 其余190个属序列占42.16%(图 6)。在OTUs的分布上, 变形菌门、放线菌门和酸杆菌门为印度块菌×云南松菌根际土壤细菌的优势类群, 黄杆菌属、根瘤菌属和假黄色单胞菌属为其优势属。
|
| 图 5 根际细菌OTUs在不同门的分布 Fig. 5 OTUs distribution in bacterial phylum |
|
| 图 6 根际细菌OTUs在不同属的分布 Fig. 6 OTUs distribution in bacterial genus |
研究结果表明, 印度块菌-云南松菌根际土壤细菌类群具有较高的多样性, 物种种类丰富, 优势菌群集中。变形菌门、放线菌门和酸杆菌门细菌是印度块菌-云南松菌根际土壤细菌的优势类群, 黄杆菌属、根瘤菌属和假黄色单胞菌属细菌是印度块菌-云南松菌根际土壤细菌。
3 讨论块菌的代谢产物影响其周围的土壤理化性质以及共生植物根部微生物的类群, 使得块菌生长并形成子囊果的地方, 大部分草本植物枯死, 形成无草或少草的火烧区式的“菌塘”。菌塘土壤微生物种群和块菌之间的关系紧密而复杂。土壤微生物通过影响真菌的生态适应性, 从而影响真菌的生长、分布及子囊果的形成和气味成分的形成[29]。菌塘内和菌塘外的土壤细菌类群的结构不同, 黑孢块菌的菌塘内, 隶属于厚壁菌门、放线菌门和黄杆菌科的细菌为优势类群, 其种类和数量远远高于菌塘外土壤细菌的种类和数量[30]。
块菌不仅影响菌塘内的细菌种群结构, 同时也会影响菌根际细菌的群落结构。在过去十年, 块菌菌根合成的研究技术已经取得了长远的进步, 获得诸多组合式样的菌根合成案例[15-17]。随着菌根技术的发展, 人们越来越认识到, 除了非生物因素的影响, 根际促生细菌(PGPR)对菌根的合成是否成功扮演着不可缺乏的角色[31]。随着研究深入, 在人工合成菌根的过程中, 添加根际促生细菌成为一种现代且有效的生物技术手段[32]。尽管很多根际促生细菌的相关研究已经报道, 但块菌菌根促生细菌的相关研究鲜有报道[32-33], 关于印度块菌的菌根促生细菌研究还未见报道。本研究首次揭示了印度块菌×云南松菌根际细菌的种群组成和结构特征, 为研究印度块菌的菌根促栖菌奠定了基础。
根据本次研究结果, 印度块菌×云南松菌根际可培养细菌以假单胞菌为绝对优势类群。假单胞菌为革兰氏阴性细菌, 能在植物根际土壤中大量增殖, 许多菌株对植物有抑制病害、促进生长的作用, 其中荧光假单胞菌(P. fluorescens)成为近几十年来研究报道最多、最具应用价值前景的一类生防菌[34]。假单胞菌也是沙漠块菌(Terfezia claveryi)菌根际土壤可培养细菌的优势类群, 且其中孟氏假单胞菌(P. mandelii)明显的提高了菌根的侵染率, 被认为是一种良好的菌根促生细菌被用于菌根合成生产中[35]。假单胞菌除在块菌菌根际土壤里是优势类群, 也是波氏块菌(T. borchii)菌根根尖以及意大利白块菌和沙漠块菌(Te. boudieri)子囊果里的优势类群[36-38]。假单胞菌是印度块菌×云南松菌根际可培养细菌的优势类群, 在印度块菌的菌根促生细菌的研究时, 假单胞菌为最理想的候选类群。
采用高通量测序技术, 扩增细菌16S rRNA V1—V3区进行测序分析, 印度块菌×云南松菌根际土壤细菌类群具有很高的多样性, 其中变形菌门、放线菌门和酸杆菌门是其优势类群。同样采用高通量测序技术研究发现, 夏块菌(T. aestivum)的菌根际土壤细菌同样是以放线菌门和变形菌门为优势类群[20]。放线菌门、变形菌门、拟杆菌门细菌也是黑孢块菌菌根际土壤细菌的优势类群[39]。在黑孢块菌的子囊果成熟过程中, 菌根际微生物虽有动态变化, 但都是以α-变形菌门和γ-变形菌门细菌为优势类群[40]。以上研究所取土壤类型、块菌和共生宿主植物都不一致, 但菌根际土壤细菌优势类群具有相似性, 变形菌门和放线菌门细菌为菌根际土壤细菌优势类群。本研究结果为印度块菌的菌根合成、种植园精细管理以及根际土壤微生物功能的研究奠定了基础, 明确了方向。
当研究菌根际土壤微生物多样性时, 相比新一代的高通量测序的方法, 传统平板培养的方法会错过很多类群和重要信息。但是, 对于根际细菌的功能研究, 以及细菌对菌根真菌的促生作用的研究, 都离不开对细菌的传统平板培养方法。因此, 本实验不仅采用高通量测序的方法, 全面揭示了印度块菌菌根际土壤微生物的种群组成, 同时采用传统的平板培养的方法, 对其可培养细菌进行了分离并鉴定, 为菌根际细菌的功能研究, 以及菌根促生菌的筛选提供重要数据。
致谢: 中国科学院昆明植物研究所刘培贵研究员给予支持, 山东省农业科学院乔鹏博士帮助样品采集, 特此致谢。| [1] | Hiltner L. über neuere erfahrungen und probleme auf dem gebiet der bodenbakteriologie und unter besonderer berücksichtigung der gründüngung und brache. Arb der Dtsch Landwirtsch Gesellschaft, 1904, 98: 59–78. |
| [2] | Hartmann A, Schmid M, Wenzel W, Hinsinger P. Rhizosphere 2004-perspectives and challenges-a tribute to Lorenz hiltner. Munich, Germany: GSF-National Research Center for Environment and Health, 2005. |
| [3] | Foster R C. The ultrastructure of the rhizoplane and rhizosphere. Annual Review of Phytopathology, 1986, 24: 211–234. DOI:10.1146/annurev.py.24.090186.001235 |
| [4] | Chen L, Brookes P C, Xu J M, Zhang J B, Zhang C Z, Zhou X Y, Luo Y. Structural and functional differentiation of the root-associated bacterial microbiomes of perennial ryegrass. Soil Biology and Biochemistry, 2016, 98: 1–10. DOI:10.1016/j.soilbio.2016.04.004 |
| [5] | Williams A V, Nevill P G, Krauss S L. Next generation restoration genetics:applications and opportunities. Trends in Plant Science, 2014, 19(8): 529–537. DOI:10.1016/j.tplants.2014.03.011 |
| [6] | Monfort-Salvador I, García-Montero L G, Grande M A. Impact of calcium associated to calcareous amendments on ectomycorrhizae in forests:a review. Journal of Soil Science and Plant Nutrition, 2015, 15(1): 217–231. |
| [7] | Izumi H, Anderson I C, Alexander I J, Killham K, Moore E R. Endobacteria in some ectomycorrhiza of Scots pine (Pinus sylvestris). FEMS Microbiology Ecology, 2006, 56(1): 34–43. DOI:10.1111/fem.2006.56.issue-1 |
| [8] | Colin Y, Nicolitch O, Van Nostrand J D, Zhou J Z, Turpault M P, Uroz S. Taxonomic and functional shifts in the beech rhizosphere microbiome across a natural soil toposequence. Scientific Reports, 2017, 7(1): 9604. DOI:10.1038/s41598-017-07639-1 |
| [9] | Wallander H. Uptake of P from apatite by Pinus sylvestris seedlings colonised by different ectomycorrhizal fungi. Plant and Soil, 2012, 218(1/2): 249–256. |
| [10] | Rillig M C, Aguilar-Trigueros C A, Bergmann J, Verbruggen E, Veresoglou S D, Lehmann A. Plant root and mycorrhizal fungal traits for understanding soil aggregation. New Phytologist, 2014, 205(4): 1385–1388. |
| [11] | Thioulouse J, Prin Y, Duponnois R. Multivariate analyses in soil microbial ecology:a new paradigm. Environmental and Ecological Statistics, 2012, 19(4): 499–520. DOI:10.1007/s10651-012-0198-z |
| [12] | Soudzilovskaia N A, Douma J C, Akhmetzhanova A A, Van Bodegom P M, Cornwell W K, Moens E J, Treseder K K, Tibbett M, Wang Y P, Cornelissen J H C. Global patterns of plant root colonization intensity by mycorrhizal fungi explained by climate and soil chemistry. Global Ecology and Biogeography, 2015, 24(3): 371–382. DOI:10.1111/geb.2015.24.issue-3 |
| [13] | Kües U, Martin F. On the road to understanding truffles in the underground. Fungal Genetics and Biology, 2011, 48(6): 555–560. DOI:10.1016/j.fgb.2011.02.002 |
| [14] | Tang Y J, Liu R S, Li H M. Current progress on truffle submerged fermentation:a promising alternative to its fruiting bodies. Applied Microbiology and Biotechnology, 2015, 99(5): 2041–2053. DOI:10.1007/s00253-015-6379-6 |
| [15] | Wan S P, Yu F Q, Tang L, Wang R, Wang Y, Liu P G, Wang X H, Zheng Y. Ectomycorrhizae of Tuber huidongense and T. liyuanum with Castanea mollissima and Pinus armandii. Mycorrhiza, 2016, 26(3): 249–256. DOI:10.1007/s00572-015-0663-2 |
| [16] | Wei J, Yan W, Agerer R. Three Tuber ectomycorrhizae associated with Chinese Pine in North China-morpho-anatomical and molecular-phylogenetic analyses. Nova Hedwigia, 2014, 98(3/4): 409–424. |
| [17] | Geng L Y, Wang X H, Yu F Q, Deng X J, Tian X F, Shi X F, Xie X D, Liu P G, Shen Y Y. Mycorrhizal synthesis of Tuber indicum with two indigenous hosts, Castanea mollissima and Pinus armandii. Mycorrhiza, 2009, 19(7): 461–467. DOI:10.1007/s00572-009-0247-0 |
| [18] | Hashidoko Y, Itoh E, Yokota K, Yoshida T, Tahara E. Characterization of five phyllosphere bacteria isolated from Rosa Rugosa leaves, and their phenotypic and metabolic properties. Bioscience, Biotechnology, and Biochemistry, 2014, 66(11): 2474–2478. |
| [19] | Splivallo R, Deveau A, Valdez N, Kirchhoff N, Frey-Klett P, Karlovsky P. Bacteria associated with truffle-fruiting bodies contribute to truffle aroma. Environmental Microbiology, 2015, 17(8): 2647–2660. DOI:10.1111/1462-2920.12521 |
| [20] | Benucci G M N, Bonito G M. The truffle microbiome:species and geography effects on bacteria associated with fruiting bodies of hypogeous pezizales. Microbial Ecology, 2016, 72(1): 4–8. DOI:10.1007/s00248-016-0755-3 |
| [21] | Gryndler M, Soukupová L, Hršelová H, Gryndlerová H, Borovičika J, Streiblová E, Jansa J. A quest for indigenous truffle helper prokaryotes. Environment Microbiology Reports, 2013, 5(3): 346–352. DOI:10.1111/1758-2229.12014 |
| [22] | Dorofeev A G, Grigor'eva N V, Kozlov M N, Kevbrina M V, Aseeva V G, Nikolav Y A. Approaches to cultivation of "nonculturable" bacteria:cyclic cultures. Microbiology, 2014, 83(5): 450–461. DOI:10.1134/S0026261714050087 |
| [23] | Brock T D. The study of microorganisms in situ:progress and problems. Society for General Microbiology, 1987, 41: 1–17. |
| [24] | Mavromatis K, Land M L, Brettin T S, Quest D J, Copeland A, Clum A, Goodwin L, Woyke T, Lapidus A, Klenk H P, Cottingham R W, Kyrpides N C. The fast changing landscape of sequencing technologies and their impact on microbial genome assemblies and annotation. PLoS One, 2012, 7(12): e48837. DOI:10.1371/journal.pone.0048837 |
| [25] | Siqueira J F, Fouad A F Jr, Rôças I N. Pyrosequencing as a tool for better understanding of human microbiomes. Journal of Oral Microbiology, 2012, 4: 10743. DOI:10.3402/jom.v4i0.10743 |
| [26] | Thompson J D, Gibson T J, Plewniak F, Jeanmougin F, Higgins D G. The Clustal_X Windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Research, 1997, 25(24): 4876–4882. DOI:10.1093/nar/25.24.4876 |
| [27] | Hall T A. BioEdit:a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucleic Acids Symposium Series, 1999, 41(2): 95–98. |
| [28] | Tamura K, Dudley J, Nei M, Kumar S. MEGA4:Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0. Molecular Biology and Evolution, 2007, 24(8): 1596–1599. DOI:10.1093/molbev/msm092 |
| [29] | Streiblová E, Gryndlerová H, Gryndler M. Truffle brûlé:an efficient fungal life strategy. FEMS Microbiology Ecology, 2012, 80(1): 1–8. DOI:10.1111/j.1574-6941.2011.01283.x |
| [30] | Mello A, Ding G C, Piceno Y M, Napoli C, Tom L M, DeSantis T Z, Andersen G L, Smalla K, Bonfante P. Truffle brûlés have an impact on the diversity of soil bacterial communities. PLoS One, 2013, 8(4): e61945. DOI:10.1371/journal.pone.0061945 |
| [31] | Kamei A, Dolai A K, Kamei A. Role of hydrogen cyanide secondary metabolite of plant growth promoting rhizobacteria as biopesticides of weeds. Global. Global Journal of Science Frontier Research:D, 2014, 14(6): 109–112. |
| [32] | Beneduzi A, Ambrosini A, Passaglia L M P. Plant Growth-Promoting Rhizobacteria (PGPR):their potential as antagonists and biocontrol agents. Genetics and Molecular Biology, 2012, 35(4 Suppl): 1044–1051. |
| [33] | Goudjal Y, Zamoum M, Meklat A, Sabaou N, Mathieu F, Zitouni A. Plant-growth-promoting potential of endosymbiotic actinobacteria isolated from sand truffles (Terfezia leonis Tul.) of the Algerian Sahara. Annals of Microbiology, 2016, 66(1): 91–100. DOI:10.1007/s13213-015-1085-2 |
| [34] | Yan W R, Zhao T C, Xiao T B, Xiao M, Zhao Z X, Chen M C. Applications of biocontrol bacterial in plant disease control. Genomics and Applied Biology, 2013, 32(4): 533–539. |
| [35] | Navarro-Ródenas A, Berná L M, Lozano-Carrillo C, Andrino A, Morte A. Beneficial native bacteria improve survival and mycorrhization of desert truffle mycorrhizal plants in nursery conditions. Mycorrhiza, 2016, 26(7): 769–779. DOI:10.1007/s00572-016-0711-6 |
| [36] | Picceri G G, Leonardi P, Iotti M, Gallo M, Baldi F, Zambonelli A, Amicucci A, Vallorani L, Piccoli G, Ciccimarra G, Arshakyan M, Burattini S, Falcieri E, Chiarantini L. Bacteria-produced ferric exopolysaccharide nanoparticles as iron delivery system for truffles (Tuber borchii). Applied Microbiology and Biotechnology, 2018, 102(3): 1429–1441. DOI:10.1007/s00253-017-8615-8 |
| [37] | Barbieri E, Guidi C, Bertaux J, Frey-Klett P, Garbaye J, Ceccaroli P, Saltarelli R, Zambonelli A, Stocchi V. Occurrence and diversity of bacterial communities in Tuber magnatum during truffle maturation. Environment Microbiology, 2007, 9(9): 2234–2246. DOI:10.1111/emi.2007.9.issue-9 |
| [38] | Dib-Bellahouel S, Fortas Z. Activity of the desert truffle Terfezia boudieri Chatin, against associated soil microflora. African Journal of Microbiology Research, 2014, 8(32): 3008–3016. DOI:10.5897/AJMR |
| [39] | Antony-Babu S, Deveau A, Van Nostrand J D, Zhou J, Le Tacon F, Robin C, Frey-Klett P, Uroz S. Black truffle-associated bacterial communities during the development and maturation of Tuber melanosporum ascocarps and putative functional roles. Environment Microbiology, 2014, 16(9): 2831–2847. DOI:10.1111/emi.2014.16.issue-9 |
| [40] | Deveau A, Antony-Babu S, Le Tacon F, Robin C, Frey-Klett P. Temporal changes of bacterial communities in the Tuber melanosporum ectomycorrhizosphere during ascocarp development. Mycorrhiza, 2016, 26(5): 389–399. DOI:10.1007/s00572-015-0679-7 |
 2018, Vol. 38
2018, Vol. 38