文章信息
- 刘国红, 刘波, 王阶平, 朱育菁, 车建美, 陈倩倩, 陈峥
- LIU Guohong, LIU Bo, WANG Jieping, ZHU Yujing, CHE Jianmei, CHEN Qianqian, CHEN Zheng.
- 养猪微生物发酵床芽胞杆菌空间分布多样性
- Spatial distribution pattern and community diversity of the Bacillus-like species in a fermentation bed system for pig rearing
- 生态学报. 2017, 37(20): 6914-6932
- Acta Ecologica Sinica. 2017, 37(20): 6914-6932
- http://dx.doi.org/10.5846/stxb201607261528
-
文章历史
- 收稿日期: 2016-07-26
- 网络出版日期: 2017-06-01
生猪养殖过程造成严重的环境污染, 以福建省为例, 2011年生猪存栏1297.80万头, 按每天每头猪排放粪尿加冲洗猪栏污水约25 kg计算, 全年排污量高达11842.425万t, 成为农业面源污染的主要污染源之一[1]。近年来, 微生物发酵床应用于养猪, 采用垫料(椰糠、谷壳、菌糠、锯末等配合)铺垫猪舍, 接入微生物菌剂, 猪粪尿直排其上, 通过微生物原位发酵, 分解猪粪、消除臭味, 从而实现生猪养殖的零排放, 解决养猪污染的问题[2-4]。
微生物发酵床养猪思路最早可以追溯到中国360年前张履祥辑补《沈氏农书》记载的猪圈垫草饲养方法[5]。现代微生物发酵床养殖研究最早见于日本, 1970年日本建立了第一个以木屑为垫料的发酵床[6], 中国香港的Tiquia等研究了发教床垫料翻堆的作用[7], 德国的Kaufmann等研究了育肥猪发酵床猪粪营养平衡[8]。加拿大Biotech公司于1985年推出一个以秸秆为垫料的发酵床系统[9], 克罗地亚的Margeta等分析了本国养猪发酵床优缺点及其发展前景[10], 波兰的Knecht等研究发现发酵床猪舍猪寄生虫病流行低于漏缝地板猪舍[11], 刘波等设计了2100m2的大栏发酵床智能化养猪系统的猪舍[12]。
关于发酵床微生物特性研究报道较少, 刘波等研究了发酵床微生物群落脂肪酸生物标记多样性[13]。郑雪芳等分析了发酵床垫料微生物亚群落的分化[14]。张学峰等揭示了不同深度发酵床垫料土著稳定微生物菌群[15]。王迪分离鉴定了发酵床的芽胞杆菌(Bacillus)[16]。林莉莉等研究了发酵床猪舍环境与猪体表微生物分布状况[17]。张庆宁从微生物发酵床筛选出了分解猪粪的芽胞杆菌[18]。刘让等筛选芽胞杆菌作为发酵床发酵菌种[19]。
微生物发酵床对猪病具有一定的生防作用, 郑雪芳等研究发现微生物发酵床养猪基质垫层形成的稳定微生物群落结构能抑制大肠杆菌特别是致病性大肠杆菌的生长[20]。毕小艳研究了猪用发酵床垫料中微生物动态变化及对猪免疫力的影响[21]。以往的研究表明, 芽胞杆菌作为发酵床的优势菌群, 具有生长优势强, 耐发酵高热, 能够产生多种与猪粪降解相关的酶类[18], 具有降解猪粪[16]、抑制病原[22]、清除臭味[23]等的作用;随着在发酵床饲养时间的延长, 从对应猪分离的大肠杆菌的抗药性逐渐降低[18]。
研究发酵床芽胞杆菌特性, 了解发酵床芽胞杆菌空间分布多样性, 对于研究发酵床微生物群落功能、猪粪降解机理、猪病生防机理等具有重要意义。目前, 关于微生物发酵床芽胞杆菌种类结构, 数量分布, 空间格局等的研究未见报道。本研究以2100 m2的微生物发酵床大栏养猪为研究对象, 通过空间格局采样, 用可培养法分离芽胞杆菌方法, 对发酵床芽胞杆菌种类分布、数量分布、空间分布的多样性进行分析, 利用空间分布型、多样性指数评估养猪微生物发酵床中芽胞杆菌的空间分布特征, 为发酵床管理、猪粪资源化利用、猪病的生防等提供研究基础。
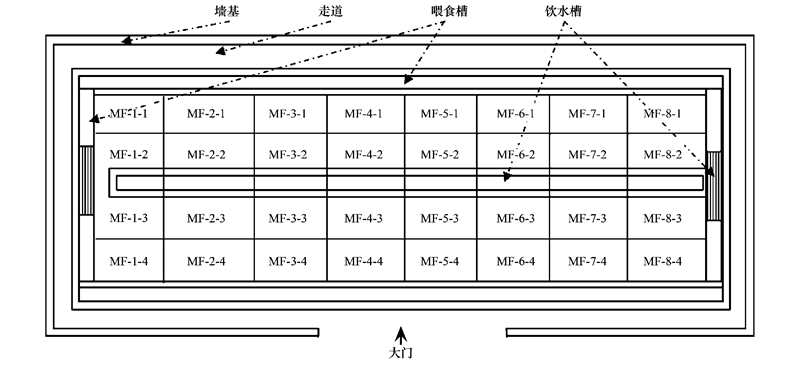
1 材料与方法 1.1 样品的采集2015年1月26日, 从福建省农业科学院福清现代设施农业样本工程示范基地大栏养猪微生物发酵床采集样品, 发酵床垫料已使用1a至1.5a左右。该大栏发酵床猪场建筑面积为2100 m2(35 m × 60 m), 发酵床槽面积1910 m2, 除两个隔离栏, 养殖面积为1600 m2, 发酵床深度80 cm, 发酵床垫料由70%椰糠和30%谷壳组成。发酵床饲养1600头育肥猪, 饲养密度为1头/m2(图 1)。

|
| 图 1 大栏微生物发酵床 Fig. 1 The piggery bedding |
采样方法:将大栏发酵床宽度方向划分4栏(行), 长度方向划分8栏(列), 每个栏采用五点取样方法采集0-20 cm的样品并混合获得栏的垫料样品, 共采集32个空间的32个样品(图 2), 装入无菌聚氯乙烯塑料瓶, 带回实验室, 并立即进行芽胞杆菌菌株的分离与保存。
|
| 图 2 发酵床空间采集格局 Fig. 2 Samples information collected from fermentation bed in the piggrey |
PCR仪和凝胶成像分析仪(Bio-Rad公司);恒温培养箱(BI-250AG)购自施都凯仪器设备(上海)有限公司;2×PCR Master Mix购自上海铂尚生物技术有限公司;引物由上海铂尚生物技术有限公司合成。LB培养基:胰蛋白胨10.0 g, 酵母提取物5.0 g, 氯化钠5.0 g, pH 7.2-7.4, 琼脂15.0 g, 水1.0 L。
1.3 微生物发酵床芽胞杆菌分离与鉴定通过梯度稀释涂布法分离获得微生物发酵床样品中的芽胞杆菌, 根据菌落形态特征等进行芽胞杆菌种类归类、统计计数及纯化, 采用-80 ℃甘油冷冻法进行保存。采用Tris-饱和酚法提取芽胞杆菌基因组DNA。采用通用细菌16S rRNA引物进行扩增、测序, 主要参考Liu等描述的方法[24]。16S rRNA基因扩增引物为27F(5′-AGAGTTTGATCCTGGCTCAG-3′)和1492R(5′-GGTTACCTTGTTACGACTT-3′)。检测出有条带的菌株PCR产物送至上海铂尚生物技术有限公司进行测序。将测序所得16S rRNA序列在韩国网站(EZtaxon-e.ezbiocloud.net)上进行序列比对分析[25], 初步判断得出芽胞杆菌种的分类地位。根据Tindall等描述, 当16S rRNA相似性 > 97%时可定义为同一个分类单元[26]。
1.4 微生物发酵床样方芽胞杆菌种类与数量空间分布微生物发酵床划分成横向4个单元, 纵向8个单元, 共取样32个空间样本。统计各空间样本芽胞杆菌种类数、数量总和、最大值、最小值、平均值等, 比较发酵床空间样本芽胞杆菌种类与数量的变化, 绘制直方图分析芽胞杆菌种类在微生物发酵床空间样本中出现频次和数量。
1.5 微生物发酵床芽胞杆菌空间分布型基于微生物发酵床4行8列采集方案, 构建数据矩阵, 统计每行样本芽胞杆菌平均值和方差, 利用聚集度指标和回归分析法, 分析芽胞杆菌样方空间分布型(表 1)。
| 聚集度指标 Aggregation indices | 方程 Equation | 注释 Note | 判别 Discrimination |
| 平均拥挤度(m*)Mean Crowding intensity | 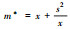
|
x为平均数, s2为方差 | |
| I指标I index |  |
x为平均数, s2为方差 | 当I < 0时为均匀分布, 当I=0时为随机分布, 当I > 0时为聚集分布 |
| m*/m指标m*/m index | 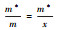
|
m*为平均拥挤度, x为平均数 | 当m*/m < 1时为均匀分布;当m*/m=1时为随机分布;当m*/m > 1时为聚集分布 |
| CA指标CA index | 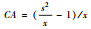
|
s2为方差, x为平均数 | 当CA < 0时为均匀分布, 当CA =0时为随机分布, 当CA > 0时为聚集分布 |
| 扩散系数(C)Diffusion coefficient | C=s2/x | 当C < 1时为均匀分布, 当C = 1时为随机分布, C > 1时为聚集分布 | |
| 负二项分布K指标K index of negative binomial distribution | K=x2/(s2-x) | 当K < 0时为均匀分布, 当K=0时为随机分布, 当K > 0时为聚集分布 | |
| m*-m回归分析法m*-m regression analysis | m*=α+βx | 当b < 1时, 为均匀分布;当b =1时, 为随机分布;当b > 1时, 为聚集分布 | |
| Talor幂法则Taylor′s power law | lgs2=lga+blgx | 当b→0时为均匀分布, b=1时为随机分布, b > 1时为聚集分布 |
以芽胞杆菌种类为样本, 以样本为指标, 构建数据矩阵, 利用生物统计软件PRIMER v5(Primer-E Ltd., Plymouth, United Kingdom [2001])进行计算、统计微生物发酵床芽胞杆菌种类出现频次、种类数量、丰富度指数(D)、均匀度指数(J′)、优势度指数(λ)、Shannon-Wiener指数(H′)和Hill指数(N1)。
以芽胞杆菌为样本, 以多样性指数为指标, 构建数据矩阵, 通过生物统计软件SPSS 16.0, 以欧氏距离为尺度, 用类平均法进行微生物发酵床芽胞杆菌多样性指数聚类分析。多样性指数公式如下:
1) 丰富度指数(D)
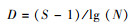
|
2) 均匀度指数(J′)
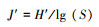
|
3) 优势度指数(λ)

|
4) Shannon-Wiener指数(H′)
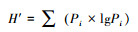
|
5) Hill指数(N1)
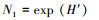
|
式中, Pi=Ni/N。Ni为第i种芽胞杆菌的数量, S为芽胞杆菌占据的单元总数, N为芽胞杆菌种类个体总数。
2 结果与分析 2.1 微生物发酵床芽胞杆菌的分离与鉴定实验结果见表 2。根据菌落形态特征区分, 从32份微生物发酵床空间样品中分离获得了芽胞杆菌452株, 通过16S rRNA基因序列比对分析, 所有芽胞杆菌菌株相似性皆大于97.0%, 鉴定为48个种, 隶属于芽胞杆菌纲的2个科即芽胞杆菌科(Bacillaceae)和类芽胞杆菌科(Paenibacillaceae), 8个属即芽胞杆菌属(Bacillus)(30种)、类芽胞杆菌属(Paenibacillus)(5种)、赖氨酸芽胞杆菌属(Lysinibacillus)(6种)、短芽胞杆菌属(Brevibacillus)(3种)、鸟氨酸芽胞杆菌属(Ornithinibacillus)(1种)、大洋芽胞杆菌属(Oceanibacillus)(1种)、少盐芽胞杆菌属(Paucisalibacillus)(1种)和纤细芽胞杆菌属(Gracilibacillus)(1种)。
| 编号 No. | 菌株编号 Strain no | 16S rRNA相似性/% 16S rRNAsimilarity | 物种名 Species name | 种类数 Number |
| [1] | FJAT-41456 | 98.1 | 高地芽胞杆菌 Bacillus altitudidns | 30 |
| [2] | FJAT-41678 | 99.3 | 解淀粉芽胞杆菌 Bacillus amyloliquefaciens | |
| [3] | FJAT-41614 | 99.5 | 阿氏芽胞杆菌 Bacillus aryabhattai | |
| [4] | FJAT-41708 | 100 | 蜡状芽胞杆菌 Bacillus cereus | |
| [5] | FJAT-41599 | 99.5 | 环状芽胞杆菌 Bacillus circulans | |
| [6] | FJAT-41623 | 99.7 | 克劳氏芽胞杆菌 Bacillus clausii | |
| [7] | FJAT-41407 | 99.10 | 蚯蚓芽胞杆菌 Bacillus eiseniae | |
| [8] | FJAT-41644 | 99.0 | 弯曲芽胞杆菌 Bacillus flexus | |
| [9] | FJAT-42923 | 99.3 | 海口芽胞杆菌 Bacillus haikouensis | |
| [10] | FJAT-41639 | 99.5 | 嗜盐噬糖芽胞杆菌 Bacillus halosaccharovorans | |
| [11] | FJAT-41679 | 99.8 | 霍氏芽胞杆菌 Bacillus horneckiae | |
| [12] | FJAT-41709 | 97.9 | 土地芽胞杆菌 Bacillus humi | |
| [13] | FJAT-41627 | 99.3 | 印空研芽胞杆菌 Bacillus isronensis | |
| [14] | FJAT-41629 | 99.7 | 柯赫芽胞杆菌 Bacillus kochii | |
| [15] | FJAT-41609 | 99.3 | 地衣芽胞杆菌 Bacillus licheniformis | |
| [16] | FJAT-41224 | 99.4 | 黄海芽胞杆菌 Bacillus marisflavi | |
| [17] | FJAT-41714 | 99.8 | 嗜常温芽胞杆菌 Bacillus mesophilum | |
| [18] | FJAT-41604 | 99.5 | 甲基营养型芽胞杆菌 Bacillus methylotrophicus | |
| [19] | FJAT-41602 | 98.2 | 尼氏芽胞杆菌 Bacillus nealsonii | |
| [20] | FJAT-41408 | 99.5 | 烟酸芽胞杆菌 Bacillus niacini | |
| [21] | FJAT-41206 | 98.8 | 海床类芽胞杆菌 Bacillus oceanisediminis | |
| [22] | FJAT-41638 | 99.0 | 蔬菜芽胞杆菌 Bacillus oleronius | |
| [23] | FJAT-41641 | 99.7 | 根际芽胞杆菌 Bacillus rhizosphaerae | |
| [24] | FJAT-41494 | 99.6 | 暹罗芽胞杆菌 Bacillus siamensis | |
| [25] | FJAT-41608 | 99.9 | 青贮窖芽胞杆菌 Bacillus siralis | |
| [26] | FJAT-41518 | 99.9 | 枯草芽胞杆菌 Bacillus subtilis | |
| [27] | FJAT-41219 | 99.9 | 特基拉芽胞杆菌 Bacillus tequilensis | |
| [28] | FJAT-41593 | 98.2 | 泰门芽胞杆菌 Bacillus timonensis | |
| [29] | FJAT-41252 | 99.5 | 越南芽胞杆菌 Bacillus vietnamensis | |
| [30] | FJAT-41633 | 99.8 | 厦门芽胞杆菌 Bacillus xiamenensis | |
| [31] | FJAT-41635 | 99.9 | 波茨坦短芽胞杆菌 Brevibacillus borstelensis | 3 |
| [32] | FJAT-41467 | 99.2 | 居湖短芽胞杆菌 Brevibacillus limnophilus | |
| [33] | FJAT-41640 | 99.6 | 硝化短芽胞杆菌 Brevibacillus nitrificans | |
| [34] | FJAT-41653 | 99.0 | 海洋纤细芽胞杆菌 Gracilibacillus marinus | 1 |
| [35] | FJAT-41606 | 98.5 | 清国酱赖氨酸芽胞杆菌 Lysinibacillus chungkukjangi | 6 |
| [36] | FJAT-41646 | 97.5 | 堆肥赖氨酸芽胞杆菌 Lysinibacillus composti | |
| [37] | FJAT-41611 | 99.6 | 纺锤形赖氨酸芽胞杆菌 Lysinibacillus fusiformis | |
| [38] | FJAT-41610 | 99.7 | 耐盐赖氨酸芽胞杆菌 Lysinibacillus halotolerans | |
| [39] | FJAT-41612 | 99.6 | 长赖氨酸芽胞杆菌 Lysinibacillus macroides | |
| [40] | FJAT-41607 | 99.3 | 锰矿土赖氨酸芽胞杆菌 Lysinibacillus manganicus | |
| [41] | FJAT-42930 | 99.4 | 淤泥大洋芽胞杆菌 Oceanobacillus caeni | 1 |
| [42] | FJAT-41619 | 98.7 | 拾蛤鸟氨酸芽胞杆菌 Ornithinibacillus scapharcae | 1 |
| [43] | FJAT-41201 | 99.9 | 巴伦氏类芽胞杆菌 Paenibacillus barengoltzii | |
| [44] | FJAT-41656 | 99.0 | 人参土芽胞杆菌 Paenibacillus ginsengiterrae | |
| [45] | FJAT-41484 | 98.3 | 伊利诺伊类芽胞杆菌 Paenibacillus illinoisensis | |
| [46] | FJAT-41526 | 99.5 | 牛奶类芽胞杆菌 Paenibacillus lactis | |
| [47] | FJAT-41673 | 98.4 | 饲料类芽胞杆菌 Paenibacillus pabuli | |
| [48] | FJAT-41371 | 98.3 | 小球状少盐芽胞杆菌 Paucisalibacillus globulus | 1 |
| FJAT是FuJian Agaricultural Technology(福建农业科技)的缩写 | ||||
实验结果见表 3。微生物发酵床划分成横向4个单元、纵向8个单元, 共取样32个空间样本, 分离鉴定统计各空间样本中芽胞杆菌的种类和数量, 结果表明:(1) 各空间样本芽胞杆菌种类差异很大, 从最多的14种(MF-1-3) 到最少的5种(MF-6-3);(2) 芽胞杆菌分布在发酵床空间差异很大, 有些芽胞杆菌种类分布在多个空间样本中, 如阿氏芽胞杆菌(Bacillus aryabhattai)可以分布在空间样本MF-1-1、MF-1-2、MF-1-3、MF-1-4等19个空间样本中, 有些种类只分布在特定的空间样本中, 如海口芽胞杆菌(Bacillus haikouensis)仅分布在空间样本MF-5-3, 居湖短芽胞杆菌(Brevibacillus limnophilus)仅分布在空间样本MF-4-1等;(3) 芽胞杆菌单个菌株在一个空间样本中的最大含量是环状芽胞杆菌(Bacillus circulans), 分布数量为50.0×106个/g(MF-1-4);含量最小的是印空研芽胞杆菌(Bacillus isronensis)等, 分布数量为0.01×106个/g(MF-4-3)。
| 空间样本 Sample | 菌株编号 Strain no | 鉴定种类 Closest species | 含量/(×106个/g) Content | 空间样本 Sample | 菌株编号 Strain no | 鉴定种类 Closest species | 含量/(×106个/g) Content |
| MF-1-1 12种 | FJAT-41614 | Bacillus aryabhattai | 0.3 | MF-4-3 7种 | FJAT-41215 | Bacillus altitudinis | 0.5 |
| FJAT-41599 | Bacillus circulans | 5.0 | FJAT-41209 | Bacillus isronensis | 0.01 | ||
| FJAT-41616 | Bacillus altitudinis | 1.0 | FJAT-41214 | Bacillus aryabhattai | 0.05 | ||
| FJAT-41609 | Bacillus licheniformis | 8.0 | FJAT-41220 | Bacillus kochii | 2.7 | ||
| FJAT-41604 | Bacillus methylotrophicus | 1.0 | FJAT-41216 | Bacillus licheniformis | 0.2 | ||
| FJAT-41602 | Bacillus nealsonii | 2.3 | FJAT-41219 | Bacillus tequilensis | 1.3 | ||
| FJAT-41608 | Bacillus siralis | 0.2 | FJAT-41213 | Bacillus subtilis | 1.0 | ||
| FJAT-41611 | Lysinibacillus fusiformis | 0.1 | MF-4-4 7种 | FJAT-41495 | Bacillus kochii | 0.1 | |
| FJAT-41610 | Lysinibacillus halotolerans | 0.1 | FJAT-41488 | Bacillus eiseniae | 0.5 | ||
| FJAT-41612 | Lysinibacillus macroides | 0.1 | FJAT-41489 | Bacillus licheniformis | 0.3 | ||
| FJAT-41606 | Lysinibacillus chungkukjangi | 1.0 | FJAT-41494 | Bacillus siamensis | 0.07 | ||
| FJAT-41607 | Lysinibacillus manganicus | 0.3 | FJAT-41491 | Bacillus tequilensis | 2.5 | ||
| MF-1-2 | FJAT-41620 | Bacillus aryabhattai | 3.0 | FJAT-41492 | Bacillus subtilis | 0.2 | |
| 11种 | FJAT-41622 | Bacillus circulans | 2.0 | FJAT-41496 | Gracilibacillus marinus' | 0.1 | |
| FJAT-41623 | Bacillus clausii | 1.0 | MF-5-1 7种 | FJAT-41504 | Bacillus altitudinis | 0.3 | |
| FJAT-41625 | Bacillus altitudinis | 1.0 | FJAT-41498 | Bacillus kochii | 0.8 | ||
| FJAT-41629 | Bacillus kochii | 2.0 | FJAT-41499 | Bacillus licheniformis | 1.5 | ||
| FJAT-41630 | Bacillus licheniformis | 1.0 | FJAT-41503 | Bacillus rhizosphaerae | 0.1 | ||
| FJAT-41621 | Bacillus methylotrophicus | 5.0 | FJAT-41511 | Bacillus tequilensis | 1.8 | ||
| FJAT-41633 | Bacillus xiamenensis | 0.1 | FJAT-41506 | Lysinibacillus halotolerans | 0.7 | ||
| FJAT-41627 | Bacillus isronensis | 1.0 | FJAT-41510 | Ornithinibacillus scapharcae | 1.2 | ||
| FJAT-41632 | Lysinibacillus halotolerans | 0.7 | MF-5-2 11种 | FJAT-41519 | Bacillus altitudinis | 0.02 | |
| FJAT-41619 | Ornithinibacillus scapharcae | 0.1 | FJAT-41512 | Bacillus amyloliquefaciens | 0.3 | ||
| MF-1-3 | FJAT-41636 | Bacillus nealsonii | 10.0 | FJAT-41513 | Bacillus cereus | 0.04 | |
| 14种 | FJAT-41652 | Bacillus aryabhattai | 5.0 | FJAT-41515 | Bacillus fiexus | 0.18 | |
| FJAT-41642 | Bacillus circulans | 2.0 | FJAT-41523 | Bacillus clausii | 0.12 | ||
| FJAT-41639 | Bacillus halosaccharovorans | 2.0 | FJAT-41527 | Bacillus kochii | 0.02 | ||
| FJAT-41643 | Bacillus licheniformis | 2.0 | FJAT-41520 | Bacillus licheniformis | 0.48 | ||
| FJAT-41648 | Bacillus methylotrophicus | 2.0 | FJAT-41524 | Bacillus siamensis | 0.01 | ||
| FJAT-41638 | Bacillus oleronius | 3.0 | FJAT-41518 | Bacillus subtilis | 0.08 | ||
| FJAT-41641 | Bacillus rhizosphaerae | 0.1 | FJAT-41516 | Bacillus tequilensis | 0.09 | ||
| FJAT-41635 | Brevibacillus borstelensis | 0.1 | FJAT-41526 | Paenibacillus lactis | 0.15 | ||
| FJAT-41640 | Brevibacillus nitrificans | 0.2 | MF-5-3 8种 | FJAT-42924 | Bacillus altitudinis | 0.1 | |
| FJAT-41644 | Bacillus fiexus | 1.0 | FJAT-42919 | Bacillus aryabhattai | 0.3 | ||
| FJAT-41649 | Lysinibacillus composti | 2.1 | FJAT-42929 | Bacillus clausii | 0.1 | ||
| FJAT-41650 | Lysinibacillus halotolerans | 0.2 | FJAT-42926 | Bacillus kochii | 0.4 | ||
| FJAT-41637 | Ornithinibacillus scapharcae | 0.2 | FJAT-42925 | Bacillus licheniformis | 0.5 | ||
| MF-1-4 12种 | FJAT-41656 | Paenibacillus ginsengiterrae | 20.0 | FJAT-42923 | Bacillus haikouensis | 1.0 | |
| FJAT-41658 | Bacillus aryabhattai | 20.0 | FJAT-42921 | Bacillus subtilis | 5.0 | ||
| FJAT-41667 | Bacillus altitudinis | 0.2 | FJAT-42930 | Oceanobacillus caeni | 0.2 | ||
| FJAT-41668 | Bacillus circulans | 50.0 | MF-5-4 7种 | FJAT-41532 | Bacillus altitudinis | 0.12 | |
| FJAT-41662 | Lysinibacillus halotolerans | 1.0 | FJAT-41536 | Bacillus clausii | 0.3 | ||
| FJAT-41663 | Bacillus halosaccharovorans | 4.0 | FJAT-41542 | Bacillus kochii | 0.1 | ||
| FJAT-41654 | Bacillus licheniformis | 0.4 | FJAT-41537 | Bacillus licheniformis | 1.1 | ||
| FJAT-41660 | Bacillus methylotrophicus | 0.6 | FJAT-41529 | Bacillus rhizosphaerae | 0.1 | ||
| FJAT-41666 | Bacillus nealsonii | 0.2 | FJAT-41541 | Bacillus tequilensis | 0.1 | ||
| FJAT-41661 | Bacillus oleronius | 1 | FJAT-41539 | Lysinibacillus halotolerans | 0.6 | ||
| FJAT-41659 | Bacillus rhizosphaerae | 0.1 | MF-6-1 6种 | FJAT-41223 | Bacillus methylotrophicus | 0.29 | |
| FJAT-41653 | Gracilibacillus marinus | 0.1 | FJAT-41225 | Bacillus aryabhattai | 9.2 | ||
| MF-2-1 8种 | FJAT-41679 | Bacillus horneckiae | 1.0 | FJAT-41236 | Bacillus circulans | 0.3 | |
| FJAT-41684 | Bacillus circulans | 4.3 | FJAT-41234 | Bacillus kochii | 0.1 | ||
| FJAT-41682 | Bacillus clausii | 2.0 | FJAT-41235 | Bacillus licheniformis | 2.6 | ||
| FJAT-41674 | Bacillus halosaccharovorans | 2.0 | FJAT-41224 | Bacillus marisflavi | 0.1 | ||
| FJAT-41683 | Bacillus altitudinis | 3.0 | MF-6-2 6种 | FJAT-41552 | Bacillus altitudinis | 1.0 | |
| FJAT-41670 | Bacillus licheniformis | 5.0 | FJAT-41548 | Bacillus clausii | 0.2 | ||
| FJAT-41673 | Paenibacillus pabuli | 0.2 | FJAT-41545 | Bacillus licheniformis | 1.7 | ||
| FJAT-41678 | Bacillus amyloliquefaciens | 2.0 | FJAT-41551 | Bacillus methylotrophicus | 0.1 | ||
| MF-2-2 8种 | FJAT-41694 | Bacillus aryabhattai | 2.0 | FJAT-41549 | Bacillus tequilensis | 1.7 | |
| FJAT-41689 | Bacillus circulans | 1.0 | FJAT-41544 | Brevibacillus borstelensis | 0.1 | ||
| FJAT-41690 | Bacillus clausii | 1.0 | MF-6-3 5种 | FJAT-41244 | Bacillus methylotrophicus | 0.57 | |
| FJAT-41700 | Bacillus halosaccharovorans | 7.0 | FJAT-41255 | Bacillus aryabhattai | 2.4 | ||
| FJAT-41695 | Bacillus altitudinis | 2.0 | FJAT-41248 | Bacillus tequilensis | 0.06 | ||
| FJAT-41687 | Bacillus kochii | 0.2 | FJAT-41242 | Bacillus kochii | 1.0 | ||
| FJAT-41699 | Bacillus methylotrophicus | 0.5 | FJAT-41252 | Bacillus vietnamensis | 1.0 | ||
| FJAT-41703 | Ornithinibacillus scapharcae | 0.4 | MF-6-4 7种 | FJAT-41143 | Bacillus clausii | 1.4 | |
| MF-2-3 10种 | FJAT-41708 | Bacillus cereus | 1.0 | FJAT-41146 | Lysinibacillus macrolides | 0.1 | |
| FJAT-41705 | Bacillus rhizosphaerae | 1.0 | FJAT-41142 | Bacillus siamensis | 1.0 | ||
| FJAT-41709 | Bacillus humi | 1.0 | FJAT-41147 | Bacillus licheniformis | 2.6 | ||
| FJAT-41712 | Bacillus aryabhattai | 3.0 | FJAT-41150 | Bacillus tequilensis | 1.0 | ||
| FJAT-41713 | Bacillus circulans | 1.0 | FJAT-41149 | Bacillus cereus | 1.0 | ||
| FJAT-41718 | Bacillus halosaccharovorans | 1.0 | FJAT-41145 | Bacillus subtilis | 1.0 | ||
| FJAT-41715 | Bacillus kochii | 2.0 | MF-7-1 7种 | FJAT-41167 | Bacillus altitudinis | 3.0 | |
| FJAT-41706 | Bacillus licheniformis | 3.0 | FJAT-41175 | Bacillus circulans | 23.0 | ||
| FJAT-41714 | Bacillus mesophilum | 1.0 | FJAT-41172 | Bacillus kochii | 2.0 | ||
| FJAT-41707 | Bacillus methylotrophicus | 1.7 | FJAT-41168 | Bacillus licheniformis | 6.0 | ||
| MF-2-4 10种 | FJAT-41729 | Bacillus clausii | 2.0 | FJAT-41166 | Bacillus subtilis | 1.0 | |
| FJAT-41728 | Bacillus isronensis | 4.0 | FJAT-41176 | Bacillus tequilensis | 4.0 | ||
| FJAT-41736 | Bacillus rhizosphaerae | 2.0 | FJAT-41174 | Ornithinibacillus scapharcae | 1.0 | ||
| FJAT-41739 | Lysinibacillus halotolerans | 1.0 | MF-7-2 7种 | FJAT-41295 | Bacillus methylotrophicus | 0.05 | |
| FJAT-41734 | Bacillus aryabhattai | 3.0 | FJAT-41299 | Bacillus aryabhattai | 0.11 | ||
| FJAT-41735 | Bacillus circulans | 2.0 | FJAT-41305 | Bacillus circulans | 0.01 | ||
| FJAT-41726 | Bacillus altitudinis | 2.0 | FJAT-41307 | Bacillus kochii | 0.41 | ||
| FJAT-41722 | Bacillus kochii | 3.0 | FJAT-41302 | Bacillus licheniformis | 4.02 | ||
| FJAT-41727 | Bacillus licheniformis | 0.7 | FJAT-41301 | Lysinibacillus halotolerans | 0.2 | ||
| FJAT-41731 | Bacillus methylotrophicus | 0.9 | MF-7-3 7种 | FJAT-41178 | Bacillus circulans | 0.3 | |
| MF-3-1 | FJAT-41752 | Bacillus kochii | 1.0 | FJAT-41180 | Bacillus clausii | 0.4 | |
| 8种 | FJAT-41754 | Bacillus siralis | 2.0 | FJAT-41181 | Lysinibacillus halotolerans | 0.1 | |
| FJAT-41742 | Gracilibacillus marinus | 4.0 | FJAT-41182 | Bacillus kochii | 1.6 | ||
| FJAT-41746 | Bacillus aryabhattai | 2.0 | FJAT-41184 | Bacillus licheniformis | 1.5 | ||
| FJAT-41750 | Bacillus altitudinis | 2.0 | FJAT-41179 | Bacillus methylotrophicus | 0.3 | ||
| FJAT-41741 | Bacillus licheniformis | 6.0 | FJAT-41190 | Bacillus subtilis | 0.1 | ||
| FJAT-41747 | Bacillus methylotrophicus | 0.5 | MF-7-4 5种 | FJAT-41309 | Bacillus kochii | 0.16 | |
| FJAT-41740 | Ornithinibacillus scapharcae | 0.1 | FJAT-41312 | Bacillus altitudinis | 0.03 | ||
| MF-3-2 | FJAT-41382 | Bacillus aryabhattai | 2.0 | FJAT-41317 | Bacillus methylotrophicus | 0.21 | |
| 8种 | FJAT-41375 | Bacillus circulans | 2.0 | FJAT-41318 | Bacillus aryabhattai | 0.2 | |
| FJAT-41374 | Bacillus halosaccharovorans | 1.0 | FJAT-41319 | Bacillus amyloliquefaciens | 0.2 | ||
| FJAT-41386 | Bacillus licheniformis | 15.0 | MF-8-1 7种 | FJAT-41323 | Bacillus methylotrophicus | 0.18 | |
| FJAT-41380 | Bacillus methylotrophicus | 0.6 | FJAT-41322 | Bacillus aryabhattai | 1.2 | ||
| FJAT-41372 | Bacillus rhizosphaerae | 0.4 | FJAT-41331 | Bacillus circulans | 0.1 | ||
| FJAT-41392 | Lysinibacillus halotolerans | 0.2 | FJAT-41328 | Bacillus kochii | 0.1 | ||
| FJAT-41371 | Paucisalibacillus globulus | 0.2 | FJAT-41324 | Bacillus licheniformis | 2.9 | ||
| MF-3-3 7种 | FJAT-41399 | Bacillus aryabhattai | 0.2 | FJAT-41329 | Lysinibacillus halotolerans | 0.3 | |
| FJAT-41393 | Bacillus clausii | 0.4 | FJAT-41330 | Bacillus amyloliquefaciens | 0.1 | ||
| FJAT-41398 | Bacillus halosaccharovorans | 0.1 | MF-8-2 8种 | FJAT-41595 | Bacillus methylotrophicus | 0.3 | |
| FJAT-41395 | Bacillus kochii | 0.1 | FJAT-41598 | Bacillus altitudinis | 0.1 | ||
| FJAT-41396 | Bacillus licheniformis | 3.0 | FJAT-41596 | Bacillus cereus | 0.2 | ||
| FJAT-41397 | Bacillus rhizosphaerae | 1.0 | FJAT-41590 | Bacillus clausii | 0.9 | ||
| FJAT-41400 | Bacillus circulans | 1.0 | FJAT-41585 | Bacillus licheniformis | 1.6 | ||
| MF-3-4 9种 | FJAT-41410 | Bacillus cereus | 0.2 | FJAT-41586 | Bacillus siamensis | 0.1 | |
| FJAT-41409 | Bacillus aryabhattai | 0.2 | FJAT-41597 | Bacillus tequilensis | 0.1 | ||
| FJAT-41412 | Bacillus clausii | 0.4 | FJAT-41593 | Bacillus timonensis | 0.1 | ||
| FJAT-41407 | Bacillus eiseniae | 0.2 | MF-8-3 6种 | FJAT-41333 | Bacillus cereus group | 0.01 | |
| FJAT-41413 | Bacillus kochii | 0.4 | FJAT-41334 | Bacillus kochii | 0.03 | ||
| FJAT-41404 | Bacillus licheniformis | 0.2 | FJAT-41335 | Bacillus methylotrophicus | 0.11 | ||
| FJAT-41403 | Bacillus methylotrophicus | 0.2 | FJAT-41336 | Bacillus amyloliquefaciens | 0.01 | ||
| FJAT-41408 | Bacillus niacini | 0.2 | FJAT-41338 | Bacillus aryabhattai | 0.06 | ||
| FJAT-41445 | Lysinibacillus halotolerans | 0.1 | FJAT-41343 | Bacillus licheniformis | 1.56 | ||
| MF-4-1 6种 | FJAT-41458 | Bacillus altitudinis | 0.2 | MF-8-4 11种 | FJAT-41205 | Bacillus amyloliquefaciens | 2.1 |
| FJAT-41463 | Bacillus kochii | 0.4 | FJAT-41199 | Bacillus clausii | 0.3 | ||
| FJAT-41457 | Bacillus licheniformis | 1.3 | FJAT-41203 | Bacillus eiseniae | 0.1 | ||
| FJAT-41459 | Bacillus methylotrophicus | 0.6 | FJAT-41191 | Bacillus licheniformis | 0.7 | ||
| FJAT-41467 | Brevibacillus limnophilus | 0.2 | FJAT-41206 | Bacillus oceanisediminis | 0.1 | ||
| FJAT-41455 | Ornithinibacillus scapharcae | 0.3 | FJAT-41195 | Bacillus siamensis | 0.5 | ||
| MF-4-2 9种 | FJAT-41476 | Bacillus circulans | 0.1 | FJAT-41192 | Bacillus subtilis | 1.1 | |
| FJAT-41474 | Bacillus altitudinis | 0.5 | FJAT-41202 | Brevibacillus borstelensis | 0.1 | ||
| FJAT-41468 | Bacillus kochii | 0.1 | FJAT-41198 | Lysinibacillus halotolerans | 2.2 | ||
| FJAT-41469 | Bacillus licheniformis | 0.8 | FJAT-41197 | Ornithinibacillus scapharcae | 0.2 | ||
| FJAT-41473 | Bacillus methylotrophicus | 7.0 | FJAT-41201 | Paenibacillus barengoltzii | 0.1 | ||
| FJAT-41475 | Bacillus siralis | 1.0 | |||||
| FJAT-41470 | Lysinibacillus halotolerans | 1.0 | |||||
| FJAT-41484 | Paenibacillus illinoisensis | 10.0 | |||||
| FJAT-41483 | Paucisalibacillus globulus | 10.0 |
基于表 3统计, 微生物发酵床芽胞杆菌种类空间出现频次分析结果见图 3。芽胞杆菌种类微生物发酵床32个空间样本中出现频次差异显著, 有的种类广泛分布在空间样本中, 有的种类分布在少数空间样本中, 其分布广泛性可分为3类:第Ⅰ类为广分布种类, 有8个种分布在14-29个空间样本中, 分别为地衣芽胞杆菌(Bacillus licheniformis)(29样本)、柯赫芽胞杆菌(B. kochii)(23样本)、甲基营养型芽胞杆菌(B. methylotrophicus)(21样本)、高地芽胞杆菌(B. altitudidns)(18样本)、阿氏芽胞杆菌(B. aryabhattai)(19样本)、环状芽胞杆菌(B. circulans)(16样本)、耐盐赖氨酸芽胞杆菌(Lysinibacillus halotolerans)(14样本)和克劳氏芽胞杆菌(B. clausii)(14样本)。第Ⅱ类为寡分布种类, 有8个种分布在5-10个空间样本中, 它们是特基拉芽胞杆菌(B. tequilensis)(10样本)、根际芽胞杆菌(B. rhizosphaerae)(8样本)、拾蛤鸟氨酸芽胞杆菌(Ornithinibacillus scapharcae)(8样本)、嗜盐噬糖芽胞杆菌(B. halosaccharovorans)(7样本)、解淀粉芽胞杆菌(B. amyloliquefaciens)(6样本)、蜡状芽胞杆菌(B. cereus)(6样本)、枯草芽胞杆菌(B. subtilis)(8样本)和暹罗芽胞杆菌(B. siamensis)(5样本)。第Ⅲ类为少分布种类, 其余的32个种分布在1-4个空间样本中。特别注意地是类芽胞杆菌和短芽胞杆菌在这都属于少分布种类, 常见的蜡状芽胞杆菌和枯草芽胞杆菌属于寡分布类型的种类。
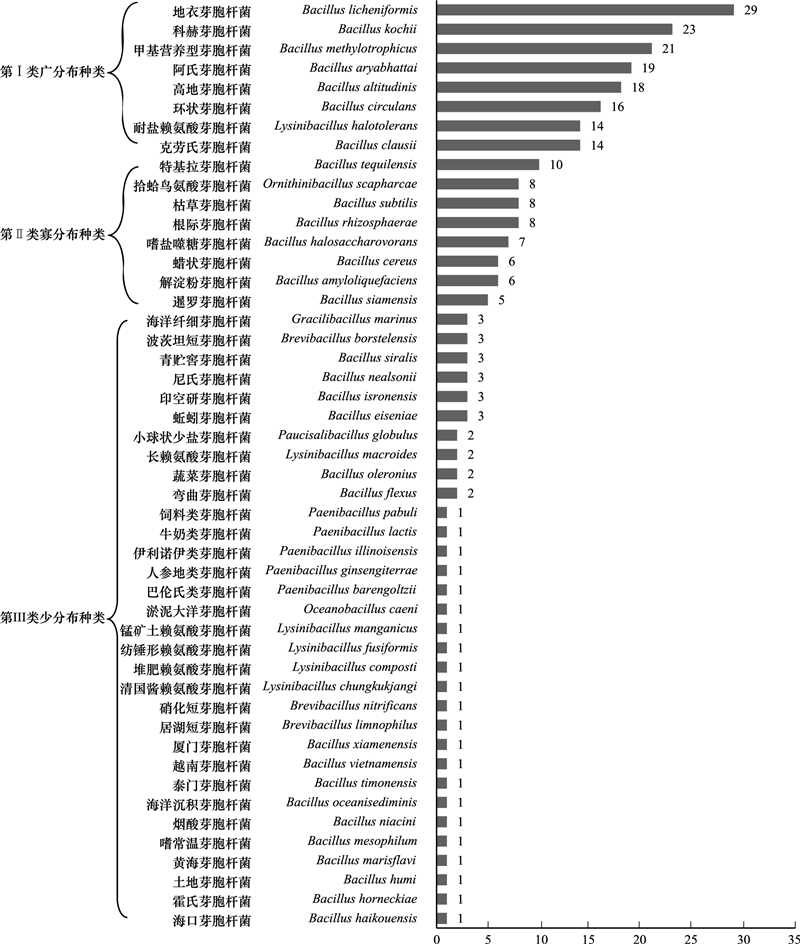
|
| 图 3 微生物发酵床空间样本芽胞杆菌出现频次 Fig. 3 Occurrence frequency of Bacillus-like in the piggery bedding |
基于表 3统计, 微生物发酵床芽胞杆菌数量分布结果见图 4。微生物发酵床48种芽胞杆菌数量最大值94.11×106个/g, 最小值为0.1×106个/g, 平均值为8.96×106个/g。根据数量分布可将其分为4类, 第Ⅰ类为高含量组, 优势种群, 占比16.7%, 数量分布在(17-94)×106个/g之间, 属于该类的芽胞杆菌有环状芽胞杆菌、地衣芽胞杆菌、阿氏芽胞杆菌、甲基营养型芽胞杆菌、人参土芽胞杆菌(Paenibacillus ginsengiterrae)、柯赫芽胞杆菌、高地芽胞杆菌和嗜盐噬糖芽胞杆菌;第Ⅱ类为中含量组, 常见种群, 占比14.6%, 数量分布在(8-13)×106个/g之间, 属于该类的芽胞杆菌包含特基拉芽胞杆菌、克劳氏芽胞杆菌、尼氏芽胞杆菌(B. nealsonii)、小球状少盐芽胞杆菌(Paucisalibacillus globulus)、伊利诺伊类芽胞杆菌(Paenibacillus illinoisensis)、枯草芽胞杆菌和耐盐赖氨酸芽胞杆菌;第Ⅲ类为寡含量组, 寡见种群, 占比18.76%, 数量分布在(3-5)×106个/g之间, 属于该类的为印空研芽胞杆菌(B. isronensis)、根际芽胞杆菌、解淀粉芽胞杆菌(B. amyloliquefaciens)、海洋纤细芽胞杆菌(Gracilibacillus marinus)、蔬菜芽胞杆菌(Bacillus oleronius)、拾蛤鸟氨酸芽胞杆菌(Or. scapharcae)、青贮窖芽胞杆菌(Bacillus siralis)、蜡状芽胞杆菌和堆肥赖氨酸芽胞杆菌(Lysinibacillus composti);第Ⅳ类为低含量组, 偶见种群, 占比50%, 数量分布在(2-0.1)×106个/g之间, 属于该类的芽胞杆菌有蜡状芽胞杆菌、暹罗芽胞杆菌(Bacillus siamensis)、海口芽胞杆菌(B. haikouensis)、霍氏芽胞杆菌(Bacillus horneckiae)、土地芽胞杆菌(Bacillus humi)、嗜常温芽胞杆菌(Bacillus mesophilum)、越南芽胞杆菌(Bacillus vietnamensis)、清国酱赖氨酸芽胞杆菌(Lysinibacillus chungkukjangi)、波茨坦短芽胞杆菌(Brevibacillus borstelensis)、锰矿土赖氨酸芽胞杆菌(Lysinibacillus manganicus)、烟酸芽胞杆菌(Bacillus niacini)、居湖短芽胞杆菌(Brevibacillus limnophilus)、硝化短芽胞杆菌(Brevibacillus nitrificans)、长赖氨酸芽胞杆菌(L. macroides)、淤泥大洋芽胞杆菌(Oceanibacillus caeni)、饲料类芽胞杆菌(Paenibacillus pabuli)、牛奶类芽胞杆菌(Paenibacillus lactis)、弯曲芽胞杆菌(B. flexus)、黄海芽胞杆菌(B. marisflavi)、海床类芽胞杆菌(B. oceanisediminis)、泰门芽胞杆菌(B. timonensis)、厦门芽胞杆菌(B. xiamenensis)、纺锤形赖氨酸芽胞杆菌(L. fusiformis)和巴伦氏类芽胞杆菌(Paenibacillus barengoltzii)。
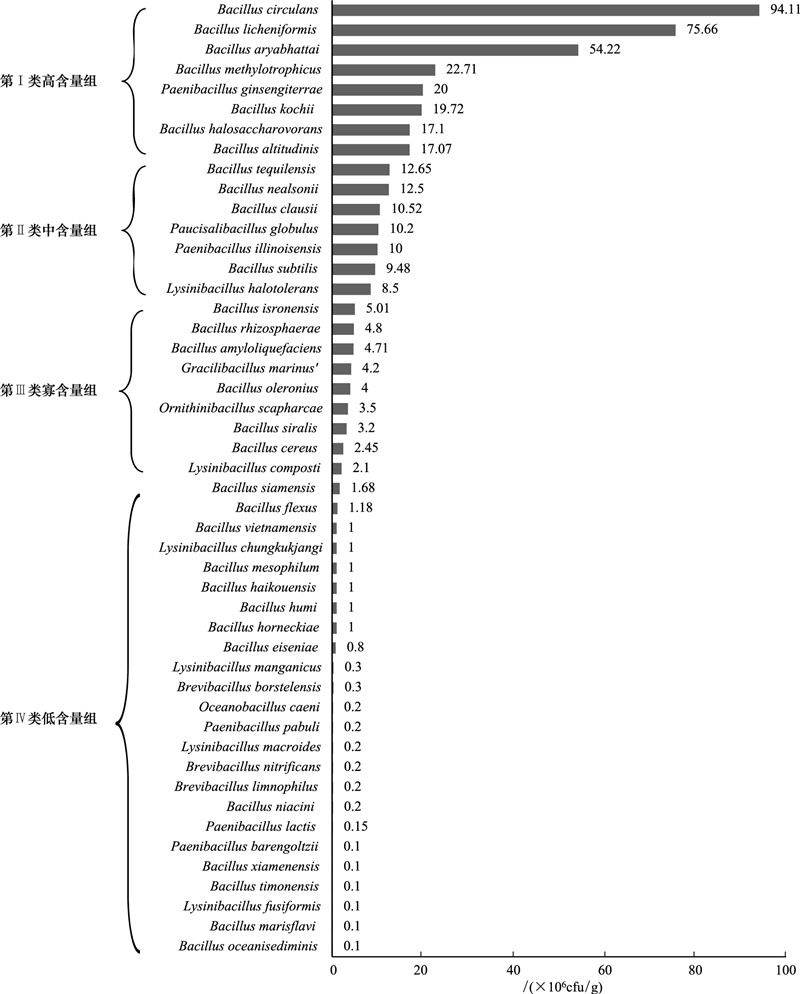
|
| 图 4 微生物发酵床芽胞杆菌数量分布多样性 Fig. 4 Distribution diversity of Bacillus-like species in the piggery bedding |
基于表 3, 将4横8纵分割的32个空间样本单元内的各芽胞杆菌数量按单元分别作总和统计, 考察芽胞杆菌作为一个种群在发酵床的空间分布型, 统计结果见表 4。结果表明:微生物发酵床空间样本单元芽胞杆菌数量分布差异很大, 其中样本中数量最高的是97.6×106个/g, 位于空间样本M-4-1, 最低的是0.800×106个/g, 位于空间样本M-7-4, 平均值为13.77×106个/g。利用表 3数据统计空间分布型指数见表 5。空间分布型的聚集度测定结果表明, 芽胞杆菌I指标=22.6183 > 0, 为聚集分布;平均拥挤度(m*/m)指标=2.4212 > 1, 为聚集分布;CA指标=1.4212 > 0, 为聚集分布;扩散系数(C)=23.6183 > 1, 为聚集分布;负二项分布K指标=1.0778 > 0, 为聚集分布, 表明芽胞杆菌在发酵床中分布为聚集分布。
| 采样位置 Location | 列坐标 Column coordinate/(×106个/g) | 平均值 Average | 方差 Variance | ||||||||
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | ||||
| 行坐标 Row-coordinate | 1 | 19.4000 | 19.5000 | 18.6000 | 3.0000 | 6.4000 | 12.5900 | 40.0000 | 4.8800 | 15.5463 | 172.6288 |
| 2 | 16.9000 | 14.1000 | 21.4000 | 30.5000 | 1.4900 | 4.8000 | 4.8000 | 3.4000 | 12.1738 | 157.3118 | |
| 3 | 29.9000 | 15.7000 | 5.8000 | 5.7600 | 7.6000 | 5.0300 | 4.4000 | 1.7800 | 9.4963 | 85.1001 | |
| 4 | 97.6000 | 20.6000 | 2.1000 | 3.7700 | 2.4200 | 8.1000 | 0.8000 | 7.5000 | 17.8613 | 1142.4909 | |
| 样方行 Sample row | 拥挤度m* Congestion degree | I指标I index | m*/m指标m*/m index | CA指标CA index | 扩散系数C Diffusion coefficient | K指标K index |
| 1 | 25.8085 | 9.7499 | 1.6071 | 0.6071 | 10.7499 | 1.6471 |
| 2 | 24.1162 | 10.9300 | 1.8289 | 0.8289 | 11.9300 | 1.2064 |
| 3 | 17.4526 | 8.0706 | 1.8602 | 0.8602 | 9.0706 | 1.1625 |
| 4 | 79.9375 | 61.7225 | 4.3886 | 3.3886 | 62.7225 | 0.2951 |
| 平均值 Average | 36.8287 | 22.6183 | 2.4212 | 1.4212 | 23.6183 | 1.0778 |
研究拥挤度(m*)与平均值(m)之间的关系, 用m*-m回归分析, 建立m*-m回归式m*=a+bm, 结果表明, m*-m回归方程(IWAO)为m*=-47.1208+5.9076m, r=0.7795;a=-47.1208 < 0, 表明芽胞杆菌微生物个体群之间相互排斥, b=5.9076 > 1, 表明芽胞杆菌空间分布型为聚集分布。运用幂法则分析表明, Taylor幂法则方程为lg(s)=-1.2876+3.1966lg(x), r=0.8233, b=3.1966 > 1, 为聚集分布。
综上所述, 养猪微生物发酵床芽胞杆菌空间分布为聚集分布。
2.4 微生物发酵床芽胞杆菌多样性指数 2.4.1 微生物发酵床芽胞杆菌空间分布多样性基于表 4统计, 芽胞杆菌总体多样性指数结果见表 6。芽胞杆菌含量范围为(0.01-9.411)×107个/g;48种芽胞杆菌总含量达4.41×108个/g;丰富度指数(D)、优势度指数(λ)、Shannon-Wiener指数(H′)、均匀度指数(J′)分别为10.49276、0.26342、1.3589、0.98027。芽胞杆菌数量达1.5×107个/g以上的种类有环状芽胞杆菌(9.411×107个/g)、地衣芽胞杆菌(7.566×107个/g)、阿氏芽胞杆菌(5.4223×107个/g)、甲基营养型芽胞杆菌(2.271×107个/g)、人参土芽胞杆菌(2.000×107个/g)、柯赫芽胞杆菌(1.972×107个/g)、嗜盐噬糖芽胞杆菌(1.710×107个/g)。
| 项目 Item | 数量 Account |
| 空间样方数 Samples | 32 |
| 芽胞杆菌含量 Content of Bacillus /(×107个/g) | 4.4062 |
| 丰富度指数 Richness index(D) | 0.4928 |
| 优势度指数 Simpson diversity index(λ) | 0.2634 |
| Shannon-Wiener指数 Shannon-Wiener index(H′) | 1.3589 |
| 均匀度指数 Pielou′s evenness index(J′) | 0.9803 |
芽胞杆菌种类多样性指数基于表 3计算, 结果见表 7。芽胞杆菌Shannon-Wiener指数范围0-2.88, 最大的种类为地衣芽胞杆菌(2.88), 最小的种类为海口芽胞杆菌、黄海芽胞杆菌、海洋芽胞杆菌等22种芽胞杆菌;优势度指数范围0.08-1, 最大的种类是黄海芽胞杆菌、海洋芽胞杆菌等22种芽胞杆菌, 最小的种类是地衣芽胞杆菌(0.08);Hill指数范围1-17.75, 最大的种类是地衣芽胞杆菌(17.75), 最小的种类是海口芽胞杆菌、黄海芽胞杆菌、海洋芽胞杆菌等22种芽胞杆菌;芽胞杆菌丰富度指数范围0-7.71, 最大的种类是, 最小的种类为海口芽胞杆菌、黄海芽胞杆菌、海洋芽胞杆菌等22种芽胞杆菌;均匀度指数范围0-1, 最大的种类是长赖氨酸芽胞杆菌和波茨坦短芽胞杆菌, 最小的种类是黄海芽胞杆菌、海洋芽胞杆菌等22种芽胞杆菌。
| 种类 Species | 分布频次 Frequency distribution | 含量 Content/(107个/g) | 丰富度指数(D) Richness index | 均匀度指数(J′) Pielou′s evenness index | Shannon-Wiener指数(H′) Shannon-Wiener index | 优势度指数(λ) Simpson index | Hill指数(N1) Hill index |
| Bacillus licheniformis | 29 | 7.566 | 6.47 | 0.85 | 2.88 | 0.08 | 17.75 |
| Bacillus kochii | 23 | 1.972 | 7.38 | 0.81 | 2.55 | 0.10 | 12.85 |
| Bacillus methylotrophicus | 21 | 2.271 | 6.40 | 0.75 | 2.27 | 0.16 | 9.72 |
| Bacillus aryabhattai | 19 | 5.322 | 4.53 | 0.70 | 2.06 | 0.20 | 7.84 |
| Bacillus altitudinis | 18 | 1.707 | 5.99 | 0.82 | 2.36 | 0.12 | 10.59 |
| Bacillus circulans | 16 | 9.411 | 3.30 | 0.54 | 1.50 | 0.35 | 4.49 |
| Bacillus clausii | 14 | 10.52 | 5.52 | 0.87 | 2.30 | 0.12 | 10.01 |
| Lysinibacillus halotolerans | 14 | 0.850 | 6.07 | 0.86 | 2.28 | 0.13 | 9.77 |
| Bacillus tequilensis | 10 | 12.65 | 3.55 | 0.78 | 1.80 | 0.19 | 6.07 |
| Bacillus rhizosphaerae | 8 | 0.480 | 4.46 | 0.74 | 1.55 | 0.27 | 4.70 |
| Bacillus subtilis | 8 | 0.948 | 3.11 | 0.71 | 1.47 | 0.33 | 4.34 |
| Ornithinibacillus scapharcae | 8 | 0.350 | 5.59 | 0.82 | 1.71 | 0.23 | 5.55 |
| Bacillus halosaccharovorans | 7 | 1.710 | 2.11 | 0.81 | 1.57 | 0.26 | 4.80 |
| Bacillus amyloliquefaciens | 6 | 0.471 | 3.23 | 0.63 | 1.13 | 0.39 | 3.09 |
| Bacillus cereus | 6 | 0.245 | 5.58 | 0.69 | 1.23 | 0.35 | 3.42 |
| Bacillus siamensis | 5 | 0.168 | 7.71 | 0.62 | 1.00 | 0.45 | 2.72 |
| Bacillus isronensis | 3 | 0.501 | 1.24 | 0.47 | 0.51 | 0.68 | 1.67 |
| Bacillus eiseniae | 3 | 0.080 | 0.00 | 0.82 | 0.90 | 0.47 | 2.46 |
| Bacillus nealsonii | 3 | 1.250 | 0.79 | 0.51 | 0.56 | 0.67 | 1.74 |
| Brevibacillus borstelensis | 3 | 0.030 | 0.00 | 1.00 | 1.10 | 0.33 | 3.00 |
| Bacillus siralis | 3 | 0.320 | 1.72 | 0.76 | 0.83 | 0.49 | 2.29 |
| Gracilibacillus marinus | 3 | 0.420 | 1.39 | 0.20 | 0.22 | 0.91 | 1.25 |
| Bacillus flexus | 2 | 0.118 | 6.04 | 0.62 | 0.43 | 0.74 | 1.53 |
| Lysinibacillus macrolides | 2 | 0.020 | 0.00 | 1.00 | 0.69 | 0.50 | 2.00 |
| Paucisalibacillus globulus | 2 | 1.020 | 0.43 | 0.14 | 0.10 | 0.96 | 1.10 |
| Bacillus oleronius | 2 | 0.400 | 0.72 | 0.81 | 0.56 | 0.63 | 1.75 |
| Bacillus haikouensis | 1 | 0.100 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Bacillus horneckiae | 1 | 0.100 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Bacillus humi | 1 | 0.100 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Bacillus marisflavi | 1 | 0.010 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Bacillus mesophilum | 1 | 0.100 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Bacillus niacini | 1 | 0.020 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Bacillus oceanisediminis | 1 | 0.010 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Bacillus timonensis | 1 | 0.100 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Bacillus vietnamensis | 1 | 0.100 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Bacillus xiamenensis | 1 | 0.010 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Brevibacillus limnophilus | 1 | 0.020 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Brevibacillus nitrificans | 1 | 0.020 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Lysinibacillus chungkukjangi | 1 | 0.100 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Lysinibacillus composti | 1 | 0.210 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Lysinibacillus fusiformis | 1 | 0.010 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Lysinibacillus manganicus | 1 | 0.030 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Oceanobacillus caeni | 1 | 0.020 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Paenibacillus barengoltzii | 1 | 0.010 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Paenibacillus ginsengiterrae | 1 | 2.000 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Paenibacillus illinoisensis | 1 | 1.000 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Paenibacillus lactis | 1 | 0.015 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
| Paenibacillus pabuli | 1 | 0.020 | 0.00 | 0.00 | 0.00 | 1.00 | 1.00 |
芽胞杆菌多样性指数聚类分析, 根据表 7数据, 以欧式距离为尺度, 用类平均法进行系统聚类, 作图 5。当λ=17时, 可将其分为2类:
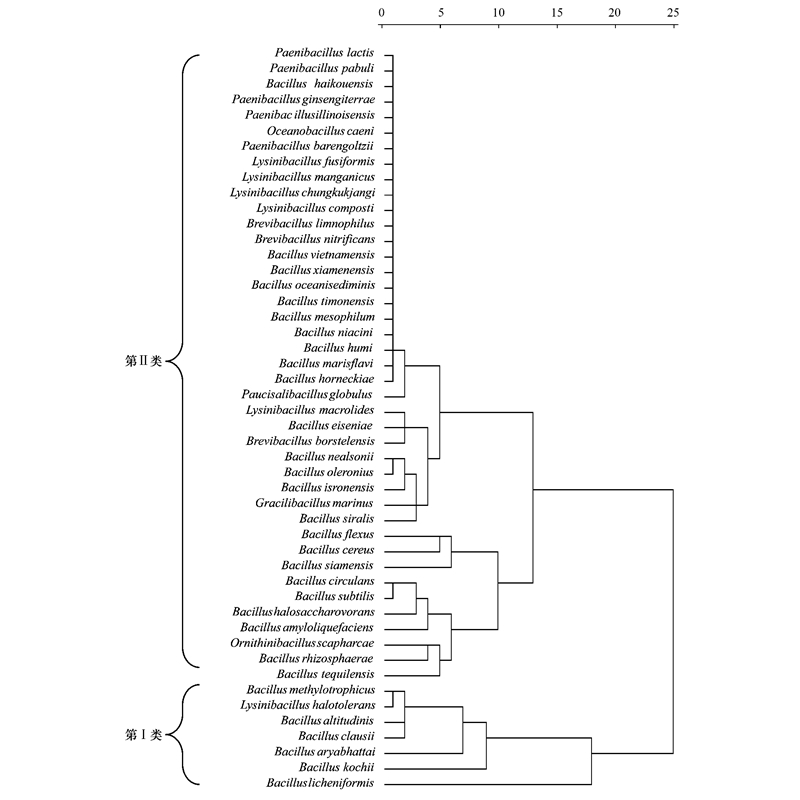
|
| 图 5 芽胞杆菌种类分布聚类分析 Fig. 5 Distribution analysis of Bacillus-like speceis |
第Ⅰ类为高丰富度高含量类型, 包括了7个种, 即甲基营养型芽胞杆菌、耐盐赖氨酸芽胞杆菌、高地芽胞杆菌、克劳氏芽胞杆菌、阿氏芽胞杆菌、科赫芽胞杆菌、地衣芽胞杆菌。
第Ⅱ类为较低丰富度较低含量类型, 该类分为2个亚类, 第一亚类为中等丰富度中等含量的类型, 包括了10个种, 即弯曲芽胞杆菌、蜡状芽胞杆菌、暹罗芽胞杆菌、环状芽胞杆菌、枯草芽胞杆菌、嗜盐噬糖芽胞杆菌、解淀粉芽胞杆菌、拾蛤鸟氨酸芽胞杆菌、根际芽胞杆菌、特基拉芽胞杆菌。第二亚类为低丰富度低含量的类型, 其余的31个种。
3 讨论微生物发酵床养猪是近几年发展起来的一种现代化生态养猪模式和技术体系, 目前在我国各地得到了广泛应用。微生物发酵床能高效分解猪粪、消除恶臭, 并最终将猪粪转化形成人工腐殖质, 而后者又可以进一步研制成微生物肥料、生物农药、生物育苗与栽培基质等高值产品, 因此, 同时实现了养猪污染的原位微生物治理和猪粪的资源化利用。在此过程中, 微生物起到了关键作用。前期, 作者所在课题组利用高通量宏基因组学技术分析了发酵床垫料中的微生物多样性, 发现发酵床垫料中的微生物多样性极其丰富, 而且存在着种类多样的芽胞杆菌(Bacillus-like)(数据待发表)。
芽胞杆菌是一类重要的微生物资源, 种类繁多, 可以产生多种具有抑菌功能物质, 如Ⅰ和Ⅱ类羊毛硫抗生素(lantibiotics)[27]。因此, 芽胞杆菌对猪群具有抗菌防病功能, 具有作为益生菌开发的潜力。Manhar等研究发现枯草芽胞杆菌AMS6具有耐强酸(pH 2.0) 和胆汁(0.3%)、高抗菌活性、抗病原菌黏附和较强的纤维素降解能力等益生菌特性, 作为饲料添加剂可以增强动物纤维素消化和肠道健康[28]。Lopetuso等综述了益生克劳氏芽胞杆菌的临床应用效果、益生菌与肠道屏障相互作用、重建肠道平衡的可能作用机理[29]。大量研究表明, 芽胞杆菌还具有高效降解猪粪中的有机物、抗生素、药物等及除臭、减少氨气排放等功能[30-34]。
尽管发酵床垫料芽胞杆菌种群及其空间分布的研究较少报道, 但国内外研究者在其他含有猪粪环境(如猪粪耗氧、厌氧堆肥)开展的相关研究也发现了类似结果。如, Yi等利用可培养、限制性片段长度多态性(PCR-RFLP)和变性剂梯度凝胶电泳(PCR-DGGE)法分析发现猪粪堆肥过程中可培养芽胞杆菌的数量和时空分布特性随着温度的改变发生了显著地变化, 枯草芽胞杆菌和蜡样芽胞杆菌为优势种, 在各阶段, 芽胞杆菌的多样性均较低;在高温阶段堆肥各层的芽胞杆菌含量最高, 而在各阶段的堆肥中间层含量均最低[35]。Guo等采用PCR-RFLP法研究发现猪粪堆肥降温阶段堆肥中间层芽胞杆菌属是优势种群, 底层优势种群则为梭菌属[36]。Li等分析发现牛粪高温堆肥阶段芽胞杆菌属、地芽胞杆菌属(Geobacillus)和尿素芽胞杆菌属(Ureibacillus)是优势属, 热脱氮地芽胞杆菌(G. thermodenitrificans)是优势种;在高温后期, 就地堆肥地芽胞杆菌(G. toebii)和领地尿素芽胞杆菌(U. terrenus)是优势种[37]。Guo等也发现猪粪高温堆肥过程中芽胞杆菌属是各层最常见种群[38]。He等研究表明芽胞杆菌种类在鸡粪堆肥各个阶段均有较高的含量[39]。本文研究结果与上述不完全一致, 发酵床垫料的优势属与上述相同亦为芽胞杆菌属, 但优势种群不一致。本研究亦获得了猪粪堆肥过程中的优势种枯草芽胞杆菌和蜡质芽胞杆菌, 但该2种芽胞杆菌非发酵床的优势种群。牛粪高温堆肥的优势种未在养猪发酵床垫料中发现, 其种群结构与猪粪的差异很大。本研究发现养猪发酵床中仅次于芽胞杆菌属的种群为赖氨酸芽胞杆菌属, 而该属在猪粪和牛粪堆肥过程皆不为优势属。微生物发酵床芽胞杆菌种类分布多样性指数不均一, 可能与发酵床的发酵等级和芽胞杆菌种类对发酵床成分的适应性和利用有关。有些芽胞杆菌种类出现富集成为优势种, 几乎存在于整个发酵床, 如地衣芽胞杆菌, 但有些适应力弱导致其数量少, 仅在某些位置存在。这说明垫料发酵程度对微生物种类分布有很大的影响。薛超等发现连作会造成土壤微生物的选择适应性, 出现一些种群富集, 而一些种群数量降低的现象[40], 进而导致不同茬次营养基质中微生物对碳源数量的利用能力出现显著差异[41]。微生物发酵床主要成分为椰糠和谷壳等高纤维材料, 所构建出的生存环境适合于能降解纤维的微生物生存。叶少文等分析了微生物发酵床垫料不同深度酶活, 发现垫料中纤维素酶活和半纤维素酶活性很高, 揭示了一类微生物的生存空间[42]。
在养猪微生物发酵床芽胞杆菌优势种群中, 地衣芽胞杆菌能够产生纤维素酶、果胶酶、淀粉酶、蛋白酶等[43-44], 这些酶作用能为地衣芽胞杆菌的生存提供碳源和氮源能量来源, Mc Carthy等发现地衣芽胞杆菌是猪粪固体堆肥过程的优势可培养微生物, 这就印证了本研究中地衣芽胞杆菌是养猪发酵床垫料中优势种群结果的可靠性[45]。Islas-Espinoza等发现地衣芽胞杆菌能降解猪粪中难降解的合成抗生素磺胺类药物, 表明养猪微生物发酵床中大量地衣芽胞杆菌的存在在猪粪降解过程扮演着重要角色[32]。阿氏芽胞杆菌具有坚强的生命力, 分布范围广。优势种高地芽胞杆菌是一种多功能芽胞杆菌, 能产生碱性蛋白酶[46], 可以用于固体发酵的添加菌剂[47]。优势种阿氏芽胞杆菌和高地芽胞杆菌的强大存在可能与发酵床的发酵健康程度密切相关。环状芽胞杆菌在微生物发酵床的广泛存在与发酵床具有生防抗病作用有一定的相关性。环状芽胞杆菌作为发酵床的优势种群是一种重要的动物益生菌, 它不仅能产生抑制猪病原菌的物质和表面活性剂[48], 同时, 还可以作为堆肥发酵程度检测指标[49]。优势种嗜盐噬糖芽胞杆菌源于盐碱土壤分离, 关于此菌报道甚少, 属于芽胞杆菌脂肪酸群Ⅲ, 特性就是可以在偏盐碱性环境生存[50]。蓝江林等发现微生物发酵床垫料含盐量很高, 且pH普遍在8.0-9.0之间, 随着发酵时间的增加, 垫料中的盐浓度和pH皆呈上升状态[51]。因此, 发酵床垫料的高盐碱度很可能是导致耐(嗜)盐碱芽胞杆菌种类存在的主要原因。优势种科赫芽胞杆菌文献报道较少, 该菌最初是从乳制品分离获得, 有着非常宽广的生长条件, 如温度范围10-40 ℃、酸碱度范围pH 6.0-10.5、盐度范围0%-10% NaCl[52], 该菌的生长特性适应微生物发酵床发酵过程生长条件极端变化而广泛分布。
微生物发酵床含有丰富的微生物资源, 通过微生物发酵床芽胞杆菌种群多样性分析, 获得了大量的芽胞杆菌资源, 这为进一步挖掘新功能和新物种提供了重要来源。关于芽胞杆菌种群变化与微生物发酵床生态功能的关联有待于进一步探讨和研究。
| [1] | 包武, 李元钦, 钱午巧, 蔡元呈, 邹以强, 朱光荣, 陈钧生. 福建省生猪养殖污染状况及治理对策. 安徽农学通报, 2013, 19(18): 90, 105–94, 105. |
| [2] | 郑雪芳, 刘波, 林营志, 蓝江林, 刘丹莹. 利用磷脂脂肪酸生物标记分析猪舍基质垫层微生物亚群落的分化. 环境科学学报, 2009, 29(11): 2306–2317. |
| [3] | 赵冬青, 吴建平, 段淇斌. 生物发酵床养猪效能和猪肉营养成分影响研究. 草业学报, 2012, 21(2): 212–218. DOI:10.11686/cyxb20120227 |
| [4] | 李买军, 马晗, 郭海宁, 尹微琴, 王小治, 封克, 顾洪如. 养猪场发酵床垫料及下层土壤中酶活性变化特性研究. 农业环境科学学报, 2014, 33(4): 777–782. DOI:10.11654/jaes.2014.04.023 |
| [5] | 张履祥. 沈氏农书. 北京: 中华书局, 1956: 50–50. |
| [6] | Gadd J. Unnel housing of pigs in Livestock Environment Ⅳ//Fourth International Symposium. Michigan: American Society of Agricultural Enginneers, 1993: 1040–1048. |
| [7] | Tiquia S M, Tam N F Y, Hodgkiss I J. Effects of turning frequency on composting of spent pig-manure sawdust litter. Bioresource Technology, 1997, 62(1/2): 37–42. |
| [8] | Kaufmann R, Heller W, Bieri M. Nutrient balance in the biobed system for fattening pigs. Agrarforschung, 1997, 4(1): 25–28. |
| [9] | Connor M L. Update on alternative housing systems for pigs. Manitoba Swine Seminar Proceedings, 1995(8): 93–96. |
| [10] | Margeta V, Kralik G, Hanžek D, Margeta P. Deep-litter pigkeeping-Croatian perspectives. Acta Agraria Kaposvariensis, 2010, 14(2): 209–213. |
| [11] | Knecht D, Popiołek M, Zaleśny G. Does meatiness of pigs depend on the level of gastro-intestinal parasites infection?. Preventive Veterinary Medicine, 2011, 99(2/4): 234–239. |
| [12] | 刘波, 蓝江林, 唐建阳, 史怀. 微生物发酵床菜猪大栏养殖猪舍结构设计. 福建农业学报, 2014, 29(5): 505–509. |
| [13] | 刘波, 郑雪芳, 朱昌雄, 蓝江林, 林营志, 林斌, 叶耀辉. 脂肪酸生物标记法研究零排放猪舍基质垫层微生物群落多样性. 生态学报, 2008, 28(11): 5488–5498. DOI:10.3321/j.issn:1000-0933.2008.11.033 |
| [14] | 郑雪芳, 刘波, 林营志, 蓝江林, 刘丹莹. 利用磷脂脂肪酸生物标记分析猪舍基质垫层微生物亚群落的分化. 环境科学学报, 2009, 29(11): 2306–2317. |
| [15] | 张学峰, 周贤文, 陈群, 魏炳栋, 姜海龙. 不同深度垫料对养猪土著微生物发酵床稳定期微生物菌群的影响. 中国兽医学报, 2013, 33(9): 1458–1462. |
| [16] | 王迪. 猪用生物发酵床垫料中微生物群落多样性变化及芽孢杆菌分离与鉴定[D]. 武汉: 华中农业大学, 2012. |
| [17] | 林莉莉, 姜雪, 冯聪, 范亮, 王际辉, 张彧. 发酵床养猪猪舍环境与猪体表微生物分布状况的研究. 安徽农业科学, 2010, 38(34): 19530–19532. DOI:10.3969/j.issn.0517-6611.2010.34.113 |
| [18] | 张庆宁, 胡明, 朱荣生, 任相全, 武英, 王怀忠, 刘玉庆, 王述柏. 生态养猪模式中发酵床优势细菌的微生物学性质及其应用研究. 山东农业科学, 2009(4): 99–105. |
| [19] | 刘让, 陈少平, 张鲁安, 苏贵成, 李岩. 生态养猪发酵益生菌的分离鉴定及体外抑菌试验研究. 国外畜牧学:猪与禽, 2010, 30(2): 62–64. |
| [20] | 郑雪芳, 刘波, 蓝江林, 苏明星, 卢舒娴, 朱昌雄. 微生物发酵床对猪舍大肠杆菌病原生物防治作用的研究. 中国农业科学, 2011, 44(22): 4728–4739. DOI:10.3864/j.issn.0578-1752.2011.22.022 |
| [21] | 毕小艳. 猪用发酵床垫料中微生物动态变化及对猪免疫力的影响[D]. 长沙: 湖南农业大学, 2011. |
| [22] | 卢舒娴. 养猪发酵床垫料微生物群落动态及其对猪细菌病原生防作用的研究[D]. 福州: 福建农林大学, 2011. |
| [23] | 张金龙. 猪发酵床养殖中芽孢杆菌菌株的筛选、鉴定及产蛋白酶条件的优化[D]. 雅安: 四川农业大学, 2009. |
| [24] | Liu B, Liu G H, Hu G P, Chen M C. Bacillus mesonae sp. nov., isolated from the root of Mesona chinensis. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(10): 3346–3352. |
| [25] | Kim O S, Cho Y J, Lee K, Yoon S H, Kim M, Na H, Park S C, Jeon Y S, Lee J H, Yi H, Won S, Chun J. Introducing EzTaxon-e: a prokaryotic 16S rRNA Gene sequence database with phylotypes that represent uncultured species. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(3): 716–721. |
| [26] | Tindall B J, Rosselló-Móra R, Busse H J, Ludwig W, Kämpfer P. Notes on the characterization of prokaryote strains for taxonomic purposes. International Journal of Systematic and Evolutionary Microbiology, 2010, 60(1): 249–266. DOI:10.1099/ijs.0.016949-0 |
| [27] | Barbosa J, Caetano T, Mendo S. Class Ⅰ and class Ⅱ lanthipeptides produced by Bacillus spp. Journal of Natural Products, 2015, 78(11): 2850–2866. DOI:10.1021/np500424y |
| [28] | Manhar A K, Bashir Y, Saikia D, Nath D, Gupta K, Konwar B K, Kumar R, Namsa N D, Mandal M. Cellulolytic potential of probiotic Bacillus subtilis AMS6 isolated from traditional fermented soybean (Churpi): an in-vitro study with regards to application as an animal feed additive. Microbiological Research, 2016, 186-187: 62–70. DOI:10.1016/j.micres.2016.03.004 |
| [29] | Lopetuso L R, Scaldaferri F, Franceschi F, Gasbarrini A. Bacillus clausii and gut homeostasis: state of the art and future perspectives. Expert Review of Gastroenterology & Hepatology, 2016, 10(8): 943–948. |
| [30] | Maeda K, Hanajima D, Toyoda S, Yoshida N, Morioka R, Osada T. Microbiology of nitrogen cycle in animal manure compost. Microbial Biotechnology, 2011, 4(6): 700–709. DOI:10.1111/mbt.2011.4.issue-6 |
| [31] | Kuroda K, Waki M, Yasuda T, Fukumoto Y, Tanaka A, Nakasaki K. Utilization of Bacillus sp. strain TAT105 as a biological additive to reduce ammonia emissions during composting of swine feces. Bioscience, Biotechnology, and Biochemistry, 2015, 79(10): 1702–1711. DOI:10.1080/09168451.2015.1042831 |
| [32] | Islas-Espinoza M, Reid B J, Wexler M, Bond P L. Soil bacterial consortia and previous exposure enhance the biodegradation of sulfonamides from pig manure. Microbial Ecology, 2012, 64(1): 140–151. DOI:10.1007/s00248-012-0010-5 |
| [33] | Gutarowska B, Matusiak K, Borowski S, Rajkowska A, Brycki B. Removal of odorous compounds from poultry manure by microorganisms on perlite-bentonite carrier. Journal of Environmental Management, 2014, 141: 70–76. |
| [34] | Hanajima D, Haruta S, Hori T, Ishii M, Haga K, Igarashi Y. Bacterial community dynamics during reduction of odorous compounds in aerated pig manure slurry. Journal of Applied Microbiology, 2009, 106(1): 118–129. DOI:10.1111/jam.2008.106.issue-1 |
| [35] | Yi J, Wu H Y, Wu J, Deng C Y, Zheng R, Chao Z. Molecular phylogenetic diversity of Bacillus community and its temporal-spatial distribution during the swine manure of composting. Applied Microbiology and Biotechnology, 2012, 93(1): 411–421. DOI:10.1007/s00253-011-3425-x |
| [36] | Guo Y, Zhang J L, Yan Y F, Wu J, Zhu N W, Deng C Y. Molecular phylogenetic diversity and spatial distribution of bacterial communities in cooling stage during swine manure composting. Asian-Australasian Journal of Animal Sciences, 2015, 28(6): 888–895. DOI:10.5713/ajas.14.0882 |
| [37] | Li R, Li L Z, Huang R, Sun Y F, Mei X L, Shen B, Shen Q R. Variations of culturable thermophilic microbe numbers and bacterial communities during the thermophilic phase of composting. World Journal of Microbiology and Biotechnology, 2014, 30(6): 1737–1746. DOI:10.1007/s11274-013-1593-9 |
| [38] | Guo Y, Zhang J L, Deng C Y, Zhu N W. Spatial heterogeneity of bacteria: evidence from hot composts by culture-independent analysis. Asian-Australasian Journal of Animal Sciences, 2012, 25(7): 1045–1054. DOI:10.5713/ajas.2011.11341 |
| [39] | He Y M, Xie K Z, Xu P Z, Huang X, Gu W J, Zhang F B, Tang S H. Evolution of microbial community diversity and enzymatic activity during composting. Research in Microbiology, 2013, 164(2): 189–198. DOI:10.1016/j.resmic.2012.11.001 |
| [40] | 薛超, 黄启为, 凌宁, 高雪莲, 曹云, 赵青云, 何欣, 沈其荣. 连作土壤微生物区系分析、调控及高通量研究方法. 土壤学报, 2011, 48(3): 612–618. DOI:10.11766/trxb201007120287 |
| [41] | 邹春娇, 齐明芳, 马建, 武春成, 李天来. Biolog-ECO解析黄瓜连作营养基质中微生物群落结构多样性特征. 中国农业科学, 2016, 49(5): 942–951. DOI:10.3864/j.issn.0578-1752.2016.05.014 |
| [42] | 叶少文, 车建美, 刘波, 王阶平, 陈倩倩, 刘国红, 陈峥, 唐建阳. 微生物发酵床垫料酶活性变化研究. 福建农业学报, 2016, 31(1): 52–56. |
| [43] | 尹红梅, 吴迎奔, 张德元, 王震, 陈薇, 贺月林. 发酵床中耐高温地衣芽胞的分离鉴定及产酶分析. 家畜生态学报, 2012, 33(6): 97–102. |
| [44] | 孙碧玉, 邵继海, 秦普丰, 汤浩, 黄红丽. 养猪发酵床中净水芽胞杆菌的分离及其固体发酵研究. 环境工程, 2014, 32(11): 60–63. |
| [45] | Mc Carthy G, Lawlor P G, Coffey L, Nolan T, Gutierrez M, Gardiner G E. An assessment of pathogen removal during composting of the separated solid fraction of pig manure. Bioresource Technology, 2011, 102(19): 9059–9067. DOI:10.1016/j.biortech.2011.07.021 |
| [46] | Vijay Kumar E, Srijana M, Kiran Kumar K, Harikrishna N, Reddy G. A novel serine alkaline protease from Bacillus altitudinis GVC11 and its application as a dehairing agent. Bioprocess and Biosystems Engineering, 2011, 34(4): 403–409. DOI:10.1007/s00449-010-0483-x |
| [47] | Madhuri A, Nagaraju B, Harikrishna N, Reddy G. Production of alkaline protease by Bacillus altitudinis GVC11 using castor husk in solid-state fermentation. Applied Biochemistry and Biotechnology, 2012, 167(5): 1199–1207. DOI:10.1007/s12010-012-9570-6 |
| [48] | Mukherjee S, Das P, Sivapathasekaran C, Sen R. Antimicrobial biosurfactants from marine Bacillus circulans : extracellular synthesis and purification. Letters in Applied Microbiology, 2009, 48(3): 281–288. DOI:10.1111/lam.2009.48.issue-3 |
| [49] | Subba Rao C, Madhavendra S S, Sreenivas Rao R, Hobbs P J, Prakasham R S. Studies on improving the immobilized bead reusability and alkaline protease production by isolated immobilized Bacillus circulans (MTCC 6811) using overall evaluation criteria. Applied Biochemistry and Biotechnology, 2008, 150(1): 65–83. DOI:10.1007/s12010-008-8147-x |
| [50] | 刘波, 刘国红, 林乃铨. 基于脂肪酸生物标记芽胞杆菌属种类的系统发育. 微生物学报, 2014, 54(2): 139–158. |
| [51] | 蓝江林, 宋泽琼, 刘波, 史怀, 黄素芳, 林娟. 微生物发酵床不同腐熟程度垫料主要理化特性. 福建农业学报, 2013, 28(11): 1132–1136. DOI:10.3969/j.issn.1008-0384.2013.11.012 |
| [52] | Seiler H, Schmidt V, Wenning M, Scherer S. Bacillus kochii sp. nov., isolated from foods and a pharmaceuticals manufacturing site. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(5): 1092–1097. |
 2017, Vol. 37
2017, Vol. 37




