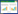文章信息
- 杨喜书, 章群, 薛丹, 吕金磊, 黄镇宇, 卢丽锋.
- YANG Xishu, ZHANG Qun, XUE Dan, LÜ Jinlei, HUANG Zhenyu, LU Lifeng.
- 中国近海细鳞鯻线粒体控制区的遗传多样性
- Genetic analysis of Terapon jarbua in coastal waters of China by using mitochondrial DNA control region sequencing
- 生态学报. 2018, 38(5): 1852-1859
- Acta Ecologica Sinica. 2018, 38(5): 1852-1859
- http://dx.doi.org/10.5846/stxb201701180152
-
文章历史
- 收稿日期: 2017-01-18
- 网络出版日期: 2017-11-21
细鳞鯻(Terapon jarbua)隶属鲈形目(Percoiformes)鯻科(Teraponidae)鯻属, 是广泛分布于印度-西太平洋海域的沿岸性浅海鱼类;中国产于东海、南海及台湾海峡[1]。细鳞鯻易于海钓和捕捞, 年产量较高;肉质细腻、营养丰富, 是我国重要经济鱼类[2-3]。近些年由于过度捕捞、生境退化等因素的影响, 野生细鳞鯻种群数量严重下降。遗传多样性是物种对环境适应能力和进化潜力的反映, 同时也是制定物种保护和种质资源开发利用的基础[4]。目前国内外对于细鳞鯻的研究多集中于生理生态、毒理及系统发育等领域[5-7];关于细鳞鯻的分子遗传研究, 国外Lavergne等[8]曾用线粒体COI基因对印度洋亚丁湾种群结构进行过简单分析, 国内Liu等人[9]利用线粒体COI探讨了台湾海域细鳞鯻种群遗传多样性情况;而对中国沿海细鳞鯻种群遗传多样性和遗传结构的研究未见报道, 因此仅靠国内和国外有限的资料并不足以反映中国沿海细鳞鯻遗传分化的全貌。
线粒体DNA (mitochondrial DNA, mtDNA)具有结构简单、遵循母系遗传、进化速度快等特点, 是动物种群遗传和分子系统学研究的理想分子标记。mtDNA内不同区域的进化速率不同;其中, 细胞色素C氧化酶I基因(COI)为蛋白质编码基因, 因进化速率适中、较为保守等特点常用作DNA条形码进行物种鉴定[10];而线粒体控制区(control region, CR)为非编码区, 以其变异位点多、进化速率最快常用于分析鱼类的种内遗传分化情况[11-13]。为此本研究以线粒体控制区作为分子标记测定中国沿海5地点102尾细鳞鯻, 旨在了解中国沿海细鳞鯻的遗传多样性、遗传结构和种群历史动态, 为其种质资源的保护和开发利用提供科学依据。
1 材料与方法 1.1 实验材料样本为2011年—2015年采自中国沿海福建平潭16尾(Pingtan, 编号为PT1—16), 广东硇洲岛22尾(Naozhou Island, 编号为NZ1—22), 广东乌石镇25尾(Wushi, 编号为WS1—25), 广西合浦17尾(Hepu, 编号为HP1—17), 海南博鳌22尾(Boao, 编号为BA1—22), 共计5个地点102尾。样本均从近海作业的渔船上直接购买的野生鱼类, 采集后置于95%乙醇中固定保存。具体采样点见图 1。
取鱼背肌肉约100mg, 晾干后采用高盐法[15]提取基因组DNA。PCR引物为本实验室自行设计:CrF:5′-YHCRCCAYYRRYYCCCAAAGCT-3′, CrR:5′-TYTCACRGGGRYGCGGATACTTGC -3′。进行PCR扩增后, 将经1%的琼脂糖凝胶电泳检测条带清晰的PCR产物送至北京六合华大基因有限公司切胶纯化, 在ABI3730DNA自动测序仪上测序。
1.3 数据处理在MEGA 6.0[16]中人工校对序列, 计算碱基组成、变异位点、转换与颠转比、群体内与群体间遗传距离, 构建系统邻接树。通过DnaSP 5.0[17]计算单倍型数、核苷酸多样性(Pi)、单倍型多样性(Hd)、遗传分化系数(Fst)和基因流(Nm)值。通过TCS[18]构建单倍型网络图。根据Arlequin[19]进行分子变异分析(AMOVA)、Tajima′s D 和Fu′s Fs 中性检验, 以及核苷酸不配对分析, 获得τ值。利用公式τ=2ut和T= t ×(代时), 估算出扩张时间;其中τ为扩张时间参数;u=μk, μ为线粒体控制区的变异速率, k表示序列长度;t表示自扩张以来所经历的代数;T值即为最终所求的扩张时间, 代时为该物种的生殖周期[20]。
2 结果与分析 2.1 细鳞鯻线粒体控制区序列的遗传多样性和遗传分化经人工校对后获得102个体962bp的部分线粒体控制区序列, 其中A、T、C、G的含量分别为32.5%、31.1%、22.6%和13.8%, A+T含量(63.6%)明显高于C+G含量(36.4%)。转换/颠换比为9.14。共检测到205个变异位点, 135个简约信息位点, 定义102个单倍型(编号H1—H102), 总体单倍型多样性(haplotype diversity, Hd)为1.000, 核苷酸多样性(nucleotide diversity, Pi)为0.022。另外, 5个群体的Hd 均为1.000;Pi为0.014—0.028, 详见表 1。各地理群体内和群体间平均遗传距离范围在0.015—0.029之间(表 2)。群体间遗传分化系数(Fst)在-0.014—0.041之间(P>0.005), 表明群体间无明显遗传分化, 基因交流频繁[21-22]。
| 群体 Population |
数量 Number |
变异位点数 Variable sites |
单倍型数 Number of haplotype |
单倍型多样性 Haplotype diversity, Hd |
核苷酸多样性 Nucleotide diversity, Pi |
| 平潭 Pingtan, PT | 16 | 64 | 16 | 1.000 ± 0.022 | 0.014 ± 0.001 |
| 硇洲岛 Naozhou, NZ | 22 | 108 | 22 | 1.000 ± 0.014 | 0.024 ± 0.004 |
| 博鳌 Boao, BA | 22 | 123 | 22 | 1.000 ± 0.014 | 0.028 ± 0.004 |
| 乌石镇 Wushi, WS | 25 | 96 | 25 | 1.000 ± 0.011 | 0.021 ± 0.003 |
| 合浦 Hepu, HP | 17 | 85 | 17 | 1.000 ± 0.020 | 0.015 ± 0.004 |
| 总体 Total | 102 | 205 | 102 | 1.000 ± 0.001 | 0.022 ± 0.002 |
| 群体 Population | 平潭 PT | 合浦 HP | 硇洲岛 NZ | 乌石镇 WS | 博鳌 BA |
| 平潭 PT | 0.015 | 0.040 | 0.061 | 0.033 | 0.041 |
| 合浦 HP | 0.016 | 0.015 | 0.010 | -0.004 | 0.036 |
| 硇洲岛 NZ | 0.021 | 0.020 | 0.025 | -0.014 | 0.005 |
| 乌石镇 WS | 0.019 | 0.018 | 0.023 | 0.022 | -0.001 |
| 博鳌 BA | 0.023 | 0.023 | 0.027 | 0.025 | 0.029 |
| *, **, ***分别表示统计上显著(P < 0.05)、较显著(P < 0.01)和极显著(P < 0.001) | |||||
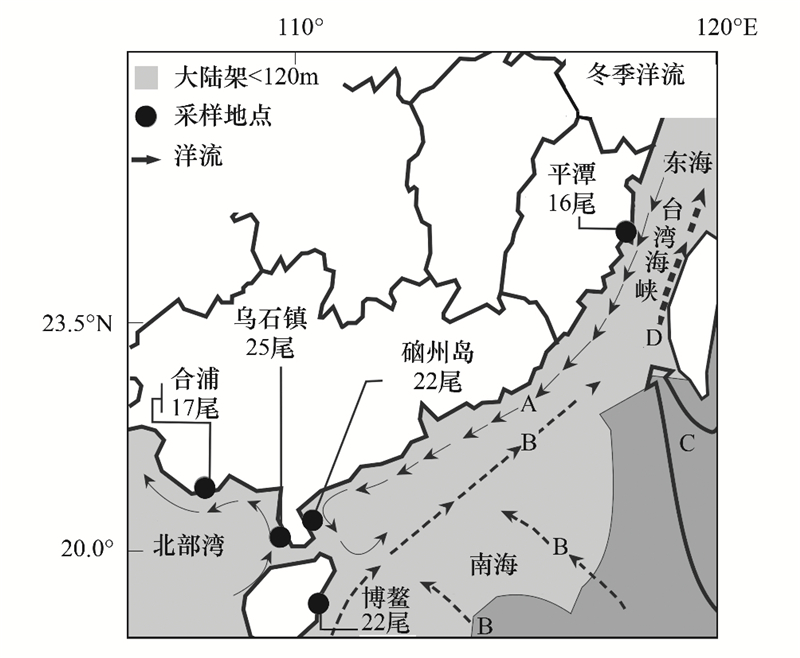
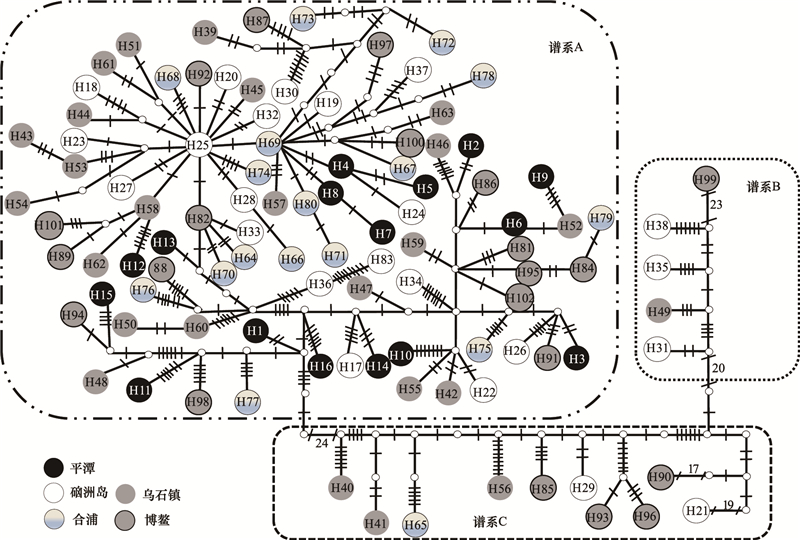
在系统邻接树(图 2)和单倍型网络图(图 3)上都出现3个谱系:谱系A、B和C, 其中谱系A(Lineage A)由平潭、硇洲岛、乌石镇、合浦和博鳌87个个体组成;谱系B(Lineage B)由硇洲岛、乌石镇及博鳌5个个体组成, 谱系C(Lineage C)由硇洲岛、乌石镇、合浦及博鳌10个个体组成, 谱系间遗传分化(表 3)极为显著(Fst=0.508—0.698, P < 0.001), 表明具有明显的谱系结构, 但没有明显的地理聚群。单倍型网络图为非典型星状, 拥有一个中心单倍型(硇洲岛-H25, 位于Lineage A中)。根据谱系间的净遗传距离(Da=0.024—0.032), 以线粒体控制区变异速率为(3—10)%/百万年[23], 估算出谱系的分歧时间大约在1.07百万年—0.24百万年前(即早-中更新世)。
|
| 图 2 基于线粒体控制区序列建立的102个细鳞鯻单倍型邻接树 Fig. 2 NJ trees of 102 haplotypes of T.Jarbua based on mtDNA control region sequences 分支上数字为大于60%的bootstrap支持率 |
|
| 图 3 基于NetWork软件构建的细鳞鯻群体单倍型网络图 Fig. 3 Parsimony network of haplotypes of T. jarbua 图中每个圈代表一个单倍型, 圈中编号为单倍型类型(编号H1—H102);枝上线条(或数字)表示突变步骤 |
| 分组 Group | 遗传分化系数 F-statistics, Fst |
净遗传距离 Net genetic distance, Da |
分化时间 Differentiation time |
| Lineage A和Lineage B | 0.698*** | 0.031 | 1.03百万年—0.31百万年前 |
| Lineage A和Lineage C | 0.692*** | 0.032 | 1.07百万年—0.32百万年前 |
| Lineage B和Lineage C | 0.508*** | 0.024 | 0.8百万年—0.24百万年前 |
另外, 由于85.3%的单倍型集中分布在Lineage A中, 因此对Lineage A进行遗传多样性及遗传分化分析(表 4)、分子方差分析(表 5), 结果显示Lineage A中种群间的变异占总变异的1.97%。另外, Lineage A中5个地理群体之间的遗传分化系数Fst在-0.021—0.068之间(P >0.005)。由此说明Lineage A中的5个群体之间基因交流频繁, 不存在遗传分化情况, 属于一个随机交配群体。
| 群体 Population |
数目 number |
单倍型多样性 Haplotype diversity, Hd |
核苷酸多样性 Nucleotide diversity, Pi |
遗传分化系数(Fixation index, Fst) 和基因流值(gene flow, Nm ) |
||||
| PT | NZ | BA | WS | HP | ||||
| 平潭 PT | 16 | 1.000 ± 0.022 | 0.014 ± 0.001 | — | 6.853 | -167.167 | 14.652 | 7.697 |
| 硇洲岛 NZ | 17 | 1.000 ± 0.020 | 0.011 ± 0.002 | 0.068 | — | 13.068 | -500.5 | 24.31 |
| 博鳌 BA | 17 | 1.000 ± 0.020 | 0.013 ± 0.001 | -0.003 | 0.037 | — | 62 | 16.741 |
| 乌石镇 WS | 21 | 1.000 ± 0.015 | 0.012 ± 0.001 | 0.033 | -0.001 | 0.008 | — | -83.833 |
| 合浦 HP | 16 | 1.000 ± 0.022 | 0.010 ± 0.001 | 0.061 | -0.021 | 0.029 | -0.006 | — |
| 总计 Total | 87 | 1.000 ± 0.002 | 0.012 ± 0.001 | — | — | — | — | — |
| 对角线下为遗传分化系数Fst, 对角线上方为基因流值Nm | ||||||||
| 变异来源 Source of variation |
df | 方差总和 Sum of squares |
变异组分 Variance components |
变异比例/% Variation |
| 种群间 Among populations | 4 | 31.708 | 0.118 | 1.97 |
| 种群内 Within populations | 82 | 481.717 | 5.875 | 98.03 |
| 总计 Total | 86 | 513.425 | 5.993 |
Lineage A的核苷酸错配图呈现出一条不完全平滑但只有一个明显顶峰的单峰型曲线(图 4), Tajima′s D和Fu′s Fs检验(表 6)均为显著的负值(P < 0.01), 表明未显著偏离种群扩张模型, 种群扩张大约发生在25.4万年—7.6万年前(即中-晚更新世)。而Lineage B和Lineage C由于数量较少不进行中性检验, 两者核苷酸错配图均呈非单峰分布(图 4)。中国近海5个群体的总体核苷酸错配图呈多峰分布, 中性检验Fu′s Fs为显著负值, Tajima′s D为非显著负值, 离差平方和(sum of squares of deviations, SSD)和粗糙指数(Raggedness index, r)值均较小且不显著。一般在相同条件下, Fu′s Fs检验对群体的近期扩张比较敏感[24], 同时结合单倍型网络图, 可以认为中国近海细鳞鯻曾出现种群扩张, 时间约为16.9万年—5.06万年前(即中-晚更新世)。
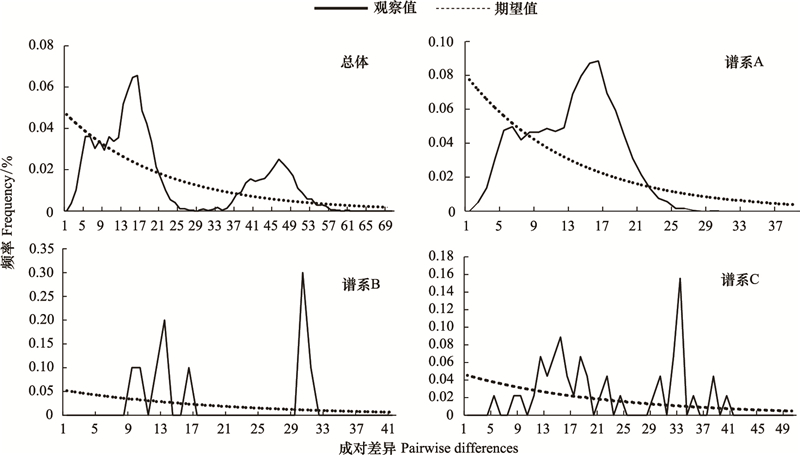
|
| 图 4 细鳞鯻各分支核苷酸错配图 Fig. 4 Mismatch distribution of T. jarbua of each Lineage |
| 谱系 Lineage | Tajima′s D检验 | Fu′s Fs 检验 | 扩张时间参数τ The time of the expansion |
离差平方和SSD Sum of Squares of Deviations |
粗糙指数 Raggedness index |
|||
| D | P | Fs | P | |||||
| 谱系 Lineage A | -2.089 | 0.004 | -24.331 | 0.001 | 14.640 | 0.004 | 0.003 | |
| 总体 Total | -1.621 | 0.019 | -23.961 | 0.001 | 9.744 | 0.008 | 0.002 | |
| D和Fs各为所在检验所得的数值, P为显著性 | ||||||||
判断一个群体的遗传多样性高低, 常用单倍型多样性(Hd )和核苷酸多样性(Pi )作为评价指标[25]。中国近海细鳞鯻总体呈现出较高的遗传多样性特征(Hd =1.000, Pi =0.022);高于同海域分布的鱼类花鲈(Lateolabrax japonicus)(Hd =0.901, Pi =0.003)[26]、蓝点马鲛(Scomberomorus niphonius)(Hd =0.702, Pi =0.0028)[27]和黑鲷(Acanthopagrus schlegeli)(Hd =0.979, Pi =0.009)[28], 仅次于真鲷(Pagrosomus major)(Hd =0.998, Pi =0.025)[29]。表明中国近海细鳞鯻的遗传多样性较高, 具有一定的开发潜力。本研究5个群体中单倍型多样性较高(均为1.000), 与Liu研究的台湾海峡细鳞鯻高单倍型多样性相一致[9];但台湾海峡细鳞鯻呈现出低遗传多样性的特征可能与选取的COI基因变异较为保守及研究样本数量有关。Grant等人[30]认为高单倍型多样性和高核苷酸多样性可能是物种中存在两个分化的亚种或相互独立的种群发生了两次接触形成的, 或者是种群呈现出稳定、发展壮大的模式且未经历瓶颈效应或种群扩张所致。结合中国细鳞鯻种群动态, 认为其遗传多样性高一方面可能是由3个谱系造成, 另一方面可能与种群所处环境不均一性或种群生活生理特性有关[31-32]。物种的生理和生活习性是该物种在特定生态环境下经过长期的自然选择、进化的结果, 表现出该物种对环境的适应性[33]。通常认为, 遗传多样性较高则表明该物种对环境变化的适应能力较强[34];细鳞鯻广泛分布于南海、东海及台湾海峡, 是一种对温盐适应能力较强、能生活于河口咸淡水环境中的鱼类, 因此推测复杂多变的海洋环境及较强的生活生理特征成为细鳞鯻种群高遗传多样性的原因之一。
3.2 细鳞鯻的遗传分化中国近海细鳞鯻群体间存在3个分化显著的谱系, 这种分歧发生在1.07百万年—0.24百万年前(即早-中更新世)。更新世时期出现冰期与间冰期交替的气候变化, 从而对边缘海的面积和结构、生物的分布范围产生巨大的影响[35];冰期海平面的急剧下降使得边缘海之间出现陆桥导致海盆的生物群体产出隔离, 促使近海鱼类谱系分化和物种形成[36-37]。但中国近海细鳞鯻没有明显的地理结构, 究其原因则可能是细鳞鯻成鱼进入较深海域产卵, 受精卵和孵化成的仔鱼均不具备游泳能力而随海流流动, 幼鱼在内海港湾肥育, 而成鱼则移向外海[1, 3]。另外, 中国沿岸流流向较为复杂(冬季从北到南流到转为夏季从南到北方向流动), 黑潮的一个分支流经巴士海峡进入南海成为南海暖流, 后又经台湾海峡向东北流入东海[12, 38](见图 1洋流流向), 使得南海、台湾海峡及东海之间的水体交换活跃, 加上海洋开放的环境中屏障较少, 因而不同地理群体的细鳞鯻其卵、幼体和成体均可随着洋流的运动进行自由扩散从而扩大分布范围, 以致于地理距离相对较远的群体之间也能够进行基因交流。更新世冰期, 西北太平洋大陆架形成很多隔离[39], 隔离使得东海与南海间的细鳞鯻出现了分化, 但由于没有足够长的时间积累变异, 再加上冰期后海平面的上升使得海域再次相连而出现二次接触而形成这种谱系结构, 这种情况与南海康氏马鲛(Scomberomorus commerson)[40]谱系分化的现象相似。
3.3 细鳞鯻种群历史动态中性检验以及单倍型网络图显示中国近海细鳞鯻出现了种群扩张的情况。赵松龄等[41]认为在近30万年来, 东海曾发生过5次海侵, 海水向陆地侵入;曾从盛[42]认为在晚更新世时期, 受气候的变化, 闽东北沿海曾发生过4次海侵, 但海侵的强度都不大, 影响的范围较有限;海侵之后伴随的是持续上万至数千年时间的海退, 海水退出本区域导致海平面大幅度升降波动。另外, 伴随着更新世晚期气候变化导致的盐度、温度及洋流模式的改变等都可能导致细鳞鯻群体出现扩张。结合中国近海细鳞鯻高单倍型多样性和高核苷酸多样性的特征, 可知细鳞鯻3个分支在1.07百万年—0.24百万年前出现分化和隔离, 细鳞鯻在25.4百万年—5.06万年前(合并谱系A的扩张时间及中国近海细鳞鯻谱系扩张时间)出现种群扩张使得分化的谱系出现二次接触, 谱系中的个体得以进行基因交流。这也解释了中国近海细鳞鯻在系统邻接树上出现了3个分化显著的谱系但谱系间不存在明显地理结构的情况。
| [1] | 朱元鼎, 张春霖, 成庆泰. 东海鱼类志. 北京: 科学出版社, 1963: 335–336. |
| [2] | 王忠良, 黄建盛, 陈刚, 张健东, 刘诗亮. 野生细鳞鯻(Therapon jarbua)肌肉主要营养成分分析及评价. 广东海洋大学学报, 2013, 33(4): 1–6. |
| [3] | 李忠炉. 北部湾口海域细鳞鯻渔业生物学特性及开发保护策略初步研究[D]. 湛江: 广东海洋大学, 2008. http://d.wanfangdata.com.cn/Thesis_Y1405586.aspx |
| [4] | Liao X L, Yu X M, Tong J G. Genetic diversity of common carp from two largest Chinese lakes and the Yangtze River revealed by microsatellite markers. Hydrobiologia, 2006, 568(1): 445–453. DOI:10.1007/s10750-006-0222-0 |
| [5] | 张健东, 宋蓓玲, 陈刚. 细鳞鯻年龄与生长的研究. 海洋科学, 2002, 26(7): 62–66. |
| [6] | Krishnakumari L, Varshney P K, Gajbhiye S N, Govindan K, Nair V R. Toxicity of some metals on the fish Therapon jarbua (Forsskal, 1775). Indian Journal of Marine Sciences, 1983, 12(1): 64–66. |
| [7] | 周佳怡, 章群, 唐优良, 余帆洋, 赵爽. 中国近海鯻科鱼类系统发育初探. 海洋渔业, 2010, 32(4): 351–355. |
| [8] | Lavergne E, Calvè I, Meistertzheim A L, Charrier G, Zajonz U, Laroche J. Complex genetic structure of a euryhaline marine fish in temporarily open/closed estuaries from the wider Gulf of Aden. Marine Biology, 2014, 161(5): 1113–1126. DOI:10.1007/s00227-014-2404-z |
| [9] | Liu S Y V, Huang I H, Liu M Y, Lin H D, Wang F Y, Liao T Y. Genetic stock structure of Terapon jarbua in Taiwanese waters. Marine and Coastal Fisheries:Dynamics, Management, and Ecosystem Science, 2015, 7(1): 464–473. DOI:10.1080/19425120.2015.1074966 |
| [10] | Hebert P D N, Cywinska A, Ball S L, Dewaard J R. Biological identifications through DNA barcodes. Proceedings of the Royal Society B:Biological Sciences, 2003, 270(1512): 313–321. DOI:10.1098/rspb.2002.2218 |
| [11] | 张艳萍, 杜岩岩, 王太, 虎永彪, 娄忠玉, 焦文龙. 秦岭细鳞鲑群体遗传结构. 生态学报, 2014, 34(17): 4950–4956. |
| [12] | 苏军虎, 张艳萍, 娄忠玉, 刘焕章, 焦文龙, 杨建宝, 魏彦明. 基于线粒体控制区序列的黄河上游厚唇裸重唇鱼种群遗传结构. 生态学报, 2012, 32(13): 4191–4198. |
| [13] | 庆宁, 丘城锋, 廖伟群, 马天峰, 梁晓旭, 列金妮. 华南沿海西部美丽小条鳅基于线粒体控制区的种群遗传变异及亲缘地理格局. 生态学报, 2010, 30(1): 258–264. |
| [14] | 孙海平. 中国近海区域海洋. 北京: 海洋出版社, 2006: 97–98. |
| [15] | Aljanabi S M, Martinez I. Universal and rapid salt-extraction of high quality genomic DNA for PCR-based techniques. Nucleic Acids Research, 1997, 25(22): 4692–4693. DOI:10.1093/nar/25.22.4692 |
| [16] | Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6:molecular evolutionary genetics analysis version 6.0. Molecular Biology and Evolution, 2013, 30(12): 2725–2729. DOI:10.1093/molbev/mst197 |
| [17] | Librado P, Rozas J. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data. Bioinformatics, 2009, 25(11): 1451–1452. DOI:10.1093/bioinformatics/btp187 |
| [18] | Bandelt H J, Forster P, Röhl A. Median-joining networks for inferring intraspecific phylogenies. Molecular Biology and Evolution, 1999, 16(1): 37–48. DOI:10.1093/oxfordjournals.molbev.a026036 |
| [19] | Excoffier L, Lischer H E L. Arlequin suite ver 3.5:a new series of programs to perform population genetics analyses under Linux and Windows. Molecular Ecology Resources, 2010, 10(3): 564–567. DOI:10.1111/men.2010.10.issue-3 |
| [20] | Nei M, Tajima F. DNA polymorphism detectable by restriction endonucleases. Genetics, 1981, 97(1): 145–163. |
| [21] | Shaklee J B, Tamaru C S, Waples R S. Speciation and evolution of marine fishes studied by the electrophoretic analysis of proteins. Pacific Science, 1982, 36(2): 141–157. |
| [22] | Wright S. The interpretation of population structure by F-statistics with special regard to systems of mating. Evolution, 1965, 19(3): 395–420. DOI:10.1111/evo.1965.19.issue-3 |
| [23] | 郜星晨, 章群, 薛丹, 宫亚运, 曹艳, 韩博平. 基于线粒体控制区部分序列的南海大斑石鲈遗传多样性分析. 海洋科学, 2016, 40(7): 41–45. DOI:10.11759//hykx20141118001 |
| [24] | Fu Y X. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection. Genetics, 1997, 147(2): 915–925. |
| [25] | Bonin A, Nicole F, Pompanon F, Miaud C, Taberlet P. Population adaptive index:a new method to help measure intraspecific genetic diversity and prioritize populations for conservation. Conservation Biology, 2007, 21(3): 697–708. DOI:10.1111/cbi.2007.21.issue-3 |
| [26] | 刘明月. 不同海域中国花鲈遗传多样性的研究及营养成分分析[D]. 南京: 南京师范大学, 2010. |
| [27] | 曹艳, 章群, 宫亚运, 吕金磊, 杨喜书. 基于线粒体COI序列的中国沿海蓝点马鲛遗传多样性. 海洋渔业, 2015, 37(6): 485–493. |
| [28] | 赵爽, 章群, 乐小亮, 彭博, 许忠能, 韦桂峰, 李贵生. 中国近海5个黑鲷地理群体的遗传变异. 海洋科学, 2010, 34(2): 75–79. |
| [29] | 乐小亮, 章群, 赵爽, 范凤娟. 中国近海真鲷遗传变异的线粒体控制区序列分析. 广东农业科学, 2010, 37(2): 136–139. |
| [30] | Grant W A S, Bowen B W. Shallow population histories in deep evolutionary lineages of marine fishes:insights from sardines and anchovies and lessons for conservation. Journal of Heredity, 1998, 89(5): 415–426. DOI:10.1093/jhered/89.5.415 |
| [31] | Nei M. Molecular Evolutionary Genetics. New York: Columbia University Press, 1987: 92–145. |
| [32] | 张丽艳, 苏永全, 王航俊, 王军. 台湾海峡鲐鱼种群遗传结构. 生态学报, 2011, 31(23): 7097–7103. |
| [33] | 陈迪. 长江刀鲚遗传多样性的细胞色素b基因序列分析[D]. 广州: 暨南大学, 2006. |
| [34] | Markert J A, Champlin D M, Gutjahr-Gobell R, Grear J S, Kuhn A, McGreevy T J Jr, Roth A, Bagley M J, Nacci D E. Population genetic diversity and fitness in multiple environments. BMC Evolutionary Biology, 2010, 10(1): 205. DOI:10.1186/1471-2148-10-205 |
| [35] | Dynesius M, Jansson R. Evolutionary consequences of changes in species' geographical distributions driven by Milankovitch climate oscillations. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(16): 9115–9120. DOI:10.1073/pnas.97.16.9115 |
| [36] | Tang W X, Lshimatsu A, Fu C Z, Yin W, Li G, Chen H, Wu Q H, Li B. Cryptic species and historical biogeography of eel gobies (Gobioidei:Odontamblyopus) along the Northwestern Pacific coast. Zoological Science, 2010, 27(1): 8–13. DOI:10.2108/zsj.27.8 |
| [37] | Kimura M. Paleogeography of the Ryukyu Islands. Tropics, 2000, 10(1): 5–24. DOI:10.3759/tropics.10.5 |
| [38] | 李立, 苏纪兰, 许建平. 南海的黑潮分离流环. 热带海洋, 1997, 16(2): 42–57. |
| [39] | Wang P X. Response of Western Pacific marginal seas to glacial cycles:paleoceanographic and sedimentological features. Marine Geology, 1999, 156(1/4): 5–39. |
| [40] | 曹艳, 章群, 宫亚运, 吕金磊, 杨喜书. 中国南海康氏马鲛线粒体COI序列遗传变异分析. 南方水产科学, 2016, 12(5): 53–60. |
| [41] | 赵松龄, 秦蕴珊. 中国东部沿海近三十万年以来的海侵与海面变动. 中国第四纪研究, 1985, 6(2): 97–103. |
| [42] | 曾从盛. 闽东北沿海晚第四纪海侵与海面变动. 福建师范大学学报:自然科学版, 1997, 13(4): 94–101. |
 2018, Vol. 38
2018, Vol. 38