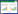文章信息
- 袁向华, 周艳玲, 宋清姿, 脱红梅, 马沁沁, 王一丁
- YUAN Xianghua, ZHOU Yanling, SONG Qingzi, TUO Hongmei, MA Qinqin, WANG Yiding.
- 蚯蚓吞食过程中土壤理化性质与放线菌多样性的变化特征
- Variations of soil physical-chemical properties and the diversity of actinomycetes during the process of swallowing of earthworms
- 生态学报. 2017, 37(4): 1199-1210
- Acta Ecologica Sinica. 2017, 37(4): 1199-1210
- http://dx.doi.org/10.5846/stxb201606211204
-
文章历史
- 收稿日期: 2016-06-21
- 修订日期: 2016-11-10
随着环境破坏的加剧, 人们对土壤生态环境的保护意识日益增强, 探究土壤与生物间的相互关系显得尤为重要。蚯蚓是土壤中的主要动物类群, 通过吞食作用破碎土壤, 并利用肠道内的活性酶降解土壤有机物, 使土壤肥效增加[1-2]。放线菌作为微生物活性物质的主要生产者[3], 为宿主提供活性物质, 在蚯蚓吞食土壤过程中主要负责腐殖酸的形成[4], 对土壤肥力及质量的改善至关重要。
蚯蚓对土壤肥力的改良主要通过吞食作用完成, 含有大量微生物的土壤经蚯蚓的吞食作用进入肠道, 再以蚓粪形式排出, 可将肠道内容物、蚓粪视作特殊生境下的土壤[5]。土壤理化性质是表征土壤肥力质量的重要指标[6], 有报道指出蚯蚓吞食过程中土壤理化性质及土壤放线菌数量均发生改变[7-9], 但放线菌多样性变化特征及两者之间的相关性未见报道。探明蚯蚓吞食过程中土壤放线菌多样性与土壤理化性质的变化特征及相关性, 可揭示该过程中放线菌多样性变化与土壤理化性质的动态关系, 为土壤的生物修复提供理论依据。本研究将蚯蚓生活土壤、肠道内容物及蚓粪视作蚯蚓吞食作用的前、中、后阶段, 分离纯化3个阶段中的放线菌, 采用克隆文库法分析3个阶段中放线菌的多样性, 并探究土壤理化性质与放线菌多样性的变化特征, 为阐明蚯蚓、土壤理化性质、放线菌多样性的相关性提供理论依据, 对环境保护、土壤生态维护具有重要应用价值。
1 材料与方法 1.1 样品采集及处理研究样地位于四川省成都市青白江区大同镇某耕地(30°41′33"N, 104°13′37"E), 属亚热带季风性湿润气候, 土壤类型为水稻土类, 采取水旱轮作方式, 种植作物为水稻、油菜、莴笋等。
采用S形布点法[10]于0—20 cm采集耕层土壤, 视该土壤为蚯蚓生活土壤, 并收集蚯蚓, 经鉴定, 该种蚯蚓为白颈环毛蚓(Pheretima californica)。将土壤混匀并分为3部分, 分别用于测定土壤理化性质、提取土壤总DNA和收集新鲜蚓粪, 新鲜蚓粪收集方法参考Haynes等[11]的方法。所有样品收集后迅速带回实验室进行实验。
除净蚯蚓体表泥土后, 75%乙醇体表灭菌、无菌水漂洗3次, 解剖获得肠道内容物后混匀。
1.2 基本理化性质测定除去蚯蚓生活土壤、肠道内容物及蚓粪中的杂物, 自然风干后碾磨过直径2 mm和0.15 mm筛, 按国标法测定样品的基本理化性质[10], 每个指标3个重复, SPSS 18.0对基本理化性质进行显著性检验。
1.3 可培养放线菌的分离、鉴定及系统发育分析 1.3.1 可培养放线菌的分离、鉴定制备3种样品的水溶液, 40℃、140 r/min震荡30 min, 每个样品3个平行, 参照史学群的优化方法分离、纯化放线菌[12], 按阮继生的方法鉴定、合并相同菌株[13]。
1.3.2 可培养放线菌16S rDNA序列测定酶法提取菌体总DNA, 以细菌16S rRNA基因通用引物27F(5′-AGAGTTTGATCATGGCTCAG-3′)和1540R(5′-AAGGAGGTGATCCAACCGCA-3′)[14]进行16S rDNA PCR扩增, 扩增体系:12.5 μL 2×PCR Mix;引物各1 μL;DNA 0.5 μL;双蒸水10 μL, 反应程序:94℃预变性30 s, 94℃变性10 s, 55℃退火1 min, 72℃延伸1 min, 30个循环, 72℃延伸10 min[15]。PCR产物经电泳检测后送上海生物工程技术公司测序。
1.3.3 可培养放线菌的系统发育分析通过BLAST程序选取与所得16S rDNA序列相近的模式菌株基因序列, 用MEGA 5.0根据N-J法构建系统发育树, 自举值为1000[16], Acidimicrobium ferrooxidans (U75647.1) 作外群。
1.4 放线菌16S rDNA克隆文库的构建 1.4.1 放线菌16S rDNA的扩增、纯化SoilGen DNA Kit提取总DNA并作为模板, 放线菌16S rRNA基因特异性引物F243(5′-GGATGAGCCCGCGGCCTA-3′);R513(5′-CGGCCGCGGCTGCTGGCACGTA-3′)[17]扩增3种样品的放线菌16S rDNA, PCR体系及程序参照韩俊的方法[18], 产物经电泳检测后再纯化。
1.4.2 放线菌16S rDNA克隆文库的构建及阳性克隆筛选16S rDNA连至pMD19-T载体上(TaKaRa), 连接体系:pMD19-T Vector 1.0 μL, SolutionⅠ5.0 μL, Template 4.0 μL。连接产物转化感受态E. coli DH5α, 蓝白斑筛选后[19]随机挑取阳性克隆送成都擎科梓熙生物技术有限公司测序。
1.5 免培养放线菌的系统发育分析在线去除16S rDNA中的载体序列(http://www.ncbi.nlm.nih.gov/tools/vecscreen/)、嵌合体(Chimeria)[20](http://comp-bio.anu.edu.au/bellerophon/bellerophon.pl), Clustal-X比对序列(multiple alignment)[21], BioEdit 7.0分析比对结果的相似性[22], 将相似性≥97.0%的序列划为同一操作分类单元(Operational taxonomic unit, OTU)[23], 剔除非放线菌OTU, 从每个OTU中选一条序列按上文方法构建进化树。
1.6 放线菌多样性分析、多样性与理化性质的相关性分析采用SPADE(Species prediction and diversity estimation)(http://chao.stat.nthu.edu.tw/softwareCE.html.)通过Estimated sample coverage、Species richness 、Shannon Index、Simpson Index指数评估文库中放线菌的多样性, 用Analytic Rarefaction version(http://strata.uga.edu/software/index.html)绘制稀释性曲线[24], Office Excel 2003统计并处理数据, SPSS 18.0进行理化性质主成分分析及理化性质与放线菌多样性的相关性分析。
2 结果与分析 2.1 基本理化性质的分析由表 1可知, 3种样品的pH均接近中性, 肠道内容物pH略低;肠道内容物和蚓粪中的全氮、钾和速效钾含量相差不明显, 但均显著高于蚯蚓生活土壤, 肠道内容物和蚓粪中的速效钾含量尤为偏高, 分别为0.52 g/kg和0.53 g/kg, 是生活土壤的1.4倍;有机质和速效磷含量在生活土壤、肠道内容物和蚓粪中依次递增, 其中肠道内容物和蚓粪中的速效磷含量分别是生活土壤的3.6和1.7倍;3种样品的全磷含量无明显差异;速效氮含量在肠道内容物、蚓粪和生活土壤中依次递减。
| 样品Samples | pH(H2O) | 有机质Total organic C/(g/kg) | 全氮Total N/(g/kg) | 速效氮Available N /(g/kg) | 全磷Total P/(g/kg) | 有效磷Available P/(g/kg) | 全钾Total K/(g/kg) | 速效钾Available K/(g/kg) |
| 生活土壤 Living soil | 6.84 | 50.6160±2.4912c | 0.9890±0.0022b | 0.0652±0.0061c | 1.1800±0.0191a | 0.0270±0.0006c | 11.7850±0.0724b | 0.3771±0.0181b |
| 肠道内容物 Intestinal contents | 6.77 | 64.0047±2.9046b | 1.0810±0.0015a | 0.2364±0.0198a | 1.2271±0.0111a | 0.0769±0.0005b | 15.4733±0.4692a | 0.5202±0.0019a |
| 蚓粪Feces | 6.81 | 72.0453±1.8017a | 1.0853±0.0016a | 0.1130±0.0056b | 1.0607±0.0669a | 0.0804±0.0003a | 15.9203±0.6884a | 0.5352±0.0243a |
| 数据为平均值±标准差(n=3) .同列数据后标注的不同字母表示差异显著(P<0 .05) | ||||||||
|
| 图 1 生活土壤中可培养放线菌的系统发育树 Fig. 1 The phylogenetic tree of culturable actinobacteria from living soil |
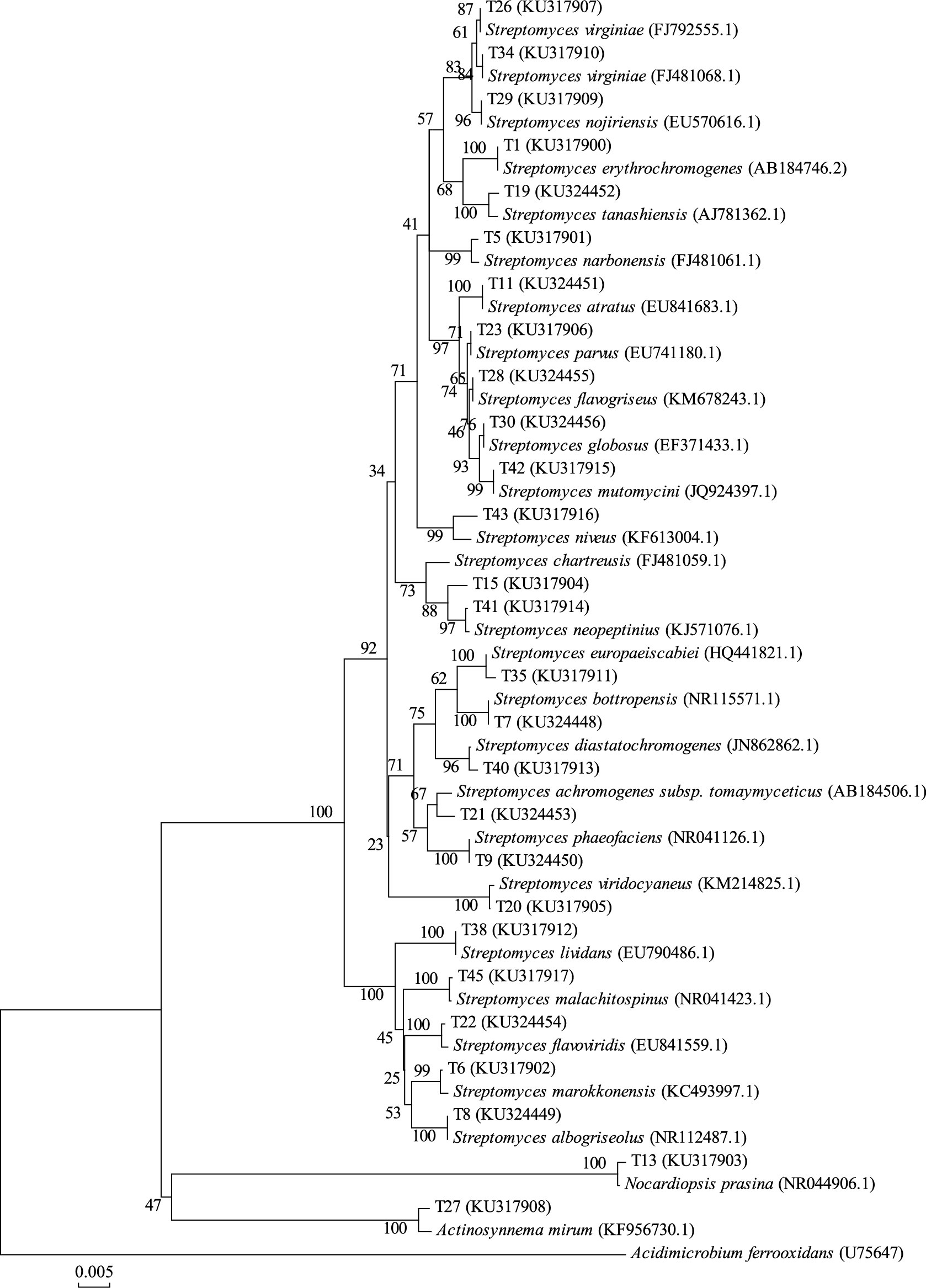
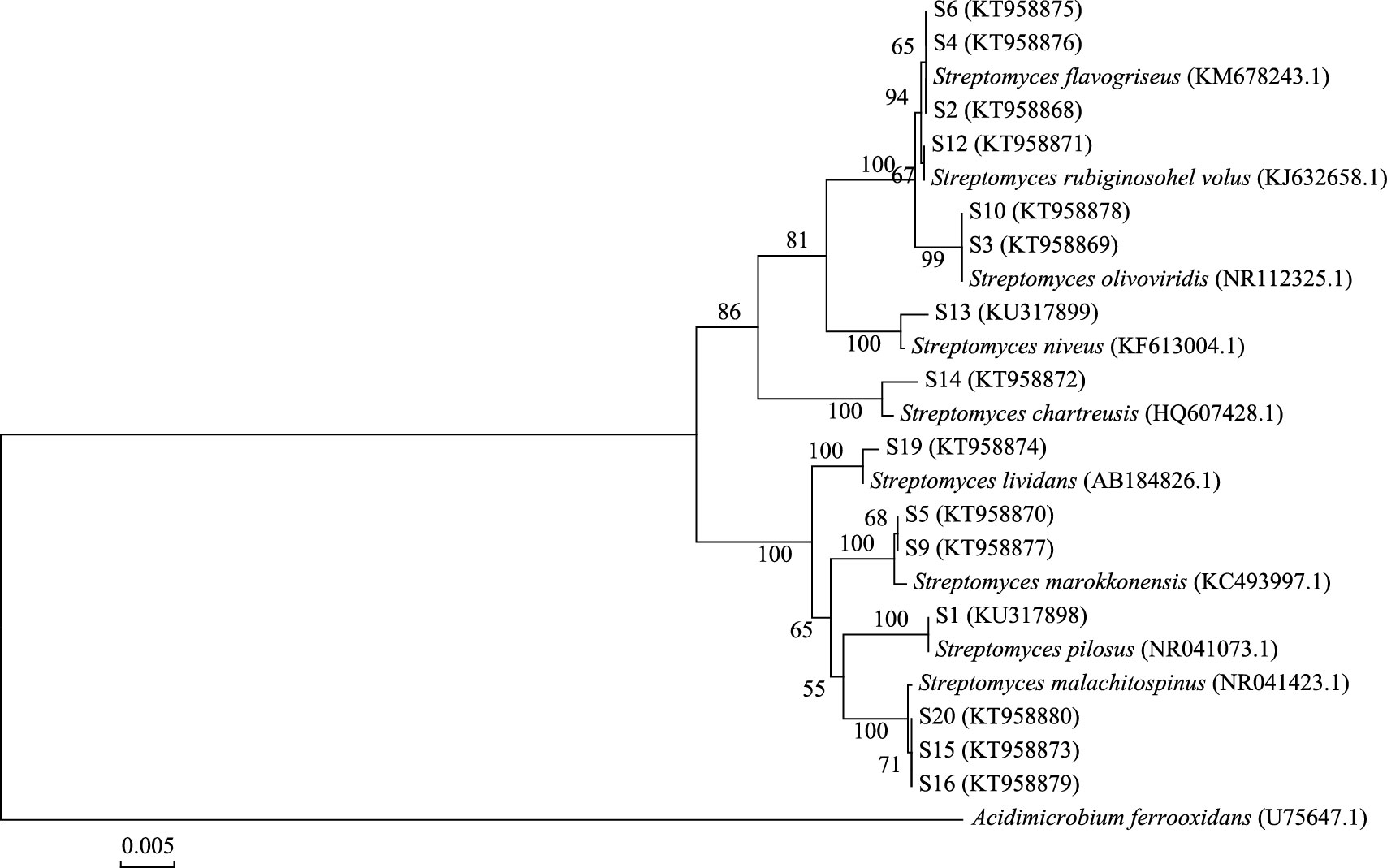
可培养放线菌16S rDNA系统进化树显示, 蚯蚓生活土壤中的27株放线菌(图 1), 分布于3个属:链霉菌属Streptomyces(25株), 拟诺卡氏菌属Nocardiopsis(1株), 束丝放线菌属Actinosynnema(1株)。从肠道内容物和蚓粪分别获得15株和17株放线菌(图 2, 图 3), 均为链霉菌Streptomyces。其中T23、T26、T28是生活土壤和蚓粪的共有菌株, S4、S5、S10是生活土壤和肠道内容物的共有菌株, F5、F25是肠道内容物与蚓粪的共有菌株。综上可知, 蚯蚓吞食作用使可培养放线菌的种类及数量降低;链霉菌在蚯蚓吞食土壤的3个阶段中均是优势菌;链霉菌是土壤中分布最广、数量最多的放线菌[25]。
2.3 克隆文库的构建放线菌16S rRNA基因特异性引物扩增片段约为270bp, 与预计片段大小一致, 经连接、转化、检验后, 用平板保存。
|
| 图 2 肠道内容物中可培养放线菌的系统发育树 Fig. 2 The phylogenetic tree of culturable actinobacteria from intestinal soil |
281条阳性克隆序列经检验未发现嵌合体(Chimeria), 蚯蚓生活土壤、肠道内容物、蚓粪克隆文库中分别含有40、20、30个OTUs(表 2)。
2.5 文库中放线菌多样性分析放线菌多样性分析结果如表 2:蚯蚓生活土壤、肠道内容物、蚓粪克隆文库的覆盖率(C)分别为20.0%、80.3%和66.7%;物种丰富度(Species richness)分别为256.0、44.0和44.5;Shannon指数(Shannon Index)分别为5.279、2.658和3.789;Simpson指数(Simpson Index)分别为0.02815、0.18785和0.05037。
| 样品Sample | 总克隆数TotalClones | OTU数OTUs | 覆盖率Coverage/% | 物种丰度Species richness | Shannon指数Shannon index | Simpson指数Simpson index |
| 生活土壤Living soil | 45 | 40 | 20 | 256 | 5.279 | 0.02815 |
| 肠道内容物Intestinal contents | 61 | 20 | 80.3 | 44.0 | 2.658 | 0.18785 |
| 蚓粪Feces | 51 | 30 | 66.7 | 44.5 | 3.789 | 0.05037 |
肠道内容物克隆文库覆盖率最高, 该文库涵盖肠道内容物中80.3%的放线菌, 生活土壤和蚓粪克隆文库包含相应环境中20.0%和66.7%的放线菌。物种丰度是评估环境中放线菌种类数的一个重要指标, 对比丰富度指数可知, 生活土壤中放线菌种类最多, 几乎是肠道内容物和蚓粪中放线菌种类的5.8倍。香农指数越大说明放线菌的多样性越高, 物种丰度和香农指数的一致性共同表明, 生活土壤放线菌的多样性最高, 蚓粪放线菌的多样性居中, 肠道内容物放线菌的多样性最低。辛普森指数越大则均匀度越高, 可见肠道内容物放线菌的均匀度最高, 蚓粪次之, 生活土壤放线菌的均匀度最低。
|
| 图 3 蚓粪中可培养放线菌的系统发育树 Fig. 3 The phylogenetic tree of culturable actinobacteria from earthworm feces |
稀释性曲线显示(图 4), 肠道内容物克隆文库的曲线已呈平缓, 蚓粪克隆文库的曲线趋于平缓, 而生活土壤克隆文库的曲线几乎呈直线。据Kemp[26]等的理论可知, 肠道内容物克隆文库基本涵盖蚯蚓肠道内容物所有种类的放线菌, 蚓粪克隆文库涵盖蚓粪中大多数种类的放线菌, 但生活土壤克隆文库只涵盖蚯蚓生活土壤中少数种类的放线菌。这一结果映证蚯蚓吞食作用使土壤放线菌的多样性降低, 放线菌的多样性在生活土壤、蚓粪和肠道内容物中依次递减, 生活土壤中有更丰富的放线菌资源。
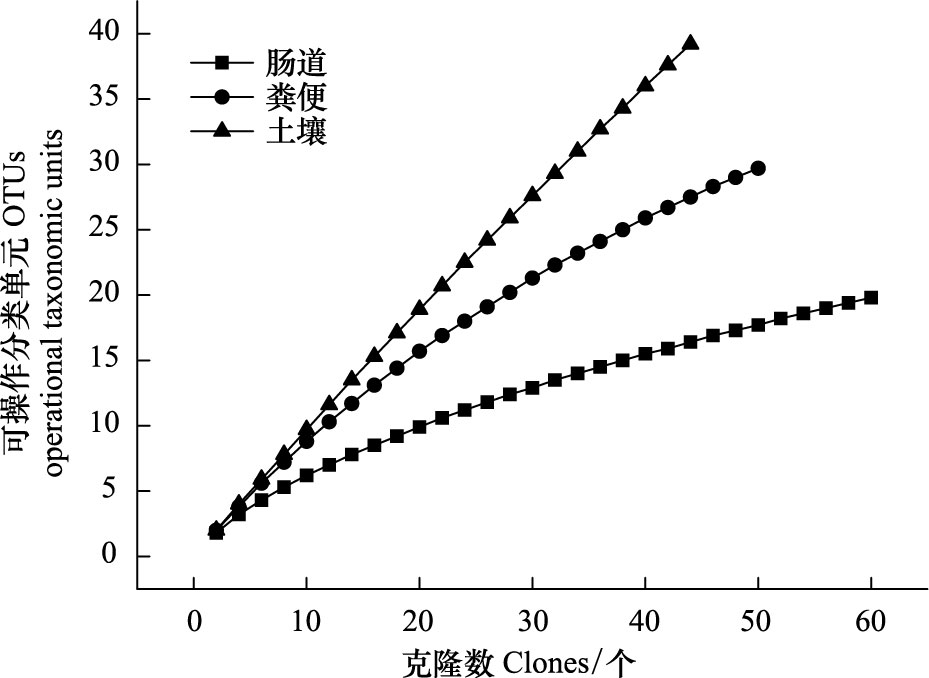
|
| 图 4 3种样品中不可培养放线菌的稀释性曲线 Fig. 4 Rarefaction curve of unculturable actinobacteria at 3 soil samples |
免培养放线菌的系统进化树显示, 蚯蚓生活土壤克隆文库含有40个OTUs(45个克隆子), 其中11个OTUs(11个克隆子)属未知放线菌, 1个OTU(1个克隆子)属于酸微菌目中的酸微菌科(Acidimicrobiaceae), 28个OTUs(33个克隆子)分布于放线菌目的10个科(图 5), 分别为假诺卡氏菌科(Pseudonocardiaceae)、类诺卡氏菌科(Nocardioidaceae)、诺卡氏菌科(Nocardiaceae)、微杆菌科(Microbacteriaceae)、放线菌科(Actinomycetaceae)、小单孢菌科(Micromonosporaceae)、分支杆菌科(Mycobacteriaceae)、链霉菌科(Streptomycetaceae)、冢村氏菌科(Tsukamurellaceae)、动球菌科(Kineococcaceae)。诺卡氏菌科含7个克隆子(15.6%), 是生活土壤中的优势放线菌群, 类诺卡氏菌科和链霉菌科分别含6个(13%)和5个克隆子(11%), 是丰度中等菌群, 微杆菌科、冢村氏菌科和动球菌科各含1个克隆子(2%)。
肠道内容物克隆文库含有20个OTUs(61个克隆子), 其中2个OTUs属未知放线菌(2个克隆子), 18个OTUs(59个克隆子)分布于放线菌目的6个科(图 6), 分别是类诺卡氏菌科(Nocardioidaceae)、链霉菌科(Streptomycetaceae)、诺卡氏菌科(Nocardiaceae)、微杆菌科(Microbacteriaceae)、分支杆菌科(Mycobacteriaceae)和小单孢菌科(Micromonosporaceae)。微杆菌科含48个克隆子(79%), 是肠道内容物中的优势放线菌群, 诺卡氏菌科含4个克隆子(6.6%), 是丰度中等菌群, 分支杆菌科含1个克隆子(1.6%), 余下3科各含2个克隆子(3.3%)。
蚓粪克隆文库含有30个OTUs(51个克隆子), 其中5个OTUs属未知放线菌(6个克隆子), 25个OTUs分布于放线菌目的6个科(图 7), 分别是类诺卡氏菌科(Nocardioidaceae)、链霉菌科(Streptomycetaceae)、诺卡氏菌科(Nocardiaceae)、微杆菌科(Microbacteriaceae)、分枝杆菌科(Mycobacteriaceae)和糖霉菌科(Glycomycetaceae)。链霉菌科含15个克隆子(29%), 是蚓粪中的优势放线菌群, 微杆菌科含13个克隆子(25.5%), 是丰度中等菌群, 糖霉菌科仅含1个克隆子(2%)。
综上可知, 蚯蚓生活土壤(吞食前)中, 放线菌多样性最高, 未知放线菌数量最多;土壤经吞食作用进入蚯蚓肠道(吞食中)导致放线菌多样性降至最低, 未知放线菌数量骤减;土壤以蚓粪(吞食后)形式排出, 放线菌多样性回升, 未知放线菌数量明显增加。此外, 3个阶段中优势菌群也有明显波动, 诺卡氏菌科、微杆菌科和链霉菌科分别是吞食前、中、后期的优势菌群, 且诺卡氏菌科在3个阶段均有广泛分布。
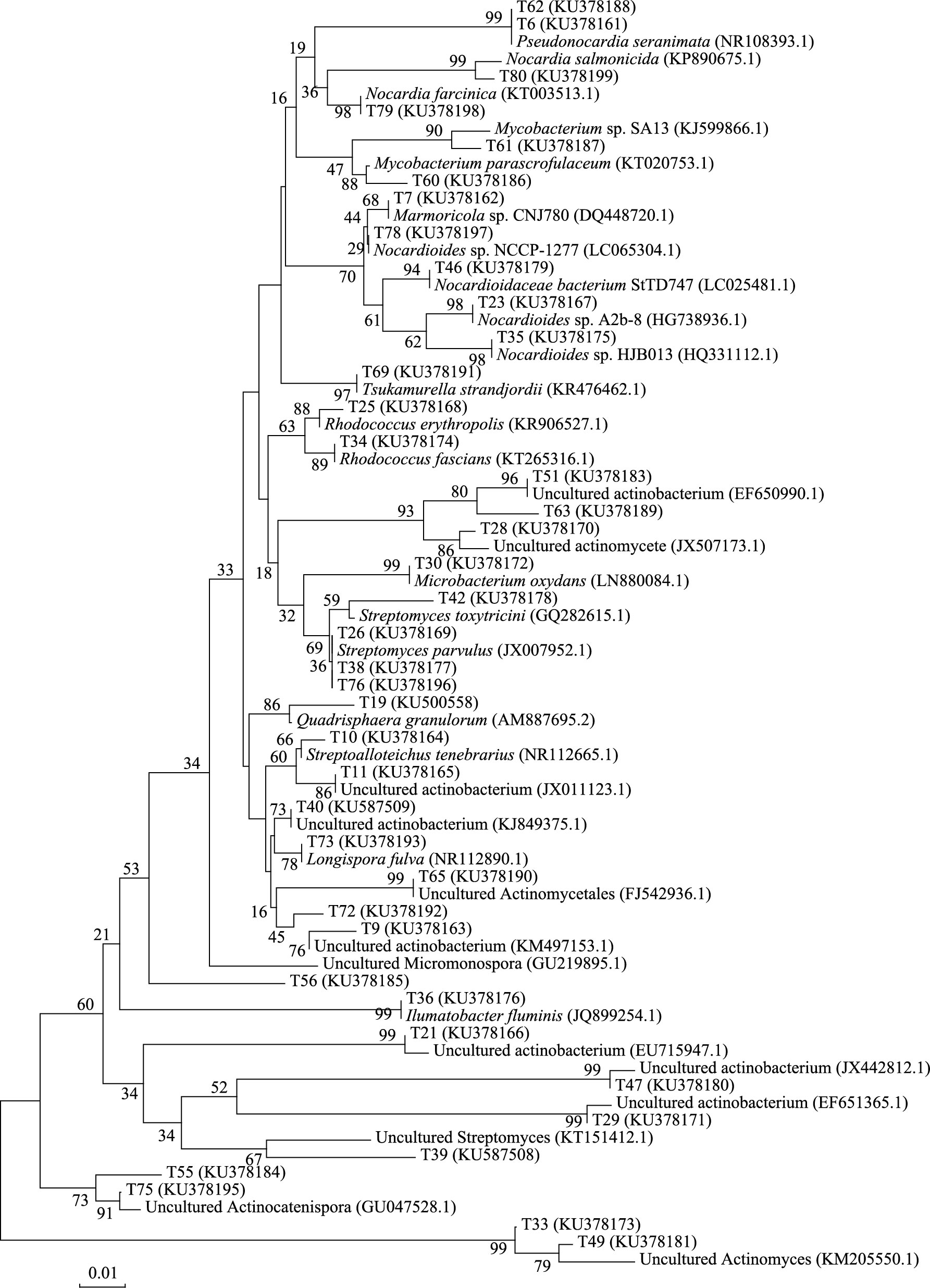
|
| 图 5 生活土壤16S rDNA克隆文库序列构建系统发育树 Fig. 5 The phylogenetic tree structured by 16S rDNA clone library from living soil |
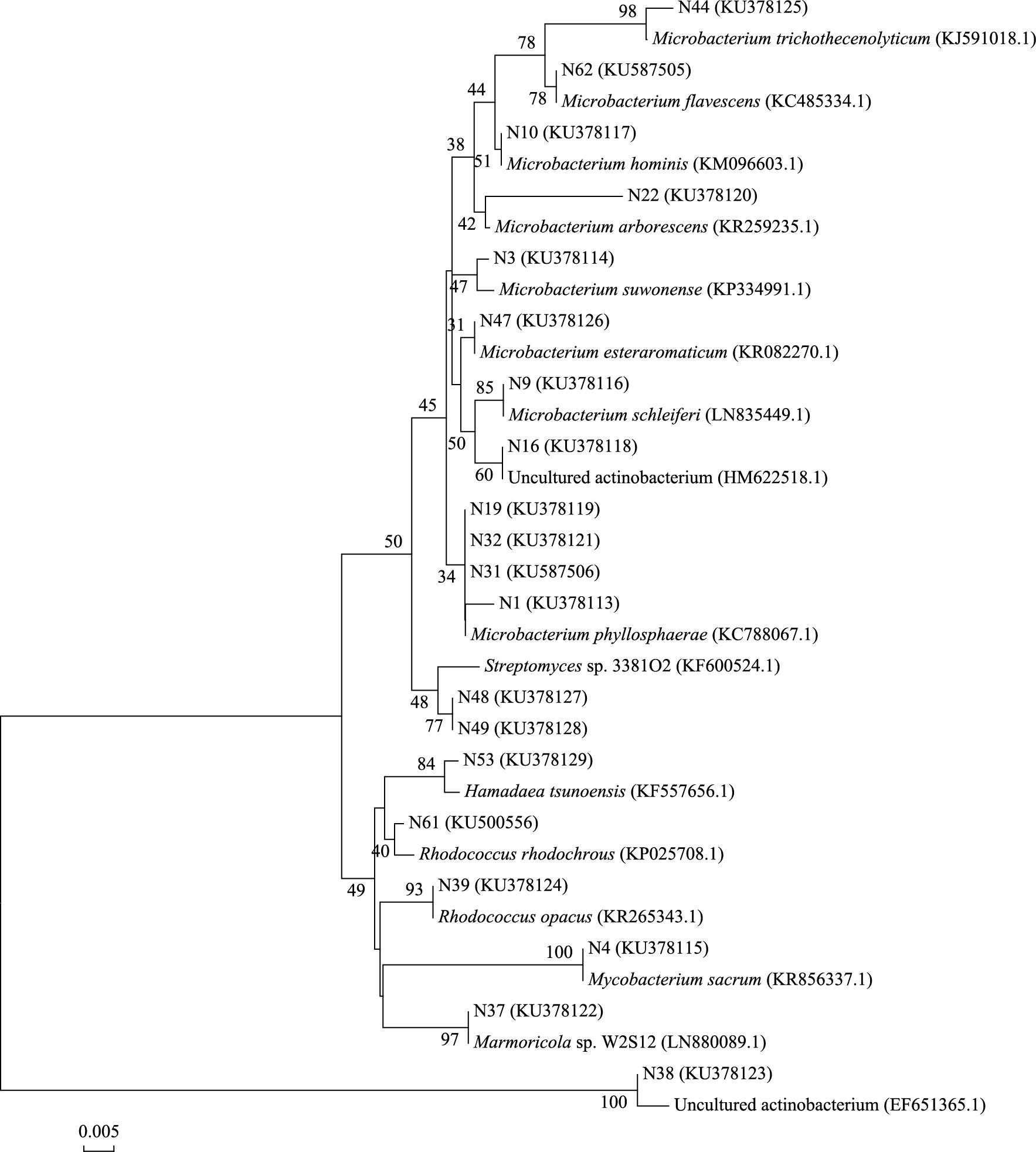
|
| 图 6 肠道内容物16S rDNA克隆文库序列构建系统发育树 Fig. 6 The phylogenetic tree structured by 16S rDNA clone library from intestinal soil |
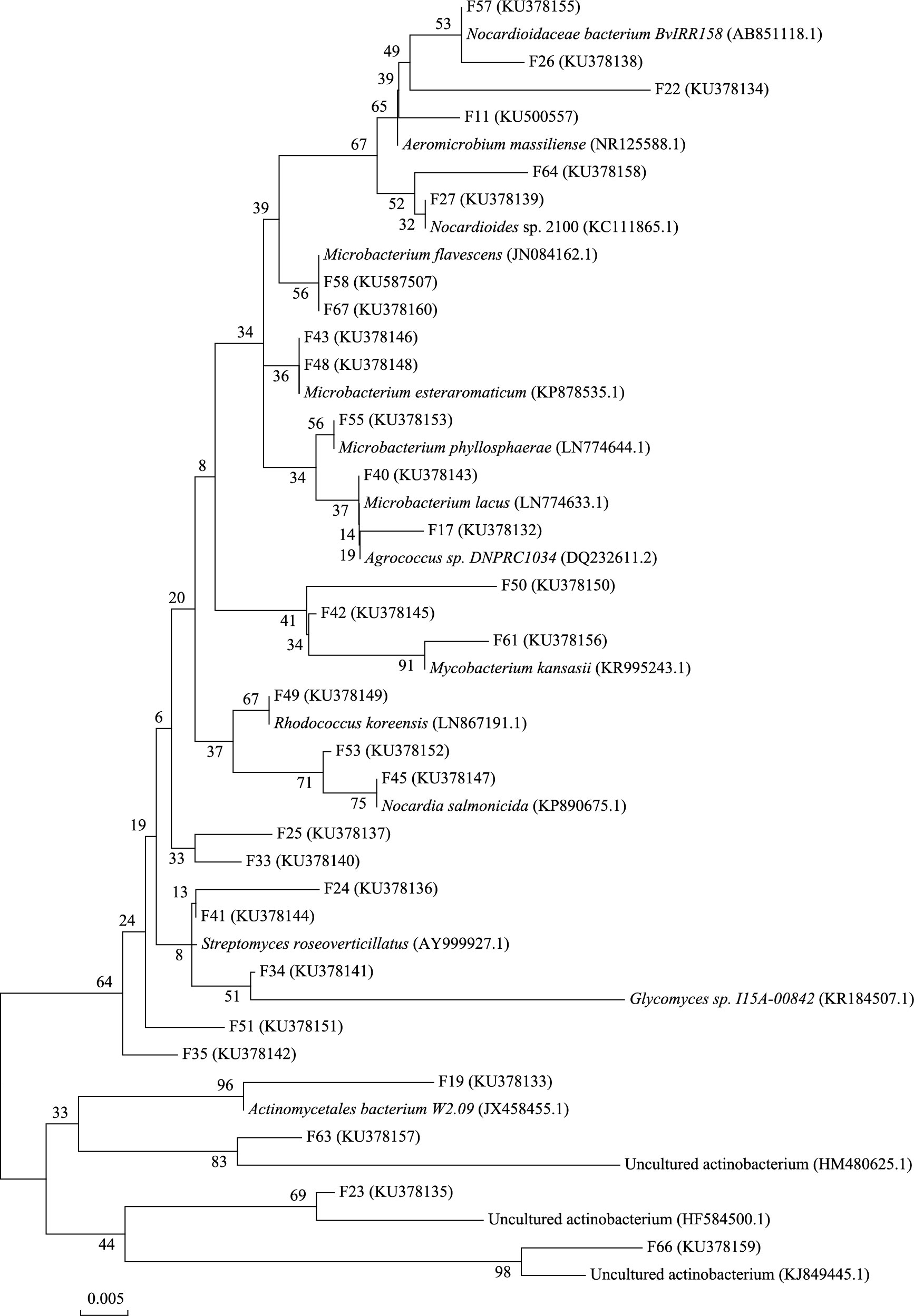
|
| 图 7 蚓粪16S rDNA克隆文库序列构建系统发育树 Fig. 7 The phylogenetic tree structured by 16S rDNA clone library from earthworm feces |
蚯蚓吞食过程中, 土壤各理化性质共同影响放线菌多样性, 因而选择主成分分析法进行多元统计分析, 利用降维思想找出主要影响因子。根据主成分分析原理, 当累积方差贡献率大于85%时可基本反映系统的变异信息。对8个土壤基本理化因子进行主成分分析, 结果(表 3)显示8个土壤基本理化因子可分为2个成分, 特征根均大于1。主成分1中全氮、有效磷、全钾、速效钾、有机质载荷量均超过0.900, 说明主成分1为全氮、有效磷、钾素和有机质的综合因子, 其中全氮、有效磷的载荷量在主成分1中最大;主成分2中全磷、速效氮、pH载荷量较大, 其中全磷的载荷量最大。主成分1贡献率为78.835%, 主成分2的贡献率为21.465%, 主成分1贡献率最大, 说明全氮、有效磷、全钾、速效钾和有机质对蚯蚓吞食过程中放线菌多样性变化起主要作用。
| 因子 Factors | 成分 Components | |
| 1 | 2 | |
| 全氮Total N | 1.000 | 0.009 |
| 速效钾Available K | 0.997 | -0.077 |
| 有机质Totalorganic C | 0.932 | -0.362 |
| 速效氮Available N | 0.709 | 0.705 |
| 特征根 Eigenvalue | 6.283 | 1.717 |
| 累积贡献率 Cumlative/% | 78.535 | 100.000 |
| 有效磷Available P | 0.999 | -0.049 |
| 全钾Total K | 0.997 | -0.080 |
| pH | -0.817 | -0.577 |
| 全磷Total P | -0.508 | 0.862 |
| 贡献率 Percent/% | 78.535 | 21.465 |
根据主成分分析结果, 分析理化性质与多样性的相关性, 结果如表 4所示, 可知放线菌的物种丰富度与土壤有效磷含量呈显著负相关性, 相关系数为-0.998;与土壤全氮含量呈极显著负相关性, 相关系数为-1, 可知土壤有效磷和全氮含量升高使土壤放线菌物种数降低。另外pH、有机质、速效氮、速效钾、全钾等含量均与放线菌多样性具有深刻的相关性, 说明蚯蚓吞食土壤过程中, 土壤理化性质与放线菌多样性相互影响。
3 讨论 3.1 土壤理化性质的变化本研究发现, 蚯蚓生活土壤(吞食前)各理化指标含量均偏低, 肠道内容物(吞食中)速效氮含量最高, 蚓粪(吞食后)的有机质、全氮、全钾、有效磷、速效钾含量均最高, 说明蚯蚓吞食土壤使土壤有效成分明显增加, 能显著增强土壤肥力, 可将蚓粪作为优质有机肥应用于土壤改良培肥与作物增产[27]。土壤理化性质的主成分分析及其与放线菌多样性的相关性发现, 在蚯蚓吞食土壤过程中, 土壤的全氮、有效磷含量可能与放线菌多样性密切相关。可见, 土壤肥力增加除受到蚯蚓胃的物理破碎、肠道矿化作用影响外[28-30], 还受到肠道放线菌种类的影响。
| 项目Item | pH | 有机质Total organic C | 全氮Total N | 速效氮Available N | 全磷 Total P | 速效钾Available K | 有效磷Available P | 全钾 Total K |
| 覆盖率Coverage | -0.925 | 0.828 | 0.977 | 0.849 | -0.302 | 0.954 | 0.963 | 0.954 |
| 物种丰度Species richness | 0.823 | -0.928 | -1.000** | -0.717 | 0.498 | -0.996 | -0.998* | -0.996 |
| Shannon指数Shannon index | 0.987 | -0.68 | -0.903 | -0.947 | 0.079 | -0.862 | -0.876 | -0.861 |
| Simpson指数Simpson index | -0.952 | 0.27 | 0.607 | 0.989 | 0.385 | 0.536 | 0.56 | 0.534 |
| *两者之间在P<0 .05水平上显著相关;**两者之间在P<0 .01水平上极显著相关 | ||||||||
本研究纯培养法和免培养法均证实, 放线菌的多样性在生活土壤(吞食前)、蚓粪(吞食后)和肠道内容物(吞食中)中依次递减。原因可能是土壤进入厌氧环境导致放线菌多样性降低[31];同时, 蚯蚓依靠吞食大量富含微生物的土壤颗粒维持生存[32], 吞食过程中肠道内的纤维素酶、蛋白酶和磷酸酶可能会使土壤中部分敏感微生物减少[33-34];并且通过克隆文库法发现, 生活土壤中少量存在的微杆菌科(Microbacteriaceae)在肠道内容物中为绝对优势放线菌, 而生活土壤和肠道内容物中少量存在的链霉菌属(Streptomycetaceae)在粪便中为优势放线菌, 推测土壤中部分放线菌在肠道中大量扩增, 导致肠道内容物和蚓粪放线菌多样性降低。蚓粪放线菌多样性略高于肠道内容物, 可能因新鲜蚓粪所处的有氧环境导致放线菌多样性回升。本研究中放线菌多样性的变化趋势与Furlong等[9]通过克隆文库法进行研究的结果一致。Parthasarati等人通过可培养法所得结果与本研究不一致[4], 可能由于蚯蚓种类及培养基不同造成[35]。
主成分分析表明, 土壤全氮、有效磷、全钾、速效钾和有机质对蚯蚓吞食过程中放线菌多样性变化影响较大;土壤理化性质与放线菌多样性的相关性分析表明土壤有效磷、全氮含量升高导致放线菌多样性显著降低。两种分析均表明在蚯蚓吞食过程中放线菌多样性受土壤理化性质的影响。
经克隆文库法发现, 蚯蚓生活土壤(吞食前)、肠道内容物(吞食中)和蚓粪(吞食后)中的放线菌主要分布在放线菌目, 与Knapp等人的研究结果一致[35]。此外, 蚯蚓吞食过程3个阶段的优势菌群有明显波动, 诺卡氏菌科为吞食前优势菌群;微杆菌属在吞食前仅占2%, 在中期数量骤增至79%, 是优势菌群, 在后期数量骤减至25.5%, 这与Huang报道的一致[36], 推测微杆菌属放线菌有助于植物性食物的分解;吞食中期链霉菌科分布很少, 后期数量明显增加, 环毛蚓的肠道环境可能利于微杆菌科放线菌的繁殖, 对链霉菌科放线菌的生存、繁殖有明显的抑制作用。
纯培养法是获得功能放线菌的必要手段, 本研究共获得59株放线菌, 链霉菌占96.6%, 蚯蚓肠道、蚓粪两种特殊生境中获得的链霉菌可能具备更强的活性和功能。有研究报道, 图 2中Streptomyces flavogriseus能产抗霉素[37], 可见其亲缘菌S2、S4、S6很可能具有产抗霉素的能力, 有待后续研究。
综上, 本研究通过可培养法与克隆文库法探明了蚯蚓吞食前、中、后阶段土壤放线菌多样性及土壤理化性质的变化特征, 主成分分析与相关性分析可知全氮、有效磷含量显著影响放线菌多样性;进一步完善了蚯蚓、土壤和放线菌3者间的相互关系, 为动物-微生物联合治理环境、保护土壤生态环境提供理论依据。
| [1] | Shan J, Liu J, Wang Y F, Yan X Y, Guo H Y, Li X Z, Ji R. Digestion and residue stabilization of bacterial and fungal cells, protein, peptidoglycan, and chitin by the geophagous earthworm Metaphire guillelmi. Soil Biology and Biochemistry, 2013, 64: 9–17. DOI:10.1016/j.soilbio.2013.03.009 |
| [2] | Bernard L, Chapuis-Lardy L, Razafimbelo T, Razafindrakoto M, Pablo A L, Legname E, Poulain J, Brüls T, O'Donohue M, Brauman A, Chotte J L, Blanchart E. Endogeic earthworms shape bacterial functional communities and affect organic matter mineralization in a tropical soil. The ISME Journal, 2012, 6(1): 213–222. DOI:10.1038/ismej.2011.87 |
| [3] | 姜怡, 唐蜀昆, 张玉琴, 娄恺, 徐丽华. 放线菌产生的生物活性物质. 微生物学通报, 2007, 34(1): 188–190. |
| [4] | Parthasarathi K, Ranganathan L S, Anandi V, Zeyer J. Diversity of microflora in the gut and casts of tropical composting earthworms reared on different substrates. Journal of Environmental Biology, 2007, 28(1): 87–97. |
| [5] | Egert M, Marhan S, Wagner B, Scheu S, Friedrich M W. Molecular profiling of 16S rRNA genes reveals diet-related differences of microbial communities in soil, gut, and casts of Lumbricus terrestris L.(Oligochaeta:Lumbricidae). FEMS Microbiology Ecology, 2004, 48(2): 187–197. DOI:10.1016/j.femsec.2004.01.007 |
| [6] | Sparling G P. Ratio of microbial biomass carbon to soil organic matter carbon as a sensitive indicator of changes in soil organic matter. Australian Journal of Soil Research, 1992, 30(2): 195–207. DOI:10.1071/SR9920195 |
| [7] | Decaëns T, Mariani L, Lavelle P. Soil surface macrofaunal communities associated with earthworm casts in grasslands of the Eastern Plains of Colombia. Applied Soil Ecology, 1999, 13(1): 87–100. DOI:10.1016/S0929-1393(99)00024-4 |
| [8] | 张立宏, 许光辉. 微生物和蚯蚓的协同作用对土壤肥力影响的研究. 生态学报, 1990, 10(2): 116–120. |
| [9] | Furlong M A, Singleton D R, Coleman D C, Whitman W B. Molecular and culture-based analyses of prokaryotic communities from an agricultural soil and the burrows and casts of the earthworm Lumbricus rubellus. Applied and Environmental Microbiology, 2002, 68(3): 1265–1279. DOI:10.1128/AEM.68.3.1265-1279.2002 |
| [10] | 刘光崧. 土壤理化分析与剖面描述. 北京: 中国标准出版社, 1996. |
| [11] | Haynes R J, Fraser P M. A comparison of aggregate stability and biological activity in earthworm casts and uningested soil as affected by amendment with wheat or lucerne straw. European Journal of Soil Science, 1998, 49(4): 629–636. DOI:10.1046/j.1365-2389.1998.4940629.x |
| [12] | 史学群, 宋海超, 刘柱. 海南省土壤拮抗放线菌分离方法初探. 中国农学通报, 2006, 22(10): 431–435. |
| [13] | 阮继生. 放线菌快速鉴定与系统分类. 北京:科学出版社, 2011. |
| [14] | 蒋国彪, 马沁沁, 方志轩, 雍彬, 刘欣林, 王一丁. 一株高效溶磷小麦内生菌的筛选与鉴定. 四川师范大学学报:自然科学版, 2012, 35(1): 122–126. |
| [15] | 宋清姿, 王一丁, 周艳玲, 马沁沁, 袁向华. 耕作土壤中蚯蚓肠道可培养放线菌的多样性研究. 四川师范大学学报:自然科学版, 2016, 39(2): 284–288. |
| [16] | Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. MEGA5:molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Molecular Biology and Evolution, 2011, 28(10): 2731–2739. DOI:10.1093/molbev/msr121 |
| [17] | Wu J T, Wang L, Ma F, Yang J X, Li S Y, Li Z. Effects of vegetative-periodic-induced rhizosphere variation on the uptake and translocation of metals in Phragmites australis (Cav.) Trin ex. Steudel growing in the Sun Island Wetland. Ecotoxicology, 2013, 22(4): 608–618. DOI:10.1007/s10646-013-1052-2 |
| [18] | 韩俊, 刘海军, 原志敏, 姚俊. 石油污染土壤可培养放线菌遗传多样性分析. 环境科学与技术, 2014, 37(3): 32–36. |
| [19] | 冯鸽, 许姗姗, 王超, 严立恩, 张璇, 李静. 黄河三角洲滨海湿地非培养放线菌多样性. 微生物学通报, 2013, 40(9): 1570–1579. |
| [20] | Huber T, Faulkner G, Hugenholtz P. Bellerophon:a program to detect chimeric sequences in multiple sequence alignments. Bioinformatics, 2004, 20(14): 2317–2319. DOI:10.1093/bioinformatics/bth226 |
| [21] | Thompson J D, Gibson T J, Plewniak F, Jeanmougin F, Higgins D G. The CLUSTAL-X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Research, 1997, 25(24): 4876–4882. DOI:10.1093/nar/25.24.4876 |
| [22] | Hall T A. BioEdit:a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucleic Acids Symposium Series, 1999, 41: 95–98. |
| [23] | Stach J E M, Maldonado L A, Masson D G, Ward A C, Goodfellow M, Bull A T. Statistical approaches for estimating actinobacterial diversity in marine sediments. Applied and Environmental Microbiology, 2003, 69(10): 6189–6200. DOI:10.1128/AEM.69.10.6189-6200.2003 |
| [24] | 谢晴宜, 屈直, 马海霞, 洪葵. 克隆文库分析红树林土壤中放线菌的多样性. 热带作物学报, 2012, 32(8): 1466–1470. |
| [25] | 徐丽华, 杨宇容, 姜成林. 云南土壤放线菌生态分布的研究. 微生物学报, 1996, 36(3): 220–226. |
| [26] | Kemp P F, Aller J Y. Bacterial diversity in aquatic and other environments:what 16S rDNA libraries can tell us. FEMS Microbiology Ecology, 2004, 47(2): 161–177. DOI:10.1016/S0168-6496(03)00257-5 |
| [27] | 崔玉珍, 牛明芬. 蚯蚓粪对土壤的培肥作用及草莓产量和品质的影响. 土壤通报, 1998, 29(4): 156–157. |
| [28] | Elvira C, Sampedro L, Benítez E, Nogales R. Vermicomposting of sludges from paper mill and dairy industries with Eisenia andrei:a pilot-scale study. Bioresource Technology, 1998, 63(3): 205–211. DOI:10.1016/S0960-8524(97)00145-4 |
| [29] | Parthasarathi K, Ranganathan L S. Longevity of microbial and enzyme activity and their influence on NPK content in pressmud vermicasts. European Journal of Soil Biology, 1999, 35(3): 107–113. DOI:10.1016/S1164-5563(00)00114-X |
| [30] | Carpenter D, Hodson M E, Eggleton P, Kirk C. Earthworm induced mineral weathering:preliminary results. European Journal of Soil Biology, 2007, 43(S1). |
| [31] | Krist fek V, Tajovsky K, Pizl V. Ultrastructural analysis of the intestinal content of earthworm Lumbricus rubellus Hoffm.(Annelida, Lumbricidae). Acta Microbiologica et Immunologica Hungarica, 1994, 41(3): 283–290. |
| [32] | Bohlen P J, Scheu S, Hale C M, McLean M A, Migge S, Groffman P M, Parkinson D. Non-native invasive earthworms as agents of change in northern temperate forests. Frontiers in Ecology and the Environment, 2004, 2(8): 427–435. DOI:10.1890/1540-9295(2004)002[0427:NIEAAO]2.0.CO;2 |
| [33] | Tiunov A V, Scheu S. Microfungal communities in soil, litter and casts of Lumbricus terrestris L.(Lumbricidae):a laboratory experiment. Applied Soil Ecology, 2000, 14(1): 17–26. DOI:10.1016/S0929-1393(99)00050-5 |
| [34] | Scheu S, Schlitt N, Tiunov A V, Newington J E, Jones H T. Effects of the presence and community composition of earthworms on microbial community functioning. Oecologia, 2002, 133(2): 254–260. DOI:10.1007/s00442-002-1023-4 |
| [35] | Knapp B A, Podmirseg S M, Seeber J, Meyer E, Insam H. Diet-related composition of the gut microbiota of Lumbricus rubellus as revealed by a molecular fingerprinting technique and cloning. Soil Biology and Biochemistry, 2009, 41(11): 2299–2307. DOI:10.1016/j.soilbio.2009.08.011 |
| [36] | Huang K, Li F S, Wei Y F, Fu X Y, Chen X M. Effects of earthworms on physicochemical properties and microbial profiles during vermicomposting of fresh fruit and vegetable wastes. Bioresource Technology, 2014, 170: 45–52. DOI:10.1016/j.biortech.2014.07.058 |
| [37] | Carlson S, Tanouye U, Omarsdottir S, Murphy B T. Phylum-specific regulation of resistomycin production in a Streptomyces sp. via microbial coculture. Journal of Natural Products, 2015, 78(3): 381–387. DOI:10.1021/np500767u |
 2017, Vol. 37
2017, Vol. 37