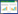文章信息
- 王姝, 周道玮.
- WANG Shu, ZHOU Daowei.
- 植物表型可塑性研究进展
- Research on phenotypic plasticity in plants: an overview of history, current status, and development trends
- 生态学报. 2017, 37(24): 8161-8169
- Acta Ecologica Sinica. 2017, 37(24): 8161-8169
- http://dx.doi.org/10.5846/stxb201611242412
-
文章历史
- 收稿日期: 2016-11-24
- 网络出版日期: 2017-08-15
2. 中国科学院东北地理与农业生态研究所, 长春 130102
2. Northeast Institute of Geography and Agroecology, Chinese Academy of Sciences, Changchun 130102, China
基因型变异和表型变异, 都被认为是进化学变化的来源。表型可塑性, 是表型变异的重要来源之一。尽管最初被认为是对基因型准确表达的干扰, 随着几篇开创性综述的发表[1-4], 表型可塑性的重要性开始广受关注。植物可以典型地反映环境条件对表型的影响, 以植物为对象的研究成为对可塑性知识的主要贡献。从现象描述, 到对可塑性的进化及其进化-生态学意义的探讨, 植物表型可塑性研究不仅涉及研究对象的不同层面和水平、包含各种非生物与生物环境因子, 还与物种的生长与适应策略等理论相联系, 使可塑性的概念渗入越来越多的研究领域, 成为新兴的生态-进化-发育生物学的核心内容[5]。
尽管如此, 人们仍远未完全理解表型可塑性的原因和结果[6]。这从很大程度上缘于可塑性研究在最基本问题(如可塑性的量化、适应性等[7-9])上缺少一致性, 需要一篇宏观视角的综述性文章对可塑性的研究内容、进展, 以及研究中存在的主要问题进行系统的论述, 以促进未来研究向更有利的方向发展。然而, 目前很多文章集中于与可塑性相关的某个或某些研究领域的进展, 缺少从整体角度介绍可塑性研究情况的综述。基于以上考虑, 本文避免了大量具体研究实例的陈列, 试图理出一条可塑性研究发展的脉络, 介绍研究内容、途径和简史的同时, 阐述可塑性的机制、适应性、进化及进化-生态学意义等主要方面的研究进展及发展趋势, 希望有利于对表型可塑性及相关问题的深入理解和探索, 推动植物可塑性研究在国内的发展。
1 表型可塑性研究 1.1 研究内容表型可塑性, 指一个基因型在不同环境下产生不同表型的现象或能力[1, 10], 也被定义为一个基因型在环境作用下能表达的表型范围[11], 或有机体在响应环境过程中改变表型的能力[5]。表型可塑性可按不同方式分类, 比如基于目标特征的特性(如形态学, 生理学, 行为等), 发育系统所解读的环境信号的特性(如食物, 种群密度, 稳定和光周期等), 在生态学背景中有机体的表现(躲避捕食者/防御, 扩散和资源获取)等;还可依据表型是可逆[12]还是不可逆[13], 对环境信号有敏感性的时期在发育早期[14]还是晚期[15], 是由个体大小或生长速率不同造成的表观可塑还是异速关系变化的真正的可塑[16]。当然, 任何分类方式都不可能穷举涉及可塑性的生物过程多样性的所有现象, 不同分类下的可塑性也并非严格地相互独立[17]。
可塑性的表达方式有很多, 最基本的一种是计算不同环境下特征平均值差异, 很多目前常用的公式衍生于此。此外, 还可直接用方差分析(ANOVA/ANCOVA)得出的环境因子导致的特征变异程度评价可塑性[18]。Valladares等总结了量化可塑性常用的17种方法, 并提出了相对距离可塑性指标和环境标准化可塑性指标, 认为其既与其他指标有较强的相关度, 又在比较不同物种时有较高的敏感性[19](表 1)。
| 可塑性指标 Index of plasticity |
计算方法 Calculation |
复合性 Complexity |
参考文献 References |
| 总变异系数Coefficient of variation-total (CVt) | 标准差/平均值(对于每个基因型的全部数据系列)(*) | 简单 | 丰富 |
| 响应范围的斜率Slope of norm of reaction | 因变量与环境的回归斜率 | 中等 | [20] |
| 可塑响应的斜率Scope of plastic response (D) | 高水平与低水平资源下的平均值的差值 | 简单 | [21] |
| 响应系数Response Coefficient (RC) | 高水平资源平均值与低水平资源平均值的比率 | 简单 | [22] |
| 环境平均值的变异系数Coefficient of variation over the environments, based on means (CVm) | 平均值的标准差/平均值的均值 | 简单 | [23-24] |
| 环境中值的变异系数Coefficient of variation over the environments, based on medians (CVm d) | 中值的标准差/中值的均值 | 简单 | [19] |
| 总可塑性Grand plasticity (Pi) | 平均值的标准差/以生物量为共变量而调整后的平均值的均值 | 简单 | [25] |
| 基于最小平方均值的可塑性指标Phenotypic Plasticity Index, based on least square means (PPF) | 100×(一个环境中的最小平方均值-另一个环境中的最小平方均值)/前一个环境中的最小平方均值 | 中等 | [26] |
| 基于最大和最小均值的可塑性指标Phenotypic Plasticity Index, based on maximum and minimum means (PIv) | (最大平均值-最小平均值)/最大平均值 | 简单 | [27-29] |
| 基于最大和最小中值的可塑性指标Phenotypic Plasticity Index, based on maximum and minimum medians (PImd) | (最大中值-最小中值)/最大中值 | 简单 | [19] |
| 基于最小平方均值的最大和最小值的可塑性指标 Phenotypic Plasticity Index, based on maximum and minimum least square means (PILSM) |
(最小平方平均值的最大值-最小值)/最大值 | 中等 | [19] |
| 相对特征范围Relative Trait Range (RTR) | (环境梯度一端的平均值-相反一端的平均值)/最大值的绝对值 | 简单 | [30] |
| 表型可塑性指标Phenotypic Plasticity Index (PIR) | (最大平均值-最小平均值)最大生长速率下的平均值 | 中等 | [31] |
| 表型惰性Phenotypic Inertia (PIN) | (∑(存活i*表现i))/(n*SD)计算n个环境的每个(i)环境 | 复杂 | [32] |
| 相对距离可塑性指标Relative Distances Plasticity Index (RDPI) | 不同环境的同一基因型的个体间表型距离的绝对值, 除以两个表型值的和(*) | 中等 | [19] |
| 简化的相对距离可塑性指标Simplified Relative Distances Plasticity Index (RDPIs) | 不同环境的同一基因型的平均值之间表型距离的绝对值, 除以两个表型平均值的和 | 简单 | [19] |
| 环境标准化可塑性指标Environmentally Standardized Plasticity Index (ESPI) | (最大平均值-最小平均值)/最大值和最小值所在的环境值之间的绝对距离(*) | 简单 | [19] |
| 个体间距离的环境标准化可塑性指标Environmentally Standardized Plasticity Index for individual distances (ESPIID) | 不同环境的同一基因型的个体间表型距离的绝对值, 除以环境值之间的绝对距离(*) | 复杂 | [19] |
| *除有星号标记的以外, 所有指标都基于一个环境内不同个体特征平均值 | |||
可塑性的研究对象是有机体个体, 因此离不开具体特征, 包括整体和构件水平的性状或特征。环境诱导的表型变化(表型可塑)常常是在构件(指植物个体重复的、经常有半自主性的结构和功能的子单元)水平上发生的, 植物整体的可塑性是环境诱导的所有构件的局部响应加上构件之间沟通和行为整合带来的相互作用效应的总和[33]。在同样的环境变异的影响下, 不同构件特征的局部响应可能有很大差异。因此可塑性研究是以特征为具体目标, 从形态、生理特征和生物量特征的描述到大量功能特征、结构特征、生活史特征等的评价。
早期可塑性研究主要在室内或野外条件下, 研究植物对单一环境因子或者两个以上环境因子交互作用的响应[34-36]。诱导可塑响应的非生物环境因素包括光照、温度、土壤营养和水分等;生物环境因素包括密度或竞争、动物采食、根瘤菌及微生物等。自然条件下, 物种生境各种环境因子的变化对物种的影响是复杂的:环境因子本身有高度的时间、空间异质性;某一环境因子信号对有机体的影响还依赖于其可预测性, 可靠性及有机体的响应时间等, 这些也是限制可塑性发展的因素[37-38]。研究环境背景复杂性的重要意义早已被反复强调, 这一点也是可塑性研究要解决的主要问题之一。
1.3 研究简史表型可塑性研究始于Woltereck对水蚤头部高度响应于营养水平变化的观测[39]。但作为一个严肃的科学领域, 表型可塑性研究的兴起较慢, 至少部分由于19世纪30、40年代强调基因型与表型之间关系的种群遗传学理论的发展[40-41]。随着进化综合理论的提出, 一些论述表型可塑性的重要文章发表于进化生物学领域, 但现代表型可塑性研究的开拓还归功于1965年Bradshaw的综述[1]。20世纪80年代, 进化的量化遗传学和最优化理论的发展导致表型可塑性研究的爆发, 大多基于进化学观点。这个时期以解决研究领域中各种问题的积极争论为特点, 很多争论涉及可塑性本身是否是自然选择的目标(以及类似“可塑性基因”是否有意义的问题)[41]。目前争论仍在持续, 但新想法、新技术的提出和旧想法的修改, 为表型可塑性研究领域增添了新的争论。进化-发育生物学的兴起, 使人们对表型变异产生的过程(相对于关注变异的大致结果)更感兴趣。同时, 表型可塑性背后的发育、分子机制也是一个活跃领域[42-43]。可塑性的成本与限制, 表型整合与可塑性之间的关系等成为人们感兴趣的问题。早期研究主要强调可塑性的进化, 关于可塑性在进化过程(种群特化、分化及物种形成等)、生态学问题(物种入侵性、多样性和分布等)中重要作用的研究已经从理论假设阶段发展到实证阶段。
2 研究进展 2.1 表型可塑性的遗传机制表型可塑性被认为受遗传控制, 包括基因和表观遗传调控机制[44]。基因水平的调控模式主要有结构基因和调控基因两种[45-46]。其中, 结构基因是构成性表达的, 表达水平直接受环境影响, 不同等位基因对环境的敏感性也有很大变化。这种观点认为, 对特征值的直接选择作用导致特征可塑性强烈的相关响应, 可塑性是很多结构基因的环境敏感性相结合的结果, 没有特定的“可塑性基因”[46]。相对照, 调控基因模式的支持者认为结构基因的表达是由调节基因介导的, 例如编码控制多种结构基因表达水平的转录因子的基因。调节基因起作用的机制是, 来自外部环境的知觉信息被有机体转换成内部可感受的信号, 刺激神经内分泌系统召集不同的转录因子, 激活不同基因[47], 产生不同的基因产物及最终表型, 从而形成有机体对不同环境信息的响应。如果调节基因的活性在环境控制下, 它们就可作为可塑性基因, 对结构基因的表达进行环境依赖性的控制, 可塑响应的形式就可独立于平均值而进化[23, 45, 48]。
可塑性受表观遗传学机制调控的证据是代际间可传递的可塑性[49], 说明表型可塑性有可遗传性[50]。表观遗传学过程, 如DNA甲基化, 可以通过DNA修饰控制基因表达。Bossdorf等把表观遗传学研究领域的实例包含到表型可塑性的生态学研究中[51]。例如, 去甲基化试剂处理对拟南芥不同基因型开花时间、开花时大小和植物生物量对营养水平的敏感性有显著影响[52]。
2.2 表型可塑性的发育过程机制除了遗传学上的控制, 可塑性还是有机体发育过程自我调节的结果。其本质是, 有机体在整个生命周期中, 通过发育过程与环境之间不断的交互作用, 发育程序的改变产生的生理、形态等方面的调整和变化[5]。无论从短期还是长期看, 可塑响应都是一个动态过程, 包括环境条件的持续影响、有机体内对环境信号的感知和整合, 以及一系列内在的传导途径的作用[53]。这方面研究已经形成一个新的交叉学科领域——生态-进化-发育生物学, 旨在理解有机体的基因组与环境之间的交互作用如何形成发育中的个体, 以及这种对环境有响应性的发育过程如何影响生态学过程和进化[54]。植物生态-发育学研究的目标首先是精确地判断植物如何感受并响应于真实世界中的环境变化[54-56]。研究指向复合的、环境信号和基因调控网络之间交互作用的本质, 并强调将现实环境变异包含到发育过程研究的重要性。这个领域中, 植物激素, 作为通过复杂的分子交互作用产生重要生态学响应的协调因子, 引起了研究者们新的兴趣[53, 57-58]。
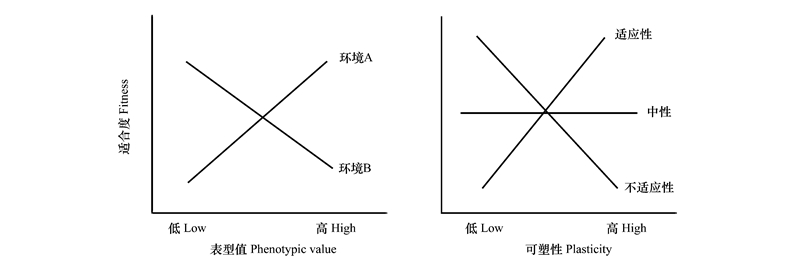
2.3 表型可塑性的适应性由于对不同方法的评价、遗传机制及可塑性受自然选择影响的方式的观点不同, 人们对适应性表型可塑性的理解受到阻碍[8]。可塑性的适应性依赖于具体环境、物种和特征[59]。假设一种可塑响应在一种环境下是适应性的, 其在另一种环境下可能是非适应性的[60];一些特征的响应是适应性的, 有利于适合度的提高, 另一些则是对生理过程或资源限制的消极响应[16, 38]。即便是单独一个特征, 其对同一个环境因子也可能同时有多种正向和负向响应趋势[38]。自然条件下, 表型可塑性不仅来自多种积极和消极环境因素的综合影响, 还是有机体通过自身的调控机制缓冲、平衡、抵抗或促进环境影响的整合性结果[38]。实际上, 是不同情况下可测的积极响应相对于消极响应的程度决定了特征可塑性的适应性。因此, 可塑性的适应性意义有高度可变性[38, 61], 常用的对可塑性的适应性的评价方法(图 1)与可塑性成本评价方法的混淆使问题更加复杂化。控制实验条件下的研究背景由于不包括所有潜在的环境选择力, 可塑性的总体适应性意义经常是不确定的[59]。最好评价自然种群中的可塑性及其结果[62], 并探索哪些条件下可塑性有最大的适应性价值, 以及这种适应性是相对于特定环境或阶段还是适用于有机体整个生命周期[59]。
与可塑性的适应性相关但不同的另一个问题是, 可塑性的成本和限制。无论可塑性是适应性的, 还是非适应性的(不适应性的和中性的), 都可能涉及产生和维持的成本和限制[37-38]。评估可塑性成本的方法是将环境中的适合度与特征值及可塑性进行相关分析(多重回归模型)[11, 37, 63-64] (图 1):一个环境下的特征值和环境之间的特征值差异都作为预测变量;控制特征平均值时, 通过适合度与环境之间特征值差异的偏回归系数估计可塑性成本[65]。当系数是负值时, 表示有成本产生;系数是正值时, 有收益产生。然而, 经验研究发现, 可塑性的成本和限制是高度可变的, 经常较弱, 难以被检测到[64, 66]。因为可塑性, 包括适应性可塑, 其产生往往同时带来收益与成本[37], 相关研究结果是收益与成本的总和;只有当成本大于收益时, 可塑性成本才能被检测到。而分离可塑性的限制、成本和收益是困难的, 可能由于各种因素的交互作用及其背景依赖性[67]。
2.5 表型可塑性的进化表型可塑性有遗传基础, 可以不依赖于平均值而进化的观点已得到普遍认可[10, 68]。Baldwin认为自然选择可以作用于可塑性的变化方向, 使可塑性程度增强或不变[69-70], Simpson将其定义为“Baldwin效应”[71]。然而, Waddington认为环境诱导的适应性可塑程度会被反复出现的环境选择降低, 变成可继承的表型(即渠道化), 被称为“遗传同化”[72-73]。哪一种机制在自然界中更普遍还不清楚。Bradshaw提出了支持可塑性的条件, 即适应性可塑发生的情况, 包括破坏性选择, 方向性选择和稳定性选择, 以及不支持可塑性的条件[1]。尽管有大量关于可塑性进化的理论假设, 仍缺少实验数据的支持。大多数研究只关注两个环境, 缺乏环境范围的数据, 例如自然种群普遍经历的环境和选择压力的变化。
2.6 表型可塑性的进化-生态学意义可塑性被认为可能是潜在的、推动宏观进化的重要机制[7, 74]。“可塑性先行”假设认为, 起初, 当外界环境发生变化时, 有机体可以通过某些特征的可塑响应使表型在新的环境下发生改变, 以免由于难以适应环境变化而死亡。随后, 通过表型与环境之间不断的交互作用, 发育系统的可塑性产生对这种表型改变的适应(包括表型适应和遗传适应)或遗传同化[75-76]。因为普遍缺乏来自自然种群的综合性检验, 这个假设目前还存在争议。另一方面, 正兴起的生态-发育生物学领域中, 可塑性在促进多样化与物种形成中的重要性已被普遍接受[54]。但表型可塑可能阻碍还是促进作用于多样化的选择仍是很多进化生物学家们长期争论的问题。
关于其生态学意义, 表型可塑性被认为对一些入侵性植物的建植与散布有重要贡献[1, 77-80], 支持[81-82]与不支持[83-85]的研究证据都有。Hendry认为可塑性有时候有助于在新环境下的定植和对生境环境变化的响应, 但在这些背景中可塑响应不总是必要或足够的[59]。这从一定程度上反映了数据的欠缺:关于面对环境变化时可塑性对种群动态贡献的研究很少。还需要关于可塑性与物种当前(当代或多代)生境适应、物种交互作用等问题之间关系的广泛研究[85]。另外, 对于这类问题以及全球变化背景下的可塑响应[11]的探讨, 可塑性的变化可能比可塑性程度更重要[85]。
3 研究展望虽然可塑性被认为是有机体适应环境的主要机制之一[86], 且已有大量相关研究, 人们距完全理解表型可塑性的原因和结果仍很远[6]。这主要有几个方面原因:首先, 人们对可塑性量化和自然选择对反应规范的影响等方面的问题还缺少共识[7], 在可塑性的适应性和进化意义的问题上还没有清晰、一致的结论[8-9]。一方面, 尽管很多定义中暗示表型可塑性是有机体通过生理、形态等方面的调整应对环境变化的能力, 其量化表达方式仍基于特征的表型差异。然而, 表型变与不变背后, 都既可能是一种自我调节能力的不足或失败, 也可能是相对更成功的积极响应。表型的可塑性和稳定性都可反映这种能力的高低, 两个过程的概念和量化方式都需重新考虑。另一方面, 可塑性必然包含环境影响和个体响应两个方面, 表型差异不显著可能因为个体响应的方向与环境影响的方向相反, 相互抵消[38]。从表型变异中区分环境影响和有机体响应两个组分, 是有效衡量有机体响应环境能力的前提。这种区分也同时有利于可塑性的适应性及成本的评价。此外, 无论可塑响应的产生, 还是其成本和限制, 不仅依赖于具体构件和特征, 还受制于特征之间的相关性, 即表型整合作用。衡量一个物种的可塑性及其适应性, 要从整体角度综合性评价多种特征的局部响应及整合作用。
其次, 可塑性的环境背景和有机体响应的复杂性。自然环境包括多种因素的共同影响和同时、异时的交互作用, 室内控制实验中往往只观测对单一环境因子的可塑响应, 且与植物生境的真实情况有很大差距[34, 53]。同时, 有机体响应于环境的过程包括多方面多水平的特征调控和变化及其交互作用, 且有特征、物种依赖性[1, 33]。要准确判断可塑性的形式, 研究其生态学和进化学意义, 必须更好地把握表型表达的环境背景的复杂性。例如, 通过更真实的环境背景下的野外实验和/或复杂的多因子实验[53, 87-89], 研究与特征响应相联系的具体环境情况, 包括环境变异的类型、空间尺度、时间安排、有效信号和可靠性、响应的速度和成本及可逆转性等[88]。另外, 物种对当下环境的可塑响应离不开其生境背景及早期环境经历的影响, 例如引发或训练、胁迫记忆[6, 85]。可塑性研究不仅要关注诱导可塑性当时的环境背景, 还应联系物种经历过的环境事件及生境特点[85]。
目前, 表型可塑性研究对象同时向更微观与更宏观水平发展。微观方向上, 结合与基因型、表观基因型有关的知识很重要, 很多细节有待阐述[6];细胞可塑性研究也开始出现[90]。还需更多研究提供在分子和细胞水平上与可塑响应产生相关的信号感受、传导途径和机制上的解释, 以及对个体内和代际之间各种表型特征如何准确表达的理解[87]。宏观上, 可塑性研究正朝向与更大尺度上的生态-进化学过程和现象相联系的方向发展。全球气候变化与植物可塑性的关系受到越来越多关注[11, 91]。另外, 很多可塑性研究局限于个别物种, 忽略了物种之间复杂的交互作用网络。需要将可塑性概念整合到多营养级关系, 如食物网或生态群落中[6]。鉴于种群内潜在的大量物种交互作用和有可塑性表现的个体特征, 这可能很有挑战性。
致谢: 感谢东北师范大学生命科学学院草地科学研究所王岭教授在本文写作中给予的帮助和建议。| [1] | Bradshaw A D. Evolutionary significance of phenotypic plasticity. Advances in Genetics, 1965, 13: 115–155. |
| [2] | Schlichting C D. The evolution of phenotypic plasticity in plants. Annual Review of Ecology and Systematics, 1986, 17(1): 667–693. DOI:10.1146/annurev.es.17.110186.003315 |
| [3] | West-Eberhard M J. Phenotypic plasticity and the origins of diversity. Annual Review of Ecology and Systematics, 1989, 20: 249–278. DOI:10.1146/annurev.es.20.110189.001341 |
| [4] | Scheiner S M. Genetics and evolution of phenotypic plasticity. Annual Review of Ecology and Systematics, 1993, 24(1): 35–68. DOI:10.1146/annurev.es.24.110193.000343 |
| [5] | Pfennig D W. Ecological evolutionary developmental biology//Kliman R M, ed. Encyclopedia of Evolutionary Biology. Oxford:Academic Press, 2016:474-481. |
| [6] | Ellers J, Stuefer J F. Frontiers in phenotypic plasticity research:new questions about mechanisms, induced responses and ecological impacts. Evolutionary Ecology, 2010, 24(3): 523–526. DOI:10.1007/s10682-010-9375-4 |
| [7] | Pigliucci M. Phenotypic Plasticity:Beyond Nature and Nurture. Baltimore, MD:Johns Hopkins University Press, 2001. |
| [8] | Via S, Gomulkiewicz R, De Jong G, Scheiner S M, Schlichting C D, Van Tienderen P H. Adaptive phenotypic plasticity:consensus and controversy. Trends in Ecology and Evolution, 1995, 10(5): 212–217. DOI:10.1016/S0169-5347(00)89061-8 |
| [9] | DeWitt T J, Scheiner S M. Phenotypic Plasticity:Functional and Conceptual Approaches. Oxford:Oxford University Press, 2004. http://onlinelibrary.wiley.com/doi/10.1002/ajhb.20088/full |
| [10] | Pigliucci M. Evolution of phenotypic plasticity:where are we going now?. Trends in Ecology & Evolution, 2005, 20(9): 481–486. |
| [11] | Nicotra A B, Atkin O K, Bonser S P, Davidson A M, Finnegan E J, Mathesius U, Poot P, Purugganan M D, Richards C L, Valladares F, Van Kleunen M. Plant phenotypic plasticity in a changing climate. Trends in Plant Science, 2010, 15(12): 684–692. DOI:10.1016/j.tplants.2010.09.008 |
| [12] | Sunobe T, Nakazono A. Sex change in both directions by alteration of social dominance in Trimma okinawae (Pisces:Gobiidae). Ethology and Sociobiology, 1993, 94(4): 339–345. |
| [13] | Greene E. Diet-induced developmental polymorphism in a caterpillar. Science, 1989, 243(4891): 643–646. DOI:10.1126/science.243.4891.643 |
| [14] | Crews D. Sex determination:where environment and genetics meet. Evolution and Development, 2003, 5(1): 50–55. DOI:10.1046/j.1525-142X.2003.03008.x |
| [15] | Applebaum S W, Heifetz Y. Density-dependent physiological phase in insects. Annual Review of Entomology, 1999, 44(1): 317–341. DOI:10.1146/annurev.ento.44.1.317 |
| [16] | Weiner J. Allocation, plasticity and allometry in plants. Perspectives in Plant Ecology, Evolution and Systematics, 2004, 6(4): 207–215. DOI:10.1078/1433-8319-00083 |
| [17] | Fusco G, Minelli A. Phenotypic plasticity in development and evolution:facts and concepts. Philosophical Transactions of the Royal Society B:Biological Sciences, 2010, 365(1540): 547–556. DOI:10.1098/rstb.2009.0267 |
| [18] | Richter S, Kipfer T, Wohlgemuth T, Guerrero C C, Ghazoul J, Moser B. Phenotypic plasticity facilitates resistance to climate change in a highly variable environment. Oecologia, 2012, 169(1): 269–279. DOI:10.1007/s00442-011-2191-x |
| [19] | Valladares F, Sanchez-Gomez D, Zavala M A. Quantitative estimation of phenotypic plasticity:Bridging the gap between the evolutionary concept and its ecological applications. Journal of Ecology, 2006, 94(6): 1103–1116. DOI:10.1111/jec.2006.94.issue-6 |
| [20] | Schlichting C D, Pigliucci M. Phenotypic Evolution:A Reaction Norm Perspective. Sunderland:Sinauer Associates Inc., 1998. |
| [21] | Stearns S C. The Evolution of Life Histories. Oxford:Oxford University Press, 1992. |
| [22] | Poorter H, Nagel O. The role of biomass allocation in the growth response of plants to different levels of light, CO2, nutrients and water:a quantitative review. Australian Journal of Plant Physiology, 2000, 27(6): 595–607. |
| [23] | Schlichting C D, Levin D A. Phenotypic plasticity:an evolving plant character. Biological Journal of the Linnean Society, 1986, 29(1): 37–47. DOI:10.1111/bij.1986.29.issue-1 |
| [24] | Schlichting C D, Levin D A. Phenotypic plasticity of annual Phlox:tests of some hypotheses. American Journal of Botany, 1984, 71(2): 252–260. DOI:10.2307/2443753 |
| [25] | Navas M L, Garnier E. Plasticity of whole plant and leaf traits in Rubia peregrina in response to light, nutrient and water availability. Acta Oecologica, 2002, 23(6): 375–383. DOI:10.1016/S1146-609X(02)01168-2 |
| [26] | Cheplick G P. Genotypic variation and plasticity of clonal growth in relation to nutrient availability in Amphibromus scabrivalvis. Journal of Ecology, 1995, 83(3): 459–468. DOI:10.2307/2261599 |
| [27] | Gratani L, Meneghini M, Pesoli P, Crescente M F. Structural and functional plasticity of Quercus ilex seedlings of different provenances in Italy. Trees, 2003, 17(6): 515–521. DOI:10.1007/s00468-003-0269-8 |
| [28] | Balaguer L, Martínez-Ferri E, Valladares F, Pérez-Corona M E, Baquedano F J, Castillo F J, Manrique E. Population divergence in the plasticity of the response of Quercus coccifera to the light environment. Functional Ecology, 2001, 15(1): 124–135. DOI:10.1046/j.1365-2435.2001.00505.x |
| [29] | Valladares F, Wright S J, Lasso E, Kitajima K, Pearcy R W. Plastic phenotypic response to light of 16 congeneric shrubs from a panamanian rainforest. Ecology, 2000, 81(7): 1925–1936. DOI:10.1890/0012-9658(2000)081[1925:PPRTLO]2.0.CO;2 |
| [30] | Richardson A D, Ashton P M S, Berlyn G P, McGroddy M E, Cameron I R. Within-crown foliar plasticity of western hemlock, Tsuga heterophylla, in relation to stand age. Annals of Botany, 2001, 88(6): 1007–1015. DOI:10.1006/anbo.2001.1538 |
| [31] | Robinson D. Phenotypic plasticity in roots and root systems:constraints, compensations and compromises. Aspects of Applied Biology, 1989(22): 49–55. |
| [32] | Milberg P, Lamont B B, Pérez-Fernández M A. Survival and growth of native and exotic composites in response to a nutrient gradient. Plant Ecology, 1999, 145(1): 125–132. DOI:10.1023/A:1009817804815 |
| [33] | De Kroon H, Huber H, Stuefer J F, Van Groenendael J M. A modular concept of phenotypic plasticity in plants. New Phytologist, 2005, 166(1): 73–82. DOI:10.1111/j.1469-8137.2004.01310.x |
| [34] | Sultan S E. Phenotypic plasticity for plant development, function and life history. Trends in Plant Science, 2000, 5(12): 537–542. DOI:10.1016/S1360-1385(00)01797-0 |
| [35] | Kurashige N S, Agrawal A A. Phenotypic plasticity to light competition and herbivory in Chenopodium album (Chenopodiaceae). American Journal of Botany, 2005, 92(1): 21–26. DOI:10.3732/ajb.92.1.21 |
| [36] | Quero J L, Villar R, Marañón T, Zamora R. Interactions of drought and shade effects on seedlings of four Quercus species:physiological and structural leaf responses. New Phytologist, 2006, 170(4): 819–834. DOI:10.1111/nph.2006.170.issue-4 |
| [37] | DeWitt T J, Sih A, Wilson D S. Costs and limits of phenotypic plasticity. Trends in Ecology & Evolution, 1998, 13(2): 77–81. |
| [38] | Van Kleunen M, Fischer M. Constraints on the evolution of adaptive phenotypic plasticity in plants. New Phytologist, 2005, 166(1): 49–60. DOI:10.1111/j.1469-8137.2004.01296.x |
| [39] | Juday C. Weitere experimentelle Untersuchungen über Artveränderung, speziellüber das Wesen quantitativer Artunterschiede bei Daphnien. By R. Woltereck, Vehr. der Deutschen Zool. Gesellsch. 1909, 19:110-172. Science, 1910, 32(819):344-345. |
| [40] | Pigliucci M. Phenotypic plasticity:Beyond nature and nutrure//Fox C W, Roff D A, Fairbairn D J, eds. Evolutionary Ecology:Concepts and Case Studies. New York:Oxford University Press, 2001. |
| [41] | Sarkar S. From the Reakionsnorm to the evolution of adaptive plasticity:A historical sketch, 1909-1999//DeWitt T J, Schiener S M, eds. Phenotypic Plasticity:Functional and Conceptual Approaches. New York:Oxford University Press, 2004:10-30. |
| [42] | Schlichting C D, Smith H. Phenotypic plasticity:linking molecular mechanisms with evolutionary outcomes. Evolutionary Ecology, 2002, 16(3): 189–211. DOI:10.1023/A:1019624425971 |
| [43] | Cossins A, Fraser J, Hughes M, Gracey A. Post-genomic approaches to understanding the mechanisms of environmentally induced phenotypic plasticity. Journal of Experimental Biology, 2006, 209(12): 2328–2336. DOI:10.1242/jeb.02256 |
| [44] | Kelly S A, Panhuis T M, Stoehr A M. Phenotypic plasticity:molecular mechanisms and adaptive significance. Comprehensive Physiology, 2012, 2(2): 1417–1439. |
| [45] | Schlichting C D, Pigliucci M. Control of phenotypic plasticity via regulatory genes. The American Naturalist, 1993, 142(2): 366–370. DOI:10.1086/285543 |
| [46] | Via S. Adaptive phenotypic plasticity:target or by-product of selection in a variable environment?. American Naturalist, 1993, 142(2): 352–365. DOI:10.1086/285542 |
| [47] | Nijhout H F. Development and evolution of adaptive polyphenisms. Evolution and Development, 2003, 5(1): 9–18. DOI:10.1046/j.1525-142X.2003.03003.x |
| [48] | Schlichting C D, Pigliucci M. Gene regulation, quantitative genetics and the evolution of reaction norms. Evolutionary Ecology, 1995, 9(2): 154–168. DOI:10.1007/BF01237754 |
| [49] | Jablonka E, Raz G. Transgenerational epigenetic inheritance:prevalence, mechanisms, and implications for the study of heredity and evolution. The Quarterly Review of Biology, 2009, 84(2): 131–176. DOI:10.1086/598822 |
| [50] | Richards C L, Bossdorf O, Pigliucci M. What role does heritable epigenetic variation play in phenotypic evolution?. BioScience, 2010, 60(3): 232–237. DOI:10.1525/bio.2010.60.3.9 |
| [51] | Bossdorf O, Richards C L, Pigliucci M. Epigenetics for ecologists. Ecology Letters, 2008, 11(2): 106–115. |
| [52] | Bossdorf O, Arcuri D, Richards C L, Pigliucci M. Experimental alteration of DNA methylation affects the phenotypic plasticity of ecologically relevant traits in Arabidopsis thaliana. Evolutionary Ecology, 2010, 24(3): 541–553. DOI:10.1007/s10682-010-9372-7 |
| [53] | Sultan S E. Plant developmental responses to the environment:eco-devo insights. Current Opinion in Plant Biology, 2010, 13(1): 96–101. DOI:10.1016/j.pbi.2009.09.021 |
| [54] | Sultan S E. Development in context:the timely emergence of eco-devo. Trends in Ecology & Evolution, 2007, 22(11): 575–582. |
| [55] | Gilbert S F. Ecological developmental biology:developmental biology meets the real world. Developmental Biology, 2001, 233(1): 1–12. DOI:10.1006/dbio.2001.0210 |
| [56] | Dusheck J. Evolutionary developmental biology:It's the ecology, stupid!. Nature, 2002, 418(6898): 578–579. DOI:10.1038/418578a |
| [57] | Gilroy S, Trewavas A. Signal processing and transduction in plant cells:the end of the beginning?. Nature Reviews Molecular Cell Biology, 2001, 2(4): 307–314. DOI:10.1038/35067109 |
| [58] | Mopper S, Wang Y Y, Criner C, Hasenstein K. Iris hexagona hormonal responses to salinity stress, leafminer herbivory, and phenology. Ecology, 2004, 85(1): 38–47. DOI:10.1890/02-0705 |
| [59] | Hendry A P. Key questions on the role of phenotypic plasticity in eco-evolutionary dynamics. Journal of Heredity, 2016, 107(1): 25–41. DOI:10.1093/jhered/esv060 |
| [60] | DeWitt T J, Robinson B W, Wilson D S. Functional diversity among predators of a freshwater snail imposes an adaptive trade-off for shell morphology. Evolutionary Ecology Research, 2000, 2(2): 129–148. |
| [61] | Auld J R, Relyea R A. Adaptive plasticity in predator-induced defenses in a common freshwater snail:altered selection and mode of predation due to prey phenotype. Evolutionary Ecology, 2011, 25(1): 189–202. DOI:10.1007/s10682-010-9394-1 |
| [62] | Nussey D H, Wilson A J, Brommer J E. The evolutionary ecology of individual phenotypic plasticity in wild populations. Journal of Evolutionary Biology, 2007, 20(3): 831–844. DOI:10.1111/jeb.2007.20.issue-3 |
| [63] | Van Tienderen P H. Evolution of generalists and specialist in spatially heterogeneous environments. Evolution, 1991, 45(6): 1317–1331. |
| [64] | Scheiner S M, Berrigan D. The genetics of phenotypic plasticity. Ⅷ. The cost of plasticity in Daphnia pulex. Evolution, 1998, 52(2): 368–378. DOI:10.1111/evo.1998.52.issue-2 |
| [65] | Lande R, Arnold S J. The measurement of selection on correlated characters. Evolution, 1983, 37(6): 1210–1226. DOI:10.1111/evo.1983.37.issue-6 |
| [66] | Sultan S E, Spencer H G. Metapopulation structure favors plasticity over local adaptation. The American Naturalist, 2002, 160(2): 271–283. DOI:10.1086/341015 |
| [67] | Auld J R, Agrawal A A, Relyea R A. Re-evaluating the costs and limits of adaptive phenotypic plasticity. Proceedings of the Royal Society B:Biological Sciences, 2010, 277(1681): 503–511. DOI:10.1098/rspb.2009.1355 |
| [68] | Scheiner S M. Plasticity as a selectable trait:reply to Via. The American Naturalist, 1993, 142(2): 371–373. DOI:10.1086/285544 |
| [69] | Baldwin J M. A new factor in evolution. The American of Naturalist, 1896, 30(354): 441–451. DOI:10.1086/276408 |
| [70] | Baldwin J M. Development and Evolution. London:MacMillan Co, 1902. |
| [71] | Simpson G G. The Baldwin effect. Evolution, 1953, 7(2): 110–117. DOI:10.1111/evo.1953.7.issue-2 |
| [72] | Waddington C H. The Strategy of the Genes. London:George Allen & Unwin Ltd., 1957. |
| [73] | Waddington C H. Genetic assimilation. Advances in Genetics, 1961, 10: 257–293. |
| [74] | West-Eberhard M J. Developmental Plasticity and Evolution. Oxford:Oxford University Press, 2003. |
| [75] | Pigliucci M, Murren C J, Schlichting C D. Phenotypic plasticity and evolution by genetic assimilation. Journal of Experimental Biology, 2006, 209(12): 2362–2367. DOI:10.1242/jeb.02070 |
| [76] | Crispo E. The Baldwin effect and genetic assimilation:revisiting two mechanisms of evolutionary change mediated by phenotypic plasticity. Evolution, 2007, 61(11): 2469–2479. DOI:10.1111/evo.2007.61.issue-11 |
| [77] | Riis T, Lambertini C, Olesen B, Clayton J S, Brix H, Sorrell B K. Invasion strategies in clonal aquatic plants:are phenotypic differences caused by phenotypic plasticity or local adaptation?. Annals of Botany, 2010, 106(5): 813–822. DOI:10.1093/aob/mcq176 |
| [78] | Sakai A K, Allendorf F W, Holt J S, Lodge D M, Molofsky J, With K A, Baughman S, Cabin R J, Cohen J E, Ellstrand N C, McCauley D E, O'Neil P, Parker I M, Thompson J N, Weller S G. The population biology of invasive species. Annual Review of Ecology and Systematics, 2001, 32(1): 305–332. DOI:10.1146/annurev.ecolsys.32.081501.114037 |
| [79] | Novak S J, Mack R N, Soltis D E. Genetic variation in Bromus tectorum (Poaceae):population differentiation in its North American range. American Journal of Botany, 1991, 78(8): 1150–1161. DOI:10.2307/2444902 |
| [80] | Baker H G. The evolution of weeds. Annual Review of Ecology and Systematics, 1974, 5: 1–24. DOI:10.1146/annurev.es.05.110174.000245 |
| [81] | Funk J L. Differences in plasticity between invasive and native plants from a low resource environment. Journal of Ecology, 2008, 96(6): 1162–1173. DOI:10.1111/jec.2008.96.issue-6 |
| [82] | Davidson A M, Jennions M, Adrienne B, Nicotra A B. Do invasive species show higher phenotypic plasticity than native species and, if so, is it adaptive? A meta-analysis. Ecology Letters, 2011, 14(4): 419–431. DOI:10.1111/ele.2011.14.issue-4 |
| [83] | Hulme P E. Phenotypic plasticity and plant invasions:is it all Jack?. Functional Ecology, 2008, 22(1): 3–7. |
| [84] | Palacio-López K, Gianoli E. Invasive plants do not display greater phenotypic plasticity than their native or non-invasive counterparts:a meta-analysis. Oikos, 2011, 120(9): 1393–1401. DOI:10.1111/more.2011.120.issue-9 |
| [85] | Wang S, Callaway R M, Zhou D W, Weiner J. Experience of inundation or drought alters the responses of plants to subsequent water conditions. Journal of Ecology, 2017, 105(1): 176–187. DOI:10.1111/1365-2745.12649 |
| [86] | Valladares F, Gianoli E, Gómez J M. Ecological limits to plant phenotypic plasticity. New Phytologist, 2007, 176(4): 749–763. DOI:10.1111/nph.2007.176.issue-4 |
| [87] | Sultan S E. Promising directions in plant phenotypic plasticity. Perspectives in Plant Ecology, Evolution and Systematics, 2004, 6(4): 227–233. DOI:10.1078/1433-8319-00082 |
| [88] | Bradshaw A D. Unravelling phenotypic plasticity-Why should we bother?. New Phytologist, 2006, 170(4): 644–648. DOI:10.1111/nph.2006.170.issue-4 |
| [89] | Bradshaw A D, Hardwick K. Evolution and stress-genotypic and phenotypic components. Biological Journal of the Linnean Society, 1989, 37(1/2): 137–155. |
| [90] | Pera M F, Tam P P L. Extrinsic regulation of pluripotent stem cells. Nature, 2010, 465(7299): 713–720. DOI:10.1038/nature09228 |
| [91] | Matesanz S, Gianoli E, Valladares F. Global change and the evolution of phenotypic plasticity in plants. Annals of the New York Academy of Sciences, 2010, 1206(1): 35–55. DOI:10.1111/nyas.2010.1206.issue-1 |
 2017, Vol. 37
2017, Vol. 37