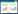文章信息
- 秦立厚, 张茂震, 钟世红, 于晓辉.
- QIN Lihou, ZHANG Maozhen, ZHONG Shihong, YU Xiaohui.
- 森林生物量估算中模型不确定性分析
- Model uncertainty in forest biomass estimation
- 生态学报. 2017, 37(23): 7912-7919
- Acta Ecologica Sinica. 2017, 37(23): 7912-7919
- http://dx.doi.org/10.5846/stxb201609281973
-
文章历史
- 收稿日期: 2016-09-28
- 网络出版日期: 2017-08-14
2. 浙江农林大学浙江省森林生态系统碳循环与固碳减排重点实验室, 临安 311300;
3. 浙江农林大学环境与资源学院, 临安 311300;
4. 北京林业大学理学院, 北京 100083
2. Zhejiang Provincial Key Laboratory of Carbon Cycling in Forest Ecosystems and Carbon Sequestration, Zhejiang Agriculture & Forestry University, Lin'an 311300, China;
3. School of Environmental & Resource Sciences, Zhejiang Agriculture & Forestry University, Lin'an 311300, China;
4. College of Science, Beijing Forest University, Beijing 100083, China
随着人类活动及现代化工业的迅速发展, 特别是化石燃料的燃烧、森林的滥伐、草原开垦等, 对地球生态系统产生着巨大的影响[1]。作为全球陆地生态系统中的最大有机碳库[2], 森林在调节全球碳平衡、减缓大气中温室气体浓度上升和维持全球气候稳定等方面具有不可替代的作用。作为反映森林生态系统生产力的重要指标, 森林生物量在计算过程中含有众多不确定性来源, 生物量的估算无论是在区域尺度上还是国家尺度上都存在极大的不确定性[3]。如何计算这些不确定性以提高森林生物量估算的准确度已成为森林生物量研究的一大挑战。
不确定性是指不精确性、模糊性、不明确性等概念总称[4]。在森林资源监测体系中, 森林资源连续清查提供了可靠的样地、样木定期观测数据。基于这些数据, 通过单木生物量模型可以获得样地森林生物量, 并可以推算到各尺度。但是从数据采集、模型建立到尺度上推过程中都包含一定的不确定性[5]。估算结果精度是否满足要求, 必须量化和分析这些不确定性。生物量估算通常包括三方面的不确定性:测量不确定性、模型不确定性、抽样不确定性。Shettles等[6]研究指出3种不确定性中, 模型不确定性所占比重较大, 约占总不确定性的70%。鉴于此, 本文仅研究不确定性较大的模型不确定性, 而测量和抽样不确定性, 本研究暂不考虑。模型不确定性来源主要有4个方面:输入变量本身的不确定性[7-8]、模型函数形式的错误设定[9]、模型残差变异[10]以及模型参数误差[11]等。输入变量本身的不确定性主要指变量(胸径、树高等)的测量误差, 测量误差主要受仪器精度、测量条件、以及人为等因素影响。与其余误差相比, 测量误差对单木生物量估算的影响相对较小[12]。模型形式的错误设定主要是因为缺少合适的模型验证数据[9]或者缺乏建模技术[13]使得生物量模型的形式设定错误。随着生物量模型研究的不断深入, 生物量模型的形式也正逐渐确定。如Araújo等[14]利用127棵实测样地数据对常用的14个生物量模型进行了验证分析, 结果表明方程式FW=aDb/(1-M)和FW=aDbHc(式中FW为生物量鲜重, D为胸径, M为含水率, H为树高)对于数据的拟合效果最好。国内王轶夫等[15], 蔡会德等[16]也对不同模型的精度进行了对比。与前两种不确定性来源相比模型残差变异以及模型参数误差研究相对较少, 在国内更是少见。模型残差变异主要与模型的拟合精度有关, 通常模型的拟合精度以决定系数(R2)来表示, R2越接近于1, 表示模型的精度越好。模型的残差变异可以以残差的标准差来度量, 但是由于不同研究的研究区及数据不同对于研究结果也有一定的差异[17-18]。与模型的残差变异相比模型的参数误差引起的不确定性相对较小, 而且模型参数误差与建模样本的数量有关[17]。目前国内对于模型不确定性对森林生物量估算的影响研究较少, 傅煜等[19-20]对森林生物量计算过程中的抽样误差和模型误差进行了研究。
本文以浙江省临安市森林资源连续清查数据为数据源, 以临安市为研究区, 通过建立了特定形式的一元、二元生物量模型来估算研究区杉木(Cunninghamia lanceolata)生物量, 并分析了两种模型形式下模型残差变异、模型参数误差带来的不确定性及模型总不确定性, 以期为提高森林生物量估计精度提供依据。
1 研究区概况与数据来源 1.1 研究区概况临安市(118°51′—119°52′E, 29°56′—30°23′N)地处浙江省西北部, 隶属杭州市, 东西长约100 km, 南北宽约70.8 km, 总面积3126 km2, 研究区地理位置如图 1所示。
|
| 图 1 研究区位置和样地分布图 Fig. 1 Location of the study area and the plots distribution |
临安市地形复杂、海拔高低悬殊, 因此气候基本呈垂直变化分布, 光、温、水地域差异明显。该市地处亚热带季风气候区, 温暖湿润, 四季分明, 光照充足, 雨量充沛, 全年平均气温16.4℃, 全年日照时数1847.3 h, 全年降雨量1628.6 mm。适宜各种林木生长, 植物资源丰富。
全市林业用地面积26.1047万hm2, 活立木总蓄积量830.0101万m3, 森林覆盖率76.55%, 其中有林地面积24.0389万hm2, 有林地蓄积量8230.5676万m3。森林资源分布格局为西高东低。植被区划属亚热带东部常绿阔叶林区, 森林类型主要有常绿落叶阔叶混交林、针叶阔叶混交林、常绿阔叶林、马尾松林、杉木林、毛竹林以及灌丛等类型。
1.2 样地清查数据研究区地面调查数据为2009年临安市森林资源连续清查样地数据, 全市共有固定样地109个, 样地为面积0.08 hm2的正方形。所有样地共有样木8179棵, 其中杉木2535棵, 其统计特征见表 1。由于样地数据中缺乏树高因子, 因此文中树高通过陈丽聪等[21]的树高曲线来求得。
| 株数 Number |
最大值 Max/cm |
最小值 Min/cm |
平均值 Mean/cm |
标准差 SD/cm |
| 2535 | 33.7 | 5.0 | 10.45 | 3.9 |
建立杉木地上部分生物量模型所用数据为52棵浙江省杉木地上部分生物量实测数据。数据收集采用收获法对样木生物量进行测定。全部样木都实测胸径、地径, 将样木伐倒后, 测量其树干长度(树高), 分树干、树枝、树叶称鲜重, 并分别抽取样品带回实验室, 在85℃恒温下烘干至恒重, 称量样本干重, 算出含水率, 然后换算出样木各组分的干重, 汇总得到立木生物量。建模数据统计情况见表 2。
| 项目 Item | 最大值 Max | 最小值 Min | 平均值 Mean | 标准差 SD |
| 胸径 DBH/cm | 37.1 | 4.2 | 17.39 | 7.99 |
| 树高 H/m | 21.1 | 4.2 | 10.58 | 4.07 |
| 地上部分生物量 Above ground biomass/kg | 430 | 2.5 | 85.58 | 80.56 |
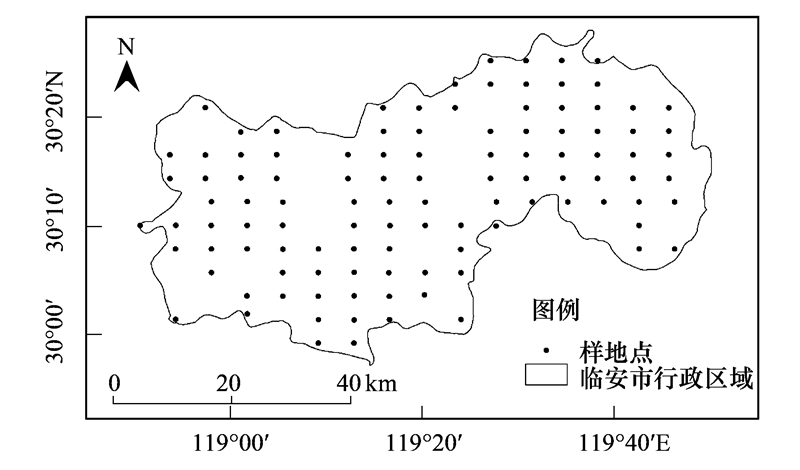
为了计算模型残差变异引起的不确定性, 本研究首先需要用52株杉木生物量实测数据拟合杉木单木生物量模型。再通过生物量预测值与实测值的差值来计算由残差变异引起的不确定性。设单木生物量实测值为B, 生物量预测模型为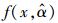
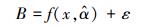
|
(1) |
上式中, 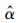
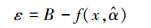
|
(2) |
对于模型残差变异引起的不确定性, 可以通过残差的标准差来衡量。本研究发现残差的标准差随单木生物量的增加而增加, 呈一定的线性关系。因此, 可以通过拟合残差的标准差与单木生物量的线性关系来计算残差变异不确定性, 即:
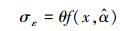
|
(3) |
式中, σε为残差的标准差, θ为拟合参数。在计算σε时, 本文采用Hosmer和Lemeshow [22]所使用的六步法(six-step procedure)来计算。具体步骤可概括为:(1)将生物量实测数据按升序进行排列, 计算单株样木的生物量预测值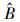
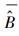
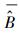
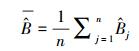
|
(4) |
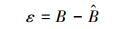
|
(5) |
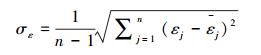
|
(6) |
式中, 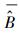
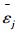
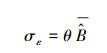
|
(7) |
θ为要拟合的参数。
2.2 模型参数不确定性计算对于模型参数引起的不确定性, 可以采用一阶泰勒级数线性化处理对参数的不确定性进行计算, Box[23]运用泰勒级数一阶展开项计算了非线性模型的参数误差, 取得较好的效果。若函数f(x)在x0处存在n阶导数, 当x趋近于x0时, f(x)可表示为:
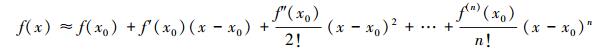
|
(8) |
那么, 对于生物量模型B= f(x, α)泰勒级数一阶展开式可以表示为:
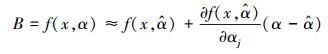
|
(9) |
式中, 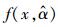

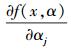
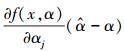
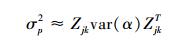
|
(10) |
式中, 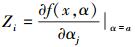
如果间接测量的量是直接测量所得到的各个测量值的函数。则间接测量的不确定度可表示为各个直接测量不确定度的合成不确定度[25]。若间接测量的函数关系为:
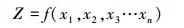
|
(11) |
则Z的不确定度可以表示为:
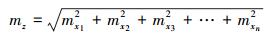
|
(12) |
式中mz表示总不确定度, mxi表示第i个变量的不确定度。根据此式, 单木生物量计算总不确定性σtree可以表示为:
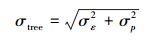
|
(13) |
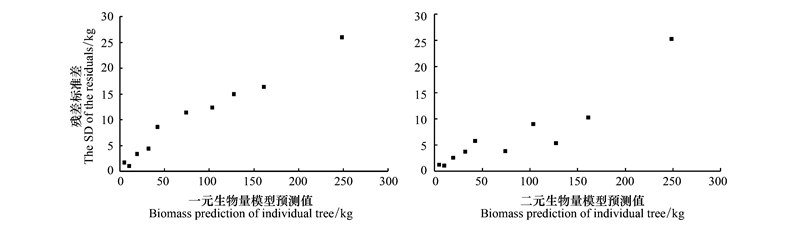
通过52棵杉木生物量实测数据对一元生物量模型进行参数求解, 最终拟合的一元生物量模型为:B=0.074D2.371, 模型的决定系数R2=0.975。基于一元生物量模型估算的临安市杉木生物量密度为6.94 Mg/hm2。图 2左图为一元生物量模型残差标准差与单木生物量预测值的散点图, 从图中可以看出两者基本成线性关系, 残差标准差随着单木生物量的增加而增加。对于一元生物量模型残差变异引起的不确定性, 最后拟合模型为: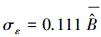
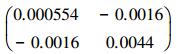
|
| 图 2 残差的标准差与生物量预测值的关系 Fig. 2 The relationship between the SD of the residuals and biomass prediction |
对于二元生物量模型, 最后拟合的生物量模型为:B=0.086D1.979H0.419, 模型的决定系数R2=0.984。基于二元生物量模型估算的临安市杉木生物量密度为7.71 Mg/hm2。图 2右图为二元生物量模型残差标准差与单木生物量预测值的散点图。从图中可以看出, 二元生物量模型残差标准差与单木生物量预测值的关系与一元生物量模型有相同的趋势。残差变异不确定性模型为: 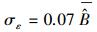
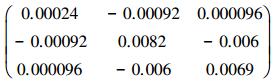
为了体现建模样本数量对模型引起的生物量估算结果的影响, 本文随机抽取了建模样本N=30, N=40, N=52时模型参数不确定性。表 3为建模样本分别为30, 40, 52时模型参数引起的不确定性。由表可知当建模样本由30增加到40再增加到52时, 一元模型参数不确定性相应为20.26%, 16.19%和14.4%。二元模型参数不确定性由13.09%减少到9.4%再减少到8.53%。说明建模样本对于模型参数引起的不确定性影响较为显著。随着样本数量的增加模型参数不确定性逐渐降低。
| 模型 Model | N=30 不确定性 Uncertainty |
N=40 不确定性 Uncertainty |
N=52 不确定性 Uncertainty |
| 一元模型 Unary model | 20.26% | 16.19% | 14.4% |
| 二元模型 Binary model | 13.09% | 9.4% | 8.53% |
表 4为不同建模样本下模型残差变异引起的生物量不确定性。在计算不同建模样本数对模型残差变异不确定性的影响时, 不同建模样本间保持相同的分组数, 通过改变每组的样本进行讨论。表 4显示, 当建模样本分别为30, 42, 48时, 一元生物量模型的残差变异引起的不确定性分别为15.2%, 12.3%, 11.7%, 二元生物量模型的不确定性为13.3%, 9.4%, 8.7%。可见, 随着建模样本的增加模型残差变异不确定性也相应变小。
| 模型 Model | N=30 | N=42 | N=48 | |||||
| 不确定性 Uncertainty |
决定系数 R2 |
不确定性 Uncertainty |
决定系数 R2 |
不确定性 Uncertainty |
决定系数 R2 |
|||
| 一元模型 Unary model | 15.2% | 0.967 | 12.3% | 0.976 | 11.7% | 0.935 | ||
| 二元模型 Binary model | 13.3% | 0.936 | 9.4% | 0.955 | 8.7% | 0.859 | ||
| 表中决定系数是指公式(7)拟合的决定系数 | ||||||||
由于本研究建模样本较少, 在分组计算模型残差变异时每组仅有5株, 样本数量较少。为了体现不同分组数据对模型残差变异的影响, 本文分别计算了每组8棵、10棵、15棵时残差变异不确定性。需要指出的是为了保证每种分组方式具有一定的组数, 分组时部分数据与上一组是重复的, 不同分组情况下重复的株数分别为3棵, 4棵, 5棵。不同分组情况下, 模型残差变异引起的不确定性如表 5所示。
| 模型 Model |
每组8棵 8 trees in each group | 每组10棵 10 trees in each group | 每组15棵 15 trees in each group | |||||
| 不确定性 Uncertainty |
决定系数 R2 |
不确定性 Uncertainty |
决定系数 R2 |
不确定性 Uncertainty |
决定系数 R2 |
|||
| 一元模型 Unary model | 11.9% | 0.975 | 11.9% | 0.972 | 12.1% | 0.974 | ||
| 二元模型 Binary model | 9.2% | 0.851 | 8.7% | 0.803 | 9.4% | 0.894 | ||
| 表中决定系数是指公式(7)拟合的决定系数 | ||||||||
从表 5可知, 当分组株数分别为8、10、15时, 一元生物量模型残差变异引起的不确定性分别为11.9%、11.9%、12.1%, 要高于每组5棵时的11.1%, 但相差不大。二元生物量模型残差变异引起的不确定性分别为9.2%、8.7%、9.4%, 高于每组5棵时的不确定性。不同的分组虽然对模型参数引起的不确定性有一定影响, 但影响较小。
4 结论与讨论本文分别计算了生物量模型中所包含的2种不确定性, 从以上结果可得出如下结论:(1)在2种不确定性来源中模型参数不确定性对估计结果影响最大, 模型残差变异引起的不确定性较小。(2)模型残差变异不确定性、参数不确定性与建模样本有关, 随着建模样本的增加, 两种不确定性相应减少。在进行生物量建模时, 可以通过增加建模样本来提高单木生物量模型的估计精度, 减小模型不确定性对森林生物量不确定性的影响。
目前区域森林植被生物量估计研究是目前研究的热点问题, 该领域还面临着两大挑战[26], 其中一个就是由于数据不准确、方法不得当, 使得森林生物量的估计存在众多不确定性。由于森林管理和决策的质量高低受森林生物量估计中所包含的不确定性影响, 生物量的估算迫切需要提高估测技术、数据质量来提高生物量估算的精度。本文分别计算模型各不确定性来源对森林生物量估算结果的影响, 其意义正在于此。与其他研究相比, 本研究还存在以下差异:(1)本研究模型残差变异的不确定性要小于国外现有研究。如Chen等[17], Chave等[18]曾对热带雨林地区生物量模型残差变异引起的误差坐了估算, 两者的结果分别为37.8%和31.3%。与本文的研究结果差距较大, 这可能由两方面原因造成:一是研究区与研究对象的不同, Chen、Chave中所用到的生物量模型为热带所有树种的生物量模型, 由于各树种之间木材密度或其他树种特征差异较大, 导致了较大的生物量残差。而本文生物量模型建模对象仅为单一树种——杉木。二是建模样本的差异。如Chen等[17]所用的建模样本为4004棵, 胸径范围为5—212cm[27], 而本研究中最大胸径为37.1cm远远小于Chen等建模样本。(2).本研究模型参数误差引起的不确定性较大。可能的原因为模型参数误差与建模样本数量有关。Chen等[17]利用4004棵样木数据计算了模型形式为a(ρD2H)b模型参数带来的不确定性, 研究表明当样木数据集足够大时由模型参数引起的不确定性很小仅为0.74%, 但样木数量减少到40棵时模型参数误差增加到5.69%。另外, 测算模型参数误差导致的不确定性有赖于生物量模型参数的协方差阵。本文杉木的建模数据52株, 由此推算整个研究区上万株林木的模型参数不确定性, 建模数据量可能偏小, 导致模型参数不确定性的度量结果偏大[19];而且本文生物量建模精度较高, 使得模型残差变异不确定性相应减小, 从而导致了模型参数误差引起的不确定性要大于模型残差变异引起的不确定性。
本研究生物量建模样本较少, 对于分组样本数对残差变异不确定性的影响本文最多讨论到每组15株的情况, 更高样本数对残差变异不确定性是否有影响尚需深入研究。
| [1] | Keeling C D. Climate change and carbon dioxide:an introduction. Proceedings of the National Academy of Sciences of the United States of America, 1997, 94(16): 8273–8274. DOI:10.1073/pnas.94.16.8273 |
| [2] | Dixon R, Solomon A M, Brown S, Houghton R E A, Trexier M C, Wisniewski J. Carbon pools and flux of global forest ecosystems. Science, 1994, 263(5144): 185–190. DOI:10.1126/science.263.5144.185 |
| [3] | Tans P P, Fung I Y, Takahashi T. Observational constraints on the global atmospheric CO2 budget. Science, 1990, 247(4949): 1431–1438. DOI:10.1126/science.247.4949.1431 |
| [4] | Shi W Z. Principles of Modeling Uncertainties in Spatial Data and Spatial Analyses. Boca Raton: CRC Press, 2009. |
| [5] | Cohen R, Kaino J, Okello J A, Bosire J O, Kairo J G, Huxham M, Mencuccini M. Propagating uncertainty to estimates of above-ground biomass for Kenyan mangroves:a scaling procedure from tree to landscape level. Forest Ecology and Management, 2013, 310: 968–982. DOI:10.1016/j.foreco.2013.09.047 |
| [6] | Shettles M, Hilker T, Temesgen H. Examination of uncertainty in per unit area estimates of aboveground biomass using terrestrial LiDAR and ground data. Canadian Journal of Forest Research, 2016, 46(5): 706–715. DOI:10.1139/cjfr-2015-0265 |
| [7] | Kitahara F, Mizoue N, Yoshida S. Evaluation of data quality in Japanese national forest inventory. Environmental Monitoring and Assessment, 2009, 159: 331–331. DOI:10.1007/s10661-008-0632-8 |
| [8] | Westfall J A, Patterson P L. Measurement variability error for estimates of volume change. Canadian Journal of Forest Research, 2007, 37(11): 2201–2210. DOI:10.1139/X07-082 |
| [9] | Breidenbach J, Antón-Fernández C, Petersson H, McRoberts R E, Astrup P. Quantifying the model-related variability of biomass stock and change estimates in the norwegian national forest inventory. Forest Science, 2014, 60(1): 25–33. DOI:10.5849/forsci.12-137 |
| [10] | Lessard V C, McRoberts R E, Holdaway M R. Diameter growth models using minnesota forest inventory and analysis data. Forest Science, 2001, 47(3): 301–310. |
| [11] | Ståhl G, Holm S, Gregoire T G, Gobakken T, Næsset E, Nelson R. Model-based inference for biomass estimation in a LiDAR sample survey in Hedmark County, Norway. Canadian Journal of Forest Research, 2010, 41(1): 96–107. |
| [12] | 赵平安, 张茂震, 陈金星, 金雨菲, 郭含茹, 何卫安. 区域森林碳分布空间估计误差定量分析. 西南林业大学学报, 2013, 33(1): 60–65, 70-70. |
| [13] | Berger A, Gschwantner T, McRoberts R E, Schadauer K. Effects of measurement errors on individual tree stem volume estimates for the Austrian national forest inventory. Forest Science, 2014, 60(1): 14–24. DOI:10.5849/forsci.12-164 |
| [14] | Araújo T M, Higuchi N, de Carvalho Júnior J A. Comparison of formulae for biomass content determination in a tropical rain forest site in the state of Pará, Brazil. Forest Ecology and Management, 1999, 117(1-3): 43–52. DOI:10.1016/S0378-1127(98)00470-8 |
| [15] | 王轶夫, 孙玉军. 马尾松生物量模型的对比研究. 中南林业科技大学学报, 2012, 32(10): 29–33. |
| [16] | 蔡会德, 农胜奇, 张伟, 江锦峰, 熊晓斐, 刘峰. 广西主要树种立木生物量模型的研建. 林业资源管理, 2014(4): 58–61. |
| [17] | Chen Q, Laurin G V, Valentini R. Uncertainty of remotely sensed aboveground biomass over an African tropical forest:propagating errors from trees to plots to pixels. Remote Sensing of Environment, 2015, 160: 134–143. DOI:10.1016/j.rse.2015.01.009 |
| [18] | Chave J, Condit R, Aguilar S, Hernandez A, Lao S, Perez R. Error propagation and scaling for tropical forest biomass estimates. Philosophical Transactions of the Royal Society B:Biological Sciences, 2004, 359(1443): 409–420. DOI:10.1098/rstb.2003.1425 |
| [19] | 傅煜, 雷渊才, 曾伟生. 区域尺度杉木生物量估计的不确定性度量. 林业科学, 2014, 50(12): 79–86. |
| [20] | 傅煜, 雷渊才, 曾伟生. 单木生物量模型估计区域尺度生物量的不确定性. 生态学报, 2015, 35(23): 7738–7747. |
| [21] | 陈丽聪, 邓华锋, 黄国胜, 程志楚. 不同起源马尾松与杉木林分树高曲线的拟合及对比. 西北农林科技大学学报:自然科学版, 2014, 42(1): 57–64. |
| [22] | Hosmer D W Jr, Lemeshow S. Applied Logistic Regression. New York: John Wiley & Sons, 1989. |
| [23] | Box M J. Bias in nonlinear estimation. Journal of the Royal Statistical Society. Series B (Methodological), 1971, 33(2): 171–201. |
| [24] | Beck J V, Arnold K J. Parameter Estimation in Engineering and Science. New York: John Wiley & Sons, 1977. |
| [25] | 李金海. 误差理论与测量不确定度评定. 北京: 中国计量出版社, 2003. |
| [26] | Wang G X, Oyana T, Zhang M Z, Adu-Prah S, Zeng S Q, Lin H, Se J Y. Mapping and spatial uncertainty analysis of forest vegetation carbon by combining national forest inventory data and satellite images. Forest Ecology and Management, 2009, 258(7): 1275–1283. DOI:10.1016/j.foreco.2009.06.056 |
| [27] | Chave J, Réjou-Méchain M, Búrquez A, Chidumayo E, Colgan M S, Delitti W B C, Duque A, Eid T, Fearnside P M, Goodman R C, Henry M, Martínez-Yrízar A, Mugasha W A, Muller-Landau H C, Mencuccini M, Nelson B W, Ngomanda A, Nogueira E M, Ortiz-Malavassi E, Pélissier R, Ploton P, Ryan C M, Saldarriaga J G, Vieilledent G. Improved allometric models to estimate the aboveground biomass of tropical trees. Global Change Biology, 2014, 20(10): 3177–3190. DOI:10.1111/gcb.2014.20.issue-10 |
 2017, Vol. 37
2017, Vol. 37