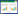文章信息
- 杨丽平, 常会会, 李杰, 张智斌, 黄原
- YANG Liping, CHANG Huihui, LI Jie, ZHANG Zhibin, HUANG Yuan.
- 基于DNA复合条形码技术的蝗虫肠道共生真菌多样性研究
- Study of the biodiversity in intestinal symbiotic fungi in grasshoppers species by using DNA meta-barcoding
- 生态学报. 2017, 37(20): 6905-6913
- Acta Ecologica Sinica. 2017, 37(20): 6905-6913
- http://dx.doi.org/10.5846/stxb201606211216
-
文章历史
- 收稿日期: 2016-06-21
- 网络出版日期: 2017-06-01
DNA复合条形码是指利用高通量测序技术同时获得很多物种的条形码序列以进行群落分类单元组成的鉴定方法[1]。该技术可以对大量混合样本快速地进行物种鉴定, 避免了单物种DNA条形码鉴定的低效率, 其快速、可重复、高效等特点让更多的科研人员和项目管理者能够轻松进行生物多样性评估。随着高通量测序技术的发展、条形码数据库的完善和生物信息学软件的开发, DNA复合条形码技术已成为微生物多样性检测的常用技术。
DNA复合条形码从诞生以来已普遍应用于微生物群落的多样性研究, 在共生真菌多样性研究方面, Schmidt[2]通过对德国弗勒斯海姆的16个土壤样本的真菌进行内转录间隔区ITS1(internal transcribed spacer)区域的PCR扩增, 将扩增产物进行高通量第二代测序, 结果得到3320个操作分类单元OTU(operational taxonomic units), 同时表明群落相似性随着地理位置的疏远逐渐降低;Siddique[3]采用复合条形码技术, 对3个不同地区设有梯度海拔的欧洲桦木树叶的真菌群落多样性进行了分析, 总结出一个关于真菌全长ITS的双末端测序的准确且有效的数据处理流程;燕勇等[4]通过对3株真菌的rDNA-ITS序列进行分析, 探讨基于ITS多态性的序列分析在真菌鉴定中的应用;隋心等[5]利用Tiangen DP330土壤提取试剂盒对土壤总DNA进行提取并对真菌的ITS-PCR体系进行优化, 建立了最佳ITS-PCR扩增体系。此外, DNA复合条形码在动物多样性[6]、植物多样性[7]、动物食性分析和排泄物[8]等方面都有广泛的应用。
蝗虫是世界性的农业害虫, 在我国分布范围广、种群数量大。近年来, 受异常气候及生态环境恶化等因素的影响, 我国蝗虫灾害的发生逐年加重, 严重影响农业、畜牧业的可持续发展和农田、草原生态环境质量[9]。了解蝗虫肠道内共生真菌, 掌握共生菌与宿主的关系以及肠道真菌群落组成, 对于利用寄生真菌或真菌代谢产生的有机物防治蝗虫有潜在的应用价值。
目前对昆虫肠道共生真菌的研究有限。国内有文献报道了取食不同品种茶叶的茶尺蠖幼虫肠道真菌[10]和黑翅土白蚁(Odontotermes formosanus)肠道中分离得到了一株产漆酶内生真菌[11];国外有文献报道了在哥斯达黎加的热带雨林中以木质为生的甲虫肠道中的细菌和真菌, 得到真菌3门16目24科40属, 且木霉属是最丰富的真菌属[12]。关于蝗虫肠道共生真菌多样性的研究就比较少。
本文选择真菌核糖体RNA基因簇的内转录间隔区(ITS)作为条形码标记, 用DNA复合条形码技术对蝗虫肠道共生真菌组成和群落结构进行了初步研究, 用α和β多样性以及聚类分析比较了不同蝗虫肠道中真菌多样性组成的差别, 初步揭示了蝗虫肠道真菌的多样性。
1 材料与方法 1.1 样品采集研究使用的蝗虫标本于2015年9—11月间采自秦岭南五台山和陕西师范大学校园(表 1)。实验前使蝗虫饥饿过夜, 排空体内食物残渣, 用已灭菌的镊子从蝗虫的头胸部背间膜将肠道轻轻取出并立即浸泡在提前已装2/3无水乙醇的50 mL的离心管中, 同时将虫体一并放入管中等待鉴定, 保存于-20℃冰箱中用于DNA的提取。
| 物种 Species |
采样地 Location |
样本编码 Sample code |
数量 Quantity |
采样时间 Date |
鉴定结果 Identification result |
|
| 斑翅蝗科 | 疣蝗Trilophidia annalata | 南五台 | N-A | 3 | 09-13 | 9纲12目 |
| Oedipodidae | 校园 | X-A | 3 | 09-21 | 10纲12目 | |
| 锥头蝗科 | 短额负蝗Atractomorpha sinensis | 南五台 | N-B | 3 | 09-13 | 6纲8目 |
| Pyrgomorphidae | 校园 | X-B | 3 | 09-21 | 7纲9目 | |
| 剑角蝗科 | 中华剑角蝗Acrida cinerea | 南五台 | N-C | 1 | 09-13 | 6纲8目 |
| Acrididae | 校园 | X-C | 1 | 09-21 | 8纲9目 | |
| 网翅蝗科 | 中华雏蝗Chorthippus chinensis | 南五台 | N-D | 3 | 09-15 | 4纲5目 |
| Arcypteridae | 素色异爪蝗Euchorthippus unicolor | 南五台 | N-G | 3 | 10-21 | 5纲7目 |
| 斑腿蝗科 | 秦岭小蹦蝗Pedopodisma tsi nlingensis | 南五台 | N-E | 1 | 09-15 | 2纲3目 |
| Catantopidae | 峨眉腹露蝗Fruhstorferiola omei | 南五台 | N-F | 1 | 10-21 | 2纲2目 |
| 短角外斑腿蝗Xenocatantops brachycerus | 南五台 | N-H | 1 | 10-21 | 5纲5目 |
将浸泡在无水乙醇中的蝗虫肠道用已灭菌过的镊子取出, 轻放在吸水纸上, 此过程需在无菌操作台进行, 待无水乙醇挥发干净后, 采用酚-氯仿方法[13]对其进行总DNA提取, 得到11个DNA样品, 样品标号如表 1所示:
1.3 PCR扩增以及测序根据White[14]等类似研究所采用的引物, 实验扩增真菌ITS的正向引物为ITS5:5′-GGAAGTAAAAGTCGTAACAAGG-3′, 反向引物为ITS4:5′-TCCTCCGCTTATTGATAT GC-3′, 扩增长度约为750bp。为了能够同时对多个样品进行测序, 以及在后续分析中区分每个样品序列, 在每个样品的5′端都加了一个7bp的标签序列。
实验中PCR体系为20 μL, 包括10 μL的2×Taq PCR StarMix with Loading Dye, 1.0 μL的DNA模版, 上游引物(10 μM)和下游引物(10 μM)各1 μL, 7 μL的双蒸水。ITS的扩增循环程序是:98℃ 5 min预热, 27个循环:98℃ 30 s, 47.6℃ 45 s, 72℃ 60 s, 72℃ 5 min延伸, 4℃保温。
PCR产物送往上海派森诺生物科技有限公司进行高通量测序, ITS采用454 FLX+平台进行测序。
2 数据处理和分析 2.1 原始序列处理:质量过滤和嵌合体的去除首先, 将测序得到的原始数据以sff格式保存, sff文件是一种二进制文件, 用mothur[15]软件中的sffinfo命令从中提取fasta序列文件和qual质量文件, 这一过程就已经对原始数据进行了初步的质量过滤。然后, 根据序列中用于区分样品的一段碱基序列信息提取每个样品的有效序列。
由于PCR扩增可能会产生嵌合体序列, 测序过程中会产生点突变等测序错误, 为了保证结果的准确性, 需要对有效序列进一步的过滤和去除嵌合体。实验用Qiime[16]软件进行序列过滤, 用mothur软件去除嵌合体序列。
2.2 OTU分类和注释使用Qiime软件中的uclust[17]对所得到的优质序列按照0.97的相似度进行OTU聚类, 选取每个OTU中最长序列为代表序列;将ITS的代表序列与数据库Unite比对, 获得每个OTU分类学信息。然后, 根据注释得到的精简OTU列表, 用excel和mothur软件做目水平上的群落百分比堆积图和样品间Venn图。
2.3 群落多样性分析根据物种丰度, 用mothur软件中的summary.single命令, 求出每个样品的Chao1, ACE, Shannon和Simpson指数。其中Chao1和ACE指数是群落丰富度指数, 二者的值越大, 说明群落丰富度越高;Shannon和Simpson指数是群落多样性指数, Simpson指数越大, 说明群落多样性越低, Shannon指数越大, 说明群落多样性越高。
用Qiime软件, 根据各样品的物种进化和丰度信息, 进行Unifrac[18-19]分析, 得到样品间差异距离矩阵, 然后进行非度量多维尺度法NMDS(non-metric multi-dimensional scaling)分析。
2.4 聚类分析根据精简后的OTU列表, 用R软件中的pheatmap程序包进行属水平上的聚类分析, 并绘制出heatmap[20-21]图。
3 结果 3.1 测序质量评价经最初的质量过滤与拼接后真菌ITS得到的有效序列有131207条, 根据序列质量控制标准, 经优化处理后共获得130600条序列, 优质序列所占比例为99.54%。各个样本的序列数及所占比例见下表 2。
| 样本 Sample |
有效序列 Effective sequence |
优质序列 High quality sequence |
% |
| N-A | 10786 | 10539 | 97.71 |
| N-B | 11187 | 11181 | 99.95 |
| N-C | 14258 | 14228 | 99.79 |
| N-D | 13357 | 13339 | 99.87 |
| N-E | 12251 | 12167 | 99.31 |
| N-F | 9896 | 9893 | 99.97 |
| N-G | 14499 | 14494 | 99.97 |
| N-H | 12592 | 12518 | 99.41 |
| X-A | 13130 | 13057 | 99.44 |
| X-B | 7415 | 7355 | 99.19 |
| X-C | 11836 | 11829 | 99.94 |
| 合计Total | 131207 | 130600 | 99.54 |
将ITS鉴定所得的优质序列与数据库比对, 共鉴定了5门16纲29目40属, 其中各样本组成情况见表 1。在总层次上OTU的分类情况如图 1所示, ITS鉴定得到N-A和N-B的共有OTU是282, 特有OTU分别是478和268;N-C和N-D的共有OTU是6, 特有OTU分别是220和85;N-E和N-F的共有OTU是6, 特有OTU分别是101和88;N-G和N-H共有OTU是1, 特有OTU分别是214和105;X-A和X-B的共有OTU是1, 特有OTU分别是933和544;X-B和X-C的共有OTU是19, 特有OTU分别是526和186, 同时还可以看出, 各样本之间存在着明显的菌群多样性变化。
|
| 图 1 ITS在总层次上组间OTU的Venn图 Fig. 1 The Venn diagram of interblock for ITS in the administrative level 图中N-A—N-H分别代表南五台每个样本的总和, X-A—X-C分别代表学校每个样本的总和 |
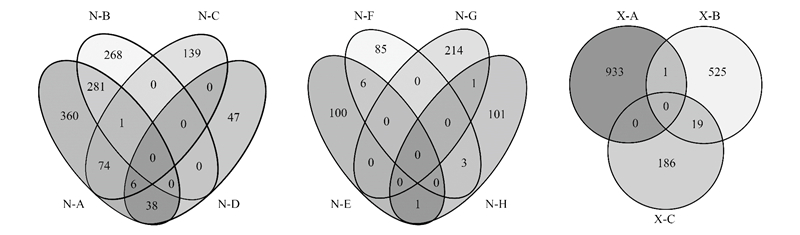
根据OTU列表, 可以用样品在各分类水平(门、纲、目、科、属)上的物种组成比例情况, 来反应样品在不同分类水平上的群落结构。对不同样本的真菌群落组成比较可知(图 2), 真菌银耳目(Tremellales)在N-A、N-B、N-G、N-H、X-A和X-B样本中微生物群落结构所占比例最高, 均约50%, 而在N-E、N-F和X-C样本中所占比例较低, 不超过30%;真菌木耳目(Auriculariales)在N-D样本中群落结构所占比例最高达50%, 其余目在各个样本中所占比例相对较少。由此可见, 真菌中银耳目和木耳菌目在蝗虫肠道中微生物群落结构最丰富。
|
| 图 2 不同蝗虫肠道真菌在目水平上的群落结构组成 Fig. 2 The community composition of Fungi at the Order level among different grasshopper species |
根据OTU聚类分析结果, 采用Chao1, ACE, Simpson, Shannon评估指数分别对样本进行多样性分析[22]。Chao1和ACE指数表明N-A和X-A的群落丰富度相对最高, N-E、N-F和N-H的群落丰富度则相对最低;Simpson和Shannon指数表明N-A和X-A的群落多样性相对最高, 而N-D、N-E和N-F的则相对最低。综合以上可以得出:N-A和X-A的群落丰富度和群落多样性相对最高, 而N-E和N-F的则相对最低。各样本多样性指数见表 3。
| 样本 Samples |
Chao1指数 Chao1 index |
ACE指数 ACE index |
Simpson 指数 Simpson index |
Shannon 指数 Shannon index |
| N-A | 907.88 | 896.97 | 0.99 | 8.23 |
| N-B | 652.43 | 661.59 | 0.99 | 7.61 |
| N-C | 283.23 | 260.55 | 0.98 | 6.27 |
| N-D | 213.00 | 199.52 | 0.90 | 5.06 |
| N-E | 121.25 | 114.88 | 0.95 | 5.01 |
| N-F | 97.00 | 97.17 | 0.96 | 5.42 |
| N-G | 275.64 | 275.70 | 0.98 | 6.31 |
| N-H | 163.33 | 128.78 | 0.98 | 5.69 |
| X-A | 1124.45 | 1127.59 | 0.99 | 8.30 |
| X-B | 666.28 | 659.88 | 0.99 | 7.90 |
| X-C | 270.18 | 254.40 | 0.96 | 5.75 |
用样品之间物种进化和丰度信息, 进行Unifrac分析, 得到样品间差异距离矩阵, 然后用差异距离矩阵进行NMDS分析, 得到NMDS图, 如图 3所示, 图中两点之间的距离越近说明两者之间的微生物群落差异性越小, 即二者的相似性越大。NMDS图显示:群落差异较小的组别为N-A (-1.04) 和X-A (-1.05), 其次为N-B(0.97) 和X-B (0.90)、N-E(-0.80) 和N-F (-0.76)、N-D(-0.24) 和N-G(-0.06), 群落差异较大的组别为X-C(0.39) 和N-C(0.27)。

|
| 图 3 各样品在对群落结构影响最大两因素下的NMDS图 Fig. 3 The NMDS diagram for each sample in the two most important factors that affect the community structure |
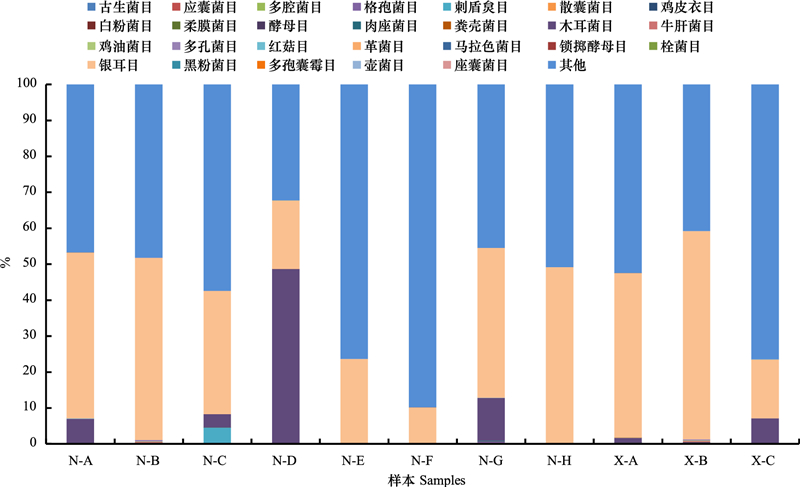
在属水平上对11个样本OTU分类进行聚类, 如图 4所示。由图可知, N-B和X-B聚为一类, 说明不同环境下短额负蝗肠道共生真菌在属水平上群落相似性较高;而N-D和N-F聚为另一类, 表明中华雏蝗和峨眉腹露蝗两物种的肠道共生真菌群落相似性较高。从属的丰富度分布来看, 布勒掷孢酵母属(Bullera)、铁艾酵母属(Tilletiopsis)、油脂酵母属(Lipomyces)、内疣衣属(Ochrolechia)、外瓶霉属(Exophiala)在11个样本间所占比例较高, 相对丰度在2—3之间, 为优势菌属;而青霉菌属(Penicilliumo)和镰刀菌属(Fusarium)这两个属是劣势菌属。从青霉菌属中可以筛选到对昆虫有明显杀虫活性的物质, 如桔青霉素、红青霉素和青霉酸等;从镰刀菌属中也可筛选到对昆虫有明显杀虫活性的物质, 如T-2霉素、玉米赤霉烯酮和单端孢素等。但是这两种劣势菌属在用传统培养法研究结果中却是优势菌属[23], 其主要原因可能是蝗虫肠道中的真菌很多不能以常规培养方法所培养, 或者因为丰度太低或生长太慢没有被分离纯化, 从而使得培养法所获得的种类远低于高通量方法获得的种类。
|
| 图 4 各样品肠道真菌在属水平上的热图 Fig. 4 The heatmap diagram for Fungi at the Genera level |
目前, 对昆虫肠道微生物区系的研究大多是针对细菌多样性研究, 而对肠道真菌多样性的研究报道还比较少[24]。本文以复合条形码方法研究了蝗虫成虫肠道真菌多样性, 从以下几方面来进行结果分析与讨论。
(1) 肠道微生物与蝗虫生活环境的关系
从类群组成和群落结构来看, 校园中的疣蝗、短额负蝗和中华剑角蝗要比分布在南五台山的相应蝗虫的真菌类群多些;且斑翅蝗科的真菌类群要比锥头蝗科及剑角蝗科类群多, 从总层次上也可以得出相似的结论。这表明环境的变化会对同种蝗虫的真菌多样性造成一定影响。有研究表示, 根据栖息地多样性与昆虫的肠道微生物以及昆虫本身的多样性呈正相关。这说明昆虫肠道内的微生物多样性以及它们的功能与昆虫所生活的栖息地有密切关系, 这些结果对如何减少栖息地生物多样性可能会影响生活的物种可以发挥的生态功能具有重要意义[25]。
(2) 肠道微生物与蝗虫食性的关系
α多样性是在一个特定区域或生态系统内的多样性, 常用微生物的群落丰富度(物种数)和多样性(均匀度)来度量, 也就是估计所鉴定的微生物类群中物种的数量以及各样本的均匀度。在α多样性分析中, 两地的疣蝗的肠道微生物群落丰富度和多样性最高, 秦岭小蹦蝗和峨眉腹露蝗的肠道微生物群落丰富度和多样性最低, 这可能是由于它们的食性差异造成的。研究表明, 食性对昆虫肠道微生物的影响是复杂的[26], 每个蝗虫都有属于自身的摄食特性, 不同种蝗虫就会由于食性的不同而导致肠道真菌群落丰富度和多样性有所差异。
(3) 肠道微生物组成与分类系统的关系
β多样性是不同生态系统之间的多样性比较, 是物种组成在不同环境或群落间的变化, 它是生物多样性的重要组成部分, 与许多生态学和进化生物学问题密切相关, 常用相似性来度量。在此, 主要是用来说明不同科和不同取样地的蝗虫肠道微生物群落组成的差异, 群落差异越小即越相似, 说明β多样性越低;反之, 差异越大即越不相似, 说明β多样性越高。从各样本之间的NMDS图来看, 疣蝗和短额负蝗两个物种在不同环境下, 其肠道内共生真菌群落结构组成相似, 不以取样地的改变而变化。中华雏蝗和素色异爪蝗、秦岭小蹦蝗和峨眉腹露蝗的真菌群落结构组成也很相似, 但是中华剑角蝗却因为环境的不同两者之间的真菌群落组成相似性较低, 且中华剑角蝗的真菌群落组成与其他物种的相似性也较低。由此可见, 同科的蝗虫肠道真菌群落结构相似, 不同科的则不相似, 中华剑角蝗除外。对于以上这个结论, 同科的蝗虫摄食习惯可能相近, 而不同科的蝗虫摄食习惯则不同, 因此, 会造成同科的蝗虫肠道真菌群落组成相似, 不同科的则不相似。
DNA复合条形码技术扩大了微生物多样性的研究范围, 为水体和土壤环境中的微生物多样性研究打开了一扇大门, 尤其可以更加快速高效的用于研究动物肠道微生物多样性。如Christian Milani等[27]通过扩增16SrDNA序列用Ion Torrent PGM技术研究人类肠道微生物多样性, 结果显示通过设计PCR引物组可以很好地检测双歧杆菌以及人类肠道微生物群落的其他菌群, 并且表明复条形码技术是一个快速、准确、便宜的方法, 适合于调查人类肠道微生物群落组成;Shelomi等[28]第一次将高通量测序技术和条形码相结合用于研究两类竹节虫的脂肪体、唾液腺、前肠和后中肠的微生物多样性, 结果显示大多数细菌属于螺旋体属(厚壁菌门)的菌株, 并且主要在孤雌生殖物种竹节虫的后中肠, 在R. artemis中肠段和Peruphasma schultei (Pseudophasmatidae)之间没有显著性差异;Zhu等[29]通过对大熊猫新鲜粪便的16S rRNA基因序列分析, 发现梭菌属(Clostridium)的13个OTU中有7个是大熊猫肠道特有的菌群, 宏基因组分析结果进一步证实了梭菌能降解纤维素与半纤维素。这些研究都表明DNA复合条形码技术在肠道微生物鉴定的可行性和有效性, 期望此方法以后更广泛的应用到蝗虫肠道微生物的研究当中, 有助于我们了解蝗虫肠道内真菌群落的差异、共生真菌的功能作用和对蝗虫的生长发育和代谢造成的影响, 找到蝗虫生物防治的新方法, 达到预防和控制的目的。
5 结论(1) 从类群组成可知, ITS共鉴定了5门16纲29目40属2786 OTU真菌。
(2) 从群落组成分析可知, 在南五台和校园两个地区银耳目和木耳菌目属于侵入蝗虫体内的两种主要虫生真菌。
(3)α多样性分析结果可知斑翅蝗科的真菌群落丰富度和多样性最高, 斑腿蝗科的真菌群落丰富度和多样性最低。
(4)β多样性分析结果可知同科的蝗虫肠道真菌群落结构相似, 不同科的则不相似, 中华剑角蝗除外。
(5) 聚类分析结果可知两地的短额负蝗聚集到一起, 网翅蝗科的中华雏蝗和斑腿蝗科的峨眉腹露蝗聚集到了一起, 表明它们两两之间的真菌群落相似性较高。在此分析中发现布勒掷孢酵母属(Bullera)、内疣衣属(Ochrolechia)和外瓶霉属(Exophiala)3个属是优势菌属;而青霉菌属(Penicilliumo)和镰刀菌属(Fusarium)这2个属是劣势菌属。
| [1] | 罗亚皇, 刘杰, 高连明, 李德珠. DNA条形码在生态学研究中的应用与展望. 植物分类与资源学报, 2013, 35(6): 761–768. |
| [2] | Schmidt P A, Bálint M, Greshake B, Bandow C, Römbke J, Schmitt I. Illumina metabarcoding of a soil fungal community. Soil Biology and Biochemistry, 2013, 65: 128–132. DOI:10.1016/j.soilbio.2013.05.014 |
| [3] | Siddique A B, Unterseher M. A cost-effective and efficient strategy for Illumina sequencing of fungal communities: a case study of beech endophytes identified elevation as main explanatory factor for diversity and community composition. Fungal Ecology, 2016, 20: 175–185. DOI:10.1016/j.funeco.2015.12.009 |
| [4] | 燕勇, 李卫平, 高雯洁, 沈志英, 王恒辉, 陈黎霞. rDNA-ITS序列分析在真菌鉴定中的应用. 中国卫生检验杂志, 2008, 18(10): 1958–1961. DOI:10.3969/j.issn.1004-8685.2008.10.007 |
| [5] | 隋心, 冯富娟, 娄鑫, 韩士杰. 土壤微生物总DNA的提取以及土壤真菌ITS-PCR体系的建立. 中国酿造, 2011, 30(9): 166–169. |
| [6] | Hajibabaei M, Shokralla S, Zhou X, Singer G A C, Baird D J. Environmental barcoding: a next-generation sequencing approach for biomonitoring applications using river benthos. PLoS One, 2011, 6(4): e17497. DOI:10.1371/journal.pone.0017497 |
| [7] | 唐敏, 伊廷双, 王欣, 谭美华, 周欣. Metabarcoding技术在植物鉴定和多样性研究中的应用. 植物分类与资源学报, 2013, 35(6): 769–773. |
| [8] | Soininen E M, Zinger L, Gielly L, Bellemain E, Bråthen K A, Brochmann C, Epp L S, Gussarova G, Hassel K, Henden J A, Killengreen S T, Rämä T, Stenøien H K, Yoccoz N G, Ims R A. Shedding new light on the diet of Norwegian lemmings: DNA metabarcoding of stomach content. Polar Biology, 2013, 36(7): 1069–1076. DOI:10.1007/s00300-013-1328-2 |
| [9] | 孟涛, 任炳忠. 蝗虫资源开发与利用的研究进展. 北华大学学报:自然科学版, 2002, 3(6): 485–490. |
| [10] | 黄长礼. 茶尺蠖取食不同品种茶树的生理效应及其肠道真菌的研究[D]. 福州: 福建师范大学, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10394-1015719796.htm |
| [11] | 赵凯, 常志威, 张小燕, 郝妍, 吴桐, 平文祥, 周东坡. 白蚁肠道共生微生物多样性及其防治方法研究现状. 应用与环境生物学报, 2012, 18(2): 331–337. |
| [12] | Rojas-jiménez K, Hernández M. Isolation of fungi and bacteria associated with the guts of tropical wood-feeding coleoptera and determination of their lignocellulolytic activities. International Journal of Microbiology, 2015, 2015: 285018. |
| [13] | 金晶, 彭颖, 李晓波. 快速提取肠道微生物基因组DNA的方法. 现代生物医学进展, 2007, 7(1): 100–103. |
| [14] | White T J, Bruns T D, Lee S B, Taylor J W. Amplification and direct sequencing of fungal ribosomal RNA Genes for phylogenetics//Innis M A, Gelfand D H, Sninsky J J, White T J, eds. PCR Protocols: A Guide To Methods and Applications. San Diego: Academic Press, 1990: 315-322. |
| [15] | Schloss P D, Westcott S L, Ryabin T, Hall J R, Hartmann M, Hollister E B, Lesniewski R A, Oakley B B, Parks D H, Robinson C J, Sahl J W, Stress B, Thallinger G G, Van Horn D J, Weber C F. Introducing mothur: open-source, platform-independent, community-supported software for describing and comparing microbial communities. Applied and Environmental Microbiology, 2009, 75(23): 7537–7541. DOI:10.1128/AEM.01541-09 |
| [16] | Caporaso J G, Kuczynski J, Stombaugh J, Bittinger K, Bushman F D, Costello E K, Fierer N, Peña A G, Goodrich J K, Gordon J I, Huttley G A, Kelley S T, Knights D, Koenig J E, Ley R E, Lozupone C A, McDonald D, Muegge B D, Pirrung M, Reeder J, Sevinsky J R, Turnbaugh P J, Walters W A, Widmann J, Yatsunenko T, Zaneveld J, Knight R. QIIME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335–336. DOI:10.1038/nmeth.f.303 |
| [17] | Edgar R C. Search and clustering orders of magnitude faster than BLAST. Bioinformatics, 2010, 26(19): 2460–2461. DOI:10.1093/bioinformatics/btq461 |
| [18] | Lozupone C, Knight R. Unifrac: a new phylogenetic method for comparing microbial communities. Applied and Environmental Microbiology, 2005, 71(12): 8228–8235. DOI:10.1128/AEM.71.12.8228-8235.2005 |
| [19] | Lozupone C, Hamady M, Knight R. UniFrac-an online tool for comparing microbial community diversity in a phylogenetic context. BMC Bioinformatics, 2006, 7: 371. DOI:10.1186/1471-2105-7-371 |
| [20] | Škuta C, Bartněk P, Svozil D. InCHlib-interactive cluster heatmap for web applications. Journal of Cheminformatics, 2014, 6: 44. DOI:10.1186/s13321-014-0044-4 |
| [21] | Deng W K, Wang Y B, Liu Z X, Cheng H, Xue Y. HemI: a toolkit for illustrating heatmaps. PLoS One, 2014, 9(11): e111988. DOI:10.1371/journal.pone.0111988 |
| [22] | Lin Y T, Whitman W B, Coleman D C, Chih-Yu C. Molecular characterization of soil bacterial community in a perhumid, low mountain forest. Microbes and Environments, 2011, 26(4): 325–331. DOI:10.1264/jsme2.ME11114 |
| [23] | 陈君芝, 涂璇, 程凡, 陈剑锋, 邓张双. 中华剑角蝗肠道共生真菌的分离鉴定及抑菌活性筛选. 三峡大学学报自然科学版, 2014, 36(3): 108–112. |
| [24] | 俞和韦, 王中康, 刘莉, 夏玉先, 殷幼平, 袁青, 曹月青, 彭国雄. 贡嘎蝠蛾幼虫肠道真菌多样性分析. 微生物学报, 2008, 48(4): 439–445. |
| [25] | Schmid R B, Lehman R M, Brözel V S, Lundgren J G. Gut Bacterial Symbiont Diversity within Beneficial Insects Linked to Reductions in Local Biodiversity. Annals of the Entomological Society of America, 2015, 108(6): 993–999. DOI:10.1093/aesa/sav081 |
| [26] | 相辉, 黄勇平. 肠道微生物与昆虫的共生关系. 应用昆虫学报, 2008, 45(5): 687–693. DOI:10.7679/j.issn.2095-1353.2008.149 |
| [27] | Milani C, Hevia A, Foroni E, Duranti S, Turroni F, Lugli G A, Sanchez B, Martín R, Gueimonde M, Van Sinderen D, Margolles A, Ventura M. Assessing the fecal microbiota: an optimized ion torrent 16S rRNA gene-based analysis protocol. PLoS One, 2013, 8(7): e68739. DOI:10.1371/journal.pone.0068739 |
| [28] | Shelomi M, Lo W S, Kimsey L S, Kuo C H. Analysis of the gut microbiota of walking sticks (Phasmatodea). BMC Research Notes, 2013, 6: 368. DOI:10.1186/1756-0500-6-368 |
| [29] | Zhu L F, Wu Q, Dai J Y, Zhang S N, Wei F W. Evidence of cellulose metabolism by the giant panda gut microbiome. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(43): 17714–17719. DOI:10.1073/pnas.1017956108 |
 2017, Vol. 37
2017, Vol. 37