文章信息
- 王蓥燕, 卢圣鄂, 陈小敏, 李跃飞, 辜运富
- WANG Yingyan, LU Sheng'e, CHEN Xiaomin, LI Yuefei, GU Yunfu.
- 若尔盖高原湿地泥炭沼泽土亚硝酸盐还原酶(nirK)反硝化细菌群落结构分析
- Analyzing the nitrate reductase gene (nirK) community in the peat soil of the Zoige Wetland of the Tibetan Plateau
- 生态学报. 2017, 37(19): 6607-6615
- Acta Ecologica Sinica. 2017, 37(19): 6607-6615
- http://dx.doi.org/10.5846/stxb201607151442
-
文章历史
- 收稿日期: 2016-07-15
- 网络出版日期: 2017-05-27
反硝化作用主要是由反硝化细菌介导在厌氧条件下将氮氧化合物(NO3-和NO2-)作为离子受体转化为气态氮(NO、N2、N2O)的生物异化过程[1]。此过程的中间产物NO和N2O导致温室效应和臭氧层空洞[1]。反硝化细菌种群丰富, 仅凭16S rRNA基因难以真实反映环境中反硝化细菌的系统发育特征即脱氮显型不能通过系统发育进行推断, 需要利用编码反硝化酶的功能基因作为分子标记来进行反硝化菌的研究[2-3]。研究表明, 反硝化反应涉及4种反硝化关键酶, 分别是硝酸盐还原酶, 亚硝酸盐还原酶和异化亚硝酸盐还原酶以及氧化亚氮还原酶。其中, 由亚硝酸盐还原酶调控的将亚硝酸盐还原成NO的反应是区分硝酸盐呼吸菌和反硝化菌的关键步骤[4]。亚硝酸盐还原酶包括2种不同结构形态, 一种由含有铜基(Cu-nir)的nirK基因编码, 另一种由含有亚铁血红素cdl(cd1-nir)的nirS基因编码[2]。其中nirK 基因存在于许多亲缘关系较远的菌株中;而nirS 基因反硝化细菌以假单胞菌占优势[4], 可见, 将nirK 基因作为分子标记可以更全面真实的反应环境中的反硝化菌群结构。近年来, nirK基因作为分子标记被广泛用于不同生境反硝化细菌群落结构及反硝化活性的研究, 如:森林土壤、草甸土、海洋底泥以及农业土壤等, 且生境不同对nirK反硝化菌群起决定作用的环境因子也不同[5-9]。故利用nirK基因作为分子标记研究若尔盖高原湿地的反硝化菌群结构和多样性, 对于深入认识若尔盖高原湿地反硝化作用的微生物调控机制具有重要意义。
湿地, 被称作为“地球之肾”, 在调控全球元素的生物地球化学循环中起着重要作用。若尔盖高原湿地位于青藏高原东北隅, 是世界上为数不多的低纬度永久冻土湿地, 具有高海拔、高紫外辐射、高有机质含量等特点[10]。同时, 若尔盖高原湿地也是我国乃至全球面积最大的高原泥炭沼泽群[11], 是中国生物多样性研究关键地区, 也是全球气候变化的热点地区[12]。目前, 研究人员针对该区域的碳循环特征[13-14]及相关微生物调控机制[15]进行了较系统地研究, 而对该区域温室气体N2O排放的微生物调控机制研究却鲜见报道。
本文结合限制性酶切片段长度多态性(RFLP)技术、克隆文库及测序等手段对若尔盖高原湿地泥炭沼泽土中nirK反硝化细菌的群落结构和多样性进行分析, 并通过冗余分析(RDA)来探索影响nirK反硝化细菌的关键环境因子。以期揭示若尔盖高原湿地生态系统反硝化活性与反硝化菌群间的内在联系, 为深入认识该生境的反硝化作用微生物调控机制提供基础理论依据。
1 材料与方法 1.1 研究去概况若尔盖高原湿地位于青海-西藏高原东部边缘的四川、甘肃和青海省的三省交界地区(32°20′—34°00′N, 101°30′—103°30′E;面积3.94×104 hm2, 平均海拔高度3500 m)。在青藏高原影响下该地区特殊的大陆性高原气候环境, 四季不分, 长冬无夏, 降水多, 湿度大, 霜冻期极长。年平均温度为1.1 ℃ (年最高温度为10.9 ℃, 年最低温度-10.3 ℃), 年平均降水量为560—860 mm[16]。沼泽土全年被水和植被覆盖, 主要植被为水生植物和莎草科的水生-中生植物(木里苔草、毛果苔草和乌拉苔草), 土壤母质为均质粉砂和粘土[10]。相关采样点信息见表 1。
| 样品编号 Sample code |
采样点 Sampling site |
经度 Longitude |
纬度 latitude |
海拔 Altitude/m |
土壤温度 Soil temperature/℃ |
| 1 | 麦溪 | 102°49′ 02.8″ | 33°54′57.0″ | 3487 | -5 |
| 2 | 阿西 | 102°48′ 48.2″ | 33°54′58.2″ | 3452 | -6 |
| 3 | 分区 | 102°48′ 59.4″ | 33°55′21.2″ | 3462 | -5 |
2012年8月在查阅有关资料的基础上, 选取阿西、麦溪和分区3个面积最大的典型牧区为研究区域, 采集这3个区域被乌拉苔草覆盖的泥炭沼泽土为研究样品, 按照“梅花型”原则进行采样, 样地面积约0.15 hm2, 布设3个采样点, 用土钻取10—20 cm的土样, 按四分法进行混匀, 分别取1 kg混后土样, 得到三等份样品, 用无菌PET树脂袋封装, 放于冰盒中带回实验室。土样于-20 ℃冰箱保存以备后续分析。
1.3 土壤理化性质与反硝化活性土壤含水量(WC)在105 ℃烘干48 h后检测[17]。有效氮(AN)通过碱解扩散法进行分析[18]。土壤有机质(SOM)、pH值、总氮(TN)、总磷(TP)、有效磷(AP)、总钾(TK)、有效钾(AK)采用Liu等人[19]的方法进行分析。土壤理化性质见表 2。
| 样品编号 Sample code |
水含量 WC/% |
pH | 土壤有机碳 SOC/(g/kg) |
总氮 TN/(g/kg) |
总磷 TP/(g/kg) |
总钾 TK/(g/kg) |
有效氮 AN/(mg/kg) |
有效磷 AP/(mg/kg) |
有效钾 AK/(mg/kg) |
| 1 | 5.12±0.09b | 7.86±0.03a | 281.25±1.39b | 5.29±0.05b | 1.43±0.14a | 7.44±0.03a | 1025.88±1.95b | 26.01±0.26a | 99.83±0.40a |
| 2 | 6.28±0.10a | 7.61±0.02a | 303.09±0.96a | 5.91±0.07a | 1.41±0.17a | 6.79±0.08b | 1079.43±3.03a | 22.63±0.11c | 96.67±0.10b |
| 3 | 4.88±0.04b | 6.85±0.04b | 239.14±0.04c | 4.55±0.06c | 1.32±0.17b | 7.30±0.06a | 993.00±3.05c | 24.72±0.11b | 98.33±0.22a |
| WC:Water content;SOC:Soil organic carbon;TN:Total nitrogen;TP:Total phosphorus;TK:Total potassium;AN:Available nitrogen;AP:Available phosphorus;AK:Available potassium;结果:平均值±标准误;每栏数据在P < 0.05水平下的显著差异 | |||||||||
土壤反硝化活性测定采用乙炔抑制法[20]:称取新鲜土壤样品25 g (以干土计)放入250 mL三角瓶, 添加25 mL溶液(包含1 mmol/L葡萄糖和1 mmol/L KNO3)。用惰性气体氦气冲洗三角瓶, 抽取瓶中土样上方10%的气体, 添加抽取气体同体积的无丙酮乙炔气体, 同时以不加乙炔气体的样品为对照。在20 ℃、225 r/min条件下振荡土壤泥浆, 在1 h和5 h用注射器抽取气样, 用气相测谱仪分析气样。
N2O浓度测定:N2O浓度用美国安捷伦公司生产的Agilent-7890A气相色谱仪测定, 检测器为电子捕获器, 分离柱内填充是Porapak Q, 计算机程序控制定量管(1 mL)和通气阀自动进样, 将空气样品中水分在进入检测器前反吹掉, 载气为甲烷-Ar混合气, 流速为60 mL/min。分离柱温度、进样口温度和检测器温度分别为60 ℃、100 ℃和300 ℃。
1.4 土壤总DNA提取利用Fast DNA spin kit for soil试剂盒(MP BIO, Inc., Irvine, CA, USA)根据制造商提供的说明进行提取。洗脱后总DNA体积为50 μL。利用1%的琼脂糖凝胶电泳确认总DNA的条带的单一性, 同时用Nano-200核酸检测仪(AoSheng, Hangzhou, China)检测浓度。
1.5 nirK基因的扩增所用引物为:nirK517F (5′-TTYGTSTAYCACTGCGCVCC-3′)和nirK1055R (5′-GCYTC-GATCAGRTTR TGGTT-3′)[21]。PCR扩增程序采用TOUCHDOWN程序。选用50 μL反应体系, 分别是PCR Mix 25 μL, DNA模板取6 μL, 引物(10 μmol/L)各1 μL, 超纯水补足50 μL。扩增程序:95 ℃预变性5 min, 94 ℃ 30 s, 57— 47℃ 45 s(每个3循环温度降低2 ℃), 72 ℃ 1 min, 15个循环;其它条件不变, 在47 ℃退火温度下继续扩增25个循环, 终延伸72 ℃ 7 min。取产物2.0 μL在含EB的1%琼脂糖凝胶上水平电泳检测, 120 V电泳30 min。用凝胶成像系统(Bio-Rad, USA)拍照检测。PCR产物置于4 ℃冰箱保存。
1.6 克隆与RFLP分析利用PCR Clean-UpTM试剂盒(MO BIO Labs, Solana Beach, CA, USA)对扩增PCR产物进行纯化, 纯化产物通过pGEM-T载体(Promega)进行克隆。根据“蓝白斑”(抑制剂:X-Gal、Ampr和IPTG)对克隆子进行筛选。3个样品平板培养基共挑取270个白色克隆子。利用nirK基因引物(如1.5所示)进行扩增, 获得250个nirK基因阳性克隆子。
将PCR产物用Msp Ⅰ和Ava Ⅱ限制性内切酶(Promega)进行双酶切。酶切体系总体积为10 μL, 包括:2 U的限制性内切酶、8 μL的PCR产物和限制缓冲剂。酶切产物通过琼脂糖凝胶电泳80V 60min进行分离, 所用琼脂糖凝胶浓度为2%。
根据限制性片段多样性条带的类型, 选取15个代表克隆子送上海生工生物有限公司进行序列测定并将获得的基因序列提交NCBI以获取基因登录号。序列登录号为:KX018494-KX018508。
1.7 数据处理基础数据处理利用Excel 2010进行, 单因素方差分析和皮尔逊相关分析利用SPSS 21.0 (SPSS Inc. Chicago)完成。nirK反硝化细菌群落结构多样性通过Shannon多样性指数(H), 丰富度(S)、均匀度(Eh)[22]和覆盖率(C)[23]进行计算。利用MEGA 5.0软件, 通过最大相邻法构建nirK基因序列系统发育树。利用CANOCO5.0软件对土壤理化性质和nirK基因反硝化群落结构进行冗余分析(RDA)。
2 结果与分析 2.1 土壤理化性质与反硝化活性3个土壤样品的理化性质见表 2。其中, 阿西地区样品的水含量、反硝化活性、土壤有机质、总氮和有效氮最高;而麦溪地区样品pH值、总磷、总钾、有效磷和有效钾是最高的。相较于以上两个地区样品, 分区地区样品的理化性质值相对较低。
反硝化活性范围为:33.52—52.77 ng N2O-N g-1 ds h-1, 最高值出现于阿西地区, 最低值位于分区地区(图 1)。皮尔逊相关分析显示:反硝化活性与土壤有机碳、总氮和丰富度呈显著正相关(表 3)。

|
| 图 1 若尔盖高原湿地泥炭沼泽土的反硝化活性 Fig. 1 Potential denitrifying activity of the peat marsh soil in Zoige plateau wetland 图中小写字母表示反硝化活性在P < 0.05水平上的显著差异 |
| 水含量WC | pH | 土壤碳 含量 SOC |
总氮 TN |
总磷 TP |
总钾 TK |
有效氮 AN |
有效磷 AP |
有效钾 AK |
反硝化 活性 PDA |
|
| 反硝化活性(PDA) | 0.889 | 0.796 | 0.998* | 0.998* | 0.835* | -0.665 | 0.969 | -0.519 | -0.426 | 1 |
| Shannon-Wiener 多样性指数(H) |
0.998* | 0.491 | 0.889 | 0.941 | 0.550 | -0.906 | 0.988 | -0.815 | -0.749 | 0.918 |
| 丰富度(S) | 0.916 | 0.756 | 0.992 | 1.000** | 0.799 | -0.711 | 0.982 | -0.572 | -0.481 | 0.998* |
| 均匀度(Eh) | 0.993 | 0.535 | 0.912 | 0.957 | 0.592 | -0.884 | 0.994 | -0.785 | -0.714 | 0.937 |
| WC:Water content;SOC:Soil organic carbon;TN:Total nitrogen;TP:Total phosphorus;TK:Total potassium;AN:Available nitrogen;AP:Available phosphorus;AK:Available potassium;PDA:Potential denitrifying activity;表中数据为:相关系数(r);*P < 0.05;** P < 0.01 | ||||||||||
限制性片段多态性分析显示(表 4), 3个地区共包括15个OTUs, 不同地区所包含的OTU类型存在一定差异。其中阿西地区所包含OTU类型最多, 麦溪次之, 分区最少。其中OTU1、2、5、8、11、12为3个地区共有, OTU13、OTU14为阿西地区特有;OTU7为麦溪地区特有;OTU15为分区地区特有。
| 限制性内切酶 Restriction enzyme |
样品编号 Sample codes |
OTU类型 OTU type |
覆盖率(C) Coverage ratio |
多样性指数(H) Shannon-wiener Diversity index |
丰度(S) Richness |
均匀度(Eh) Evenness |
| Msp Ⅰ | 1 | 12 | 0.9539±0.02a | 2.59±0.02b | 3.12±0.04b | 0.87±0.02a |
| 和Ava Ⅱ | 2 | 13 | 0.9653±0.03a | 2.91±0.04a | 3.38±0.07a | 0.92±0.02a |
| 3 | 10 | 0.9346±0.02b | 2.49±0.06b | 2.81±0.02 c | 0.85±0.02a | |
| 结果为平均值±标准误; 每栏数据在P < 0.05的水平下显著差异 | ||||||
克隆文库的覆盖率用于分析土壤微生物群落的多样性, 文库中的特有OTU类型越多则多样性的覆盖率越低[24]。本文3个地区各自的克隆文库的覆盖率都高于91%(表 4), 充分说明所构建的克隆文库几乎全部覆盖地区里的nirK基因种类。同时, 克隆文库也可以代表若尔盖高原湿地泥炭沼泽土中nirK反硝化群落的特征。
基于RFLP数据计算nirK基因群落的Shannon多样性指数(H)、丰富度(S)以及均匀度(Eh)(表 4)。nirK基因反硝化细菌群落多样性在3个采样点的差异不明显。Shannon-Wiener多样性指数的变化范围为:2.91—2.49, 其中阿西采样点多样性指数、丰富度和均匀度最高, 麦溪次之, 分区最小。通过表 3皮尔逊相关分析显示:香侬-威尔多样性指数与水含量显著相关(P < 0.05)。丰富度与总氮极显著相关(P < 0.05)。而均匀度与各理化性质的相关性不明显。
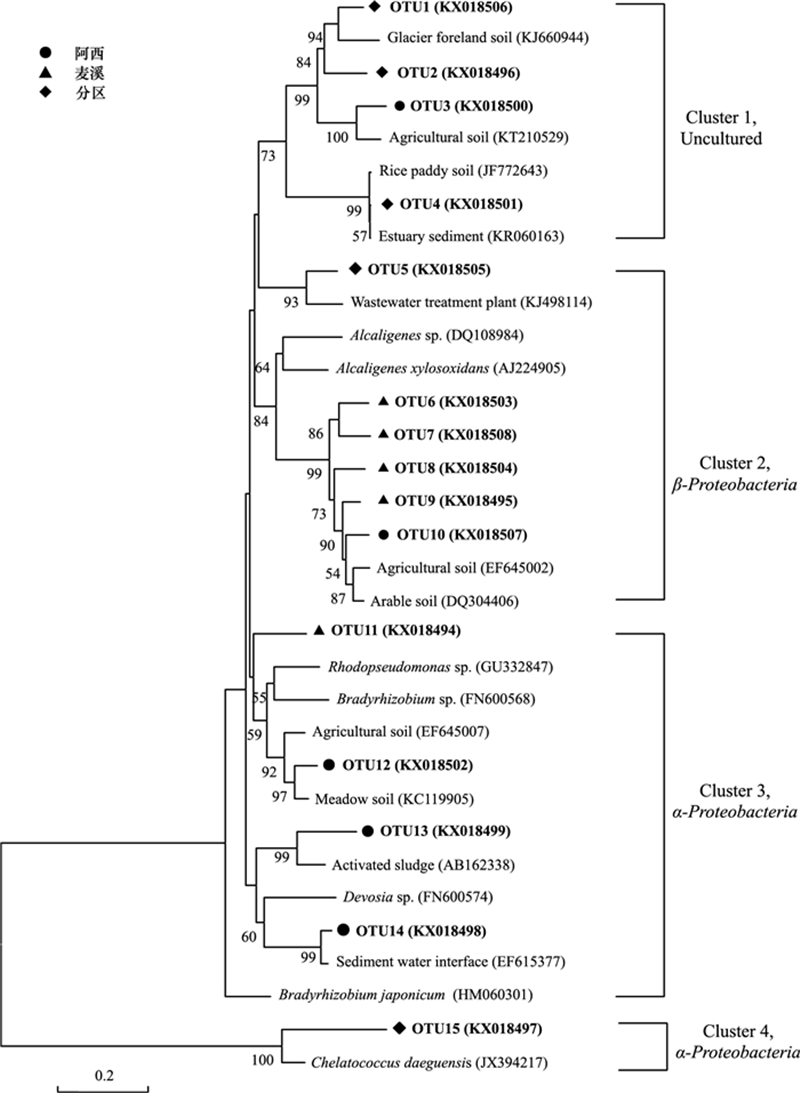
2.3 系统发育地位分析基于RFLP图谱, 同一RFLP类型下选取一个克隆序列作为一个OTU。已测序的15个阳性克隆序列长度均约为530 bp。根据Palmer的方法[25], 将82%作为序列相似性标准, 可以将nirK序列定位到属的水平。系统发育分析显示:15条序列中, 10条与变形门细菌相似(图 2), 包括Alpha-Proteobacteria和Beta-Proteobacteria两个纲。还有5条序列无法确定其分类地位。
|
| 图 2 若尔盖高原湿地泥炭沼泽土nirK反硝化细菌的系统发育分析 Fig. 2 Phylogeny analysis of the nirK denitrifiers of the peat marsh soil in Zoige plateau wetland 在树节上的数字代表 1 000取样的百分比, 仅显示大于50%的情况 |
Alphaproteobacteria纲是若尔盖泥炭沼泽土最优势的nirK反硝化菌群, 占克隆子总数的51.0%, 有5个OTU属于这个类群(Cluster3、Cluster4)。其中, 丰度最高的OTU12, 在250个正确插入片段的克隆子中占有43个, 与GenBank可培养反硝化细菌Rhodopseudomonas sp. 2—8 (GU332847) 在核酸水平上具有85%的序列相似性。此代表序列来自不同的生境, 如沼泽土、农田土壤、酸性土壤、水稻土等。丰度第二的OTU14包括27个克隆子, 占克隆子总数的10.8%, 多来源于海底底泥、农业土壤等。OTU13的源于城市污水处理厂。此外, 属于OTU12、OTU13和OTU14的克隆子主要来自阿西地区, 显示阿西采样点中的优势种群属于Alphaproteobacteria纲。
Betaproteobacteria 纲包含5个OTU, 占克隆子总数的38%。OTU8最丰富, 该OTU占克隆子总数的14.4%, 与可培养反硝化细菌Achromobacter xylosoxidan (AB969826) 具有84%的序列相似性。其相似序列来源于草甸土和农田土壤, 相似度在82%—89%之间。此外, OTU6—9的克隆子大部分来自麦溪地区, 显示麦溪采样点中的优势种群属于Betaproteobacteria纲。还有部分OTU无法确定其分类地位(Cluster 1), 占克隆子总数的11%。分区采样点的克隆序列大多属于此类。以上OTU相似的序列大多来自于冰川, 海滩、高山贫营养湖、农业土壤、河口、内陆湖底泥等, 序列相似度在85%—89%之间。
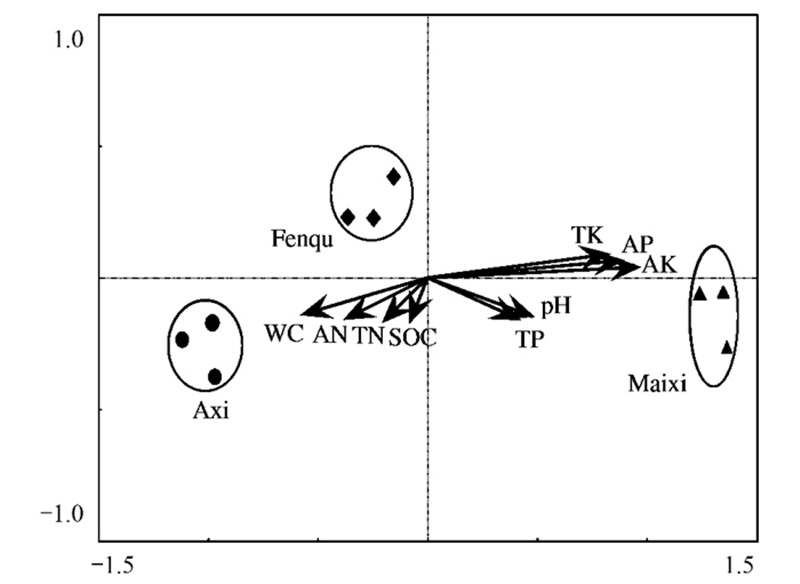
2.4 nirK反硝化细菌群落结构与土壤理化性质的冗余分析若尔盖高原湿地泥炭沼泽土nirK反硝化细菌群落与土壤理化性质之间的RDA相关性见图 3。分析结果显示:第一排序轴的特征值为0.97, 第二排序轴特征值为0.02。由图可知, 第一排序轴与有效磷、有效钾关系最为密切, 均达到极显著水平(P < 0.01)。第二轴与土壤有机碳、总氮、总磷和有效氮较为密切, 均达到显著水平(P < 0.05)。总体而言, 本实验中有效钾、有效磷对nirK基因反硝化群落结构的影响最显著。此外, 由采样点投射到各理化性质上的投影点可知, 阿西地区含高有机质, 麦溪地区含高有效钾、有效磷, 分区地区各理化性质居中。而各采样点的连线段长度表示各采样点nirK反硝化细菌群落的相似程度, 即麦溪地区与其他两个地区的连接距离较长, 则其nirK反硝化菌群有明显的差异。
|
| 图 3 若尔盖高原湿地泥炭沼泽土nirK反硝化细菌群落结构的RDA排序图 Fig. 3 RDA ordination analysis of the nirK denitrifier communities in the peat marsh soil in Zoige plateau wetland WC:水分含量Water content;SOC:土壤有机碳Soil organic carbon;TN:总氮Total nitrogen;TP:总磷Total phosphorus;TK:总钾Total potassium;AN:有效氮Available nitrogen;AP:有效磷Available phosphorus;AK:有效钾Available potassium |
环境因子如土壤含水量, pH和营养成分等是影响反硝化作用和反硝化细菌种群组成和多样性的重要因素, 本文皮尔逊相关分析显示:反硝化活性与土壤有机质、总氮和总磷都呈极显著相关(表 3)。已有研究表明[26], 反硝化活性的高低取决于提供给细菌生长的有机质的质量与数量。在污水灌溉的农田里检测反硝化活性结果显示, 影响反硝化活性的主要环境因子是总氮含量[27]。White[28]等人在研究被水覆盖40a的泥炭沼泽土反硝化活性时发现:总磷含量与反硝化活性呈极显著相关性。上述研究结果与本文结果相似, 显示若尔高高原湿地的反硝化活性受土壤理化性质特别是有机质、氮素和磷素的有效性的明显影响。
系统发育分析显示nirK反硝化菌群主要属于变形门。此外, 若尔盖高原湿地不同地区泥炭沼泽土中的nirK反硝化菌群存在明显差异。其中, 阿西地区分离的克隆子序列主要属于α-变形门(Cluster 3-4), 少部分属于β-变形门。在α-变形门中, 以慢生型大豆根瘤菌属(Bradyrhizobium japonicum)、红假单胞菌属(Rhodopseudomonas)、苍白杆菌属(Ochrobactrum)还有德沃斯氏菌属(Devosia)为主。与本文结果相似, Priemé等[5]研究土耳其沼泽化潮土中亚硝酸盐微生物群落得出慢生型大豆根瘤菌(Bradyrhizobium japonicum, AJ002516) 是该生境中的优势反硝化微生物类群。Saito等研究也发现定向施无机肥的水稻土中nirK反硝化菌群除以上慢生型大豆根瘤菌属外还有红假单胞菌属(Rhodopseudomonas, YP782977) 以及苍白杆菌属(Ochrobactrum, YP00137298)[29]。但德沃斯氏菌属(Devosia)未见相关报道。麦溪地区反硝化菌群主要为β-变形门, 少部分为α-变形门, 且主要为β-变形门的产碱杆菌属(Alcaligenes)。与本文类似, Prieme等人研究土耳其沼泽土壤中的反硝化细菌类群时, 也得出β-变形门细菌相对较少[5]。而分区地区的主要反硝化菌群不能确定其种属地位。结合本文的反硝化活性和系统发育分析结果, 若尔盖高原湿地泥炭沼泽土的反硝化活性可能主要由α-变形门细菌调控。与本文研究相似, Katsuyama等人在研究不同森林土的反硝化活性和调控菌群时亦发现α-变形门的反硝化菌是调控该类生境反硝化活性的主要菌群[30]。综上, 若尔盖高原湿地泥炭沼泽土中存在多样化的反硝化菌群, 原因可能是这些泥炭沼泽土微域生境条件如理化性质、含水量和含氧量及氧化还原电位存在差异而导致[3]。
环境因子是影响微生物群落结构变化的重要因子, 研究土壤微生物群落结构与土壤理化性质间的相互关系对于深入认识土壤微生物在生态系统中扮演的角色和功能具有重要意义[31]。RDA分析显示有效钾和有效磷是影响若尔盖高原湿地泥炭沼泽土nirK反硝化细菌群落结构的主要因子。Zhang等[32]对青藏高原草甸土nirK反硝化细菌群落研究发现, 温度、水含量以及C/N比是影响nirK反硝化细菌群落结构的主要因子。而Xie等[33]研究青藏高原草甸土上放牧对反硝化细菌群落结构的影响发现土壤有机碳和硝态氮是影响nirK反硝化细菌群落结构的重要因子。而与本文结果相似, 如Barta等[34]研究发现森林土壤中有效磷对nirK反硝化细菌群落结构有显著影响(r=0.83, P < 0.001)。Xue等[35]研究表明有效钾是影响有机农业土壤nirK反硝化细菌群落的重要因子。此外, 相关研究也表明:当有效磷和有效钾作为土壤限制营养因子时, 会诱导溶磷溶钾微生物分泌相关酶类释放土壤中的磷、钾元素, 改善土壤微域生境, 进而反过来调控这些微生物的种群和数量[36-37]。综上, 磷、钾元素作为生命活动的重要营养因子, 它们与受微生物调控的氮素生物地球化学循环可能存在非常复杂的作用关系, 这些关系的解释尚须进一步研究证实。
本文分析了若尔盖高原湿地泥炭沼泽土反硝化活性和nirK反硝化细菌群落结构和多样性及影响因子。研究显示nirK基因反硝化菌群主要属于变形门菌, 该区域的反硝化活性主要受α-变形门反硝化菌群影响。土壤有效钾和有效磷是影响nirK基因反硝化菌群落结构的主要环境因子。
| [1] | Conrad R. Soil microorganisms as controllers of atmospheric trace gases (H2, CO, CH4, OCS, N2O and NO). Microbiology Reviews, 1996, 60(4): 609–640. |
| [2] | Zumft W G. Cell biology and molecular basis of denitrification. Microbiology and Molecular Biology Reviews, 1997, 61(4): 533–616. |
| [3] | Wolsing M, Priemé A. Observation of high seasonal variation in community structure of denitrifying bacteria in arable soil receiving artificial fertilizer and cattle manure by determining T-RFLP of nir gene fragments. FEMS Microbiology Ecology, 2004, 48(2): 261–271. DOI:10.1016/j.femsec.2004.02.002 |
| [4] | Braker G, Zhou J Z, Wu L, Devol A H, Tiedje J M. Nitrite reductase genes (nirK and nirS) as functional markers to investigate diversity of denitrifying bacteria in pacific northwest marine sediment communities. Applied and Environmental Microbiology, 2000, 66(5): 2096–2104. DOI:10.1128/AEM.66.5.2096-2104.2000 |
| [5] | Priemé A, Braker G, Tiedje J M. Diversity of nitrite reductase (nirK and nirS) gene fragments in forested upland and wetland soils. Applied and Environmental Microbiology, 2002, 68(4): 1893–1900. DOI:10.1128/AEM.68.4.1893-1900.2002 |
| [6] | Keil D, Meyer A, Berner D, Poll C, Shcützenmeister A, Piepho H P, Vlasenko A, Philippot L, Schloter M, Kandeler E, Marhan S. Influence of land-use intensity on the spatial distribution of N-cycling microorganisms in grassland soils. FEMS Microbiology Ecology, 2011, 77(1): 95–106. DOI:10.1111/fem.2011.77.issue-1 |
| [7] | Scala D J, Kerkhof L J. Nitrous oxide reductase (nosZ) gene-specific PCR primers for detection of denitrifiers and three nosZ genes from marine sediments. FEMS Microbiology Letter, 1998, 162(1): 61–68. DOI:10.1111/fml.1998.162.issue-1 |
| [8] | Yoshida M, Ishii S, Otsuka S, Senoo K. Temporal shifts in diversity and quantity of nirS and nirK in a rice paddy field soil. Soil Biology and Biochemistry, 2009, 41(10): 2044–2051. DOI:10.1016/j.soilbio.2009.07.012 |
| [9] | Robertson G P, Paul E A, Harwood R R. Greenhouse gases in intensive agriculture: contributions of individual gases to the radiative forcing of the atmosphere. Science, 2000, 289(5486): 1922–1925. DOI:10.1126/science.289.5486.1922 |
| [10] | Huo L L, Chen Z K, Zou Y C, Lu X G, Guo J W, Tang X G. Effect of Zoige alpine wetland degradation on the density and fractions of soil organic carbon. Ecological Engineering, 2013, 51: 287–295. DOI:10.1016/j.ecoleng.2012.12.020 |
| [11] | 张晓云, 吕宪国, 顾海军. 若尔盖湿地面临的威胁、保护现状及对策分析. 湿地科学, 2005, 3(4): 292–297. |
| [12] | 罗清, 彭国照. 若尔盖及其邻近地区气候变化对湿地生态环境的影响. 高原山地气象研究, 2008, 28(3): 44–48. |
| [13] | 陈槐, 高永恒, 姚守平, 吴宁, 王艳芬, 罗鹏, 田建卿. 若尔盖高原湿地甲烷排放的时空异质性. 生态学报, 2008, 28(7): 3425–3437. |
| [14] | 王德宣. 若尔盖高原泥炭沼泽二氧化碳、甲烷和氧化亚氮排放通量研究. 湿地科学, 2010, 8(3): 220–224. |
| [15] | Zhang G S, Tian J Q, Jiang N A, Guo X P, Wang Y F, Dong X Z. Methanogen community in Zoige wetland of Tibetan plateau and phenotypic characterization of a dominant uncultured methanogen cluster ZC-I. Environmental Microbiology, 2008, 10(7): 1850–1860. DOI:10.1111/j.1462-2920.2008.01606.x |
| [16] | Zhou W J, Lu X F, Wu Z K, Deng L, Jull A J T, Donahue D, Beck W. Peat record reflecting Holocene climatic change in the Zoigê Plateau and AMS radiocarbon dating. Chinese Science Bulletin, 2002, 47(1): 66–70. DOI:10.1360/02tb9013 |
| [17] | Hartemink A E, Janssen B H, Buresh R J, Jama B. Soil nitrate and water dynamics in Sesbania fallows, weed fallows, and maize. Soil Science Society of America Journal, 1996, 60(2): 568–574. DOI:10.2136/sssaj1996.03615995006000020033x |
| [18] | 鲁如坤. 土壤农业化学分析方法. 北京: 中国农业科技出版社, 2000: 125–510. |
| [19] | Liu H, Dasgupta P K. Analytical chemistry in a drop. Solvent extraction in a microdrop. Analytical Chemistry, 1996, 68(11): 1817–1821. |
| [20] | Dambreville C, Hallet S, Nguyen C, Morvan T, Germon J C, Philippot L. Structure and activity of the denitrifying community in a maize-cropped field fertilized with composted pig manure or ammonium nitrate. FEMS Microbiology Ecology, 2006, 56(1): 119–131. DOI:10.1111/fem.2006.56.issue-1 |
| [21] | Chen Z, Luo X Q, Hu R G, Wu M N, Wu J S, Wei W X. Impact of long-term fertilization on the composition of denitrifier communities based on nitrite reductase analyses in a paddy soil. Microbial Ecology, 2010, 60(4): 850–861. DOI:10.1007/s00248-010-9700-z |
| [22] | Hill T C J, Walsh K A, Harris J A, Moffett B F. Using ecological diversity measures with bacterial communities. FEMS Microbiology Ecology, 2003, 43(1): 1–11. DOI:10.1111/fem.2003.43.issue-1 |
| [23] | Good I J. The population frequencies of species and the estimation of population parameters. Biometrika, 1953, 40(3/4): 237–264. DOI:10.2307/2333344 |
| [24] | Qiu X Y, Wu L Y, Huang S H, McDonel P E, Palumbo A V, Tiedje J M, Zhou J Z. Evaluation of PCR-generated chimeras, mutations, and heteroduplexes with 16S rRNA gene-based cloning. Applied and Environmental Microbiology, 2001, 67(2): 880–887. DOI:10.1128/AEM.67.2.880-887.2001 |
| [25] | Palmer K, Drake H L, Horn M A. Genome-derived criteria for assigning environmental narG and nosZ sequences to operational taxonomic units of nitrate reducers. Applied and Environmental Microbiology, 2009, 75(15): 5170–5174. DOI:10.1128/AEM.00254-09 |
| [26] | Jahangir M M R, Khalil M I, Johnston P, Cardenas L M, Hatch D J, Butler M, Barrett M, O'flaherty V, Richards K G. Denitrification potential in subsoils: a mechanism to reduce nitrate leaching to groundwater. Agriculture, Ecosystems & Environment, 2012, 147: 13–23. |
| [27] | Guo G X, Deng H, Qiao M, Yao H Y, Zhu Y G. Effect of long-term wastewater irrigation on potential denitrification and denitrifying communities in soils at the watershed scale. Environmental Science and Technology, 2013, 47(7): 3105–3113. |
| [28] | White J R, Reddy K R. Nitrification and denitrification rates of Everglades wetland soils along a phosphorus-impacted gradient. Journal of Environmental Quality, 2003, 32(6): 2436–2443. DOI:10.2134/jeq2003.2436 |
| [29] | Saito T, Ishii S, Otsuka S, Nishiyama M, Senoo K. Identification of novel Betaproteobacteria in a succinate-assimilating population in denitrifying rice paddy soil by using stable isotope probing. Microbes and Environments, 2008, 23(3): 192–200. DOI:10.1264/jsme2.23.192 |
| [30] | Katsuyama C, Kondo N, Suwa Y, Yamagishi T, Itoh M, Ohte N, Kimura H, Nagaosa K, Kato K. Denitrification activity and relevant bacteria revealed by nitrite reductase gene fragments in soil of temperate mixed forest. Microbes and Environments, 2008, 23(4): 337–345. DOI:10.1264/jsme2.ME08541 |
| [31] | Ruiz-Rueda R O, Trias M R, Garcia-Gil L J, Bañeras L. Diversity of the nitrite reductase gene nirS in the sediment of a free-water surface constructed wetland. International Microbiology, 2007, 10(4): 253–260. |
| [32] | Zhang Y G, Li D Q, Wang H M, Xiao Q M, Liu X D. The diversity of denitrifying bacteria in the alpine meadow soil of Sanjiangyuan natural reserve in Tibet Plateau. Chinese Science Bulletin, 2006, 51(10): 1245–1254. DOI:10.1007/s11434-006-1245-7 |
| [33] | Xie Z, Le Roux X, Wang C P, Gu Z K, An M, Nan H Y, Chen B Z, Li F, Liu Y J, Du G Z, Feng H Y, Ma X J. Identifying response groups of soil nitrifiers and denitrifiers to grazing and associated soil environmental drivers in Tibetan alpine meadows. Soil Biology and Biochemistry, 2014, 77: 89–99. DOI:10.1016/j.soilbio.2014.06.024 |
| [34] | Bárta J, Melichová T, Vaněk D, Picek T, Šantrůč ková H. Effect of pH and dissolved organic matter on the abundance of nirK and nirS denitrifiers in spruce forest soil. Biogeochemistry, 2010, 101(1/3): 123–132. |
| [35] | Xue K, Wu L Y, Deng Y, He Z L, Van Nostrand J, Robertson P G, Schmidt T M, Zhou J Z. Functional gene differences in soil microbial communities from conventional, low-input, and organic farmlands. Applied and Environmental Microbiology, 2013, 79(4): 1284–1292. DOI:10.1128/AEM.03393-12 |
| [36] | 王斐, 李菊梅, 戴建军, 马义兵. 有机结合态磷肥对土壤微生物及磷酸酶的影响. 化肥工业, 2013, 40(3): 3–9. |
| [37] | McGill W B, Cole C V. Comparative aspects of cycling of organic C, N, S and P through soil organic matter. Geoderma, 1981, 26(4): 267–286. DOI:10.1016/0016-7061(81)90024-0 |
 2017, Vol. 37
2017, Vol. 37