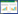文章信息
- 李鹏, 李永春, 史加亮, 郑宪清, 武国干, 蒋玮, 赵凯, 明凤, 潘爱虎, 吕卫光, 唐雪明
- LI Peng, LI Yongchun, SHI Jialiang, ZHENG Xianqing, WU Guogan, JIANG Wei, ZHAO Kai, MING Feng, PAN Aihu, LÜ Weiguang, TANG Xueming.
- 水稻秸秆还田时间对土壤真菌群落结构的影响
- Rice straw return of different decomposition days altered soil fungal community structure
- 生态学报. 2017, 37(13): 4309-4317
- Acta Ecologica Sinica. 2017, 37(13): 4309-4317
- http://dx.doi.org/10.5846/stxb201603110431
-
文章历史
- 收稿日期: 2016-03-11
- 网络出版日期: 2017-02-23
2. 复旦大学生命科学学院, 上海 200438;
3. 浙江农林大学环境与资源学院, 临安 311300;
4. 德州市农业科学研究院, 德州 253000;
5. 上海市农业科学院生态环境保护研究所, 上海 201403
2. School of Life Science of Fudan University, Shanghai 200438, China;
3. School of Environmental and Resources of Zhejiang Agriculture and Forestry University, Lin'an 311300, China;
4. Dezhou Academy of Agricultural Sciences, Dezhou 253000, China;
5. Institute of Eco-Environment and Plant Protection of Shanghai Academy of Agricultural Sciences, Shanghai 201403, China
土壤微生物是土壤生物区系中重要的功能组分和土壤生物群落的重要类群, 对土壤的形成发育、物质循环和肥力演变等具有重大影响[1], 是维持土壤资源持续性和生态系统功能的关键因素[2]。土壤微生物参与土壤有机质的分解及腐殖质的形成等过程[3], 是土壤养分循环的主要驱动者, 因而土壤微生物的生物量、活性和群落结构是土壤质量的重要方面[4]。土壤微生物对土壤有机质的变化十分敏感, 能对土壤生态机制变化和环境胁迫做出反应, 土壤微生物群落结构的组成和变化情况, 是评价土壤健康程度或者土壤质量的早期重要指标[5]。在农田生态系统中还田秸秆是土壤有机质的重要来源之一[6], 作物秸秆富含碳、氮、磷、钾等营养元素, 是宝贵的自然资源。据估算世界每年产生约34亿t作物秸秆, 因各种因素废弃或者焚烧的秸秆量占总秸秆量的60%以上[7], 造成了养分资源的巨大浪费, 并严重污染了环境。我国秸秆总产量居世界首位, 其总产量已达6亿t, 如何合理利用秸秆资源, 保护环境, 是关系到农业可持续发展的一个关键问题[8]。秸秆还田可以改善土壤结构[9], 提高土壤有机碳含量[10]和土壤微生物活性[11], 进而提高作物产量。
作物秸秆主要是由木质素、纤维素、半纤维素等聚结的致密的碳水化合物组成[12], 微生物作为产生纤维素酶类、木质素酶类的主要来源, 在秸秆物质的降解过程中发挥了巨大的作用, 添加秸秆为土壤微生物提供了丰富的碳源和氮源, 促进了微生物的生长繁殖, 提高了土壤微生物活性, 特别是增加了真菌和细菌比例[13], 引起土壤微生物群落结构的变化[14-15]。目前相关研究表明, 秸秆还田会导致土壤细菌结构以及多样性发生显著变化[16-18]。土壤真菌由于具有较高的胞外酶活性且其酶种类比较全面, 使其有很强的纤维素降解能力, 在秸秆还田的过程中发挥重要作用, Bardgett等[19]研究表明, 在秸秆降解的途径中, 真菌的分解作用占据优势。Marschner等[20]采用磷酸脂肪酸(PLFA)和尼龙网袋法对小麦秸秆降解早期(30 d内)的土壤进行分析, 结果表明在秸秆降解1—2周时, 土壤真菌的群落结构发生了显著得变化。由于秸秆在土壤中的自然降解是个长期过程, 但目前自然条件下秸秆还田对土壤真菌群体数量和群落结构的长期影响尚鲜见报道。为此, 本研究通过设置水稻秸秆还田不同时间(90, 180, 270, 360 d), 采用聚合酶链反应(polymerase chain reaction, PCR)-变性梯度凝胶电泳(denaturing gradient gel electrophoresis, DGGE)及荧光定量PCR方法研究真菌群体数量和种群结构的演变趋势, 并利用冗余分析(Redundancy Analysis -RDA)研究土壤真菌的群落结构变化与土壤性质的关联, 为揭示土壤真菌对秸秆还田引起的微生境变化的适应与演变趋势, 维持土壤生态系统功能的稳定提供理论基础。
1 材料与方法 1.1 试验设计与样品采集土壤样品采自上海市崇明区长江农场秸秆还田长期定位试验田(31°30′ N, 121°31′ E)。试验于2013年10月进行秸秆还田试验, 稻秸还田量为6000 kg/hm2(依据当地水稻产量6000 kg/hm2, 并考虑根茎残茬折算), 稻秸秆经切碎后耕埋, 耕深10 cm, 并设置秸秆不还田对照。选定小区面积33 m2, 重复3次。分别在秸秆还田后90、180、270 d和360 d采集秸秆还田和未秸秆还田土壤样品。采用五点取样法采集0—10 cm土壤, 混匀分装, 一部分样品用液氮速冻后在-20℃保存, 供分子生物学研究;另一部分用于土壤基本理化性质分析。土壤理化性质参照文献[21]进行分析, 结果见表 1。
| 处理 Treatment | pH (1:2.5H2O) | 有机碳 Organic C /(g/kg) | 总氮 Total N /(g/kg) | 碱解氮 Alk-hydr N /(mg/kg) | 速效磷 Available P/ (mg/kg) | 速效钾 Available K/ (mg/kg) |
| C90 | 7.31±0.05b | 15.48±1.27c | 1.45±0.04a | 168.53±11.46b | 11.29±1.08b | 121.97±8.84a |
| T90 | 7.28±0.07b | 16.82±0.64c | 1.47±0.05a | 149.77±5.65c | 10.37 ±0.60b | 98.78±5.34b |
| C180 | 7.26±0.12b | 15.82±1.22c | 1.45±0.03a | 187.89±9.60a | 13.69 ±0.62a | 100.76±8.28b |
| T180 | 7.41±0.05b | 17.27±1.21c | 1.40±0.07a | 161.28±13.21bc | 14.76 ±0.49a | 108.26±4.83a |
| C270 | 7.41±0.03b | 15.82±0.51c | 1.43±0.08a | 184.67±11.56ab | 13.74 ±0.53a | 121.58±7.29a |
| T270 | 7.59±0.18ab | 20.40±1.95b | 1.38±0.06a | 177.97±5.80ab | 13.89 ±0.97a | 101.70±7.72b |
| C360 | 7.45±0.06b | 16.40±0.88c | 1.39±0.07a | 170.34±8.56b | 13.89 ±0.76a | 100.57±6.55b |
| T360 | 7.60±0.19a | 25.80±2.78a | 1.35±0.05a | 160.66±10.53bc | 14.23 ±0.89a | 111.79±7.41ab |
| T代表处理(秸秆还田土壤)Treat (Straw decomposition soil);C代表对照(秸秆未还田土壤)Control (No straw decomposition soil); 同列不同小写字母表示差异显著(P<0.05) | ||||||
采用the ISOIL for Beads Beating kit(NipponGeneCo., Ltd., Toyama, Japan)试剂盒提取土壤总DNA, 称取0.5 g于-20 ℃保存的土壤样品按试剂盒说明书进行土壤DNA提取, 用30 μL灭菌水溶解DNA, 用1.0%琼脂糖凝胶电泳检测所提取的DNA片段大小, 并用NanoDrop ND-1000核酸蛋白测定仪(NanoDrop Technologies, Inc.)测定DNA的浓度和质量, DNA样品于-20℃保存。
1.3 土壤真菌群落结构的PCR-DGGE分析应用Veriti梯度PCR仪(Thermo Fisher Scientific Inc)进行真菌18S rRNA基因的PCR扩增, 每个样品3次重复。以提取的总DNA为模板, 引物为NS7-GC和NS8[22]。PCR体系如下:无菌双蒸水31.7 μL, 10×Buffer (-MgCl2) 5.0 μL, dNTPs(各2.5 mmol/L)5.0μL, 25 mmol/L MgCl2 3.0 μL, 引物1(10 μmol/L)和引物2(10 μmol/L)各0.5 μL, 牛血清血蛋白(BSA-10 mg/mL)1.0 μL, 3%去离子甲酰胺1.0 μL , Taq酶(5U/μL-宝生物工程(大连)有限公司)0.3 μL, DNA模板(20ng/μL)2.0μL。PCR反应条件:反应条件:94℃预变性3 min;95℃变性45 s, 50℃退火45 s, 72℃延伸45 s(35个循环); 最后72℃延伸10 min。
使用DCodeTM Universal Mutation Detection System(Bio-Rad Laboratory, Inc.), 在变性剂梯度为30%—60%的聚丙烯酰胺凝胶上进行变性梯度凝胶电泳(DGGE)。使用40%丙烯酰胺/双丙烯酰胺凝胶(37.5:1), 电泳缓冲液为1×TAE。电压80V, 60℃, 电泳13 h, 采用1:10000(v/v)SYBR Green染色30 min, 染色结果用Gel DocTM EQ (Bio-Rad Laboratory,Inc.)凝胶成像系统进行图像分析。
将DGGE图谱中的主要条带进行切胶, DNA回收, 然后再分别以不带GC夹的引物NS7/NS8对回收条带DNA进行PCR扩增, 将扩增产物利用pMD 18-T载体进行克隆测序(生工生物工程(上海)股份有限公司)。登陆NCBI通过Blast软件将11条测序序列与GenBank中的已知序列比对来确定测定序列的微生物种类。利用Sequin软件向Genbank提交11条测序序列, 并获得登录号(KU510560-KU510570)。利用MEGA 5.0软件基于DGGE条带序列, 进行Bootstrap验证系统发育分析, 邻接法(Neighbor-joining, N-J)构建系统发育树。
1.4 土壤真菌群体数量的荧光定量PCR分析应用ABI StepOne Plus扩增仪(Thermo Fisher Scientific Inc)对真菌ITS rRNA基因进行荧光定量PCR扩增, 引物为NSⅠ1和58A2R[23], 每个样品3次重复。荧光定量PCR反应体系10 μL, 引物NSⅠ1和58A2R (10 μmol/L)各0.25 μL, 2×SYBR Premix Ex TaqTM (宝生物工程(大连)有限公司)5 μL, 模板4 μL, 用无菌ddH2O补足至10 μL。采用三步法进行荧光定量PCR:95 ℃预变性3 min;94 ℃, 30 s;53 ℃, 30 s;72 ℃, 45 s;40个循环。
以混合土样总DNA为模板进行真菌ITS rRNA基因PCR扩增。将100 μL PCR产物用1.5%琼脂糖凝胶电泳, 切下含有ITS rRNA基因的条带, 用AxyPrep DNA凝胶回收试剂盒(爱思进生物技术(杭州)有限公司)纯化。使用pMD 18-T载体(宝生物工程(大连)有限公司)连接PCR产物, 连接产物转化到大肠杆菌DH5α感受态细胞, 在氨苄青霉素平板上进行蓝白斑筛选阳性克隆。取部分阳性转化菌液送生工生物工程(上海)股份有限公司进行测序, 作为氨氧化古菌荧光定量PCR分析的标准DNA。利用Axygen AP-MN-P-50(爱思进生物技术(杭州)有限公司)试剂盒提取重组质粒DNA, 质粒DNA浓度使用Nanodrop ND-1000测定。根据已知重组质粒全序列长度和阿伏伽德罗常数计算对应的ITS rRNA基因拷贝数, 以10倍梯度对重组质粒进行梯度稀释(拷贝数为1010—106), 进行定量PCR扩增, 每个梯度3次重复, 制作标准曲线。
1.5 数据处理与统计分析采用SPSS 18.0统计软件IBM Group, Inc.进行数据处理, 使用新复极差法(SSR)多重比较检验差异显著性。采用Quantity One 4.4软件(Bio-Rad Laboratory,Inc.)进行DGGE图谱分析, 并采用未加权算术平均对群法(unweighted pair-group method using arithmetic averages, UPGMA)法对DGGE图谱进行聚类分析。运用Dice Coefficient法根据电泳图谱对样品进行相似性分析。使用香农-威纳指数(Shannon-Wiener index H)、丰度指数(R)和均匀度指数(E)对真菌群落多样性进行分析[24], 其计算公式为:
H=-∑(ni/N)lg(ni/N)
E=H/lnR
式中, ni为每条电泳条带光密度峰值;N是同一泳道中所有条带光密度峰值总和。电泳条带光密度的峰值通过分析软件Quantity one进行读取。R为每一泳道总的条带数。
使用CANOCO 4.5.1软件(Microcomputer Power, Ithaca, USA)对DGGE揭示的细菌群落结构与环境参数进行冗余分析(Redundancy Analysis-RDA)[25]。
2 结果与分析 2.1 秸秆还田不同时间土壤真菌的群体数量变化由图 1可以看出, 秸秆还田和秸秆未还田对照土壤的真菌ITS rRNA基因拷贝数在2.61×107— 6.46×107拷贝/克干土。随着秸秆还田时间的增加, 土壤真菌群体数量呈现上升趋势, 360 d的秸秆还田土壤真菌群体数量最高, 90 d的秸秆还田土壤真菌群体数量最低;90 d和270 d的秸秆还田与各自对照相比, 土壤真菌群体数量没有显著差异;180 d和360 d秸秆还田与各自对照相比, 土壤真菌群体数量显著增加。
|
| 图 1 秸秆还田不同时间土壤真菌的ITS rRNA基因拷贝数变化 Fig. 1 Copy numbers of ITS rRNA genes in the soil DNA extracts as determined by quantitative PCR under different days of rice straw decomposition (mean±SE) 不同小写字母表示不同时间处理间差异显著(P<0.05) |
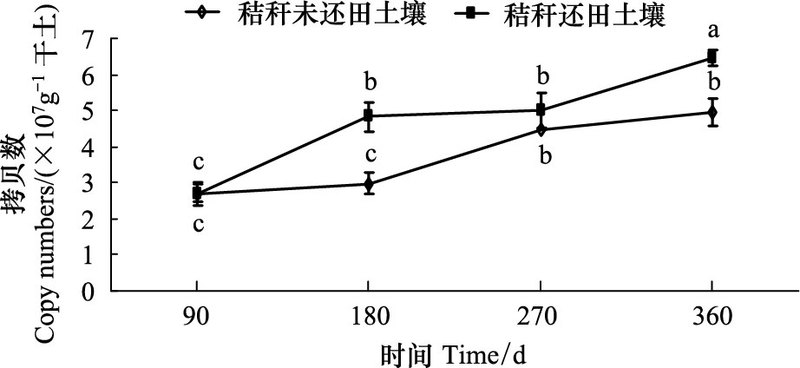
采用真菌特异性引物NS7-GC/NS8对18S rRNA基因进行PCR扩增, 纯化后进行DGGE分析(图 2)。图 2中可以看出, 随着秸秆还田时间的延长, PCR-DGGE图谱出现了较明显的差异, 表明土壤真菌群落结构发生明显变化。常见菌群如条带c, e, f, g, h, k在90, 180, 270、360 d的秆还田和秸秆未还田的土壤对照中均存在, 但条带的亮度发生明显的变化, 说明常见菌群数量发生了变化;随着秸秆还田时间的增加, 原有的优势菌群如条带a和b减弱或消失, 新的优势菌群如条带d、i、j出现或增强。

|
| 图 2 秸秆还田不同时间土壤真菌群落结构的PCR-DGGE分析 Fig. 2 PCR-DGGE analysis of soil fungal community structures in different days of rice straw decomposition T:处理(秸秆还田土壤)Treat (Straw decomposition soil);C:对照(秸秆未还田土壤)Control (No straw decomposition soil);Ⅰ、Ⅱ、Ⅲ:3次重复;图中小写字母代表测序DGGE条带 |
对秸秆还田不同时间土壤真菌的DGGE图谱条带进行多样性指数分析表明(表 2), 秸秆还田对真菌群落结构具有明显影响, 随着秸秆还田时间的增加, 多样性指数(H、R和E)均有升高趋势, 在360 d时, 秸秆还田土壤真菌多样性指数达到最高。
| 处理 Treatment | 香农-威纳指数(H)
Shannon-Wiener index | 丰富度(R)
Richness | 均匀度(E)
Evenness |
| C90 | 2.31±0.18b | 25.32±0.39ab | 0.94±0.12a |
| T90 | 2.28±0.14b | 26.91±0.53a | 0.95±0.10a |
| C180 | 1.99±0.09b | 21.84±0.65b | 0.82±0.03b |
| T180 | 2.71±0.10ab | 25.44±0.76ab | 0.93±0.02a |
| C270 | 2.14±0.15b | 20.65±0.56b | 0.91±0.03a |
| T270 | 2.60±0.12a | 26.29±0.87a | 1.00±0.08a |
| C360 | 2.45±0.09b | 25.66±0.54ab | 0.98±0.03a |
| T360 | 2.80±0.10a | 28.82±0.32a | 1.01±0.02a |
| T代表处理(秸秆还田土壤);C代表对照(秸秆未还田土壤); 同列不同小写字母表示差异显著(P<0.05) | |||
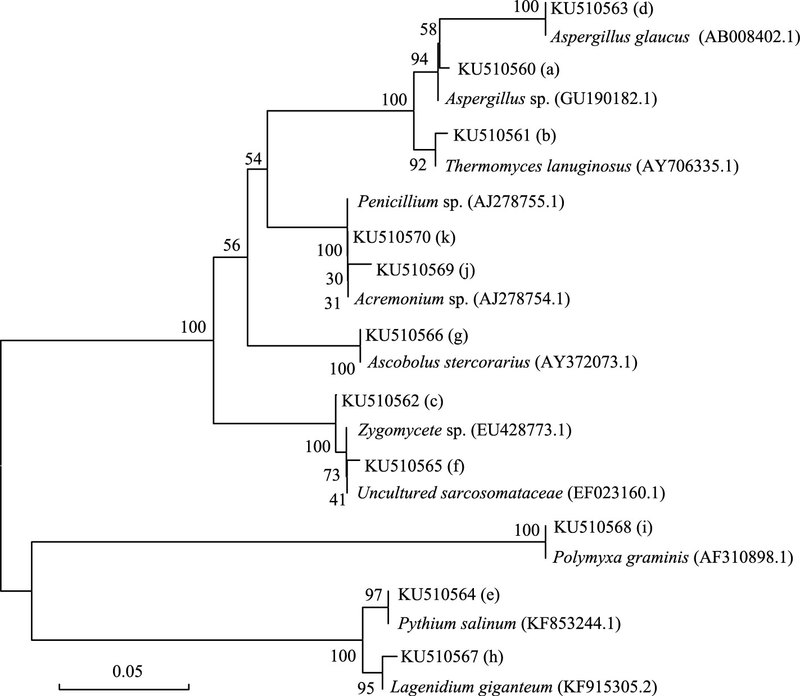
选取了11条DGGE图谱主要条带进行序列切胶回收测序(图 2), 分析秸秆还田后土壤真菌的群落演变过程。在Genebank中进行Blast序列比对, 利用MEGA 5.0构建系统发育树(图 3)。结果表明, 常见种群如条带c, e, f, g, h, k分别与接合菌(Zygomycete sp.)、盐腐霉菌(Pythium salinum)、肉盘菌(Uncultured Sarcosomataceae)、牛粪盘菌(Ascobolus stercorarius)、大链壶菌(Lagenidium giganteum)、青霉菌(Penicillium sp.)聚在一起;原有的优势种群如条带a和b与曲霉属真菌(Aspergillus sp.)和疏绵状丝孢菌(Thermomyces lanuginosus)聚在一起;新的优势种群如条带d、i、j与灰绿曲霉菌(Aspergillus glaucus)、禾谷多粘菌(Polymyxa graminis)、枝顶孢霉菌(Acremonium sp.)聚在一起。
|
| 图 3 秸秆还田不同时间土壤真菌DGGE条带序列的系统发育树 Fig. 3 Phylogenetic tree based on soil fungal DGGE profiles in different days of rice straw decomposition |
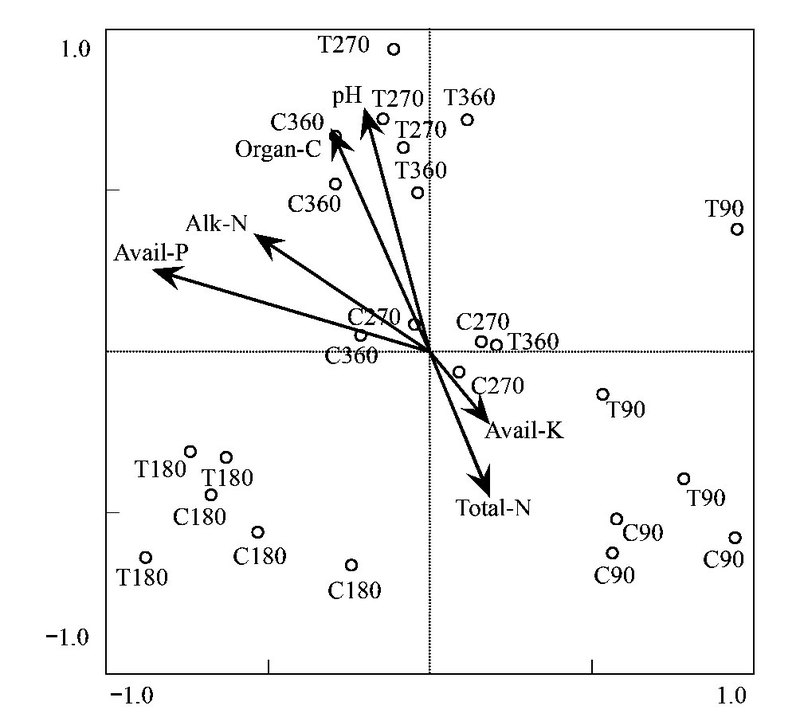
以秸秆还田不同时间土壤真菌DGGE图谱和土壤理化性质为两个变量组, 进行RDA分析(图 4, 表 3)。结果表明, 水稻秸秆还田后, 土壤真菌群落结构发生明显改变, 而随着秸秆还田时间的延长, 不同阶段土壤真菌群落差异明显。90 d和180 d秸秆还田和对照土壤样地较为接近, 说明其真菌群落结构相似度较高, 秸秆还田早期还未对真菌群落结构造成明显影响;270 d和360 d的秸秆还田和对照土壤样地差异较大, 说明其真菌群落的优势菌种发生变化, 群落结构相似度较低, 随着秸秆还田时间的增加, 秸秆还田已经引起真菌群落结构显著变化。
|
| 图 4 秸秆还田不同时间土壤真菌DGGE条带与土壤性质的冗余分析 Fig. 4 RDA of soil fungal DGGE profiles and soil chemistry properties in different days of rice straw decomposition T:代表处理(秸秆还田土壤)Treat (Straw decomposition soil); C:代表对照(秸秆未还田土壤) Control (No straw decomposition soil).Avail-K:速效钾Available K;Avail-P:速效磷Available P;Organ C:有机碳Organic carbon;Alk-N:碱解氮Alkalize-Hydrolyzed N; Total-N:总氮Total nitrogen;pH:pH值 |
| 项目Items | 排序轴1 Axes 1 | 排序轴2 Axes 2 | 排序轴3 Axes 3 | 排序轴4 Axes 4 |
| 特征值Eigenvalue | 0.309 | 0.144 | 0.067 | 0.041 |
| 物种-环境相关性Species-environment correlations | 0.867 | 0.885 | 0.875 | 0.909 |
| 累计百分比方差Cumulative percentage variance | ||||
| 数据物种of species data | 30.9 | 45.2 | 52.0 | 56.1 |
| 物种-环境关系of species-environment relation | 51.0 | 74.8 | 85.9 | 92.7 |
由表 3可知, RDA排序3个排序轴的特征值分别为0.309、0.144和0.067, 真菌群落组成与环境因子3个排序轴的相关系数分别为0.867、0.885和0.875。前两轴的特征值占总特征值80.7%, 说明排序效果很好。前两轴的物种与环境相关系数较高, 共解释了物种-环境关系总方差的74.8%。图 4表明, 土壤有机碳、pH值和速效磷对真菌群落结构的影响达到显著水平(P<0.05), 是引起土壤真菌群落结构及多样性变异的主要因素, 而速效钾和总氮的影响最小。
3 讨论本研究采用RDA分析了秸秆还田和对照土壤的6个环境因子对土壤真菌群落结构的影响, 发现土壤土壤有机碳、pH值和速效磷与真菌群落变化的相关性大于其他环境因子, 这与近来研究发现的土壤的pH值和有机碳是影响土壤微生物群落结构的重要因素相一致[26-27]。Sun等[18]研究认为, 土壤pH值是影响微生物群落结构最重要的因素之一。Lu等[28]研究发现, 革兰氏阴性细菌的丰富度受到pH值的显著影响。在本研究中, 对照土壤pH值呈先降低再升高的趋势, 而秸秆还田土壤pH值呈现逐渐升高的趋势, 在360 d时pH值均达到最大, 表明秸秆还田可以缓解土壤酸化, 这也与兰丽丽等[29]实验结果一致。Liu等[30]研究表明, 作物秸秆直接还田可以增加土壤有机碳含量, 从而提高土壤肥力。本研究结果表明, 相对于秸秆未还田对照, 随着秸秆还田时间的增加, 秸秆还田土壤有机碳的含量显著增加, 在还田360 d时达到最高值2.58%, 而总氮和碱解氮出现下降的趋势, 史央等[31]研究结果表明, 稻草秆物料经过一年的腐解, 仍有42.26%未被降解。因次本研究结果原因可能是秸秆还田后经过360 d的腐解, 水稻秸秆未完全被腐解, 土壤微生物继续可以分解秸秆, 促进秸秆碳释放, 使土壤有机碳含量持续增加, 约为还田90 d的近一倍。同时秸秆的C/N的比值影响秸秆腐解的速度, 水稻秸秆C/N约为100:1, 显著大于微生物自身的C/N(25:1到30:1之间), 微生物除了合成自身生长所需要的C/N, 为了利用多余的碳, 会继续从土壤中吸收氮, 从而造成土壤总氮和碱解氮的损失。张四海等[32]采用磷脂脂肪酸(PLFA)法测定土壤微生物生物量, 结果表明, 添加秸秆配合施入磷素对微生物总生物量、真菌数量和真菌/细菌比值具有显著的促进作用。本研究中土壤速效养分如速效磷的累积与真菌群落结构的变化具有较强的相关性, 可能是由于大量速效养分的明显变化对土壤真菌产生了胁迫作用, 影响了优势种群的分布, 具体的机理目前还没有明确, 有待于进一步研究。
由真菌ITS rRNA基因拷贝数的荧光定量PCR结果可知, 水稻秸秆还田使得土壤真菌群体数量发生显著变化, 180 d和360 d的秸秆还田与各自对照相比, 土壤真菌群体数量发生显著增加;而随着时间增加, 秸秆未还田对照土壤的真菌群体数量表现为升高的趋势, 原因推测是水稻秸秆未还田试验地的上茬作物是小麦, 小麦收获后少量秸秆可能残留在试验地中, 从而为秸秆未还田试验地的土壤真菌提供了有机碳源, 促进了真菌的生长。90 d秸秆还田和对照相比, 土壤真菌群体数量未发生显著增加, 表明秸秆还田早期未对其真菌群落结构造成明显影响;而270 d秸秆还田和对照相比, 土壤真菌群体数量也未发生显著变化, 原因可能是第二年7月底采样时, 上海崇明降雨较多, 土壤含水量和土壤温度较高, Brockett等[33]和Buyer等[34]研究表明土壤含水量和土壤温度可以显著影响微生物群落结构, 由此我们推测此时期的土壤含水量和土壤温度对真菌群体数量的影响超过了秸秆还田。
本研究中秸秆还田不同时间的土壤真菌DGGE图谱的聚类分析和RDA分析均表明, 90 d的秸秆还田和对照的土壤样品聚为一类, 同时其真菌群体数量和多样性指数(H、R和E)与对照均未表现明显差异, 说明秸秆还田前期未对其真菌群落结构造成明显影响, 与90 d定量PCR分析得出的秸秆还田和对照的真菌群体数量结果相一致。随着秸秆还田时间的增加, 真菌群体数量和多样性指数(H、R和E)均表现升高的趋势, 并在360 d时达到最高。刘骁蒨[35]进行DGGE条带测序发现, 秸秆还田和施肥的优势土壤真菌包含可以降解纤维素的半知菌纲的枝顶孢属(Acremonium sp.)。本研究对DGGE的11条特征条带进行序列切胶回收测序, 结果表明, 土壤真菌主要种群包括了工业中用以可以生产纤维素酶的真菌:青霉菌(Penicillium sp.)、曲霉属真菌(Aspergillus sp.)和枝顶孢霉属(Acremonium sp.)[36], 其中青霉菌(Penicillium sp.)在秸秆还田过程中均为优势菌群, 而枝顶孢霉属(Acremonium sp.)在90 d时作为新的优势菌群出现, 推测其在降解纤维素的过程中, 种群数量迅速增加。因而本研究结果为从这种特定的土壤微生境中分离生产纤维素酶活力较高的真菌提供了依据。
4 结论综上所述, 水稻秸秆还田可以增加土壤真菌群体数量和多样性指数, 还田270 d和360 d的真菌群落结构发生了显著变化, 土壤有机碳、pH和速效磷是引起土壤真菌群落结构及多样性变异的主要因素。
| [1] | Torsvik V, Øvreås L, Thingstad T F. Prokaryotic diversity:magnitude, dynamics, and controlling factors. Science, 2002, 296(5570): 1064–1066. DOI:10.1126/science.1071698 |
| [2] | Sparling G P. Soil microbial biomass, activity and nutrient cycling as indicators of soil health//Pankhurst C E, Doube B M, Gupta V V S R, eds. Biological Indicators of Soil Health. Wallingford, UK:CAB International, 1997:97-119. |
| [3] | Gil S V, Meriles J, Conforto C, Basanta M, Radl V, Hagn A, Schloter M, March G J. Response of soil microbial communities to different management practices in surface soils of a soybean agroecosystem in Argentina. European Journal of Soil Biology, 2011, 47(1): 55–60. DOI:10.1016/j.ejsobi.2010.11.006 |
| [4] | Zelles L. Fatty acid patterns of phospholipids and lipopolysaccharides in the characterisation of microbial communities in soil:a review. Biology and Fertility of Soils, 1999, 29(2): 111–129. DOI:10.1007/s003740050533 |
| [5] | Dilly O, Munch J C. Ratios between estimates of microbial biomass content and microbial activity in soils. Biology and Fertility of Soils, 1998, 27(4): 374–379. DOI:10.1007/s003740050446 |
| [6] | Riffaldi R, Levi-Minzi R, Saviozzi A, Benetti A. Adsorption on soil of dissolved organic carbon from farmyard manure. Agriculture, Ecosystems & Environment, 1998, 69(2): 113–119. |
| [7] | 陈洪章. 秸秆资源生态高值化理论与应用. 北京: 化学工业出版社, 2006. |
| [8] | 韩鲁佳, 闫巧娟, 刘向阳, 胡金有. 中国农作物秸秆资源及其利用现状. 农业工程学报, 2002, 18(3): 87–91. |
| [9] | 劳秀荣, 孙伟红, 王真, 郝艳如, 张昌爱. 秸秆还田与化肥配合施用对土壤肥力的影响. 土壤学报, 2003, 40(4): 618–623. DOI:10.11766/trxb200111300421 |
| [10] | 王虎, 王旭东, 田宵鸿. 秸秆还田对土壤有机碳不同活性组分储量及分配的影响. 应用生态学报, 2014, 25(12): 3491–3498. |
| [11] | Mary B, Recous S, Darwis D, Robin D. Interactions between decomposition of plant residues and nitrogen cycling in soil. Plant and Soil, 1996, 181(1): 71–82. DOI:10.1007/BF00011294 |
| [12] | 徐广, 刁治民, 曾智科, 王坚, 肖前青. 微生物降解秸杆的研究进展与前景展望. 科技资讯, 2007(22): 137–137. DOI:10.3969/j.issn.1672-3791.2007.22.119 |
| [13] | Frey S D, Six J, Elliott E T. Reciprocal transfer of carbon and nitrogen by decomposer fungi at the soil-litter interface. Soil Biology and Biochemistry, 2003, 35(7): 1001–1004. DOI:10.1016/S0038-0717(03)00155-X |
| [14] | Holland E A, Coleman D C. Litter placement effects on microbial and organic matter dynamics in an agroecosystem. Ecology, 1987, 68(2): 425–433. DOI:10.2307/1939274 |
| [15] | Baumann K, Marschner P, Smernik R J, Baldock J A. Residue chemistry and microbial community structure during decomposition of eucalypt, wheat and vetch residues. Soil Biology and Biochemistry, 2009, 41(9): 1966–1975. DOI:10.1016/j.soilbio.2009.06.022 |
| [16] | Rui J P, Peng J J, Lu Y H. Succession of bacterial populations during plant residue decomposition in rice field soil. Applied and Environmental Microbiology, 2009, 75(14): 4879–4886. DOI:10.1128/AEM.00702-09 |
| [17] | Fu Q L, Liu C, Ding N F, Lin Y C, Guo B, Luo J F, Wang H L. Soil microbial communities and enzyme activities in a reclaimed coastal soil chronosequence under rice-barley cropping. Journal of Soils and Sediments, 2012, 12(7): 1134–1144. DOI:10.1007/s11368-012-0544-7 |
| [18] | Sun B, Wang X Y, Wang F, Jiang Y J, Zhang X X. Assessing the relative effects of geographic location and soil type on microbial communities associated with straw decomposition. Applied and Environmental Microbiology, 2013, 79(11): 3327–3335. DOI:10.1128/AEM.00083-13 |
| [19] | Bardgett R D, Frankland J C, Whittaker J B. The effects of agricultural management on the soil biota of some upland grasslands. Agriculture, Ecosystems & Environment, 1993, 45: 25–45. |
| [20] | Marschner P, Umar S, Baumann K. The microbial community composition changes rapidly in the early stages of decomposition of wheat residue. Soil Biology and Biochemistry, 2011, 43(2): 445–451. DOI:10.1016/j.soilbio.2010.11.015 |
| [21] | 鲁如坤. 土壤农业化学分析方法. 北京: 中国农业科技出版社, 2000. |
| [22] | Okubo A, Sugiyama S I. Comparison of molecular fingerprinting methods for analysis of soil microbial community structure. Ecological Research, 2009, 24(6): 1399–1405. DOI:10.1007/s11284-009-0602-9 |
| [23] | Mitchell J I, Zuccaro A. Sequences, the environment and fungi. Mycologist, 2006, 20(2): 62–74. DOI:10.1016/j.mycol.2005.11.004 |
| [24] | 张金屯. 数量生态学. 北京: 科学出版社, 2004. |
| [25] | Braak C J F, Smilauer P. CANOCO reference manual and canodraw for windows user's guide:software for canonical community ordination (version 4.5). Ithaca:Microcomputer Power, 2002. |
| [26] | Rousk J, Brookes P C, Bååth E. Contrasting soil pH effects on fungal and bacterial growth suggest functional redundancy in carbon mineralization. Applied and Environmental Microbiology, 2009, 75(6): 1589–1596. DOI:10.1128/AEM.02775-08 |
| [27] | Griffiths R I, Thomson B C, James P, Bell T, Bailey M, Whiteley A S. The bacterial biogeography of British soils. Environmental Microbiology, 2011, 13(6): 1642–1654. DOI:10.1111/j.1462-2920.2011.02480.x |
| [28] | Lu P, Lin Y H, Yang Z Q, Xu Y P, Tan F, Jia X D, Wang M, Xu D R, Wang X Z. Effects of application of corn straw on soil microbial community structure during the maize growing season. Journal of Basic Microbiology, 2015, 55(1): 22–32. DOI:10.1002/jobm.v55.1 |
| [29] | 兰丽丽. 添加不同作物秸秆对土壤酸度变化的影响[D]. 长春: 吉林农业大学, 2015. |
| [30] | Liu E K, Yan C R, Mei X R, He W Q, Bing S H, Ding L P, Liu Q, Liu S, Fan T L. Long-term effect of chemical fertilizer, straw, and manure on soil chemical and biological properties in northwest China. Geoderma, 2010, 158(3/4): 173–180. |
| [31] | 史央. 红壤中秸秆降解的微生物演替及应用研究. 南京: 南京师范大学, 2003. |
| [32] | 张四海, 黄健, 骆争荣, 董曙光, 王意锟, 朱强根, 张龙, 金爱武. 添加秸秆和磷素对土壤微生物群落的影响. 应用生态学报, 2014, 25(3): 797–802. |
| [33] | Brockett B F T, Prescott C E, Grayston S J. Soil moisture is the major factor influencing microbial community structure and enzyme activities across seven biogeoclimatic zones in western Canada. Soil Biology and Biochemistry, 2012, 44(1): 9–20. DOI:10.1016/j.soilbio.2011.09.003 |
| [34] | Buyer J S, Teasdale J R, Roberts D P, Zasada I A, Maul J E. Factors affecting soil microbial community structure in tomato cropping systems. Soil Biology and Biochemistry, 2010, 42(5): 831–841. DOI:10.1016/j.soilbio.2010.01.020 |
| [35] | 刘骁蒨. 秸秆还田方式与施肥对水稻土壤微生物学特性的影响[D]. 雅安: 四川农业大学, 2013. |
| [36] | 谢天文, 刘晓风, 袁月祥, 闫志英, 贺蓉娜, 廖银章. 真菌产纤维素酶的诱导物及其调控机理研究进展. 应用与环境生物学报, 2010, 16(3): 440–444. |
 2017, Vol. 37
2017, Vol. 37