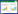文章信息
- 杨亚东, 张明才, 胡君蔚, 张凯, 胡跃高, 曾昭海
- YANG Yadong, ZHANG Mingcai, HU Junwei, ZHANG Kai, HU Yuegao, ZENG Zhaohai.
- 施氮肥对华北平原土壤氨氧化细菌和古菌数量及群落结构的影响
- Effects of nitrogen fertilizer application on abundance and community structure of ammonia oxidizing bacteria and archaea in a North China agricultural soil
- 生态学报. 2017, 37(11): 3636-3646
- Acta Ecologica Sinica. 2017, 37(11): 3636-3646
- http://dx.doi.org/10.5846/stxb201603220512
-
文章历史
- 收稿日期: 2016-03-22
- 网络出版日期: 2017-02-22
硝化作用是农田氮素循环的重要环节, 也是土壤氮素转化的关键过程[1]。氨氧化作用是硝化作用的第一步反应, 也是整个过程的限速步骤[2]。在很长一段时间内, 来自变形菌纲的氨氧化细菌(AOB)[2-3]被认为是其唯一承担者, 直到氨氧化古菌(AOA)[4-5]的发现, 才证实了氨氧化过程是由AOB和AOA两类微生物共同承担。Leininger等[6]首次报道了原始土壤和农业土壤中AOA在数量上远高于AOB, 再次证实了AOA是参与土壤环境中氨氧化过程的重要微生物。
前人的研究表明, AOB和AOA数量和群落组成受氨的有效性[3]、温度[7]、土壤pH[8]、土壤含水量[9]和施肥措施[10]等因素影响。Zhou等[11]研究表明施用氮肥可以提高AOB数量, 而施用有机肥则增加了AOA多样性;张苗苗等[12]研究显示长期施用氮肥降低了AOB多样性, 显著提高了AOA数量;Di等[13]发现草地土壤中AOA数量远高于AOB, 而施用动物尿液(富含NH3)显著促进了AOB数量的增加;Chen等[14]研究结果也证实了施肥对AOA数量影响强于对AOB的影响, 且影响AOA群落组成;Long等[15]研究却发现施氮肥对AOB和AOA数量影响不显著;关于施氮水平对AOB和AOA的影响研究方面, Zhong等[16]研究了温室蔬菜不同施氮量对AOB和AOA的影响, 结果表明土壤施氮量增加后, AOB和AOA的数量均下降, 且增施氮肥对AOB群落结构的影响强于对AOA群落结构的影响。施用氮肥对土壤AOB和AOA的影响因氮肥类型和施氮量水平不同存在显著差异, 而施氮水平对华北地区小麦生产系统土壤AOB和AOA数量和群落组成的影响仍不清楚, 亟待研究。
华北平原是我国重要的粮食主产区, 长期小麦-玉米集约种植模式需要大量的肥水投入, 带来温室气体(N2O、NO)排放和地下水污染等问题[10], 给区域造成很大的生态压力。研究不同施氮水平对土壤AOB和AOA数量和群落结构的影响可以进一步明确氮肥转化的微生物机制。本研究以连续5a的田间定位试验为基础, 以华北平原小麦季土壤为研究对象, 采用荧光定量PCR(Real-time quantitative PCR, q-PCR)、末端限制性片段长度多样性(T-RFLP)和基因克隆文库技术, 比较了4种施氮水平下小麦土壤AOB和AOA数量和群落组成的差异。旨在阐明华北平原小麦土壤中AOB和AOA对氮肥的响应机制, 为土壤科学施肥、维护土壤微生物多样性和农业可持续发展等提供依据。
1 材料与方法 1.1 试验设计和土壤样品采集试验地位于河北省沧州市吴桥县中国农业大学吴桥实验站(37°37′N, 116°22′E)。该地区属暖温带半湿润大陆性季风气候, 土壤类型为中壤质潮土, 成土母质为河流冲积物。年平均气温12.9℃, 全年积温(≥ 0℃)为4, 826℃, 无霜期201 d, 降雨量为562 mm, 日照时数2724 h。氮肥梯度试验起始于2010年, 种植模式为冬小麦-夏玉米一年两熟, 冬小麦季施氮量设置4个梯度, 分别为不施氮肥(0 kg N/hm2, CK), 施低水平氮肥(75 kg N/hm2, N1), 施中水平氮肥(150 kg N/hm2, N2) 和施高水平氮肥(225 kg N/hm2, N3), 磷肥施用量90 kg P2O5/hm2, 钾肥施用量90 kg K2O/hm2。氮肥、磷肥和钾肥分别为尿素、磷酸二氢钙和硫酸钾, 肥料均基施。小区面积为24 m2, 重复3次, 完全随机区组排列。
土壤样品采集于2015年6月冬小麦收获后, 采用对角线五点采样法, 每个小区采集5个点0—20 cm土壤。同一处理的土壤样品混合成一个样品, 除去碎石、根系及其他杂物, 过2 mm筛, 将土样分成两部分, 一部分迅速用锡箔纸包裹后液氮冷冻, 用冰盒保存带回实验室后-80℃保存, 用于DNA提取及后续分析;另一部分装入自封袋后带回实验室, 用鲜土样测定铵态氮和硝态氮含量, 剩余土壤自然风干后测定土壤pH、有机质和全氮含量。
1.2 土壤理化性质测定土壤pH采用电位法(水:土=2.5:1) 测定, 土壤全氮采用浓硫酸-蒸馏滴定法测定, 土壤铵态氮和硝态氮用2 mol/L KCl溶液浸提新鲜土样后, 采用流动分析仪测定(SAN++, Skalar, Holland), 土壤有机质采用重铬酸钾容量法测定[17]。
1.3 土壤总DNA提取取0.2—0.5 g冷冻土壤样品, 用E.Z.N.A.® Soil DNA Kit(Omega, USA)试剂盒提取土壤总DNA。用1%琼脂糖凝胶电泳检测总DNA完整性, 用NANO Quant(Tecan, Switzerland)测定提取DNA浓度和纯度。
1.4 总细菌16S rDNA、AOB和AOA amoA基因的扩增与荧光定量PCR分析分别用引物BACT1369F/PROK1541R、amoA-1F/amoA-2R和Arch-amoAF/Arch-amoAR扩增细菌16S rDNA、AOB和AOA amoA基因(表 1)。PCR体系为50 μL, 分别含10× Buffer 5.0 μL, dNTP(各2.5 μmol/L)4.0 μL, 上下游引物(10 μM)各1.0 μL, DNA模板(1—10 ng)1 μL, rTaq(5 U/μL)1.0 μL, 最后用ddH2O补至50.0 μL。PCR程序:95℃预变性3 min;95℃变性30 s, 55℃退火30 s, 72℃延伸45 s, 35个循环;最后72℃延伸5 min。回收PCR产物连接至pMD18-T载体, 转化至大肠杆菌DH5α感受态中, 经Amp+、IPTG和X-gal的LB平板筛选阳性克隆, 测序分析。
| 靶标 Target | 引物名称 Primer | 序列(5′—3′)
Sequence (5′—3′) | 片段长度 Length (bp) | 荧光定量PCR程序 Real-time PCR profiles | 参考文献 References |
| 细菌 Bacteria | BACT1369F PROK1541R | 5′-CGGTGAATACGTTCYCGG-3′
5′-AAGGAGGTGATCCRGCCG CA-3′ | 173 | 95℃ 10 min; 95℃ 10 s, 60℃ 1 min, 35 cycles | [17] |
| 氨氧化细菌 AOB | amoA-1F amoA-2R | 5′-GGGGTTTCTACTGGTGGT-3′
5′-CCCCTCKGSAAAGCCTTCTTC-3′ | 491 | 95℃ 10 min; 95℃ 10 s, 55℃ 30 s, 68℃ 40 s, 40 cycles | [17] |
| 氨氧化古菌 AOA | Arch-amoAF Arch-amoAR | 5′-STAATGGTCTGGCTTAGACG-3′
5′-GCGGCCATCCATCTGTATGT-3′ | 635 | 95℃ 10 min; 95℃ 15 s, 55℃ 30s, 68℃ 40 s, 40 cycles | [17]. |
| Y表示C或T;R表示A或G;K表示G或T;S表示C或G;AOB:氨氧化细菌Ammonia oxidizing bacteria;AOA:氨氧化古菌Ammonia oxidizing archaea | |||||
标准曲线的制作:提取测序正确的细菌16S rDNA、AOB和AOA amoA基因的阳性克隆质粒, 用NANO Quant(Tecan, Switzerland)测定质粒浓度, 按10倍梯度稀释质粒至103—108拷贝数/微升, -80℃保存, 用于制备标准曲线。
荧光定量PCR在ABI 7300(ABI, USA)上进行, 反应体系为20 μL, 分别含SYBR® Premix Ex Taq Ⅱ(Thi RNaseH Plus)10.0 μL, 上下游引物各0.8 μL(10 μM), ROX Reference Dye(50x)0.4 μL, DNA模板2.0 μL(1—10 ng), 用ddH2O补至20.0 μL。PCR引物及程序如表 1所示。
1.5 末端限制性片段长度多样性分析对AOB和AOA进行T-RFLP分析, 选用amoA-1F/amoA-2R和Arch-amoAF/Arch-amoAR扩增AOB和AOA amoA基因, 其中引物amoA-1F和Arch-amoAF的5′端用6-FAM进行荧光标记, PCR体系和程序同1.4所示。回收PCR产物后, 用限制性内切酶Rsa Ⅰ(Taraka, 大连)分别酶切AOB和AOA amoA基因。反应体系为20 μL, 分别含10× T Buffer 2 μL, 0.1% BSA 2 μL, Rsa Ⅰ(10 U/μL)1 μL, PCR产物DNA 10 μL, 最后用ddH2O补至20.0 μL。37℃水浴酶切3 h, 重复3次, 用乙醇沉淀法纯化酶切产物, 最后将3个重复混匀成一个样品。取回收的酶切产物0.3 μL、分子量内标(GS500LIZ)0.5 μL和甲酰胺9.5 μL混合均匀后加入96孔板中, 95℃变性5 min, 4℃冷却后离心, 1× Buffer缓冲液上机检测, 采用ABI 3730(ABI, USA)测序仪测定分析。用GeneMarker V 2.2(ABI, USA)分析电泳图中各峰的片段长度和峰面积, 忽略长度小于50 bp和峰面积小于1%的峰。将峰的信号强度标准化, 每种末端限制性片段(T-RF)类型的相对丰度用百分比形式表示。
1.6 克隆和测序将4个土壤样品的DNA混合后做模板, 采用amoA-1F/amoA-2R和Arch-amoAF/Arch-amoAR扩增AOB和AOA amoA基因, 体系和扩增条件同1.4所示, 引物不添加荧光标记。扩增产物回收后, 分别连接至pMD18-T载体(Taraka, 大连)上, 转化至大肠杆菌DH5α感受态中, 经筛选后分别选取了53和56个阳性克隆进行测序分析。测序结果经分析比对后, 将同源性≥97%的序列归为一类, 选一条用于构建系统发育树。将具有代表性的AOB和AOA amoA基因序列提交至NCBI数据库中, 获AOB和AOA登录号分别为KU365731—KU365744和KU365745—KU365753。
1.7 数据分析数据的方差分析和相关性分析用SPSS 20.0软件(SPSS 20.0 for Windows, SPSS Inc, USA)完成;土壤总细菌、AOB和AOA数量的数据取对数后, 用SigmaPlot 12.5软件制图;测序核苷酸序列用DNAMAN 6.0软件分析, 并用MEGA 5.0软件构建系统进化树, 选用邻-接法(Neighbour-Joining);用末端限制性片段(T-RF)的长度和相对含量数据进行主成分分析, 由Canoco 5.0软件完成。
2 结果 2.1 土壤基础理化性质不同施氮量0—20 cm土层土壤中除有机质含量差异不显著(14.96—15.49 g/kg, P>0.05) 外, 其他理化性质存在显著差异(表 2)。随施氮量增加, 土壤pH呈显著下降趋势(8.46—8.29), 依次为N3<N2<N1<CK(P<0.05)。增施氮肥提高了土壤全氮(0.86—1.04 g/kg)、铵态氮(0.92—1.54 mg/kg)和硝态氮含量(3.76—24.29 mg/kg)。N3和N2土壤全氮含量显著高于N1和CK(P<0.05);N3土壤铵态氮含量显著高于N2、N1和CK(P<0.05);不同施氮处理土壤硝态氮含量均存在显著差异, 依次为CK<N1<N2<N3(P<0.05)。随着施氮量增加, 土壤碳氮比下降, CK显著高于N2和N3, N1显著高于N3(P<0.05)。
| 处理 Treatment | pH | 有机质 Total organic matter / (g/kg) | 全氮含量 Total Nitrogen / (g/kg) | 铵态氮 NH4+-N / (mg/kg) | 硝态氮 NO3--N / (mg/kg) | 碳氮比 C/N |
| 不施氮肥No N fertilization, CK | 8.46±0.01 a | 14.96±0.87 a | 0.86±0.06 b | 0.92±0.09 c | 3.76±0.44 d | 10.14±0.73 a |
| 施低水平氮肥 Low N fertilization rate, N1 | 8.38±0.01 b | 15.18±.050 a | 0.91±0.01 b | 1.24±0.12 b | 6.36±0.23 c | 9.65±0.29 ab |
| 施中水平氮肥 Moderate N fertilization rate, N2 | 8.34±0.02 c | 15.25±0.26 a | 0.99±0.02 a | 1.22±0.15 b | 7.92±0.22 b | 8.95±0.05 bc |
| 施高水平氮肥 High N fertilization rate, N3 | 8.29±0.01 d | 15.49±0.41 a | 1.04±0.02 a | 1.54±0.08 a | 24.29±0.69 a | 8.67±0.37 c |
| 不同字母表示处理间差异显著(P<0.05), 数据为平均值±标准差, n=3;CK 0 kg N/hm2, N1 75 kg N/hm2, N2 150 kg N/hm2, N3 225 kg N/hm2 | ||||||
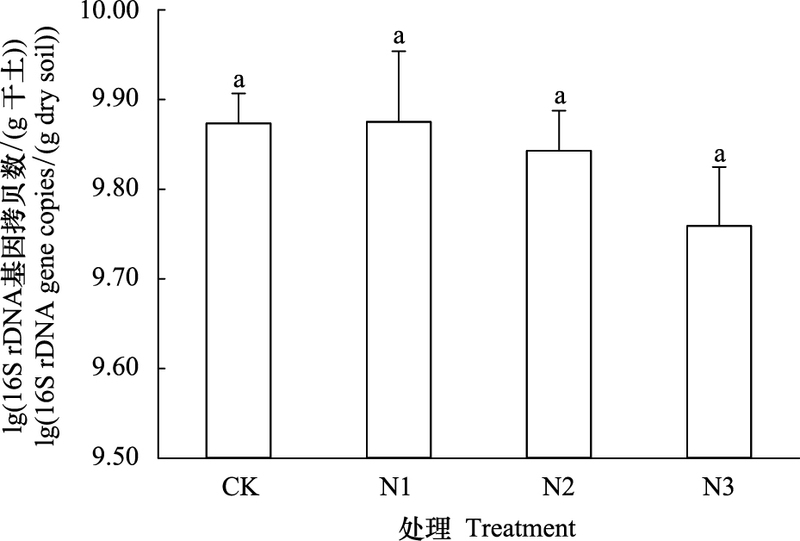
以16S rDNA和amoA基因为靶标, 用荧光定量PCR测定土壤总细菌、AOB和AOA数量。结果显示, 不同施氮处理土壤总细菌数量在5.74×109—7.50×109拷贝数/g干土, 处理间差异不显著(P>0.05, 图 1)。不同施氮处理土壤AOB数量在8.89×106—2.66×107拷贝数/g干土, 不同施氮处理AOB数量均差异显著, 随施氮量增加, AOB数量显著增加(P<0.05)。与CK相比, N1、N2、N3中AOB数量分别提高了64%、150%和199%。AOA数量在3.83×108—7.78×108拷贝数/g干土, CK、N1和N2中AOA数量显著高于N3(P<0.05, 图 2)。相关性分析结果显示, 总细菌数量仅与硝态氮(r=-0.582, n=12, P<0.05) 呈显著负相关关系。AOB数量与全氮(r=0.838, n=12, P<0.01) 和铵态氮(r=0.597, n=12, P<0.05) 呈显著正相关关系, 与土壤pH(r=-0.859, n=12, P<0.01) 和碳氮比(r=-0.784, n=12, P<0.01) 呈显著负相关关系。AOA数量与硝态氮(r=0.926, n=12, P<0.01) 和土壤pH(r=0.594, n=12, P<0.05) 呈显著正相关关系, 与铵态氮(r=-0.717, n=12, P<0.05) 呈显著负相关关系。
|
| 图 1 不同施氮处理细菌16S rDNA基因拷贝数 Fig. 1 Abundance of bacteria 16S rDNA gene under different N fertilizer application rate treatments |
|
| 图 2 不同施氮处理氨氧化细菌和氨氧化古菌amoA基因拷贝数 Fig. 2 Abundance of ammonia oxidizing bacteria and archeae amoA genes under different N fertilizer application rate treatments |
不同施氮处理土壤AOA数量均显著高于AOB数量, AOA与AOB数量比值在81.72—14.38之间, 随施氮量增加, AOA与AOB数量比值呈下降趋势。荧光定量结果表明, 增施氮肥能显著提高小麦土壤AOB数量, 对总细菌和AOA数量影响不显著。此外, 总细菌、AOB和AOA数量与土壤各环境因子的相关性存在明显差异。
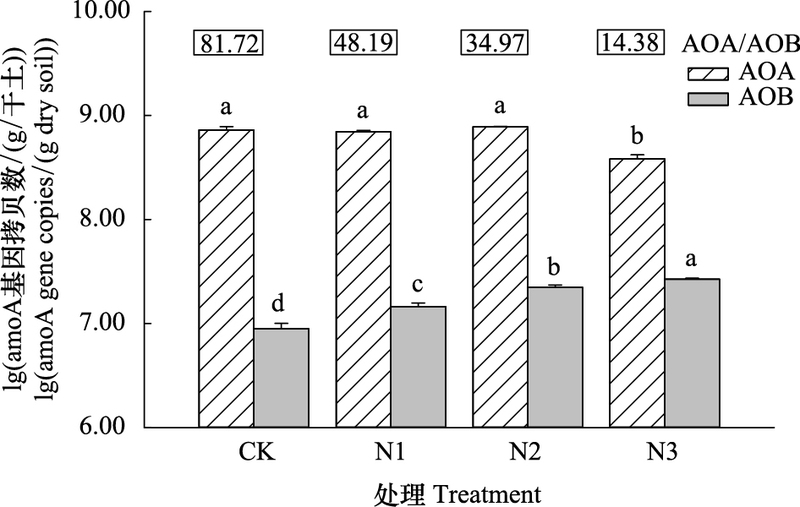
2.3 AOB和AOA的群落结构选用限制性内切酶RsaⅠ分别对AOB和AOA进行T-RFLP分析。4个处理共得到10个AOB的T-RF片段长度(图 3a), 其中96、210、251、244、255、270、274和380 bp在4个处理中均存在, 63 bp只存在于CK、N1和N2中, 248 bp只存在于N2和N3中。长度为210、251、255、274和380 bp的T-RFs为主要片段, 分别占总片段的3.91%—11.38%、26.60%—29.71%、26.97%—39.49%、6.50%—11.20%和11.87%—17.76%, 合计占总片段的92.74%—93.34%。这些片段所代表的AOB在土壤中占优势, 其中251、255和380 bp长度所占比例均超过10%, 为占绝对优势的3个AOB类群。
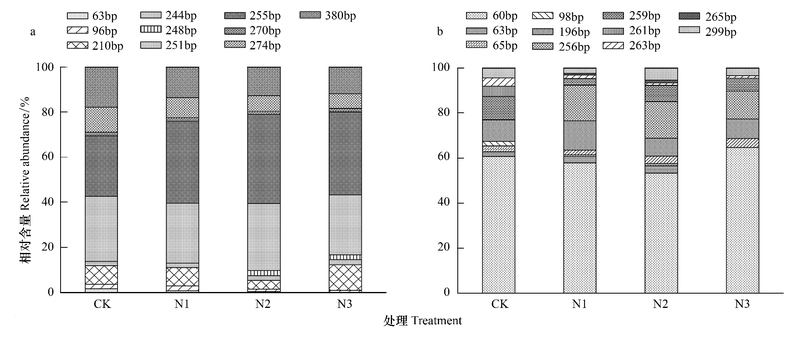
|
| 图 3 不同施氮处理氨氧化细菌(a)和古菌(b)amoA基因T-RFs的相对丰度 Fig. 3 Relative abundance of T-RFs of ammonia oxidizing bacteria (a) and archaea (b) amoA genes restricted by RsaⅠ under different N fertilizer application rate treatments |
4个处理共得到11个AOA的T-RF片段长度(图 3b), 其中60、98、196、259、261、263和299 bp在4个处理中均存在, 63和65 bp只存在于CK、N1和N2中, 256 bp只存在于N1、N2和N3中, 261 bp只存在于CK中, 265 bp只存在于N1和N2中。长度为60、196、256、259和299 bp的T-RFs为主要片段, 分别占总片段的53.26%—64.76%、7.79%—12.88%、12.26%—16.17%、2.94%—10.45%和2.46%—5.34%, 合计占总片段的85.13%—94.91%。这些片段所代表的AOA在土壤中占优势, 其中60和256 bp长度所占比例均超过10%, 为占绝对优势的2个AOA类群。
2.4 AOB和AOA amoA基因序列的系统发育树构建经测序比对分析后, 分别得到14和9个AOB和AOA amoA基因操作分类单元(operational taxonomic unit, OTU)。AOB amoA基因序列两两同源性在69.92%—96.54%之间, 14条序列的同源性为91.77%;AOA amoA基因序列两两同源性在77.48%—96.22%之间, 9条序列的同源性为87.44%。系统进化分析结果显示:AOB amoA基因序列聚类在两个Cluster中, 其中, AOB 12聚类在N. communis Cluster中, 其余AOB都聚类在Cluster 3中。聚类在Cluster 3中的AOB分布在Cluster 3a和Cluster 3b两个分支中, 除片段长度为210 bp的T-RF聚类在Cluster 3b外, 其他片段长度T-FRs均聚类于Cluster 3a(图 4);所有AOA amoA基因序列都聚类在Cluster S中, 分布在4个分支中。其中, 196 bp T-RF聚类在分支Ⅰ和Ⅲ中, 60 bp T-RF聚类在分支Ⅱ中, 63 bp T-RF聚类在分支Ⅱ和Ⅲ中, 299 bp T-RF聚类在分支Ⅰ和Ⅳ中(图 5)。
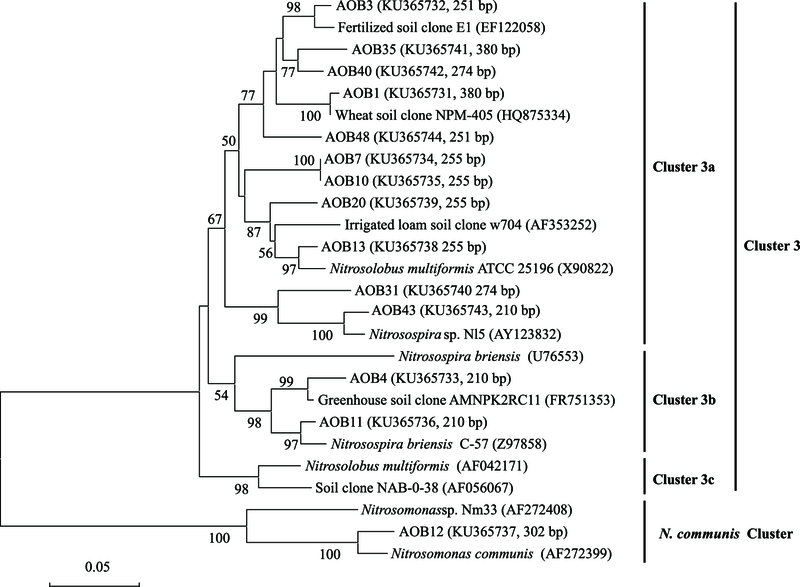
|
| 图 4 氨氧化细菌amoA基因系统发育树 Fig. 4 Phylogenetic tree of ammonia oxidizing bacteria amoA gene sequences |
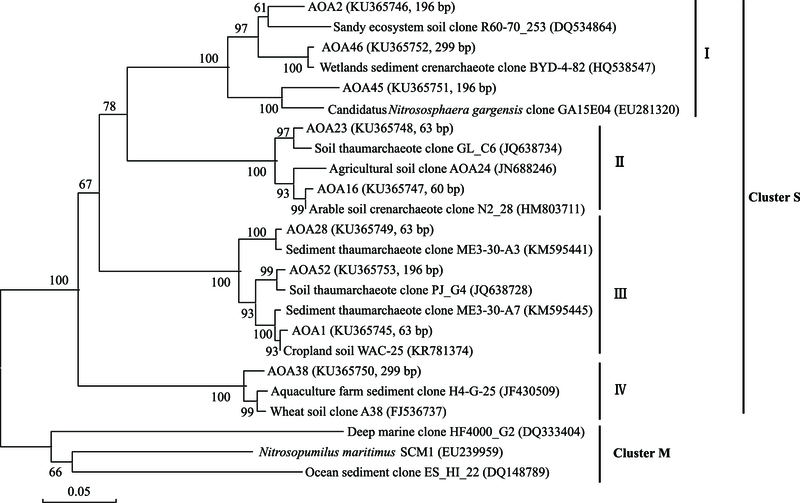
|
| 图 5 氨氧化古菌amoA基因系统发育树分析 Fig. 5 Phylogenetic tree of ammonia oxidizing archaea amoA gene sequences |
基于T-RFs的主成分分析(PCA)结果显示, 第一、二排序轴是主成分轴。对AOB群落变异的解释量达到95.16%, 其中轴1和轴2分别为76.81%和18.35%(图 6a);对AOA群落变异的解释量达到94.44%, 其中轴1和轴2分别为77.22%和17.22%(图 6b)。处理CK、N1、N2和N3的AOB都明显分开, 分布在4个象限中;而4个处理的AOA群落结构差异弱于AOB, 其中, N1和N2聚在一起(第三象限), 与CK和N3明显分开。PCA结果表明:施氮量显著影响小麦土壤AOB和AOA的群落结构, 且不同施氮处理的AOB群落结构差异强于AOA。

|
| 图 6 不同施氮处理氨氧化细菌(a)和古菌(b)amoA基因T-RFs主成分分析 Fig. 6 Principal component analysis (PCA) of T-RFs of ammonia oxidizing bacteria (a) and archaea (b) amoA genes under different N fertilizer application rate treatments |
土壤总细菌数量是反映土壤总微生物活性的一个重要参数。本研究中土壤总细菌16S rDNA基因在5.74×109—7.50×109拷贝数/g干土之间, 与Shen等[17]研究发现细菌数量在3.39×109—1.23×1010拷贝数/g干土之间的结果相似, 相较于He等[8]在水稻土壤中细菌数量1010拷贝数/g干土数量级低, 且总细菌数量仅与硝态氮呈显著负相关关系。小麦与水稻土壤类型和理化性质差异可能是引起总细菌数量差异的主要原因。此外, 本研究不同施氮水平土壤总细菌数量差异不显著, 与He等[8]和Shen等[17]研究结果一致, 表明土壤总细菌数量对施氮肥响应不明显。
AOB和AOA是土壤氮循环的关键微生物, 直接影响着土壤健康、肥力状况和生产力等。本研究不同施氮量处理AOA数量均高于AOB, AOA与AOB数量比值在81.72—14.38之间, 与Leininger等[6]、He等[8]、Shen等[17]和Jia等[18]研究结果相符, 进一步证实了碱性土壤更适合AOB。AOB数量在8.89×106—2.66×107拷贝数/g干土, 与He等[8]、Chen等[14]和Wessén等[19]研究中AOB数量级一致, 比Shen等[16]、Wang等[20]和Yao等[21]研究中AOB高出2—3个数量级。AOA数量在3.83×108—7.78×108拷贝数/g干土, 与Chen等[14]研究中水稻土壤AOA数量级一致, 比He等[8]、Shen等[17]和Wang等[20]研究中AOA数量高出1—2个数量级。此外, 本研究中AOB数量随施氮量增加而显著增加, 而AOA数量差异不显著, 与Di等[22]研究发现高氮草地土壤中AOB数量随施氮量增加而增加一致, 而与Zhong等[16]研究表明随着施氮量增加, AOB和AOA数量均下降不同。
He等[10]研究表明土壤pH是造成AOB和AOA数量差异的主要原因, 而Chen等[14]认为肥料是引起AOA数量变化的主要原因, 土壤pH和肥料均对AOB数量产生影响;Di等[22]认为硝态氮含量是引起AOB数量增加的原因;但Zhong等[22]在温室条件下却得到相反的结果, 认为硝态氮含量是导致AOB数量下降的原因, 而AOA数量下降则可能是由于土壤pH降低造成。本研究中AOB数量与土壤pH呈极显著负相关关系, 与全氮含量呈极显著正相关关系, AOB数量上升可能是土壤pH下降和肥料作为底物导致, 与Di等[22]结果一致。AOA数量与土壤pH呈显著正相关关系, 而与全氮含量相关性不显著, AOA数量的降低, 可能是由施氮量增加引起的土壤pH下降造成, 而非氮肥作为底物造成, 与He等[8]和Chen等[14]研究结果相符。不同施氮处理土壤pH均呈碱性, 且长期增施氮肥引起土壤pH降低导致了AOA数量下降和AOB数量上升, 与Shen等[23]报道的AOB适合在中性至碱性、氮素含量丰富的土壤中生存, 而AOA适合在酸性土壤中生存的结果一致。本研究中不同施氮处理AOA在数量上占优势, 随着施氮量增加, AOB数量显著上升, AOA数量上的优势减弱, 表明AOB对氮肥更敏感, 高氮条件下土壤中氨氧化过程的主要承担者可能是AOB而非AOA, 与Jia等[18]和Shen等[23]结论一致。
3.2 施氮肥对小麦土壤AOB和AOA群落组成的影响本研究用限制性内切酶RsaⅠ酶切AOB和AOA amoA基因经后测序分别得到了10和11个T-RFs, 与Zhou等[11]研究紫土中AOB用Acc Ⅱ酶切得到11个T-RFs, 莫旭华等[24]研究小麦土壤中AOA用AfaⅠ酶切得到10个T-RFs的结果一致;但Chen等[25]研究发现草原土壤中AOB用MobⅠ酶切后仅得到4个T-RFs, Chen等[14]研究发现水稻土壤中AOA用MobⅠ酶切后仅得到4个T-RFs。T-RFs数量不同, 可能是选用内切酶不同导致, 也可能是由于土壤类型差异引起。
不同施氮处理中, T-RF长度为63、96、274、380、248和255 bp的AOB类群所占比例差异较大, 且随施氮量的增加表现出不同的变化趋势;而T-RF长度为256和265 bp的AOA类群差异较大, 且4个处理中AOA均含有2个绝对优势的类群(>10%), 表明小麦土壤中AOB的群落组成受施氮量的影响较大, 而AOA具有较稳定性的群落结构,与Shen等[17]碱性土壤条件下得到的结果一致, 而与He等[8]、Chen等[14]、Ying等[26]酸性土壤条件下得到的结果相反。另外, PCA分析也显示4个处理AOB比AOA群落结构差异性更大, AOB群落结构更易受氮肥的影响。这些结果都证实了施肥引起AOB和AOA群落结构的变化, 且碱性条件下AOB比AOA对氮肥施用量更敏感。
3.3 施氮肥小麦土壤中AOB和AOA的系统发育本研究中绝大多数AOB都聚类在Cluster 3的两个分支中, 除AOB 12属于Nitrosomonas属外, 其他AOB都聚类在Nitrosospira属中, 与莫旭华等[24]和Chu等[27]在小麦土壤中得到的AOB都属于Nitrosospira属的结果相符;也与水稻土壤[14]、黄土旱塬黑垆土壤[28]和草原[25]等土壤中AOB都主要聚类在Nitrosospira属中的结果一致;符合Kowalchuk等[29]报道的土壤环境中氨氧化细菌以Nitrosospira属为主, 而非Nitrosomonas属的结论。He等[8]、Chen等[14]和Shen等[16]在水稻土壤中均发现了聚类于Cluster S和Cluster M的AOA类群, pH越低, 聚类于Cluster M的AOA类群越多, 并且Cluster S在农田和草地生态系统中占绝对优势。本研究中所有AOA均聚类于Cluster S中, 未发现聚类在Cluster M的AOA类群, 与Chen等[25]和Shen等[30]的结果一致, 与农田生态系统中聚类于Cluster S的AOA占绝对优势的结果相符, 本研究中土壤pH呈碱性是导致所有AOA都分布在Cluster S中的主要原因。
4 结论本研究对长期施用不同氮肥水平下小麦季土壤氨氧化微生物的群落结构和丰度特征进行了研究, 结果发现, 随施氮量增加引起土壤pH下降及其他理化性质的改变, 施氮量增加显著提高AOB的数量, 对AOB群落结构的影响强于对AOA群落结构的影响, 表明该地区小麦土壤中AOB比AOA对施用氮肥的响应更敏感。
致谢: 本研究得到农业部都市农业(北方)重点实验室马兰青教授、杨明峰副教授的帮助, 特此致谢。| [1] | 贺纪正, 张丽梅. 氨氧化微生物生态学与氮循环研究进展. 生态学报, 2009, 29(1): 406–415. |
| [2] | Prosser J I. Autotrophic nitrification in bacteria. Advances in Microbial Physiology, 1989, 30: 125–181. |
| [3] | Kowalchuk G A, Stephen J R. Ammonia-oxidizing bacteria:a model for molecular microbial ecology. Annual Review of Microbiology, 2001, 55(1): 485–529. DOI:10.1146/annurev.micro.55.1.485 |
| [4] | Venter J C, Remington K, Heidelberg J F, Halpern A L, Rusch D, Eisen J A, Wu D Y, Paulsen I, Nelson K E, Nelson W, Fouts D E, Levy S, Knap A H, Lomas M W, Nealson K, White O, Peterson J, Hoffman J, Parsons R, Baden-Tillson H, Pfannkoch C, Rogers Y H, Smith H O. Environmental genome shotgun sequencing of the Sargasso Sea. Science, 2004, 304(5667): 66–74. DOI:10.1126/science.1093857 |
| [5] | K nneke M, Bernhard A E, De La Torre J R, Walker C B, Waterbury J B, Stahl D A. Isolation of an autotrophic ammonia-oxidizing marine archaeon. Nature, 2005, 437(7058): 543–546. DOI:10.1038/nature03911 |
| [6] | Leininger S, Urich T, Schloter M, Schwark L, Qi J, Nicol G W, Prosser J I, Schuster S C, Schleper C. Archaea predominate among ammonia-oxidizing prokaryotes in soils. Nature, 2006, 442(7104): 806–809. DOI:10.1038/nature04983 |
| [7] | Tourna M, Freitag T E, Nicol G W, Prosser J I. Growth, activity and temperature responses of ammonia-oxidizing archaea and bacteria in soil microcosms. Environmental Microbiology, 2008, 10(5): 1357–1364. DOI:10.1111/j.1462-2920.2007.01563.x |
| [8] | He J Z, Shen J P, Zhang L M, Zhu Y G, Zheng Y M, Xu M G, Di H J. Quantitative analyses of the abundance and composition of ammonia-oxidizing bacteria and ammonia-oxidizing archaea of a Chinese upland red soil under long-term fertilization practices. Environmental Microbiology, 2007, 9(9): 2364–2374. DOI:10.1111/emi.2007.9.issue-9 |
| [9] | Hastings R C, Butler C, Singleton I, Saunders J R, McCarthy A J. Analysis of ammonia-oxidizing bacteria populations in acid forest soil during conditions of moisture limitation. Letters in Applied Microbiology, 2000, 30(1): 14–18. DOI:10.1046/j.1472-765x.2000.00630.x |
| [10] | Strauss S L, Reardon C L, Mazzola M. The response of ammonia-oxidizer activity and community structure to fertilizer amendment of orchard soils. Soil Biology and Biochemistry, 2014, 68: 410–418. DOI:10.1016/j.soilbio.2013.10.016 |
| [11] | Zhou Z F, Shi X J, Zheng Y, Qin Z X, Xie D T, Li Z L, Guo T. Abundance and community structure of ammonia-oxidizing bacteria and archaea in purple soil under long-term fertilization. European Journal of Soil Biology, 2014, 60: 24–33. DOI:10.1016/j.ejsobi.2013.10.003 |
| [12] | 张苗苗, 王伯仁, 李冬初, 贺纪正, 张丽梅. 长期施加氮肥及氧化钙调节对酸性土壤硝化作用及氨氧化微生物的影响. 生态学报, 2015, 35(19): 6362–6370. |
| [13] | Di H J, Cameron K C, Shen J P, Winefield C S, O'Callaghan M, Bowatte S, He J Z. Ammonia-oxidizing bacteria and archaea grow under contrasting soil nitrogen conditions. FEMS Microbiology Ecology, 2010, 72(3): 386–394. DOI:10.1111/fem.2010.72.issue-3 |
| [14] | Chen X, Zhang L M, Shen J P, Wei W X, He J Z. Abundance and community structure of ammonia-oxidizing archaea and bacteria in an acid paddy soil. Biology and Fertility of Soils, 2011, 47(3): 323–331. DOI:10.1007/s00374-011-0542-8 |
| [15] | Long X E, Chen C R, Xu Z H, Linder S, He J Z. Abundance and community structure of ammonia oxidizing bacteria and archaea in a Sweden boreal forest soil under 19-year fertilization and 12-year warming. Journal of Soils and Sediments, 2012, 12(7): 1124–1133. DOI:10.1007/s11368-012-0532-y |
| [16] | Zhong W H, Bian B Y, Gao N, Min J, Shi W M, Lin X G, Shen W S. Nitrogen fertilization induced changes in ammonia oxidation are attributable mostly to bacteria rather than archaea in greenhouse-based high N input vegetable soil. Soil Biology and Biochemistry, 2016, 93: 150–159. DOI:10.1016/j.soilbio.2015.11.003 |
| [17] | Shen J P, Zhang L M, Zhu Y G, Zhang J B, He J Z. Abundance and composition of ammonia-oxidizing bacteria and ammonia-oxidizing archaea communities of an alkaline sandy loam. Environmental Microbiology, 2008, 10(6): 1601–1611. DOI:10.1111/emi.2008.10.issue-6 |
| [18] | Jia Z J, Conrad R. Bacteria rather than archaea dominate microbial ammonia oxidation in an agricultural soil. Environmental Microbiology, 2009, 11(7): 1658–1671. DOI:10.1111/emi.2009.11.issue-7 |
| [19] | Wessén E, Nyberg K, Jansson J K, Hallin S. Responses of bacterial and archaeal ammonia oxidizers to soil organic and fertilizer amendments under long-term management. Applied Soil Ecology, 2010, 45(3): 193–200. DOI:10.1016/j.apsoil.2010.04.003 |
| [20] | Wang Y, Zhu G B, Song L Y, Wang S Y, Yin C Q. Manure fertilization alters the population of ammonia-oxidizing bacteria rather than ammonia-oxidizing archaea in a paddy soil. Journal of Basic Microbiology, 2014, 54(3): 190–197. DOI:10.1002/jobm.v54.3 |
| [21] | Yao H Y, Gao Y M, Nicol G W, Campbell C D, Prosser J I, Zhang L M, Han W Y, Singh B K. Links between ammonia oxidizer community structure, abundance, and nitrification potential in acidic soils. Applied and Environmental Microbiology, 2011, 77(13): 4618–4625. DOI:10.1128/AEM.00136-11 |
| [22] | Di H J, Cameron K C, Shen J P, Winefield C S, O'Callaghan M, Bowatte S, He J Z. Nitrification driven by bacteria and not archaea in nitrogen-rich grassland soils. Nature Geoscience, 2009, 2(9): 621–624. DOI:10.1038/ngeo613 |
| [23] | Shen J P, Zhang L M, Di H J, He J Z. A review of ammonia-oxidizing bacteria and archaea in Chinese soils. Frontiers in Microbiology, 2012, 3: 296–296. |
| [24] | 莫旭华, 史荣久, 李慧, 郑佳, 王元芬, 徐慧. 华北典型旱地小麦土壤amoA基因的PCR-RFLP分析. 生态科学, 2009, 28(1): 49–55. |
| [25] | Chen Y L, Xu Z W, Hu H W, Hu Y J, Hao Z P, Jiang Y, Chen B D. Responses of ammonia-oxidizing bacteria and archaea to nitrogen fertilization and precipitation increment in a typical temperate steppe in Inner Mongolia. Applied Soil Ecology, 2013, 68: 36–45. DOI:10.1016/j.apsoil.2013.03.006 |
| [26] | Ying J Y, Zhang L M, He J Z. Putative ammonia-oxidizing bacteria and archaea in an acidic red soil with different land utilization patterns. Environmental Microbiology Reports, 2010, 2(2): 304–312. DOI:10.1111/emi4.2010.2.issue-2 |
| [27] | Chu H Y, Fujii T, Morimoto S, Lin X G, Yagi K, Hu J L, Zhang J B. Community structure of ammonia-oxidizing bacteria under long-term application of mineral fertilizer and organic manure in a sandy loam soil. Applied and Environmental Microbiology, 2007, 73(2): 485–491. DOI:10.1128/AEM.01536-06 |
| [28] | 刘桂婷, 程林, 王保莉, 赵其国, 曲东. 长期不同施肥对黄土旱塬黑垆土氨氧化细菌多样性的影响. 中国农业科学, 2010, 43(13): 2706–2714. DOI:10.3864/j.issn.0578-1752.2010.13.010 |
| [29] | Kowalchuk G A, Stephen J R, De Boer W, Prosser J I, Embley T M, Woldendorp J W. Analysis of ammonia-oxidizing bacteria of the beta subdivision of the class Proteobacteria in coastal sand dunes by denaturing gradient gel electrophoresis and sequencing of PCR-amplified 16S ribosomal DNA fragments. Applied and Environmental Microbiology, 1997, 63(4): 1489–1497. |
| [30] | Shen X Y, Zhang L M, Shen J P, Li L H, Yuan C L, He J Z. Nitrogen loading levels affect abundance and composition of soil ammonia oxidizing prokaryotes in semiarid temperate grassland. Journal of Soils and Sediments, 2011, 11(7): 1243–1252. DOI:10.1007/s11368-011-0375-y |
 2017, Vol. 37
2017, Vol. 37