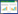文章信息
- 刘姣, 刘长发, 李盛德, 李晋, 陶韦, 李璐瑶, 马悦欣
- LIU Jiao, LIU Changfa, LI Shengde, LI Jin, TAO Wei, LI Luyao, MA Yuexin.
- 翅碱蓬对盐沼沉积物微生物生物量及β-氨氧化细菌群落的影响--以双台河口为例
- Effects of Suaeda heteroptera on microbial biomass and the community structure of β-ammonia-oxidizing bacteria in salt marsh sediments: The case of the Shuangtai estuary
- 生态学报[J]. 2016, 36(24): 8081-8090
- Acta Ecologica Sinica[J]. 2016, 36(24): 8081-8090
- http://dx.doi.org/10.5846/stxb201505070946
-
文章历史
- 收稿日期: 2015-05-07
- 网络出版日期: 2016-04-12
2. 辽宁省高校近岸海洋环境科学与技术重点实验室, 大连 116023;
3. 大连海洋大学理学院, 大连 116023;
4. 盘锦海洋与水产研究所, 盘锦 124010
2. Key Laboratory of Marine Environmental Research of Liaoning Higher Education, Dalian 116023, China;
3. School of Science, Dalian Ocean University, Dalian 116023, China;
4. Institute of Ocean and Fisheries of Panjin, Panjin 124010, China
盐沼在营养转化和滞留中起重要作用。这些生态系统的特点是具有由根贡献大量碳的高净初级生产力[1-2]。总碳从植物根转移到沉积物,包括渗出液、分泌物、溶解产物和粘液,影响根附近土壤微生物群落,影响主要的营养供应和改变沉积物的理化性质[3]。这些有机质的输入支持密集的微生物群落,盐沼植被沉积物微生物集合体的丰度和活力显示植物专一的属性[4]。
Ria de Aveiro盐沼沉积物上不同盐生植物种类调节细菌群落的丰度、垂直分布和组成。沉积物与植物物种相互作用的物理性质(深度、水分含量、粒径分布)调节细菌总细胞丰度[5]。Empordà和Doñana湿地挺水性植物影响其根际细菌16S rRNA基因丰度[6]。辽河三角洲湿地不同植被类型土壤微生物中细菌、放线菌和真菌三大类微生物数量以及土壤微生物生物量碳(MBC)分布都表现为表层(0—10 cm)大于其它两层(10—20 cm和20—30 cm)。包括翅碱蓬在内的八种植被类型土壤中三大类微生物数量以及土壤MBC不同,其中翅碱蓬土壤中放线菌、真菌和MBC含量相对较低[7]。
硝化作用,硝化细菌顺序氧化氨为亚硝酸盐和硝酸盐,在河口和大陆架沉积物它链接有机氮化物的矿化作用和其最终通过反硝化作用或厌氧氨氧化以气态产物去除[8]。在位于Savannah的Skidaway岛的盐沼生态系统研究设施中高互花米草Spartina alterniora沉积物潜在硝化速率比矮互花米草沉积物的高10倍,而无植被沉积物的潜在硝化速率位于两者之间[9]。Lucio del Cangrejo盐沼芦苇Phragmites australis根际潜在硝化速率与无植被沉积物有显著差异[6]。硝化细菌由系统发育和生理上不同氨氧化细菌(AOB)和亚硝酸氧化细菌组成。其中由AOB催化的氨氧化是硝化作用的限制性步骤,因此前人对盐沼硝化细菌的研究主要针对AOB进行[6, 9-10]。高互花米草沉积物AOB氨单加氧酶基因(amoA)丰度比矮互花米草沉积物的高1—2个数量级,也比无植被沉积物的高[9]。Wequetequock-Pawcatuck盐沼高互花米草和狐米草Spartina patens沉积物β-AOB amoA 丰度显著高于矮互花米草[10]。Trias等2012研究表明,Empordà和Doñana湿地挺水性植物影响其根际AOB丰度[6]。植被类型不同程度地影响湿地沉积物AOB群落结构和多样性[11-13]。
双台河口滨海潮滩湿地位于辽宁省盘锦市辽东湾北部,已被列入国际重要湿地名录,拥有大面积的浅海海域、翅碱蓬滩涂、芦苇田和水稻田,是我国高纬度地区最大的滨海河口湿地。目前对辽河口(主要范围位于双台河口国家级自然保护区)芦苇湿地沉积物硝化作用、AOB数量和群落研究表明,芦苇根际效应对硝化作用有促进作用[14]。AOB多样性指数时空分布存在较大差异,盐分、总氮和有机质含量是AOB数量时空分布的重要影响因素[15]。不同盐度胁迫下AOB数量和群落结差异较大[16]。但翅碱蓬Suaeda heteroptera对盐沼微生物生物量及β-氨氧化细菌群落的影响尚未见报道。本试验以双台河口为例,研究翅碱蓬对盐沼沉积物微生物生物量、16S rRNA基因丰度、潜在硝化速率、β-AOB丰度和群落结构的影响,为该生境的生态修复技术提供参考。
1 材料与方法 1.1 样品采集于2013年7月4日、8月14日、9月26日和11月22日,在双台河口潮滩湿地选取3个翅碱蓬生长良好,密度均匀的采样站位,S1(40°55.821′N,121°46.764′E)、S2(40°55.409′N,121°46.886′E)和S3(40°55.573′N,121°46.796′E)(图 1),采集各站位10—15 cm沉积物样品,并在S1和S2附近选取无植被覆盖的裸滩区域,采集同样深度的沉积物样品。采集的样品置于灭菌的自封袋内,低温保存带回实验室。
|
| 图 1 采样站位地理位置图 Fig. 1 Location of sampling sites |
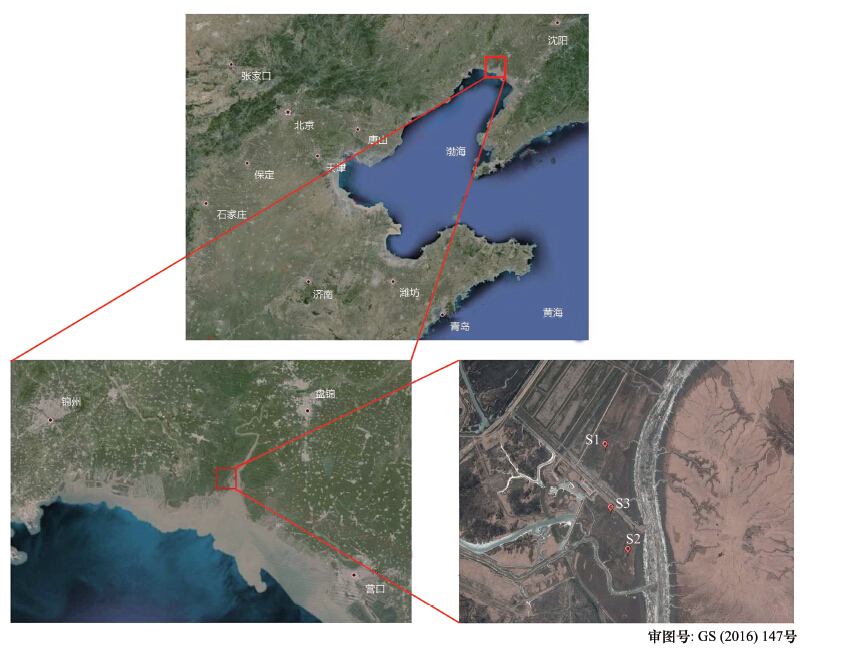
沉积物MBC和MBN测定采用熏蒸提取法[17],其中熏蒸处理为25℃真空条件下培养24 h,提取过程所用浸提液为0.5 mol/L K2SO4溶液(土液比1:2.5)。浸提液中总有机碳的测定采用总有机碳测定仪(TOC VCPH,日本岛津)进行。MBC(mg/kg湿土)和MBN(mg/kg湿土)分别由下式求得:
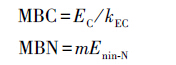
|
式中,Ec为熏蒸与未熏蒸沉积物总有机碳的差值,kEC为转化系数,取0.45;Enin-N为熏蒸与未熏蒸沉积物的茚三酮反应态氮的差值,m为转化系数,取值5.0
1.2.2 细菌16S rRNA基因丰度的定量利用实时荧光定量PCR(qPCR) 测定沉积物细菌16S rRNA基因丰度。使用试剂盒[基因组DNA快速抽提试剂盒(土壤),生工生物工程(上海)股份有限公司]提取沉积物总DNA,由生工生物工程(上海)股份有限公司使用细菌16S rRNA基因通用引物对F357/R518[18]进行PCR扩增,将纯化后的PCR产物克隆到大肠杆菌质粒中,用蓝白平板斑筛选出阳性克隆子,经过夜培养后提取出质粒DNA,作为质粒标准品。质粒标准品的原始浓度为1.34×109 copies/μL,对质粒标准品进行梯度稀释并扩增,20 μL反应体系含Green-2-Go qPCR mastermix (2×) 10 μL,引物(10 μmol/L)各0.5 μL,模板DNA 2 μL,ddH2O 7 μL。扩增程序为95℃酶活化10 min,95℃变性15 s,60℃退火60 s,40个循环。扩增结束后根据各稀释度的已知起始拷贝数及相应Ct值作标准曲线。以沉积物DNA样品为模板进行PCR扩增,扩增条件与标准品相同,根据标准曲线计算样品基因拷贝数。以每克湿土中所含有的16S rRNA基因拷贝数表示沉积物中细菌16S rRNA基因丰度。
1.2.3 潜在硝化速率测定参考Moin等[10]的加铵培养法稍加修改测定沉积物潜在硝化速率。称取约10 g沉积物样品,置于装有100 mL NH4Cl培养液(25 mg N/L,盐度5 PSU)的三角瓶中,用保鲜膜封口,并在上面扎几个孔,放置于恒温振荡器上震荡24 h (25℃,150 r/min),每隔4 h取1次培养液1 mL离心(每次取液后用等量培养液补足),用营养盐全自动分析仪(AutoAnalyzer 3)(德国Bran Luebbe公司)测定上清液中NO2--N(萘乙二胺分光光度法)、NO3--N的含量(镉柱还原-萘乙二胺分光光度法)。依据指数期NO2--N和NO3--N 的增加计算潜在硝化速率。
1.2.4 β-AOB丰度定量采用引物对为amoA-1F/amoA-2R[19]qPCR对β-AOB amoA拷贝数进行测定,以每克湿土中所含有的amoA拷贝数表示沉积物中β-AOB丰度,方法同1.2.2 。
1.2.5 β-AOB群落结构分析采用DGGE对沉积物β-AOB的群落结构进行分析,用β-AOB的16S rRNA引物进行巢式PCR扩增,第1次PCR扩增所用引物对为βAMOf/βAMOr[20]。50 μL反应体系含10×PCR buffer 5 μL,dNTP(2.5 mmol/L) 4 μL,MgCl2(25 mmol/L) 3 μL,引物(10 mmol/L)各2 μL,Taq酶(5 U/μL) 0.5 μL,DNA模板 3 μL,ddH2O补足50 μL。扩增程序为94℃预变性5 min;94℃变性30 s,55℃退火30 s,72℃延伸90 s,30个循环;72℃延伸5 min。第二次PCR扩增上游引物为CTO189f-A/B-GC和CTO189f-C-GC,下游引物为CTO654r[21]。50 μL反应体系与第1次PCR相同。扩增程序为94℃预变性5 min;94℃变性1 min,65℃退火1 min(每个循环降低0.5℃),72℃延伸1 min,20个循环;94℃变性1 min,55℃退火1 min,72℃延伸1 min,10个循环;72℃延伸10 min。将PCR产物上样含45%—55%梯度变性剂的6%聚丙烯酰胺凝胶,在TAE中电泳16 h(温度60℃,电压70 V),然后用Genefinder核酸染料避光染色30 min,凝胶成像仪观察。将优势条带切下,洗脱,用不带GC夹的相同引物进行PCR再扩增,产物送生工生物工程(上海)股份有限公司进行测序。将DGGE条带测序结果在GeneBank中进行BLAST比对,找出最相似序列,用系统发育分析软件包MEGA4.0进行统计和聚类分析[22]。采用邻接法(neighbor-joining method)构建系统发育树,并通过自举分析(bootstrap)进行置信度检测,自举数据集为1000次。采用Quantity One软件对DGGE图谱进行分析,得到每个条带的灰度值,计算β-AOB群落的香农-维纳指数(Shannon-Wiener)多样性指数(H′),H′=-∑Pi×lnPi=-∑(ni/N)×ln(ni/N),Pi代表第i条带的灰度占样品总灰度的比率,ni代表第i条带的灰度值,N代表该泳道的条带总灰度值。
1.3 数据处理用SPSS16.0软件对不同采样日期裸滩和翅碱蓬沉积物微生物生物量、16S rRNA基因丰度、潜在硝化速率和β-AOB丰度数据进行单因素方差分析和Tukey多重比较,对同一采样日期两种生境数据进行独立样本t检验,当P < 0.05时存在显著差异。
2 结果 2.1 MBC和MBN裸滩沉积物MBC随时间没有显著差异,但翅碱蓬沉积物MBC呈现一定的时间动态,8月和9月较7月显著升高(P < 0.05,图 2);所有采样月份翅碱蓬沉积物MBC显著高于裸滩(P < 0.05,图 2)。裸滩沉积物MBN 8月最高,7月和11月较低(P < 0.05,图 3),翅碱蓬沉积物MBN 8和9月显著高于7和11月(P < 0.05,图 3)。当所有采样日期的数据结合分析时,翅碱蓬沉积物MBN显著高于裸滩(图 3,P < 0.05)。

|
| 图 2 沉积物微生物生物量碳 Fig. 2 Microbial biomass carbon in salt marsh sediments 裸滩(0)不同小写字母表示不同采样日期差异显著(P < 0.05),翅碱蓬(A0)不同大写字母表示不同采样日期差异显著( P < 0.05),星号表示同一采样日期两种生境差异显著( P < 0.05) |
|
| 图 3 沉积物微生物生物量氮 Fig. 3 Microbial biomass nitrogen in salt marsh sediments 裸滩(0)不同小写字母表示不同采样日期差异显著(P < 0.05),翅碱蓬(A0)不同大写字母表示不同采样日期差异显著( P < 0.05),星号表示同一采样日期两种生境差异显著( P < 0.05) |
qPCR结果显示,裸滩沉积物16S rRNA基因丰度范围是2.22×109— 4.07×109拷贝/g (湿重),翅碱蓬沉积物16S rRNA基因丰度范围是2.82×109 — 5.57×109 拷贝/g (湿重)(图 4)。两种生境16S rRNA基因丰度随时间变化趋势基本一致,均是7月、8月和9月显著高于11月(图 4,P < 0.05)。当所有采样日期的数据结合分析时,翅碱蓬植被显著影响沉积物细菌16S rRNA基因丰度(图 4,P < 0.05)。
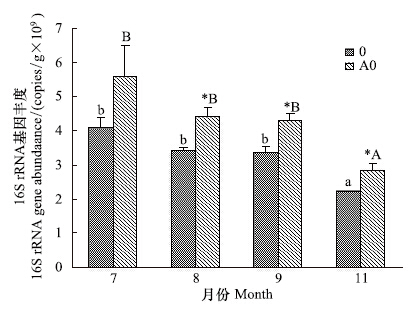
|
| 图 4 沉积物16S rRNA基因丰度 Fig. 4 Abundance of 16S rRNA gene in salt marsh sediments |
裸滩沉积物潜在硝化速率波动范围为0.42—0.59 mgN kg-1 h-1;翅碱蓬沉积物潜在硝化速率波动范围为0.59—0.99 mgN kg-1 h-1(图 5)。两种生境潜在硝化速率随时间没有显著差异。当所有采样日期的数据结合分析时,翅碱蓬沉积物潜在硝化速率显著高于裸滩(图 5,P < 0.05)。
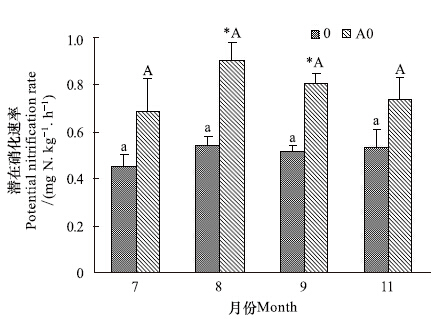
|
| 图 5 沉积物潜在硝化速率 Fig. 5 Potential nitrification rate in salt marsh sediments |
实时荧光定量PCR结果表明,裸滩沉积物β-AOB amoA丰度范围是3.14×105—4.78×105拷贝/g(湿重),8月显著高于9月和11月(图 6,P < 0.05);翅碱蓬地点的丰度范围是5.46×105—7.45 ×105 拷贝/g (湿重),7月和8月略高于9月和11月,但并无显著差异(图 6)。当所有采样日期的数据结合分析时,翅碱蓬沉积物β-AOB amoA丰度显著高于裸滩(图 6,P < 0.05)。
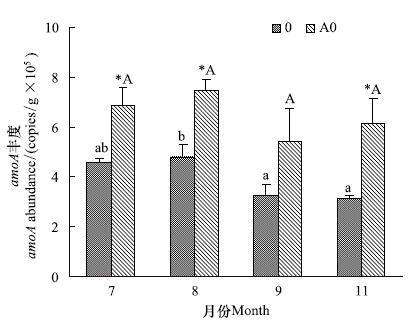
|
| 图 6 沉积物β-AOB amoA 丰度 Fig. 6 Abundance of β-AOB amoA in salt marsh sediments |
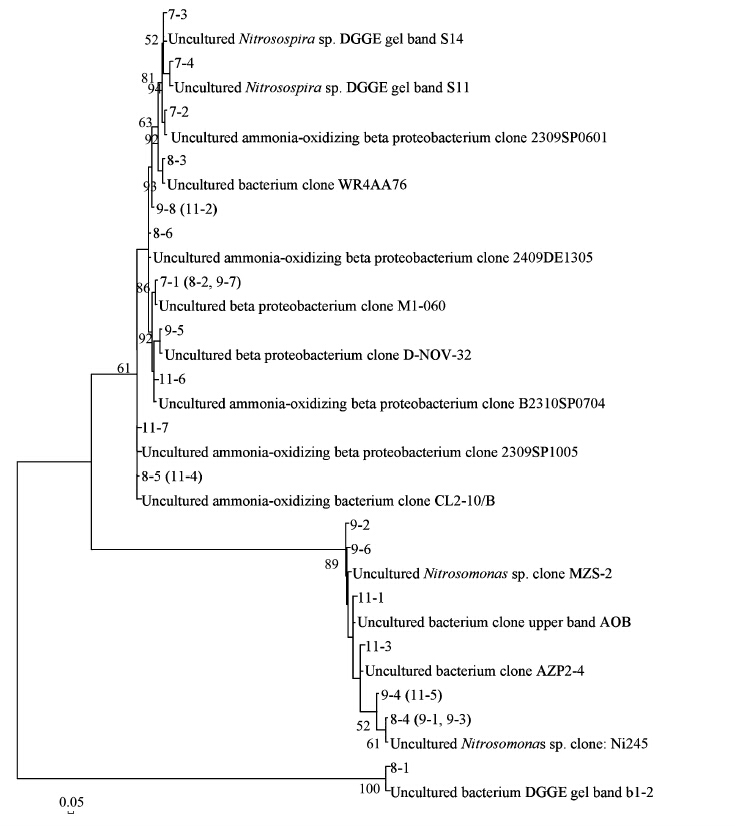
沉积物的DGGE结果见图 7,裸滩沉积物DGGE条带数目为3—5条,翅碱蓬沉积物为3—8条,两种生境β-AOB优势种群组成有一定差异,条带7—4、8—3、9—2、9—6、9—7、11—5、11—6和11—7是有植被沉积物特异性条带。总计对25条DGGE优势条带序列((Genebank登录号:KT259283—KT259305,)进行BLAST分析,将不同月份获得的相似性较高的序列合并构建系统发育树(图 8)。结果表明,7月2条带(7—3和7—4)显示与亚硝化螺菌属Nitrosospira 成员的较高的相似性(98—99%),8月、9月和11月6条带(8—4、9—1、9—3、9—6、9—8和11—2)与未培养亚硝化单胞菌属Nitrosomonas sp.克隆相似性97—99%。其中条带9—6与来源于葡萄牙Douro河河口沉积物尚未培养Nitrosomonas sp.克隆MZS—2相似性97%[23]。两种生境细菌群落的香农维纳多样性指数见表 1,除了8月份,翅碱蓬沉积物细菌群落多样性较高。
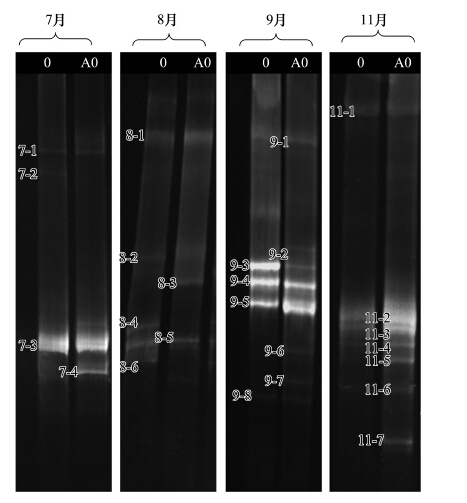
|
| 图 7 巢式PCR扩增CTO片段的DGGE指纹图谱 Fig. 7 DGGE profile of nested PCR-amplified CTO fragments 0-裸滩沉积物,A0-翅碱蓬沉积物 |
|
| 图 8 沉积物β-AOB DGGE序列的系统发育树 Fig. 8 Phylogenetic tree of β-AOB derived from the sequences of the excised DGGE bands in salt sediments |
| 采样日期 Sampling date | 裸滩沉积物(0) Bare mudflat sediment | 翅碱蓬沉积物(A0) Suaeda heteroptera sediment |
| 7月 July | 0.83 | 0.99 |
| 8月 August | 1.60 | 1.09 |
| 9月 September | 1.43 | 1.84 |
| 11月 November | 1.35 | 1.83 |
湿地土壤的微生物生物量碳氮受植被类型的影响,如黄河故道湿地的碱蓬滩地和芦苇沼泽[24]及扎龙湿地芦苇生境和草甸生境的MBC和MBN[25],辽河三角洲不同植被类型土壤(柽柳、草地、芦苇、玉米、森林、碱蓬、翅碱蓬和翅芦苇)的MBC含量均不相同[7]。本研究中翅碱蓬生长区沉积物MBC和MBN显著高于裸滩,说明植物对MBC和MBN有利。翅碱蓬湿地MBC时间变化趋势与盘锦芦苇湿地MBC的基本一致[26]。翅碱蓬沉积物MBC和MBN 7月份较低,8月和9月显著升高,可能与其生长有关,翅碱蓬6—7月份生长最快,需要从土壤中摄取大量营养物质,在一定程度上形成了与微生物间的营养需求竞争,致使微生物可获得营养量降低,限制了微生物的生长和繁殖,8月上旬生长缓慢,8月中下旬开花,此时大量植物根系活动能力增强,使得土壤有机质矿化作用增大,土壤养分充足,根系分泌物丰富,土壤微生物生物量较高。
前人研究表明,qPCR可用于测定海湾和沿海泻湖沉积物样品的细菌16S rRNA基因丰度[27-28]。Empordà 和Doñana湿地挺水性植物(芦苇,川蔓藻Ruppia maritima,流苏菜莨Ruppia cirrhosa和双穗雀稗Paspalum distichum)根际细菌16S rRNA基因丰度高于无植被沉积物[6]。本研究中翅碱蓬沉积物中细菌16S rRNA基因丰度显著高于裸滩,九龙江河口湿地有类似结果,红树林沉积物的16S基因丰度显著高于裸滩[13],说明植物可促进细菌的生长。本试验裸滩沉积物细菌16S rRNA基因丰度范围与Shinji和Nakaumi湖表层沉积物的细菌16S rRNA基因丰度范围一致[28],但高于九龙江河口湿地裸滩(30 cm)沉积物[13],低于胶州湾表层(0—5 cm)沉积物和九龙江河口红树林自然保护区裸滩(0—5 cm和5—20 cm)沉积物[27, 29],其原因之一可能是采样深度的不同,沉积物深度不同,总碳(TC)、总氮(TN)和TC/TN不同,细菌16S rRNA基因丰度不同[29]。
Dollhopf等[9]发现高互花米草沉积物的潜在硝化速率高于裸滩。本研究有类似的结果,翅碱蓬沉积物潜在硝化速率高于裸滩。因为植物根系有助于沉积物充氧[30],可能有助于促进硝化作用[31]。Empordà湿地Basses d′en Coll寡盐泻湖和Doñana国家公园Lucio del Cangrejo 盐沼芦苇根际潜在硝化速率显著高于无植被沉积物[6]。本试验翅碱蓬沉积物的潜在硝化速率高于Wequetequock-Pawcatuck盐沼互花米草及狐米草沉积物(0—2 cm)的潜在硝化速率[10];裸滩沉积物的潜在硝化速率与珠江口表层沉积物的相同[32]。
植被的类型影响湿地沉积物中β-AOB amoA基因的丰度,Wequetequock-Pawcatuck盐沼互花米草及狐米草沉积物(0—2 cm)的β-AOB amoA丰度有明显差异[10]。Trias等[6]研究表明,与无植被沉积物比较,Empordà 和Doñana湿地挺水性植物(芦苇,川蔓藻,流苏菜莨和双穗雀稗)根际AOB丰度增加。Zhang等[29]研究表明,互花米草的入侵影响九龙江河口沉积物(0—5 cm)amoA丰度。珠江河口红树林和芦苇表层湿地沉积物AOB(最大或然数法)数量高于裸地沉积物[32]。本研究翅碱蓬植被显著影响沉积物β-AOB amoA丰度,表明植物有利于AOB的生存。本研究区β-AOB amoA丰度范围与用qPCR测定的其它河口或盐沼表层(0—5 cm)沉积物的β-AOB amoA丰度范围一致[8, 10, 33-37],高于莱州湾河口潮滩表层(0—5 cm)沉积物的β-AOB amoA丰度[38],但低于污染程度较为严重的香港米埔自然保护区河口泥滩沉积物(0—15 cm)的β-AOB amoA丰度[39-40]。
沉积物潜在硝化速率与β-AOB amoA丰度是否显著相关,不同学者研究结果并不一致。有的盐沼沉积物[9]和河口沉积物[35, 41]潜在硝化速率与β-AOB amoA丰度呈显著正相关关系。Wequetequock-Pawcatuck盐沼不同植物生长区沉积物潜在硝化速率与β-AOB amoA丰度相关关系不同,只有高互花米草沉积物两者显著相关,矮互花米草和狐米草沉积物两者之间不相关[10]。其他河口沉积物[34, 42]潜在硝化速率与β-AOB amoA丰度之间没有相关性。本研究中翅碱蓬沉积物和裸滩沉积物两者之间也没有显著相关关系(翅碱蓬沉积物r=0.148,P=0.647;裸滩 r=-0.085,P=0.841)。实际上考虑到潜在硝化速率不代表原位速率和因此不能够准确反映出硝化种群存在是很重要的。也许是潜在速率试验期间的条件(如氧气、铵或盐度)不是对所有硝化细菌都是最适的。在未来的研究中,除了丰度之外,测量基因表达可能有助于更好地量化起作用的种群[10]。
关于植被对湿地沉积物β-AOB群落的影响,不同学者针对不同植被得出的结论并不一致。互花米草入侵对九龙江河口沉积物(0—5 cm和5—20 cm)β-AOB群落组成没有影响[29]。在Scheldt河口潮汐淡水沼泽植物对沉积物(0,5,10,20 cm)β-AOB类群影响较小[11]。Li等[12]研究表明,红树林树木强烈影响米埔自然保护区湿地沉积物(1—2 cm,20—21 cm)β-AOB群落多样性。九龙江河口湿地裸滩沉积物(30 cm)β-AOB多样性高于红树林沉积物[13]。从九龙江河口裸滩沉积物获得序列属于Nitrosospira,而红树林沉积物超过75%的所有细菌amoA序列和所有优势amoA序列属于Nitrosomonas[13]。Wequetequock-Pawcatuck盐沼(0—2 cm)不同植被类型(互花米草及狐米草沉积物)沉积物的β-AOB amoA克隆文库获得的序列属于Nitrosospira[10]。本实验从裸滩和翅碱蓬沉积物获得的序列属于Nitrosospira和Nitrosomonas,翅碱蓬植被对β-AOB群落组成和多样性均有一定的影响。
4 结论不同采样日期裸滩沉积物MBC、翅碱蓬地点的β-AOB amoA丰度和两种生境潜在硝化速率没有显著差异;翅碱蓬沉积物MBC、裸滩沉积物的β-AOB amoA丰度和两种生境MBN及细菌16S rRNA基因丰度呈现时间变化。结合所有采样日期的数据分析表明,翅碱蓬植被显著影响沉积物MBC、MBN、细菌16S rRNA基因丰度、潜在硝化速率和β-AOB amoA丰度;植物对β-AOB 群落组成和多样性均有一定的影响。
| [1] | Schubauer J P, Hopkinson C S. Above-and belowground emergent macrophyte production and turnover in a coastal marsh ecosystem, Georgia. Limnology and Oceanography , 1984, 29 (5) : 1052–1065. DOI:10.4319/lo.1984.29.5.1052 |
| [2] | Mendonça A, Duarte A C, Santos E B H. Spectroscopic properties of sedimentary humic acids from a salt marsh (Ria de Aveiro, Portugal): comparison of sediments colonized by Halimione portulacoides (L.) Aellen and non-vegetated sediments. Biogeochemistry , 2004, 69 (2) : 159–174. DOI:10.1023/B:BIOG.0000031078.38692.62 |
| [3] | Singh B K, Millard P, Whiteley A S, Murrell J C. Unravelling rhizosphere-microbial interactions: opportunities and limitations. Trends in Microbiology , 2004, 12 (8) : 386–393. DOI:10.1016/j.tim.2004.06.008 |
| [4] | Burke D J, Hamerlynck E P, Hahn D. Interactions among plant species and microorganisms in salt marsh sediments. Applied and Environmental Microbiology , 2002, 68 (3) : 1157–1164. DOI:10.1128/AEM.68.3.1157-1164.2002 |
| [5] | Oliveira V, Santos A L, Aguiar C, Santos L, Salvador  C, Gomes N C M, Silva H, Rocha S M, Almeida A, Cunha Â. Prokaryotes in salt marsh sediments of Ria de Aveiro: Effects of halophyte vegetation on abundance and diversity. Estuarine, Coastal and Shelf Science , 2012, 110 : 61–68. DOI:10.1016/j.ecss.2012.03.013 |
| [6] | Trias B, Ruiz-Rueda O, García-Lledó A, Vilar-Sanz A, López-Flores R, Quintana X D, Hallin S, Bañeras L. Emergent macrophytes act selectively on ammonia-oxidizing bacteria and archaea. Applied and Environmental Microbiology , 2012, 78 (17) : 6352–6356. DOI:10.1128/AEM.00919-12 |
| [7] | 赵先丽, 周广胜, 吕国红. 辽河三角洲不同植被类型土壤微生物特征研究. 土壤通报 , 2009, 40 (6) : 1266–1269. |
| [8] | Santoro A E, Francis C A, De Sieyes N R, Boehm A B. Shifts in the relative abundance of ammonia-oxidizing bacteria and archaea across physicochemical gradients in a subterranean estuary. Environmental Microbiology , 2008, 10 (4) : 1068–1079. DOI:10.1111/j.1462-2920.2007.01547.x |
| [9] | Dollhopf S L, Hyun J H, Smith A C, Adams H J, O'Brien S, Kostka J E. Quantification of ammonia-oxidizing bacteria and factors controlling nitrification in salt marsh sediments. Applied and Environmental Microbiology , 2005, 71 (1) : 240–246. DOI:10.1128/AEM.71.1.240-246.2005 |
| [10] | Moin N S, Nelson K A, Bush A, Bernhard A E. Distribution and diversity of archaeal and bacterial ammonia oxidizers in salt marsh sediments. Applied and Environmental Microbiology , 2009, 75 (23) : 7461–7468. DOI:10.1128/AEM.01001-09 |
| [11] | Laanbroek H J, Speksnijder A G C L. Niche separation of ammonia-oxidizing bacteria across a tidal freshwater marsh. Environmental Microbiology , 2008, 10 (11) : 3017–3025. DOI:10.1111/emi.2008.10.issue-11 |
| [12] | Li M, Cao H L, Hong Y G, Gu J D. Spatial distribution and abundances of ammonia-oxidizing archaea (AOA) and ammonia-oxidizing bacteria (AOB) in mangrove sediments. Applied Microbiology and Biotechnology , 2011, 89 (4) : 1243–1254. DOI:10.1007/s00253-010-2929-0 |
| [13] | Luo Z X, Qiu Z Z, Wei Q S, Du Laing G, Zhao Y L, Yan C Z. Dynamics of ammonia-oxidizing archaea and bacteria in relation to nitrification along simulated dissolved oxygen gradient in sediment-water interface of the Jiulong river estuarine wetland, China. Environmental Earth Sciences , 2014, 72 (7) : 2225–2237. DOI:10.1007/s12665-014-3128-6 |
| [14] | 白洁, 陈春涛, 赵阳国, 田伟君, 董晓, 尹宁宁. 辽河口湿地沉积物硝化细菌及硝化作用研究. 环境科学 , 2010, 31 (12) : 3011–3017. |
| [15] | 白洁, 董晓, 赵阳国. 辽河口芦苇湿地土壤氨氧化菌的时空变化. 中国环境科学 , 2011, 31 (11) : 1870–1874. |
| [16] | 钟丽华, 董晓, 白洁, 王妍, 赵阳国. 辽河口芦苇湿地氨氧化菌群落结构与土壤盐分的关系. 中国海洋大学学报 , 2013, 43 (4) : 94–99. |
| [17] | 吴金水, 林启美, 黄巧云, 肖和艾. 土壤微生物生物量测定方法及其应用. 北京: 气象出版社, 200654–74. |
| [18] | Muyzer G, de Waal E C, Uitterlinden A G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA. Applied and Environmental Microbiology , 1993, 59 (3) : 695–700. |
| [19] | Rotthauwe J H, Witzel K P, Liesack W. The ammonia monooxygenase structural gene amoA as a functional marker: Molecular fine-scale analysis of natural ammonia-oxidizing populations. Applied and Environmental Microbiology , 1997, 63 (12) : 4704–4712. |
| [20] | McCaig A E, Embley T M, Prosser J I. Molecular analysis of enrichment cultures of marine ammonia oxidisers. FEMS Microbiology Letters , 1994, 120 (3) : 363–367. DOI:10.1111/fml.1994.120.issue-3 |
| [21] | Kowalchuk G A, Stephen J R, De Boer W, Prosser J I, Embley T M, Woldendorp J W. Analysis of ammonia-oxidizing bacteria of the beta subdivision of the class Proteobacteria in coastal sand dunes by denaturing gradient gel electrophoresis and sequencing of PCR-amplified 16S ribosomal DNA fragments. Applied and Environmental Microbiology , 1997, 63 (4) : 1489–1497. |
| [22] | Tamura K, Dudley J, Nei M, Kumar S. MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) Software Version 4. 0. Molecular Biology and Evolution , 2007, 24 (8) : 1596–1599. DOI:10.1093/molbev/msm092 |
| [23] | Magalhães C, Bano N, Wiebe W J, Hollibaugh J T, Bordalo A A. Composition and activity of beta-Proteobacteria ammonia-oxidizing communities associated with intertidal rocky biofilms and sediments of the Douro River estuary, Portugal. Journal of Applied Microbiology , 2007, 103 (4) : 1239–1250. DOI:10.1111/jam.2007.103.issue-4 |
| [24] | 朱新玉. 黄河故道湿地土壤质量因子与景观类型的耦合关系. 资源科学 , 2015, 37 (1) : 85–93. |
| [25] | 张静, 马玲, 丁新华, 陈旭日, 马伟. 扎龙湿地不同生境土壤微生物生物量碳氮的季节变化. 生态学报 , 2014, 34 (13) : 3712–3719. |
| [26] | 赵先丽, 周广胜, 周莉, 吕国红, 贾庆宇, 谢艳兵. 盘锦芦苇湿地土壤微生物生物量C的季节动态. 土壤通报 , 2008, 39 (1) : 43–46. |
| [27] | Dang H Y, Li J, Chen R P, Wang L, Guo L Z, Zhang Z N, Klotz M G. Diversity, abundance, and spatial distribution of sediment ammonia-oxidizing betaproteobacteria in response to environmental gradients and coastal eutrophication in Jiaozhou Bay, China. Applied and Environmental Microbiology , 2010, 76 (14) : 4691–4702. DOI:10.1128/AEM.02563-09 |
| [28] | Tsuboi S, Amemiya T, Seto K, Itoh K, Rajendran N. The ecological roles of bacterial populations in the surface sediments of coastal lagoon environments in Japan as revealed by quantification and qualification of 16S rDNA. World Journal of Microbiology and Biotechnology , 2013, 29 (5) : 759–774. DOI:10.1007/s11274-012-1231-y |
| [29] | Zhang Q F, Peng J J, Chen Q, Li X F, Xu C Y, Yin H B, Yu S. Impacts of Spartina alterniflora invasion on abundance and composition of ammonia oxidizers in estuarine sediment. Journal of Soils and Sediments , 2011, 11 (6) : 1020–1031. DOI:10.1007/s11368-011-0369-9 |
| [30] | Mendelssohn I A, McKee K L, Patrick W H Jr. Oxygen deficiency in Spartina alterniflora roots: metabolic adaptation to anoxia. Science , 1981, 214 (4519) : 439–441. DOI:10.1126/science.214.4519.439 |
| [31] | An S, Joye S B. Enhancement of coupled nitrification-denitrification by benthic photosynthesis in shallow estuarine sediments. Limnology and Oceanography , 2001, 46 (1) : 62–74. DOI:10.4319/lo.2001.46.1.0062 |
| [32] | 王玉萍, 王立立, 李取生, 叶妹. 珠江河口湿地沉积物硝化作用强度及影响因素研究. 生态科学 , 2012, 31 (3) : 330–334. |
| [33] | Mosier A C, Francis C A. Relative abundance and diversity of ammonia-oxidizing archaea and bacteria in the San Francisco Bay estuary. Environmental Microbiology , 2008, 10 (11) : 3002–3016. DOI:10.1111/emi.2008.10.issue-11 |
| [34] | Zheng Y L, Hou L J, Newell S, Liu M, Zhou J L, Zhao H, You L L, Cheng X L. Community dynamics and activity of ammonia-oxidizing prokaryotes in intertidal sediments of the Yangtze estuary. Applied and Environmental Microbiology , 2014, 80 (1) : 408–419. DOI:10.1128/AEM.03035-13 |
| [35] | Bernhard A E, Tucker J, Giblin A E, Stahl D A. Functionally distinct communities of ammonia-oxidizing bacteria along an estuarine salinity gradient. Environmental Microbiology , 2007, 9 (6) : 1439–1447. DOI:10.1111/emi.2007.9.issue-6 |
| [36] | Cao H L, Hong Y G, Li M, Gu J D. Diversity and abundance of ammonia-oxidizing prokaryotes in sediments from the coastal Pearl River estuary to the South China Sea. Antonie van Leeuwenhoek , 2011, 100 (4) : 545–556. DOI:10.1007/s10482-011-9610-1 |
| [37] | Jin T, Zhang T, Ye L, Lee O O, Wong Y H, Qian P Y. Diversity and quantity of ammonia-oxidizing archaea and bacteria in sediment of the Pearl River Estuary, China. Applied Microbiology and Biotechnology , 2011, 90 (3) : 1137–1145. DOI:10.1007/s00253-011-3107-8 |
| [38] | Zhang X L, Agogué H, Dupuy C, Gong J. Relative abundance of ammonia oxidizers, denitrifiers, and anammox bacteria in sediments of hyper-nutrified estuarine tidal flats and in relation to environmental conditions. Clean-Soil, Air, Water , 2014, 42 (6) : 815–823. DOI:10.1002/clen.v42.6 |
| [39] | Cao H L, Li M, Hong Y G, Gu J D. Diversity and abundance of ammonia-oxidizing archaea and bacteria in polluted mangrove sediment. Systematic and Applied Microbiology , 2011, 34 (7) : 513–523. DOI:10.1016/j.syapm.2010.11.023 |
| [40] | Wang Y F, Feng Y Y, Ma X J, Gu J D. Seasonal dynamics of ammonia/ammonium-oxidizing prokaryotes in oxic and anoxic wetland sediments of subtropical coastal mangrove. Applied Microbiology and Biotechnology , 2013, 97 (17) : 7919–7934. DOI:10.1007/s00253-012-4510-5 |
| [41] | Bernhard A E, Landry Z C, Blevins A, de la Torre J R, Giblin A E, Stahl D A. Abundance of ammonia-oxidizing archaea and bacteria along an estuarine salinity gradient in relation to potential nitrification rates. Applied and Environmental Microbiology , 2010, 76 (4) : 1285–1289. DOI:10.1128/AEM.02018-09 |
| [42] | Caffrey J M, Bano N, Kalanetra K, Hollibaugh J T. Ammonia oxidation and ammonia-oxidizing bacteria and archaea from estuaries with differing histories of hypoxia. The ISME Journal , 2007, 1 (7) : 660–662. DOI:10.1038/ismej.2007.79 |
 2016, Vol. 36
2016, Vol. 36