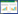文章信息
- 宋飏, 黄原
- SONG Yang, HUANG Yuan.
- DNA复合条形码在太白山土壤动物多样性研究中的应用
- The application of DNA metabarcoding in the study of soil animal diversity in Taibai Mountain
- 生态学报[J]. 2016, 36(14): 4531-4539
- Acta Ecologica Sinica[J]. 2016, 36(14): 4531-4539
- http://dx.doi.org/10.5846/stxb201412122475
-
文章历史
- 收稿日期: 2014-12-12
- 网络出版日期: 2015-10-30
DNA复合条形码技术(metabarcoding)是一种结合了DNA条形码技术和高通量测序技术的快速鉴定混合样本中物种的方法[1-3]。DNA条形码技术通过一段短的DNA序列来鉴定物种,由Hebert等人于2003年提出[4]。传统的Sanger测序只能对单一的DNA分子进行测序,而高通量测序技术可以分别对混合DNA中的每一条DNA分子进行测序。两种技术的结合运用可以获得混合样本中大量的含有分类学信息的DNA片段的序列,然后将这些序列与合适的数据库比对即可确定其代表的类群。随着高通量测序技术的持续发展与分析软件的不断完善,在分析生物多样性时,DNA复合条形码技术可以作为代替传统方法的一个选择[2-3]。
DNA复合条形码技术已普遍使用于微生物群落的研究,并且正越来越多的应用到动植物多样性研究中[1, 5]。在土壤动物方面:Douglas等人构建了7个节肢动物群落,通过对这些节肢动物群落的DNA复合条形码研究证明了该方法可以精确估计群落内的系统发育多样性和群落间的差异性[2];Bienert等人对2个不同地点的土壤样品进行了研究,发现这一方法能很好地区分这2个地点的蚯蚓群落组成,并且与使用传统方法得到的结果一致[6];Yang等人对基于该技术分析土壤小型动物多样性的3种数据处理流程进行了分析比较,结果表明3种流程的处理结果基本一致[7];魏攀利用该技术对小五台山国家级自然保护区1m2草地中无脊椎动物多样性进行了研究,共调查得到125种无脊椎动物[8];Yang等人使用该方法对采自土壤和落叶层的样本与采自地上的样本进行了比较,结果表明,在研究生物多样性格局时,两类样本可以得到相似的生态学信息[9]。此外,还有许多关于土壤线虫的研究[10-12]。
目前, 科学界已经认识到土壤是地球上生物多样性最丰富的生境之一[13]。虽然土壤生物多样性的研究已有很长时间,但是,关于土壤生物多样性本身的很多基础科学问题仍然不清楚。从某种意义上说,对土壤生物多样性的研究还处于初期阶段[14]。
我国对土壤动物的研究主要集中在东北、亚热带地区、热带雨林地区以及新疆地区,对于中西部地区的土壤动物研究还很少[15-16]。太白山为秦岭主峰,位于动物古北界与东洋界的交汇和过渡地带,是华北、华中和横断山脉3个植物区系的交汇处,自然植被带完整,生态系统复杂多样,生物资源丰富,在海拔3400m以上存有完整的第四纪古冰川遗迹,是一个保存完好的自然生态系统,是我国生物多样性保护的关键区域[17]。
本文选取18S核糖体小亚基RNA(18S rRNA)基因和细胞色素C氧化酶亚基I(COI)基因的部分序列作为分子标记,应用DNA复合条形码技术对秦岭太白山地区5种不同生境(高山草甸,针叶林,落叶小叶林,落叶阔叶林,农田)的中小型土壤动物多样进行研究,并讨论该方法目前应用于中小型土壤动物研究的优缺,为进一步探讨秦岭中小型土壤动物多样性格局以及秦岭生物多样性保护提供科学依据。
1 研究方法 1.1 样本的采集和分离于2014年6月上旬在太白山自然保护区及周围选取高山草甸、针叶林、落叶小叶林、落叶阔叶林和农田5种生境进行取样。每种生境选取10m×10m的3个样方,在每个样方内随机采取3份土样,采样深度为0—15cm。将土样带回实验室,用Tullgren漏斗进行分离(40W白炽灯分离48 h)。将收集到的土壤动物保存在100%乙醇中,用于总DNA的提取。样地详细信息见表 1。
| 生境 Habitat |
采样地点 Sampling position |
海拔 Elevation/m |
备注 Remark |
| 高山草甸Alpine meadow | 33.95785°N, 107.76079°E | 3604 | 高山草甸 |
| 33.95935°N, 107.76438°E | 3598 | 高山草甸 | |
| 33.96813°N, 107.77204°E | 3602 | 高山草甸 | |
| 针叶林Coniferous forest | 33.97859°N, 107.76949°E | 3334 | 太白红杉林 |
| 33.98616°N, 107.75208°E | 3066 | 巴山冷杉林 | |
| 33.99572°N, 107.73326°E | 2904 | 巴山冷杉林 | |
| 落叶小叶林 | 34.01117°N, 107.72562°E | 2726 | 糙皮桦林 |
| Deciduous small-leaved forest | 34.01562°N, 107.71668°E | 2742 | 糙皮桦林 |
| 34.01762°N, 107.70982°E | 2517 | 红桦林 | |
| 落叶阔叶林 | 34.08043°N, 107.69726°E | 1326 | 锐齿栎林 |
| Deciduous broad-leaved forest | 过仙桥附近* | - | 锐齿栎林 |
| 34.08844°N, 107.68707°E | 1171 | 栓皮栎林 | |
| 农田Farmland | 34.88408°N, 107.68707°E | 1171 | 玉米地 |
| 34.04218°N, 107.34136°E | 1169 | 秦艽田 | |
| 34.09689°N, 107.65855°E | 856 | 麦田 | |
| *采样时该地点GPS无法定位 | |||
将一个样方分离到的所有土壤动物混合放入一个1.5mL离心管,使用血液/细胞/组织基因组DNA提取试剂盒(天根生化科技有限公司)进行总DNA提取,得到15份总DNA。将每种生境的3份总DNA进行等浓度混合,得到用于后续PCR的5份总DNA。
1.3 PCR及测序PCR反应体系为25μL,包括2×Taq PCR StarMix with Loading Dye 12.5μL,灭菌超纯水9.5μL,正向引物(10μmol/L)1.0μL,反向引物(10μmol/L)1.0μL,总DNA 1.0μL。
18S rRNA基因部分序列(大约350bp)的扩增使用正向引物#3:5′-GYGGTGCATGGCCGTTSKTRGTT-3′和反向引物#5_RC:5′-GTGTGYACAAAGGBCAGGGAC-3′[18]。为了同时对多个样本进行测序,在每个样本的正向引物5′端加上了7bp的barcode序列,用于后续分析中区分序列来自哪个样本。PCR的循环程序为:95℃10min;10个循环:95℃10s,57℃30s(每个循环降低0.5℃),72℃60s;30个循环:95℃10s,55℃30s,75℃60s;最后在72℃下延长5min。
COI基因部分序列(大约313bp)的扩增使用正向引物mlCOlintF:5′-GGWACWGGWTGAACWGT WTAYCCYCC-3′[19]和反向引物jgHCO2198:5′-TANACYTCNGGRTGNCCRAARAAYCA-3′[20](用N替换了原序列中的I)。同样在每个样本的正向引物5′端加上了7bp的barcode序列。PCR的循环程序为:16个循环:95℃10s,62℃30s(每个循环降低1℃),72℃60s;25个循环:95℃10s,46℃30s,72℃30s;最后在72℃下延长5min。
每份总DNA扩增3次,然后将3份PCR产物等浓度混合。PCR产物的测序使用Illumina MiSeq平台,采用250PE测序策略,由上海派森诺生物科技有限公司完成。
1.4 数据处理 1.4.1 原始数据处理步长为1的5bp窗口从序列起始位置开始滑动,要求窗口中碱基平均质量≥Q20,低于该值则截断序列。然后使用FLASH(http://ccb.jhu.edu/software/FLASH/)对通过质量过滤的序列进行拼接。使用Qiime[21]对拼接后的序列进一步过滤,舍弃含有N碱基、长度< 150bp、barcode有错配和引物错配碱基数> 1的序列。使用mothur[22]去除嵌合体序列,得到用于后续分析的序列。
1.4.2 OTU聚类及注释使用Qiime将序列按相似度0.97聚类为OTU(Operational Taxonomic Unit)[23],选取每个OTU最长的序列作为代表序列。将18S rRNA基因和COI基因的OTU代表序列分别与Silva[24]数据库(115版)和BOLD[25]数据库(截止到Release 5.00-v1)进行比对,获得每个OTU的分类学信息。使用两个标记的OTU列表生成物种列表,然后合并,删去不属于土壤动物的类群。使用Excel绘制目水平上的群落组成百分比堆积图。使用R软件VennDiagram程序包绘制Venn图。
1.4.3 α多样性分析使用R软件vegan程序包对各样本的Chao1丰富度指数,ACE丰富度指数,Shannon-Wiener多样性指数和Simpson多样性指数进行计算。
1.4.4 群落相似性分析使用R软件vegan程序包对各样本之间的Bray-Curtis距离进行计算,然后使用ape程序包基于Bray-Curtis距离对样本进行主坐标分析(Principal Coordinates Analysis; PCoA)。
1.4.5 聚类分析根据物种列表,使用R软件pheatmap程序包将科水平上的分类信息进行聚类分析并绘制Heatmap图。
2 结果 2.1 原始数据处理与OTU聚类经最初的质量过滤与拼接后得到611959序列,其中18S rRNA基因309167条,COI基因302792条。经Qiime与mothur进一步过滤后得到用于后续分析的序列,共437313条,其中18S rRNA基因211436条,COI基因225877条。合格序列聚类后,18S rRNA基因共得到1653个OTU,COI基因共得到2138个OTU。
2.2 种类组成本文的土壤动物分类参照《中国土壤动物检索图鉴》[26]。调查共鉴定到土壤动物3门9纲28目199科,其中18S rRNA基因鉴定到2门8纲23目101科,COI基因鉴定到3门7纲16目128科,两者共同鉴定到2门6纲12目31科。高山草甸包含21目136科,针叶林包含20目141科,落叶小叶林包含21目122科,落叶阔叶林包含21目138科,农田包含23目132科。各样本所包含的类群及各类群在各样本中所含序列条数见附表。
对各样本做Venn图(图 1)可得,各样本在科水平共有的类群数为84,在高山草甸、针叶林、落叶小叶林、落叶阔叶林、农田各自总类群数中所占比例分别为61.76%、59.57%、68.85%、60.87%、63.64%。高山草甸独有的类群数为9,占其总类群数6.62%;针叶林独有的类群数为13,占其总类群数9.22%;落叶小叶林独有的类群数为7,占其总类群数5.74%;落叶阔叶林独有的类群数为10,占其总类群数7.25%;农田独有的类群数为7,占其总类群数5.30%。
|
| 图 1 各样本在科水平的Venn图 Fig. 1 Venn diagram for families of samples |
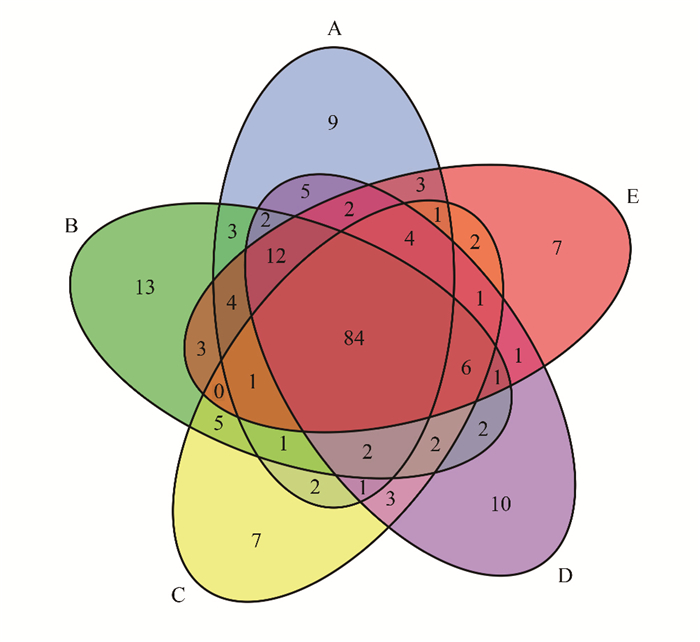
如图 2所示,在目水平上,真螨目、弹尾目和蜘蛛目的丰度在各个样本中都较高,其中蜘蛛目在落叶小叶林中所占比例达64.62%。鳞翅目在高山草甸、针叶林和农田中的丰度较高,而在落叶小叶林和阔叶林中丰度较低。半翅目在高山草甸、针叶林和落叶小叶林中丰度较高,而在落叶阔叶林和农田中丰度较低。双尾目在落叶阔叶林中所占比例达61.04%,而在其他生境中含量较低。条马陆目在针叶林中含量较高,鞘翅目在农田中含量较高。其余类群在各生境中含量均较低。
|
| 图 2 目水平上各类群的序列丰度在各样本中所占的百分比 Fig. 2 Percentage of sequence aboundance of taxa for order in each sanple |
各生境的土壤动物群落Chao1丰富度指数为:针叶林>落叶小叶林>落叶阔叶林>高山草甸>农田;ACE丰富度指数为:针叶林>落叶阔叶林>高山草甸>落叶小叶林>农田;Shannon-Wiener多样性指数为:针叶林>高山草甸>农田>落叶阔叶林>落叶小叶林;Simpson多样性指数为:针叶林>高山草甸>农田>落叶阔叶林>落叶小叶林。各生境多样性指数见表 2。
| 生境 Habitat |
Chao1指数 Chao1 index |
ACE指数 ACE index |
Shannon-Wiener指数 Shannon-Wiener index |
Simpson指数 Simpson index |
| 高山草甸Alpine meadow | 151.33 | 157.90 | 2.63 | 0.87 |
| 针叶林Coniferous forest | 174.83 | 165.70 | 2.81 | 0.89 |
| 落叶小叶林Deciduous small-leaved forest | 162.60 | 149.29 | 1.67 | 0.57 |
| 落叶阔叶林Deciduous broad-leaved forest | 157.71 | 161.09 | 1.92 | 0.62 |
| 农田Farmland | 145.57 | 146.03 | 2.52 | 0.84 |
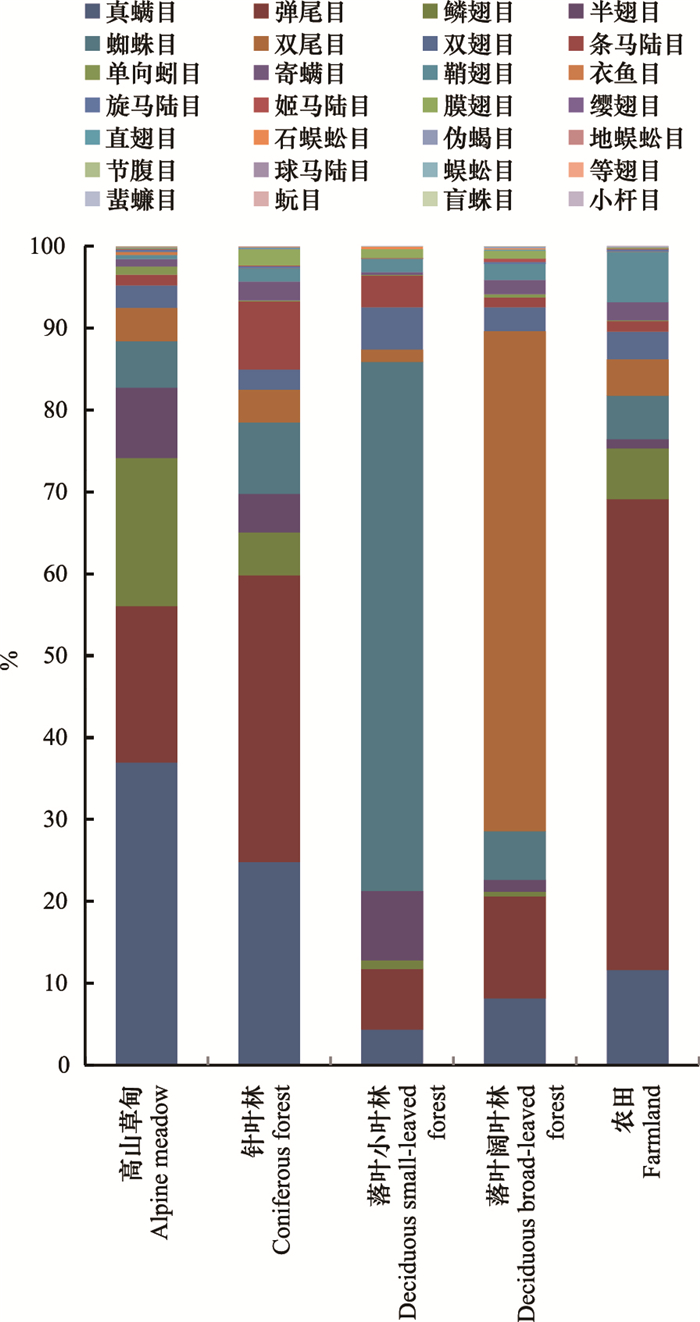
Bray-Curtis距离可以作为衡量群落间相似性的指标,Bray-Curtis距离越小,表示两个样本的群落组成越相似。表 3为各样本之间的Bray-Curtis距离。根据各样本之间的Bray-Curtis距离,可以做PCoA分析。样本的相似性越高,它们在PCoA图中的距离就越近。图 3为根据各样本间的Bray-Curtis距离,选取贡献率最大的主坐标组合所做的PCoA图。图 3显示土壤动物群落组成在高山草甸、针叶林、农田3种生境中的相似性较高,落叶小叶林和落叶阔叶林的土壤动物群落组成与这3种生境的相似性较低,落叶小叶林与落叶阔叶林之间的土壤动物群落组成相似性也较低。
| 样本 Sample |
高山草甸 Alpine meadow |
针叶林 Coniferous forest |
落叶小叶林 Deciduous small- leaved forest |
落叶阔叶林 Deciduous broad- leaved forest |
| 针叶林Coniferous forest | 0.4030 | |||
| 落叶小叶林Deciduous small-leaved forest | 0.7639 | 0.6910 | ||
| 落叶阔叶林Deciduous broad-leaved forest | 0.6550 | 0.6169 | 0.7784 | |
| 农田Farmland | 0.5268 | 0.4367 | 0.7602 | 0.6449 |
|
| 图 3 PCoA分析 Fig. 3 PCoA analysis |
Heatmap图可以直观地将数据值的大小以颜色的深浅表示出来,同时可根据需要将数据进行物种或样本间的相似性聚类。图 4为使用欧式距离和最长聚类法所作的Heatmap图,展现了科水平各类群在各样本中的丰度分布,同时也在科水平对各样本进行了聚类。可以看到,高山草甸与针叶林首先聚类,然后与农田聚类,然后与落叶阔叶林聚类,最后与落叶小叶林聚类。

|
| 图 4 聚类分析Heatmap图 Fig. 4 Heatmap of cluster analysis |
从类群列表来看,两个分子标记鉴定到的类群没有完全重叠,两者共同鉴定到的类群只有31科。而18S rRNA基因单独鉴定到70科,COI基因单独鉴定到97科,远大于它们共同鉴定到的类群数。即使是在目水平上,两者鉴定到的类群也有很大的区别。这是由于不同分子标记的通用引物在不同类群中的扩增能力不同与数据库的覆盖度不同造成的[18-19, 27]。因此单独使用一种分子标记来鉴定样本中含有的类群会造成对类群数的低估,同时使用多种分子标记可以更全面的鉴定样本中所含类群[10, 28]。考虑到数据库的可用性,对于后生动物来说,较好的分子标记有COI、18S rRNA和28S rRNA基因[18, 25, 29-30]。
尽管采样地点的海拔跨度高达2500m以上,生境类型发生了剧烈的变化,但从各样本群落组成来看,在目水平,真螨目、弹尾目和蜘蛛目在各生境中所占的比例都较大,在科水平,5种生境的共有类群达84种,在各生境类群数中所占比例大多超过60%(在针叶林中为59.57%),这表明太白山中小型土壤动物中大多数类群为广布类群。另外,各生境均含有类群数低于10%的特有类群,表明海拔与生境的变化会对太白山中小型土壤动物的群落组成造成一定的影响。这与以往的研究结果一致[31-33]。
在α多样性分析中,虽然Chao1指数与ACE指数同为丰富度指数,但它们的排序结果却有细微的差别,落叶小叶林中的土壤动物群落Chao1指数排在落叶阔叶林和高山草甸之前,而ACE指数却排在了落叶阔叶林和高山草甸之后。这是因为ACE指数的计算公式中加入了一个定义“优势”类群的阈值,这个阈值在vegan程序包中为类群包含10条序列,而Chao1指数并没有这个规则[34]。因此可以推断落叶小叶林土壤动物群落中包含较多的低丰度类群(所含序列数< 10)。在DNA复合条形码技术中,Shannon-Wiener和Simpson多样性指数是基于一个类群所含序列数计算的,而不是基于一个类群的个体数。Carew等人[28]指出,在一个样本中,一个物种的序列数和个体数是正相关的,但序列的频率并不能完美的反映物种的频率。Piñol等人[35]的研究也表明该技术的定量潜力有限,只有定性的研究是可靠的。因此,Shannon-Wiener和Simpson多样性指数仅能作为一个参考,不能通过这两个指数很好的判断土壤动物群落的多样性。定量问题是目前该技术用于土壤动物研究的局限性。
从各样本之间的Bray-Curtis距离与PCoA图来看,针叶林与高山草甸的土壤动物群落组成最为相似,针叶林与农田次之。针叶林、高山草甸与农田三者的土壤动物群落组成相似性较高,而落叶小叶林和落叶阔叶林的土壤动物群落组成与这3种生境的相似性都较低,落叶小叶林和落叶阔叶林的土壤动物群落组成相似性也较低。聚类分析得到了与PCoA分析相符的结果。这种相似性的形成可能与各生境中的气温和土壤的理化条件相关[32],有待后续的实验。
根据以往的土壤动物研究可知,在一次调查中会得到成千上万的土壤动物,且土壤动物包含的类群很多,鉴定这些土壤动物需要多人长时间作业。对于其它的环境样本,也存在同样的问题。而通过DNA复合条形码技术,则可以快速简便的完成大量样本的分类鉴定,与传统方法相比,省时省力,这也是该技术的优势所在。随着高通量测序技术的不断发展和分析方法的不断完善,DNA复合条形码技术的高成本效益和全面性将使更容易的大尺度监测生物多样性趋势成为可能[36-37]。
| [1] | Coissac E, Riaz T, Puillandre N. Bioinformatic challenges for DNA metabarcoding of plants and animals[J]. Molecular Ecology , 2012, 21 (8) : 1834–1847. DOI:10.1111/j.1365-294X.2012.05550.x |
| [2] | Yu D W, Ji Y, Emerson B C, Wang X Y, Ye C X, Yang C Y, Ding Z L. Biodiversity soup:metabarcoding of arthropods for rapid biodiversity assessment and biomonitoring[J]. Methods in Ecology and Evolution , 2012, 3 (4) : 613–623. DOI:10.1111/mee3.2012.3.issue-4 |
| [3] | Ji Y Q, Ashton L, Pedley S M, Edwards D P, Tang Y, Nakamura A, Kitching R, Dolman P M, Woodcock P, Edwards F A, Larsen T H, Hsu W W, Benedick S, Hamer K C, Wilcove D S, Bruce C, Wang X Y, Levi T, Lott M, Emerson B C, Yu D W. Reliable, verifiable and efficient monitoring of biodiversity via metabarcoding[J]. Ecology Letters , 2013, 16 (10) : 1245–1257. DOI:10.1111/ele.12162 |
| [4] | Hebert P D N, Cywinska A, Ball S L, deWaard J R. Biological identifications through DNA barcodes[J]. Proceedings of the Royal Society of London B:Biological Sciences , 2003, 270 (1512) : 313–321. DOI:10.1098/rspb.2002.2218 |
| [5] | 马兰. DNA微型条形码和复合条形码研究进展[J]. 农业灾害研究 , 2013, 3 (6) : 58–60. |
| [6] | Bienert F, De Danieli S, Miquel C, Coissac E, Poillot C, Brun J J, Taberlet P. Tracking earthworm communities from soil DNA[J]. Molecular Ecology , 2012, 21 (8) : 2017–2030. DOI:10.1111/j.1365-294X.2011.05407.x |
| [7] | Yang C X, Ji Y Q, Wang X Y, Yang C Y, Yu D W. Testing three pipelines for 18S rDNA-based metabarcoding of soil faunal diversity[J]. Science China Life Sciences , 2013, 56 (1) : 73–81. DOI:10.1007/s11427-012-4423-7 |
| [8] | 魏攀.宏条形码研究小五台山国家自然保护区一平方米草地中无脊椎动物多样性[D].北京:中国科学院大学, 2013. |
| [9] | Yang C X, Wang X Y, Miller J A, Blécourt M D, Ji Y Q, Yang C Y, Harrison R D, Yu D W. Using metabarcoding to ask if easily collected soil and leaf-litter samples can be used as a general biodiversity indicator[J]. Ecological Indicators , 2014, 46 : 379–389. DOI:10.1016/j.ecolind.2014.06.028 |
| [10] | Porazinska D L, Giblin-Davis R M, Faller L, Farmerie W, Kanzaki N, Morris K, Powers T O, Tucker A E, Sung W, Thomas W K. Evaluating high-throughput sequencing as a method for metagenomic analysis of nematode diversity[J]. Molecular Ecology Resources , 2009, 9 (6) : 1439–1450. DOI:10.1111/men.2009.9.issue-6 |
| [11] | Porazinska D L, Sung W, Giblin-Davis R M, Thomas W K. Reproducibility of read numbers in high-throughput sequencing analysis of nematode community composition and structure[J]. Molecular Ecology Resources , 2010, 10 (4) : 666–676. |
| [12] | Darby B J, Todd T C, Herman M A. High-throughput amplicon sequencing of rRNA genes requires a copy number correction to accurately reflect the effects of management practices on soil nematode community structure[J]. Molecular Ecology , 2013, 22 (21) : 5456–5471. DOI:10.1111/mec.12480 |
| [13] | Bardgett R D. The Biology of Soil:A Community and Ecosystem Approach. Oxford: Oxford University Press, 2005. |
| [14] | 时雷雷, 傅声雷. 土壤生物多样性研究:历史、现状与挑战[J]. 科学通报 , 2014, 59 (6) : 493–509. |
| [15] | 马文明, 陈智华, 吴鹏飞. 我国土壤动物生态学研究进展[J]. 安徽农业科学 , 2008, 36 (22) : 9644–9645. |
| [16] | 吾玛尔·阿布力孜, 古丽布斯坦·努尔买买提, 阿布都肉苏力·吐孙, 木开热木·阿吉木, 吴松林. 新疆阜康绿洲不同生境土壤动物群落多样性及其季节动态[J]. 生态学杂志 , 2013, 32 (6) : 1412–1420. |
| [17] | 李先敏, 余玲江. 太白山自然保护区的生物多样性及其威胁因素研究[J]. 林业调查规划 , 2005, 30 (6) : 6–9. |
| [18] | Machida R J, Knowlton N. PCR primers for metazoan nuclear 18S and 28S ribosomal DNA sequences[J]. PLoS ONE , 2012, 7 (9) : e46180. DOI:10.1371/journal.pone.0046180 |
| [19] | Leray M, Yang Y J, Meyer C P, Mills S C, Agudelo N, Ranwez V, Boehm J T, Machida R J. A new versatile primer set targeting a short fragment of the mitochondrial COI region for metabarcoding metazoan diversity:Application for characterizing coral reef fish gut contents[J]. Frontiers in Zoology , 2013, 10 : 34–34. DOI:10.1186/1742-9994-10-34 |
| [20] | Geller J, Meyer C, Parker M, Hawk H. Redesign of PCR primers for mitochondrial cytochrome coxidase subunit I for marine invertebrates and application in all-taxa biotic surveys[J]. Molecular Ecology Resources , 2013, 13 (5) : 851–861. DOI:10.1111/1755-0998.12138 |
| [21] | Caporaso J G, Kuczynski J, Stombaugh J, Bittinger K, Bushman F D, Costello E K, Fierer N, Peña A G, Goodrich J K, Gordon J I, Huttley G A, Kelley S T, Knights D, Koenig J E, Ley R E, Lozupone C A, McDonald D, Muegge B D, Pirrung M, Reeder J, Sevinsky J R, Turnbaugh P J, Walters W A, Widmann J, Yatsunenko T, Zaneveld J, Knight R. QⅡME allows analysis of high-throughput community sequencing data[J]. Nature Methods , 2010, 7 (5) : 335–336. DOI:10.1038/nmeth.f.303 |
| [22] | Schloss P D, Westcott S L, Ryabin T, Hall J R, Hartmann M, Hollister E B, Lesniewski R A, Oakley B B, Parks D H, Robinson C J, Sahl J W, Stres B, Thallinger G G, Horn D J V, Weber C F. Introducing mothur:Open-source, platform-independent, community-supported software for describing and comparing microbial communities[J]. Applied and Environmental Microbiology , 2009, 75 (23) : 7537–7541. DOI:10.1128/AEM.01541-09 |
| [23] | Blaxter M, Mann J, Chapman T, Thomas F, Whitton C, Floyd R, Abebe E. Defining operational taxonomic units using DNA barcode data[J]. Philosophical Transactions of the Royal Society B:Biological Sciences , 2005, 360 (1462) : 1935–1943. DOI:10.1098/rstb.2005.1725 |
| [24] | Quast C, Pruesse E, Yilmaz P, Gerken J, Schweer T, Yarza P, Peplies J, Glöckner F O. The SILVA ribosomal RNA gene database project:improved data processing and web-based tools[J]. Nucleic Acids Research , 2013, 41 (D1) : D590–D596. DOI:10.1093/nar/gks1219 |
| [25] | Ratnasingham S, Hebert P D N.bold:The Barcode of Life Data System (http://www.barcodinglife.org). Molecular Ecology Notes, 2007, 7(3):355-364. |
| [26] | 尹文英. 中国土壤动物检索图鉴. 北京: 科学出版社, 1998. |
| [27] | Aylagas E, Borja Á, Rodríguez-Ezpeleta N. Environmental status assessment using DNA metabarcoding:towards a genetics based marine biotic index (gAMBI)[J]. PLoS ONE , 2014, 9 (3) : e90529. DOI:10.1371/journal.pone.0090529 |
| [28] | Carew M E, Pettigrove1 V J, Metzeling L, Hoffmann A A. Environmental monitoring using next generation sequencing:rapid identification of macroinvertebrate bioindicator species[J]. Frontiers in Zoology , 2013, 10 : 45–45. DOI:10.1186/1742-9994-10-45 |
| [29] | Pruesse E, Quast C, Knittel K, Fuchs B M, Ludwig W, Peplies J, Glöckner F O. SILVA:a comprehensive online resource for quality checked and aligned ribosomal RNA sequence data compatible with ARB[J]. Nucleic Acids Research , 2007, 35 (21) : 7188–7196. DOI:10.1093/nar/gkm864 |
| [30] | Creer S, Fonseca V G, Porazinska D L, Giblin-Davis R M, Sung W, Power D M, Packer M, Carvalho G R, Blaxter M L, Lambshead P J, Thomas W K. Ultrasequencing of the meiofaunal biosphere:practice, pitfalls and promises[J]. Molecular Ecology , 2010, 19 (Supplement s1) : 4–20. |
| [31] | 尹文英. 中国土壤动物. 北京: 科学出版社, 2000. |
| [32] | 白登忠, 谢寿安, 史睿杰, 成洪刚. 秦岭土壤环境变化对土壤动物群落的影响[J]. 西北林学院学报 , 2012, 27 (6) : 1–7. |
| [33] | 阴环.陕西长安光头山土壤动物群落多样性的研究[D].西安:陕西师范大学, 2004. |
| [34] | 李鑫.苏打盐碱地桑树/大豆间作的土壤微生物多样性研究[D].哈尔滨:东北林业大学, 2012. |
| [35] | Piñol J, Mir G, Gomez-Polo P, Agustí N. Universal and blocking primer mismatches limit the use of high-throughput DNA sequencing for the quantitative metabarcoding of arthropods[J]. Molecular Ecology Resources , 2015, 15 (4) : 819–830. DOI:10.1111/1755-0998.12355 |
| [36] | Wintle B A, Runge M C, Bekessy S A. Allocating monitoring effort in the face of unknown unknowns[J]. Ecology Letters , 2010, 13 (11) : 1325–1337. DOI:10.1111/j.1461-0248.2010.01514.x |
| [37] | Possingham H P, Wintle B A, Fuller R A, Joseph L N.The conservation return on investment from ecological monitoring//Lindenmayer D B, Gibbons P, eds.Biodiversity Monitoring in Australia.Melbourne:CISRO Publishing, 2012:49-58. |
 2016, Vol. 36
2016, Vol. 36