文章信息
- 钟金城, 柴志欣, 马志杰, 王永, 杨万远, 拉环
- ZHONG Jincheng, CHAI Zhixin, MA Zhijie, WANG Yong, YANG Wanyuan, LA Huan
- 野牦牛线粒体基因组序列测定及其系统进化
- Mitochondrial complete genome sequencing and phylogenetic research on wild yak
- 生态学报, 2015, 35(5): 1564-1572
- Acta Ecologica Sinica, 2015, 35(5): 1564-1572
- http://dx.doi.org/10.5846/stxb201310142481
-
文章历史
- 收稿日期:2013-10-14
- 修订日期:2014-07-14
2. 青海大学畜牧兽医科学院, 青海高原牦牛研究中心, 西宁 810016;
3. 青海大通种牛场, 西宁 810016
2. Academy of Animal Science and Veterinary Medicine of Qinghai University, Qinghai Plateau Yak Research Center, Xining 810016, China;
3. Qinghai Datong Cattle Farm, Xining 810102, China
野牦牛(Bos mutus)属于牛科(Bovidae)牛亚科(Bovinae),是青藏高原珍贵而特有的野生物种资源,为国家一级重点保护野生动物,已被《濒危野生动植物种国际贸易公约》列为严禁贸易物种。目前,野牦牛种群数量为2万余头,分布于唐古拉山、昆仑山、巴颜喀拉山等高寒奇冷、气候多变的高山寒漠地带[1]。野牦牛体质健壮、性情凶猛、其肉可食、皮可制革、毛可制绳索和编织帐篷,尾巴可制成拂尘掸和入药,粪便晒干后可当燃料,是一种“全能”物种。野牦牛是家牦牛的近缘野生种,具有极强的抗寒能力和低氧适应能力。野牦牛是宝贵的遗传资源,野牦牛与家牦牛杂交能产生显著的杂种优势,其后代在生产性能、抗逆性等方面均较家牦牛有所提高,目前已经利用野牦牛资源培育了“大通牦牛”新品种[2]。
动物线粒体基因组(mtDNA)具有进化速率快、多态性丰富、无重组等特点,被广泛应用于动物起源、演化和分类,以及群体遗传结构、亲缘关系等研究[3]。由于野牦牛分布区生态环境恶劣,交通不便,其样品难于获得,从分子水平上对野牦牛的研究很少,目前仅见几篇文献报道。王敏强等[4, 5]利用微卫星标记对6头野牦牛遗传多样性进行分析,表明野牦牛具有丰富的遗传多样性。Guo S C等[6]基于mtDNA D-loop区部分序列(636—637 bp)对13头野牦牛和250头家牦牛及GenBank中已发表的相应序列对牦牛的起源、驯化和遗传多样性等进行了分析,表明家、野牦牛均具有丰富的遗传多样性。马志杰等[7]对6头野牦牛线粒体DNA D-loop区全序列进行了测定和分析,揭示野牦牛群体具有丰富的遗传多样性。另一方面,牦牛在牛亚科物种中的分类学地位仍然存在争议。为确定野牦牛线粒体基因组全序列结构,并在此基础上探究野牦牛在牛亚科中的分类学地位,本研究对野牦牛mtDNA进行全序列测定和结构分析,并结合GenBank中家牦牛、普通牛、瘤牛、欧洲野牛的相应序列对其分类学地位进行研究,以期为进一步了解野牦牛的遗传特性、起源、演化和分类以及野牦牛遗传资源的保护与利用提供基础。
1 材料与方法 1.1 实验动物青海省大通牦牛原种场从小捕获而人工饲养的6头健康成年野牦牛,颈静脉采血15 mL,EDTA抗凝,低温保存带回实验室,置于-80℃超低温保存备用。
1.2 方法本研究的所有实验工作均在动物遗传育种学国家民委-教育部重点实验室(西南民族大学)完成。
(1)基因组DNA提取和检测
按照常规的酚-氯仿法提取野牦牛血液基因组DNA[8],用紫外分光光度计和凝胶电泳双重检测其浓度和纯度,调整终浓度到50—100 ng/μL备用。
(2)引物设计、合成
根据Guo等[6]报道的家牦牛mtDNA全序列(GenBank accession No.: AY684273),综合使用Primer Premier 5.0和oligo 6.0软件设计特异性引物(表 1),引物序列由宝生物工程(大连)有限公司(TaKaRa)合成。
| 引物 Primers |
引物序列((5'-3') Primer sequences(5'-3') |
产物长度(bp) Product of the Size |
引物 Primers |
引物序列((5'-3') Primer sequences(5'-3') |
产物长度(bp) Product of the Size |
| P1 | F: CTACAGTCTCACCGTCAACC R: GGGGTGTAGATGCTTGC |
1050 | P7 | F: CATCACTTGCCCATTTCTTAC R: ATAGAGTCCGGCGTTT |
2310 |
| P2 | F: CTTGAGCACCAGCATAATG R: CTTTCATCGTTCCCTTGC |
1550 | P8 | F: TAGCATTCGCAGTATCTC R: TTCGGATGTCTTGTTCG |
2910 |
| P3 | F: CTATATACCGCCATCTTCAG R: CTACGACATTTGGACCTTTTC |
2180 | P9 | F: TGGTACGGACGAGCAG R: AGGAGGGAGCCGAAGT |
2040 |
| P4 | F: TGCCTGCCCAGTGACAAC R: GTACTTGCTTAGGGCTTTG |
3020 | P10 | F: GTTCCGTAGCCATAGCCG R: TTGAGTCTTAGGGAGGTT |
1520 |
| P5 | F: CAAAGCCCTAAGCAAGTAC′ R: TAATAGCGGGCAAAATGG |
2250 | P11 | F: ACCAGTCGAACACCCATA R: ATTAGCCATTAGTCCATC |
800 |
| P6 | F: GAATGATTAAACGGATGCC R: GTATGGTGCCCTTGGAAA |
2020 |
(3)PCR扩增
PCR扩增体(25 μL):10×ExTaqBuffer(Mg2+Free)2.5 μL,MgCl2 (25 mmol/L)1.5μL,dNTP(2.5 mmol/L)2 μL,上、下游引物分别为(20 pmol/L)1 μL,模板DNA(25—50 ng/μL)1 μL,ExTaq酶(5 U/μL) 0.5 μL,超纯水15.5 μL。10×Ex Taq Buffer(Mg2+ Free)、MgCl2(25 mmol/L)、dNTP(2.5 mmol/L)、Ex Taq酶(5 U/μL)等均购自宝生物工程(大连)有限公司(TaKaRa)。
PCR反应程序:95℃预变性4 min;95℃变性45 s,退火温度根据片断的Tm值而定退火时间为50 s,72℃延伸时间根据片断的长短每1 kb/min设置,35个循环;72℃延伸5 min;4℃保存。产物用1.0%琼脂糖凝胶电泳,EB染色并用凝胶成像系统检测。
(4)克隆、测序
对野牦牛mtDNA基因组PCR产物进行胶回收纯化,回收的目的片段进行连接、转化,将构建的重组质粒通过PCR法转化子鉴定,以确保克隆成功。将纯化后的阳性重组质粒用3730 XL型全自动DNA测序仪进行测序,测序结果用Clustalw和DNASTAR软件包中的MegAlign程序对同源序列进行排列,并经人工仔细核查,通过Seqman进行拼接得到目的序列。
(5)生物信息学分析
首先利用Clustal X、Sequin和BLAST软件对mtDNA全长序列进行同源序列比对分析,确定各蛋白编码基因、rRNA基因及D-loop区在线粒体全基因组中的位置。用tRNAscan-SE在线分析软件对tRNA进行预测,得到其2个tRNA基因。tRNAscan-SE没有发现的tRNA基因通过与已知的牦牛(AY684273)序列比对确定,用RNAStructure和RNADraw软件绘制其二级结构图。
(6)系统进化树的构建
从GenBank中搜索下载牛亚科其它物种的mtDNA全序列(表 2),用DNASTAR软件包中的MegAlign程序将其与本研究中测定的野牦牛相应序列进行多序列比对,后用DnaSP 4.50.3和MEGA 5.0计算不同序列间的变异位点、简约信息位点数、颠换百分比、转换/颠换比率、序列间的遗传距离和差异百分比;分别用邻位相连法NJ、最小进化法ME和最大简约法MP作系统进化树。
| 物种名 Species |
序列号 Accession No. |
大小(bp) Size |
物种名 Species |
序列号 Accession No. |
大小(bp) Size |
| *为本次试验所测序列 | |||||
| 野牦牛 *( Bos mutus) | 16322 | 鬣羊( Ammotragus lervia) | NC_009510 | 16530 | |
| 牦牛( Bos grunniens) | AY684273 | 16322 | 水牛( Bubalus bubalis) | NC_006295 | 16359 |
| 瘤牛( Bos indicus) | NC_005971 | 16341 | 美洲野牛( Bison bison) | NC_012346 | 16319 |
| 普通牛( Bos taurus) | NC_006853 | 16338 | 白犀牛( Ceratotherium simum) | Y07726 | 16832 |
| 绵羊( Ovis aries) | NC_001941 | 16616 | 印度犀牛( Rhinoceros unicornis) | NC_001779 | 16829 |
| 羚羊( Pantholops hodgsonii) | NC_007441 | 16498 | 马( Equus caballus) | X79547 | 16660 |
野牦牛线粒体基因组全长为16 322 bp,介于美洲野牛(American bison,NC_012346)16 319 bp和普通牛(Bos taurus,NC_006853)16 338 bp之间。共有37个基因,包括2个rRNA基因(16S rRNA和12S rRNA,长度2 528 bp)、22个tRNA基因(长度为1 524 bp)、13个编码蛋白基因(Protein-coding genes,PCGs,长度为11420 bp),非编码D-loop区,序列长度为892 bp;分别位于L链和H链(图 1,表 3)。基因组中无间隔序列,基因间排列紧密,无内含子,37个基因间共存在69 bp重叠,长度从1—40 bp不等,其中ATPase 8和ATPase 6重叠40个碱基,是最长的重叠。

|
| 图 1 野牦牛线粒体基因组物理图谱 Fig.1 Structure of mammalian mitochondrial genome 顺时针和逆时针分别表示不同基因分布在重链或轻链上 |
| 基因名 Gene name |
位置 Location |
分布链 Distribution |
大小 Size |
碱基GC含量 content of GC base |
起始密码 Start codon |
终止密码 Stop codon |
| D-loop | 1—892 | H | 892 | 39.24 | ||
| tRNA-Phe | 893—959 | H | 67 | 43.28 | ||
| 12S rRNA | 960—1916 | H | 957 | 40.75 | ||
| tRNA-Val | 1917—1983 | H | 67 | 31.34 | ||
| 16S rRNA | 1984—3554 | H | 1571 | 37.75 | ||
| tRNA-Leu (UUR) | 3555—3629 | H | 75 | 40.00 | ||
| ND1 | 3632—4588 | H | 957 | 41.27 | ATG | TAA |
| tRNA-Ile | 4588—4656 | H | 69 | 26.09 | ||
| tRNA-Gln | 4654—4725 | L | 72 | 37.50 | ||
| tRNA-Met | 4728—4796 | H | 69 | 46.38 | ||
| ND2 | 4797—5839 | H | 1043 | 35.19 | ATA | TAG |
| tRNA-Trp | 5840—5905 | H | 66 | 37.88 | ||
| tRNA-Ala | 5907—5975 | L | 69 | 33.33 | ||
| tRNA-Asn | 5977—6050 | L | 74 | 43.24 | ||
| OL | 6051—6081 | L | 31 | 54.84 | ||
| tRNA-Cys | 6083—6149 | L | 67 | 46.27 | ||
| tRNA-Tyr | 6150—6217 | L | 68 | 36.76 | ||
| COI | 6219—7763 | H | 1545 | 41.88 | ATG | TAA |
| tRNA-Ser (UCN) | 7761—7831 | L | 71 | 40.85 | ||
| tRNA-Asp | 7836—7904 | H | 69 | 34.78 | ||
| COII | 7906—8589 | H | 684 | 37.28 | ATG | ATA |
| tRNA-Lys | 8593—8659 | H | 67 | 38.81 | ||
| ATPase8 | 8661—8861 | H | 201 | 28.86 | ATG | TAA |
| ATPase6 | 8822—9502 | H | 681 | 38.03 | ATG | TAA |
| COIII | 9502—10282 | H | 781 | 44.81 | ATG | TAA |
| tRNA-Gly | 10286—10354 | H | 69 | 36.23 | ||
| ND3 | 10355—10700 | H | 346 | 41.04 | ATA | TAA |
| tRNA-Arg | 10702—10770 | H | 69 | 21.74 | ||
| ND4L | 10771—11067 | H | 297 | 35.02 | ATG | TAA |
| ND4 | 11061—12438 | H | 1378 | 37.01 | ATG | TAA |
| tRNA-His | 12439—12508 | H | 70 | 24.29 | ||
| tRNA-Ser (AGY) | 12509—12568 | H | 60 | 35.00 | ||
| tRNA-Leu (CUN) | 12570—12639 | H | 70 | 35.71 | ||
| ND5 | 12640—14460 | H | 1821 | 39.54 | ATA | TAA |
| ND6 | 14444—14971 | L | 528 | 36.74 | ATG | TAA |
| tRNA-Glu | 14972—15040 | L | 69 | 33.33 | ||
| Cytb | 15045—16184 | H | 1140 | 41.67 | ATG | AGA |
| tRNA-Thr | 16189—16257 | H | 69 | 39.13 | ||
| tRNA-Pro | 16257—16322 | L | 66 | 42.42 |
野牦牛线粒体基因组序列的GC含量为38%,AT含量为62%。tRNA基因和蛋白编码基因的GC含量存在差异,其中22个tRNA基因GC含量变化范围在21.74%—46.38%,蛋白编码基因GC含量的变化范围在28.86%—44.81%,蛋白编码基因的GC含量较tRNA基因高。ATPase8基因GC含量为28.86%,ATPase6基因GC含量为38.03%,两者相差约9%。
牛亚科物种的线粒体基因组中碱基组成相对稳定,野牦牛以及家牦牛、瘤牛、普通牛、绵羊、羚羊、鬣羊、水牛、美洲野牛、白犀牛、印度犀牛、马的全基因组中A、C、G、T的比例分别为32.2%—33.7%、25.9%—28.5%、12.7%—14.0%、25.7%—27.4%;rRNA基因中,A、C、G、T的比例分别为36.7%—38.1%、22.2%—23.0%、15.9%—17.9%、22.7%—23.8%;tRNA基因中,A、C、G、T的比例分别为34.5%—35.7%、20.8%—22.4%、14.8%—15.5%、27.2%—29.0%;编码蛋白基因中,A、C、G、T的比例分别为31.3%—32.8%、27.3%—30.4%、11.5%—12.8%、26.1%—28.0%;D-loop区中,A、C、G、T的比例分别为27.3%—33.7%、22.7%—30.9%、11.9%—15.5%、25.4%—29.8%。
野牦牛mtDNA组成偏好于碱基A和T,A+T含量远高于C+G的含量,这与其他物种相似,表明哺乳动物线粒体基因组的结构相对保守,不仅表现在其碱基序列长度相似,且其4种碱基组成比例也十分相近,在D-loop区中长度差异大,有较多插入和缺失序列,其碱基组成比例也与其它分子标准变异相对较大,但总体碱基组成相对稳定。
2.2 野牦牛线粒体蛋白质编码基因、tRNAs和密码子使用频率野牦牛线粒体基因组有13个蛋白编码基因,包含1个细胞色素b(Cytochrome b)编码基因、2个ATP合成酶亚基(ATPase6和ATPase8)、3个细胞色素氧化酶亚基(COI、COII、COIII)和7个NADH脱氢酶亚基(ND-1、-2、-3、-4、-4L、-5、和 -6)。这些蛋白编码基因没有内含子,基因结构排列紧密,总长11420bp,占基因组全长的70%。除ND6基因之外的所有12个蛋白编码基因全部由H链编码。野牦牛线粒体基因组密码子使用情况与已公布的牛科其他物种基本一致:10个蛋白编码基因起始密码子均为ATG,仅ND2、ND3和ND5基因起始密码子为非典型的ATA,终止密码子使用最多的是TAA(10个),ND2基因终止密码子为TAG,COIII基因终止密码子为ATA,Cytb基因终止密码子为AGA。
野牦牛线粒体基因组含有22个tRNA,其中14种位于重链上,8种位于轻链上,基因长度为60—75 bp。其中包含2个tRNA-Leu (UUR和CUN)和2个tRNA-Ser (UCN和AGY)。采用软件RNAscan-SE和RNAstructure预测tRNA二级结构发现,在22种tRNA基因中只有tRNA-Ser缺少二氢尿嘧啶环,其他的均可以折叠成典型的tRNA三叶草结构(图 2),其中tRNA-Val二级结构包括:典型三叶草型,主要包括氨基酸接受臂(Acceptor stem)、反密码子臂、反密码子环(Anti-codon loop)、双氢尿嘧啶(DHU)臂、TΨC臂、TΨC环(TΨCloop)。tRNA-Ser(AGY)的二级结构:缺乏双氢尿嘧啶(DHU)臂。
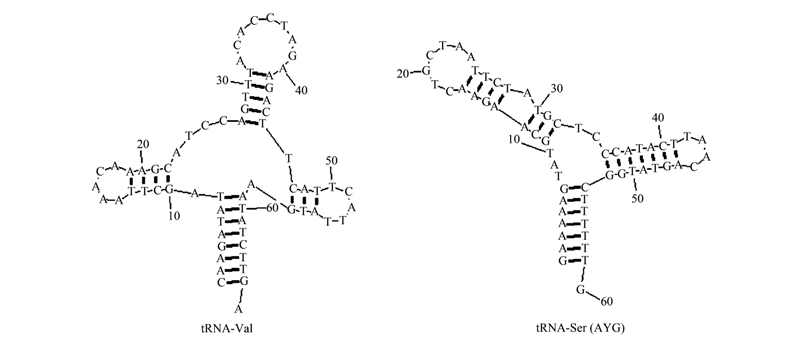
|
| 图 2 野牦牛线粒体基因组tRNA的二级结构图 Fig.2 Secondary structure of tRNA in wild yak |
为评估密码子使用情况,计算了RSCU值(表 4)。在线粒体全基因组序列中,UUA、CUA、AUA、GUU、UCU等30个密码子的RSCU均大于1,为野牦牛线粒体基因组偏好性密码子;其中A3s、T3s、C3s、G3s (A、T、G、C 的含量在第3位碱基总量中所占的比率) 分别42.38%、32.50%、32.99%、17.37%,A3s均大于T3s、C3s、G3s,表明野牦牛线粒体基因组偏爱使用碱基A或以A结尾的密码子。
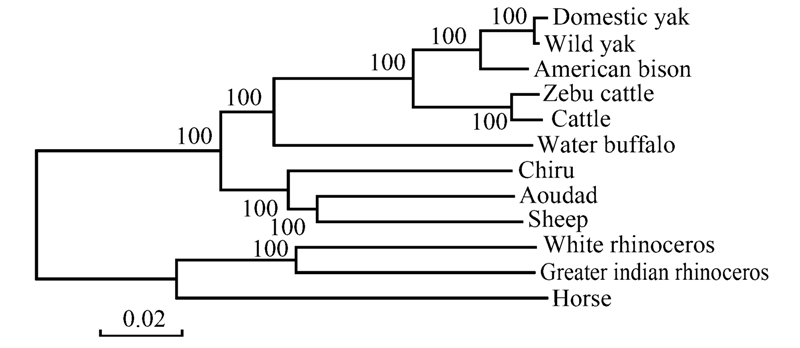
2.3 野牦牛与牛亚科其他物种间的系统进化关系以野牦牛和牛亚科其他物种的线粒体全基因组为分子标准,分别用邻位相连法NJ和最小进化法ME(图 3)和最大简约法MP(图 4)构建系统进化树。结果表明整个进化树的置信度很高,各分支的支持率均为100。3种不同方法构建的分子系统进化树基本一致,野牦牛是家牦牛的近缘野生种,亲缘关系较近两者首先相聚,形态学研究显示牦牛的前额骨、上额骨和鼻骨在组织排列上与美洲野牛和欧洲野牛相似,分子生物学等研究也表明牦牛与美洲野牛亲缘关系相近,再与美洲野牛聚为一类;普通牛与瘤牛聚为一类后与该类聚成一大类,其后与水牛相聚。最后才分别与其他物种相聚。
| 终止密码子及Trp和Met(RSCU值为1)未列入表中 | ||||||||
| 氨基酸 Amino acid |
密码子 Codon |
使用频率 Frequency of use |
相对密码子使用度 Relative synonymous codon usage |
氨基酸 Amino acid |
密码子 Codon |
使用频率 Frequency of use |
相对密码子使用度 Relative synonymous codon usage |
|
| Phe | UUU | 108 | 1.00 | Tyr | UAU | 139 | 0.97 | |
| UUC | 109 | 1.00 | UAC | 148 | 1.03 | |||
| Leu | UUA | 143 | 1.43 | His | CAU | 136 | 1.18 | |
| UUG | 73 | 0.73 | CAC | 95 | 0.82 | |||
| CUU | 75 | 0.75 | Gln | CAA | 174 | 1.34 | ||
| CUC | 95 | 0.95 | CAG | 85 | 0.66 | |||
| CUA | 171 | 1.72 | Asn | AAU | 144 | 0.97 | ||
| CUG | 41 | 0.41 | AAC | 153 | 1.03 | |||
| Ile | AUU | 137 | 0.98 | Lys | AAA | 190 | 1.47 | |
| AUC | 106 | 0.76 | AAG | 68 | 0.53 | |||
| AUA | 175 | 1.26 | Asp | GAU | 52 | 0.97 | ||
| Val | GUU | 39 | 1.05 | GAC | 55 | 1.03 | ||
| GUC | 36 | 0.97 | Glu | GAA | 101 | 1.17 | ||
| GUA | 57 | 1.53 | GAG | 71 | 0.83 | |||
| GUG | 17 | 0.46 | Cys | UGU | 34 | 0.83 | ||
| Ser | UCU | 126 | 1.25 | UGC | 48 | 1.17 | ||
| UCC | 118 | 1.17 | Arg | CGU | 22 | 0.92 | ||
| UCA | 121 | 1.20 | CGC | 25 | 1.04 | |||
| UCG | 37 | 0.37 | CGA | 35 | 1.46 | |||
| Pro | CCU | 113 | 1.14 | CGG | 14 | 0.58 | ||
| CCC | 118 | 1.19 | Ser | AGU | 48 | 0.70 | ||
| CCA | 114 | 1.15 | AGC | 90 | 1.30 | |||
| CCG | 51 | 0.52 | Gly | GGU | 25 | 0.79 | ||
| Thr | ACU | 131 | 1.18 | GGC | 34 | 1.08 | ||
| ACC | 116 | 1.05 | GGA | 45 | 1.43 | |||
| ACA | 157 | 1.41 | GGG | 22 | 0.70 | |||
| ACG | 40 | 0.36 | ||||||
| Ala | GCU | 60 | 1.23 | |||||
| GCC | 66 | 1.35 | ||||||
| GCA | 57 | 1.17 | ||||||
| GCG | 12 | 0.25 | ||||||
|
| 图 3 基于线粒体全基因组序列采用邻接 (NJ) 和最小进化 (ME) 法图构建的聚类关系 Fig.3 Based on mtDNA sequence constructed the NJ and ME phylogenetic trees |

|
| 图 4 基于线粒体全基因组序列采用最大简约 (MP) 法图构建的聚类关系 Fig.4 Based on mtDNA sequence constructed the MP phylogenetic trees |
哺乳动物线粒体全基因组大小相对稳定,全长均在16—17.5 kb间,只有极少数物种少于16 kb。基因组中无间隔序列,排列紧密,无内含子,基因的排列顺序基本一致。2个rRNA序列长度在2.5 kb左右、22个tRNA序列长度在1.5 kb左右,其序列长度和组成都比较接近[9]。长度最接近的是连接在一起的13个编码蛋白序列,其序列长度稳定在11.3 kb左右,且通过Clustalw的多序列比对,其碱基组成也十分相似,是很好的分子标准。长度差异最大的是D-loop区,其序列长度在800bp—1.7 kb,有较多的插入和缺失,可以看出线粒体基因组序列的长度差异主要存在于调控区。这些特点也表明线粒体基因组的稳定性。
本研究中,野牦牛线粒体基因组全长为16 322 bp。基因组中无间隔序列,基因间排列紧密,基因内无内含子。整个基因组由37个编码基因(2个rRNA基因、22个tRNA基因、13个编码蛋白基因)和D-loop区组成。野牦牛线粒体基因组的组成、结构与其他哺乳动物的一致。
3.2 关于野牦牛的遗传多样性自20世纪50年代以来,国内外学者从不同的研究层次对牦牛的遗传多样性进行了较为深入的探讨,揭示了家牦牛在群体内和群体间均具有丰富的遗传多样性。但对野牦牛的遗传多样性,由于其性情粗野不易捕获,且受分布范围和数量等因素限制,至今尚未进行系统地分析研究。在本研究中,将野牦牛的线粒体基因组序列与家牦牛的序列(Accession No:AY684273)进行比对,以及对6头野牦牛的mtDNA基因全序列进行SNP分析的结果均表明,野牦牛mtDNA基因序列内具有较丰富的遗传多态性,这与Guo[10]、张成福[11]、钟金城[12]等对家牦牛线粒体DNA D-loop区序列的分析结果一致,表明野牦牛群体也具有较丰富的遗传多样性,这是今后开发利用牦牛遗传资源和培育牦牛新品种的遗传基础。
3.3 关于牦牛的起源、演化及系统进化关系据研究表明,现在的家养牦牛是距今五千多年前(龙山文化时期),由我国古羌人在藏北羌塘等地区,将捕获的野牦牛驯养而来的;在人类驯养的所有家畜中,唯独牦牛的驯化史较清楚[13, 14]。现在的家养牦牛起源于我国的西藏;野牦牛是家养牦牛的近缘野生种。从目前发现的牦牛化石考证,野牦牛和家牦牛都是距今三百多万年前(更新世)生存并广为分布在欧亚大陆东北部的原始牦牛的后代。后来,由于地壳运动、气候变迁而南移至现世界屋脊——我国青藏高原,并适应高寒气候环境条件而延续下来,现今的家、野牦牛,都是同一祖先的后代,他们之间不存在祖先与后代的关系;现在的野牦牛,也不是家牦牛的始祖、始源或祖先的观点[15]。
随着分子生物学的兴起和发展,利用现代分子技术辅以考古学、历史学、形态学等对动物的驯化历史进行了深入研究。近年来通过对野牦牛和家牦牛的研究初步揭示了牦牛的驯化历史,群体间的遗传多样性及其结构等方面的内容。Guo等[6, 16]通过对250头家牦牛及13头野牦牛的D-loop部分序列进行了分析,发现存在两个较大的母系遗传分支,这两个分支的分化时间约在131000—109000年以前,早于牦牛的驯化时间,同时显示,在一个野生居群中也存在这两大遗传分支,表明所有的家养牦牛都起源于同一个野生基因库。Ho等[17]基于D-loop片段对牦牛两大分支的分化时间重新进行估计,结果显示其分化时间在75000年以前。
关于牦牛的起源、演化和动物学分类地位,一直是牦牛科学研究的热点之一。学者们依不同的研究方法和证据,提出了各自的观点和看法。在《中国牛品种志》[18]、《中国畜禽遗传资源志·牛志》[19]中,认为牦牛(Bos grunniens)与普通牛(Bos taurus)、瘤牛(Bos indicus)等均为牛属(Bos)中的不同种,他们属于种间的关系。而Gray[20]、Groves[21]、冯祚建[22]、Miyamoto[23]、Olsen[24, 25]、Geraads[26]、Kraus[27]、Wall[28]、Hassanin[29]、李齐发[30]、常洪[31]等学者分别从古生物学、形态学、分子生物学等的研究结果,认为牦牛与普通牛、瘤牛分属于不同的属,牦牛属于牦牛属(Poephagus),牦牛属包括家牦牛(Poephagus grunniens)和野牦牛(Poephagus mutus)2个种,普通牛和瘤牛为家牛属(Bos)。在本研究中,基于线粒体基因组的分子系统发生分析结果显示野牦牛与家牦牛首先相聚在一起,然后再与美洲野牛聚为一类;普通牛与瘤牛聚为一类;这两类相聚成一大类后,才与水牛聚为更大的一类,最后才分别与其他物种相聚。因此,将牦牛划分为牛亚科中一个独立属——牦牛属(Poephagus)较将牦牛作为牛属中的一个亚属或一个种更合适,牦牛属包括家牦牛(Poephagus grunniens) 和野牦牛(Poephagus mutus)2个不同的种。
4 结论野牦牛线粒体基因组的组成、结构与其他哺乳动物的相似,基因组全长为16 322 bp,基因组中无间隔序列,基因间排列紧密,基因内无内含子;整个基因组由37个编码基因(2个rRNA基因、22个tRNA基因、13个编码蛋白基因)和D-loop区组成。野牦牛具有较丰富的遗传多样性。研究结果支持牦牛属(Poephagus)为牛亚科中的一个独立属,包括家牦牛(Poephagus grunniens)和野牦牛(Poephagus mutus)2个种。
| [1] | 杨博辉, 郎侠, 阎萍, 梁春年, 孙晓萍. 中国野牦牛种质资源库体系的建立. 家畜生态学报, 2006, 27(1): 1-5. |
| [2] | 《中国畜禽遗传资源状况》编委会. 中国畜禽遗传资源状况. 北京: 中国农业出版社, 2004. |
| [3] | Wolf C, Rentsch J, Hübner P. PCR-RFLP analysis of mitochondrial DNA: a reliable method for species identification. Journal of Agricultural and Food Chemistry, 1999, 47(4): 1350-1355. |
| [4] | 王敏强. 大通牦牛部分种质特性的研究 [D]. 杨凌: 西北农林科技大学, 2000. |
| [5] | 胡江. 野牦牛、家牦牛及其杂交种的生物学特性和遗传多样性研究 [D]. 兰州: 甘肃农业大学, 2005. |
| [6] | Guo S C, Savolainen P, Su J P, Zhang Q, Qi D L, Zhou J, Zhong Y, Zhao X Q, Liu J Q. Origin of mitochondrial DNA diversity of domestic yaks. BMC Evolutionary Biology, 2006, 6(1): 73. |
| [7] | 马志杰, 钟金城, 韩建林, 徐惊涛, 窦全林, 常怀普. 野牦牛(Bos grunniens mutus)mtDNAD-Loop区的遗传多样性. 生态学报, 2009, 29(9): 4798-4803. |
| [8] | 萨姆布鲁克 J, 拉塞尔 D W. 分子克隆实验指南. 北京: 科学出版社, 2002. |
| [9] | Bernard Lemire. Mitochondrial genetics. Available from: http://www.wormbook.org/chapters/www_mitogenetics/mitogenetics.Pdf. |
| [10] | Guo S, Savolainen P, Su J, Zhang Q, Qi D, Zhou J, Zhong Y, Zhao X, Liu J. Origin of mitochondrial DNA diversity of domestic yaks. BMC Evolutionary Biology, 2006, 6(1): 73-73. |
| [11] | 张成福, 徐利娟, 姬秋梅, 信金伟, 钟金城. 西藏牦牛mtDNA D-loop区的遗传多样性及其遗传分化. 生态学报, 2012, 32(5): 1387-1395. |
| [12] | 钟金城, 柴志欣, 姬秋梅, 赵上娟, 徐丽娟, 唐懿挺, 张成福, 信金伟. 西藏牦牛的遗传多样性及其系统进化研究. 西南民族大学学报: 自然科学版, 2011, 37(3): 368-378. |
| [13] | 蔡立. 四川牦牛. 成都: 四川民族出版社, 1989. |
| [14] | 蔡立. 中国牦牛. 北京: 中国农业出版社, 1992: 45-47. |
| [15] | 钟金城. 牦牛遗传与育种. 成都: 四川科学技术出版社, 1996: 24-68. |
| [16] | 王召锋. 牦牛驯化和适应: 线粒体基因组证据 [D]. 兰州: 兰州大学, 2011. |
| [17] | Ho S Y W, Larson G, Edward S C J, HeuPink T H, Lakin K E, Holland P W H, Shapiro B. Correlating Bayesian date estimates with climatic events and domestication using a bovine case study. Biology Letters, 2008, 4(4): 370-374. |
| [18] | 中国家畜家禽品种志编委会. 中国牛品种志. 上海: 上海科学技术出版社, 1988. |
| [19] | 国家畜禽遗传资源委员会. 中国畜禽遗传资源志·牛志. 北京: 中国农业出版社, 2011. |
| [20] | Gray J E. List of the specimens of Mammalia in the collection of the British Museum. London, Trustees of the British Museum, 1843. |
| [21] | Groves C P. Systematic relationships in the Bovini (Artiodactyla: Bovidae). Zeitschrift für Zoologische Systematik und Evolutionsforschung, 1981, 19: 264-278. |
| [22] | 冯祚建, 蔡桂全, 郑昌琳. 西藏哺乳类. 北京: 科学出版社, 1986. |
| [23] | Miyamoto M M, Tanhauser S M, Laipis P J. Systematic relationship in the artiodactyls tribe Bovini (Family Bovidae), as determined from mitochondrial DNA sequences. Systematic Zoology, 1989, 38(4): 342-349. |
| [24] | Olsen S J. Fossil Ancestry of the Yak, Its cultural significance and domestication in Tibet. Proceedings of the Academy of Natural Sciences of Philadelphia, 1990, 142: 73-100. |
| [25] | Olsen S J. Confused yak taxonomy and evidence of domestication. Illinois State Museum Scientific Papers, 1991, 23: 387-393. |
| [26] | Geraads D. Phylogenetic analysis of the Tribe Bovini (Mammalia: Artiodactyl). Zoological Journal of the Linnean Society, 1992, 104: 193-207. |
| [27] | Kraus F, Jarecki L, Miyamoto M M, Tanhauser S M, Laipis P J. Mispairing and compensational changes during the evolution of mitochondrial ribosomal RNA. Molecular and Biological Evolution, 1992, 9(4): 770-774. |
| [28] | Wall D A, Davis S K, Read B M. Phylogenetic relationships in the subfamily bovinae (Mammalia: Artiodactyla) based on Ribosomal DNA. Journal of Mammalogy, 1992, 73(2): 262-275. |
| [29] | Hassanin A, Douzery E J P. Evolutionary affinities of the enigmatic Saola (Pseudoryx Ngheti Nhensis) in the context of the molecular phylogeny of Bovidae. Proceedings of Royal Society London, 1999, 266(1422): 893-900. |
| [30] | 李齐发, 李隐侠, 赵兴波, 刘振山, 张庆波, 宋大伟. 牦牛线粒体DNA 细胞色素b 基因序列测定及其起源、分类地位研究. 畜牧兽医学报, 2006, 37(11): 1118-1123. |
| [31] | 常洪. 动物遗传资源学. 北京: 科学出版社, 2009. |
 2015, Vol. 35
2015, Vol. 35