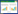文章信息
- 苗祯, 杜宗军, 李会荣, 楼妍颖, 罗玮
- MIAO Zhen, DU Zongjun, LI Huirong, LOU Yanying, LUO Wei
- 5株北极微藻藻际环境的细菌多样性
- Analysis of bacterial diversity in the phycosphere of five Arctic microalgae
- 生态学报, 2015, 35(5): 1587-1600
- Acta Ecologica Sinica, 2015, 35(5): 1587-1600
- http://dx.doi.org/10.5846/stxb201305070961
-
文章历史
- 收稿日期:2013-05-07
- 修订日期:2014-04-17
2. 中国极地研究中心, 国家海洋局极地科学重点实验室, 上海 200136
2. State Oceanic Administration Key Laboratory for Polar Science, Polar Research Institute of China, Shanghai 200136, China
微藻分泌的胞外产物对细菌食物链具有重要作用,形成了自微藻细胞向外至一定距离的对细菌生长有刺激作用的区域,类似于陆上的根际环境,这个区域被命名为“藻际环境”(phycosphere)[1]。有研究表明藻际环境的游离细菌和粘附细菌群落结构是不同的[2, 3],Fandino等研究表明在腰鞭毛藻赤潮期间游离细菌主要属于α-Proteobacteria和γ-Proteobacteria,粘附细菌主要以CFB菌群占优势[3]。Schfer 等研究了6种不同的藻株附着不同的“卫星”细菌群落,而且基因指纹显示这种“卫星”附着关系较为稳定。所有藻株附着类群的共同点就是均归属于α-Proteobacteria和CFB其中的某一个支系[4]。也有研究表明海洋藻际环境中细菌群落和海洋中的游离细菌群落在系统发育上有着明显的区别,说明两种不同生存环境下的细菌种群结构组成由不同的选择性压力所影响[5, 6]。更多的生态调查研究表明在浮游植物赤潮过程中细菌群落组成与浮游植物的组成、生长和生理状态相关[4, 5, 7, 8]。特定藻类能吸引不同细菌附生群落,显示浮游植物-细菌相互作用具有种间特异性[4, 6, 9],而菌群对某些微藻的表现出“溶藻”[10]或者“共促”机理[11]都证明了藻、菌关联之间的特异性。
北极地区是受全球变化影响最深的地区,极地海洋系统易于受到周围环境等因子的影响,已经引起科学家对这些极端区域海洋微生物的多样性和生态学作用的极大兴趣。目前极地海洋微生物多样性研究多以海冰、海水和近岸沉积物微生物群落为研究对象,对北极海域浮游细菌系统发育多样性的研究表明,浮游细菌主要分布在α-、γ-Proteobacteria、δ-Proteobacteria、ε-Proteobacteria、CFB类群、绿色非硫细菌和Verrucomicrobia这7个大的类群。这些克隆序列跟多数非培养的环境序列存在高度同源性[12]。人们利用传统培养技术和分子生物学技术开展了对北极楚科奇海、弗拉姆海峡、斯匹次卑尔根岛屿附近峡湾海冰样品的细菌系统发育多样性分析。海冰细菌主要属于α、γ-Proteobacteria、CFB、低G+C含量革兰氏阳性菌群和高G+C含量革兰氏阳性菌群[13, 14, 15]。而极区微藻也是常见的浮游植物类群,是重要的初级生产力类群,北极海域的主要优势种类为青绿藻和硅藻。它们同样在北冰洋生态系统的碳固定过程中起着重要的作用。目前人们热衷于极地快速变化环境下的特定种群(浮游植物,浮游细菌,浮游动物等)的生态学调查,而在这种迅速变化的生境中的生态系统反馈和应对机理需要更多的摸索和一些全新的理解。在此背景下,展开针对北极获得的几株优势微藻藻际环境中藻菌关联的机理的研究。
基于对北极的优势浮游植物-细菌相互作用机制的探索为目的,从北极各生境中分离出的具重要生态地位的微藻,并对与这些微藻紧密关联的微生物群落多样性进行研究,将对认识北极海域微食物环以及其中的各个生态学角色有了更深入的认知。
1 材料与方法 1.1 藻种来源及培养本研究中的5株北极微藻,信息见表 1。海洋微藻微单胞藻(Micromonas sp.)、脆杆藻(Fragilariopsis sp.)、四棘藻(Attheya septentrionalis)和海链藻(Thalassiasira sp.)购自美国国家海洋藻类和微生物中心(NCMA,Maine,USA),淡水藻小球藻(Lw 2006/68)采自2006年中国第3次北极黄河站考察,北极斯匹次卑尔根群岛黄河站(78°55′N,11°56′E)附近的夏季冰川融水坑,样品低温((6±0.5) ℃)保存回国,在实验室分离纯化并保存于中国极地研究中心藻种库。
| 微藻 Microalgae | 编号 No. |
分类 Classification |
来源 Source |
培养基 Medium |
| 脆杆藻Fragilariopsis sp. | CCMP 2297 | Bacillariophyta | 巴芬湾 | L1+Si |
| 四棘藻Attheya septentrionalis | CCMP 2084 | Bacillariophyta | 巴芬湾 | f/2 |
| 海链藻Thalassiasira sp. | CCMP 1056 | Bacillariophyta | 挪威海 | f/2 |
| 微单胞藻Micromonas sp. | CCMP 2099 | Chlorophyta | 巴芬湾 | L1 |
| 小球藻Chlorella sp. | Lw 2006/68 | Chlorophyta | 斯匹次卑尔根岛 | B |
微藻培养在光照培养箱中进行,温度为(6±0.5) ℃,光强为2000 lx,光照周期为光 ∶ 暗=12 h ∶ 12 h。所有藻株的培养基参照表 1,海水均采自北极海域,将海水通过0.45 μm的膜过滤,121 ℃,20 min进行灭菌。维生素溶液通过0.2 μm滤器进行除菌。
1.2 微藻生长曲线的测定微藻计数主要采用光学显微镜细胞计数[16]。取100 μL混匀的藻液均匀加入细胞计数板中,光学显微镜(Olympus)(10×40)观察计数。计数从第0天开始,每2 d计数1次。
1.3 藻际环境的细菌采样取不同生长期的藻液10 mL直接通过0.2 μm的聚碳酸膜(Whatman)过滤,获得游离和粘附的总细菌。取不同生长期的藻液10 mL,先经膜过滤,获得微藻细胞的粘附细菌(Attached);其中Micromonas sp.由于细胞约为2.0—3.0 μm,选用孔径为2.0 μm的滤膜,其他藻株选用3.0 μm滤膜。将滤液再通过0.2 μm的滤膜,获得藻液中的游离细菌。滤膜及样于冰箱-80 ℃保存。
1.4 DNA提取DNA提取参照Zhou等[17]的方法进行。
1.5 16S rRNA基因全长序列 PCR参照 Bosshard 等[18]的方法进行。16S rRNA基因引物序列如下:27F (5′-AGA GTT TGA TCC TGG CTC AG -3′) 和1492R (5′-GGT TAC CTT GTT ACG ACT T -3′)。PCR 反应条件为 94 ℃变性 4 min,然后在 94 ℃ 45 s,50 ℃ 45 s,72 ℃ 1 min 循环30次,72 ℃延伸10 min。
1.6 16S rRNA 基因V3 区的PCR扩增采用Shabir等[19]的方法进行。引物的序列如下:341F (5′-CGC CCG CCG CGC GCG GCG GGC GGG GCG GGG GCA CGG GGG GCC TAG GGG AGG CAG CAG-3′) 和534R (5′-ATT ACC GCG GCT GCT GG-3′),下划线处为GC钳序列。PCR扩增在Veriti Thermal Cycler(ABI)上进行。以 1 μL 16S rRNA全长序列 PCR 产物作为模板,PCR反应条件:94 ℃预变性4 min,然后是30循环(94 ℃变性30 s,58 ℃退火30 s,72 ℃延伸1 min),最后72 ℃延伸10 min。
1.7 变性梯度凝胶电泳(DGGE)800 ng—1 μg 16S rRNA基因V3区扩增产物进行DGGE分离。用D-code System电泳仪(Bio-Rad公司)进行DGGE电泳分离。制备变性梯度凝胶,使PAGE胶浓度为8%,变性梯度40%—60%(7 mol/L尿素和40%甲酰胺为100%变性),电泳缓冲液为1×TAE,PCR产物在60 ℃,200 V条件下电泳6 h。电泳完毕,用GelRedTM(Biotium)染色45—60 min。DGGE图谱通过Gel Doc 2000凝胶成像系统 (Bio-Rad,Hercules,CA,USA)获得。
将聚丙烯酰胺凝胶中在同一个水平位置的条带割下,放在PCR小管中,并加入20 μL MilliQ水,放在4 ℃冰箱过夜,以此为模板,进行16S rRNA V3 区 PCR扩增,实验条件同上。
1.8 DNA序列测序和分析按照pMD18-T载体试剂盒(Takara,大连)说明进行操作。将16S rRNA基因V3区扩增产物分别同pMD18-T载体连接,连接产物转入大肠杆菌DH5α感受态细胞中。涂布至含有氨苄青霉素SOC平板上,37 ℃培养12 h。每个平板挑取克隆,接入500 μL SOC液体培养基37 ℃培养,经 T 载体引物 PCR 扩增筛选阳性克隆,扩增引物为 M13+(5′-GTA AAA CGA CGG CCA G -3′)和 M13-(5′- CAG GAA ACA GCT ATG AC -3′),送去(南方基因测试中心)测序。测序获得的16S rRNA序列进行BLAST比对(http://www.ncbi.nlm.nih.gov),应用CLUSTALX1.8(多重序列比对程序)进行匹配比对,后用PHYLIP 3.67(进化树分析)构建进化树[20]。
1.9 聚类分析根据电泳条带的有无,运用图像分析软件Quantity One(4.6.2)分析DGGE图谱,采用UPGMA法进行聚类分析。
2 结果与分析 2.1 生长曲线测定结果5株微藻的生长曲线见图 1。从图 1中可以看出,5株微藻的生长周期约为30 d。小球藻的3个生长阶段不明显,将小球藻3个生长阶段大致划分,延滞期约为7 d,随后进入指数生长期,在21 d达到最大密度,随后略微下降并稳定生长。其他4株微藻的生长阶段较明显,脆杆藻、四棘藻、海链藻和微单胞藻的延滞期分别为前9 d、5 d、12 d和9 d,在16 d、14 d、27 d和16 d达到最大生长密度,随后进入稳定生长期。藻液分别取自各个藻株的生长延滞期、指数期和稳定期(图 1)。
|
| 图 1 5种不同微藻的生长曲线 Fig.1 The growth curve of five different microalgae involved into this study L: 延滞生长期;E: 指数生长期;S: 稳定生长期 |
从DGGE电泳图谱(图 2A,图 3A,图 4A,图 5A,图 6A)可以看出,5株微藻藻际细菌群落的条带的数目和位置有所区别,说明不同藻株的藻际细菌群落组成不同。5种藻际细菌群落的优势种均十分明显,分别对应于DGGE图谱中的亮条带。较微弱DGGE条带所对应的细菌类群的数量可能相对较低。

|
| 图 2 脆杆藻藻际细菌群落的16S rRNA基因V3区的DGGE图谱和聚类分析图(T: 总细菌;A: 粘附细菌;F:游离细菌) Fig.2 DGGE patterns and cluster analysis of 16S rRNA gene V3 region amplification fragments of bacteria community in the phycosphere of Fragilariopsis sp. |
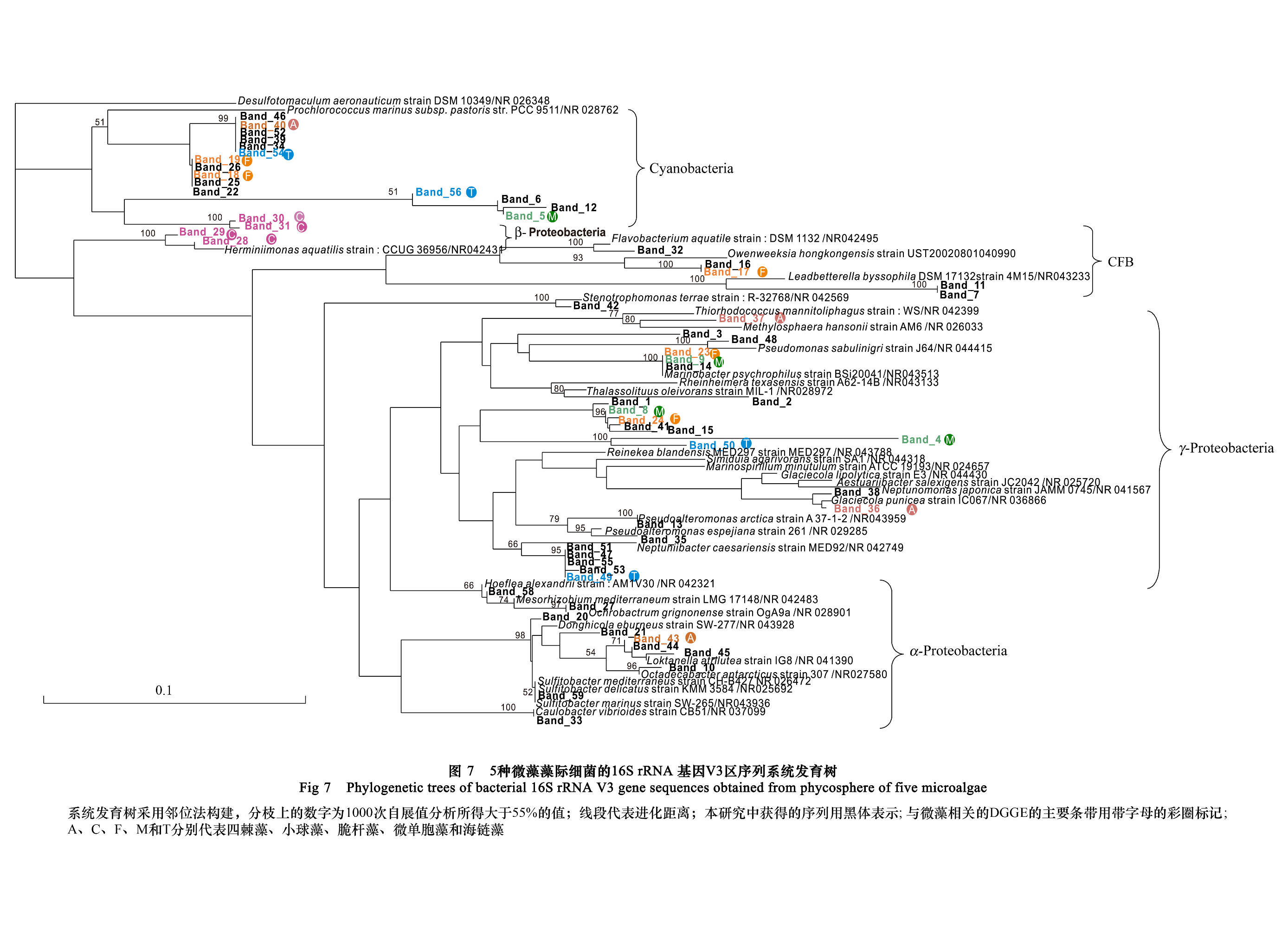
基于5株微藻藻际细菌的DGGE图谱主要条带,共获得59条克隆序列(其中一条测序失败),它们的测序结果经BLAST比对显示(表 2),5株微藻藻际细菌种群与数据库中的已知序列的相似性为87%—100%,包 括: Cyanobacteria、CFB菌群、α-Proteobacteria、β-Proteobacteria、γ-Proteobacteria(图 7)。由表 2和系统进化树
| DGGE 条带 DGGE band |
分类 Taxanomic group |
有效描述的物种的最相似序列(登录号) Most closely related sequence from a validly described bacterial species (GenBank accession No.) |
相似度/% Sequence similarity |
数据库中最相似的序列(登录号) Most closely related database sequence (GenBank accession No.) |
相似度/% Sequence similarity |
| **条带最亮; *条带比较亮 | |||||
| 微单胞藻Micromonas sp. | |||||
| Band_1 | Gammaproteobacteria | Psychromonas arctica(NR_028821) | 94 | clone Kwat47(EU035868) | 98 |
| Band_2 | Gammaproteobacteria | Thalassolituus oleivorans(NR_028972) | 94 | clone B78-116 (EU287080) | 100 |
| Band_3 | Gammaproteobacteria | Rheinheimera texasensis (NR_043133) | 94 | clone SHAB602(GQ348690) | 95 |
| Band_4* | Gammaproteobacteria | Shewanella canadensis(NR_042994) | 92 | clone Kwat47(EU035868) | 98 |
| Band_5** | Cyanobacteria | Prochlorococcus marinus subsp. Pastoris(NR_028762) |
87 | clone IB0346E7(HQ730061) | 100 |
| Band_6* | Cyanobacteria | Prochlorococcus marinus subsp. Pastoris(NR_028762) |
87 | clone IB0346E7(HQ730061) | 100 |
| Band_7 | CFB | Leadbetterella byssophila(NR_043233) | 94 | clone MEf05b11G6 (FJ828154) | 95 |
| Band_8* | Gammaproteobacteria | Shewanella canadensis (NR_042994) | 94 | clone Gap-2-29 (EU642169) | 95 |
| Band_9* | Gammaproteobacteria | Marinobacter psychrophilus(NR_043513) | 100 | ||
| Band_10 | Alphaproteobacteria | Octadecabacter antarcticus (NR_027580) | 98 | Octadecabacter sp. ANT9202(AY167337) |
99 |
| Band_11 | CFB | Leadbetterella byssophila(NR_043233) | 94 | clone MEf05b11G6 (FJ828154) | 95 |
| Band_12 | Cyanobacteria | Prochlorococcus marinus subsp. Pastoris(NR_028762) | 87 | clone IB0346G2 (HQ730072) | 100 |
| 脆杆藻Fragilariopsis sp. | |||||
| Band_13 | Gammaproteobacteria | Pseudoalteromonas arctica(NR_043959) | 100 | ||
| Band_14 | Gammaproteobacteria | Marinobacter psychrophilus(NR_043513) | 99 | Marinobacter psychrophilus (FR691434) | 99 |
| Band_15 | Gammaproteobacteria | Shewanella canadensis(NR_042994) | 93 | clone Kwat47 | 99 |
| Band_16 | CFB | Owenweeksia hongkongensis(NR_040990) | 95 | clone PI_4b12a (AY580701) | 99 |
| Band_17* | CFB | Owenweeksia hongkongensis(NR_040990) | 95 | clone PI_4b12a (AY580701) | 99 |
| Band_18** | Cyanobacteria | Halospirulina tapeticola(NR_026510) | 91 | clone CM01187X1E11(GU170707) | 99 |
| Band_19** | Cyanobacteria | Halospirulina tapeticola(NR_026510) | 91 | clone CM01187X1E11 (GU170707) | 100 |
| Band_20 | Alphaproteobacteria | Sulfitobacter marinus (NR_043936) | 99 | Sulfitobacter sp. LM-16(AJ534237) | 100 |
| Band_21 | Alphaproteobacteria | Sulfitobacter marinus (NR_043936) | 98 | Sulfitobacter sp. LM-16(AJ534237) | 100 |
| Band_22 | Cyanobacteria | Halospirulina tapeticola(NR_026510) | 91 | clone AW03F12(EF630247) | 100 |
| Band_23* | Gammaproteobacteria | Marinobacter psychrophilus(NR_043513) | 99 | Marinobacter psychrophilus (FR691434) | 100 |
| Band_24* | Gammaproteobacteria | Shewanella canadensis(NR_042994) | 93 | clone Gap-2-29(EU642169) | 95 |
| Band_25 | Cyanobacteria | Halospirulina tapeticola(NR_026510) | 91 | clone AW03F12(EF630247.) | 100 |
| Band_26 | Cyanobacteria | Halospirulina tapeticola(NR_026510) | 91 | clone AW03F12(EF630247.) | 100 |
| 小球藻Chlorella sp. | |||||
| Band_27 | Alphaproteobacteria | Mesorhizobium thiogangeticum(NR_042358) | 100 | ||
| Band_28* | Betaproteobacteria | Herminiimonas fonticola(NR_043090) | 100 | ||
| Band_29* | Betaproteobacteria | Herminiimonas fonticola(NR_043090) | 99 | ||
| Band_30** | Cyanobacteria | Halospirulina tapeticola(NR_026510) | 91 | clone Gap-3-60 (EU639772) | 100 |
| Band_31** | Cyanobacteria | Halospirulina tapeticola(NR_026510) | 91 | clone Gap-3-60 (EU639772) | 99 |
| Band_32 | CFB | Bizionia myxarmorum(NR_043121) | 98 | CLAb44(HQ230110) | 98 |
| Band_33 | Alphaproteobacteria | Brevundimonas nasdae (NR_028633) | 100 | 100 | |
| 四棘藻Attheya septentrionalis | |||||
| Band_34 | Cyanobacteria | Halospirulina tapeticola(NR_026510) | 91 | MBE6 (AF191757) | 100 |
| Band_35 | Gammaproteobacteria | Pseudoalteromonas espejian(NR_029285) | 98 | Pseudoalteromonas sp. BSi20627(EU330358) |
99 |
| Band_36 | Gammaproteobacteria | Glaciecola punicea (NR_036866) | 99 | Glaciecola sp. D94(DQ652563) | 100 |
| Band_37 | Gammaproteobacteria | Methylosphaera hansonii (NR_026033) | 96 | clone MOH_577_24(EU399445) | 99 |
| Band_38 | Gammaproteobacteria | Glaciecola punicea (NR_036866) | 98 | Glaciecola sp. D94(DQ652563) | 98 |
| Band_39* | Cyanobacteria | Halospirulina tapeticola(NR_026510) | 91 | clone PI_4g9g (AY580402) | 100 |
| Band_40** | Cyanobacteria | Halospirulina tapeticola(NR_026510) | 91 | clone PI_4g9g (AY580402) | 100 |
| Band_41 | Gammaproteobacteria | Shewanella canadensis(NR_042994) | 93 | clone Kwat47(EU035868) | 99 |
| Band_42 | Gammaproteobacteria | Stenotrophomonas acidaminiphila(NR_025104) | 100 | ||
| Band_43 | Alphaproteobacteria | Loktanella atrilutea (NR_041390) | 100 | ||
| Band_44 | Alphaproteobacteria | Loktanella salsilacus (NR_025539) | 100 | ||
| Band_45 | Alphaproteobacteria | Loktanella atrilutea (NR_041390) | 100 | ||
| 海链藻Thalassiosira sp. | |||||
| Band_46 | Cyanobacteria | Rubidibacter lacunae(NR_044104) | 91 | clone 16B_215(AM501739) | 100 |
| Band_47 | Gammaproteobacteria | Ferrimonas kyonanensis(NR_041387) | 95 | clone JL-ETNP-R18(AY726789) | 96 |
| Band_48 | Gammaproteobacteria | Pseudomonas cedrina subsp. Fulgida strain (NR_042147) |
98 | M71_D72(FM992728) | 98 |
| Band_49* | Gammaproteobacteria | Ferrimonas kyonanensis(NR_041387) | 95 | clone JL-ETNP-R18(AY726789) | 96 |
| Band_50 | Gammaproteobacteria | Ferrimonas kyonanensis(NR_041387) | 95 | clone H10-120(HQ222995) | 96 |
| Band_51* | Gammaproteobacteria | Ferrimonas kyonanensis(NR_041387) | 94 | clone OV01102/03(HM587890) | 95 |
| Band_52 | Cyanobacteria | Rubidibacter lacunae(NR_044104) | 91 | clone DT_068(DQ881210) | 100 |
| Band_53 | Gammaproteobacteria | Ferrimonas kyonanensis(NR_041387) | 95 | clone H10-120(HQ222995) | 96 |
| Band_54** | Cyanobacteria | Rubidibacter lacunae(NR_044104) | 91 | clone D05RT35 (GQ242576) | 100 |
| Band_55* | Gammaproteobacteria | Ferrimonas kyonanensis(NR_041387) | 95 | clone H10-120(HQ222995) | 96 |
| Band_56** | Cyanobacteria | Prochlorococcus marinus subsp.Pastoris(NR_028762) | 88 | clone SGPW483(GQ346754) | 97 |
| Band_58 | Alphaproteobacteria | Hoeflea alexandrii(NR_042321) | 100 | ||
| Band_59 | Alphaproteobacteria | Sulfitobacter mediterraneus(NR_026472) | 100 | ||
(图 7)可以看出,从5株微藻培养液获得的细菌数量和类群有所不同。微单胞藻、脆杆藻、四棘藻和海链藻的藻际细菌中,主要由Cyanobacteria、α-Proteobacteria和γ-Proteobacteria组成,仅微单胞藻和脆杆藻检测出CFB菌群。淡水藻株小球藻是由Cyanobacteria、CFB菌群、α-Proteobacteria、β-Proteobacteria,而无γ-Proteobacteria。无论是哪种微藻,群落的优势种都十分明显,它们分别对应于DGGE泳道中的亮条带。从DGGE图和系统进化树中可以看出,微单胞藻藻际优势菌群为Cyanobacteria和γ-Proteobacteria(图 7中M表示);脆杆藻藻际优势菌群为Cyanobacteria、CFB和γ-Proteobacteria(图 7中F表示);小球藻藻际优势类群为Cyanobacteria和β-Proteobacteria(图 7中C表示);四棘藻藻际优势类群为Cyanobacteria(图 7中A表示);海链藻藻际优势类群为Cyanobacteria和γ-Proteobacteria(图 7中T表示)。可以发现Cyanobacteria和γ-Proteobacteria在这几株微藻的藻际环境中广泛存在。
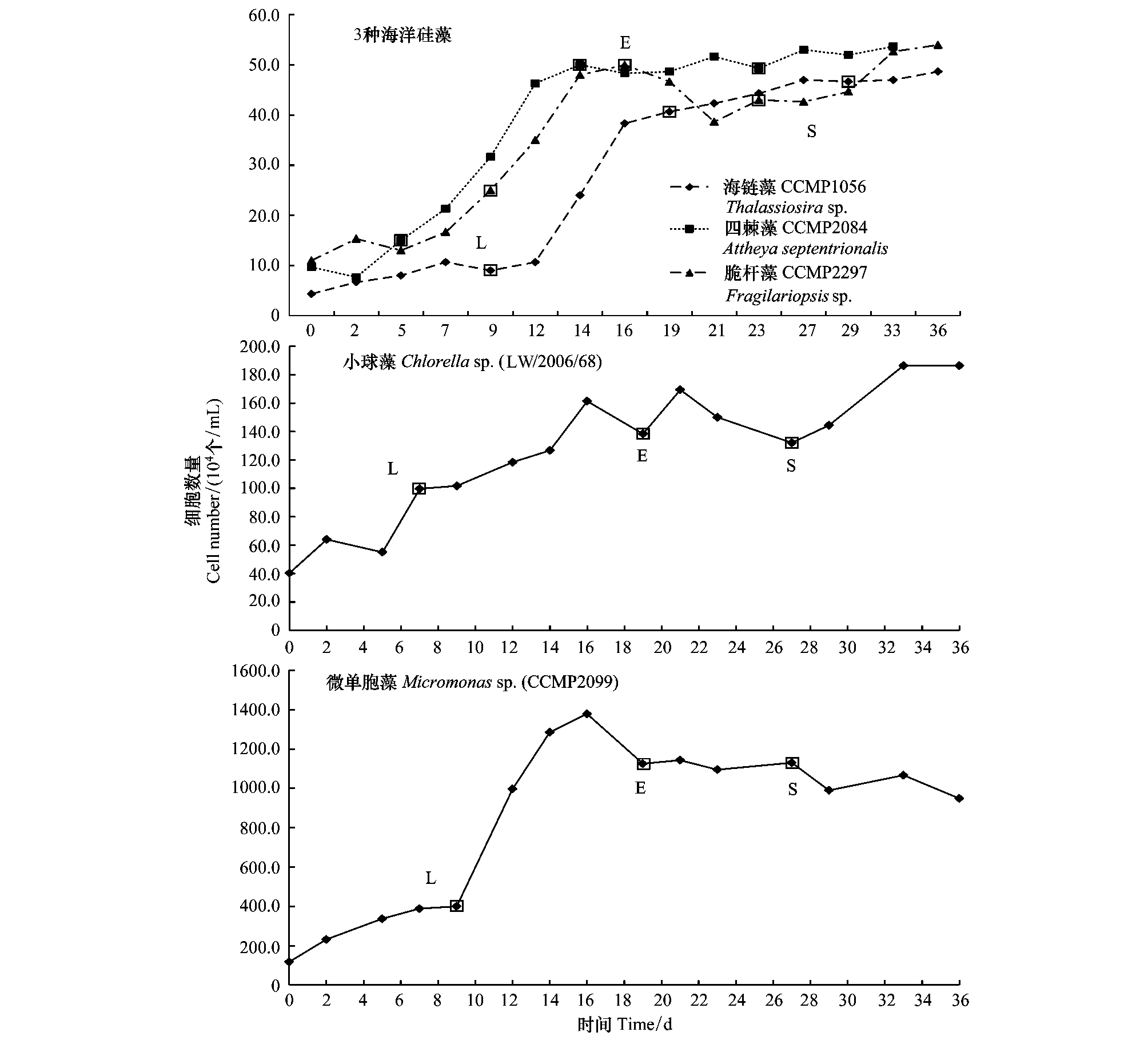
脆杆藻藻际细菌测序的DGGE条带有14条(图 2A),属于γ-Proteobacteria和Cyanobacteria的序列各有5条,属于α-Proteobacteria和CFB的序列各有2条。其中α-Proteobacteria的序列属于红细胞科的亚硫酸盐杆菌属(Sulfitobacter),γ-Proteobacteria的序列属于海洋杆菌属、希瓦菌属和假交替单胞菌科的交替假单胞菌属(Pseudoalteromonas)等属,CFB的序列属于奥文氏菌属(Owenweeksia),Cyanobacter的序列属于盐螺旋菌属(Halospirulina)。亮度较高的条带为Band_17、 Band_18、Band_19、Band_23和Band_24。Band_17属于奥文氏菌属,与香港牛尾海分离获得香港奥文氏菌(Owenweeksia hongkongensis)相似性为95%,与普拉姆岛沿岸未培养拟杆菌细菌克隆PI_4b12a相似性为99%。Band_18,Band_19属于Chroococcales目,与Halospirulina tapeticola相似度为91%,与墨西哥湾沿岸海水中未培养Cyanobacterium克隆CM01187X1E11相似性分别为99%和100%。Band_23属于海洋杆菌属,与北极海冰中的嗜冷海杆菌相似性为99%。Band_24属于Chromatiaceae纲,与海洋沉积物中加拿大希瓦菌(Shewanella canadensis)的相似性为93%,与来自密歇根湖近岸的未培养的Chromatiaceae纲的细菌克隆 Gap-2-29相似性为95%。
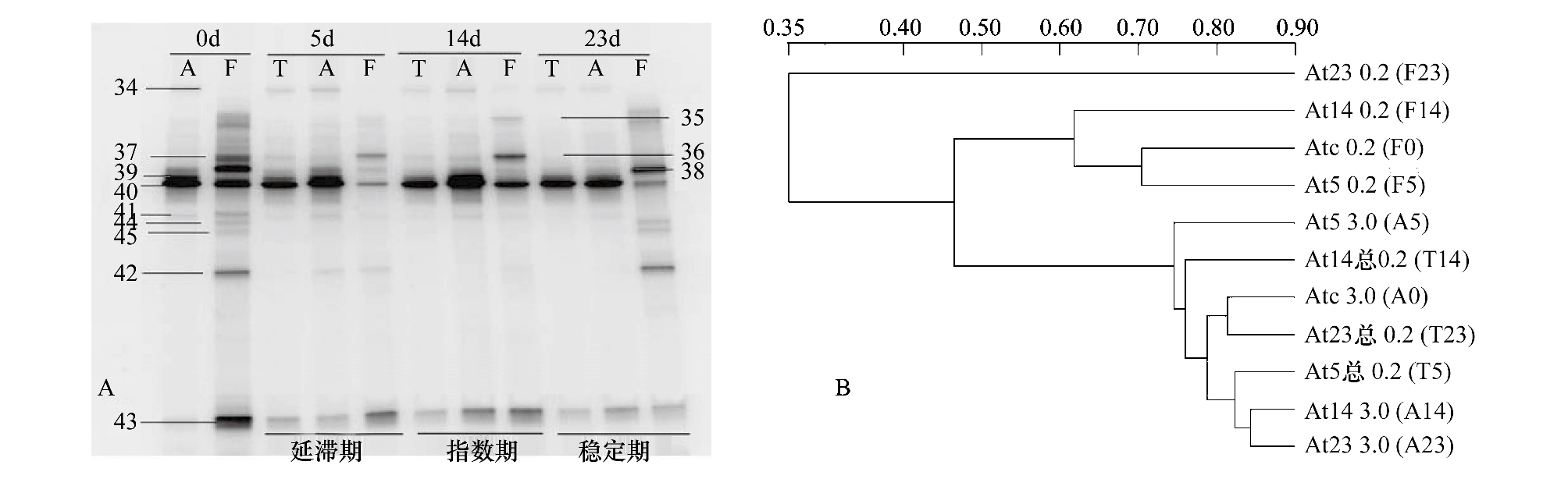
从四棘藻获得的DGGE条带有12条(图 3A),与γ-Proteobacteria相似的序列有6条,与α-Proteobacteria和Cyanobacteria相似的序列各有3条。其中α-Proteobacteria的序列属于陆丹式菌属(Loktanella),γ-Proteobacteria中的序列属于交替单胞菌科海洋杆菌属和冰居杆菌属、假交替单胞菌科的交替假单胞菌属、甲基球形菌属(Methylosphaera)和寡养单胞菌属(Stenotrophomonas)等,Cyanobacteria的序列属于盐螺旋菌属。亮度较高的条带Band_39 、Band_40均属于Chroococcales目,与已鉴定的Halospirulina tapeticola相似度为91%,与来自墨西哥湾沿岸海水未培养Cyanobacterium克隆PI_4g9g相似性为100%。
|
| 图 3 四棘藻藻际细菌群落的16S rRNA基因V3区的DGGE图谱和聚类分析图(T: 总细菌;A: 粘附细菌;F:游离细菌) Fig.3 DGGE patterns and cluster analysis of 16S rRNA gene V3 region amplification fragments of bacteria community in the phycosphere of Attheya septentrionalis |
海链藻DGGE主要条带有13条(图 4A),与γ-Proteobacteria相似的序列有7条,与α-Proteobacteria相似的序列有2条,与Cyanobacter相似的序列有4条。其中α-Proteobacteria中的序列属于红细胞科的亚硫酸盐杆菌属和赫夫勒氏菌属(Hoeflea),γ-Proteobacteria中的序列属于假单胞菌科的假单胞菌属(Pseudomonas)和铁还原单胞菌属(Ferrimonas),Cyanobacteria中的序列属于Rubidibacter属和原绿球菌属。亮度较高的条带有5条,分别是Band_49、Band_51和Band_55 属于γ-Proteobacteria,Band_54 、 Band_56 属于Cyanobacteria。Band_49、Band_51和Band_55与铁还原单胞菌属相似性最高,与日本湾Ferrimonas kyonanensis相似性分别为95%、94%和95%,Band_49与来自东部热带北太平洋的未培养γ-Proteobacteria克隆JL-ETNP-R18相似性为96%。Band_51和Band_55与来自墨西哥湾深海的未培养γ-Proteobacteria克隆OV01102/03和H10-120相似性分别为95%和96%。Band_54与Rubidibacter属相似性最高,与来自西太平洋楚克环礁海水Rubidibacter lacunae相似性为91%,与低盐度潮汐沉积物的未培养Cyanobacteria的克隆D05RT35相似性为100%。Band_56与海洋原绿球菌(Prochlorococcus marinus)相似性为87%,与海洋环境中未培养的Cyanobacterium克隆SGPW483相似性为97%。
|
| 图 4 海链藻藻际细菌群落的16S rRNA基因V3区的DGGE图谱和聚类分析图(T: 总细菌;A: 粘附细菌;F:游离细菌) Fig.4 DGGE patterns and cluster analysis of 16S rRNA gene V3 region amplification fragments of bacteria community in the phycosphere of Thalassiosira sp. |
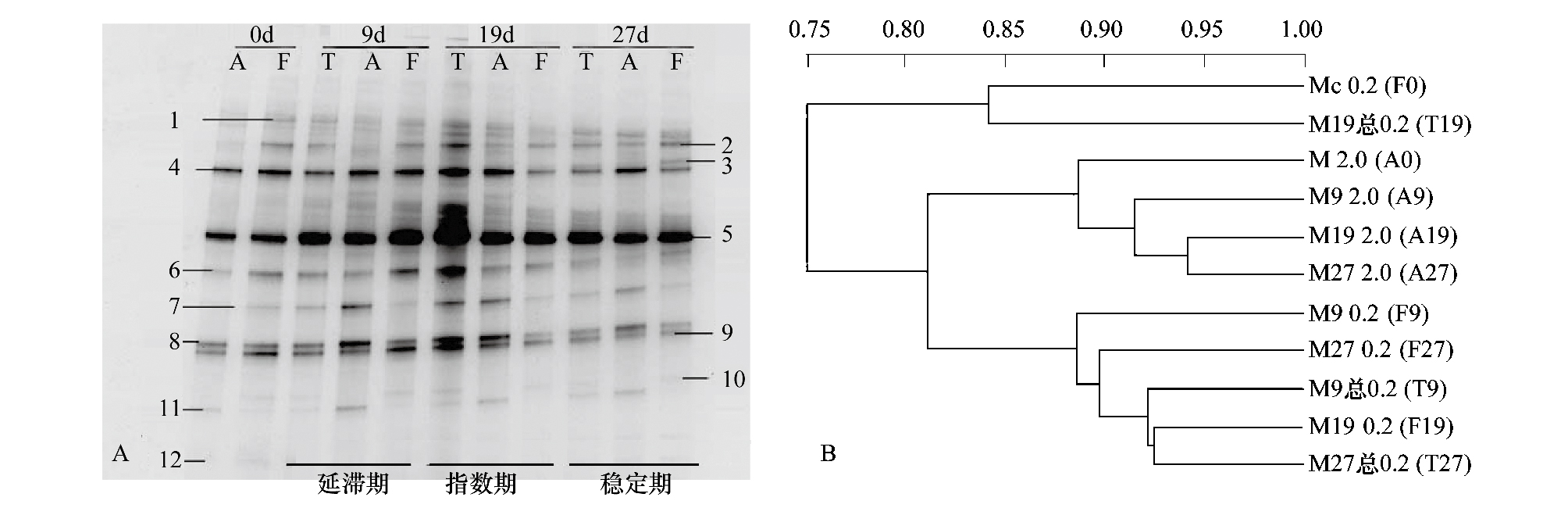
微单胞藻藻际细菌测序的DGGE条带为12条(图 5A),分别属于α-Proteobacteria(1条)、γ-Proteobacteria(6条)、 Cyanobacter(3条)和CFB(2条),其中α-Proteobacteria的序列属于红细胞科的十八杆菌属(Octadecabacter),γ-Proteobacteria的序列属于海洋杆菌(Marinobacter)、希瓦菌属(Shewanella)、嗜冷单胞菌属(Psychromonas)、深海弯菌属(Thalassolituus)和伦黑墨氏菌属(Rheinheimera)等属,CFB的序列属于Leadbetterella属,Cyanobacter的序列属于原绿球菌(Prochlorococcus)属。亮度较高的Band_4 、Band_5 、Band_8和Band_9 均属于γ-Proteobacteria。其中Band_4和Band_8与希瓦菌属(Shewanella)相似性最高,与海洋沉积物获得的加拿大希瓦菌(Shewanella canadensis)相似性分别为92%和94%,Band_4与Norweigian深海水环境的未培养细菌克隆Kwat47相似性为98%。Band_8与来自密歇根湖近岸的未培养细菌克隆Gap-2-29相似性为95%。Band_5与海洋原绿球菌(Prochlorococcus marinus)相似性为87%,与南极冰川附近海水未培养的细菌克隆IB0346E7相似性为100%。 Band_9为海洋杆菌属(Marinobacter),与来北极海冰嗜冷海杆菌(Marinobacter psychrophilus)相似性为100%。
|
| 图 5 微单胞藻藻际细菌群落的16S rRNA基因V3区的DGGE图谱和聚类分析图(T: 总细菌;A: 粘附细菌;F:游离细菌) Fig.5 DGGE patterns and cluster analysis of 16S rRNA gene V3 region amplification fragments of bacteria community in the phycosphere of Micromonas sp. |
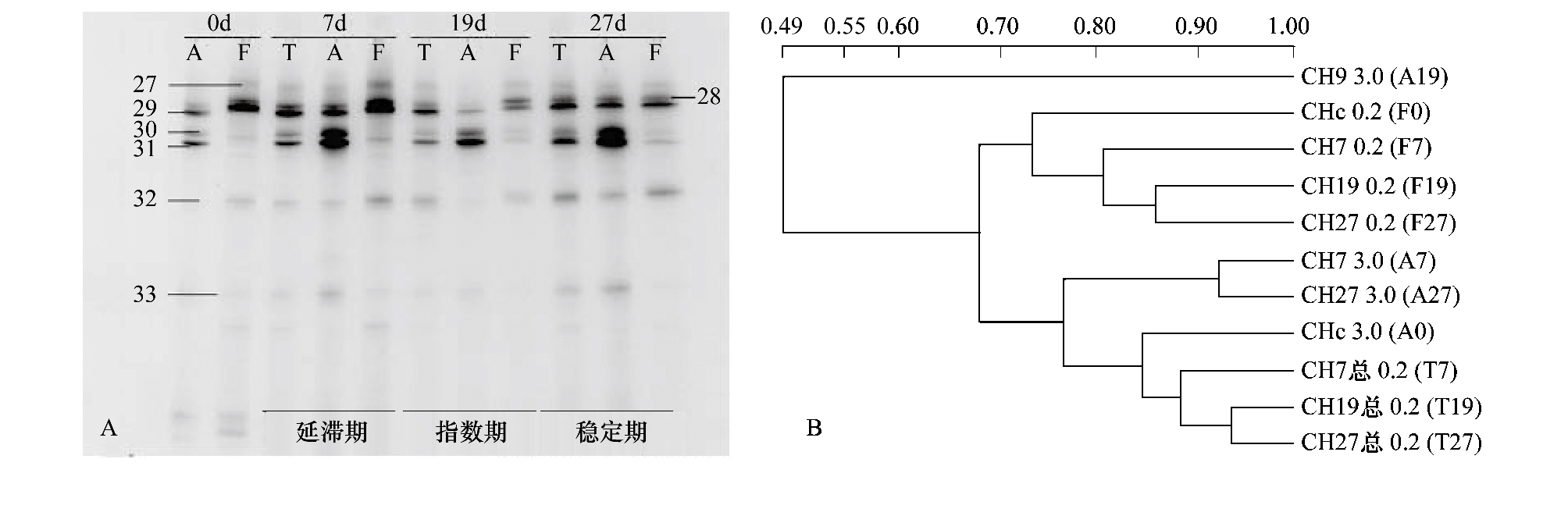
小球藻藻际细菌测序的DGGE条带有7条(图 6A),属于α-、β-Proteobacteria和Cyanobacteria的序列各有2条,仅1条属于CFB。α-Proteobacteria中的序列属于中间根瘤菌属(Mesorhizobium)和短波单胞菌属(Brevundimonas),β-Proteobacteria中的序列属于Herminiimonas属,CFB中的序列属于比自欧式属(Bizionia),Cyanobacteria中的序列属于盐螺旋菌属。亮度较高的条带Band_29、Band_30和Band_31,Band_29属于Herminiimonas属,与来自瓶装矿物水的Herminiimonas fonticola相似性为99%。Band_30、Band_31属于Chroococcales目,与Halospirulina tapeticola相似性为91%,与来自墨西哥湾沿岸海水的未培养Cyanobacterium克隆Gap-3-60相似性分别为100%和99%。
|
| 图 6 小球藻藻际细菌群落的16S rRNA基因V3区的DGGE图谱和聚类分析图(T: 总细菌;A: 粘附细菌;F:游离细菌) Fig.6 DGGE patterns and cluster analysis of 16S rRNA gene V3 region amplification fragments of bacteria community in the phycosphere of Chlorella sp. |
Cyanobacteria和α-Proteobacteria在5株微藻藻际环境中均有发现。脆杆藻、小球藻和四棘藻藻际细菌Cyanobacteria的条带相同(均为Halospirulina tapeticola),而微单胞藻和海链藻藻际细菌Cyanobacteria是有相同的条带,相同条带为Prochlorococcus marinus,与其他3株的Cyanobacteria不同。脆杆藻和海链藻藻际环境均有α-Proteobacteria的Sulfitobacter属的细菌。β-Proteobacteria只在淡水的小球藻中存在。除小球藻,其他海洋微藻均有γ-Proteobacteria存在。其中微单胞藻、脆杆藻和四棘藻藻际环境中,均有Shewanella属的细菌。而在脆杆藻、四棘藻和海链藻中有Pseudoalteromonas属的细菌。Marinobacter属的细菌在微单胞藻和脆杆藻的藻际环境中存在。
2.3 不同微藻藻际粘附细菌和游离细菌多样性微单胞藻藻际细菌16S rRNA基因DGGE电泳图谱聚类分析(图 5B)显示游离细菌和粘附细菌分别各自聚为一簇,但是两者之间的相似性指数达到80%以上,说明微单胞藻藻际游离细菌和粘附细菌无显著变化。
脆杆藻、四棘藻、海链藻和小球藻聚类分析(图 2B、图 3B、图 4B、图 6B)显示游离细菌和粘附细菌各自聚为一簇,相似性分别为45%、68%、56%和60%,说明游离细菌和粘附细菌多样性显著不同。根据图 2—图 4、图 6和表 2也可以看出,脆杆藻中游离细菌由CFB和γ-Proteobacteria组成,粘附细菌由Cyanobacteria组成;小球藻游离细菌由β-Proteobacteria组成,粘附细菌由Cyanobacteria组成;四棘藻的游离细菌由γ-Proteobacteria组成,粘附细菌由Cyanobacteria组成;海链藻的游离细菌由α-Proteobacteria和γ-Proteobacteria,粘附细菌由Cyanobacteria和α-Proteobacteria组成。
2.4 不同生长期的藻际细菌多样性脆杆藻藻际细菌聚类分析(图 2B)显示游离细菌在脆杆藻生长的延滞期(F9)、指数期(F16)与稳定期(F23)相似性在75%以上,与F0的相似性为53%,说明游离细菌的群落结构在脆杆藻不同生长期无明显变化,只是与脆杆藻刚培养时的细菌群落发生了明显变化。在延滞期时游离细菌的DGGE条带有5条,而在稳定期游离细菌的条带有8条(图 2A)。粘附细菌中,相似度最高的是稳定期(A23)和A0,为67%,与延滞期(A9)相似度最低,为50%,说明粘附细菌在脆杆藻的不同生长时期的多样性不同,在延滞期时粘附细菌的条带仅有4条,而到了稳定期粘附细菌的条带达到11条(图 2A),γ-Proteobacteria和CFB多样性增加,并且α-Proteobacteria也被检测到。生长初期游离细菌和粘附细菌的多样性有明显差异,且游离细菌多样性较为丰富。到了生长末期,游离和粘附细菌群落组成更加丰富。
四棘藻藻际细菌聚类分析(图 3B)显示游离细菌在四棘藻生长的延滞期(F5)与刚培养时(F0)相似度在80%以上,而F5和F0与指数期(F14)聚在一起,相似度在70%以上,而这3个时期的游离细菌与稳定期的相似度最低,仅为25%。粘附细菌中指数期(A14)和稳定期(A23)相似度在90%以上,与延滞期(A5)的相似度也在85%以上,说明游离细菌或粘附细菌多样性在四棘藻各个生长期并没有太大的变化。Band_39、Band_40微藻各个生长阶段的粘附状态的丰度比游离状态的多。
海链藻藻际细菌聚类分析(图 4B)显示,粘附细菌中,指数期(A19)和延滞期(A9)相似度为95%,这两个时期与稳定期的相似度为75%。游离细菌中,稳定期(F29)和指数期(F19)相似度为85%,与延滞期的相似度也在73%。说明粘附细菌和游离细菌的多样性在整个生长周期无明显变化。DGGE图谱(图 4A)显示了丰富的海链藻藻际细菌的多样性,优势关联菌群在整个生长周期无明显变化。其中主要关联菌群如(Band_49,Band_56)粘附状态的丰度比游离状态的少,而Band_51,Band_54,Band_55作为粘附状态的丰度则比游离状态的多。
微单胞藻藻际细菌聚类分析(图 5B)显示游离细菌在微单胞藻生长的延滞期(F9)、指数期(F19)和稳定期(F27)相似度在85%以上,粘附细菌在微单胞藻生长延滞期(A9)和稳定期(A27)相似度为93%,而微单胞藻延滞期和稳定期的粘附细菌与指数期(A19)相似度最低。说明微单胞藻整个生长周期中,游离细菌和粘附细菌结构稳定。
小球藻藻际细菌聚类分析(图 6B)显示游离细菌在小球藻生长的稳定期(F27)和指数期(F19)相似度最高,为86%,然后小球藻生长的延滞期和稳定期的游离细菌与延滞期(F7)聚在一起,相似度也在80%以上。粘附细菌在小球藻生长的延滞期(A7)和稳定期(A27)聚在一起,相似度为93%,小球藻在这两个时期的粘附细菌与指数期(A19)相似度最低,为49%。说明小球藻整个生长周期的关联菌群多样性和优势关联菌群均较为稳定,无显著变化,但是粘附细菌和游离细菌的多样性和优势菌群则明显不一致(图 6A)。
|
| 图 7 5种微藻藻际细菌的16S rRNA 基因V3区序列系统发育树 Fig.7 Phylogenetic trees of bacterial 16S rRNA V3 gene sequences obtained from phycosphere of five microalgae 系统发育树采用邻位法构建,分支上的数字为1000次自展值分析所得大于55%的值;线段代表进化距离;本研究中获得的序列用黑体表示;与藻相关的DGGE的主要条带用带字幕的彩圈标记;A、C、F、M和T分别代表四棘藻、小球藻、脆杆藻、微单胞藻和海链藻 |
采用PCR-DGGE方法对北极不同生境的5株微藻藻际环境微生物多样性进行了序列系统发育分析。研究表明,5株北极微藻藻际细菌具有显著的种间特异性,每个藻株的主要附生微生物群落差异显著。Schfer等通过PCR-DGGE的方法对6株海洋硅藻的附生细菌群落进行分析,6株硅藻具有不同的附生细菌群落,在6株硅藻中检测出的主要类型都是特有的,只有少数类型在多个硅藻中共同存在[4]。不同微藻藻际细菌群落结构的不同预示着微藻提供为这些细菌提供了特异性的生存条件,从而对细菌进行选择。研究的3株硅藻藻际细菌γ-Proteobacteria占优势,α-Proteobacteria占少数,在四棘藻中没有检测出CFB,这与Schfer等研究的硅藻(双尾藻、威氏海链藻、冰河拟星杆藻、聚生角毛藻和圆筛藻)的藻际细菌主要由α-Proteobacteria和CFB占优势不同,预示着尽管是相同纲的藻株,其藻际细菌群落结构差异明显。
根据表 2和图 5可以看出,微单胞藻藻际细菌为α-Proteobacteria、γ-Proteobacteria、Cyanobacteria和CFB菌群四类。β-Proteobacteria通常在淡水环境中的非常丰富,多数情况下在海洋样品中的含量是很低或者是无法检测出来的,因此β-Proteobacteria不是典型的海洋浮游细菌[21, 22]。从淡水藻小球藻藻际细菌发现β-Proteobacteria存在,但是无γ-Proteobacteria。Fisher等对3株淡水绿藻Desmidium grevillii、Hyalotheca dissiliens和Spondylosium pulchrum研究显示克隆的序列中存在β-Proteobacteria[23]。这些结果说明β-Proteobacteria在淡水环境中广泛存在,γ-Proteobacteria是典型的海洋细菌,尽管在海洋中大量存在,但是在本研究中的小球藻藻际环境中没有检测到,预示了γ-Proteobacteria可能在小球藻-细菌间的相互作用并不重要。
Grossart等研究圆海链藻和骨条藻这两株硅藻藻际细菌多样性,发现细菌主要属于α-Proteobacteria、γ-Proteobacteria 和CFB菌群3类,并且α-Proteobacteria的Roseobacter在这两株硅藻都有存在[5]。在本研究中Cyanobacteria、α-Proteobacteria、γ-Proteobacteria在3株硅藻:脆杆藻、四棘藻和海链藻藻际环境中均有分布,CFB菌群仅在脆杆藻中存在。多种藻类藻际细菌的系统发育分析反映了α-Proteobacteria、γ-Proteobacteria和CFB广泛存在。
在5株微藻藻际环境中都检测到Cyanobacteria。在多数极地环境中Cyanobacteria不仅在光合自养微生物占优势地位,而且构成了微生物生态系统的生物量。有报道在北极海洋存在Cyanobacteria,但是很少是浮游植物的主要成员[24]。
3.2 不同微藻藻际粘附细菌和游离细菌多样性微单胞藻细菌结构稳定,游离细菌和粘附细菌的群落结构组成差异并不明显,几乎由相同的种类组成,仅在丰度上存在细微的差异(游离细菌和粘附细菌的一些条带在亮度上有差异,如条带4在微单胞藻指数期时的条带)。从图 5A中可以看到,各个条带在游离细菌和粘附细菌中均存在,说明在微藻培养液中,细菌菌群在游离细菌和粘附细菌中都没有明显的差异。
脆杆藻、小球藻、四棘藻和海链藻游离细菌和粘附细菌的群落结构组成不同,游离细菌主要由α-Proteobacteria和γ-Proteobacteria,而粘附细菌主要由Cyanobacteria组成。该结果与Fandino和Grossart研究结果一致[3, 5]。在腰鞭毛藻藻际环境中,游离细菌主要由α-和γ-Proteobacteria组成,而粘附细菌则由CFB构成[3],在硅藻藻际环境中,游离细菌由α-Proteobacteria组成,粘附细菌由CFB构成[5],说明至少在这两类微藻中游离细菌、粘附细菌的组成是相似的。并且也有研究表明不同藻种来源的藻际细菌也是不同的。在海洋环境中,硅藻藻际粘附细菌主要由γ-Proteobacteria和CFB组成[25],在湖泊与河流中,硅藻藻际粘附细菌则主要由β-Proteobacteria组成[26, 27]。因此,推测微藻藻际环境中的细菌类群可能与所处的环境相关。
3.3 不同生长期藻际细菌多样性微单胞藻、小球藻、四棘藻和海链藻各生长期藻际微生物的DGGE图谱比较稳定,说明微生物多样性变化不大,主要差别仅为游离细菌和粘附细菌丰度上有所不同。该结果与Schfer的研究结果较一致,Schfer发现藻际微生物的DGGE图谱在整个生长期非常稳定,仅发现2株硅藻海链藻(Thalassiosira weissflogii)和角毛藻(Chaetoceros socialis)存在的微小差异,在不同的时间条带亮度不同。
脆杆藻藻际细菌的研究结果则显示粘附细菌的多样性在延滞期、指数期和稳定期有显著不同;游离细菌只是在接种时不同,在延滞期、指数期和稳定期细菌群落结构无明显差异。在延滞期时,脆杆藻粘附细菌的条带仅有4条,游离细菌的条带有5条,而到了稳定期粘附细菌的条带达到11条,游离细菌的条带有8条,在稳定生长期新出现了α-Proteobacteria的Sulfitobacter属和γ-Proteobacteria的Shewanella属细菌。Riemann等在研究由海链藻主导的硅藻赤潮时发现,在赤潮到达顶峰时细菌的3种主要种群消失,随后新的种群出现,主要是由特化的α-Proteobacteria和CFB相关细菌组成,其中α-Proteobacteria主要是玫瑰杆菌属(Roseobacter)的细菌,而CFB则分布在Flexibacter属、Cytophaga属和勒温氏菌属(Lewinella)[7]。Grossart等对圆海链藻的藻际细菌多样性研究表明,藻际指数期和稳定期的细菌多样性显著不同,在稳定期检测到细菌数量明显最多,α-Proteobacteria主要由Roseobacter组成,第二大类CFB则主要有Brumimicrobium属和极地杆菌属(Polaribacter)。α-Proteobacteria的数量在指数生长期由22%增长到31%,而在稳定生长期进一步增长至41%[5]。说明海链藻的藻际细菌群落结构相似,主要的关联群落主要是α-Proteobacteria和CFB组成,而脆杆藻的藻际细菌群落结构则明显不同。杨晓茹对塔玛亚历山大藻的微生物多样性研究也表明指数期的细菌多样性远低于延滞期和稳定期[8]。这些研究均与本研究一致,在微藻从刚开始培养至稳定期的转变过程中,微藻释放的有机物发现了变化,从而导致游离和粘附细菌的群落结构均发生了变化。α-Proteobacteria的多样性和数量在稳定生长期增长,预示了α-Proteobacteria更能适应稳定期的微藻藻际环境。
致谢: 感谢中国极地研究中心张瑾实验员对研究的帮助。
| [1] | Bell W, Mitchell R. Chemotactic and growth responses of marine bacteria to algal extracellular products. Biological Bulletin, 1972, 143(2): 265-277. |
| [2] | De Long E F, Franks D G, Alldredge A L. Phylogenetic diversity of aggregate-attached vs. free-living marine bacterial assemblages. Limnology and Oceanography, 1993, 38(5): 924-934. |
| [3] | Fandino L B, Riemann L, Steward G F, Long R A, Azam F. Variations in bacterial community structure during a dinoflagellate bloom analyzed by DGGE and 16S rDNA sequencing. Aquatic Microbial Ecology, 2001, 23(2): 119-130. |
| [4] | Schäfer H, Abbas B, Witte H, Muyzer G. Genetic diversity of 'satellite’ bacteria present in cultures of marine diatoms. FEMS Microbiology Ecology, 2002, 42(1): 25-35. |
| [5] | Grossart H P, Levold F, Allgaier M, Simon M, Brinkhoff T. Marine diatom species harbour distinct bacterial communities. Environmental Microbiology, 2005, 7(6): 860-873. |
| [6] | Rooney-Varga J N, Giewat M W, Savin M C, Sood S, LeGresley M, Martin J L. Links between phytoplankton and bacterial community dynamics in a coastal marine environment. Microbial Ecology, 2005, 49(1): 163-175. |
| [7] | Riemann L, Steward G F, Azam F. Dynamics of bacterial community composition and activity during a mesocosm diatom bloom. Applied and Environmental Microbiology, 2000, 66(2): 578-587. |
| [8] | 杨小茹, 苏建强, 郑小伟, 周月霞, 田蕴, 宁修仁, 郑天凌. 基于分子技术的1株产毒藻藻际细菌多样性分析. 环境科学, 2009, 30(1): 271-279. |
| [9] | Bates S S, Gaudet J, Kaczmarska I, Ehrman J M. Interaction between bacteria and the domoic-acid-producing diatom Pseudo-nitzschia multiseries (Hasle) Hasle; can bacteria produce domoic acid autonomously? Harmful Algae, 2004, 3(1): 11-20. |
| [10] | 王新, 周立红, 郑天凌, 宁修仁. 塔玛亚历山大藻藻际细菌溶藻过程. 生态学报, 2007, 27(7): 2864-2871. |
| [11] | Ferrier M, Martin J L, Rooney-Varga J N. Stimulation of Alexandrium fundyense growth by bacterial assemblages from the Bay of Fundy. Journal of Applied Microbiology, 2002, 92(4): 706-716. |
| [12] | Bano N, Hollibaugh J T. Phylogenetic composition of bacterioplankton assemblages from the Arctic Ocean. Applied and Environmental Microbiology, 2002, 68(2): 505-518. |
| [13] | Brinkmeyer R, Knittel K, Jürgens J, Weyland H, Amann R, Helmke E. Diversity and Structure of Bacterial Communities in Arctic versus Antarctic Pack Ice. Applied and Environmental Microbiology, 2003, 69(11): 6610-6619. |
| [14] | Junge K, Imhoff F, Staley T, Deming W. Phylogenetic diversity of numerically important arctic sea-ice bacteria cultured at subzero temperature. Microbial Ecology, 2002, 43(3): 315-328. |
| [15] | Groudieva T, Kambourova M, Yusef H, Royter M, Grote R, Trinks H, Antranikian G. Diversity and cold-active hydrolytic enzymes of culturable bacteria associated with Arctic sea ice, Spitzbergen. Extremophiles, 2004, 8(6): 475-488. |
| [16] | 侯建军, 黄邦钦, 戴相辉. 赤潮藻细胞计数方法比较研究. 中国公共卫生, 2004, 20(8): 907-908. |
| [17] | Zhou J Z, Bruns M A, Tiedje J M. DNA recovery from soils of diverse composition. Applied and Environmental Microbiology, 1996, 62(2): 16-322. |
| [18] | Bosshard P P, Santini Y, Grüter D, Stettler R, Bachofen R. Bacterial diversity and community composition in the chemocline of the meromictic alpine Lake Cadagno as revealed by 16S rDNA analysis. FEMS Microbiology Ecology, 2000, 31(2): 173-182. |
| [19] | Dar S A, Kuenen J G, Muyzer G. Nested PCR-denaturing gradient gel electrophoresis approach to determine the diversity of sulfate-reducing bacteria in complex microbial communities. Applied and Environmental Microbiology, 2005, 71(5): 2325-2330. |
| [20] | Luo W, Li H R, Cai M H, He J F. Diversity of microbial eukaryotes in Kongsfjorden, Svalbard. Hydrobiologia, 2009, 636(1): 233-248. |
| [21] | Nold S C, Zwart G. Patterns and governing forces in aquatic microbial communities. Aquatic Ecology, 1998, 32(1): 17-35. |
| [22] | GlöCkner F O, Fuchs B M, Amann R. Bacterioplankton compositions of lakes and oceans: A first comparison based on fluorescence in situ hybridization. Applied and Environmental Microbiology, 1999, 65(8): 3721-3726. |
| [23] | Fisher M M, Wilcox L W, Graham L E. Molecular characterization of epiphytic bacterial communities on charophycean green algae. Applied and Environmental Microbiology, 1998, 64(11): 4384-4389. |
| [24] | Vincent W F. Cyanobacterial dominance in the polar regions//Whitton B A, Potts M, eds. The Ecology of Cyanobacteria: Their Diversity in Time and Space. Dordrecht: Kluwer, 2002: 321-340. |
| [25] | Bidle K D, Azam F. Bacterial control of silicon regeneration from diatom detritus: significance of bacterial ectohydrolases and species identity. Limnology and Oceanography, 2001, 46(7): 1606-1623. |
| [26] | Grossart H P, Simon M. Bacterial colonization and microbial decomposition of limnetic organic aggregates (lake snow). Aquatic Microbial Ecology, 1998, 15(2): 127-140. |
| [27] | Böckelmann U, Manz W, Neu T R, Szewzyk U. Characterization of the microbial community of lotic organic aggregates ('river snow’) in the Elbe river of Germany by cultivation and molecular methods. FEMS Microbiology Ecology, 2000, 33(2): 157-170. |
 2015, Vol. 35
2015, Vol. 35