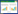文章信息
- 胡常巨, 熊金波, 陈和平, 王凯, 叶然, 崔永平, 朱建林, 张德民
- HU Changju, XIONG Jinbo, CHEN Heping, WANG Kai, YE Ran, CUI Yongping, ZHU Jianlin, ZHANG Demin
- 象山港网箱养殖区与非养殖区的细菌群落分布
- Distribution of bacterioplankton communities in cage culture and non-cultured areas of Xiangshan Bay, Ningbo, China
- 生态学报, 2015, 35(24): 8053-8061
- Acta Ecologica Sinica, 2015, 35(24): 8053-8061
- http://dx.doi.org/10.5846/stxb201406171263
-
文章历史
- 收稿日期: 2014-06-17
- 网络出版日期: 2015-05-21
2. 宁波大学建筑工程与环境学院, 宁波 315211;
3. 宁波海洋环境监测中心站, 宁波 315012
2. Faculty of Architectural, Civil Engineering and Environment, Ningbo University, Ningbo 315211, China;
3. Marine Environmental Monitoring Center of Ningbo, SOA, Ningbo 315012, China
在养殖环境中,浮游细菌既是各种有机物质的分解者和转化者,又是物质和能量的贮存者,在物质循环和能量流动中具有无可替代的作用[5, 6]。同时,有些细菌还是近海养殖的致病菌,如弧菌是最常见的条件致病菌之一。在养殖环境条件恶化时,弧菌大量增殖,可导致养殖生物感染发病[7, 8]。我们的前期工作发现,浮游细菌的群落组成与对虾养殖塘的健康状况密切相关[9],且细菌群落的变化趋势是可以预测的[10]。目前有大量研究表明微生物是以群落的形式整体承担重要的生态功能,群落响应可以综合全面地反映环境扰动[9],如微生物群落结构能对水温、pH值、溶解氧、氨氮、磷酸盐、总有机碳、化学需氧量和深度等因素的变化产生快速响应[11, 12, 13, 14, 15]。因此,在近海养殖生态系统中,水体细菌的群落结构和多样性有可能作为评价海水养殖生态系健康和稳定的重要指标[16, 17]。
象山港是浙江省重要的养殖基地[18]。然而,以往对象山港生态环境的报道多以潮间带生物、浮游动植物和底栖动物为主,对养殖环境中微生物的研究也主要集中在底泥细菌群落以及水体中浮游细菌的数量方面[3, 18, 19, 20, 21],有关浮游细菌群落结构方面的研究也较少。本研究通过16S rRNA基因焦磷酸测序技术研究象山港网箱养殖和非养殖区的浮游细菌群落,比较不同水深度下的细菌群落结构,获得驱动浮游细菌群落结构变异的环境因子,初步揭示了浮游细菌群落对养殖扰动的响应规律。结合不同细菌类群的功能,通过群落组成变化推测生态功能变化,可为网箱养殖生态健康提供评价和维护依据。
1 实验方法 1.1 实验设计和水样采集"象山港位于浙江省宁波市的东部,为东北-西南向的狭长形半封闭海湾。为研究养殖活动对浮游细菌群落的影响,2012年4月10日,选取位于象山港中西沪港内网箱养殖区(121°45′9.935″ E,29°32′20.472″ N)和非养殖区(121°46′10.056″ E,29°36′4.356″ N)两个站位,其具体时间为15:40和16:10,潮位均为低平一落,两位点相距7 km。养殖区面积约1.1 km2,拥有海水网箱2000余只,网箱规格为3 m × 3 m × 6 m,主要养殖种类为大黄鱼和鲈鱼,平均放养密度为3000尾/网箱,养殖周期为2—3a。每个区域分别采集表(水深0.5 m)、中(水深2.5 m)、底(水深8 m)3个深度的水样3 L。
每个点位设置两个重复。采集的水样置于预先灭菌容器中存于冰盒,4 h内运回实验室进行检测分析。1 L水样用于提取细菌总DNA,剩余水样用于水质指标测定。
现场测定水温、盐度和pH值。其它水质指标测定方法参照《海洋监测规范》(GB 17378.4—2007):溶解氧(DO)用碘量法;化学需氧量(COD)用碱性高锰酸钾法;叶绿素a(Chl a)采用丙酮萃取分光光度法;铵盐(NH4+)用次溴酸钠氧化法;亚硝酸盐(NO2-)分析用重氮-偶氮法;硝酸盐(NO3-)用锌镉还原法;总氮(TN)与总磷(TP)用过硫酸钾氧化法;磷酸盐(PO43-)用抗坏血酸还原磷钼蓝法;总有机碳(TOC)用总有机碳仪器法。
1.2 DNA提取分别取各样品1 L水样,用经灭菌处理的100 μm孔径的尼龙布预过滤,后用0.2 μm孔径的聚碳酸酯膜(Millipore)过滤。滤膜在无菌剪刀剪碎后用Power Soil DNA试剂盒(Mobio)提取收集物的总DNA。用NanoDrop ND-1000型分光光度计测定DNA浓度和纯度。抽提后的DNA于-80 ℃备用。
1.3 细菌16S rRNA基因扩增和454测序PCR扩增16S rRNA基因的V1-V3可变区(27F:AGAGTTTGATCMTGGCTCAG和519R: GWATTACCGCGGCKGCTG),其中正向引物上带有10 个碱基的barcode序列,用于区分不同样品,DNA模板用量为50 ng,在50 μL反应体系扩增。每个样品设置3个PCR重复,以降低扩增造成的偏差。PCR反应条件如下:94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,30个循环,72 ℃延伸10 min。扩增产物用1%琼脂糖凝胶电泳检测。将每个样品的重复扩增产物混合后纯化(TaKaRa Biotech,Japan)。最后所有样品的PCR产物等摩尔量混合,在罗氏FLX 454仪器上测序(Roche Diagnostics,Branford,CT,USA)。
1.4 测序数据处理利用Qiime(Quantitative Insights Into Microbial Ecology,http://qiime.org)流程对下机测序数据进行样品分装、去嵌合体和质量筛选;利用Uclust将相似性大于97%的序列归为同一个分类操作单元(Operational Taxonomic Units,OTUs)[22]。选取每个OTU中丰度最高的序列作为代表序列[23],通过 Greengenes数据库中对比获得OTUs的分类地位信息[24]。为避免测序深度不同造成的偏差,每个样品随机选取5700条序列(样品中最低测度深度)进行后续分析。
1.5 数据分析用R软件(v.2.11.0)中vegan软件包计算养殖水环境中浮游细菌群落的多样性,包括多样性指数(Shannon Wiener)和均匀度指数(Pilou′s Evenness)。应用冗余分析方法(Redundancy analysis,RDA)分析细菌群落与环境因子之间的关系[25],同时采用偏冗余分析(Partial redundancy analysis,pRDA),计算出水体理化因子和空间距离对群落变异的相对贡献。
用Past(v.2.03)软件中非参数多维度法(Non-metric multidimensional scaling,NMDS)分析浮游细菌的群落组成,将各样品浮游细菌群落组成在二维图上可视化。NMDS中,一般采用压力系数(Stress)评定所得到的多维构型与实际数据之间的适合度,压力系数越小,表示适合度越高[25]。根据Kruskal提出的判断标准,压力系数<0.2,适合度较好,聚类分析可靠[26]。应用群落相似性检验分析养殖和水深对浮游细菌群落分布的影响。利用SIMPER(Similarity percentage)分析找出对群落差异贡献度高的OTUs。
2 结果 2.1 水体理化性质主要结果表明:在网箱养殖区中,水温、磷酸盐、总磷、亚硝酸盐和铵盐的均值较高,化学需氧量显著高于非养殖区(t检验,P = 0.004);与网箱养殖区相比,非养殖区中叶绿素a和总有机碳较高(表 1)。在不同水体中,化学需氧量均随水深逐渐递减,而总有机碳随水深逐渐增加,其它环境因子在水深层次上分布规律不明显(表 1)。
| 站位 Sites | 水温 Temperature/ ℃ | 化学需氧量 COD/ (mg/L) | 磷酸盐 PO43-/ (mg/L) | 总磷 TP/ (mg/L) | 亚硝酸盐 NO-2/ (mg/L) | 铵盐 NH4+/ (mg/L) | 硝酸盐 NO-3/ (mg/L) | 总氮 TN/ (mg/L) | 总有机碳 TOC/ (mg/L) | 叶绿素a Chl a/ (μg/L) |
| 养殖区表层YS | 14.59 | 1.14 | 0.056 | 0.113 | 0.006 | 0.026 | 0.72 | 1.20 | 1.56 | 2.3 |
| 养殖区中层YM | 14.53 | 1.09 | 0.047 | 0.116 | 0.005 | 0.024 | 0.64 | 1.14 | 1.60 | 2.5 |
| 养殖区底层YB | 14.48 | 1.02 | 0.047 | 0.171 | 0.004 | 0.020 | 0.64 | 0.84 | 1.93 | 0.8 |
| 非养殖区表层NS | 15.52 | 0.87 | 0.043 | 0.064 | 0.004 | 0.023 | 0.69 | 0.74 | 1.84 | 2.7 |
| 非养殖区中层NM | 14.46 | 0.81 | 0.042 | 0.084 | 0.004 | 0.020 | 0.67 | 1.12 | 1.96 | 2.7 |
| 非养殖区底层NB | 14.39 | 0.77 | 0.042 | 0.162 | 0.003 | 0.022 | 0.73 | 1.64 | 2.44 | 1.8 |
| COD:Chemical oxygen demand; TP:Total phosphorus; TN:Total nitrogen; TOC:Total organic carbon; Chl a:Chlorophyll a; Y:养殖区Cage culture area; N:非养殖区Non-cultured area; S:表层Surface; M:中层Middle; B:底层Bottom | ||||||||||
象山港水体主要浮游细菌类群为γ-变形菌纲 (Gammaproteobacteria)、α-变形菌纲(Alphaproteobacteria)、 拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、其它变形菌门(Unclassified Proteobacteria)、β-变形菌纲 (Betaproteobacteria)和ε-变形菌纲(Epsilonproteobacteria),平均相对丰度分别为47.48%、39.45%、7.76%、1.32%、1.13%、0.87%和0.63%,占细菌总数的98.64%(图 1)。在网箱养殖区,拟杆菌门(P = 0.006)和放线菌门(P = 0.015)显著高于非养殖区,而γ-变形菌纲(P = 0.038)显著低于非养殖区。在网箱养殖区,α-变形菌平均相对丰度随水深逐渐降低,而γ-变形菌纲逐渐增加;在非养殖区,拟杆菌平均相对丰度随水深逐渐降低(图 1)。

|
| 图1 各样品的优势菌门 (纲) 平均相对丰度图 Fig.1 Relative abundances of the dominant phyla or classes in water samples Y:cage culture area,养殖区;N:non-cultured area,非养殖区;S:surface,表层;M:middle,中层;B:bottom,底层 |
总体而言,浮游细菌群落多样性指数(t检验,P = 0.790)和均匀度指数(t检验,P = 0.587)在网箱养殖区中的均值高于非养殖区,但无显著差异。网箱区中层水样浮游细菌的多样性指数最高,表层水最低,分别为4.808,4.104;均匀度指数也是中层水最高(0.735),表层水最低(0.652)(表 2)。在非养殖区,则是表层水多样性最高,底层水多样性最低,但是这些差别均不显著(表 2)。
| 养殖Culture | 水深Depth | 多样性指数Shannon Wiener | 均匀度指数Pilou′s Evenness |
| 网箱养殖区 | 表层 | 4.104±0.085b | 0.652±0.008b |
| Cage culture area | 中层 | 4.808±0.395a | 0.735±0.027a |
| 底层 | 4.748±0.388ab | 0.708±0.043ab | |
| 非养殖区 | 表层 | 4.602±0.115ab | 0.704±0.015ab |
| Non-cultured area | 中层 | 4.586±0.067ab | 0.698±0.016ab |
| 底层 | 4.338±0.349ab | 0.664±0.043b | |
| 养殖Culture | F | 0.078 | 0.329 |
| P | 0.790 | 0.587 | |
| 水深Depth | F | 1.571 | 1.984 |
| P | 0.283 | 0.218 | |
| 养殖×水深Culture×Depth | F | 3.034 | 3.397 |
| P | 0.123 | 0.103 | |
| 同列中不同字母代表的值表示有显著差异 | |||
利用NMDS分析浮游细菌群落组成,压力系数为0.16,说明可以较好地反映各采样点间浮游细菌群落的相似性(图 2)。各样品在图中的相对距离可以反映所对应的浮游细菌群落组成的相似程度[15],可将所有样品大致分为3类,网箱区样品为一类,非养殖区中层和底层样品成一类,非养殖区表层样品聚为一类(图 2)。此外,沿着第一排序轴网箱区和非养殖区的细菌群落分布规律均由表层向底层,具有相似的演替轨迹(图 2)。
|
| 图2 浮游细菌群落组成的非度量尺度分析NMDS二维图 (箭头表示从表层到底层的变化方向) Fig.2 Two-dimensional NMDS of bacteriolplankton community compositions (The arrows representing changes from surface to bottom) NMDS: 非参数多维度法Non-metric multidimensional scaling |
群落相似性检验分析发现网箱区和非养殖区浮游细菌群落分布差异显著(P = 0.023);不同水深层次的细菌群落差异不显著(P = 0.070),表明水深对浮游细菌群落分布影响较小。SIMPER分析表明网箱养殖区和非养殖区的细菌群落差异度为62.41。对群落差异度贡献率较大的11个OTUs(贡献率>1%),累积贡献率达到45.02%,其中海单胞菌属(Marinomonas)(2个OTUs)、褐杆菌属(Phaeobacter)(1个OTU)、红杆菌科(Rhodobacteraceae)(3个OTUs)、交替假单胞菌属(Pseudoalteromonas)(1个OTU)、Sediminicola sp.(1个OTU)、鞘脂单胞菌目(Sphingomonadales)(1个OTU)、冰居菌属(Glaciecola)(1个OTU)和弧菌科(Vibrionaceae)(1个OTU)(表 3)。对造成群落差异的主要OTUs在网箱养殖区和非养殖区进行丰度比较,发现黄杆菌科(Flavobacteriaceae)(t检验,P = 0.012)和褐杆菌属(t检验,P = 0.041)的平均相对丰度在网箱养殖区显著高于非养殖区,而交替假单胞菌属和弧菌科的平均相对丰度在非养殖区中较高。
| 操作分类单元OTUs | 分类Taxa | 贡献率Contribution/% |
| OTU 2597 | Alphaproteobacteria; Rhodobacterales; Rhodobacteraceae; Phaeobacter | 8.21 |
| OTU 1392 | Alphaproteobacteria; Rhodobacterales; Rhodobacteraceae | 6.41 |
| OTU 3757 | Alphaproteobacteria; Rhodobacterales; Rhodobacteraceae | 3.88 |
| OTU 4032 | Alphaproteobacteria; Rhodobacterales; Rhodobacteraceae | 1.16 |
| OTU 4384 | Alphaproteobacteria; Sphingomonadales | 2.36 |
| OTU 4336 | Bacteroidetes; Flavobacteriia; Flavobacteriales; Flavobacteriaceae; Sediminicola | 2.11 |
| OTU 3560 | Gammaproteobacteria; Alteromonadales; Pseudoalteromonadaceae; Pseudoalteromonas | 3.21 |
| OTU 927 | Gammaproteobacteria; Alteromonadales; Alteromonadaceae; Glaciecola | 1.88 |
| OTU 1873 | Gammaproteobacteria; Oceanospirillales; Oceanospirillaceae; Marinomonas | 3.09 |
| OTU 2203 | Gammaproteobacteria; Oceanospirillales; Oceanospirillaceae; Marinomonas | 11.12 |
| OTU 2233 | Gammaproteobacteria; Vibrionales; Vibrionaceae | 1.59 |
| SIMPER: Similarity percentage; OTUs: Operational Taxonomic Units | ||
浮游细菌群落结构的降趋势对应分析(DCA)结果显示,DCA排序前4个轴中最大值小于3,适合冗余分析[25]。对环境因子进行标准化处理、蒙特尔检验(Mantel test,permu=999)和去除膨胀因子大于20的环境因子[27],最终挑选出与群落变异显著相关的4个环境因子,COD(P = 0.003)、PO43-(P = 0.027)、NH4+(P = 0.016)和TOC(P = 0.022)。
第一排序轴(RDA1)的主要环境因子为COD(负相关,r = - 0.89)、TOC(正相关,r = 0.71)和PO43-(负相关,r = - 0.60),第二排序轴(RDA2)的主要环境因子为NH4+(正相关,r = 0.87),共同解释了21.59%的群落变异(图 3)。从箭头的连线长度看出,环境因子对浮游细菌群落结构影响的相关性为COD最大,TOC次之,PO43-和NH4+较小(图 3)。养殖区和非养殖区样品分别集中在RDA1轴的左侧和右侧,说明养殖区细菌群落变异与COD、PO43-和NH4+呈正相关,非养殖区细菌群落变异与TOC呈正相关(图 3)。
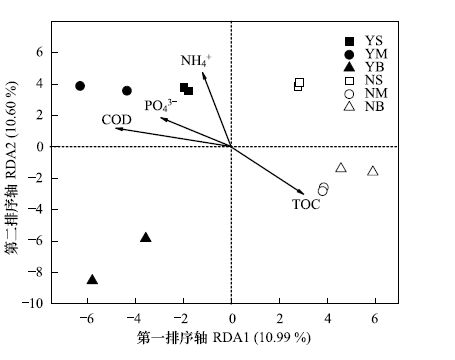
|
| 图3 网箱养殖区与非养殖区不同水深层次水样浮游细菌群落与其变异驱动的主要环境因子RDA双向排序图 Fig.3 RDA ordination biplot showed factors that driving the variation of bacterial communities between cage culture area and non-cuntured area COD: Chemical oxygen demand; TOC: Total organic carbon; RDA: Redundancy analysis |
同时,加入空间距离单独变量(Mantel test,P = 0.030)[28],结合与群落变异显著相关的环境因子(COD、TOC、PO43-和NH4+),利用pRDA分析,发现环境因子共解释了38.18%,空间距离单独解释了10.66%的群落变异,两者的相互作用仅解释了0.04%,表明空间距离与环境因子间间的自相关性可以忽略,养殖活动是导致群落变异的主要因素。
3 讨论与结论关于养殖活动对水体浮游细菌的影响,在群落水平上的研究还较少[3],其研究手段主要是依赖培养技术和基于16S rRNA基因的DGGE等分子指纹技术和克隆文库测序方法[3, 29]。这些方法费时费力,分析通量非常有限,而细菌多样性又异常丰富,因此揭示的信息非常有限,忽略了一些具有重大功能的低丰度物种的存在[30, 31]。新近出现的焦磷酸测序技术则将细菌的鉴定分析通量提高百万倍,很好得解决了通量瓶颈,对细菌群落结构信息揭示的更加全面、详细[32, 33]。本研究利用16S rRNA 基因焦磷酸测序技术分析了12个海水样品,产生了110950条序列,共检测出5752个OTUs(相当于种属水平),每个样品的序列数在5774—12877之间,涵盖的OTU数平均达680—1578,为深入探究浮游细菌群落结构与环境因子的关联性提供了数据基础。
3.1 浮游细菌多样性与群落组成维持浮游细菌在养殖水体中的多样性可以增强养殖环境生物群落生态系统的稳定性,减少养殖生物患病的风险。已有研究表明,高密度网箱增养殖活动使养殖区域及附近水体中的营养盐和有机污染物浓度较高,对细菌具有一定的选择作用,导致养殖区的细菌多样性较低[3]。本实验则显示,网箱区和非养殖区的多样性无显著差异。这种差异可能是因为网箱养殖区受污染程度较低,对浮游细菌多样性影响较小,也可能是以前研究不能检测到稀有物种造成的。
从浮游细菌种类组成来看,α-变形菌、γ-变形菌、拟杆菌为海水中主要的优势菌群,Kirchman也发现变形菌和拟杆菌是组成海洋异养细菌的两个最大的类群[34]。在以前的象山港生态研究中,利用克隆测序方法发现优势菌组成也与上述相似[35]。实验结果显示,从网箱区到非养殖区,拟杆菌门和放线菌门的相对丰度显著降低,而γ-变形菌纲的相对丰度增加,可能是网箱区域磷酸盐、总磷浓度较高所致。有研究表明α-变性菌的相对丰度随水体中总磷浓度增加显著增加,拟杆菌门的相对丰度随磷酸盐的浓度升高而增加[36]。对东海沉积物中的微生物群落结构研究也表明,随着N、P污染梯度的变化,细菌群落中γ-变形菌和δ-变形菌相对丰度发生显著变化,与污染物浓度显著相关,能够指示环境受污染程度[28]。因此,拟杆菌门、放线菌门和γ-变形菌纲的丰度对水质变化敏感,可能用于指示和评价海水养殖污染的生态效应。
3.2 浮游细菌群落空间分布已有研究表明,养殖活动能够改变浮游细菌群落结构[19],本文结果也表明网箱区与非养殖区的群落差异显著。Salvator等[37]对网箱养殖活动的调查也发现:网箱养殖活动对网箱下微生物的影响效果显著,但离网箱外50 m区域受养殖活动影响较小。本研究中尽管养殖区站位和非养殖区站位的距离较远,空间距离对群落变异的影响(10.66%贡献度)小于环境因子(38.18%贡献度)。造成群落变异的主要原因可能是养殖区和非养殖区之间水质差异较大,如化学需氧量有显著差异。有研究表明,水质情况相似的采样点,其浮游细菌群落的组成也相似[38]。本研究还发现网箱区和非养殖区的细菌群落由表层向底层分布的规律一致,但水深对群落多样性和群落结构相似性均无显著影响。李玉华等[38]通过对松花湖水体中不同空间分布的细菌群落结构研究表明,水深变化对细菌群落垂直分布有重要影响。以上结果不一致性可能是由于不同深度范围造成的,如本实验采样点最大水深只有8 m,远小于松花湖平均水深35 m。
刘晶晶等[3]对象山港网箱养殖区水体中细菌分布研究表明,与养殖场鱼类疾病相关的弧菌在网箱区中的数量均大于网箱外对照区,而本文结果中,交替假单胞菌属(Pseudoalteromonas)和弧菌科(Vibrionaceae)在非养殖区中的平均相对丰度较高。但同时发现褐杆菌属(Phaeobacter)平均相对丰度在养殖区显著高于网箱区,而褐杆菌属对以弧菌、交替假单胞菌等为代表的主要致病菌具有广泛的抗菌活性谱并且能控制致病菌的繁殖[39, 40]。因此,推测可能是褐杆菌属的高丰度存在抑制了弧菌和交替假单胞菌的繁殖,具体原因和机制有待进一步研究。
3.3 影响浮游细菌群落的主要环境因子冗余分析表明,化学需氧量、磷酸盐、铵盐和总有机碳是影响浮游细菌群落的主要环境因子。除总有机碳外,这与李秋芬等[35]应用DGGE技术对象山港中部养殖区细菌群落结构特征的研究所得到的结果一致。铵盐和磷酸盐是调控浮游细菌群落分布的重要因子,与浮游细菌群落分布密切相关[20]。高密度的水产养殖常造成水体环境中氮、磷营养盐浓度的增加[1],从而影响浮游细菌群落组成,包括条件致病菌的增殖和致病性的增加,同时增加水产动物对病原菌的敏感性,进而导致养殖病害的发生。化学需氧量是衡量海水中有机质含量的重要指标,水体中的溶解性有机物是异养细菌生长繁殖和生命活动最基本的营养源[20]。在养殖区中因剩余残饵和养殖生物的排泄物与分泌物均含有丰富的有机质,造成养殖区中的化学需氧量显著高于非养殖区,从而影响浮游细菌群落组成的分布。在水生生态系统中,溶解性总有机碳是异养细菌重要的碳源,浮游细菌可以吸收、利用水体中的溶解性有机碳,进行二次生产,其分布与总有机碳显著相关[13]。有研究表明,总有机碳与有机污染物浓度存在正相关关系[41],这与本实验结果不一致,可能是由于网箱区浮游植物的生长消耗了大量的总有机碳,而养殖动物对浮游植物的捕食,表现为养殖区的叶绿素a和总有机碳浓度较低。
综上所述,近海网箱养殖活动影响水质,进而改变细菌群落结构,且从表层至底层具有相似的演替规律。其中在网箱区增加的化学需氧量、活性磷酸盐和铵盐,总有机碳是驱动细菌群落变异的主要环境因子。造成群落差异的敏感类群如拟杆菌、放线菌和γ-变形菌可能用于指示和评价海水养殖污染程度。
| [1] | Degefu F, Mengistu S, Schagerl M. Influence of fish cage farming on water quality and plankton in fish ponds: A case study in the Rift Valley and North Shoa reservoirs, Ethiopia. Aquaculture, 2011, 316(1/4): 129-135. |
| [2] | 王冉, 刘铁铮, 王恬. 抗生素在环境中的转归及其生态毒性. 生态学报, 2006, 26(1): 265-270. |
| [3] | 刘晶晶, 曾江宁, 陈全震, 江志兵, 赵永强, 廖一波, 寿鹿, 徐晓群, 黄逸君, 杜萍. 象山港网箱养殖区水体和沉积物的细菌生态分布. 生态学报, 2010, 30(2): 377-388. |
| [4] | 金珊, 王国良, 赵青松, 郑天伦, 陈寅儿. 海水网箱养殖大黄鱼弧菌病的流行病学研究. 水产科学, 2005, 24(1): 17-19. |
| [5] | Madoni P, Braghiroli S. Changes in the ciliate assemblage along a fluvial system related to physical, chemical and geomorphological characteristics. European Journal of Protistology, 2007, 43(2): 67-75. |
| [6] | Polat S, Koray T. Planktonic dinoflagellates of the northern Levantine Basin, northeastern Mediterranean Sea. European Journal of Protistology, 2007, 43(3): 193-204. |
| [7] | Hjelm M, Riaza A, Formoso F, Melchiorsen J, Gram L. Seasonal incidence of autochthonous antagonistic Roseobacter spp. and Vibrionaceae strains in a turbot larva (Scophthalmus maximus) rearing system. Applied and Environmental Microbiology, 2004, 70(12): 7288-7294. |
| [8] | Korsnes K, Nicolaisen O, Skår C K, Nerland A H, Bergh Ø. Bacteria in the gut of juvenile cod Gadus morhua fed live feed enriched with four different commercial diets. ICES Journal of Marine Science, 2006, 63(2): 296-301. |
| [9] | Zhang D M, Wang X, Xiong J B, Zhu J L, Wang Y N, Zhao Q F, Chen H P, Guo A N, Wu J F, Dai H P. Bacterioplankton assemblages as biological indicators of shrimp health status. Ecological Indicators, 2014, 38: 218-224. |
| [10] | Xiong J B, Zhu J L, Wang K, Wang X, Ye X S, Liu L, Zhao Q F, Hou M H, Qiu Q L L, Zhang D M. The temporal scaling of bacterioplankton composition: High turnover and predictability during shrimp cultivation. Microbial Ecology, 2014: 67(2): 256-264. |
| [11] | Humayoun S B, Bano N, Hollibaugh J T. Depth distribution of microbial diversity in Mono Lake, a meromictic soda lake in California. Applied and Environmental Microbiology, 2003, 69(2): 1030-1042. |
| [12] | Yan Q Y, Yu Y H, Feng W S, Yu Z G, Chen H T. Plankton community composition in the Three Gorges Reservoir Region revealed by PCR-DGGE and its relationships with environmental factors. 环境科学学报(英文版), 2008, 20(6): 732-738. |
| [13] | 周凯, 章洁香, 张瑜斌, 卢东伟, 丁玉静, 孙省利. 深圳湾浮游细菌生物量的时空分布及其影响因素. 热带海洋学报, 2013, 32(3): 65-71. |
| [14] | 冯胜, 秦伯强, 高光. 细菌群落结构对水体富营养化的响应. 环境科学学报, 2007, 27(11): 1823-1829. |
| [15] | 吕明姬, 汪杰, 范铮, 黄艺. 滇池浮游细菌群落组成的空间分布特征及其与环境因子的关系. 环境科学学报, 2011, 31(2): 299-306. |
| [16] | González N, Anadón R, Viesca L. Carbon flux through the microbial community in a temperate sea during summer: role of bacterial metabolism. Aquatic Microbial Ecology, 2003, 33(2): 117-126. |
| [17] | Grossart H P, Simon M. Bacterioplankton dynamics in the Gulf of Aqaba and the northern Red Sea in early spring. Marine Ecology Progress Series, 2002, 239: 263-276. |
| [18] | 高爱根, 杨俊毅, 陈全震, 王自磐, 张健, 董永庭, 宁修仁. 象山港养殖区与非养殖区大型底栖生物生态比较研究. 水产学报, 2003, 27(1): 25-31. |
| [19] | 裘琼芬, 张德民, 叶仙森, 郑珍珍. 象山港网箱养殖对近海沉积物细菌群落的影响. 生态学报, 2013, 33(2): 483-491. |
| [20] | 杨季芳, 王海丽, 陈福生, 屠霄霞, 陈吉刚, 冯辉强. 象山港海域细菌的分布特征及其环境影响因素. 生态学报, 2011, 31(14): 4007-4018. |
| [21] | 温丹, 张德民, 初航. 网箱养殖海区底泥产芽孢细菌多样性. 海洋与湖沼, 2009, 40(5): 615-621. |
| [22] | Edgar R C. Search and clustering orders of magnitude faster than BLAST. Bioinformatics, 2010, 26(19): 2460-2461. |
| [23] | De Santis T Z, Hugenholtz P, Keller K, Brodie E T, Larsen N, Piceno Y M, Phan R, Andersen G L. NAST: a multiple sequence alignment server for comparative analysis of 16S rRNA genes. Nucleic Acids Research, 2006, 34(Web Server issue): W394-W399. |
| [24] | De Santis T Z, Hugenholtz P, Larsen N, Rojas M, Brodie E L, Keller K, Huber T, Dalevi D, Hu P, Andersen G L. Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB. Applied and Environmental Microbiology, 2006, 72(7): 5069-5072. |
| [25] | 刘乐冕, 杨军, 余小青, 余正, 张永雨, 田野, 张冬红. 厦门后溪流域沿城乡梯度浮游细菌多样性及其与环境因子的关系. 应用与环境生物学报, 2012, 18(4): 591-598. |
| [26] | Kruskal J B. Multidimensional scaling by optimizing goodness of fit to a nonmetric hypothesis. Psychometrika, 1964, 29(1): 1-27. |
| [27] | Braak C J F, Šmilauer P. CANOCO reference manual and CanoDraw for Windows user's guide: software for canonical community ordination (version 4. 5). Section on Permutation Methods. Microcomputer Power, Ithaca, New York, 2002. |
| [28] | Xiong J B, Ye X S, Wang K, Chen H P, Hu C J, Zhu J L, Zhang D M. Biogeography of the sediment bacterial community responds to a nitrogen pollution gradient in the East China Sea. Applied and Environmental Microbiology, 2014, 80(6): 1919-1925. |
| [29] | 杨彩霞, 王崇明, 李赟, 梁颜韬, 王娜, 蔡玉勇. 应用DGGE技术分析流清河湾扇贝养殖海区细菌群落结构的季节变化. 水产学报, 2012, 36(3): 407-413. |
| [30] | Parkes R J, Cragg B A, Wellsbury P. Recent studies on bacterial populations and processes in subseafloor sediments: A review. Hydrogeology Journal, 2000, 8(1): 11-28. |
| [31] | Nogales B, Lanfranconi M P, Piña Villalonga J M, Bosch R. Anthropogenic perturbations in marine microbial communities. FEMS Microbiology Reviews, 2011, 35(2): 275-298. |
| [32] | Muyzer G, De Waal E C, Uitterlinden A G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA. Applied and Environmental Microbiology, 1993, 59(3): 695-700. |
| [33] | Kirchman D L, Cottrell M T, Lovejoy C. The structure of bacterial communities in the western Arctic Ocean as revealed by pyrosequencing of 16S rRNA genes. Environmental Microbiology, 2010, 12(5): 1132-1143. |
| [34] | Kirchman D L. The ecology of Cytophaga-Flavobacteria in aquatic environments. FEMS Microbiology Ecology, 2002, 39(2): 91-100. |
| [35] | 李秋芬, 有小娟, 张艳, 毛玉泽, 焦海峰. 象山港中部养殖区细菌群落结构的特征及其在生境修复过程中的变化. 中国水产科学, 2013, 20(6): 1234-1246. |
| [36] | 沈燕, 高光, 汤祥明, 晁建颖. 用FISH技术检测太湖梅梁湾水体中细菌优势种群在冬夏两季的变化. 湖泊科学, 2010, 22(1): 57-62. |
| [37] | Porrello S, Tomassetti P, Manzueto L, Finoia M G, Persia E, Mercatali I, Stipa P. The influence of marine cages on the sediment chemistry in the Western Mediterranean Sea. Aquaculture, 2005, 249(1): 145-158. |
| [38] | 李玉华, 许其功, 赵越, 李强, 魏自民, 赵昕宇. 松花湖水体中不同空间分布的细菌群落结构分析. 农业环境科学学报, 2013, 32(4): 764-770. |
| [39] | Porsby C H, Nielsen K F, Gram L. Phaeobacter and Ruegeria species of the Roseobacter clade colonize separate niches in a Danish turbot (Scophthalmus maximus)-rearing farm and antagonize Vibrio anguillarum under different growth conditions. Applied and Environmental Microbiology, 2008, 74(23): 7356-7364. |
| [40] | Prado S, Montes J, Romalde J L, Barja J L. Inhibitory activity of Phaeobacter strains against aquaculture pathogenic bacteria. International Microbiology, 2010, 12(2): 107-114. |
| [41] | Accardi Dey A M, Gschwend P M. Assessing the combined roles of natural organic matter and black carbon as sorbents in sediments. Environmental Science and Technology, 2002, 36(1): 21-29. |
 2015, Vol. 35
2015, Vol. 35